Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse de l'expression de mammifères Linker-histones sous-types
Dans cet article
Résumé
Nous décrivons un ensemble de tests pour analyser les niveaux d'expression des histones H1. ARNm de gènes H1 individuels sont mesurés quantitativement par transcription inverse amorce aléatoire basé suivie par PCR en temps réel, alors que la quantification des protéines histones H1 de est obtenue par une analyse HPLC.
Résumé
Linker histone H1 se lie à la particule de noyau nucléosome et l'ADN linker, pour faciliter le pliage de la chromatine dans la structure d'ordre supérieur. H1 est essentielle pour le développement des mammifères 1 et régule l'expression génique spécifique in vivo 2-4. Parmi les protéines hautement conservées des histones, la famille des histones H1 est le groupe le plus hétérogène. Il ya 11 sous-types H1 chez les mammifères qui sont différentiellement régulés au cours du développement et dans différents types cellulaires. Ces sous-types H1 comprennent 5 somatiques H1 (H1a-e), le H1 0 remplacement, 4 germinales sous-types de cellules H1 spécifiques, et H1x 5. La présence de sous-types H1 multiples qui diffèrent de l'affinité de liaison à l'ADN et la capacité compaction de la chromatine 6-9 fournit un niveau supplémentaire de modulation de la fonction chromatine. Ainsi, l'analyse d'expression quantitative des sous-types H1 individuels, à la fois de l'ARNm et de protéines, est nécessaire pour une meilleure compréhension de la régulation de l'enseignement supérieurstructure de la chromatine ordre et la fonction.
Nous décrivons ici un ensemble de tests conçus pour analyser les niveaux d'expression des sous-types H1 individuels (Figure 1). expression des ARNm de différents gènes variants H1 est mesurée par un ensemble de très sensibles et quantitative RT-PCR (qRT-PCR), qui sont plus rapides, plus précis et nécessitent des échantillons beaucoup moins par rapport à l'approche alternative de l'analyse par Northern blot. Contrairement à la plupart des autres messages d'ARNm cellulaires, les ARNm des gènes les plus histones, y compris la majorité des gènes H1, n'ont pas une longue queue polyA, mais qui contiennent une structure en tige-boucle à la région 3 'non traduite (UTR) 10. Par conséquent, les ADNc sont préparés à partir d'ARN totaux par transcription inverse en utilisant des amorces aléatoires au lieu d'oligo-dT amorces. En temps réel PCR avec des amorces spécifiques à chaque sous-types H1 (Tableau 1) sont effectuées pour obtenir une mesure hautement quantitative des taux d'ARNm de sous-type H1 individuelles. Expression des gènes de ménage sont analysés comme des contrôles pour la normalisation.
L'abondance relative des protéines de chaque sous-type H1 et histones est obtenue par chromatographie en phase liquide inverse à haute performance (CLHP-PI) l'analyse des histones totaux extraits de cellules de mammifères 11-13. La méthode par CLHP et les conditions d'élution décrites ici donnent des séparations optimales de sous-types H1 souris. En quantifiant le profil HPLC, nous calculons la proportion relative des sous-types H1 individuels au sein de la famille H1, ainsi que de déterminer le ratio de H1 à nucléosome dans les cellules.
Protocole
1. Préparation de l'échantillon et d'extraction d'ARN
- Avant l'extraction d'ARN, toutes les surfaces de travail et les pipettes doivent être nettoyées avec de l'éthanol 70% et traité avec une solution de décontamination RNase, tels que la RNase Zap. Cette pratique réduit les risques de contamination de RNase et la dégradation des ARN. Porter des gants pour toutes les procédures.
- Pour extraire l'ARN à partir de tissus de la souris, disséquer l'organe d'intérêt de la souris euthanasiés, et lavez le tissu en phosphate glacée solution saline tamponnée (PBS: 0,13 M de NaCl, 5 mM de phosphate de sodium dibasique heptahydraté, 5 mM de sodium dihydrogénophosphate heptahydraté, pH 7,4) . Procéder immédiatement à l'extraction de l'ARN à l'étape 1.4. Si le tissu frais est de ne pas être traitées pour extraction de l'ARN, les échantillons de tissus devraient être gelés composant logiciel enfichable dans l'azote liquide immédiatement et être conservés à -80 ° C pour une utilisation ultérieure.
- Si l'ARN doit être extrait de culture de cellules adhérentes, aspirer les milieux de culture, rincer avec une quantité suffisante de PBS, et ajouter de Réactif Trizol (Invitrogen) sur la plaque et passez à l'étape 1.4. Pour les cellules cultivées en suspension, de récolter les cellules et les cellules par centrifugation. Jeter les médias, rincer les granules rapidement avec du PBS, et le culot des cellules avec centrifugation. Ajouter le réactif Trizol et passez à l'étape 1.4.
- Réactif Trizol suffisante est nécessaire pour l'obtention d'ARN de haute qualité. Utiliser 1 ml de réactif Trizol pour extraire l'ARN de 50 à 100 mg de tissu, 5 - 10 x 10 6 cellules (pour les cultures en suspension) ou par 3,5 cm (plaque pour les cultures adhérentes). Homogénéiser le tissu dans le réactif Trizol avec Polytron PT2100 homogénéisateur (ou équivalent). Passez à extraire l'ARN à partir d'échantillons de tissus ou de cellules selon le manuel du fabricant pour le réactif Trizol (Invitrogen).
- Concentration en ARN est mesurée en utilisant Nanodrop 1000 (Thermo) et de la qualité d'ARN est analysé par électrophorèse sur gel. Les fourchettes de rendement typiques de pg 1-10 de l'ARN par mg de tissu ou de 5-15 ug d'ARN pour 1 x 10 6 cellules en culture. Pour éliminer lela contamination potentielle de l'ARN à partir des traces d'ADN génomique, les échantillons d'ARN sont traités avec de la RNase-Free DNase (Sigma AMP-D1) selon les instructions du fabricant. Répétez détermination de la concentration de l'ARN et l'électrophorèse sur gel d'assurer aucune dégradation de l'ARN à partir de ce traitement. Magasin extrait l'ARN à -80 ° C.
* Remarque: l'ARN peut aussi être extrait à l'aide RNAeasy kit (Qiagen) selon le manuel du kit, ou par de l'ADN / ARN (Qiagen) si les deux ADN et l'ARN sont souhaitées.
2. Reverse Transcription PCR quantitative (qRT-PCR)
- L'ARN total subit une transcription inverse en ADNc Superscript using System III Synthèse du premier brin (Invitrogen). Depuis l'ARNm de la plupart des gènes H1 manquent poly-A-queues, il est essentiel d'utiliser des hexamères aléatoires au lieu d'oligo-dT comme amorces pour la synthèse d'ADNc. Cependant, si l'analyse d'expression de gènes avec des messages polyadénylé à de faibles niveaux est également souhaitée, un mélange de hexamères au hasard et d'oligo-dT should être utilisé dans la transcription (RT) la réaction inverse pour améliorer l'efficacité de transcription inverse d'ARNm polyadénylé.
- Effectuer la réaction de RT selon le manuel du fabricant. En bref,
- Dans un tube de 0,5 ml PCR, mélanger 5 ug d'ARN total, 1 pl de 50 ng / ul hexamères aléatoires, 1 pl de 10 mM de dNTP mix, et d'ajouter traitée au DEPC H 2 O pour rendre le volume total de réaction de 10 ul. Bien mélanger et incuber pendant 5 minutes à 65 ° C, suivi par 1 minutes d'incubation sur la glace.
- Préparer 10 ul du mélange de synthèse d'ADNc: 2 pl de tampon 10xRT, 4 pl de 25 mM MgCl 2, 2 pi de 0,1 M DTT, 1 pl de RNaseOut (40 U / pl), 1 pl de SuperScript III RT (200 U / pl) et l'ajouter à l'ARN mélange / amorce.
- Laisser incuber pendant 10 minutes à 25 ° C, suivie par 50 minutes à 50 ° C et terminer la réaction à 85 ° C pendant 5 minutes.
- Chaque réaction donne généralement de 100 à 250 ng / l de produit d'ADNc. Rangez les produits d'ADNc à -20° C ou procéder immédiatement en temps réel PCR quantitative (qPCR).
- qPCR peut quantifier avec précision les copies de séquences cibles avec une grande efficacité et la reproductibilité 14. On choisit mesurée par PCR quantitative SYBR Green colorant, ce qui donne un signal fluorescent seulement lorsqu'il intercale avec l'ADN double brin (ADNdb). Bien que n'étant pas aussi précis que Taqman 14, cette méthode est plus rentable, plus facile à adopter dans le laboratoire, et donne plus de polyvalence pour qPCR. Par conséquent, il est important d'examiner la parcelle d'amplification (figure 2A) et les courbes de fusion dérivés du produit qPCR (figure 2B) pour assurer l'efficacité et la spécificité de réaction.
| Sous-types des histones | Nomenclature souris histone | Human nomenclature des histones | ||
| Genom ne | Aucune adhésion. | Nom de gène | Aucune adhésion. | |
| Histone H1a | Hist1h1a | NM_030609 | HIST1H1A (H1.1) | NM_005325 |
| H1b Histone | Hist1h1b | NM_020034 | HIST1H1B (H1.5) | NM_005322 |
| H1c Histone | Hist1h1c | NM_015786 | HIST1H1C (H1.2) | NM_005319 |
| H1D Histone | Hist1h1d | NM_145713 | HIST1H1D (H1.3) | NM_005320 |
| H1E Histone | Hist1h1e | NM_015787 | HIST1H1E (H1.4) | NM_005321 |
| Histone H1 ° | H1f0 | NM_008197 | H1F0 | NM_005318 |
| Histone H1oo | H1foo | NM_183811 | H1FOO | NM_153833 |
| H1T Histone | Hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| H1t2 Histone | H1fnt | NM_027304 | H1FNT | NM_181788 |
| H1x Histone | H1fx | NM_198622 | H1FX | NM_006026 |
| Hils1 histones | Hils1 | NM_081792 | HILS1 | AY286318 |
Tableau 1. Histone nomenclature sous-type H1 chez la souris et l'homme.
- Conception avant et arrière amorces PCR spécifiques pour chaque gène H1 (Tableau 1). En raison de la similarité de séquence élevée chez les somatique H1, en particulier dans la région correspondant à la domaine globulaire central, il est essentiel de veiller à ce que les amorces conçues pour un sous-type H1 spécifique ne sont pas alignés avec outres gènes H1, ou croisées amplifier sous-types H1 autres. Il est également important de noter que la plupart des gènes H1 ne contiennent pas les introns. Ainsi intron couvrant amorces généralement adoptées pour la RT-PCR pour éviter la contamination génomique ne sont pas disponibles. Au lieu de cela, des échantillons d'ARN doivent être pré-traités à la DNase (voir 1.5) pour éliminer toute trace de contamination génomique. En outre, RT (-)-PCR quantitative doit être effectuée en parallèle pour valider l'absence d'une contamination génomique dans les échantillons d'ADNc.
- Amorces de conception aussi des gènes de référence internes, dont l'expression ne sont pas modifiés dans les échantillons. Souvent gènes domestiques tels que glycéraldéhyde-3-phosphate déshydrogénase (GAPDH) et bêta-actine gènes, sont choisis en tant que gènes de référence. signaux qPCR de gènes de ménage servent de témoins de normalisation.
- Préparer chaque réaction PCR (volume total 25 pi) de la manière suivante: 12,5 ul de 2x IQ SYBR Supermix vert (Bio-Rad) (dNTP contenant, 50 U / ml ADN polymérase ITAQ, 6 mM MgCl 2, SYBR Green I et 20nM fluorescéine), 2 pl de 4 ng / ul d'ADNc, 1,25 pl de 10 nM avant / mix amorce inverse, et de 9,25 l de ddH 2 O, et bien mélanger en Microseal plaque de 96 puits PCR. Utilisez Films adhésifs «B» Microseal (Bio-Rad) pour s'assurer que le couvercle de la plaque est scellée à la plaque. Appuyez sur ou homogénéiser brièvement au vortex de la plaque PCR, et centrifuger les mélanges de réaction par une centrifugation à court. Placer la plaque dans MyIQ couleur en temps réel d'un système unique de détection par PCR (Bio-Rad) pour qPCR.
- Nous utilisons les conditions suivantes: qPCR 95 ° C pendant 3 minutes, suivi par 40 cycles de 95 ° C pendant 10 secondes, 60 ° C pendant 20 secondes, 72 ° C pendant 30 secondes. Examiner les courbes d'amplification (figure 2A) pour la PCR efficacité et Ct (seuil de cycle) des valeurs. La ligne de seuil peut être réglée automatiquement par les iQ5 2.0 Système optique version de logiciel.
- L'efficacité primaire et optimale de concentration d'ADNc nécessaire peut être testée par un essai de courbe standard, dans lequel une série de dilutions d'ADN génomique is utilisé pour qPCR et les valeurs de Ct sont tracées en fonction du log de la quantité d'ADN matrice. Un test PCR en temps réel optimisé avec des amorces de haute spécificité et l'efficacité donnera une courbe linéaire standard, avec le coefficient de détermination (R 2)> 0,98. Évitez amorces avec longueur de l'amplicon plus de 200 pb, qui ont tendance à avoir une mauvaise efficacité de l'amplification.
- Parce SYBR Green détecte toute ADNdb, il est essentiel pour effectuer une course courbe de fusion suivant la qPCR faire en sorte que l'amplicon désiré, mais non dimères d'amorces ou de contaminants, sont amplifiées et détectées. Pour l'état fondu courbe d'analyse, l'instrument PCR en temps réel du programme pour chauffer les échantillons à partir de 55 ° C à 95 ° C dans 0,5 ° C par incréments avec la collecte des données. Le réglage par défaut de fusion courbe d'analyse pour MyIQ (Bio-Rad) l'instrument est la suivante: 95 ° C pendant 1 minute, 55 ° C pendant 1 minute, suivi par 81 cycles de 10 secondes au point de consigne 55 ° C, faire fondre la courbe, + 0,5 ° C Température (caméra recueille des données à chaque cycle).
- Depuis la température de fusion (Tm) de ADNdb dépend de la longueur et la teneur en GC amplicon, amplicons différents auront Tm (s). Examiner les courbes de fusion dérivés des produits de qPCR pour confirmer la température de fusion spécifique d'amplicons souhaités ainsi que l'absence de pics de bruit amplicon (figure 2B).
- Préparer double ou triple réactions pour chaque dosage pour l'analyse statistique. Comprennent des contrôles négatifs pour qRT PCR, comme RT (-)-PCR quantitative (qPCR avec l'ARN comme matrice sans transcription inverse) et qPCR sans ADNc, l'ARN ou l'ADN de source ajouté au mélange de PCR. RT (-)-qPCR peut servir de contrôle de la contamination d'ADN génomique potentiel (RT (-) dans la figure 2 & 3).
- Analyser les données qPCR avec iQ5 optique System Software version 2.0 (Bio-Rad). Normaliser les valeurs d'expression de gènes isoforme H1 à l'expression de gène de ménage (par exemple GAPDH, ß-actine, HPRT) pour obtenir des niveaux d'expression relatifs des gènes H1.
* Toutes les procédures doivent être effectuées sur la glace ou à 4 ° C.
- Disséquer les tissus de la souris et le rincer avec de la glace du PBS froid. (Si ce n'est pas procéder à l'extraction immédiatement, enclenchez congeler et conserver les échantillons comme décrit dans l'étape 1.2.) Hacher le tissu en morceaux avec une lame de rasoir. Transfert hachis d'un homogénéisateur Dounce (B pilon). Ajouter 10 ml de tampon de saccharose (0,3 M de saccharose, 15 mM NaCl, 10 mM d'HEPES [pH 7,9], 2 mM d'EDTA, 0,5 mM PMSF, complète la protéase mini cocktail inhibiteur de Tablet, ajouter frais) par gramme de tissu. Homogénéiser le tissu avec 10-15 coups.
- Transfert des homogénats à un tube de 15 ml, de spin à 500 rpm pendant 30 secondes (modèle centrifugeuse: Eppendorf 5810R); transférer soigneusement le surnageant dans un nouveau tube (jeter les débris à granulés de tissus), et centrifuger à 2000 rpm pendant 5 minutes, à sédimenter les cellules. Passez à l'étape 3.4.
- Si histones et la chromatine doivent être extraits à partir de cellules cultivées en monocouche, rinceravec du PBS, ajouter du PBS pour la boîte de culture, et de récolter les cellules en utilisant grattoir à cellules et les cellules par centrifugation. Pour les cellules cultivées en suspension, granulés cellules par centrifugation.
- Remettre en suspension le culot cellulaire dans 10 ml de tampon de saccharose supplémenté avec 0,5% de NP-40 (par gramme de tissu ou de montant de départ 10 (8) cellules). Transférer l'échantillon à un homogénéisateur Dounce (B pilon) et Dounce 10 coups à moins de 20 minutes d'incubation. À ce stade, les noyaux sont obtenus. Examiner la qualité des noyaux sous un microscope. Pellet les noyaux par centrifugation à 2000 rpm pendant 5 minutes. Jeter le surnageant.
- Reprendre le culot des noyaux dans 3 ml de tampon en sel élevée (0,35 M de KCl, 10 mM Tris [pH 7,2], 5 mM de MgCl2, 0,5 mM PMSF - ajouter frais avant chaque utilisation) pour 1 g de tissu ou de 10 (8) cellules. Transférer l'échantillon à un petit homogénéisateur Dounce (B pilon) et homogénéiser avec des coups de 5-10.
- Aliquoter la suspension dans des tubes Eppendorf 3 (1 ml chacun), incuber sur de la glace pendant 20 minutes, suivie parcentrifugation à 14000 rpm pendant 10 minutes à granulés chromatine. Jeter le surnageant.
- Ajouter 0,8 ml 0,2 H 2 SO 4 à chaque pastille chromatine. Utilisez un tube Eppendorf pilon Dounce à moudre le culot bien jusqu'à ce que la pastille est complètement dissocié. Incuber les échantillons sur une plateforme rotative à 4 ° C pendant la nuit. Histones totaux sont extraits à cette étape de traitement à l'acide.
- Centrifuger à 14000 rpm pendant 10 minutes. Transférer le surnageant (extraits des histones) dans des tubes Eppendorf deux (400 pl / tube). Jeter le culot. Ajouter 2,5 volumes (1 ml) d'éthanol glacé à chaque tube .. Conserver les échantillons à -20 ° C pendant la nuit.
- Centrifuger à 14000 rpm pendant 10 minutes à granulés histones totales, éliminer le surnageant. Laver les trois granules fois avec EtOH à 70%, laissez sur le banc pendant 20-30 minutes à l'air sec. Conserver les protéines séchées à -80 ° C ou les dissoudre dans le trou DDH 2 O et de procéder à une analyse par HPLC immédiatement. Protéines séchées peuvent être stockéesà -80 ° C pour un maximum de 1 an.
4. Analyse HPLC des histones
- Remettre en suspension le culot histone dans le volume recommandé de ddH 2 O en fonction de la capacité de la colonne en phase inverse et l'instrument de CLHP. Nous utilisons colonne en phase inverse C18 250 x 4,6 mm (Vydac) et Äktapurifier UPC 900 instrument (GE Healthcare) pour l'analyse HPLC. En général, nous dissoudre 50-100 ug des histones au total dans 100 ul de ddH 2 0 pour l'analyse.
- Centrifuger à 14000 rpm pendant 5 minutes pour éliminer les résidus insolubles. Dosage de protéine Bradford est utilisée pour déterminer la quantité de protéine d'être injecté dans la colonne. Injecter de 50 à 100 pg de protéine totale dans la colonne en phase inverse sur le système HPLC. Chargement d'une quantité excessive de protéines devrait être évitée afin de prévenir le colmatage de la colonne.
- Fractionner les histones et les histones de base avec un gradient d'acétonitrile augmente comme indiqué dans le tableau 2.
Tableau 2. Gradient croissant d'acétonitrile au fil du temps.
- L'effluent est contrôlé à 214 nm, et les profils HPLC (Figure 4) sont enregistrées et analysées à l'aide Äktapurifier UPC 900 (GE Healthcare) avec UNICORN 5,11 logiciels (GE Healthcare). Les fractions protéiques peuvent également être collectées avec la fraction d'auto-collecteur (Frac-920 - GE) pour une analyse plus poussée, par exemple la spectrométrie de SDS-PAGE et de masse.
- Les A 214 valeurs des pics de chaque sous-type H1 et H2B sont normalisées par le nombre de liaisons peptidiques de chaque protéine d'histone correspondante. La proportion relative des sous-types H1 individuels histone H1 sein de la famille, ainsi que le rapport de sous-types H1 à particules de noyau nucléosomes peut être calculée à partir de ces valeurs normalisée A 214 (figure 5).
5. Les résultats représentatifs
La liste des sous-types H1 mammifères, l'organigramme de l'ensemble et les résultats représentatifs de l'analyse d'expression de gènes particuliers de l'histone H1 sont indiqués dans le tableau 1, figure 1 et la Figure 2-5, respectivement.Figure 2A montre les courbes d'amplification typiques de réactions qPCR H1A utilisant l'ADNc préparé à partir de foie de souris et mESCs, tandis que la figure 2B montre les courbes de fusion dérivés des amplicons correspondants. La courbe de fusion présente un pic unique caractéristique à la température de fusion (Tm) à 86 ° C pendant l'amplicon H1a PCR, et n'a pas de fond pics non spécifiques, suggérant une spécificité élevée de H1a qPCR dosage. Évaluation de la parcelle d'amplification (figure 2A) montre que les réactions qPCR triple de chaque échantillon a donné des signaux compatibles avec les valeurs de Ct presque identiques, ce qui suggère une grande reproductibilité. Le manque d'amplicons accumulation de RT (-)-qPCR réactions indique que la contamination d'ADN génomique n'était pas présent, ou minimes. En utilisant les valeurs de Ct de gènes H1 et gènes de ménage, tels que la GAPDH, les niveaux relatifs d'expression d'ARN de chaque gène H1 ont été calculés. Des exemples de résultats calculés pour H1 ° et les gènes H1A sont présentés dans Figure 3. Les niveaux d'expression relatifs des ARNm H1a sont plus élevés dans mESCs rapport avec le foie de souris, alors que H1 ° l'expression est beaucoup plus élevé dans le foie que dans mESCs.
La différence dans l'expression de H1a ou H1 ° dans le foie de souris par rapport MESC adulte est également évident à partir des profils HPLC des protéines histones (Figure 4). H1 °, le H1 de différenciation spécifique, est accumulée à une grande quantité dans les tissus matures, ce qui représente 27,2% du total dans H1 foie adulte (figure 5A). En revanche, H1 ° protéine est pratiquement absent dans mESCs indifférenciées (figure 4B). D'autre part, H1a est fortement exprimé, à la fois dans les transcrits d'ARNm et de protéines, en mESCs (Figure 3 et 4B). Grâce à la quantification des pics H1 dans le profil HPLC, la proportion relative de chaque sous-type H1 individu au sein de la famille H1 est déterminée (figure 5A). En outre, les valeurs de sous-type H1 particulier (ouH1 total) par des nucléosomes peut être calculée par le rapport de l'normalisée Un 214 valeur de crête de sous-types H1 correspondants (S1 ou la somme des totale) à la moitié de la normalisée A 214 valeurs de H2B (figure 5B).
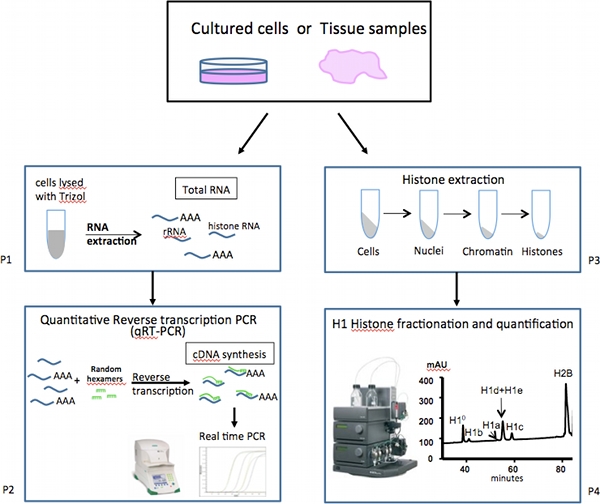
Figure 1. Diagramme d'ensemble de l'analyse d'expression de mammifères linker-histones sous-types.
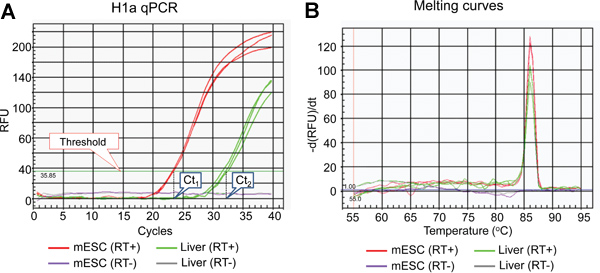
Figure 2. Les résultats représentatifs de H1a qPCR dosage. Parcelle d'amplification (A) de H1a qPCR dosage. La ligne de seuil et les valeurs de Ct fixés par le logiciel iQ5 système optique sont indiqués. (B) dérivés fondre les courbes de produits qPCR représenté dans (A).
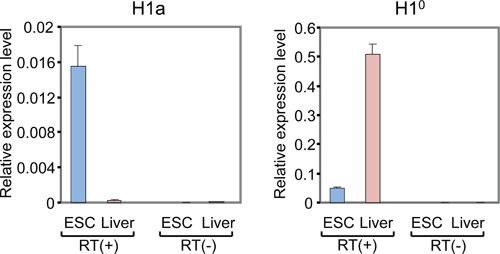
Figure 3. QRT-PCR analyse des niveaux d'ARNm de H1a et H1 ° dans mESCs et adultes mOuse foie. Y représente axe niveaux d'expression relatifs des gènes H1 à celle de la GAPDH de référence gène. qPCR avec RT (-) des échantillons d'ARN (sans transcription inverse) montre peu ou pas de signaux.
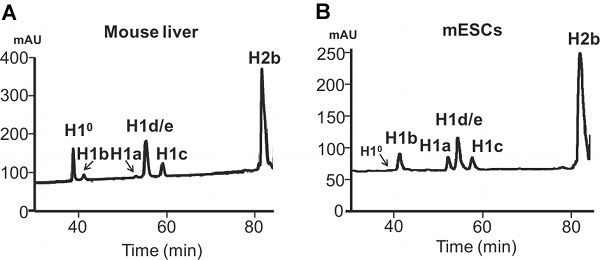
Figure 4. Analyse HPLC des histones extraites à partir des cellules de mammifères. HPLC en phase inverse analyse de 100 pg histones totaux extraits de foie de souris adultes (A) et les CES de souris (B). L'axe X: temps d'élution. L'axe Y: MAU, milli-absorption unités.
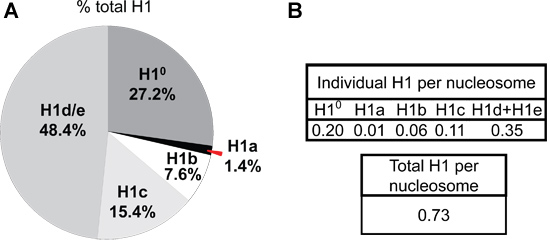
Figure 5. La composition sous-type H1 et H1 ratios par nucléosomes dans le foie de souris adulte. Les valeurs de A 214 de l'aire du pic de chaque isoforme H1 et H2B sont calculés en utilisant le logiciel UNICORN 5,11 (GE Healthcare), et normalisé par le nombre de liaisons peptidiques présents dans la protéine correspondante histone. La somme de normalisée Une214 valeurs de tous les sous-types H1 est obtenue comme la valeur totale pour le 1er semestre. Le pourcentage de H1 total pour chaque sous-type H1 (A) ainsi que le rapport de H1 à nucléosome (représenté par la moitié de la normalisée A 214 valeurs de H2B) (B) dans le foie de souris adulte sont calculés à partir du profil de CLHP représenté la figure 4A.
Discussion
L'ensemble des analyses présentées ici permettent une analyse complète des niveaux d'expression de mammifères linker sous-types d'histones. Bien conçus qRT-PCR fournir des mesures très sensibles et précises de messages d'ARN à partir des gènes de mammifères histone H1. La partie critique de qRT-PCR pour l'éditeur de liens de sous-type gènes des histones est la préparation de l'ADNc en utilisant une amorce aléatoire basé transcription inverse. ARNm des gènes les plus histones, y co...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Ce travail est soutenu par le NIH de subvention et d'un cancer GM085261 Géorgie Coalition Scholar Award distingué (à YF).
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | |
| RNase Zap | Applied Biosystems | AM9780 | |
| Réactif Trizol | Invitrogen | 15596-018 | |
| SuperScriptIII | Invitrogen | 18080-051 | |
| L'éthanol absolu | Fisher Scientific | BP2818-4 | |
| IQ SYBR Green | Bio-Rad | 170-8880 | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| Deoxyribonuclease I | Sigma | AMP-D1 | |
| Microseal plaque de 96 puits PCR | Bio-Rad | MSP-9605 | |
| Joints adhésifs «B» Microseal | Bio-Rad | MSB-1001 | |
| Saccharose | Organics Agros | AC40594 | |
| Phosphate de sodium dibasique heptahydraté (Na 2 HPO 4 · 7H 2 O) | Fisher Scientific | BP332 | |
| Le chlorure de sodium (NaCl) | Américaine bioanalytique | AB01915 | |
| Heptahydraté phosphate monosodique (NaH 2 PO 4 · 7H2O) | Fisher Scientific | BP-330 | |
| HEPES | Fisher Scientific | BP310 | |
| Complete tablette Mini cocktail inhibiteur de la protéinase | Roche Applied Science | 11836153001 | |
| EDTA | Sigma | E-5134 | |
| Fluorure de phénylméthanesulfonyle (PMSF) | Américaine bioanalytique | AB01620 | |
| Nonidet-40 (NP-40) | Américaine bioanalytique | AB01425 | |
| Le chlorure de potassium (KCl) | Fisher Scientific | BP366 | |
| Tris [hydroxyméthyl aminométhane] | Américaine bioanalytique | AB02000 | |
| Le chlorure de magnésium (MgCl2) | Fisher Scientific | BP214 | |
| L'acide sulfurique (H2SO4) | VWR | VW3648-3 | |
| L'hydroxyde d'ammonium (NH 4 OH) | Organics Agros | AC42330 | |
| Bradford Protein Assay | Bio-Rad | 500-0001 | |
| Acétonitrile | EMD | AX0145-1 | |
| L'acide trifluoroacétique (TFA) | JTBaker | 9470-01 |
Références
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon