Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Immunoprécipitation de la chromatine (ChIP) à l'aide Drosophila Tissus
Dans cet article
Résumé
Récemment la technologie de séquençage à haut débit a considérablement augmenté la sensibilité de la chromatine immunoprécipitation (ChIP) expérience et son invité application à l'aide des cellules purifiées ou de tissus disséqués. Ici, nous délimiter une méthode pour utiliser la technique ChIP avec Drosophila, qui peut traiter de l'état chromatine endogène dans un système bien caractérisé biologique.
Résumé
L'épigénétique reste un domaine en développement rapide qui étudie la façon dont l'Etat contribue à la chromatine expression différentielle des gènes dans des types cellulaires distincts à différents stades de développement. Régulation épigénétique contribue à un large éventail de processus biologiques, y compris la différenciation cellulaire au cours du développement embryonnaire et de l'homéostasie à l'âge adulte. Une stratégie essentielle dans les études épigénétiques est d'examiner comment diverses modifications des histones et des facteurs de la chromatine réguler l'expression génique. Pour résoudre ce problème, immunoprécipitation de la chromatine (ChIP) est largement utilisé pour obtenir un instantané de l'association de facteurs particuliers avec de l'ADN dans les cellules d'intérêt.
Technique de ChIP utilise couramment des cellules en culture comme matériau de départ, qui peut être obtenu en abondance et d'homogénéité pour générer des données reproductibles. Cependant, il ya plusieurs mises en garde: Tout d'abord, l'environnement de cultiver des cellules dans les boîtes de Pétri est différente de celle in vivo, ce qui peutreflète pas l'état chromatine endogène des cellules dans un organisme vivant. Deuxièmement, tous les types de cellules peuvent être cultivées ex vivo. Il ya seulement un nombre limité de lignées cellulaires, à partir de laquelle les gens peuvent obtenir suffisamment de matériel pour le dosage ChIP.
Nous décrivons ici une méthode pour faire l'expérience en utilisant des tissus de Drosophila ChIP. Le matériau de départ est disséqué les tissus d'un animal vivant, ce qui peut refléter l'état chromatine endogène. L'adaptabilité de cette méthode avec beaucoup de différents types de tissus permettra aux chercheurs de répondre beaucoup plus biologiquement questions pertinentes relatives à la régulation épigénétique in vivo 1, 2. En combinant cette méthode avec séquençage à haut débit (ChIP-seq) sera en outre permettre aux chercheurs d'obtenir un paysage épigénomique.
Protocole
(La procédure puce entière prend environ deux jours. Préparation des bibliothèques de puces pour séquençage à haut débit prend un autre 2-3 jours.)
1. Disséquer et préparer les tissus pour l'expérience ChIP (~ 1 million de cellules)
- Disséquer les tissus d'intérêt (par exemple 200 paires de Drosophila testicules) dans du PBS froid + inhibiteur de la protéase 1x (dissoudre 1 pastille de cocktail d'inhibiteurs de protéase dans 1,5 ml de PBS 1x pour obtenir une solution stock de 7x) + PMSF (concentration finale 100 pg / mL). Rincer le tissu deux fois et remettre en suspension des tissus dans 200 ul de la même solution PBS avec des inhibiteurs.
- Pour fixer l'échantillon, ajouter 5,5 pi formaldéhyde 37% (formaldéhyde chaud en bain à 37 ° C eau pendant 1 min avant utilisation). Incuber à 37 ° C pendant 15 minutes, vortex toutes les 5 minutes entre les deux.
- Placer l'échantillon sur la glace pour que le tissu reposer pendant 2 minutes. Rincer 2 fois avec 450 ul de PBS (avec les inhibiteurs de protéase et PMSF). L'échantillon peut maintenant être magasind à -20 ° C. Répéter plusieurs fois la dissection plusieurs pour obtenir suffisamment de matériel pour ChIP.
2. Préparer surnageant avec la protéine-ADN Conjugaison pour dosage ChIP
- Mélanger l'échantillon suffisamment de l'étape 1.3 dans un tube 1,75 ml Eppendorf.
- Retirez du PBS à partir échantillon de tissu. Ajouter 200 ul de tampon de lyse (50 mM Tris-HCl, pH 7,6, 1 mM de CaCl2, 0,2% de Triton X-100 ou NP-40, 5 mM de butyrate, et 1x cocktail inhibiteur de protéinase). Ajouter une nouvelle solution de stock PMSF à un tampon de lyse (PMSF concentration finale de 0,5 mM). Homogénéiser avec le bleu homogénéisateur (Cat # 749521-1590 Fisher) jusqu'à ce que les tissus se dissocient complètement sans agrégation, incuber à température ambiante (température ambiante) pendant 10 minutes.
- Sonication: sonicateur micropointe utilisation (Misonix HS-XL200) à la puissance 20. Soniquer pendant 10 sec et le reste sur la glace pendant 50 sec. Répétez 4-5 fois (temps de sonication optimale doit être déterminée de manière empirique, que plus sonication donnera des fragments plus petits. Pour ChIP utilisant des anticorps contre Histonmodifications e, nous avons l'habitude soniquer chromatine à environ 200-300 pb, car le facteur de transcription régulateur de la chromatine ou (par exemple des protéines Polycomb), nous avons l'habitude soniquer chromatine au 200-1000 pb. Cependant, la taille des fragments optimale devrait être testée empiriquement. Remarque: TOUJOURS GARDER LE TUBE ON ICE MEME PENDANT sonication EXEMPLE POUR ÉVITER DE CHAUFFE!
- Diluer l'échantillon en ajoutant 1,8 ml tampon RIPA (10 mM Tris-HCl, pH 7,6, EDTA 1 mM, SDS 0,1%, 0,1% de Na-désoxycholate, 1% de Triton X-100, avec des inhibiteurs de protéase et PMSF à la concentration identique à celle décrite précédemment). Enregistrer 40 ul comme entrée de commande en ajoutant 2 ul NaCl 5M et incuber à 65 ° C pendant la nuit (O / N) pour inverser réticulation.
- Pour conjuguer l'anticorps à des billes, ajouter 40 ul de protéines A perles dans un tube eppendorf de 1,5 ml, puis ajouter 600 ul de PBS et le rock à 4 ° C pendant 2 minutes, appliquer des perles à l'aimant et enlever le surnageant. Ajouter 100 ul de PBS et l'anticorps d'intérêt (quantité d'anticorps doit être déterminée empiquement). Incuber à température ambiante pendant 1 heure ou 4 ° C pendant 4 heures.
- Enlever le surnageant à partir de perles par aimant et ajouter l'extrait de chromatine 1mL de l'étape 2.4 aux perles. Tourner à 4 ° CO / N.
- Appliquer l'échantillon ChIP à l'aimant et enlever le surnageant. Laver les billes avec le tampon suivant à 4 ° C pendant 10 minutes chacune:
2x avec 1 ml de tampon RIPA [1.89 ml "tampon RIPA '+ 315 ul inhibiteurs de la protéase 7x + 20 uL PMSF];
2x avec 1 ml de tampon RIPA + 0,3 M de NaCl [1.89 ml tampon RIPA + 220 pi 3 M de NaCl];
2x avec 1 ml de tampon LiCl (0,25 M LiCl, 0,5% NP40, 0,5% NaDOC);
1x avec 1 ml de TE 1x + 0,2% de Triton X-100;
1x avec 1 ml de TE 1x. - Pour inverser réticulation, remettre en suspension les perles dans 100 pi de tampon TE + 3 pl SDS à 10% + 5 ul de protéinase K (20 mg / mL). Incuber à 65 ° CO / N.
- Appliquer des perles à l'aimant et de transférer le surnageant dans un nouveau tube. Le surnageant contient votre échantillon d'ADN. Laver perles avec 100 ul de TE + 0,5 MNaCl et combiner le surnageant deux.
- Phénol-chloroforme extraction de l'échantillon d'ADN. Ajouter 200 ul de phénol: Chlorofrom: IAA (25/24/1) mélange et le vortex. Centrifuger à 14k pendant 5 minutes à température ambiante. Alternativement, l'ADN peut être extrait en utilisant le kit Qiagen PCR purification.
- Transférer le surnageant / aqueuse dans un nouveau tube, et ajouter l'acrylamide linéaire pour une concentration finale de 20 pg / ml (variante 1 ul de glycogène à 20 mg / ml pour chaque 1 mL de surnageant peut également être utilisé. Le glycogène est utilisé pour qPCR ultérieure ou des analyses PCR en acrylamide linéaire est utilisée pour le séquençage des tests.) Ajouter 20 uL de 3M NaOAc et 500 ul EtOH 100%. Bien mélanger et incuber à 80 ° C pendant 10 minutes. Centrifuger à vitesse maximale pendant 20 minutes à 4 ° C.
- Enlever le surnageant et laver avec 300 EtOH 70% ul. Échantillon d'air sec et remettre en suspension dans 50 pl de tampon TE. Exemple peuvent désormais être stockés à -20 ° C. L'échantillon peut être utilisé pour la PCR quantitative (qPCR) test (Figure 1).
3. Analyser ChIP-ed ADN
3a. Analyser ChIP-ed ADN en utilisant qPCR
- Diluer l'ADN génomique à la concentration ci-dessous pour faire une courbe standard: non dilué, 1/10, 1/100, 1/1000, 1/5000.
- Pour chaque paire d'amorces, mis en place qPCR en double exemplaire en une plaque de 96 puits avec de l'ADN génomique à partir de 3a.1 étapes, le contrôle d'entrée et ChIP-ed ADN:
10 uL 2x SYBR mélange PCR (Fermentas, K0222);
1 pl 10 uM Amorce;
1 ul 10 amorce inverse uM;
x ul ChIP-ed ADN (contrôle d'entrée, ou de l'ADN génomique);
y ul sans nucléase H 2 O pour régler le volume de réaction à 20 pi. - Centrifuger la plaque de 96 puits avec Mini plaque de spinner (Labnet).
- Effectuer qPCR avec ABI 7300 PCR en temps réel du système (ou d'autres systèmes en temps réel PCR) en utilisant la condition suivante:
Étape 1: 50 ° C pendant 2 min, 1 cycle;
Etape 2: 95 ° C pendant 10 min, 1 cycle;
Etape 3: 95 ° C pendant 15 s, à 60 ° C pendant 1 min, 40 cycles;
Etape 4 (étape de dissociation): 95 ° C pendant 15 s, 60 ° C pendant 1 min, 95 ° C pendant 15 s. - Vérifiez la courbe de dissociation: un pic à la parcelle de dissociation thermique suggère un amplicon unique à partir de la réaction de PCR. Plus d'un pic indique non spécifique produit avec la paire d'amorces, les données qPCR doit pas être utilisé.
- Déterminer la phase linéaire de l'amplification exponentielle de la réaction PCR pour chaque jeu d'amorces, en calculant la formule qui résout quantité d'ADN selon l'Ct (cycle seuil) de valeur, qui est basée sur la courbe d'étalonnage en utilisant la série de l'ADN génomique dilué dans 3a. 1.
- Si la valeur de Ct ChIP-ed ADN et de commande d'entrée sont dans la plage linéaire de la valeur Ct, calculer l'enrichissement de l'ADN ChIP-ed par rapport entrée, selon le facteur de dilution d'ChIP-ed ADN et de commande d'entrée en 2,4.
3b. Amplifier ChIP-ed ADN pour le séquençage à haut débit.
- Fin de la réparation de ChIP-ed mélange d'ADN (UtilisezEpicentre d'ADN FIN-kit de réparation), mis en place sur la glace:
1-34 ADN génomique ul de l'étape 2.12 (0.1 à 5 mg)
5 ul de tampon 10x réparation Fin
5 pl 2,5 mM dNTP mix
5 pi 10 mM ATP
x pi H 2 O pour régler le volume de réaction à 49 pl
1 mix ul Enzyme Fin-Réparation (ADN polymérase T4 + T4 polynucléotide kinase)
Incuber pendant 45 minutes à température ambiante. Purifier réaction à l'aide Qiagen MinElute réaction
Kit de nettoyage. Eluer dans 30 ul tampon d'élution (EB). - Ajouter A surplombe les à l'extrémité 3 ':
30 ul du produit de l'ADN élué 3b.1 étape
5 pi de tampon NEB 10x n ° 2
10 pi 1 mM dATP
2 pl dH 2 O
3 pi 5 Fragment de Klenow U / pl (3 '→ 5' exo-)
Incuber pendant 30 minutes à 37 ° C. Purifier réaction à l'aide MinElute ReactionCleanup kit. Eluer dans 10 EB ul. - Solexa ligature linker:
10 pl de l'ADN élué à partir 3b.2 étape
10 ul ddH 2 O
2.5 μ l 10x tampon T4 DNA ligase
1 Adaptateur ul mélange oligo d'Illumina (1/10-diluted de stock)
2,5 pl ligase d'ADN T4 (400 unités / pl)
Incuber pendant 1 heure à température ambiante. Purifier réaction à l'aide MinElute ReactionCleanup kit. Eluer dans 20 EB pl. Exemple peuvent désormais être stockés à -20 ° C. - Exécutez l'échantillon d'ADN élué à partir 3b.3 étape en utilisant l'appareil E-gel. Isoler la bande 300-500 pb dans le gel et purifier en utilisant le kit Qiagen Gel Extraction (cette étape permet d'éliminer les 125 pb de ~ auto-ligaturés linkers de la ligature adaptateur oligo). Eluer dans 12 EB pl.
- Amplification de la bibliothèque en utilisant jumelé-end (PE) amorces d'Illumina:
10,5 ul de l'ADN élué à partir 3b.4 étape
12,5 ul de mélange maître (2X Phusion HF, Finnzymes)
1 pl de PE1 PCR amorce 1
1 pl de PE2 PCR amorce 2
État PCR:
98 ° C pendant 30 sec
(98 ° C 10 Sec, 65 ° C 30 Sec, 72 ° C 30 s), répètent 20 cycles
72 ° C pendant 5 minutes. - Les échantillons provenant de 3b.5 étape peut être utilisé pour ChIP-seq après avoir sélectionné l'ADN de taille appropriée (300-500 pb) en utilisant la norme de 2% gel d'agarose. Exemple peuvent désormais être stockés à -20 ° C. Il est préférable d'isoler chaque échantillon ChIP sur un seul gel pour éviter la contamination. Les échantillons peuvent être soumis à l'séquençage à haut débit.
3c. Analyse du pipeline Solexa
- Le séquençage de 25 pb lit ont été obtenus à partir de l'Analyseur de Illumina Genome (GA) de pipeline.
- Toutes les lectures ont été alignés sur le génome de la Drosophile (dm3) en utilisant le ELAND (Efficient alignement local de données de nucléotides) de logiciels, permettant jusqu'à deux mésappariements avec la séquence de référence.
- Lectures unique cartographiés ont été retenus pour l'analyse en aval. Pour de multiples lectures identiques, jusqu'à trois exemplaires ont été retenus pour réduire la possibilité de biais de l'amplification par PCR.
- La sortie du pipeline GA a été converti en données extensibles navigateur (BED) de fichiers.
- Pour générerles fichiers perruque pour télécharger le navigateur UCSC pour la visualisation, nous avons utilisé un scrip python qui a été décrit dans une publication précédente 3 avec 4 pb que la taille de la fenêtre et 160 pb que la taille des fragments d'ADN.
- Pour classer les gènes chez la drosophile dans les gènes silencieux et exprimé, nous avons utilisé un certain nombre numérique appelé RPKM (lit / par kilobase fusionné région exonique / par million mappé lit). Les gènes avec RPKM = 0 ont été classés dans le groupe silencieux et des gènes avec RPKM ≥ 1 ont été classés dans les groupes exprimées, qui peuvent être classées en trois sous-groupes comme les gènes avec une faible expression, modérée et élevée, et chaque groupe dispose d'environ le même nombre de gènes.
- Pour comparer le niveau de modification des histones et l'expression des gènes, nous avons utilisé un script python qui a été décrit dans un 3 publication précédente.
4. Les résultats représentatifs
Des exemples de ChIP-qPCR résultats en utilisant BAM (sac de billes) mutant testicules sont représentés à la figure 1 4. En bam testicules, il ya un échec dans la transition de spermatogonies proliférative à la différenciation des spermatocytes 5, 6. Nous utilisons bam testicules comme une source de cellules germinales indifférenciées, qui sont enrichis dans ce type de tissu. Gènes de différenciation nécessaire à la différenciation du sperme, comme facteur de transcription spécifique des hommes 87 (mst87F), Don Juan (dj), et l'oignon floue (FZO) ne sont pas exprimés en bam testicules. Ces gènes sont hautement enrichi avec la répression H3K27me3 modification des histones 7 (figure 1A), mais sont dépourvus de la population active H3K4me3 modification des histones 8 (figure 1B), une signature que nous avons appelé chromatine «monovalent» 7. Enrichissement de l'une des H3K27me3 ou H3K4me3 est déterminée par la normalisation à une cycline constitutivement exprimé un gène (Cyca) 4.
contenu "> ChIP-seq analyse en utilisant le même ensemble d'anticorps (c.-à-répressive H3K27me3 et H3K4me3 active) avec bam testicules mutants (Figure 2) a validé les résultats de qPCR de la figure 1. Pour les trois gènes testés différenciation terminale mst87F, dj et FZO , leurs régions génomiques sont hautement enrichi avec H3K27me3 mais pas H3K4me3 (Figure 2A à 2C). En revanche, le gène exprimé de façon constitutive Cyca a H3K4me3 significatif mais peu contraignante H3K27me3 à proximité de son site de départ de la transcription (TSS) (figure 2D).En outre, les profils de ChIP H3K27me3 et H3K4me3 près des TSS de quatre classes de gènes différentiellement exprimés sont compatibles avec la fonction de chaque modification des histones. Comme le montre la figure 3A, l'enrichissement de H3K27me3 en aval de la TSS est inversement corrélée avec le niveau de l'expression des gènes. Les gènes silencieux sont les plus H3K27me3 alors que legènes fortement exprimés n'ont pas contraignant H3K27me3. Ces données sont cohérentes avec le rôle répressif de H3K27me3 sur l'expression génique. En revanche, l'enrichissement de H3K4me3 autour des TSS a montré la corrélation inverse avec le niveau de l'expression des gènes (figure 3B), compatible avec le rôle actif de H3K4me3 sur l'expression génique.
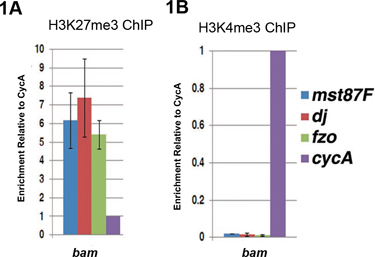
Figure 1. Analyse qPCR de ChIP-ed ADN en utilisant un anticorps contre la répression soit H3K27me3 modification des histones (A) ou actif H3K4me3 modification des histones (B) dans enrichie en cellules indifférenciées bam testicules mutantes. (A) Dans les testicules, bam gènes de différenciation (Mst87F, dj, FZO) sont enrichis avec la modification des histones H3K27me3 répressive. (B) des gènes de différenciation sont épuisées avec H3K4me3 marque active. Le niveau de l'ADN ChIP (puces à ADN / entrée) au niveau du gène cible (Mst87F, dj ou FZO) a d'abord été normalisée à la lev el de l'ADN ChIP au niveau du gène de contrôle Cyca. Les barres d'erreur indiquent l'écart-type de trois indépendante biologique répétitions.
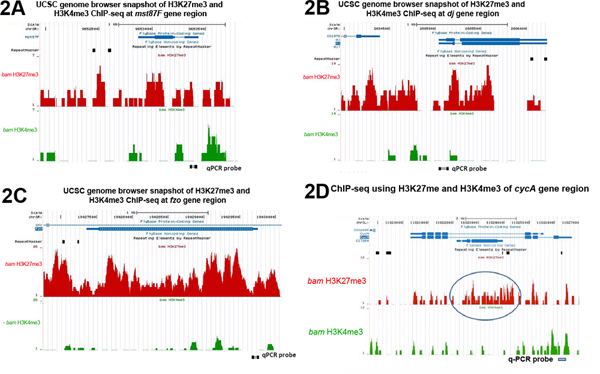
Figure 2. Instantanés UCSC Genome Browser de H3K27me3 et H3K4me3 enrichissement à travers les régions entières de la génomique (A) Mst87F (B) dj et (C) FZO, (D) des gènes Cyca. Le cercle H3K27me3 enrichi région (D) reflète l'état de la chromatine un gène qui se chevauchent CG7264, qui est humble exprimée en bam testicules (RPKM = 1), mais fortement exprimé dans les testicules de type sauvage (RPKM = 130) 8 (ChIP-seq données provenant de 7). Les sondes utilisées dans l'analyse PCR quantitative des résultats ChIP de la figure 1 sont marqués au bas de chaque parcelle. Cliquez ici pour agrandir la figure .
tp_upload/3745/3745fig3.jpg "/>
Figure 3. ChIP-seq profils en utilisant H3K27me3 et H3K4me3 en bam testicules 7. Les quatre groupes de gènes (gènes 7,509) ont été classés en fonction de leurs niveaux d'expression génique basée sur l'ARN-seq résultats 8. Les classes représentatives de gènes sont tracées pour l'enrichissement d'une modification particulière des histones, en utilisant des séquences de 3kb en amont pour 3kb aval de leurs sites de début de transcription (TSS). Cela génère un profil de l'enrichissement de (A) H3K27me3 (K27) et (B) H3K4me3 (K4) ChIP-seq analyses dans chaque groupe. Cliquez ici pour agrandir la figure .
Discussion
La polyvalence des analyses ChIP abordés dans le présent protocole peut être utilisé sur différents tissus, qui prévoit la possibilité d'étudier l'état de la chromatine dans un système biologiquement pertinentes. Expériences utilisant des cellules de ChIP systèmes de culture sont commodes à réaliser parce que grande quantité de cellules peut être facilement obtenu. Cependant, les cellules cultivées ne reflètent pas nécessairement les cellules dans un environnement multi-cellulaire. En dévelop...
Déclarations de divulgation
Nous n'avons rien à communiquer.
Remerciements
Les auteurs tiennent à remercier le laboratoire du Dr Keji Zhao (NIH / NHLBI) pour leur aide dans la fourniture de résultats de séquençage. Nous aimerions également remercier le projet du génome UCSC pour l'utilisation de Genome Browser pour visualiser séquençage cartographié lit.
Ce travail a été soutenu par le Sentier de la R00HD055052 NIH pour Independence Award et R01HD065816 du NICHD, l'Lucile Parkard Fondation, et l'Université Johns Hopkins des fonds de démarrage pour XC
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Complete cocktail Mini inhibiteur de la protéase | Roche | 11836153001 | |
| Le formaldéhyde (37%) | Supelco | 47083-U | |
| PMSF | Sigma | 78830 | |
| Kontes culot pilon | Fischer Scientific | K749521-1590 | |
| Kit de purification PCR | Qiagen | 28104 | |
| Polyacrylamide linéaire | Sigma | 56575-1ML | |
| Le glycogène | Qiagen | 158930 | |
| SYBR green / ROX qPCR Master Mix | Fermentas | K0223 | |
| Mini plaque de spinner | Labnet | Z723533 | |
| PCR en temps réel du système | Applied Biosystem | 4351101 | |
| Processeur petit volume à ultrasons | Misonix | HS-XL2000 | Modèle interrompu |
| Dynabeads, la protéine A | Invitrogen | 100-01D | |
| Dynamag aimant | Invitrogen | 123-21D | |
| Phénol: Chlorofrom: IAA | Invitrogen | 15593-049 | |
| Epicentre d'ADN FIN-kit de réparation | Biotechnologies Epicentre | ER0720 | |
| MinElute reactioKit de nettoyage n | Qiagen | 28204 | |
| Fragment de Klenow (3 '→ 5' exo-) | New England Biolabs | M0212S | |
| ADN ligase de T4 | Promega Corporation | M1794 | |
| Adaptateur oligonucléotides | Illumina | PE-400-1001 | |
| Primer Paired-End 1,0 et 2,0 | Illumina | 1001783 1001 784 | |
| Système Electorphoresis E-Gel | Invitrogen | G6512ST | |
| 2X Phusion HF Mastermix | Finnzymes | F-531 |
Références
- Kharchenko, P. V., Alekseyenko, A. A., Schwartz, Y. B., Minoda, A., Riddle, N. C., Ernst, J., Sabo, P. J., Larschan, E., Gorchakov, A. A., Gu, T. Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485 (2011).
- Filion, G. J., van Bemmel, J. G., Braunschweig, U., Talhout, W., Kind, J., Ward, L. D., Brugman, W., de Castro, I. J., Kerkhoven, R. M., Bussemaker, H. J., van Steensel, B. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Barski, A., Cuddapah, S., Cui, K., Roh, T. Y., Schones, D. E., Wang, Z., Wei, G., Chepelev, I., Zhao, K. High-resolution profiling of histone methylations in the human genome. Cell. 129, 823-837 (2007).
- Chen, X., Lu, C., Prado, J. R., Eun, S. H., Fuller, M. T. Sequential changes at differentiation gene promoters as they become active in a stem cell lineage. Development. 138, 2441-2450 (2011).
- Gonczy, P., Matunis, E., DiNardo, S. bag-of-marbles and benign gonial cell neoplasm act in the germline to restrict proliferation during Drosophila spermatogenesis. Development. 124, 4361-4371 (1997).
- McKearin, D. M., Spradling, A. C. bag-of-marbles: a Drosophila gene required to initiate both male and female gametogenesis. Genes Dev. 4, 2242-2251 (1990).
- Gan, Q., Schones, D. E., Eun, S. H., Wei, G., Cui, K., Zhao, K., Chen, X. Monovalent and unpoised status of most genes in undifferentiated cell-enriched Drosophila testis. Genome Biol. 11, 42-42 (2010).
- Gan, Q., Chepelev, I., Wei, G., Tarayrah, L., Cui, K., Zhao, K., Chen, X. Dynamic regulation of alternative splicing and chromatin structure in Drosophila gonads revealed by RNA-seq. Cell Res. 7, 763-783 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon