Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Simple mutagenèse bisulfite d'ovocytes
Dans cet article
Résumé
Mutagenèse bisulfite est l'étalon-or pour l'analyse de méthylation de l'ADN. Notre protocole modifié permet une analyse méthylation de l'ADN au niveau unicellulaire et a été spécialement conçu pour les ovocytes individuels. Il peut également être utilisé pour le clivage-embryons au stade.
Résumé
L'épigénétique englobe toutes héréditaires et des modifications réversibles de la chromatine que l'accessibilité des gènes alter, et sont donc les principaux mécanismes de régulation de la transcription du gène 1. Méthylation de l'ADN est une modification épigénétique qui agit principalement comme une marque de répression. Grâce à l'addition covalente d'un groupement méthyle sur les cytosines dans les dinucléotides CpG, on peut recruter d'autres protéines de répression et les modifications des histones pour lancer le processus impliqués dans la condensation de la chromatine et faire taire des gènes 2. Méthylation de l'ADN est essentielle pour le développement normal car elle joue un rôle essentiel dans la programmation du développement, la différenciation cellulaire, la répression des éléments rétroviraux, inactivation du chromosome X et l'empreinte génomique.
Une des méthodes les plus puissants pour l'analyse méthylation de l'ADN est la mutagenèse bisulfite. Bisulfite de sodium est un agent mutagène ADN qui désamine cytosines en uraciles. Après amplification par PCR et seque ncing, ces phénomènes de conversion sont détectés comme thymines. Cytosines méthylées sont protégés de la désamination et restent donc en cytosines, permettant l'identification de la méthylation de l'ADN au niveau individuel nucléotide 3. Développement de l'essai de mutagenèse bisulfite a progressé de ceux initialement déclarés 4-6 vers ceux qui sont plus sensible et reproductible 7. Un progrès important a été l'incorporation de petites quantités d'ADN dans un bourrelet d'agarose, protégeant ainsi l'ADN provenant du traitement bisulfite dure 8. Cette analyse de la méthylation permis à être exécuté sur des pools d'ovocytes et embryons au stade blastocyste-9. Le protocole le plus sophistiqué du bisulfite de mutagenèse à ce jour est pour individuels blastocyste-embryons au stade 10. Toutefois, étant donné que les blastocystes ont en moyenne 64 cellules (contenant 120-720 pg d'ADN génomique), cette méthode n'est pas efficace pour les études de méthylation sur les ovocytes individuels ou clivage au stade d'embryons. tente "> Prendre des indices de plongement d'agarose des montants d'ADN minute, y compris les ovocytes 11, nous présentons ici une méthode par laquelle les ovocytes sont directement intégrés dans une solution de lyse agarose et perles de récupération qui suit immédiatement et le retrait de la zone pellucide de l'ovocyte. Cela nous permet de contourner les deux principaux défis de la mutagenèse ovocyte unique: bisulfite. protégeant une infime quantité d'ADN de la dégradation et la perte subséquente au cours des étapes du protocole de nombreuses important, car les données sont obtenues à partir d'ovocytes simples, la question de la partialité PCR au sein des pools est éliminée plus. , par inadvertance la contamination de cellules du cumulus est détectable par cette méthode depuis n'importe quel échantillon avec plus d'un modèle de méthylation peuvent être exclus de l'analyse 12. Ce protocole fournit une méthode améliorée pour les analyses réussies et reproductibles de méthylation de l'ADN au niveau unicellulaire et est parfaitement adapté des ovocytes individuels ainsi que le clivage au stade d'embryons.
Protocole
JOUR 1
Préparer les solutions suivantes fraîches sur le jour de la collecte des ovocytes avec l'eau distillée stérile comme l'eau GIBCO. Pour réduire le risque de contamination par l'ADN, changer de gants souvent et utiliser des embouts de filtre. Garder des tubes incliné à l'écart en position ouverte, et tous les tubes rappel lorsqu'il n'est pas utilisé. Nous recommandons que les solutions sont faites en n +1.
3% d'agarose LMP
30 Point de fusion basse mg (LMP) Agarose
jusqu'à 1 ml H 2 O GIBCO
dissoudre @ 70 ° C
Solution de lyse
8 pi de tampon de lyse
1 pl protéinase K
1 IGEPAL ul 10%
lieu sur la glace jusqu'à ce que prêt à l'emploi
02:01 Agarose: Lysis Solution (10 pi par ovocyte individuelle, le montant est de 3 ovocytes)
20 pi 3% agarose LMP
10 Solution de lyse ul
mélanger à 70 ° C
Ly SDSSIS tampon (501 pi par ovocyte individuelle)
| 1x TE pH 7,5 | 450 pi |
| SDS à 10% | 50 pl |
| Protéinase K | 1 pl |
| 501 pi |
1. Collection d'ovocytes
- Placez les oviductes de souris disséquées dans les médias M2, et arracher les ampoules pour extraire le complexe de cellules du cumulus.
- Séparez les ovocytes à partir du complexe de cellules du cumulus en utilisant 0,3 mg / ml, solution hyaluronidase dans une chute de 30 pi de médias M2. Gardez les ovocytes dans une solution aussi longtemps qu'il le faudra pour éliminer les cellules du cumulus, comme une exposition prolongée peut les endommager. Lavez les ovocytes 3x en baisse de 30 pi de médias M2, en supprimant les cellules du cumulus périodiquement.
- Retirez la zone pellucide en utilisant une solution acide de Tyrode. Placez les ovocytes dans un baisse de 30 ul d'une solution d'abord, puis transférer à un autre 30baisse ul, comme tous les médias entraînés va diluer l'acide et de réduire son efficacité. Gardez les ovocytes dans une solution aussi longtemps qu'il le faudra pour enlever le zona, comme une exposition prolongée peut les endommager. Note: une concentration accrue de solution acide ou de la pronase tyrode peut être utilisé pour des échantillons de l'homme, en tant que zone pellucide humaine est plus résistant à un traitement avec une solution acide de Tyrode que la souris.
- Lavez les ovocytes une fois de plus dans une chute de 30 pi de médias M2.
2. Enrobage d'agarose et lyse
- Pour effectuer intégration d'agarose, placer la solution de lyse sur un heatblock 70 ° C. Ajouter le four préchauffé à agarose LMP à la solution de lyse, la production d'un agarose 2:1: solution de lyse.
- Placez un seul ovocyte sur une lame de verre propre dans un minimum de médias M2. Prendre en hausse de 10 pi de l'agarose: solution de lyse dans une extrémité de la pipette, et (sous un microscope) doucement expulser une petite quantité (~ 1 pi ou moins) sur la lame de verre, ce qui lui permet de se mélanger avec la blancheal médias. Doucement ramasser l'ovocyte dans la pointe de la pipette et de mettre tous les pl 10 dans un tube Eppendorf avec 300 ul d'huile minérale de sorte que le cordon forme une sphère.
Remarque: ce processus doit être fait assez rapidement que l'agarose va durcir si la température descend aussi peu que 5 ° C en dessous de 70 ° C. - Incuber le tube sur la glace pendant 10 minutes. Pour effectuer la lyse, enlever l'huile minérale 300 pi et ajouter 500 ul du tampon de lyse SDS. Incuber une nuit dans un bain à 50 ° C l'eau.
Note: La solution de lyse (Tableau 1) peuvent également être utilisés à cette fin.
JOUR 2
Préparer les solutions suivantes frais sur le jour de la mutagenèse de bisulfite. Pour réduire les risques de contamination par l'ADN, changer de gants souvent et utiliser des embouts de filtre. Garder des tubes incliné à l'écart en position ouverte, et tous les tubes rappel lorsqu'il n'est pas utilisé. Nous recommandons que les solutions sont faites en n +1.
| 3 M de NaOH | 2,4 g de NaOH dans 20 ml autoclavé ddH 2 O |
| NaOH 0,1 M | 0,5 ml de 3M en 14,5 ml autoclavé ddH O 2 |
| 0,3 M de NaOH | 1,5 ml de 3M dans 13,5 ml autoclavé ddH O 2 |
2.5 Solution de bisulfite de M
- 3,8 g de bisulfite de sodium
5,5 ml GIBCO distillée H 2 O
1 ml 3 M de NaOH
dissoudre la température ambiante @ - 110 mg d'hydroquinone
1 ml GIBCO distillée H 2 O
dissoudre @ 90 ° C (pour seulement aussi longtemps que nécessaire pour dissoudre, mélanger régulièrement)
Lorsque complètement dissous, mélanger la solution (a) et (b)
* Tenir à l'écart de la lumière *
3. Mutagenèse bisulfite
- Supprimer complètement le tampon de 500 pl de lyse SDS et ajouter 300 ul d'huile minérale (~ 20 heures). Tout en restant un tampon de lyse va diluer ee agarose quand il est chauffé et le bourrelet est plus sensible à la dissolution dans les étapes ultérieures. Procéder à la mutagenèse bisulfite immédiatement, ou conserver à -20 ° C pour un maximum de 5 jours.
- Le cas échéant, retirer les ovocytes du congélateur et laisser décongeler (seulement jusqu'à ce que agarose perle est relativement translucide). Incuber pendant 2,5 minutes sur un bloc de 90 ° C de chaleur, après quoi Incuber sur de la glace pendant 10 minutes.
Remarque: Ne pas mélanger ou remuer, étendre plus de 2,5 minutes, ou fluctuer la température. - Pour effectuer la dénaturation, retirer l'huile minérale et ajouter 1 ml de NaOH 0.1M dans chaque tube, film et inverser 5-6 fois.
- Incuber pendant 15 minutes dans un bain-marie à 37 ° C, en inversant tous les 3-4 minutes. Le bourrelet doit flotter dans le NaOH.
- Pour effectuer le traitement au bisulfite, tourner doucement le tube, puis retirez le NaOH et ajouter 300 ul d'huile minérale et 500 une solution de bisulfite ul. Incuber le tube pendant 3,5 heures dans un bain-marie à 50 ° C. * Tenir à l'écart de la lumière *
Note: La longueur de l'incubation peut être nécessaire de déterminé de manière empirique pour le gène d'intérêt. - Pour effectuer désulfonation, incuber sur de la glace pendant 3 minutes, puis retirez l'huile minérale et de la solution de bisulfite, tourner doucement, et ajouter 1 ml 0,3 M NaOH. Flick et inverser 5-6 fois.
- Incuber pendant 15 minutes dans un bain-marie à 37 ° C, en inversant tous les 3-4 minutes. Le bourrelet doit flotter dans le NaOH.
- Laver les échantillons, en commençant par tourner doucement, puis retirez le NaOH et ajouter 1 ml 1x TE pH 7,5. Agiter pendant 5-10 minutes à température ambiante (sur un agitateur). Essorage doux à nouveau, puis retirez le TE 1x. Répétez cette opération de lavage à deux reprises.
- Ajouter 1 ml autoclavé ddH 2 O. Agiter pendant 5-10 minutes à température ambiante (sur un agitateur). Essorage doux, puis retirez le H 2 O. Répétez ddH de lavage 2 O à deux reprises.
- Contrôler le pH du surnageant; il devrait être pH 5,0. Si encore trop basique, laver de nouveau avec H 2 O. Retirez tous les surnageant, ne laissant que l'agarose êtreannonce.
4. 1 er et 2 nd cycle de l'amplification par PCR
- Préparer 1 er tour de PCR ** mélange pendant le lavage **
| 10 Primer uM extérieur avant | 0,5 pl |
| 10 Inverser Primer uM extra-atmosphérique | 0,5 pl |
| 240 ng / ml ARNt | 1 pl |
| H 2 O | 13 pi |
Ajouter à Illustra perles Prêt-à-Go Hot Start PCR
Faites glisser délicatement le solide agarose talon dans le tube de PCR (~ 10 pi)
Chauffer à 70 ° C et mélanger
Ajouter 25 ul d'huile minérale
Total: 50 pi
- Amplifier
Remarque: Un exemple de conditions de cyclisme pour la souris est SNRPN dénaturation pendant 2 minutes à 94 ° C, suivie de 40 cycles de 30 secondes à 94 ° C, 1 minute à 50 ° C, et 1minute à 68 ° C, et une finale de 10 minute étape d'élongation à 68 ° C. La température de recuit pendant 1 er tour de PCR pour la souris H19 et Peg3 est de 50 ° C. - Préparer 2 e cycle de PCR mélange
| 10 Primer uM intérieur avant | 0,5 pl |
| 10 Inverser Primer uM intérieure | 0,5 pl |
| H 2 O | 19 pl |
Ajouter à Illustra perles Prêt-à-Go Hot Start PCR
Ajouter 5 pi 1 produit ronde st comme un modèle. Chauffer le produit r 1 ronde à 70 ° C pendant 1 minute à ramollir l'agarose. Veiller au pipetage en dessous de la couche d'huile minérale.
Ajouter 25 ul d'huile minérale
Total: 50 pi
Remarque: des séquences d'amorces imbriquées pour SNRPN, H19, et Peg3 peut être trouvée dans le marché Velker et al 10,12.
- Amplifier
Note: les conditions vélo pour la souris est SNRPN dénaturation pendant 2 minutes à 94 ° C, suivie de 40 cycles de 30 secondes à 94 ° C, 1 minute à 50 ° C, et 1 minute à 68 ° C et une élongation finale 10 minutes étape à 68 ° C 10. Souris H19 et Peg3 besoin d'un 50 ° C la température de recuit pour 2 PCR ème tour. - Comme un test de diagnostic, des deuxièmes échantillons rondes peut être coupé avec une enzyme de restriction qui est la méthylation ou spécifiques de la souche.
| 2 produit e tour | 4 pl |
| Enzyme de restriction | 1 pl |
| Tampon | 1 pl |
| H 2 O | 4 pl |
- Les produits de digestion électrophorèse sur gel d'acrylamide 8%. Les bandes hétérogènes représentent plus d'un sequence.
5. TA Cloning et Colony PCR
- Pour cloner 2 produit ème tour, la première manche à 70 ° C pendant 1 minute pour adoucir l'agarose, puis ligation dans le vecteur en utilisant le Promega pGEM-T Vector System (Fisher Scientific Cat # A1360).
| 2 e ronde PCR | 1 pl |
| pGEMT-EASY vecteur | 1 pl |
| Ligase | 1 pl |
| H 2 O | 2 pl |
| Tampon de ligature 2x | 5 pl |
Incuber une nuit à 4 ° C en machine de PCR.
- Décongeler des cellules compétentes E. coli sur la glace pendant 15 minutes (Zymo Research Corp Cat # T3009). Ajouter 3 pi réaction de ligature à 8 pi de E. coli et la ligature incuber sur de la glace pendant 15 minutes.
- Le choc thermique pendant 40 secondes dans un bain-marie à 42 ° C,et incuber sur de la glace pendant 2 minutes. Ajouter 60 ul moyen SOC et incuber à 37 ° C pendant 1 heure (dans un shaker).
- Placer tout le mélange réactionnel sur une plaque LB / agar / IPTG / Xgal / Amp et la plaque à incuber à 37 ° C pendant la nuit.
- Préparer une PCR sur colonie mélange
| 20 Primer avant uM M13 | 0,7 pl |
| 20 Primer inverse uM M13 | 0,7 pl |
| 5X Visez vert tampon Taq | 7,0 pl |
| DNTP 10 mM | 0,7 pl |
| ADN-polymérase Taq | 0,28 pi |
| H 2 O | 25,62 pi |
| 35 Total ul |
Ajouter 35 ul colonie PCR Master Mix dans un tube PCR. Choisissez une colonie bactérienne blanche de la plaque avec une pointe de pipette, et il tourbillon dans la réaction de PCR.
- Amplification avec une dénaturation pendant 10 minutes à 94 ° C, suivie de 30 cycles de 45 secondes à 94 ° C, 30 secondes à 57 ° C, et 1 minute à 72 ° C, et une étape finale d'élongation de 10 minutes à 72 ° C. Électrophorèse 4 pi sur un gel d'agarose 1,5%. Envoyer ~ 30 ul du produit de PCR pour le séquençage.
Remarque: Pour les ovocytes, 5 colonies produits de PCR sont séquencés. - Une fois les résultats de séquençage sont obtenus, les profils de méthylation peut être lu. Tout ce qui restait d'origine CG en tant que CG a été méthylé, et toute CG d'origine qui est maintenant un TG a été méthylée.
6. Les résultats représentatifs
Dans notre travail, nous avons imprimé test de méthylation dans des ovocytes et des embryons individuels (Figure 1). Après amplification par PCR nichée utilisant des amorces bisulfite convertis, il est possible de confirmer une conversion réussie en visualisant une taille de fragment correct sur un gel d'agarose (figure 2). Un ovocyte individuellereprésente un allèle parental, et en théorie, a un profil de méthylation imprimé. En tant que tel, le second tour des produits de PCR peuvent être testés pour la contamination non intentionnelle. Une enzyme de restriction sensible à la méthylation de l'ADN (tels que HinfI ou DpnII) peut être utilisé pour digérer le second cycle de PCR produit afin de déterminer si elle contient un allèle méthylé ou non méthylé (figure 3). Un C méthylé dans la séquence de reconnaissance enzymatique est clivé en un C non méthylé qui est converti en T n'est plus reconnu par l'enzyme et est non coupé. Tout échantillon d'ovocytes MII contenant à la fois méthylé et non méthylés allèles doivent être jetés, car il est révélateur de la contamination de cellules du cumulus (figure 3). Après la ligature et la transformation, réussie colonie amplification par PCR peut être visualisé sur un gel d'agarose pour s'assurer que les échantillons avec la taille de produit correct sont envoyés pour le séquençage (Figure 4). Enfin, la séquence de cinq cl individuelleceux provenant d'un ovocyte MII devrait produire cinq modèles de méthylation identiques et les taux de conversion identiques nonCpG (figure 5a). Tous les échantillons qui contiennent plus d'un motif doit être éliminée (figure 5b). Depuis ovulés ovocytes MII ont deux copies du chromosome ou un organisme rattaché polaire, il ya une possibilité pour l'obtention de deux configurations de séquences similaires (Figure 5c). Nous recommandons en supprimant les données à partir d'ovocytes qui ont des motifs de méthylation très dissemblables puisque la contamination de cellules du cumulus ne peut pas être exclu.

Figure 1. Schéma de l'essai mutagenèse ovocytes bisulfite unique.
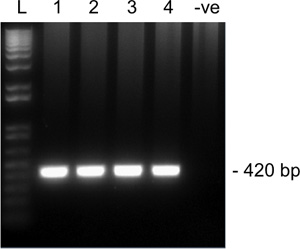
Les résultats représentatifs Figure 2. De 2 ème cycle d'amplification pour SNRPN à partir d'un chantLe MII ovocyte sur un gel d'agarose 1,5%. Lanes 1-4 sont quatre célibataires ovocytes MII et la piste 5 est un contrôle négatif (pas d'ovocytes). Taille de l'amplicon prévue pour SNRPN est de 420 pb. L, échelle.
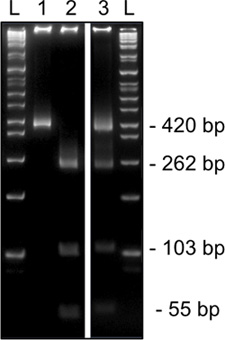
Les résultats représentatifs Figure 3. De 2 ème cycle de digestion de restriction spécifique de méthylation pour SNRPN à partir d'un seul ovocyte MII sur un gel d'acrylamide 8%. La digestion de restriction HinfI diagnostic montre l'ADN non méthylé qui abrite un T qui abolit le site de restriction (420bp, piste 1) ou l'ADN méthylé qui contient un site de reconnaissance au sein de C (coupe, 262, 103 et 54 pb, piste 2). Digestion montrant à la fois des sites méthylés et non méthylés enzyme de restriction (bandes coupées & uncut, piste 3) sont révélateurs de la contamination de cellules du cumulus. L, échelle.
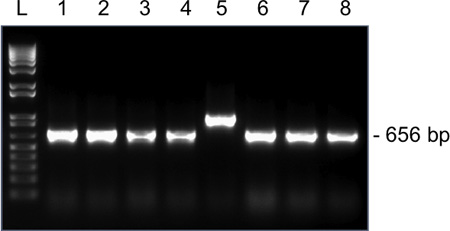
Figure 4. Les résultats représentatifs pour la colonie d'amplification par PCR pour SNRPN à partir d'un seul ovocyte MII sur un gel d'agarose 1,5%. Taille de l'amplicon attendu suite de la ligature des SNRPN dans le vecteur pGEM-T Easy et en utilisant M13 amorces sens et antisens est 656 pb. Lane 1-8, 1-8 amplicons provenant de clones. Clone 5 a une taille de l'amplicon incorrecte et ne doit pas être envoyé pour le séquençage.

Figure 5. Représentant résultats du séquençage pour SNRPN à partir d'un seul ovocyte MII. SNRPN est méthylé dans les ovocytes. Les cercles noirs indiquent CpG méthylés. Les cercles blancs indiquent CpG non méthylés. CpG nombre et l'emplacement est représentatif d'une souris femelle B6 souche. a) prévue résultats du séquençage pour SNRPN à partir d'un seul ovocyte MII. Seul un seul brin d'ADN devrait s'amplifier dans les cinq clones. Les ovocytes avec un modèle de méthylation seule et même boniment de conversion non-CpGn devrait être inclus dans les analyses (de conversion pour cent des non-CpG indiquée à droite a été calculé comme le nombre de non-CpG cytosines converties en thymine comme un pourcentage du total des non-CpG cytosines). b) les résultats de séquençage pour SNRPN à partir d'un seul ovocyte MII à la contamination de cellules du cumulus. Notez la dissemblance entre les Etats et les modèles de méthylation de conversion indiquant amplification brins multiples. c) Le séquençage résultats pour SNRPN à partir d'un seul ovocyte MII avec des copies de chromosomes ou de l'inclusion des deux corps polaire.
Discussion
Ce test seul ovocyte contient de nombreuses étapes avec un certain nombre qui sont essentiels et nécessitent des soins spéciaux. La première est le lavage des ovocytes. Il est particulièrement important de se laver chaque ovocyte à plusieurs reprises dans un milieu frais tombe suite à un traitement hyaluronidase pour enlever le plus les cellules du cumulus plus grand nombre possible. En outre, lors du transfert des ovocytes à la solution acide de Tyrode pour zone pellucide retrait assurez-vous du milieu environn...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par l'Université de Western Ontario, le Département d'obstétrique et de gynécologie, et une subvention ER06-02-188 à partir de la recherche et l'innovation Ministryof, bourse de nouveau chercheur. MMD a été soutenue par un programme de formation des IRSC sur la reproduction, le développement des jeunes et l'impact sur les bourses d'études supérieures de la santé (REDIH).
matériels
Table des réactifs spécifiques et des équipements.
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Collection d'ovocytes | |||
| Hyaluronidase | Sigma | H4272 | |
| Acide de Tyrode | Sigma | T1788 | |
| Protéinase K | Sigma | P5568 | |
| IGEPAL 10% | Bioshop | NON999.500 | |
| Solution de lyse | |||
| Tris, pH 7,5 | Bioshop | TRS001.5 | |
| LiCl | Sigma | L9650 | & Nbsp; |
| EDTA pH 8,0 | Sigma | E5134 | |
| LiDS | Bioshop | LDS701.10 | |
| TNT | Invitrogen | P2325 | |
| Tampon de lyse SDS | |||
| PH 7,5 TE | Bioshop (Tris) Sigma (EDTA) | TRS001.5 E5134 | |
| SDS à 10% | Bioshop | SDS001.500 | |
| Conversion au bisulfite | |||
| Hydroxyde de sodium | Sigma | S8045 | |
| Hydrogénosulfite de sodium (bisulfite de sodium) | Sigma | 243973 | ; |
| Hydroquinone | Sigma | H9003 | |
| Bas point de fusion (LMP) Agarose | Sigma | A9414 | |
| Huile minérale | Sigma | M8410 | |
| Milieu M2 | Sigma | M7167 | |
| GIBCO eau distillée | Invitrogen | 15230-196 | |
| Autoclavée bidistillée (jj) de l'eau | |||
| PCR | |||
| Illustra Hot Mix Démarrer RTG | GE Healthcare | 28-9006-54 | |
| 240 ng / ml ARNt de levure | Invitrogen | 15401-011 | |
| 5x tampon vert réaction GoTaq | Proméga | M7911 | |
| Intérieure et extérieure amorces imbriquées | Sigma | ||
| La ligature | |||
| Promega vecteur pGEM-T Facile | Fisher Scientific | A1360 | |
| TA Cloning | |||
| Compétentes cellules de E. coli | Zymo Research Corp | T3009 | |
| Équipement | |||
| Microscope à dissection | |||
| 70 ° C et 90 ° C à chaleur blocs | |||
| 37 ° C et 50 ° C Bain-marie (42 ° C pour les transformations) | |||
| Bascule | |||
| Machine à PCR | |||
Références
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170 (2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l. Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon