Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Fabrication et utilisation de puces à micro-environnement (MEArrays)
Dans cet article
Résumé
Un procédé de criblage combinatoire fonctionnelle pour obtenir un aperçu des effets de la composition moléculaire du microenvironnement sur les fonctions cellulaires est décrite. La méthode tire parti des microréseaux à base de technologies permettant de créer des tableaux de définis microenvironnements combinatoires qui soutiennent l'adhésion cellulaire et l'analyse fonctionnelle.
Résumé
Les interactions entre les cellules et leur microenvironnement environnante avoir des conséquences fonctionnelles de comportement cellulaire. Le niveau de la cellule unique, microenvironnements distincts peuvent imposer la différenciation, la migration et la prolifération des phénotypes, et sur le niveau des tissus du microenvironnement processus aussi complexe que la morphogenèse et la tumorigenèse 1. Non seulement la cellule et son contenu moléculaire de l'impact des micro-environnements les cellules à l'intérieur, mais il ne faut l'élasticité 2 et 3 de la géométrie des tissus. Définie comme la somme de cellule à cellule, ECM-, et solubles dans les interactions de facteurs, en plus des caractéristiques physiques, le micro-environnement est complexe. Les phénotypes de cellules dans un tissu sont partiellement en raison de leur teneur en génomique et en partie en raison des interactions combinatoires avec le microenviroment. Un défi majeur est de relier des combinaisons spécifiques de composants micro-environnement des comportements distinctifs. ent "> Ici, nous présentons le micro-puces à ADN (MEArray) plate-forme de criblage cellulaire fonctionnelle des interactions avec les micro-environnements combinatoires 4. La méthode permet le contrôle simultané de la composition moléculaire et le module d'élasticité, et combine l'utilisation de puces à ADN largement disponibles technologies micromodelage et. écrans MEArray nécessitent aussi peu que 10.000 cellules par réseau, ce qui facilite les études fonctionnelles des types de cellules rares, telles que les cellules souches adultes. Une limitation de cette technologie est que microenvironnements tissulaires entières peuvent pas être complètement récapitulé sur MEArrays. Cependant, la comparaison des réponses dans le même type de cellule à de nombreux micro-environnements connexes, par exemple les combinaisons deux à deux des protéines de la MEC qui caractérisent un tissu donné, donnera un aperçu de la manière dont les composants du microenvironnement obtenir des phénotypes spécifiques de tissus fonctionnels.
MEArrays peuvent être imprimés en utilisant une grande variété de recombinaison growth facteurs, des cytokines, des protéines purifiées et ECM, et des combinaisons de ceux-ci. La plate-forme est uniquement limitée par la disponibilité des réactifs spécifiques. MEArrays se prêtent à temps caduc analyse, mais sont le plus souvent utilisées pour les analyses de point final de fonctions cellulaires qui sont mesurables avec des sondes fluorescentes. Par exemple, la synthèse de l'ADN, l'apoptose, l'acquisition d'états différenciés, ou la production de produits de gènes spécifiques sont habituellement mesurés. En bref, le débit de base d'une expérience MEArray est de préparer des lames recouvertes de substrats d'impression et de préparer la plaque de maître de protéines qui doivent être imprimés. Puis les tableaux sont imprimés avec un robot de puces à ADN, des cellules sont autorisés à fixer, croître en culture, puis sont fixés chimiquement après avoir atteint le point limite expérimental. Dosages fluorescents ou colorimétriques, imagées avec des microscopes traditionnels ou les scanners puces à ADN, sont utilisés pour révéler pertinentes phénotypes moléculaires et cellulaires (figure 1).
Protocole
1. Impression Substrata Préparation
La décision d'utiliser polydiméthylsiloxane (PDMS)-enduit ou de polyacrylamide (PA)-lames recouvertes dépend des paramètres importants de la conception expérimentale. Le module d'élasticité des deux polymères peuvent être ajustées pour imiter les raideurs des différents tissus en modifiant le rapport base / PDMS de durcissement, et le rapport d'acrylamide / bis-acrylamide de PA. PDMS peuvent imiter les tissus plus rigides dans la gamme de 1-10MPa (cartilage, par exemple, la cornée et les parois des artères), et PA peuvent imiter les tissus mous de l'ordre de 100Pa-100kPa (par exemple, du sein, du cerveau, du foie et de la prostate) 5. PDMS est peu coûteux, facile à préparer, et la géométrie des traits imprimés sera identique à la tête des aiguilles d'impression. Ainsi, la taille et la forme des fonctions peuvent être contrôlées avec précision en utilisant des épingles avec des géométries différentes pointes. PDMS est plus hydrophobe que PA, ce qui provoque des difficultés lors de la manipulation des cellules et immunoscontenant pas, et peut être incompatible avec certaines lignées cellulaires. Parce que PA est un hydrogel et un natif non-encrassement de la surface, les cellules ne fixer à des endroits où il n'y a que des protéines d'adhésion cellulaire de soutien. La géométrie des traits imprimés sur des gels de PA ne suit précisément la géométrie de la tête d'épingle; habituellement, ils deviennent des cercles en raison de la diffusion, quelle que soit la géométrie de tête d'épingle qui est utilisé. Impression des paramètres de contact diamètre de temps et la broche peut être déterminée empiriquement pour la taille caractéristique optimale sur des gels de l'AP.
Polydiméthylsiloxane (PDMS)
- Dans un gobelet en plastique jetable combiner Sylgard 184 de base d'élastomère de silicone avec l'agent de durcissement à un ratio de 10:1, mélanger vigoureusement avec une langue de bois ou de plastique abaisse puis dégazer dans une cloche à vide à température ambiante pendant 30 min.
- Un centre de lame de microscope standard sur le mandrin à vide actionnée d'une tournette, puis bruine 0,5 ml du polymère élastomère mélangé sur le centre de la surface de glissement. Centrifuger à6000 rpm pendant 60 sec.
- Durcir les lames revêtues de PDMS dans un four à 70 ° C ou sur une plaque chaude numérique (protégé de la poussière) pendant 4 heures à une nuit.
- Diapositives durcis peuvent être utilisées immédiatement ou stockées pendant plusieurs mois dans une boîte de diapositive qui est scellé dans un sac Ziploc en plastique et conservés dans un tiroir. Le PDMS attire la poussière et doit donc être bien protégé de la circulation de l'air ambiant.
- Remarque: Nitrile ou d'autres gants sans latex doivent être portés lorsque l'on travaille avec le kit élastomère PDMS. Contact accidentel avec des gants en latex inhibe la polymérisation PDMS.
Polyacrylamide (PA)
- Gravure NaOH: Placer les lames sur un bloc chauffant à 80 ° C. Ajouter 1 ml de NaOH 0,1 N sur chaque diapositive, en prenant soin de couvrir toute la surface de glissement. Laissez-le s'évaporer NaOH (un film blanc doit se former sur la surface de glissement). Depuis le gel PA ne peut attacher solidement sur les surfaces gravées NaOH, le gel PA va se détacher pendant le séchage de l'étape, si le surf diapositive entièreAs n'est pas couverte par NaOH. Si la surface de la lame n'a pas été complètement couverte, répéter en ajoutant 1 ml de NaOH 0,1 N. Les diapositives peuvent ensuite être stockés à température ambiante (TA) pendant plusieurs jours. Note: Une alternative à la gravure NaOH est de la couche d'ozone ou de plasma nettoyer les lames.
- 3-aminopropyltriéthoxysilane (APES) revêtement: Dans une hotte, placer les lames dans une boîte de 15 ml et ajouter 300 ul APES sur chaque diapositive NaOH gravé. Laissez les APES réagir avec le NaOH glisse pendant 5 min. Le dépassement de ce temps provoquera des problèmes de lavage n'a pas réagi réactif APES. Laver soigneusement avec de l'eau APES désionisée deux à trois fois sur les deux côtés des lames. Si le lavage n'est pas complète, APES seront oxydés par le glutaraldéhyde pour former un dépôt brun sur coulisse dans l'étape 1,8.
- Oxydation glutaraldéhyde: Aspirer toutes les solutions des surfaces de glissement. Dans chaque plat 15 cm, ajouter 25 ml de glutaraldéhyde à 0,5% dans du PBS. Réagir pendant 30 min dans un endroit sombre. Après 30 minutes, aspirer tout le glutaraldéhyde et l'utilisation non-lint du travaillingettes Atory (Kimwipes par exemple) pour bien sécher les diapositives. Les diapositives peuvent ensuite être stockés à température ambiante pendant une journée.
- Préparation du gel: Après la préparation des mélanges PA, y compris l'acrylamide, le bis-acrylamide et ddH 2 O en s'accordant avec le tableau ci-dessous, dégazer pendant 30 min, puis placez mélanges PA sur la glace pour ralentir la polymérisation. Ajouter APS et TEMED et mélangez bien droit avant d'effectuer les gels. Pipet mélanges PA sur les surfaces de glissement et les placer 24 mm x 50 mm, numéro 1 des lamelles sur le dessus de l'Autorité palestinienne. Evitez d'appuyer sur la lamelle et lame de verre ensemble et éviter la formation de bulles. Pour les gels> 40.000 Pa utiliser 100 pi, pour les autres gels utiliser 350 pi.
| Module désiré (Pa) | % D'acrylamide | Bis-acrylamide% | L'acrylamide en stock 40% (ml) | Bis-Acrylamide en stock 2% (ml) | De l'eau déminéralisée (ml) | APS (pl) | TEMED (pl) |
| 480 ± 160 | 3 | 0,06 | 0,75 | 0,3 | 8,95 | 100 | 10 |
| 4470 ± 1190 | 5 | 0,15 | 1,25 | 0,75 | 8 | 100 | 10 |
| 40.400 ± 2390 | 8 | 0,48 | 2 | 2,4 | 5,6 | 100 | 10 |
Adapté de 6,7.
- Laissez le gel PA polymériser pendant 2 heures, puis retirez lamelles dans de l'eau déminéralisée.
- Laver les lames gel PA dans de grandes jarres Coplan dans l'eau pendant la nuit (~ 8 h) pour enlever les acrylamides n'ayant pas réagi.
- Lames sèches dans un four à 37 ° C pendant 2-4 heures ou jusqu'à ce que PA gel durcit complètement.
- PA gel diapositives peuvent être conservés à 4 ° C pendant un mois dans une boîte de coulissement étanche.
2. Préparation Plate protéines Maître
- Toutes les protéines should être aliquotés dans des actions de solutions 10X dans les tampons recommandées par le fournisseur et conservés à -80 ° C. La plupart des protéines ECM sont solubles dans l'eau déminéralisée, mais le pH peut être ajusté avec des gouttes d'acide acétique. La plupart des facteurs de croissance, des cytokines et des récepteurs recombinants domaines extracellulaires sont préparées dans du PBS avec BSA, mais les conditions du fabricant peuvent varier. Filtrer les aliquotes de protéines à travers une um 0,45 mm nylon 4-seringue filtre (Nalgene) avant le stockage.
- Concevoir une plaque maître conformément aux combinaisons de protéines désirées et des dilutions. Les cellules adhérentes généralement compter sur la présence d'au moins un ECM compatible avec la médiation d'adhésion cellulaire, mais des anticorps dirigés contre des épitopes de surface des cellules peut également servir de médiateur attachement parfois. C'est une bonne idée d'ajouter un colorant libre FITC ou une protéine fluorophore conjugué à au moins un puits de sorte que les tableaux peuvent être facilement orientée plus tard.
- Préparez la plaque de maître par dilution des combinaisons de protéines avec un tampon d'impression composé de 100 mMTris-acetate/20% glycerol/0.05% de Triton-100X pH 5,2. En général, chaque puits d'une plaque de 384 puits ne contient pas plus de 10 ul.
- Enregistrer le contenu de chaque puits de chaque plaque maître dans un fichier de données délimité par des tabulations de base et fournir à chaque plaque de maître avec un numéro d'identification unique. Une date à six chiffres suivi par les initiales du concepteur sert souvent l'objet (MMDDYYinitial). Parce que les volumes sont faibles et, des aliquotes de protéine peuvent être utilisés efficacement pour générer un grand nombre de plaques répétés. Il est recommandé que les plaques maîtres sont stockés à -80 ° C et chaque plaque maître doit subir plus de deux cycles gel-dégel.
3. Impression MEArray
- MEArrays peuvent être imprimés avec les plus classiques robots impression de biopuces. Les imprimantes qui utilisent des broches Quill silicone ou broches en acier inoxydable fonctionnent bien, mais la viscosité protéine peut être problématique. Imprimantes capillaires sont idéales robots impression microarray pour cette application, car ils travaillentbien avec les solutions de protéines visqueux.
- Pour atteindre une bonne puissance statistique dans un tableau de 10 à 12 points répétés de chaque micro-environnement est recommandée. Une telle conception permettra une comparaison de l'activité dans un microenvironnement par rapport à l'autre dans le même tableau en utilisant de simples T-statistiques de test. Dunnette essai peut être utilisé pour comparer l'activité dans un environnement de contrôle avec d'autres micro-environnements. Cette conception fonctionne mieux quand un phénotype fonctionnel a été associé avec le micro-régulation avant d'effectuer les expériences MEArray.
- L'humidité doit être maintenue autour de 50%. Contrôle de l'humidité est importante, car un faible taux d'humidité peut sécher la solution à l'intérieur des broches ou dans les puits de la plaque de maître provoquant le dépôt inefficace sur les substrats d'impression. L'humidité peut être contrôlé efficacement par drapage du robot avec les non-poreuse des bâches en plastique et en utilisant à la fois un humidificateur et un déshumidificateur réglé pour maintenir une humidité de 50%. Plattens d'impression peuvent être refroidis useful pour la préservation de certaines protéines, mais il faut être prudent afin d'éviter la formation de condensation sur les diapositives.
- Chaque tableau imprimé doivent être étiquetés avec des étiquettes de diapositives congélateur encodées à l'épreuve avec un numéro de série qui se compose de la plaque d'identification de maîtrise suivie d'un nombre à trois chiffres (MMDDYYinitial-nnn). Comme chaque tableau est utilisé ou distribué, les détails de leurs traitements expérimentaux devraient être maintenus dans une base de données. Le suivi des dates d'impression et le nombre de cycles gel-dégel des plaques maîtres aidera à identifier les conditions optimales pour le maintien de la reproductibilité.
- MEArrays imprimés doivent être stockés dans des boîtes scellées de diapositives à -20 ° C pendant pas plus d'un mois. Reproductibilité décline sensiblement par la suite.
4. La culture de cellules de mammifères sur MEArrays pour l'analyse fonctionnelle
- Fixez chambres de culture: Pour limiter le volume des médias et des numéros de culture de cellules requises sur les MEArrays, une chambre en plastique est fis pour entourer la matrice imprimé. Pour les tableaux nombreuses, une seule chambre d'une diapositive 2-chambre (Nunc) qui contient une zone de 4,2 cm 2 peut être utilisé. Retirez les chambres de la chambre de coulissement manufacturés et coupez les en deux chambres avec une lame de rasoir. Utiliser une seringue de 3 ml d'appliquer un mince cordon de silicone aquarium (DAP) pour le bord d'une chambre de presse et sur la surface d'un MEArray. Évitez de placer la chambre d'aquarium appliqué du silicone sur les caractéristiques du tableau.
- Obturation et de rinçage: MEArrays doivent être bien rincés pour éliminer les monomères n'ayant pas réagi, qui peuvent être toxiques pour les cellules. Si PDMS lames enduites ont été utilisés, puis entre les régions dans les caractéristiques imprimées doivent d'abord être bloquée avec un revêtement anti-fouling pour empêcher l'adhésion cellulaire; incuber dans les tableaux 1% Pluronic F108 (BASF) dans de l'eau ou de BSA 2% en l'eau pendant 15 min sous vide. PA diapositives gel ne nécessite pas d'étape de blocage. Dans tous les cas, rincez tableaux avec des milieux de culture de cellules trois fois pendant cinq minutes (choix des médias dépendsur les cellules utilisées, mais l'utilisation d'antibiotiques est recommandée peu importe le support ou cellules). Gels PA désirez plus d'incubation de 30 minutes dans les médias pour réhydrater le gel.
- La fixation des cellules: Quatre à cinq tableaux peuvent s'adapter à l'intérieur d'un 15 chambres simples cm stérile boîte de Pétri. Couvrir le plat de Pétri avec un couvercle pour maintenir les tableaux stérile. Ajouter la moitié du volume final de la MEArray médias en ajoutant les cellules dans un milieu à une concentration finale de 10.000 à 1.000.000 cellules / ml. Les cellules se fixent sur les caractéristiques imprimées à des vitesses différentes en fonction de la composition de la micro-imprimés. Vérifier la fixation uniforme en consultant les tableaux à travers un microscope inversé stade en 15 à 20 minutes d'intervalle. En agitant doucement les MEArrays d'avant en arrière, les cellules attachées de manière à motifs peuvent être distinguées des cellules flottantes, seules.
- L'élimination des cellules non liées: Le PA enrobées de MEArrays, les cellules non liées peuvent être aspirés et les médias peuvent être remplacés par un volume approprié. Le PDMS-enduites MEArrays, les médias ne peuvent jamais être complètement retiré du puits, car les cellules se dessèchent et meurent presque immédiatement. Ainsi le PDMS revêtues MEArrays, les cellules non liées doit être enlevée par un processus d'échanges successifs de la moitié du volume de média jusqu'à ce que toutes les cellules non liées sont éliminées, déterminé par examen microscopique. L'effet de l'arrosage de PDMS est moins important lorsque les milieux contenant du sérum est utilisé par rapport à des milieux définis, et lorsque BSA est utilisé pour bloquer les zones non imprimées par rapport à Pluronics F108.
- Les cellules peuvent être cultivées sur MEArrays placés à l'intérieur des boîtes de Pétri de 15 cm pendant plusieurs jours avec des changements de supports normaux. Les changements de supports sur des lames de PDMS doit être fait avec les modifications successives de la moitié du volume des médias.
- Fixateurs courants, comme le paraformaldéhyde et le méthanol / acétone, sont compatibles avec les systèmes MEArray. Lors de la coloration des cellules sur PA enrobées de MEArrays, fixateurs peuvent être ajoutés et emporté tout comme ils le seraient dans une procédure de coloration conventionnelle.Toutefois, lorsque la coloration des cellules sur PDMS enduits MEArrays, la surface doit rester mouillée, même pendant la fixation. Aspirer la moitié des médias et le remplacer par un fixateur. Répétez le processus plusieurs fois jusqu'à ce que le puits est rempli d'une majorité de fixateur. Après fixation, le fixateur est progressivement remplacé de la même manière avec un tampon de blocage qui est appropriée pour l'étape suivante de l'analyse.
- Immunomarquage est couramment utilisé pour analyser les fonctions cellulaires. Routines de coloration peut varier, mais lorsque l'on travaille sur les MEArrays PDMS, il faut effectuer chaque étape de lavage et aspiration comme ci-dessus, changeant progressivement les solutions et ne permettant jamais à la surface de-humide. Démouillage va causer des artefacts de coloration.
- Les chambres peuvent être enlevées à l'aide d'une lame de rasoir. Lamelles couvre peut être monté sur le dessus de MEArrays colorées à l'aide Fluoromount-G (Southern Biotech). La détection peut être réalisée avec la plupart des scanners multicolores biopuces à fluorescence ou microscopie confocale à moteurcarrelage modes d'acquisition d'images.
5. Les résultats représentatifs
Un exemple de dépôt de protéines à motif imprimé sur un PDMS-côtoyé MEArray l'aide d'épingles à pointe carrée de silicium sur une plume broche de microréseaux d'impression robot est illustré à la figure 2. Dépôt de protéines différentes qui sont imprimés peut être vérifiée par immunofluorescence en utilisant des anticorps (figure 2A). Dilutions des solutions de protéines dans la plaque maître sont le reflet de la quantité (intensité de fluorescence) qui est déposée sur la surface de substrats d'impression (Figure 2B). Les cellules doivent s'attacher aux caractéristiques imprimées de manière évidente à motifs (figure 2C).
Un exemple d'une expérience montrant que MEArray dilutions inverses de deux protéines spécifiques microenvironnement suscité profils d'expression d'une protéine de kératine manière concentration dépendante dans un multipotentes mammaire p épithéliale humainelignée cellulaire rogenitor (D920 cellules), est présenté à la figure 3. Parcelles bulles s'avèrent utiles pour déterminer si des phénotypes spécifiques sont imposées sur les caractéristiques de cellules identiques d'une série de dilutions. Par exemple, si une molécule particulière dans un microenvironnement provoque un phénotype distinct, une fois le composant instructive a été dilué suffisamment dans un contexte de ECM neutre le phénotype doit changer ou disparaître. Détection par immunofluorescence de la kératine kératine 8 et 14 protéines des filaments intermédiaires a été réalisée avec un 4200A Axon (Molecular Devices) scanner microarray. Douze séries de dilution réplique ont été imprimés sur chaque MEArray, et le journal 2 Le ratio de la kératine kératine 14 8 à intensité moyenne de fluorescence a été représentée graphiquement comme un complot bulle pour donner une idée réaliste de la variation et de la reproductibilité du signal. Ci-données d'un MEArray qui a été fixé après que les cellules ont attachés et les cellules non liées ont été emportés (figure 3A), et après 24 heures de culture (figure 3B). Pour cette analyse, relativement faible, une analyse de variance à sens unique a été utilisée pour déterminer la variance de la moyenne du signal à chaque point dans le temps, et regroupés en deux tests t bilatéraux ont été utilisés pour déterminer si les différentes dilutions de collagène de type I et humaine recombinante P- cadhérine a provoqué des changements dans l'expression de la kératine. Il n'y avait pas de variation de la moyenne entre les cellules sur les caractéristiques juste après l'attachement, mais il y avait des différences significatives dans l'expression entre les cellules de kératine après 24 heures d'exposition à des micro-environnements différents. Les tests t vérifié ce type haute concentrations de collagène I a suscité plus kératine 8 expression, alors élevées P-cadhérine concentrations suscité une forte kératine de signal 14 après 24 h. Ce résultat est cohérent avec les rapports précédents que la P-cadhérine contenant des micro-environnements imposera des K14-exprimant le phénotype myoépithéliales le bi-puissants des cellules progénitrices mammaires 4.
Un exemple d'un scanner à touted MEArray imprimé sur un gel de 40.000 Pa PA est illustré à la figure 4.

Figure 1. Un organigramme de la procédure MEArray. Tout d'abord, les substrats d'impression sont préparées soit avec PDMS ou PA. Deuxièmement, les plaques maîtres sont préparés et annotés dans une base de données. Troisièmement, les MEArrays sont imprimés et codés avec des numéros de série. Quatrièmement, chambres de culture sont attachés, les surfaces sont des blocs et / ou rincée, puis les cellules sont autorisés à fixer et les cellules non liées sont éliminées. Cinquièmement, les cellules peuvent être traitées avec des taches ou bio-essai après une période d'incubation basée sur la conception expérimentale. Enfin, Images de MEArray peuvent être obtenues et analysées avec le scanner approprié et logiciels.
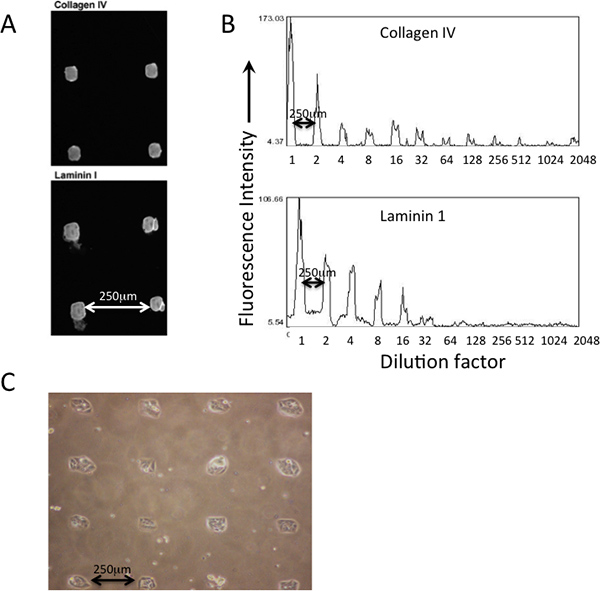
Figure 2. Deposition et l'abondance relative des protéines imprimées peut être vérifié avec immunomarquage avant la fixation des cellules. A) Les anticorps qui reconnaissent collagène de type IV et de la laminine-111 ont été utilisés pour vérifier leur présence dans les caractéristiques imprimées d'un MEArray. B) En utilisant une caractéristique intensité de pixel moyenne dans l'analyse NIH logiciel ImageJ, l'abondance relative des deux protéines à travers une série de dilutions à partir d'une solution de protéine de 200 pg / ml, peut être évaluée qualitativement. Micrographie phase C) des cellules attachées à D920 de forme carrée caractéristiques d'un imprimé à revêtement PDMS MEArray.
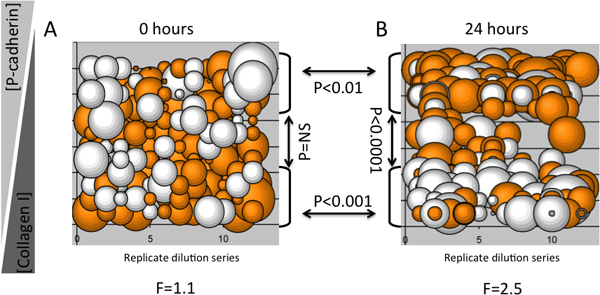
Figure 3. Un exemple d'analyse MEArray utilisant des changements dans l'expression de kératine dans une lignée de cellules progénitrices multipotentes comme une fonction du temps et du microenvironnement. Chaque bulle représente les rapports de la kératine kératine 8 et 14 niveaux de protéines de 10 à 15 cellules attachées à une feature dans un MEArray. L'expression a été déterminée avec des sondes d'immunofluorescence. A) montre les ratios de kératine dans les cellules juste après l'attachement, et B) montre les ratios de kératine après 24 heures sur un tableau qui a été étalée en parallèle. La concentration maximale de deux protéines était de 200 pg / ml et diluée 2 fois. Le diamètre d'une bulle représente l'amplitude du log 2 de la kératine rapport 8 et 14 kératine intensité moyenne, et le codage de couleur orange et blanc indique des valeurs> 0 et <0, respectivement. Valeurs de F pour une ANOVA et P-valeurs des tests t et des supports avec des flèches d'identifier les populations comparées, sont présentés.

Figure 4. Un exemple d'une analyse MEArray acquises en utilisant un mode d'acquisition de carrelage sur un microscope confocal à balayage laser. HCC1569 cellules nous a permis d'intégrer l'ADN analogique EdU pendant 4 heures avant la fixation.DAPI (bleu) et Edu (rouge) sont affichés.
Discussion
La méthode présentée ici permet MEArray analyses fonctionnelles des cellules et les interactions combinatoires microenvironnement 4. MEArray analyse combine l'utilisation des technologies de base micromodelage, biologie cellulaire, et des robots d'impression microarray et des dispositifs d'analyse qui sont disponibles dans les installations multi-utilisateurs nombreux. MEArray écrans sont compatibles avec la plupart des types de cellules adhérentes, mais sans sérum formulations de milieux pe...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
ML est pris en charge par la NIA (R00AG033176 et R01AG040081) et Laboratoire de recherche et de développement en scène, US Department of Energy contrat # DE-AC02-05CH11231.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires (optionnel) |
| Lames de verre 25 mm x 75 mm | VWR | 48311-600 | |
| Verre de lamelles (n ° 1) 24 mm x 50 mm | VWR | 48393-241 | |
| Bac de coloration (ou un bocal Coplan) | VWR | 25461-003 | |
| Des boîtes de Pétri (15 cm) | BD Falcon | 351058 | |
| NaOH (1,0 N) | Sigma-Aldrich | S2567 | |
| APES (> 98% (3-aminopropyl) triéthoxysilane) | Sigma-Aldrich | A3648 | |
| Glutaraldéhyde | Sigma-Aldrich | G7651 | 50% dans l'eau |
| APS (> persulfate d'ammonium à 98%) | SIGMA-Aldrich | A3678 | Préparer solution à 10% de travail avec ddH 2 O |
| TEMED (N, N, N ', N'-tétraméthyléthylènediamine) | Sigma-Aldrich | T9281 | |
| L'acrylamide (40%) | Sigma-Aldrich | A4058 | |
| Bis-acrylamide (2% v / v) | Fisher BioReagents | BP1404-250 | |
| Seringue 0,45 um filtre à 4 mm en nylon | Nalgene | 176-0045 | |
| FITC | Sigma-Aldrich | F4274 | |
| PDMS (polydiméthylsiloxane) | Dow Corning | 3097358-1004 | Kit Sylgard élastomère 184 via Ellsworth Adhesives |
| 2-chambre diapositives | NUNC | 177380 | |
| Pluronic F108 | BASF | 30089186 | |
| Aqumastic arium | Dow Corning | DAP 00688 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Gobelets en plastique jetables | |||
| Abaisse-langues | |||
| Les gants en nitrile | |||
| Boîtes en plastique lames de microscope | |||
| Tournette | WS-400B-6NPP/LITE | Technologies Corporation Laurell | |
| Four | |||
| Plaque de cuisson numérique | |||
| Plaques 384 puits | Une marque appropriée pour le robot puces à ADN | ||
| Robot d'impression microarray | |||
| Phase inversée et microscope à fluorescence | |||
| Axon microarray scanners | Molecular Devices | Plusieurs configurations existent |
Références
- Bissell, M. J., Labarge, M. A. Context, tissue plasticity, and cancer: are tumor stem cells also regulated by the microenvironment. Cancer Cell. 7, 17-23 (2005).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev. Cell. 6, 483-495 (2004).
- LaBarge, M. A. Human mammary progenitor cell fate decsions are products of interactions with combinatorial microenvironments. Integrative Biology. 1, 70-79 (2009).
- Kim, H. N. Patterning Methods for Polymers in Cell and Tissue Engineering. Annals of biomedical engineering. , (2012).
- Boudou, T., Ohayon, J., Picart, C., Pettigrew, R. I., Tracqui, P. Nonlinear elastic properties of polyacrylamide gels: implications for quantification of cellular forces. Biorheology. 46, 191-205 (2009).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current protocols in cell biology. Chapter 10, Unit 10 (2010).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat. Methods. 2, 119-125 (2005).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol. Syst. Biol. 2, 37 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon