Method Article
Multi-cible approche de traitement parallèle pour Gene à la structure Détermination de la polymérase de l'influenza PB2 Subunit
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Structure basée sur la conception de médicaments joue un rôle important dans le développement de médicaments. La poursuite des cibles multiples en parallèle augmente considérablement les chances de succès pour la découverte de plomb. L'article qui suit met en évidence la façon dont le Centre de génomique structurelle Seattle pour maladies infectieuses utilise une approche multi-cible pour la détermination gène à la structure de la grippe A de sous-unité PB2.
Résumé
Pandémies de souches de virus influenza hautement virulentes peuvent provoquer la morbidité et de la mortalité répandue dans les populations humaines à travers le monde. Aux États-Unis seulement, une moyenne de 41.400 morts et 1,86 million d'hospitalisations sont causées par une infection de virus de la grippe chaque année 1. Des mutations ponctuelles dans la polymérase de base protein 2 sous-unité (PB2) ont été liés à l'adaptation de l'infection virale chez l'homme 2. Les résultats de ces études ont mis en évidence l'importance biologique de PB2 comme un facteur de virulence, mettant ainsi en évidence son potentiel en tant que cible thérapeutique antivirale.
Le programme de génomique structurale mis de l'avant par l'Institut national des allergies et maladies infectieuses (NIAID) fournit du financement à Emerald Bio et trois autres institutions du Nord-Ouest du Pacifique qui, ensemble, forment le Centre de génomique structurelle Seattle pour les maladies infectieuses (SSGCID). Le SSGCID se consacre à fournir à la communauté scientifique threstructures protéiques e-dimensionnelles de NIAID catégorie pathogènes AC. Rendre cette information structurelle à la disposition de la communauté scientifique sert à accélérer la structure basée sur la conception de médicaments.
Structure basée sur la conception de médicaments joue un rôle important dans le développement de médicaments. La poursuite des cibles multiples en parallèle augmente considérablement les chances de succès pour la nouvelle découverte de plomb en ciblant une voie ou une famille de protéine entière. Emerald Bio a développé un pipeline de traitement parallèle haut débit, multi-cible (MTPP) pour la détermination gène à la structure pour soutenir le consortium. Nous décrivons ici les protocoles utilisés pour déterminer la structure de la sous-unité PB2 de quatre grippe A souches différentes.
Protocole
Un aperçu du protocole est présenté dans la Figure 1.
Biologie moléculaire
1. Construire Conception
Utilisez un logiciel de compositeur Gene concevoir construction des protéines et des séquences de gènes synthétiques conçus codons. L'utilisation de logiciels de compositeur Gene a été offert en détail ailleurs 3.
- Utilisez le module de visualisation d'alignement et de construction module de conception de comparer les alignements de séquences de protéines et de définir construction des protéines. Alignez séquence d'acides aminés de la cible à la fois les éléments structurels primaires et 3D à partir des homologues dans la Protein Data Bank (PDB), si disponible (Figure 2).
- Utiliser les informations d'alignement pour faire des dessins de construct structure guidées en choisissant nouvelles extrémités basé sur la conservation de la structure primaire et des structures 3D des homologues.
- Conception insert PCR (iPCR) et le vecteur PCR (RVPC) amplimers (borne amorces).
- Utilisation Gene CL'algorithme de protéine à l'ADN de omposer, arrière-traduire la séquence d'acides aminés de synthèse dans la séquence d'acide nucléique d'ingénierie codon.
- Utilisez le tableau d'usage des codons bon (CUT) pour optimiser la séquence pour l'expression dans E. coli.
- Pratiquement cloner insérer dans pET28 vecteur modifié pour intégrer un tag histidine N-terminal 6x et la protéine de fusion Smt3/SUMO qui permet la purification facile.
- Placez l'ordre des gènes synthétiques avec de l'ADN 2.0 et l'ordre des amorces de Integrated DNA Technologies.
2. Polymerase incomplète Primer Extension (PIPE) Clonage
- Préparer amorces et les gènes
- Centrifuger les plaques fournies par le fabricant contenant des amorces à 1000 rpm pendant 1 min.
- Apportez concentration d'amorce à 100 um et ajouter un tampon TE 50 pi.
- Diluer amorces à 10 um avec déminéralisée (DI) dans une plaque à fond en V à 96 puits.
- Centrifuger le gène fourni par le fournisseur dans un tube de 1,5 ml à 1300 rpm pendant 1 min.
- Utilisation de tampon TE, amener la concentration de l'ADN de chaque tube à 50 ng / pl.
- Dans des tubes de 1,5 ml, faire des dilutions de chaque amorce à 10 ng / ul.
- Boutique amorces et des gènes à -20 ° C lorsqu'il n'est pas utilisé.
- Préparer Insérer PCR (iPCR)
- Décongelez un flacon de PFU Master Mix sur la glace; garder les gènes et les amorces à la température ambiante.
- Créer un plan d'affectation des puits de plaque à un ensemble d'amorces et de construire.
- Ajouter 13 ul d'eau DI dans chaque puits d'une plaque PCR 96 puits.
- Ajouter 5 ul de l'avant et 5 pi d'amorce inverse à chaque réaction dans la plaque de 96 puits selon le plan de la plaque, en veillant à changer conseils entre chaque puits.
- Ajouter 2 ul de chaque gène de pleine longueur à son bien approprié selon le plan de la plaque.
- Ajouter 25 ul de PFU master mix à chaque puits, en veillant à changer conseils entre chaque puits.
- Vélo les réactions en utilisant les conditions de PCR suivantes:
- 95 ° C 2 min
- 95 ° C 30 sec
- 50 ° C 45 sec
- 68 ° C 3 min
- 4 ° C ∞
- Répétez les étapes bd pour 25 cycles.
- Transférer 10 pl de chaque réaction iPCR à une nouvelle plaque PCR 96 puits.
- Ajouter 3 pi de colorant de charge 6X pour chaque échantillon.
- Séparez chaque échantillon sur un% TAE BET gel d'agarose 1 à 110 V à côté d'un bps échelle d'ADN 100-500 pour confirmer amplification du fragment.
- Produit en iPCR à -20 ° C lorsqu'il n'est pas utilisé (éviter un gel dégel autant que possible).
3. Préparer PCR Vector (RVPC)
- Commencez culture de nuit de transformer E. coli avec pET28 vecteur plasmidique.
- Inoculer deux tubes de 5 ml de bouillon 2-YT avec 50 pg / ml de kanamycine.
- Cultivez cultures d'une nuit à 37 ° C dans un shaker à 220 rpm.
- Isoler les cultures après une nuit de croissance par centrifugation à 3000 rpm pendant 15 min.
- Utiliser un Qiagen QIAprep Spin Kit Miniprep pour extraire pET28 vecteur de culots bactériens selon les instructions du fabricant.
- Configuration restriction digestions enzymatiques de pET28 extraits plasmide.
- Ajouter 2,2 ul de tampon BamHI 10X et 1 pl de BamHI et HindIII à 20 pi de pET28 vecteur.
- Incuber réaction pendant 1 heure à 37 ° C.
- Produit de digestion séparés sur un gel.
- Reportez-vous à l'étape 2.2.10.
- Couper la bande de vecteur de gel et de purifier à l'aide du kit d'extraction de gel QIAquick selon les instructions du fabricant.
- L'utilisation d'un NanoDrop, de quantifier la concentration de l'ADN.
- Diluer couper vecteur à 10 ng / ul. Conserver à -20 ° C lorsqu'il n'est pas utilisé.
- Préparer amorces RVPC.
- Centrifugeuse IDT oligonucliotides fournis pendant 1 min à 1300 rpm.
- Apportez concentration à 100 microns avec de l'eau DI.
- Préparer 10 uM dilution des deux amorces sens et antisens dans un tube de 1,5 ml.
- Boutique amorces et des dilutions d'amorces à -20 ° C.
- Décongeler Pfu Master Mix sur la glace et dégel matrice et les amorces à la température ambiante.
- réactions RVPC d'installation dans une plaque PCR 96 puits:
- Dans la première rangée d'une plaque de 96 puits combinent 60 pl de deux amorces de RVPC avant et arrière et 24 pl d'digéré pET28 gabarit (10 ng / ul).
- Avec une pipette multicanal 12-tip, transférer 12 pi de l'apprêt et le mélange maître de modèle à rester chaque puits de la plaque. Cela devrait se traduire par 12 pi de l'apprêt et master mix de modèle dans chaque puits de la plaque.
- Ajouter 13 ul d'eau DI à chaque puits.
- Ajouter 25 ul de PFU Master Mix dans chaque puits.
- Vélo les réactions à travers les conditions de PCR utilisées à l'étape 2.2.7.
- Piscine toutes les réactions RVPC dans un tube Falcon de 15 ml.
- Vérifiez amplification de fragments en séparant 10 pi de produit de PCR en commun sur un gel (durée prévue du digéré vecteur pET28est d'environ 6 kb).
- Reportez-vous à l'étape 2.2.10.
- Préparer fusionner plaques.
- Aliquote 3 pi de produit RVPC dans chaque puits d'une plaque à fond en V à 96 puits.
- plaques de conserver à -20 ° C jusqu'à la fusion avec le produit iPCR.
4. Fusionner iPCR et RVPC produits
- produits IPCR de dégel et pré-aliquotée RVPC 96 puits fusionnent plaque à la température ambiante.
- Ajouter 3 pi de chaque produit iPCR à son bien respective de la plaque de fusion.
- Transformez fusionner plaque dans Top Ten des cellules compétentes chimiquement.
- Ajouter 2 pi de chaque réaction de fusion en un seul tube de 50 pi de cellules chimiquement compétentes fournis par le fabricant et procéder avec le protocole fourni par le fabricant.
- Préparer cultures d'une nuit pour chaque construction de la plaque de transformation.
- Aliquote de 5 bouillon de TB ml (avec 50 pg / ml de kanamycine) à partir d'un réservoir stérile ml 25 dans chaque puits d'un bloc de puits profonds.
- Utiliser Sterile technique, vous obtenez une colonie isolée de chaque plaque de transformation et inoculer le bien approprié du bloc puits profond.
- Couvrir le bloc avec une couverture AirPore.
- Secouez bloc à 220 rpm à 37 ° C pendant la nuit.
- Sédimenter les cellules par centrifugation du bloc pendant 30 min à 4000 rpm.
- Décanter le surnageant et tapotez le dessus du bloc sécher avec une serviette en papier.
- Mini-prep utilisant un Qiagen 96 puits appareil à vide selon les instructions du fabricant.
5. Préparation des stocks de glycérol de constructions succès clonés
- Transform réussi à cloner la séquence d'ADN validés dans BL21 (DE3) chimiquement des cellules compétentes conformément aux instructions du fabricant.
- Pour chaque construction, choisissez une seule colonie isolée de l'(DE3) transformation et inoculer BL21 dans 1 ml de 2-YT bouillon (avec 50 pg / ml de kanamycine).
- Secouez cultures à 3-4 heures pour 220 rpm à 37 ° C.
- Étiqueter un 1,5 mlvisser tube de bouchon avec le numéro unique d'identification d'assemblage, de la souche de cellules, et la date. Ajouter 500 ul de 50% de glycérol et 500 pi de culture cellulaire et inverser plusieurs fois. Stocker immédiatement du stock de glycérol sur la glace sèche ou dans un congélateur à -80 ° C.
6. Test d'expression
| Lysis Buffe r | Tampon de lavage | tampon d'élution |
| 50 mM de NaH 2 PO 4 à pH 8,0 NaCl 300 mM 10 mM d'imidazole 1% de Tween 20 MgCl2 2 mM 0,1 pl / ml benzonase 1 mg / ml de lysozyme | 50 mM de NaH 2 PO 4 à pH 8,0 NaCl 300 mM 20 mM d'imidazole 0,05% de Tween 20 | Tris 25 mM, pH 8,0 NaCl 300 mM 250 mM imidazole 0,05% de Tween 20 |
* Ajouter benzonase, lysozyme,et l'inhibiteur de la protéase immédiatement avant la lyse.
- Morts à un échantillon à partir du stock de glycérol sur la kanamycine agar sélectif et incuber une nuit à 37 ° C.
- Démarrer une pré-culture non inducteur dans un bloc de 96 puits à fond rond; inoculer 1,2 ml de bouillon de TB (avec 50 mg / ml de kanamycine) complémenté avec 0,5% de glucose avec un E. fraîchement cultivés coli isolent. Croître pendant la nuit en agitant à 220 rpm à 37 ° C.
- Après une nuit de croissance, commencer cultures d'induction en inoculant 1,2 ml de bouillon de TB (avec 50 mg / ml de kanamycine) supplémenté avec Novagen Overnight Express System 1 (selon le protocole du fabricant) avec 40 pl de la pré-culture.
- Cultiver les cultures d'induction à petite échelle à 20 ° C pendant 48 heures, en agitant à 220 rpm.
- Récolte des cellules par centrifugation à 4000 rpm pendant 15 min, verser le surnageant et conserver à -20 ° C pendant au moins 1 heure avant le traitement.
- Dans le bloc 96 puits, remettre en suspension les culots cellulaires dans un tampon de lyse 300 pi.
- Incuber les cellules dans un tampon de lyse à la température ambiante pendant 30 min suivie d'une lyse mécanique par agitation vigoureuse pendant 30 minutes à température ambiante.
- Clarifier le lysat brut par centrifugation pendant 30 min à 4000 rpm à 4 ° C.
- Utiliser une pipette multi-canal pour transférer 200 pl du lysat clarifié (fraction soluble) à un plateau de 96 puits à fond plat (Qiagen). Pour chaque puits contenant un échantillon, ajouter 40 billes Ni-NTA magnétiques ul (Qiagen).
- Agiter doucement la plaque sur une bascule pendant 1 heure à 16 ° C.
- Placer la plaque sur une plaque de poste magnétique (Qiagen) et enlever la fraction soluble non lié. Prenez soin de ne pas la pipette sur une des perles de Ni-NTA.
- Retirer la plaque de la plaque de poste et remis en suspension doucement les perles dans un tampon de lavage 200 pi. Introduire à la pipette de haut en bas pendant 30 secondes, puis placez la plaque arrière sur la plaque poste.
- Retirez le tampon de lavage et répétez l'étape 6.12.
- Retirer la plaque de la plaque de poste et éluer le Ni-NTA lié TAprotéines rget par lavage avec 50 tampon d'élution ul pendant 5 min.
- Revenir plaque de fond plat à la plaque magnétique de poste et de les transférer à l'élution d'une plaque à fond en V à 96 puits fraîche.
- Transférer 20 ul de l'élution sur une plaque de v-bas frais de 96 puits et de réagir avec 1 pl Ulp1 protéase.
- Selon le protocole du fabricant, d'analyser le Ulp1 fraction élue et élue + par électrophorèse capillaire en utilisant un LabChip 90.
- Sinon, toutes les fractions du test d'expression peuvent être analysées par SDS-PAGE.
7. Grand fermentation à l'échelle
- Utiliser un embout de pipette stérile pour obtenir une égratignure d'un stock de glycérol, inoculer 100 ml de bouillon de TB (avec 50 mg / ml de kanamycine) et de croître pendant la nuit. Agiter à 220 tpm et 37 ° C.
- Après une nuit de croissance, développez pré-culture en inoculant 1 L de bouillon de TB avec des solutions auto-induction EMD (voir le protocole du fabricant) (avec 50 mg / ml de kanamycine) dans un flacon dérouté 2 avec 10 ml de lapré-culture (dilution 1:100).
- Agiter les cultures expansées 1 L à 37 ° C; changer la température de l'incubateur agité à 20 ° C lorsque la densité optique de 0,6 (DO 600) est atteinte.
- Après une nuit de croissance, jetez un représentant aliquote de 10 ml de chaque construction pour les tests d'expression.
- cellule de récolte coller par centrifugation à 5000 rpm pendant 15 min et éliminer le surnageant.
- cellule de congélation coller à -80 ° C.
EPURATION DE PROTÉINES
Tampons:
| tampon de lyse | Tampon A (équilibration) | Buffer B (élution) | Dimensionnement tampon de colonne |
| Tris 25 mM, pH 8,0 NaCl 200 mM 0,5% Glycérine 0,02% de CHAPS 10 mM d'imidazole 1 mM PTCE 50 mM d'arginine 5 benzonase ul À 100 mg lysozyme 3 comprimés inhibiteurs de la protéase (sans EDTA) | Tris 25 mM, pH 8,0 NaCl 200 mM 10 mM d'imidazole 1 mM PTCE 50 mM d'arginine 0,25% Glycérine | Tris 25 mM, pH 8,0 NaCl 200 mM 200 mM d'imidazole 1 mM PTCE | Tris 25 mM, pH 8,0 NaCl 200 mM 1% Glycérine 1 mM PTCE |
* Ajouter benzonase, lysozyme, et des comprimés inhibiteurs de la protéase à chaque échantillon de 150 ml immédiatement avant la lyse.
8. lyse cellulaire
- Faire 2 L de tampon de lyse, ne pas ajouter lysozyme, comprimés inhibiteurs de la protéase ou benzonase (chaque échantillon sera lysée séparément dans 150 ml de tampon de lyse).
- cellule de dégel et remettre en suspension coller dans un tampon de lyse à une masse 1:5 ratio de volume en agitant vigoureusement pendant 30 min à 4 ° C. Cassez les morceaux en vrac des côtés du récipient à l'aide d'une spatule propre. Pendant cette période de temps préparer Ni et DialyTampons sis
- Sur la glace, la lyse des cellules en utilisant un appareil à ultrasons Misonix (70% de puissance, 2 sec on / off 1 sec impulsions pendant 3 min) et doucement conteneur de turbulence pour éviter la surchauffe. Enregistrer une petite (200 pl) aliquote de lysat brut pour une analyse ultérieure.
- Clarifier le lysat brut par centrifugation à 18.000 g pendant 35 min à 4 ° C, recueillir le surnageant et sauver un petit (200 pi) aliquote pour une analyse ultérieure. Magasin granulés à 4 ° C jusqu'à ce qu'il soit confirmé la protéine a été lysées dans la fraction soluble.
9. Setup Maker Protéines Pre-run
- Avec le fabricant de protéines allumé et le logiciel ouvert, initialiser l'instrument.
- Une fois initialisé, fixez un 5,0 ml GE HisTrap colonne Nickel-chélate FF Healthcare (colonne Ni) sur une ligne distincte du portique pour chacun des échantillons.
- Exécuter volumes de colonne 3-4 (CV) de tampon d'équilibrage à travers chaque colonne.
- Premier l'équilibrage et les lignes de tampon d'élution.
- Equilibrmangé les colonnes en aspirant tampon A travers la colonne une fois.
10. Nickel 1 (Ni1) Colonne
- Laver chaque colonne avec 20 ml d'eau Milli-Q pour éliminer le tampon de stockage. Exécuter 5 ml de tampon B et 25 ml de tampon A pour atteindre l'équilibre.
- Chargez le lysat clarifié contenant la protéine solubilisée dans les colonnes à un taux de 2 ml / min puis suivre par un lavage à 15 ml avec le tampon A.
- On élue la protéine liée à un gradient progressif avec des tampons A et B par les ratios suivants respectueusement: 5 ml 95:5, 60:40 5 ml, 10 ml 0:100. Recueillir chaque fraction d'élution séparément.
- Analyser: fractions éluées, lysat brut, lysat clarifié et écoulement continu par SDS-PAGE. fractions de piscine contenant de la protéine et en utilisant un Nanodrop pour mesurer A 280 pour déterminer approximativement la quantité de protéine présente.
11. Ulp1 Clivage
- Gardez une petite aliquote (250 pi) de la piscine Ni1 de colonne pour l'analyse de gel ultérieur. Apportez le restede la piscine Ni1 à 10 ml et ajouter ubiquitine protéase 1 (Ulp1) à 1 pl / 5 mg de protéine totale pour enlever l'étiquette d'affinité His-Smt.
- Dialyser la piscine Ni1 + Ulp1 contre 2 L de tampon A pendant 4 heures à 4 ° C en 10 kDa de coupure de poids moléculaire (MWCO) sur une plaque d'agitation à 4 ° C.
- Après dialyse, exécutez SDS-PAGE de Ni1 piscine et Ni1 piscine + Ulp1 pour déterminer si Ulp1 clivage a réussi.
12. Nickel 2 (Ni2) Colonne
- Charger protéine clivée au cours de la même colonne de Ni et répéter les étapes 9.3 à une vitesse d'écoulement réduite de 1 ml / min. La balise hors clivée se lie à la colonne et la protéine cible tagless va maintenant Flow-Through. Recueillir le flux continu dans un récipient frais.
- Laver la colonne Ni avec un tampon de 3 ml Un suivi par 5 ml de tampon B pour éluer toute sa protéine marquée et non spécifiquement lié. Recueillir chaque fraction séparément.
- Exécuter SDS-PAGE de Ni2 accréditives, laver et Ni2 fractions d'élution pour vérifier Ulp1 clivage et qui protein est présent dans l'effluent. Utilisation d'un Nanodrop pour mesurer A 280 pour déterminer approximativement la présence de la protéine.
13. Concentrant
- Concentrer le flux continu Ni2 (et Ni2 élution si la protéine est présente) à 5 ml avec un Ultra 10 kDa tube de centrifugeuse MWCO Amicon. Tourner dans des intervalles de 10 min à 4000 rpm à 4 ° C. Mélanger avec une pipette entre chaque tour de roue pour empêcher la protéine de trop se concentrer sur la membrane.
14. Chromatographie d'exclusion stérique (SEC)
- Mettre en place un S-100 10/30 GL colonne Sephacryl (GE Healthcare) en équilibrant avec un tampon SEC 200 ml à un débit de 0,5 ml / min sur un système AKTApurifier (GE Healthcare).
- Préparer superliaisons 10 ml pour une utilisation sur la colonne SEC conformément aux instructions du fabricant.
- En utilisant une seringue de 5 ml, charger les échantillons sur superliaisons et commencer la course SEC.
- Surveiller la trace absorbance UV à 280 nm tout en collectant petit volume fractions.
- Exécuter fractions SEC par SDS-PAGE.
- Mutualiser les fractions SEC montrant les bandes d'intensité les plus élevés.
- Concentrez fractions SEC communs. Reportez-vous à l'étape 13.1.
- protéines aliquote dans 100 échantillons ul, gel rapide dans l'azote liquide et conserver à -80 ° C.
CRISTALLISATION
15. la cristallisation des protéines
- Pré-remplir chaque réservoir d'une plaque de cristallisation 96 puits Compact Jr (Emerald Bio) avec 80 pl de l'écran de cristallisation (Emerald Bio) de son choix.
- Diluer protéine avec dimensionnement tampon à 2-20 mg / ml et conserver dans la glace.
- Verser 0,4 l de protéines et 0,4 ul de l'écran de cristallisation dans chacun des 96 puits et couvrir avec du ruban d'étanchéité cristalline (Manco).
- Conservez la plaque à 16 ° C tout en vérifiant pour la cristallisation des protéines périodiquement au cours des prochaines semaines sous un microscope à dissection.
16. Cristal récolte
- Créer un cryoprotecteur à partir de la liqueur mère et de l'éthylène glycol. Couper le ruban transparent qui recouvre le puits avec le cristal de protéine cible. Pour un puits vide, ajouter 1,6 ul de la condition de cristallisation correspondante et se combiner avec 0,4 l de l'éthylène-glycol qui donne une concentration finale de 20% d'éthylène glycol et 80% de cristallisation état. Remarque: pour optimiser diffraction cristal essayer différents cryoprotecteurs tels que le glycérol, les huiles, les polyéthylène glycols MW faibles, et / ou à des pourcentages de la cryoprotectant variable.
- Avant de récolter refroidir une rondelle ALS-style dans un vase de Dewar rempli d'azote liquide et fermer le couvercle.
- Récolter le cristal en plaçant une boucle cryogénique avec le diamètre intérieur correspondant à la taille du cristal sur une baguette de cristal magnétique (Hampton Research) et enlever le directement à partir de la solution ainsi.
- Plonger immédiatement la boucle cryogénique avec le cristal récolté dans le puis plongez cryoprotecteur dans la rondelle ALS-style à clignoter geler le crystal. Répéter pour un nombre souhaité de cristaux.
17. Cristal dépistage / Collecte de données
- Une fois la récolte terminée utiliser une baguette rondelle à placer le couvercle rondelle cryo magnétique sur la SLA rondelle. Avec des pinces coudées, retourner la rondelle à l'envers.
- Transférer la rondelle à un Rigaku ACTEUR Dewar, visser une Pusher Puck sur la rondelle, et percer le couvercle laissant dans le vase de Dewar avec des épingles face.
- En utilisant le logiciel JDirector, écran chaque cristal sous les paramètres suivants: fente de faisceau fixé à 0,5 degrés, la distance du détecteur fixé à 50 mm, l'étape d'image à 70 degrés, et la longueur d'exposition réglé à 30 sec.
- Exécuter MOSFLM sur les images de test que vous avez prises avec JDirector pour déterminer quelle est la meilleure stratégie est de cristal et de collecte de données.
- Recueillir un ensemble de données complet basé sur vos résultats de MOSFLM.
18. Traitement de données / Détermination de la structure
- Exécuter XDS / XSCALE 4 à traiter l'ensemble des données.
- Ouvrez le logiciel de bain CCP4.
- Exécuter Phaser 5 pour calculer une solution de remplacement moléculaire en utilisant un modèle de recherche d'homologie élevé, lorsqu'il y en a. Dans ce cas, nous avons utilisé le 3CW4 PDBID comme un modèle de recherche 6.
- Exécuter Refmac 7 pour affiner votre modèle moléculaire contre la réflexion observée recueillies dans l'ensemble de données. Résolution finale devrait être basé hors de la coquille et plus déterminée par les paramètres suivants: R facteur> 50%, I / sigma> 2, et l'exhaustivité> 90%.
- Construire un modèle de densité électronique en 3 dimensions avec le logiciel graphique moléculaire COOT 8.
- Avant de déposer la structure de l'APB valider avec le logiciel de MolProbity 9 pour vérifier que la qualité de la structure est appropriée pour le dépôt.
Résultats
Les résultats suivants illustrent les résultats attendus du protocole décrit, et dans le cas de PB2, les résultats observés.
Utiliser Gene Compositeur, cinq séquences d'acides aminés cibles pleine longueur de la sous-unité PB2 polymérase du virus de l'influenza ont été conçus (Figure 2). Les séquences PB2 ont été traduits en arrière et soumis à de nombreuses étapes d'ingénierie 3, entraînant des codons séquences harmonisées optimisés pour l'expression dans E. coli. des produits IPCR (Figure 3b), un total de trente-quatre constructions ont été clonés avec succès dans un système de vecteur pET28 10 modifié avec un N-terminal 6x His-Smt tag de fusion en utilisant PIPE clonage 3 comme indiqué sur la Figure 3a. Un résumé des flux de clonage est présenté dans la Figure 4.
Après le succès du clonage, l'expression des protéines micro-échelle de chaque construction a été testé dans BL21 (DE3) E.cellules de E. coli. Les cellules ont été cultivées dans un milieu de TB complétée par Novagen Nuit 1 moyen Express (selon le protocole du fabricant) de 48 h à 20 ° C dans un incubateur réglé agitation à 220 rpm. Après la croissance, les cellules ont été récoltées et testées pour l'expression de la protéine soluble utilisant électrophorèse capillaire avec un étrier LabChip 90. Quatorze des trente-quatre PB2 constructions ont conduit à la protéine cible soluble et est entré fermentation à grande échelle. Cultures à grande échelle de chaque construction ont été cultivées dans un milieu de TB complété avec Nuit 1 moyen Express Novagen selon le protocole du fabricant. Après la croissance, les cellules ont été récoltées par centrifugation et stockés à -80 ° C. Expression de protéines à grande échelle de chaque culture a été confirmée par une analyse SDS-PAGE (figure 5) avant de procéder à la purification à grande échelle.
La protéine Maker a été utilisé pour effectuer la purification parallèle des quatorze constructions PB2. Les lysats clarifiés d'all quatorze constructions ont été réalisées à travers une colonne nickel-chélate. Après avoir déterminé les fractions contenant la protéine cible par SDS-PAGE, les fractions correspondantes ont été regroupées pour chaque échantillon et la concentration de chacun a été déterminé par une lecture A280. Retrait de l'étiquette His-Smt 6x a été menée par l'ajout de Ulp1 suivie d'une dialyse pendant la nuit et une deuxième colonne de nickel. Confirmation de la suppression de l'étiquette His-Smt a été menée par SDS-PAGE (figure 6), et chaque échantillon a été concentré avec une kDa tube à centrifuger Amicon Ultra 10. Après concentration à l'aide des tubes à centrifuger Amicon Ultra, chaque échantillon a été effectuée sur une colonne dimensionnement pour atteindre la pureté cristallographique. Une seconde concentration a été réalisée pour augmenter la concentration en protéine à un niveau nécessaire pour la cristallisation. Les quatorze constructions ont été purifiés avec succès et inclus dans des essais de cristallisation.
La cristallisation a été initié par le dégel précédemment frprotéines ozen. La cristallisation a été effectuée dans une pièce climatisée à 16 ° C avec des plaques spécialement conçues (Emerald Bio) pour s'asseoir baisse de la diffusion de vapeur (Figure 7). Sélection initiale a été menée avec quatre écrans à matrice clairsemée; JCSG +, pacte, magicien plein, et CryoFull (Emerald Bio), suivant une stratégie Newman prolongée. 0,4 pi de solution de protéines a ensuite été mélangé avec 0,4 l de crystallant (ou solution de réservoir) du réservoir correspondant en utilisant des plaques de cristallisation Jr 96 puits compacts (Emerald Bio). Sur les quatorze échantillons purifiés neuf d'entre eux a donné des cristaux appropriés pour les études de diffraction (figure 8). Un ensemble de données diffraction en interne ont été recueillies sur cinq des neuf constructions cristallisées au Cu Ka longueur d'onde en utilisant un générateur Rigaku SuperBright FR-E + anode tournante de rayons X équipé d'optiques Osmic VariMAX HF et un 944 détecteur Saturn + CCD (Figure 9 ). Chaque jeu de données a été traitée avec XDS / XSCALE 4 < / Sup> et redimensionnée à une résolution finale. Les tentatives pour résoudre les structures de remplacement moléculaire ont été réalisées avec Phaser 5 de la CCP4 Suite 7. Les modèles finaux ont été obtenus après raffinement dans REFMAC 7 et reconstruction manuelle avec Foulque 11. Les structures ont été évaluées et corrigées pour la géométrie et de remise en forme avec MolProbity 9. Un total de quatre structures de la sous-unité PB2 ont été déterminés (Figure 10) et déposé dans l'APB. Figure 11 illustre le résultat global à chaque étape du pipeline MTPP.

Figure 1. Vue d'ensemble du gène à la structure SSGCID voie pour le traitement parallèle multi-cibles à Emerald Bio.
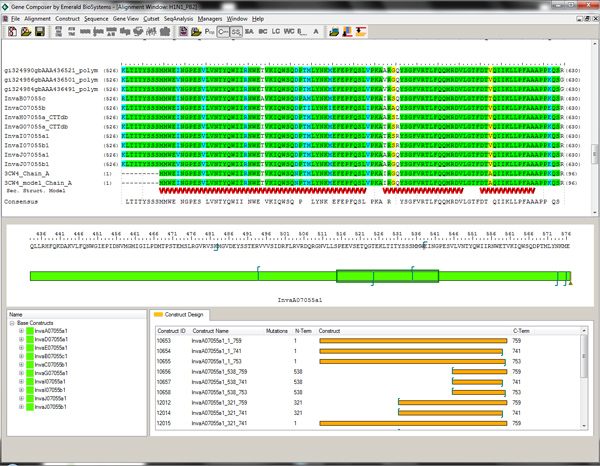
Figure 2. Visualisation de l'alignement et la protéine selon module de conception dans les logiciels de compositeur Gene. La base d'acides aminés construire de cible est représenté en vert (fenêtre du milieu) et la structure troncatures guidées de constructions alternatives sont indiquées en or (fenêtre du bas). Un alignement de plusieurs PB2 séquences virales de la grippe est représentée par rapport à la séquence et les éléments de structure secondaire du domaine C-terminal de 3CW4 PDBID. La connaissance de la structure de domaine et des éléments de structure secondaire permet troncatures N-terminaux à être choisis au sein de l'Composer Conception Module de Gene par un clic droit sur le résidu d'acide aminé souhaité. Cliquez ici pour agrandir la figure .
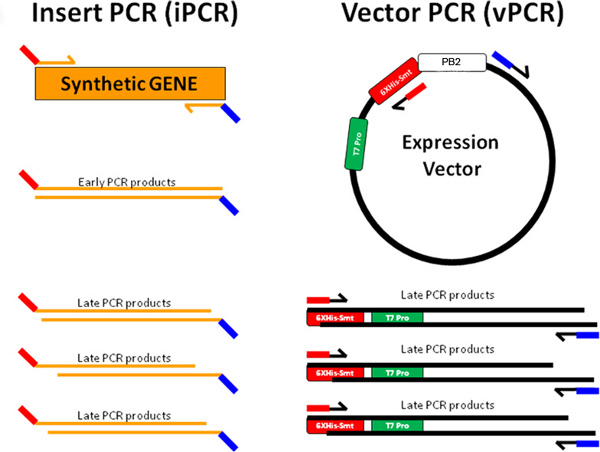
Figure 3a. PIPE clonage est illustré dans lequel l'insert de gène synthétique (orange) est amplifié par conçus avant (lignes rouge-orange) et inverser amorces (lignes orange bleu) pour générer des instructions INSERT matériau PCR. Le vecteur d'expression est amplifiée avec des lignes arrière (rouge-noir ) et à terme (lignes bleu-noir) pour générer des amorces PCR matériel vecteur. Les séquences des produits de IPCR terminaux sont complémentaires aux séquences terminales des produits RVPC (rouge de iPCR complète de RVPC rouge et bleu de iPCR compléments bleu de RVPC). Ceci permet aux produits de IPCR et RVPC pour recuire pour former des plasmides qui sont répliquées après transformation dans l'hôte BL21 (DE3) E. chimiquement compétente cellules de E. coli.

Figure 3b. Analyse sur gel d'agarose de iPCR production ts de la sous-unité PB2. échecs IPCR peuvent être considérées comme faibles bandes ou graisseux, tandis que les produits IPCR réussies sont représentés par des bandes robustes. la qualité du produit iPCR peut généralement être corrélée avec la réussite du clonage. Des marqueurs de poids moléculaires sont en kilodaltons. Figure est reproduit à partir de Raymond et al., 2011 12.

Figure 4. Gene étapes d'ingénierie de cible PB2 de protéines ont été effectuées en utilisant un logiciel de compositeur Gene. Après la séquence d'acide nucléique d'ingénierie a été établi pour chaque cible, les constructions alternatives de protéines 6-7 ont été conçus pour chacun. Traitement parallèle multi-cibles dans les étapes initiales de la conception des gènes et le clonage a donné lieu à 34 constructions, dont 14 étaient des cibles viables qui produisent des protéines solubles dans E. coli.
re 5 "src =" / files/ftp_upload/4225/4225fig5.jpg "/>
Figure 5. Représentant analyse SDS-PAGE de fermentation à grande échelle montrant l'expression de la protéine robuste (taille attendue de 25,76 kDa), environ 50% soluble (piste 4) et environ 50% clivage de tag 6x His-Smt de protéine éluée (piste 7).
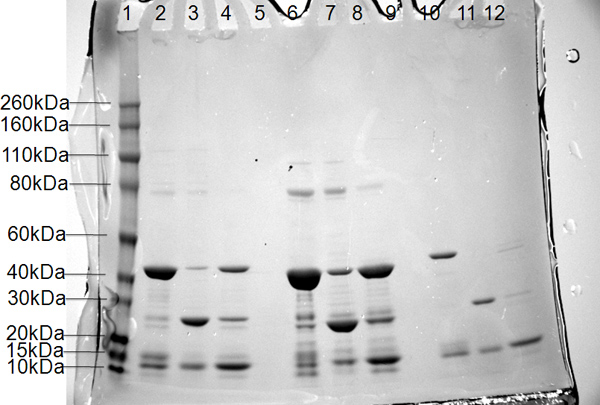
Figure 6. Résultats SDS-PAGE pour trois constructions de la polymérase sous-unité PB2 Lane 1, les marqueurs de poids moléculaire (étiqueté à gauche en kDa);. Pistes 2, 6 et 10, les protéines commun de Nickel colonne 1, lignes 3, 7 et 11, dynamique de la protéine clivée dans le tampon A partir Nickel 2, lignes 4, 8 et 12, le retrait de l'étiquette 6x His-Smt dans le tampon B de Nickel 2.
d/4225/4225fig7.jpg "/>
Figure 7. Un schéma de diffusion de la vapeur par la méthode de la goutte assise. L'méthode de la goutte assise pour la cristallisation des protéines tombe dans la catégorie de la diffusion de vapeur. Cette méthode est basée sur un échantillon purifié de la protéine et précipitant à l'équilibre avec un réservoir contenant de plus des conditions similaires dans une concentration plus élevée. Comme l'eau se vaporise à partir de l'échantillon des protéines et des transferts vers le réservoir, la concentration en agent précipitant augmente à un niveau optimal pour la cristallisation de la protéine.

Figure 8. cristaux de protéine de sous-unité de polymérase PB2 à partir d'une souche du virus de la grippe.

Figure 9. L'image de diffraction aux rayons X de la polymérase PB2 sous-unité à partir d'unla souche du virus de la grippe.

Figure 10. Diagrammes de ruban des molécules dans l'unité asymétrique cristallographique de 4 PB2 structures. Des structures secondaires de couleur dans le motif arc en ciel avec des codes PDB correspondant. (A) 3K2V (A/Yokohama/2017/2003/H3N2) (b) 3KHW (A / Mexique / InDRE4487/2009/H1N1) (c) 3KC6 (A/Vietnam/1203/2004/H5N1) (d) 3L56 (A/Vietnam/1203/2004/H5N1).

Figure 11. analyse des résultats pour la grippe PB2 cibles par les méthodes décrites. L'structure pipeline de détermination est illustrée en cinq étapes: détermination clonage, la solubilité, la purification, la cristallisation et la structure.
Discussion
Multi-Target Parallel Processing
Structure basée sur la conception de médicaments joue un rôle important dans la découverte de médicaments. Le SSGCID se consacre à fournir à la communauté scientifique des structures protéiques de trois dimensions à partir de NIAID catégorie pathogènes AC. Rendre cette information structurelle largement disponibles finira par servir à accélérer la structure basée sur la conception de médicaments.
La première étape critique de l'approche MTPP est de conception construction. Constructions multiples de chaque protéine cible augmente la probabilité de succès de détermination de la structure et des augmentations redressement. Il est inévitable que certaines constructions de protéines vont échouer au cours des étapes du pipeline. La mise en œuvre de la méthode de clonage PIPE en charge la méthode MTPP en permettant la génération de nombreuses constructions en format 96 puits sans travail des étapes de purification intensive. Jumelage clonage PIPE avec la capacité d'analyser l'expression des protéines dans le même format de 96 puits (Étrier LabChip 90) accélère encore le flux global. Le jumelage de ces méthodes permet d'identifier rapidement des constructions qui produisent des protéines solubles qui assure le succès de la production de protéines à grande échelle et de purification.
Un aspect essentiel à la réussite de la MTPP haut débit est le Créateur de protéines (brevet américain n ° 6,818,060, Emerald Bio) instrument. La protéine Maker est un système de chromatographie liquide parallèle à 24 canaux spécialement conçu pour accroître l'efficacité de la production de protéines à haut débit et des applications de recherche de pipelines génomique structurale connexes. Selon le protocole décrit précédemment pour la protéine Maker, les avantages sont évidents par rapport à un système de FPLC sur une seule ligne. Une seule personne peut purifier jusqu'à 48 cibles en parallèle dans un délai de huit heures. En revanche, une seule personne utilisant un système FPLC sur une seule ligne ne peut purifier un maximum de quatre cibles dans le même délai. Les hauts niveaux de pureté pour chaque cibleréalisé avec la protéine Maker est un facteur critique dans la réussite future de la cristallisation des protéines pour l'analyse de la structure.
Limitations et dépannage
Résoudre des structures tridimensionnelles par cristallographie aux rayons X est un effort multi-étagée avec de nombreux défis, dont l'un est l'incapacité d'obtenir de grandes quantités de protéine cible soluble. Une stratégie qui peut être mis en œuvre pour résoudre le problème de solubilité est l'utilisation d'un hôte d'expression alternatif comme E. cellules de E. coli sont incapables de réaliser plusieurs importantes modifications post-traductionnelles eucaryotes. Expression dans diverses levures, lignées de cellules d'insectes et de mammifères qui sont capables d'effectuer ces modifications post-traductionnelles sont souvent une alternative appropriée. protéines cibles sont parfois exprimées, mais totalement insoluble dans les conditions de lyse standard. La protéine Maker peut être une ressource précieuse pour le dépistage rapide des conditions de lyse cellulaire alternativescomme décrit dans Smith et al. 2011 13. Cette stratégie est souvent nécessaire de maintenir des cibles se déplaçant à travers le pipeline. En tout pipeline génomique structurale, protocoles standardisés peuvent ne pas convenir à toutes les cibles qui vient à travers le pipeline et les objectifs peuvent nécessiter une optimisation individuelle. Par exemple, nous avons choisi d'utiliser 20% d'éthylène glycol pour chaque cryoprotecteur. Dans les cas que cette condition n'est pas approprié, cryoprotectants ou les concentrations d'autres peuvent avoir besoin d'être testé.
En raison de la nature unique de chaque protéine cible individuelle, limitant la vitesse et imprévisible étape dans la détermination de la structure est la cristallisation. Les décalages de pipelines MTPP le taux de réussite généralement faible de cristallisation des protéines avec l'optimisation des écrans à matrice clairsemée initiales. Chaque cristal initial frapper des écrans de matrices creuses disponibles dans le commerce est optimisée avec un constructeur E-Screen (Emerald Bio). L'écran d'optimisation est conçu arolide l'état du tube en cristal initiale, modifiant les concentrations des tampons, des sels, et les additifs. Écrans optimisation réussie donner des cristaux appropriés pour les études de diffraction et de la détermination de la structure.
Le programme de génomique structurale mis de l'avant par l'Institut national des allergies et maladies infectieuses (NIAID) fournit du financement à Emerald Bio et trois autres institutions du Nord-Ouest du Pacifique qui sont ensemble le SSGCID (Emerald Bio, SeattleBiomed, l'Université de Washington et Pacific Northwest National Laboratory) . Chaque membre du consortium a été choisi pour son expertise dans l'application des technologies state-of-the-art nécessaires à la réalisation des objectifs du programme de génomique structurale NIAID. À ce jour, SSGCID a déposé 461 structures dans le classement APB comme le septième plus grand contributeur dans le monde, et en 2011, la plus productive. Les protocoles et méthodologies du SSGCID sont fournis avec l'intention de bénéficier de lacommunauté scientifique et la perpétuation de la recherche sur les maladies infectieuses.
Déclarations de divulgation
Les auteurs sont des employés de Emerald Bio, Inc.
Remerciements
Les auteurs tiennent à remercier tous les membres du consortium SSGCID. Réalisation des objectifs de la SSGCID est rendue possible par les efforts considérables déployés par tous les membres de l'équipe à Emerald Bio. Cette recherche a été financée au titre du contrat fédéral n ° HHSN272200700057C de l'Institut national des allergies et des maladies infectieuses, les Instituts de la Santé et le ministère de la Santé et des Services sociaux nationale.
matériels
| Name | Company | Catalog Number | Comments |
| Primers | IDT | ||
| Genes | DNA 2.0 | ||
| TE buffer | Qiagen | provided in kit | |
| 96-well half skirt PCR plates | VWR | 10011-248 | |
| PFU Master Mix | |||
| 6X Orange Loading Dye | Fermentas | R0631 | |
| 10X TAE | Teknova | T0280 | |
| Agarose | Sigma-Aldrich | A9414-10G | |
| pET28 vector | |||
| 2-YT Broth | VWR | 101446-848 | |
| Kanamycin | Teknova | K2151 | |
| Restriction Enzymes | Fermentas | ||
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Top 10 chemically comp cells | Invitrogen | C4040-06 | |
| Disposable Troughs (Sterile, 25 ml) | VWR | 89094-662 | |
| Airpore covers (Rayon films for bio cultures) | VWR | 60941-086 | |
| 24-well blocks | VWR | 13503-188 | |
| QIAvac 96 | Qiagen | 19504 | |
| BL21(DE3) cells chemcomp (phageR) | NEB | C2527H | |
| 50% Glycerol | VWR | 100217-622 | |
| TB Media | Teknova | T7060 | |
| IPTG | Sigma-Aldrich | ||
| 1 M Tris pH 8.0 | Mediatech | 46-031-CM | |
| 5 M NaCl | Teknova | S0251 | |
| Glycerol | Aldrich | G7893-4L | |
| CHAPS | JT Baker | 4145-01 | |
| Imidazole | Sigma | 56749-1KG | |
| TCEP | Amresco | K831-10G | |
| L-arginine | Amresco | 0877-500G | |
| Benzonase | EMD | 70746-3 | |
| Lysozyme | USB | 1864525GM | |
| 10 kDa MWCO dialysis tubing | Thermo | 68100 | |
| Amicon Ultra 10 ka MWCO concentrators | Millipore | UFC901024 | |
| HisTrap FF columns | GE | 17-5255-01 | |
| HiTrap Chelating columns | GE | 17/0408-01 | |
| Compact Jr crystallization plates | Emerald Bio | EBS-XJR | |
| Crystalization screens | Emerald Bio | ||
| Ethylene Glycol 100% | Emerald Bio | EBS-250-EGLY | |
| Crystal Wand Magnetic Straight | Hampton Research | HR4-729 | |
| Mounted CryoLoop 0.1-0.2 mm | Hampton Research | HR4-955 | |
| ALS style puck | |||
| Puck Wand | |||
| Bent Tongs | |||
| Puck Pusher |
Références
- Lowen, A. C., Mubareka, S., Steel, J., Palese, P. Influenza virus transmission is dependent on relative humidity and temperature. PLoS Pathogens. 3 (10), 1470-1476 (2007).
- Yamada, S., et al. Biological and structural characterization of a host-adapting amino acid in influenza virus. PLoS Pathog. 6, e1001034 (2010).
- Lorimer, D., Raymond, A., Walchli, J., Mixon, M., Barrow, A., Wallace, E., Grice, R., Burgin, A., Gene Stewart, L. Composer: database software for protein construct design, codon engineering, and gene synthesis. BMC Biotechnol. 9, 36 (2009).
- Kabsch, W. Integration, scaling, space-group assignment and post-refinement. Acta Cryst. D. 66, 125-132 (2010).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. J. Phaser crystallographic software. Appl. Cryst. 40, 658-674 (2007).
- Kuzuhara, T., Kise, D., et al. Structural basis of the influenza A virus RNA polymerase PB2 RNA-binding domain containing the pathogenicity-determinant lysine 627 residue. J. Biol. Chem. 284, 6855-6860 (2009).
- Murshudov, G. N., Skubàk, P., Lebedev, A. A., Pannu, N. S., Steiner, R. A., Nicholls, R. A., Winn, M. D., Long, F., Vagin, A. A. REFMAC5 for the refinement of macromolecular crystal structures. Acta Cryst. D. 67, 355-367 (2011).
- Cowtan, K. Recent developments in classical density modification. Acta Cryst. D. 66, 470-478 (2010).
- Chen, V. B., Arendall, W. B., Headd, J. J., Keedy, D. A., Immormino, R. M., Kapral, G. J., Murray, L. W., Richardson, J. S., Richardson, D. C. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Cryst. D. 66, 12-21 (2010).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol. Cell. 5, 865-876 (2000).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Cryst. D. 66, 486-501 (2010).
- Raymond, A. C., Haffner, T. E., Ng, N., Lorimer, D., Staker, B. L., Stewart, L. J. Gene design, cloning and protein-expression methods for high-value targets at the Seattle Structural Genomics Center for Infectious Disease. Acta Cryst. F. 67, 992-997 (2011).
- Smith, E. R., Begley, D. W., Anderson, V., Raymond, A. C., Haffner, T. E., Robinson, J. I., Edwards, T. E., Duncan, N., Gerdts, C. J., Mixon, M. B., Nollert, P., Staker, B. L., Stewart, L. J. The Protein Maker: an automated system for high-throughput parallel purification. Acta Cryst. F. 67, 1015-1021 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon