Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un outil analytique qui quantifie les changements de morphologie cellulaire en trois dimensions des images de fluorescence
Dans cet article
Résumé
Nous avons développé une plate-forme logicielle qui utilise Imaris Neuroscience, ImarisXT et MATLAB pour mesurer les changements dans la morphologie d'une forme indéfinie pris trois dimensions fluorescence confocale de cellules individuelles. Cette nouvelle approche peut être utilisée pour quantifier les changements dans la forme des cellules qui suivent l'activation du récepteur et représente donc un outil possible supplémentaire pour la découverte de médicaments.
Résumé
Les outils les plus courants de logiciels d'analyse disponibles pour mesurer images de fluorescence sont des données en deux dimensions (2D) qui s'appuient sur des réglages manuels pour l'inclusion et l'exclusion des points de données, et la reconnaissance des formes assistée par ordinateur pour appuyer l'interprétation et les conclusions de l'analyse. Il est devenu de plus en plus important de pouvoir mesurer images de fluorescence construits à partir d'ensembles de données tridimensionnelles (3D) afin d'être en mesure de saisir la complexité de la dynamique cellulaire et comprendre les bases de la plasticité cellulaire dans les systèmes biologiques. Instruments de microscopie sophistiquées ont permis la visualisation des images 3D de fluorescence grâce à l'acquisition d'images multispectrales de fluorescence et puissant logiciel d'analyse qui reconstitue les images des cheminées à foyer commun qui fournissent ensuite une représentation en 3D des images collectées en 2D. Méthodes avancées de conception basée sur stéréologie ont progressé de l'approximation et hypothèses de l'original à base de modèle stéréologie 1, même dans des coupes tissulaires complexes 2. En dépit de ces avancées scientifiques en microscopie, il reste un besoin pour une méthode automatisée d'analyse qui exploite pleinement les données intrinsèques 3D pour permettre l'analyse et la quantification des changements complexes de la morphologie cellulaire, la localisation des protéines et le trafic des récepteurs.
Les techniques actuelles pour quantifier les images de fluorescence sont méta-Morph (Molecular Devices, Sunnyvale, CA) et Image J (NIH), qui fournissent une analyse manuelle. Imaris (Andor Technology, Belfast, Irlande du Nord) logiciel fournit la fonctionnalité, MeasurementPro qui permet la création manuelle de points de mesure qui peuvent être placés dans une image de volume ou tiré sur une série de coupes 2D pour créer un objet 3D. Cette méthode est utile pour des mesures ponctuelles seul clic pour mesurer une distance en ligne entre deux objets ou pour créer un polygone qui entoure une région d'intérêt, mais il est difficile d'appliquer à complex structures du réseau cellulaire. Tracer filament (Andor) permet la détection automatique de la 3D neuronale de type filament cependant, ce module a été développé pour mesurer les structures définies comme les neurones, qui sont constitués de dendrites, axones et des épines (structure arborescente). Ce module a été ingénieusement utilisé pour effectuer des mesures morphologiques de cellules non-neuronales 3, cependant, les données de sortie de fournir des informations d'un réseau étendu cellulaire en utilisant un logiciel qui dépend d'une forme de la cellule définie plutôt que d'être un modèle en forme amorphe cellulaire. Pour surmonter le problème de l'analyse de cellules en forme amorphe et de rendre le logiciel plus adapté à une application biologique, Imaris développé cellulaire Imaris. Il s'agissait d'un projet scientifique avec l'Eidgenössische Technische Hochschule, qui a été développé pour calculer la relation entre les cellules et organites. Bien que le logiciel permet la détection des contraintes biologiques, en forçant un noyau par celluleet l'utilisation de membranes cellulaires pour les cellules du segment, il ne peut pas être utilisé pour analyser les données de fluorescence qui ne sont pas continu, car, idéalement, il construit la surface des cellules sans les espaces vides. À notre connaissance, à l'heure actuelle aucune approche modifiable par l'utilisateur automatisé qui fournit des informations morphométriques à partir d'images de fluorescence 3D a été développé qui permet d'obtenir des informations spatiales cellulaire d'une forme non définie (Figure 1).
Nous avons développé une plate-forme d'analyse en utilisant le module de base Imaris logiciels et Imaris XT interfacé à MATLAB (Mat Works, Inc.) Ces outils permettent la mesure 3D de cellules sans une forme prédéfinie et avec incohérentes composants du réseau de fluorescence. En outre, cette méthode permettra aux chercheurs qui ont étendu l'expertise dans les systèmes biologiques, mais pas la connaissance des applications informatiques, d'effectuer la quantification des changements morphologiques dans la dynamique cellulaire.
Protocole
1. Tridimensionnelle analyse morphométrique de la cellule unique modifications phénotypiques
- Embryonnaires humaines de rein (HEK293), les cellules ont été transfectées avec l'hémagglutinine (HA)-étiqueté corticotropin libérant du récepteur du facteur-2 (CRF-R2), un G récepteurs couplés aux protéines (RCPG) comme décrit précédemment 4, 5.
- Les cellules ont été laissées sans traitement (pas de traitement, NT), stimulées avec le ligand endogène CRF-R2, facteur de libération de corticotropine, CRF (1 uM, 30 min), ou prétraité avec un sélectif CRF-R2 antagoniste, anti-sauvagine 30 (AS -30, 1 pM, 30 min) avant le traitement agoniste.
- Les cellules ont ensuite été fixées, perméabilisées et traités avec des anti-HA. CRF-R2 a été visualisée en utilisant Alexa 594 nm conjugué anti-souris (IgG 1) anticorps. DAPI a été utilisé pour visualiser la scène mitotique noyaux.
- Pour limiter la subjectivité expérimentateur, les conditions expérimentales n'étaient pas connus jusqu'à ce que les images ont été acquises et analysées.
- Nous avons acquis l'images de cellules HEK293 fixes à l'aide d'un plan-apochromatique 63x/1.4 huile DIC objectif et microscope Zeiss LSM 510 META confocal relié à un système intégré de laser cohérente à deux photons comprend un laser Verdi-V5 et un système de Mira laser 900-F.
- Pendant le processus d'acquisition de données, les cellules ont été compartimentées à la fois par sectionnement multispectrale, 488 nm et 790 nm (~ 350 nm Ex 2ph.) Et z-partitionnement (par incréments de 0,5 um) afin d'inclure les données de la membrane nucléaire externe au récepteur extracellulaire extrémités.
- Les données de fluorescence ont été traitées à l'aide Imaris premier, ce qui permet la visualisation et la segmentation des données microscopie 3D, et un modèle 3D composé de voxels cubiques a été créée pour l'analyse morphométrique.
Puis, Imaris XT module a été utilisé pour l'interface avec la langue Imaris MATLAB programme d'ordinateur pour déterminer l'emplacement des coordonnées des extensions GPCR.
Afin de prendre en compte la variabilité cellulaire, nous avons obtenu et analysé par fluorescence images takfr à partir de 22 cellules: aucun traitement (NT) (n = 7), agoniste (CRF) traitement (n = 8) et de pré-traitement avec un antagoniste (AS-30) avant le traitement agoniste (n = 7) (figure 2) .- La région d'intérêt (ROI) doit comprendre une cellule qui n'est pas en phase mitotique actif et ne ferme pas à d'autres cellules. De cette manière, l'analyse portera sur les cellules avec un seul noyau et les extensions récepteurs ne sont pas perturbé par la proximité d'autres cellules.
- La structure 3D cellulaire a été reconstruite à partir des données multispectrales de fluorescence à l'aide Imaris (v.7.1.1).
- À la suite de l'algorithme personnalisé par Imaris, la première surface de rendu est utilisé pour représenter la membrane nucléaire. Imaris détermine s'il ya plus d'un noyau dans la région d'intérêt.
- Ensuite, l'algorithme de création de points a été utilisée pour localiser l'extension CRF-R2. La détection des taches a été utilisé, car il compense le bruit de fond et l'intensité irrégulière de laréseau complexe de cellules en forme amorphe.
- Pour maximiser l'intégration de chaque unité de détection de la fluorescence de CRF-R2, le diamètre des taches a été fixé à 0,2 um, ce qui est la plus petite unité au sein de l'image d'extrapoler des informations distinctes sous la forme d'une intensité mesurée en utilisant un filtre gaussien. Filtrage place a été incorporée dans le processus de création automatisée des taches. Le logiciel, cependant, donne à l'utilisateur la possibilité d'utiliser des filtres pour définir les paramètres.
- Pour éviter la troncature des données, l'ensemble de données a été converti en 8-bits (non signé) point fixe, à 32-bit virgule.
- Les données de voxels intensités ont été échangés à repérer à l'aide des données de coordonnées Imaris XT Module interfacé avec MATLAB et l'emplacement exact de chaque point de l'espace a été déterminée en effectuant une transformation de distance à l'aide de la membrane nucléaire comme point de référence (figure 3).
- Les données résultantes peuvent être quantifiés et présentés sous forme de graphique pour sSTATISTIQUES analyse. Les comparaisons entre les groupes ont été effectuées à l'aide de deux ANOVA et Bonferroni post-test. Les données sont présentées sous forme de moyenne ± écart-type. Les différences sont considérées comme significatives à * p <0,05. Les calculs ont été réalisés avec GraphPad Prism 5,02 (figure 4).
2. Les résultats représentatifs
Pour démontrer la puissance de notre approche, nous avons quantifié les modifications cellulaires qui résultent de l'interaction de G récepteurs couplés aux protéines (RCPG) et du facteur de libération de corticotropine récepteur-2 (CRF-R2) avec son ligand CRF endogène dans les cellules HEK293 transfectées.
Nous montrons que le CRF-R2 récepteurs sont localisés dans la membrane plasmique et le projet de régions finies de la membrane des cellules (figure 2A et Movie 1). En utilisant l'analyse 2D classique, il est possible de détecter ce sous-ensemble de extracellulaires des récepteurs CRF-R2 seulement si nous analysons l'adhésion récepteur points sur la vitre couvre. Par conséquent, nous perdons toute autre information provenant de z empilés données multispectrales (figure 5).
Lorsque les cellules sont traitées avec le CRF, les récepteurs extracellulaires sont considérablement réduits, comme indiqué par la diminution de la distance des points de la membrane plasmique. Ils sont également redistribués à partir d'emplacements principalement finis en un certain nombre d'endroits discrets (figure 2B et Movie 2).
L'effet du CRF sur la distribution récepteur membranaire est empêchée par un prétraitement avec l'antagoniste du CRF-R2 spécifique, antisavagine 30 (AS-30) et nous constatons que les extensions CRF-R2 ne changent pas (figure 2C et Movie 3).
La répartition des taches distale, tracées dans les 5 um spectre de couleurs intervalles codés, est utilisée pour visualiser la distance des voxels à partir de la membrane nucléaire. Aucun traitement et antagoniste pretreatment (AS-30, 1 uM, 30 min) avant le traitement agoniste (CRF, 1 uM, 30 min) montrent aucune différence significative (ns) différence de contraction GPCR. Le traitement des cellules avec l'agoniste (CRF, 1 uM, 30 min) réduit progressivement le nombre de CRF-R2 contenant des voxels par rapport à l'absence de traitement, 0-5 um (ns), 6-15 um (** p < 0,01) et> 15 um (*** p <0,005), ou par rapport à AS-30 traitement, 0-10 um (ns), 11-15 um (** p <0,01) et> 15 um (*** p <0,005) (figure 4).
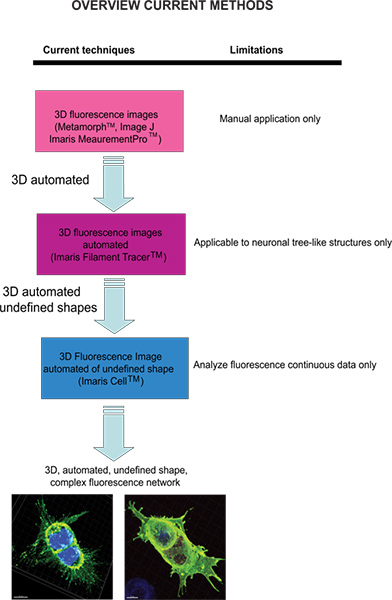
Figure 1. Schéma des techniques actuellement disponibles et leur limitation à analyser les images de fluorescence. Cliquez ici pour agrandir la figure .
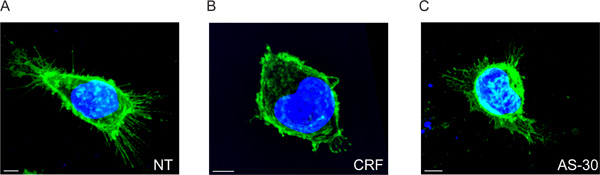
Figure 2. 3D multispectral images de fluorescence des cellules HEK293 transfectées avec HA-CRF-R2 avant et après le traitement agoniste et antagoniste. Images fusionnées montrant HA-CRF-R2-sondé utilisant des anticorps anti-HA et visualisées en utilisant Alexa 488 anticorps conjugué anti-souris (IgG 1) secondaire; DAPI a été utilisé pour visualiser les noyaux. Les images ont été acquises à balayage laser confocal (CLS) microscope. Barre d'échelle 5 microns.
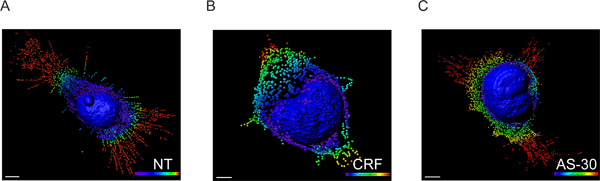
Figure 3. Modèle 3D de cellules HEK293 transfectées avec HA-CRF-R2 reconstitué à partir des images en utilisant le logiciel CLS Imaris. Rendu de la surface du noyau et de la création taches décrivant des RCPG convertis en de petites vésicules. Les données de fluorescence ont été traitées à l'aide Imaris première qui permet la visualisation et la segmentation de l'ensemble de données 3D microscopie. Puis, Imaris XT a été utilisé pour Imaris d'interface avec MATLAB. Les intensités de voxels ont été échangées contrecoordonne place. Les points du spectre codé en couleur (bleu, vert 0-5 um, 6-10 um, jaunes et rouges um 11-15> 15 um) représentent la forme à distance de la membrane nucléaire. Échelle de 5 um.
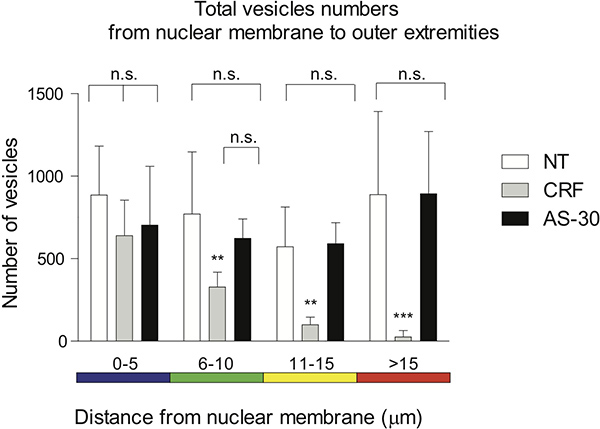
La figure 4. Représentation graphique de la distribution distale de points tracés dans le spectre de couleur codées dans des intervalles de 5 um est utilisée pour visualiser la distance des voxels à partir de la membrane nucléaire. Pas de traitement et de prétraitement avec un antagoniste (AS-30, 1 uM, 30 min) avant le traitement agoniste (CRF, 1 uM, 30 min) montrent aucune différence significative (ns) différence de contraction GPCR. Le traitement des cellules avec un agoniste (CRF, 1 uM, 30 min) réduit progressivement la distance du nombre de CRF-R2 contenant des voxels par rapport à l'absence de traitement 0-5 um (ns), 6-15 um (** p <0,01) et> 15 um (*** p <0,005), ou AS-30 de traitement 0-10 um (ns), 11-15 um (** p <0,01) et> 15 um (*** p <0,005).
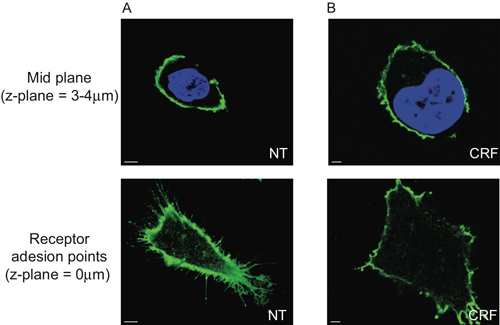
Figure 5. Limitation de l'analyse morphométrique 2D de cellules HEK 293 transfectées avec HA-CRF-R2. L'article plan médian de cellules (3-4μm-dessus de la lamelle de verre) indiquant le centre des noyaux visualisées au DAPI et HA-CRF-R2 sondé avec anti-HA et visualisées à l'aide Alexa 488 nm conjugué anti-souris (IgG 1) secondaire anticorps et acquis avec CLS montre pas de différence entre (A) aucun traitement (NT) et (B) agoniste (CRF, 1 uM, 30 min), tandis que les points d'adhérence récepteurs sont radicalement différentes.
Film 1. Tourner librement dans le modèle 3D "Surpass" Mode de cellules HEK 293 transfectées avec HA-CRF-R2, aucun traitement, pour évaluer les différences phénotypiques cellulaires entre les protéines réceptrices. La barre d'échelle de 5 à 20 um./ Files/ftp_upload/4233/4233movie1.avi "target =" _blank "> Cliquez ici pour voir le film.
Movie 2. Tourner librement dans le modèle 3D "Surpass" Mode de cellules HEK 293 transfectées avec HA-CRF-R2, traitement par agoniste, CRF (CRF, 1 uM, 30 min) pour évaluer les différences phénotypiques cellulaires entre les protéines réceptrices. La barre d'échelle de 5 à 20 um. Cliquez ici pour voir le film.
Movie 3. Librement 3D en rotation dans le "Surpass" Mode de cellules HEK 293 transfectées avec HA-CRF-R2, un prétraitement avec un antagoniste (AS-30, 1 uM, 30 min) avant le traitement agoniste (CRF, 1 uM, 30 min ) pour évaluer les différences phénotypiques cellulaires entre les protéines réceptrices. La barre d'échelle de 5 à 20 um. Cliquez ici pour voir le film.
Discussion
Nous avons montré que le traitement CRF induit un changement important dans la morphologie et la localisation de CRF-R2. Le changement de CRF-R2 a été inhibée par le traitement antagoniste sélectif. Nous avons montré que des modifications des récepteurs n'ont pas été détectés et ne peut pas être mesurée en utilisant les standards techniques 2D multispectrale. La possibilité d'étudier des images 3D complexes est essentiel d'intégrer la complexité des paramètres biologiques pour l'analyse...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Nous remercions le Centre de développement de l'imagerie biologique (BIDC) de l'Université de Californie à San Francisco pour l'utilisation de la Imaris, Imaris XT et Matlab. Nous remercions V. Kharazia pour l'assistance technique et AT Henry, LK Floren, L. Daitch pour leur contribution à la rédaction du manuscrit. Ce travail a été soutenu par un financement de l'État de Californie recherche médicale sur l'abus d'alcool et les substances grâce à l'UCSF à SEB, le National Institutes of Health: 1R21DA029966-01 et d'attribution NIH Fast Track pour cribler la collection MLSMR à SEB école, UCSF de pharmacie ( Bureau du doyen et de pharmacie clinique) et l'Ecole de Médecine (Clinical Pharmacology & Therapeutics expérimentales) pour CLHK.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires (optionnel) |
| De rein embryonnaire humain (HEK293) | American Type Culture Collection | CRL-1573 | |
| Eagle modifié par Dulbecco (DMEM) | Invitrogen | 11965118 | |
| Sérum foetal bovin (FBS) | Invitrogen | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | Invitrogen | A-11001 | |
| Anticorps monoclonal anti-HA.11 (IgG 1) | Covance | 16B12 | |
| DAPI | Vector Laboratories | H-1200 | ; |
| CRF | Sigma | C2917 | |
| Antisauvagine-30 (AS-30) | Sigma | A4727 |
Références
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon