Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Itératif d'optimisation des duplex d'ADN pour la cristallisation des complexes ADN-SEQA
Dans cet article
Résumé
Structure cristalline des complexes protéine-ADN peuvent donner une idée fonction de la protéine, mécanisme, ainsi que la nature de l'interaction spécifique. Nous rapportons ici comment optimiser la durée, la séquence et extrémités de l'ADN double brin pour la co-cristallisation avec Escherichia coli SEQA, un régulateur négatif de l'initiation de la réplication.
Résumé
Escherichia coli SEQA est un régulateur négatif de la réplication de l'ADN qui empêche prématurés événements réinitiation par séquestrants hémiméthylé grappes GATC au sein de l'origine de réplication 1. Au-delà de l'origine, SEQA se trouve à la fourche de réplication, où il organise l'ADN nouvellement répliqué par une augmentation des structures ordonnées 2. SEQA que faiblement associés avec des séquences GATC simples, mais il forme des complexes de haute affinité avec les duplexes d'ADN contenant plusieurs sites GATC. L'unité minimale fonctionnelle et structurelle de SEQA est un dimère, ce qui explique l'exigence d'au moins deux séquences GATC pour former un complexe de haute affinité avec l'ADN hémiméthylé 3. En outre, l'architecture SEQA, avec l'oligomérisation et de liaison d'ADN domaines séparés par un lieur flexible, permet la liaison de répétitions GATC séparés par jusqu'à trois spires hélicoïdales. Par conséquent, la compréhension de la fonction de SEQA au niveau moléculaire nécessite la structure analyse des SEQA lié à de multiples séquences GATC. En protéine-ADN cristallisation, l'ADN peut n'en ai pas à un effet exceptionnel sur les interactions d'emballage en fonction de la taille relative et l'architecture de la protéine et l'ADN. Si la protéine est plus grande que la plupart des empreintes d'ADN ou de l'ADN, l'empilement cristallin est principalement médié par interactions protéine-protéine. A l'inverse, lorsque la protéine est de la même taille ou plus petite que l'ADN ou ne couvre qu'une fraction de l'interaction ADN, ADN-ADN et l'ADN-protéine dominer empilement cristallin. Par conséquent, la cristallisation de complexes protéine-ADN nécessite le dépistage systématique de longueur 4 de l'ADN et l'ADN (extrémités émoussées ou faux) 5-7. Dans ce rapport, nous décrivons comment concevoir, optimiser, de purifier et cristalliser duplex d'ADN contenant des répétitions en tandem hémiméthylé GATC dans un complexe avec une variante dimère de SEQA (SeqAΔ (41-59)-A25R) pour obtenir des cristaux appropriés pour déterminer la structure.
Protocole
1. Purification des protéines
La séquence de liaison flexible reliant l'. N-(oligomérisation) et C-terminal (liaison à l'ADN) domaines de SEQA aides à la reconnaissance des répétitions hémiméthylé GATC séparées par une à trois spires de l'ADN Pour cette étude, nous avons utilisé une variante de dimère de SEQA (SeqAΔ (41-59)-A25R) avec une mutation ponctuelle dans le domaine N-terminal qui empêche davantage d'oligomérisation et un lieur raccourci qui limite liaison à l'ADN de tandem GATC répète séparés exclusivement par une tour sur l'ADN (Figure 1) 2,8.
- Transformer BL21 (DE3) par le plasmide codant pour SEQA sous le contrôle du promoteur T7,
- Plaque de la réaction de transformation de LB-agar, y compris 100 ug / ml d'ampicilline,
- Prélever des colonies mixtes pour inoculer une culture à petite échelle pendant la nuit (milieu LB avec 100 pg / ml d'ampicilline),
- Le lendemain matin, inoculer une L 1 du multimédia en utilisant une dilution 1:100 de la overnight la culture,
- Cultiver les cellules à une densité optique de 600 ~ 0,7 et induire la production de protéines par addition d'isopropyl β-D-1-thiogalactopyranoside (IPTG) à une concentration finale de 1 mM,
- Continuer l'incubation pendant 3 heures à 37 ° C sous agitation orbitale, puis récolter les cellules par centrifugation (10 min à 3300 g),
- Remettre en suspension le culot cellulaire dans un tampon de lyse A et purification par sonication,
- Effacer le lysat par centrifugation (40 min à 39000 g) et de charger le surnageant sur une colonne d'héparine équilibrée avec le tampon de purification,
- Éluer SEQA utilisant un gradient linéaire de NaCl 1 M (SEQA élue à ~ NaCl 0,7 M),
- Mettre en commun les fractions contenant de l'ensemble SEQA, diluer à réduire la force ionique de l'échantillon et de la charge dans une colonne de chromatographie échangeuse de cations équilibrée avec un tampon de purification,
- En utilisant un gradient linéaire de sel, pur élue à SEQA ~ NaCl 0,4 M,
- Mettre en commun les fractions contenant SEQA ensemble, concene (3 mg / ml) dans un tampon et un magasin de stockage.
2. Purification d'ADN
- Commande complémentaires oligonucléotides non méthylés et méthylés de votre société préférée,
- Dissoudre 1 pmol de chaque ADN simple brin lyophilisée dans 800 ul de autoclavé ddH O 2, vortex et laisser reposer pendant 10-20 min,
- Ajouter 800 ul de tampon de charge 2X préchauffé à chaque oligonucléotide,
- Pour oligonucléotides longs nucléotides 20-30, préparer un grand gel dénaturant 10% (160 x 250 x 1 mm):
* Bien mélanger 80 ml de mélange PAGE 10%, 80 TEMED ul et 800 ul par le persulfate d'ammonium gel et verser,
* Une fois polymérisé, enlever le peigne et rincer les puits avec fond ddH 2 O,
* Monter les gels sur gel de fonte y compris la plaque de refroidissement et ensuite remplir les réservoirs supérieur et inférieur avec tampon de migration (1X TBE),
* Pré-exécuter le gel à 700-750 V pour réchauffer le gel jusqu'à 55 ° C,
* Arrêter la course et rincer le puits thoroughly avec tampon. - Chauffer les oligonucléotides à 90 ° C pendant 2 min,
- Vortex et faire tourner les échantillons et immédiatement avant le chargement du gel,
- Exécutez le gel à ~ 700 V et l'arrêter une fois que votre oligonucléotide a migré à mi-chemin. (Notez que sur un gel de polyacrylamide 10% de bleu de bromophénol co-migre avec des oligonucléotides ~ 20 bases de long et le xylène cyanol FF avec ~ 60 bases de long),
- Arrêter le gel, démonter la boîte de gel et retirez les cales,
- Sur une surface plane, retirer une plaque de verre et couvrir le gel avec une pellicule de plastique,
- Tournez le gel autour, retirez la plaque de verre et couvrir d'une pellicule plastique,
- Marquez les bandes en utilisant la lumière UV et une plaque fluorescente derrière le gel de voir l'ombre d'ADN,
- Couper la bande avec une lame de rasoir en petits morceaux et de les transférer dans un tube de 15 ml stérile,
- Ajouter 9 ml de tampon d'élution et éluer une nuit à 37 ° C sous agitation,
- Transférer délicatement la solutionà un tube à centrifuger autoclave à l'aide d'une pipette de gel de chargement pour éviter le transfert de pièces d'acrylamide et ajouter 1 ml d'acétate de sodium 3 M à pH 7 (dilution 1:10) plus 25 ml d'éthanol à 100% réfrigérés (2,5 volumes),
- Incuber à -20 ° C pendant au moins 3 heures,
- Isoler et transférer le surnageant dans un tube séparé,
- Sécher le culot sur le speed-vac à feu moyen,
- Reprendre le culot dans 400 ul de autoclavé ddH 2 O et transférer dans un nouveau tube,
- Ajouter 40 ul d'acétate de sodium 3 M à pH 7 et 1 ml d'éthanol à 100% et bien mélanger (vortex) et incuber 30 min à température ambiante, puis 30 min à -20 ° C,
- Faites tourner pendant 15 min à 18000 g et jeter le surnageant,
- Rincer le culot avec 100 ul d'éthanol froid à 70% pour éliminer le sel résiduel à partir du culot et de spin pendant 6 min à 18000 g. Jeter l'éthanol et sécher le culot de speed-vac,
- Reprendre le culot dans un total de 100 ul de autoclavé ddH 2 O. Mesurer la concentration de l'oligonucléotide,
- Pour recuire les duplexes d'ADN hémiméthylé, mélanger concentrations équimolaires des simples brins complémentaires et chauffer les mélanges à 95 ° C dans un bain-marie pendant 5 min, puis les laisser refroidir lentement à température ambiante à l'intérieur du bain-marie.
3. Formation protéine-ADN et des analyses complexes
- Mélanger des volumes égaux d'purifié SeqAΔ (41-59)-A25 (81 uM) et de l'ADN hémiméthylé (81 uM),
- Incuber à température ambiante pendant 15 min et conserver à 4 ° C jusqu'à ce que vous soyez prêt à l'utiliser,
- Écran pour des conditions de cristallisation en utilisant commerciales clairsemée écrans à matrice,
- Une fois fils de cristallisation initiales ont été identifiés, d'optimiser les conditions de croissance des cristaux de qualité de diffraction,
- Cryoprotect les SEQA à ADN résultant cristaux, soit en augmentant la quantité de PEG 400 présent dans la solution de cristallisation à une concentration finale de 25% (v / v) ou l'ajout de glycérol 20% (v / v) de la solution de cristallisation,
- Scoop cristaux individuels avec une boucle en nylon, et les flash-congeler dans l'azote liquide,
- Testez la limite de diffraction de chaque cristal à 100 K.
4. Les résultats représentatifs
Pour obtenir la structure cristalline de SeqAΔ (41-59)-A25R lié à l'ADN hémiméthylé, nous consécutivement trois paramètres optimisés sur l'ADN: (i) la séparation entre les séquences GATC hémiméthylé, (ii) la longueur du duplex, et (iii) l' absence / présence de 5 'surplombs.
Tests de modification de l'électro-mobilité indiquent que SeqAΔ (41-59)-A25R se lie préférentiellement répète GATC séparés par 10.9 paires de bases (Figure 1). Par conséquent, nous avons d'abord projeté duplex 23-24 paires de bases (pb) de long contenant deux séquences GATC hémiméthylé séparés par soit 9 ou 10 points de base. Trois duplex a donné des cristaux belle forme (figure 2). Alsi aucun des cristaux diffractés à haute résolution, le 23 bps en duplex de long avec les deux sites GATC séparés de 9 points de base diffractée des rayons X mieux que le reste, ce qui indique que la séparation GATC de 9 points de base a été préféré pour la cristallisation. Par conséquent, nous avons fixé la distance inter-GATC à 9 points de base pour tous les écrans suivants.
Généralement, la cristallisation est favorisée ADN duplex pour des longueurs exactes correspondant aux spires hélicoïdales, car plusieurs molécules d'ADN peut empiler tête-à-queue pour former un ADN-B continue dans le cristal 9. Par conséquent, nous avons réduit la longueur totale des duplex de 21 points de base (soit deux spires hélicoïdales). Alors un duplex avec 21 points de base des extrémités franches n'ont pas donné des cristaux de qualité de diffraction (données non présentées), un duplex 21 points de base avec une seule extrémité protubérante 5 'nucléotides à chaque extrémité ne donner des cristaux qui diffractée à 5 Å dans notre source d'origine. L'amélioration de la limite de diffraction suggéré que duplex de bout-en-bout association a été en effet favorisertion de cristaux d'emballage.
Depuis SEQA interagit avec des séquences GATC qui sont sur la même face de l'ADN, la face opposée de la double hélice d'ADN doit être exposés au solvant. Pour améliorer encore les cristaux du complexe, on a alors modifié la séquence du duplex d'inclure un dinucléotide CG entre les deux sites GATC-gorge, de promouvoir squelette interactions avec les molécules d'ADN adjacentes dans le cristal par la face opposée du duplex, un procédé qui a été utilisé pour améliorer la cristallisation de l'ADN dans des 10 dernières années. Cependant, la limite de diffraction des cristaux cultivés avec le duplex contenant CG était identique à celles qui sont cultivées avec un duplex d'ADN similaire qui ne contient pas le dinucléotide CG (figure 3). Ce résultat indique que la rainure cœur de réseau interactions ne sont pas importants dans ce cas. Nous avons ensuite optimisé la longueur des surplombs en comparant les cristaux obtenus avec un duplex d'ADN qui ont deux nucléotides supplémentaires à chaqueL'extrémité 5 '. Ce changement a eu un effet drastique sur la morphologie des cristaux, ainsi que, la limite de diffraction, indiquant que le nucléotide supplémentaire a radicalement changé les contacts et l'organisation moléculaire cristaux améliorée (figure 3).
La structure cristalline de SeqAΔ (41-59)-A25R lié à cette dernière duplex d'ADN a confirmé que la face libre de la double hélice d'ADN ne peut avoir de contact cristal, comme prévu par l'effet limité de l'introduction d'un dinucléotide CG-. En dépit de la taille relative des protéines et de l'ADN, la plupart des interactions entre compagnons de symétrie sont médiés par des interactions protéine-protéine et protéine-ADN (figure 4). Il est intéressant, dans ce cas particulier, l'effet bénéfique de faux dinucléotide 5 'n'est pas due à la formation d'un ADN pseudo-continue. Au lieu de cela, l'extrémité 5 'du brin dénaturé les projets à l'écart des axes d'ADN et interagit avec la molécule SEQA proximale du complexe, ce qui explique pourquoi deux nucléotides w ere strictement nécessaire pour changer le cristal d'emballage 8,11.
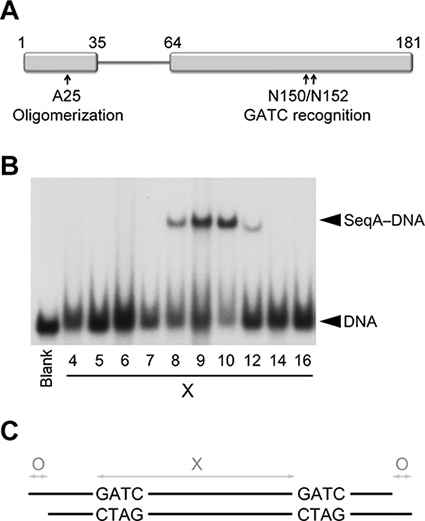
Figure 1. La liaison de SeqAΔ (41-59)-A25R à l'ADN hémiméthylé. (A) Représentation schématique des domaines de SEQA que oligomérisation des protéines médiate et liaison à l'ADN. (B) analyse électrophorétique de décalage de mobilité des SeqAΔ (41-59)-A25R avec des ADN contenant deux séquences GATC hémiméthylé séparés par un nombre croissant de paires de bases ( X). La voie la plus à gauche (marqué Blank) contient un mélange équimolaire d'ADN avec 5, 7, 12, 21, 25 et 34 paires de bases entre les deux séquences GATC en l'absence de SeqAΔ (41-59)-A25R. (C) Diagramme montrant les trois variables optimisées sur les duplexes d'ADN pour obtenir des cristaux de qualité de diffraction.
upload/4266/4266fig2.jpg "fo: content-width =" 6in "fo: src =" / files/ftp_upload/4266/4266fig2highres.jpg "/>
Figure 2. Effet de la variation inter-GATC distance. Résumé des oligonucléotides utilisés et les cristaux obtenus avec duplex pb contenant deux longues 23-24 hémiméthylé sites GATC séparés par 9 ou 10 paires de bases. Toutes les photos de cristaux ont été prises au même grossissement et la barre d'échelle indique 100 um. La limite de résolution de chaque SeqAΔ (41-59)-A25R-ADN cristal est basée sur des images de diffraction collectées sur une Rigaku RU-300 système générateur de rayons X. Cliquez ici pour agrandir la figure .

Figure 3. Effet de la variation de l'ADN se termine. Résumé de til oligonucléotides utilisés et les cristaux obtenus avec 21 pb de long duplex contenant deux sites GATC hémiméthylé séparés par 9 paires de bases et comprenant 0, 1 ou 2 nucléotides surplombe extrémité 5 '. Toutes les photos de cristaux ont été prises au même grossissement et la barre d'échelle indique 100 um. La limite de résolution de chaque SeqAΔ (41-59)-A25R-ADN cristal est basée sur des images de diffraction recueillies à X12C lignes de lumière et X29 (NSLS, BNL). Cliquez ici pour agrandir la figure .
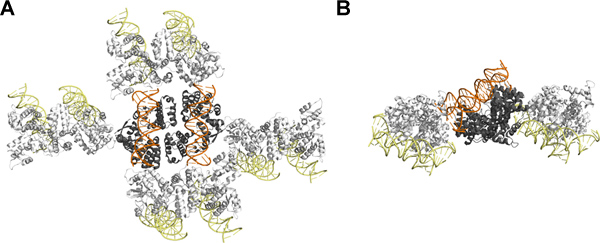
Figure 4. Emballage cristal de SeqAΔ (41-59)-A25R lié à l'ADN avec le surplomb dinucléotide: Vue du haut (A) et le côté (B). L'unité asymétrique contient deux SeqAΔ (41-59)-A25R-ADN où la protéine est représentée en gris tandis que l'ADN est représenté en orange. Symmetry liée SeqAΔ (41-59)-A25R-molécules d'ADN sont représentées en blanc pour la protéine et jaune pour l'ADN. Ce chiffre a été préparé en utilisant PyMOL 12. Ce chiffre est lié à Film 1. Cliquez ici pour agrandir la figure .
Film 1. Cliquez ici pour voir le film .
Discussion
Un des plus grands défis dans macromoléculaire cristallographie aux rayons X est d'obtenir des cristaux de qualité de diffraction. Dans le cas des complexes de protéines ou de protéines-ADN, ce défi est exacerbée en raison des variables supplémentaires qui doivent être optimisés. Il est largement admis que la longueur de l'ADN et la présence de surplombs collantes pour renforcer l'association de molécules d'ADN voisines dans un pseudo-duplex plus sont les principaux paramètres à optimiser. ...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Les auteurs tiennent à remercier le personnel PXRR au NSLS (Brookhaven National Laboratory) pour l'assistance au cours de la collecte des données et Monica Pillon de l'aide pour la purification d'ADN. Ce travail a été soutenu par les Instituts de recherche en santé (MOP 67189).
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Catalogue # | Commentaires (optionnel) |
| TRIS | Bioshop | TRS003.5 | |
| Acide éthylènediaminetétraacétique (EDTA) | Fisher Scientific | E478-500 | |
| Dithiotheitrol (TNT) | Bio Basic Inc | DB0058 | |
| NaCl | Bioshop | SOD002.10 | |
| Glycérol | Caledon | 5350-1 | |
| Saccharose | Sigma-Aldrich | S5016-500G | |
| Dodécyl sulfate de sodium (SDS) | Bioshop | SDS001.500 | |
| Urée | Bioshop | URE001.5 | |
| 40% Bis 29:1 / acrylamide | Bio Basic Inc | A0007-500ml | Stocker à 4 ° C |
| L'acide borique | EMD | BX0865-1 | |
| Xylène cyanol FF | Bio-Rad | 161-0423 | |
| Bleu de bromophénol | Bioshop | BR0222 | |
| Système double réglable Gel verticale | CBC Scientific Company Inc | SGAD-250 | |
| Indice de cristallisation écran | Hampton Research | HR2-144 | Stocker à 4 ° C |
| Écran de l'assistant cristallisation je | Emerald BioSystems | EBS-WIZ-1 | Stocker à 4 ° C |
| Écran de l'assistant cristallisation II | Emerald BioSystems | EBS-WIZ-2 | Stocker à 4 ° C |
| Écran cristallisation Classics | Qiagen | 130701 | Stocker à 4 ° C |
| Plateaux Intelliplate | Art Robbins Instruments | 102-0001-00 | |
Solutions Tampon de purification de protéines: 100 mM Tris pH 8, EDTA 2 mM, DTT 2 mM et glycérol 5%. Tampon de stockage Protéines: 20 mM Tris pH 8, NaCl 150 mM, DTT 5 mM, EDTA 0,5 mM et glycérol à 5%. Mélange chargement de gel: Ajouter 20 g de saccharose, 25 mg de bleu de bromophénol, 25 mg de xylène cyanol FF, 1 ml de 10% p / v de SDS et 10 ml de TBE 10X à 70 ml d'autoclave ddH O. 2 Remuer avec un chauffage doux jusqu'à ce que le saccharose est dissous et ajuster la finalevolume à 100 ml avec de l'autoclave ddH O. 2 Stocker à 4 ° C. Tampon de charge 2X: Ajouter 11 g d'urée à 10 ml de mélange de chargement de gel. Incorporer sur une plaque chauffante jusqu'à ce que l'urée se dissout. Aliquoter en tubes de 2 ml et conserver à 4 ° C. PAGE 10X mélange: Mélanger 420,4 g d'urée, 100 ml de TBE 10X (autoclave), 250 ml de 40% 29:1 Bis / acrylamide dans les ddH 2 O. Remuer jusqu'à ce que complètement dissous et ajuster le volume à 1 litre. Stocker dans des bouteilles foncées à 4 ° C. TBE 10X: Dissoudre 108 g de TRIS, 55 g d'acide borique et 9,3 g d'EDTA à 1 litre de ddH 2 O. Autoclave et conserver à température ambiante. Tampon d'élution: Diluer 8 ml de NaCl 5 M, 2 ml de 1 M pH TRIS 7,5, 0,4 ml d'EDTA 0,5 M pH 8 sur 200 ml de ddH 2 O. Autoclave et conserver à température ambiante. |
Références
- Campbell, J. L., Kleckner, N. E. coli oriC and the dnaA gene promoter are sequestered from dam methyltransferase following the passage of the chromosomal replication fork. Cell. 62, 967-979 (1990).
- Guarné, A. Crystal structure of a SeqA-N filament: implications for DNA replication and chromosome organization. Embo. J. 24, 1502-1511 (2005).
- Brendler, T., Austin, S. Binding of SeqA protein to DNA requires interaction between two or more complexes bound to separate hemimethylated GATC sequences. Embo. J. 18, 2304-2310 (1999).
- Jordan, S. R., Whitcombe, T. V., Berg, J. M., Pabo, C. O. Systematic variation in DNA length yields highly ordered repressor-operator cocrystals. Science. 230, 1383-1385 (1985).
- Tan, S., Hunziker, Y., Pellegrini, L., Richmond, T. J. Crystallization of the yeast MATalpha2/MCM1/DNA ternary complex: general methods and principles for protein/DNA cocrystallization. J. Mol. Biol. 297, 947-959 (2000).
- Rice, P. A., Yang, S., Mizuuchi, K., Nash, H. A. Crystal structure of an IHF-DNA complex: a protein-induced DNA U-turn. Cell. 87, 1295-1306 (1996).
- Yang, W., Steitz, T. A. Crystal structure of the site-specific recombinase gamma delta resolvase complexed with a 34 bp cleavage site. Cell. 82, 193-207 (1995).
- Chung, Y. S., Brendler, T., Austin, S., Guarne, A. Structural insights into the cooperative binding of SeqA to a tandem GATC repeat. Nucleic Acids Res. , (2009).
- Anderson, J., Ptashne, M., Harrison, S. C. Cocrystals of the DNA-binding domain of phage 434 repressor and a synthetic phage 434 operator. Proc. Natl. Acad. Sci. U.S.A. 81, 1307-1311 (1984).
- Timsit, Y., Moras, D. DNA self-fitting: the double helix directs the geometry of its supramolecular assembly. Embo J. 13, 2737-2746 (1994).
- Chung, Y. S., Guarne, A. Crystallization and preliminary X-ray diffraction analysis of SeqA bound to a pair of hemimethylated GATC sites. Acta Crystallogr Sect F Struct Biol Cryst Commun. 64, 567-571 (2008).
- DeLano, W. L. . The PyMOL Molecular Graphic Systems. , (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon