Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Bioluminescent imagerie bactérienne
Dans cet article
Résumé
Cet article décrit l'administration de Lux-étiqueté Les bactéries à des souris et ultérieures In vivo Analyse en utilisant l'imagerie par bioluminescence IVIS.
Résumé
Cette vidéo décrit l'utilisation de l'imagerie du corps entier bioluminesce (BLI) pour l'étude de la traite des bactéries chez des souris vivantes, avec un accent sur l'utilisation de bactéries en thérapie génique et cellulaire du cancer. Les bactéries présentes une classe intéressante de vecteur pour la thérapie du cancer, qui possède une capacité naturelle à se développer de manière préférentielle dans les tumeurs après administration systémique. Bactéries génétiquement pour exprimer le gène lux cassette d'autorisation BLI détection des bactéries et en même temps les sites tumoraux. L'emplacement et les niveaux de bactéries au sein des tumeurs au fil du temps peut être facilement examinés, visualisées en deux ou trois dimensions. La méthode est applicable à une large gamme d'espèces bactériennes et les types de xénogreffes de tumeurs. Cet article décrit le protocole d'analyse des bactéries bioluminescentes dans les souris porteuses de tumeurs sous-cutanées. Visualisation des bactéries commensales dans le tractus gastro-intestinal (TGI) par BLI est également décrit. Ce puissant et pas cher, en temps réel représentation stratégie d'imageriets une méthode idéale pour l'étude des bactéries in vivo dans le cadre de la recherche sur le cancer, en particulier la thérapie génique et les maladies infectieuses. Cette vidéo décrit la procédure pour l'étude de lux-étiqueté E. coli dans des souris vivantes, ce qui démontre la lecture spatiale et temporelle BLI réalisables en utilisant le système IVIS.
Protocole
1. L'induction des tumeurs
- Pour l'induction de tumeurs de routine, la dose tumorigène minimale de cellules en suspension dans 200 ul de milieu de culture sans sérum a été injecté par voie sous cutanée (sc) dans le flanc de l'infection gratuit 6-8 semaines vieilles femelles Balb / C ou athymiques souris MF1-nu/nu n = 6 (Harlan, Oxfordshire, Royaume-Uni) (1 x 10 6 cellules 4T1) en utilisant une aiguille de seringue de calibre 21. La viabilité des cellules utilisées pour l'inoculation était supérieure à 95% telle que déterminée par un comptage visuel en utilisant un hémocytomètre et d'exclusion du bleu de trypan (Gibco).
- Après la création de la tumeur, les tumeurs ont été autorisés à se développer et ont été suivis deux fois par semaine. Le volume tumoral est calculé selon la formule V = (ab 2) Π / 6, où a est le plus grand diamètre de la tumeur et b est le diamètre le plus long perpendiculaire à un diamètre.
2. Préparation bactérienne
- La souche bactérienne utilisée dans le présent ProtocoJ'étais E. coli K-12 MG1655, une protéine non-toxine-exprimant la souche, hébergeant un plasmide codant pour luxABCDE qui permet aux bactéries à détecter par BLI. E. coli MG1655 contenant le luxABCDE intégré a été cultivé en aérobiose à 37 ° C en milieu LB (Sigma-Aldrich, Irlande) additionné de 300 ug / ml d'érythromycine (Em). Le dérivé de bioluminescence MG1655 a été créé en utilisant le plasmide p16S lux qui contient le P AIDE constitutive luxABCDE opéron 1.
- Pour la préparation pour l'administration à des souris, les cultures ont été incubées dans du milieu LB à 37 ° C dans un agitateur à 200 tours par minute à la phase mi-log (densité optique à 600 nm). Les bactéries ont été récoltées par centrifugation (6000 xg pendant 5 min), lavées avec du PBS (Sigma), et dilué dans du PBS 1 × 10 7 unités formant colonies (UFC) / ml pour administration par voie intraveineuse, ou 1 x 10 10 pour gavage.
3. Bactérienne Administrationtion
- Les souris ont été divisés au hasard en groupes expérimentaux lorsque les tumeurs ont atteint environ 100 mm 3 de volume. Pour l'administration par voie intraveineuse, les souris ont reçu chacun sobres 10 6 cellules dans 100 ul, injecté directement dans la veine latérale de la queue en utilisant une aiguille de seringue 28G. Le nombre viable de chaque inoculum a été déterminé par placage rétrospective.
- Pour les études de colonisation GIT, 10 9 cellules bactériennes ont été administrés par voie orale dans 100 pi par souris par gavage, pendant trois jours consécutifs. Préexistantes commensales niveaux bactériens ont diminué avant de nourrir par addition de 5 mg / ml de streptomycine dans l'eau potable de la souris pendant 7 jours avant le début du gavage oral 1.
4. Imagerie par bioluminescence
- 2D imagerie in vivo BLI a été réalisée en utilisant la IVIS100 (étrier). Au moment après l'administration défini des points bactérienne, souris ont été anesthésiées avec étrier de XGI-8 anesthésie au gazSystème avec isofluorane 3%, et l'ensemble du corps d'analyse d'images a été réalisée dans le IVIS 100 système pendant 2-5 min à haute sensibilité.
- Pour l'imagerie 3D, souris anesthésiées ont été placés dans une navette imagerie souris à l'intérieur du système d'imagerie optique pour l'imagerie dorsale (IVIS spectre, étrier). D'acquérir des images de la luciférase bactérienne signal de la reconstruction 3D optique, des longueurs d'onde d'émission de filtre allant de 500 à 580 nm ont été utilisés avec des temps d'acquisition de bacs de 16 min 3-4 par filtre pour maximiser le rapport signal sur bruit. Dans le cadre de cette séquence d'acquisition d'images, une image de la lumière structurée a été obtenu pour définir une carte de hauteur. Cette carte était entrée diffuse la lumière d'imagerie tomographie (DLit) algorithmes de reconstructions qui ont été utilisés pour former une image 3D optique en utilisant un non-négatif au optimisation carrés 2.
- Analyse d'images: Régions d'intérêt ont été identifiés et quantifiés à l'aide du logiciel Image habitable (étrier).
5. ReprésentantRésultats
Dans cette étude, les bactéries commensales non pathogènes E. coli K-12 MG1655 exprimant l'opéron luxABCDE était IV administré à des souris porteuses de xénogreffes de tumeurs sc 4T1. Lux bactérien signal a été détectée spécifiquement dans les tumeurs des souris après administration IV-(Figure 2). Récupération de la culture de bactéries à partir d'échantillons souris valide l'existence d'une relation linéaire entre le nombre de bactéries viables et la quantité de lumière détectée (figure 3). L'imagerie in vivo du oralement administrés par des bactéries commensales dans le GIT est également réalisée en utilisant la 3D BLI.
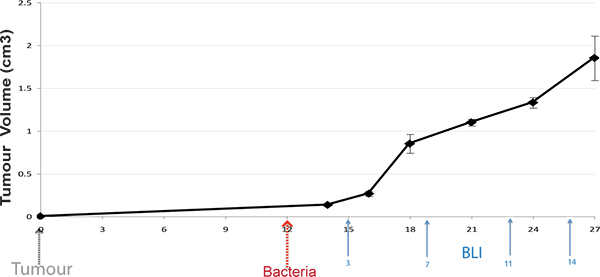
Figure 1. Protocole Timeline. Tumeurs sous-cutanées sont induits chez la souris, et les bactéries administré sur le développement des tumeurs (100 mm 3). Des souris vivantes sont BLI imâgés de moins après l'administration de divers points temporels bactérienne (flèches afficher les temps typiques).

Figure 2. L'administration de E. coli MG1655 luxABCDE à des souris porteuses de tumeurs. tumeurs sous-cutanées 4T1 ont été induites chez MF1 souris nu / nu et E. coli MG1655 luxABCDE administré sur le développement des tumeurs. Chaque animal a reçu 10 6 cellules injectées directement dans la veine latérale de la queue. Les souris ont été imagées moins quatre points dans le temps au cours de l'étude (points noirs axe z et images) avec récupération ultérieure des bactéries viables (UFC) à partir d'échantillons de tumeurs de souris sacrifiées (graphique à barres). Augmentation du nombre de bactéries et de l'expression du gène plasmidique spécifiquement dans les tumeurs a été observée au cours du temps (souris représentant illustré par point de temps). Cliquez ici pour agrandir la figure .
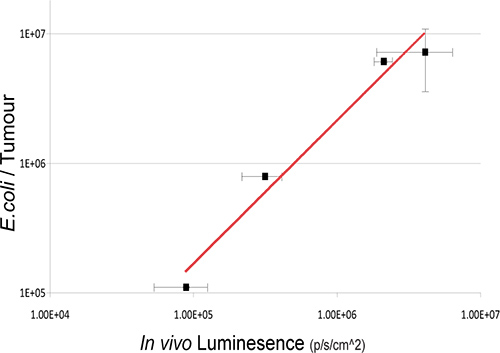
Figure 3. Relation entre le nombre de bactéries intra-tumorale et la bioluminescence. Bactéries viables dans les tumeurs ont été dénombrés par ex vivo de tumeurs culture bactérienne à la suite de BLI à divers points de temps après l'administration IV. Les valeurs de log du nombre de bactéries (ufc) par rapport aux unités bioluminesce in vivo sont représentées graphiquement. Une forte corrélation entre le nombre de bactéries et de signaux de bioluminescence bactérienne est observée R 2 = 0,9717 1.
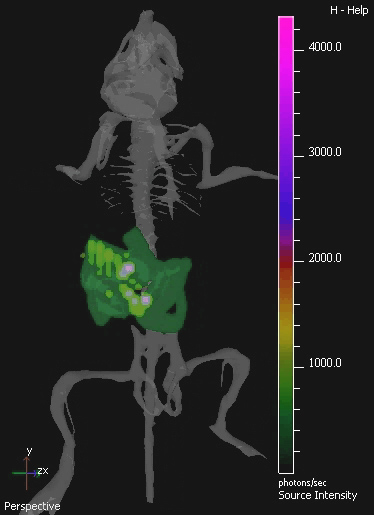
Figure 4. 3D Image De IVIS tractus gastro-intestinal murin colonisés par E. coli MG1655. Le GIT de souris a été colonisée par l'administration orale de 10 9 cfu de E. coli pendant trois jours consécutifs. Une image d'échantillon isolé à partir de la tomographie 3D de la souris colonisé est affiché.Images 3D montrent un atlas numérique de la souris sur le squelette de fournir l'enregistrement anatomique. E. coli MG1655 bioluminescence est visible en vert en bas, et de pourpre à des niveaux supérieurs.
Discussion
Dans le cadre de la thérapie génique, l'utilisation d'agents biologiques pour la livraison de gènes thérapeutiques aux patients a montré des résultats prometteurs 3-5. Comme les virus, les propriétés biologiques innés de bactéries permettre la délivrance d'ADN efficace de cellules ou de tissus, en particulier dans le cadre du cancer. Il a été montré que les bactéries sont naturellement capables de ralliement pour des tumeurs lorsqu'elles sont administrés par voie systémique r?...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Les auteurs tiennent à remercier un soutien pertinent à ce manuscrit de la Commission européenne du septième programme-cadre (PIOF-GA-2009-255466) et le Conseil de la santé irlandais de recherche (HRA_POR/2010/138). Lux-étiqueté E. coli était un cadeau aimable de M. Cormac Gahan, University College Cork.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Lignée cellulaire 4T1 | ATCC | CRL-2539 | Syngénique modèle du cancer du sein dérivée d'une façon spontanée résultant BALB / c tumeur mammaire |
| DMEM | Sigma-Aldrich | D6429 | Eagle modifié par Dulbecco à moyen |
| PBS | Sigma-Aldrich | D8537 | Phosphate Buffered Saline |
| Xenogen IVIS | Caliper Life Sciences | IVIS 100 pour l'imagerie 2D; Spectre IVIS pour la 3D. | |
| Luria Broth Miller (LB) | Sigma-Aldrich | L2542 | Milieu de croissance pour E. coli |
| Érythromycine | Sigma-Aldrich | E5389 | Antibiotique |
| Streptomycine | Sigma-Aldrich | S9137 | Antibiotique |
| Souris MF1nu/nu | Harlan (Royaume-Uni) | 069 (nu) / 070 (nu / +) | Hsd: athymique Nu-Foxn1nu |
| Souris Balb / c | Harlan (Royaume-Uni) | 066 | Haplotype H-2 d |
| Aiguille de gavage | Vet-Tech Solutions (UK) | DE009 | 22G x 38mm droite aiguille de gavage |
| Seringue pour injection IV | BD Biosciences | 309309 à 1 ml | Seringue à insuline avec 28 x G ½ pouces micro-aiguille fine IV. |
| Seringue pour inoculation de la tumeur | Braun | 9161376V | Omnifix 26 x G ½ aiguille pouces |
Références
- Cronin, M., et al. High resolution in vivo bioluminescent imaging for the study of bacterial tumour targeting. PLoS One. 7, e30940 (2012).
- Kuo, C., Coquoz, O., Troy, T. L., Xu, H., Rice, B. W. Three-dimensional reconstruction of in vivo bioluminescent sources based on multispectral imaging. J. Biomed. Opt. 12, 024007 (2007).
- Tangney, M., Ahmad, S., Collins, S. A., O'Sullivan, G. C. Gene therapy for prostate cancer. Postgrad Med. 122, 166-180 (2010).
- Morrissey, D., O'Sullivan, G. C., Tangney, M. Tumour targeting with systemically administered bacteria. Curr. Gene Ther. 10, 3-14 (2010).
- Collins, S. A., et al. Viral vectors in cancer immunotherapy: which vector for which strategy. Curr. Gene Ther. 8, 66-78 (2008).
- Yu, Y. A., Zhang, Q., Szalay, A. A. Establishment and characterization of conditions required for tumor colonization by intravenously delivered bacteria. Biotechnol. Bioeng. 100, 567-578 (2008).
- Baban, C. K., Cronin, M., O'Hanlon, D., O'Sullivan, G. C., Tangney, M. Bacteria as vectors for gene therapy of cancer. Bioeng. Bugs. 1, 385-394 (2010).
- Cronin, M., et al. Orally administered bifidobacteria as vehicles for delivery of agents to systemic tumors. Mol. Ther. 18, 1397-1407 (2010).
- van Pijkeren, J. P., et al. A novel Listeria monocytogenes-based DNA delivery system for cancer gene therapy. Hum. Gene Ther. 21, 405-416 (2010).
- Ahmad, S., et al. Induction of effective antitumor response after mucosal bacterial vector mediated DNA vaccination with endogenous prostate cancer specific antigen. J. Urol. 186, 687-693 (2011).
- Riedel, C. U., et al. Improved luciferase tagging system for Listeria monocytogenes allows real-time monitoring in vivo and in vitro. Appl Environ Microbiol. 73, 3091-3094 (2007).
- Cheng, C. M., et al. Tumor-targeting prodrug-activating bacteria for cancer therapy. Cancer Gene Ther. 15, 393-401 (2008).
- Foucault, M. L., Thomas, L., Goussard, S., Branchini, B. R., Grillot-Courvalin, C. In vivo bioluminescence imaging for the study of intestinal colonization by Escherichia coli in mice. Appl. Environ. Microbiol. 76, 264-274 (2010).
- Collins, S. A., Hiraoka, K., Inagaki, A., Kasahara, N., Tangney, M. PET Imaging For Gene & Cell Therapy. Curr. Gene Ther. , (2012).
- Tangney, M., Francis, K. P. In vivo Optical Imaging in Gene & Cell Therapy. Curr. Gene Ther. , (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon