Method Article
Une méthode de surveillance abordable VIH-1 pour la résistance aux médicaments à ressources limitées
Dans cet article
Résumé
tests de résistance des médicaments pour le VIH-1 individus infectés échec d'un traitement antirétroviral (ART) peut guider les futurs traitements et d'améliorer les résultats du traitement. Optimisation des individus et des populations résultats pour la santé de la prévalence élevée du VIH, mais les pays à ressources limitées finira par exiger le génotypage de la résistance aux médicaments abordables et accessibles et méthodes d'interprétation.
Résumé
VIH-1 de la pharmacorésistance a le potentiel de compromettre sérieusement l'efficacité et l'impact de la thérapie antirétrovirale (ART). Comme les programmes de traitement antirétroviral en Afrique sub-saharienne continuent à se développer, les personnes sous ARV doivent être étroitement surveillés pour l'émergence de la résistance aux médicaments. La surveillance de la résistance aux médicaments transmis pour suivre la transmission de souches virales déjà résistantes à l'ART est également critique. Malheureusement, les tests de résistance aux médicaments n'est pas encore facilement accessibles en situation de ressources limitées, car le génotypage est coûteux et nécessite laboratoire sophistiqué et l'infrastructure de gestion des données. Une méthode génotypique d'accès ouvert de surveillance de la résistance aux médicaments de gérer des individus et évaluer la résistance aux médicaments transmis est décrite. La méthode utilise un logiciel libre et open source pour l'interprétation des profils de résistance aux médicaments et la génération de rapports individuels des patients. Le protocole de génotypage a un taux d'amplification supérieur à 95% pour les échantillons de plasma avec avcharge IRAL> 1000 VIH-1 copies d'ARN / ml. La sensibilité diminue de manière significative pour les charges virales <copies 1000 ARN VIH-1 / ml. La méthode décrite ici a été validé contre une méthode de VIH-1 de la pharmacorésistance tests approuvés par la Food and Drug Administration (FDA), la méthode de génotypage ViroSeq. Limites de la méthode décrite ici comprennent le fait qu'il n'est pas automatique et qu'elle a également échoué pour amplifier la forme circulante recombinante CRF02_AG partir d'un panneau de validation d'échantillons, bien qu'il amplifie les sous-types A et B d'un même panneau.
Introduction
L'épidémie de VIH en Afrique australe a connu une évolution rapide 1 avec une augmentation exponentielle concomitante chez les personnes sous traitement antirétroviral (ART), en particulier en Afrique du Sud 2, 3. Comme preuve de l'impact épidémiologique des programmes de traitement à grande échelle pour réduire l'incidence 4 et l'augmentation de l'espérance de vie dans les pays à ressources limitées (RLS) 5 continue à s'accumuler, les efforts visant à accroître la couverture du traitement antirétroviral seront intensifiés. L'évolution des lignes directrices à l'utilisation du traitement comme outil de prévention 6, 7 au titre des programmes de test et traitement signifie que le nombre absolu de personnes sous traitement va encore augmenter. Un grand nombre de personnes seront sous ARV pendant de longues périodes de temps que l'espérance de vie moyenne des individus sur ART se rapproche de celui de la population non infectée du VIH 8. Le développement et la transmission de la pharmacorésistance du VIH a Always été considéré comme une menace pour les réalisations de l'art 9-12. Ainsi, il est nécessaire pour la surveillance et le suivi de la résistance aux médicaments plus rigoureux que plusieurs personnes sont engagées sur ART.
Tester la résistance aux médicaments génotypique (GRT) a été utilisé avec succès dans les pays développés, à la fois pour la surveillance ainsi que la surveillance du VIH-1 la résistance aux médicaments chez les personnes recevant un traitement antirétroviral. Dans ces contextes, GRT a été intégrée dans le continuum de soins pour le VIH-1 individus infectés. La plupart des directives internationales recommandent TJB pour les patients adultes ou pédiatriques défaut ART (première ligne et de deuxième ligne) 13-15, les patients de pédiatrie exposés à la prévention de la transmission mère-enfant (PTME), mais par la suite les régimes infectés 16, et dans les endroits où hauts niveaux de résistance aux médicaments transmis entre les individus présentant une infection aiguë 13-15. Toutefois, les exigences de coûts, de la technologie et de l'infrastructure ont limité la miseœuvre des approches similaires à la surveillance de la résistance aux médicaments dans le SJSR.
Le traitement du VIH d'Afrique du Sud et des lignes directrices de suivi ne recommande pas actuellement l'utilisation de GRT dans le choix de guidage de l'ART pour les personnes défaillantes schémas de première intention 17. Les individus sont mis basée principalement sur virologique (ARN VIH-1 charge virale) paramètres. Cependant en 2012, l'Afrique australe cliniciens du VIH Société a publié les premières lignes directrices 18 tests de résistance aux médicaments ARV Afrique du Sud. Ces lignes directrices recommandent de tester GRT pour tous les adultes à défaut de première ligne et de deuxième intention et pour les nourrissons infectés et les enfants exposés à la PTME 18. Cependant, GRT n'est pas recommandé pour les personnes de 18 infection aiguë, car il n'existe aucune preuve réelle des niveaux élevés de résistance aux médicaments transmis dans le sud de l'Afrique 19-29. Il est prévu que certaines de ces recommandations seront intégrés au fil du temps dans le tre nationalatment et suivi les directives des différents pays de la région. Déjà, en 2013 des lignes directrices de traitement en Afrique du Sud il ya maintenant recommandation du GRT au moment de l'échec de deuxième ligne pour les adultes et au moment de la première ou de deuxième ligne échec thérapeutique à base d'IP pour les enfants 30.
Il a été démontré que l'incorporation de GRT dans les directives de traitement en Afrique du Sud serait potentiellement coût-neutre. Compte tenu du coût des médicaments de deuxième ligne de traitement qui sont relativement plus chers que les médicaments de première ligne, en utilisant GRT pour identifier les patients qui ont vraiment besoin d'être passé à un traitement de deuxième ligne n'entraînera pas de coût supplémentaire pour le programme. En outre, GRT peut également identifier d'autres raisons de l'échec, de conserver les options de traitement et de générer des informations sur les profils de résistance émergents 31. Par conséquent, il est nécessaire de réduire le coût des méthodes de surveillance de la résistance aux médicaments encore plus loin afin d'améliorer l'accès, la qualité des soins d'und résultats.
Ici, nous présentons une méthode de GRT conçu pour utiliser génériques (open source) des amorces pour la transcription inverse, la réaction en chaîne par polymérase (PCR) et le séquençage (tableau 1), ainsi que les logiciels open source pour la plupart de la pharmacorésistance interprétation. Pour la gestion clinique, le protocole est complété par un examen et un signalement méthode complète avec interprétation spécialiste du rapport de la résistance aux médicaments de laboratoire avec un strict respect des directives nationales de traitement. Le protocole est divisé en quatre composantes différentes: 1) acide ribonucléique du VIH (ARN) de l'extraction, 2) la transcription inverse et Polymerase Chain Reaction (PCR) de cibles virales, 3) séquençage et 4) les méthodes de bio-informatique pour l'analyse des chromatogrammes, alignement, curation et l'interprétation des données de séquence.
Protocole
Une. Acide éthylènediaminetétraacétique (EDTA) Tout traitement du sang
Remarque: Le sang peut être traitée immédiatement après la collecte de peut être conservé à 4 ° C pendant plus de 24 h.
- Travailler dans une enceinte de sécurité biologique, permettent à l'ensemble de l'échantillon de sang EDTA pour atteindre la température ambiante.
- Pour chaque échantillon, étiqueter suffisamment cryotubes avec l'identification de l'échantillon (ID), la matière de stockage (de plasma), et la date.
- Centrifuger les échantillons pendant 10 min à 1000 x g. Ne pas utiliser les freins pour arrêter la centrifugeuse. Cela donnera trois couches (de haut en bas): plasma, les leucocytes (de la couche leucocytaire) - une couche très mince - et les érythrocytes, y compris les plaquettes.
- Aspirer délicatement le surnageant (plasma) et aliquote de 500 ml dans chaque tube cryogénique. Prenez soin de ne pas perturber la couche de cellules (couche leuco-plaquettaire) ou transférer des cellules.
- Stocker à -80 ° C jusqu'au moment de l'extraction de l'ARN ou de procéder à l'extraction d'ARN immédiatement.
- Préparer une feuille de calcul d'extraction avec les ID des échantillons à extraire y compris les contrôles de plasma positifs et négatifs.
- Pour chaque échantillon à extraire, étiqueter un tube de microcentrifugation stérile de 1,5 ml avec l'ID de l'échantillon, la date d'extraction et "ARN". Étiqueter également un tube de colonne et collecte assemblés ainsi que d'un tube de microcentrifugation de 2 ml contenant une solution de lyse de travail avec des nombres correspondants de la feuille de travail d'extraction.
- Travailler dans le cabinet Bio-sécurité, ajouter 200 ul d'échantillon dans le tube à centrifuger de 2 ml correspondant de solution de travail de lyse.
- Vortex puits et incuber pendant 10 min à température ambiante.
- Après 10 min, centrifuger brièvement le tube.
- Ajouter 800 ml d'éthanol absolu pour chacun des tubes.
- Mélanger au vortex d'impulsion et centrifuger brièvement.
- Transférer 600 pl de cette solution à l'ensemble de tube de colonne / collection correspondante. Centrifuger à 6000 xg pendant 1 min.
- Transférer la colonne dans un nouveau tube collecteur et jeter l'ancien tube collecteur contenant l'effluent. Répéter deux fois plus l'étape précédente 2.8 (ci-dessus).
- Ajouter 500 ul de tampon AW1 de lavage à chaque colonne et centrifuger à 6000 g pendant 1 min.
- Jeter le tube de filtrat et la collecte et le transfert de la colonne dans un nouveau tube de collecte.
- Ajouter 500 pi était AW2 tampon et centrifuger à 20 000 g pendant 3 min. Répétez l'étape 2.11.
- Centrifuger dans un nouveau tube de collecte à 20 000 g pendant 2 min.
- Jeter colonne filtrat et dans 1,5 ml microtube.
- Ajouter 60 ul de tampon AVE (RNase de l'eau libre) au milieu de la colonne assurer que vous n'avez pas distribuer le liquide sur le côté de la colonne.
- Incuber à température ambiante pendant 1 min.
- Centrifuger à 6000 g pendant 2 min.
- Jeter la colonne et plafonner les 1,5 ml microtubes.
- Les échantillons sont maintenant prêts pour revertranscription soi.
- Si le test doit être effectué immédiatement, conserver à 4 ° C pendant 6 h. Toutefois, si le test est d'être retardé puis placer à -80 ° C immédiatement. NB: ne pas congeler / décongeler les échantillons de plus de 3x.
3. Préparation des réactifs pour la transcription inverse
- Avant de commencer, calculer les volumes de chacun des réactifs nécessaires pour le nombre d'échantillons en cours de traitement, y compris les contrôles de plasma positifs et négatifs. Ajoutez également un contrôle réactif.
- En utilisant les volumes calculés à partir de l'étape 3.1 (ci-dessus), préparer le désoxyribonucléotide triphosphate (dNTP) amorce mélange dans un tube propre, stérile 200 pi PCR suivie par une brève impulsion vortex. Chaque échantillon devrait avoir 0,5 pi de l'amorce inverse RT21 et 0,5 pi de dNTP, voir tableau 2.
- Aliquote de 1,0 ul du mélange de dNTP amorce de tubes de PCR de 200 pi.
- Préparer la transcriptase inverse (RT) de mélange enzymatique en ajoutant 1 μ; L de tampon de transcription inverse 10x, 1 pi de 0,1 M de DTT et 2 ul de 25 mM de MgCl 2 à un tube stérile, suivi par un vortex et centrifuger brièvement, voir le tableau 3.
- Ajouter 0,5 pi de chacun des enzymes RNAseOUT et Exposant III transcriptase inverse pour le tube enzyme de mélange puis appuyez doucement le tube pour mélanger.
- Gardez les tubes avec les mélanges de dNTP amorce et mélange d'enzymes sur un bloc froid et passer à la station d'ARN.
4. Transcription inverse
- Ajouter 6 ul de l'échantillon d'ARN dans le tube de mélange de dNTP-amorce suivie par un bref vortexage pour mélanger.
- Après l'ajout de l'ARN, passer à la salle PCR avec deux dNTP / amorce / ARN et RT mélange d'enzymes mélanger des tubes sur un bloc froid ou de la glace.
- Centrifuger brièvement les tubes dNTP / amorce / ARN (mélange de l'étape 4.2) et les placer dans un thermocycleur.
- On chauffe à 65 ° C pendant 5 minutes pour dénaturer l'ARN.
- Refroidir rapidement à 4 ° C, maintenir pendant 2min.
- Pause le thermocycleur tout à 4 ° C; prendre les tubes.
- Ajouter rapidement 5 pi du mélange d'enzymes tout en gardant les tubes sur un bloc froid.
- Mélanger doucement en appuyant sur le tube puis centrifuger brièvement les tubes et revenir à l'thermocycleur.
- Maintenir les tubes à 50 ° C pendant 60 min pour inverser transcrire l'ARN, suivie par la dénaturation enzymatique à 85 ° C pendant 5 minutes pour arrêter la transcription inverse.
- Refroidir à 37 ° C. Dès la température est de 37 ° C, faire une pause et prendre le métro de la thermocycleur.
- Ajouter rapidement 0,5 pi de RNAse H pour les tubes et revenir à l'thermocycleur.
- Maintenir à 37 ° C pendant 20 min, puis refroidir à 4 ° C.
- L'ADN complémentaire (ADNc) peut être utilisé immédiatement ou peut être stocké à -20 ° C ou moins jusqu'à ce que nécessaire. Cependant, le stockage à long terme de l'ADNc devrait être à -80 ° C.
5. Préparation des réactifs pour la PCR
- Avare de départ, calculer les volumes de chacun des réactifs nécessaires pour le nombre d'échantillons en cours de traitement et les témoins. En plus des trois contrôles (positif, négatif, et réactifs), vous pouvez également ajouter un contrôle PCR (ADN du VIH). Les premier et second mélanges pour PCR rondes peuvent être préparés en même temps et le deuxième mélange maître stockés à -20 ° C jusqu'à utilisation. Mixages peuvent être enregistrés pendant environ 8 heures.
- Ajouter 18,4 ul d'eau, 2,5 ul de tampon 10 x, 1,0 ul de MgCl2, 0,5 ul de dNTP, et 0,25 ul de chacune des amorces, comme indiqué sur le tableau 4 et le vortex.
- Ajouter 0,1 ul de Platinum Taq polymérase (5U/μl) et mélanger délicatement le tube en appuyant dessus.
- Aliquotes 23 pi de mélange maître de tubes de PCR 200 pi.
- Avec les tubes de Master Mix sur un bloc froid ou mouvement de la glace de la salle de PCR.
6. PCR nichée
- Ajouter 2 ul de l'ADNc de 23 pi de la Maste de PCR 1er tourr mélanger.
- Fermer les tubes, placer les échantillons dans le thermocycleur et en utilisant les PCR conditions de cyclage suivantes: 94 ° C pendant 2 min, 30 cycles de 95 ° C pendant 30 sec, 58 ° C pendant 20 sec, et 72 ° C pendant 2 min, suivie par une extension finale à 72 ° C pendant 10 min, comme indiqué sur la figure 1.
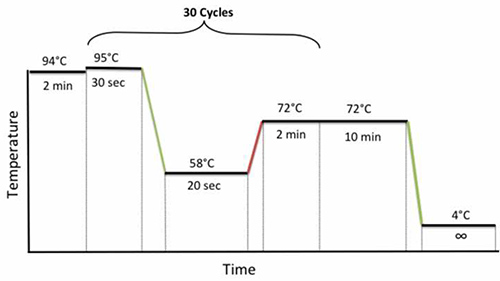
Figure 1. PCR nichée conditions de cyclage. Cliquez ici pour agrandir l'image.
- Continuer à l'étape 2e tour PCR ou stocker les produits de PCR 1er tour à -20 ° C ou moins jusqu'à ce que nécessaire à un stade ultérieur.
- Pour 2e cycle de PCR, ajouter 2 pl du produit PCR 1er tour à 23 pi de la 2e ronde PCR Master Mix und utiliser le même programme PCR sur la figure 1.
7. Gel d'électrophorèse
- Préparation de gel
- Ajouter un comprimé de 0,5 g d'agarose dans un ballon de 250 ml en verre et ajouter 50 ml de tampon TBE 1x dans le ballon.
- Chaleur au micro-ondes jusqu'à ébullition; agiter fréquemment (environ toutes les 30 secondes) jusqu'à ce que complètement solubilisé. Utiliser une poignée de silicone ou four gant pour saisir le ballon chaud. La solution d'agarose peut bouillir sur le ballon très facilement pour suivre de près ce processus.
- Refroidir à la température ambiante pendant environ 10 min.
- Verser agarose dans un plateau de coulage de gel contenant peigne de taille appropriée; gel prêt à l'emploi dans environ 20-30 min.
- Placez gel dans la chambre d'électrophorèse et de fonctionner comme recommandé par le fabricant.
- L'électrophorèse sur gel et la visualisation.
- Vortex Novel jus pendant 10 secondes avant de l'utiliser.
- Diluer 1 pl de jus Novel avec 5 pi de DÉchantillon NA et mélange.
- Diluer 3 pi de jus Novel avec 3 pi de marqueur de poids moléculaire et mélanger.
- Chargez les mélanges de sections 7.2.2 et 7.2.3 (ci-dessus) et exécutez le gel à 100 V et 400 mA pendant 40 min à évaluer l'amplification par PCR.
- Amplification positif peut être visualisé sous lumière UV 1315 fragment de pb, Figure 2.
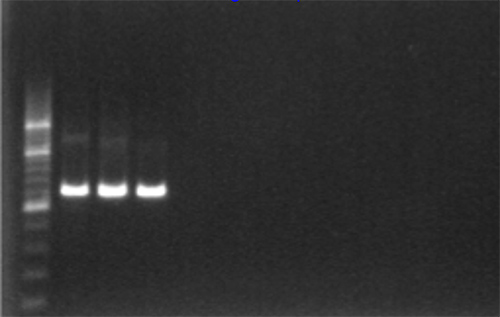
Figure 2. confirmation du gel de l'amplification PCR en utilisant 1% d'agarose électrophorèse sur gel et un pb échelle de 200. Cliquez ici pour agrandir l'image.- Il devrait y avoir aucune amplification dans les contrôles négatifs et de réactifs, ce qui indique l'absence de contamination.
8. PCR Nettoyage du produit
- En prévision de la réaction de séquençage, les produits de PCR positifs second tour sont nettoyés en utilisant le kit de purification PureLink PCR.
- Ajouter 80 pi de tampon de liaison de travail haut de coupure (B3) à 20 pi de produit de PCR et pipette mélange.
- Ajouter l'échantillon mélangé avec le tampon de liaison à une colonne de centrifugation dans un tube de collecte.
- Centrifuger la colonne à 10 000 x g pendant 1 min. Transférer la colonne dans un nouveau tube de collecte.
- Laver la colonne avec 650 pi de tampon de lavage avec de l'éthanol.
- Centrifuger la colonne à 10 000 x g pendant 1 min. Transférer la colonne dans un nouveau tube de collecte.
- Centrifuger la colonne à la vitesse maximale pendant 2-3 min pour enlever toute solution de lavage résiduelle.
- Placer la colonne spin dans un tube de 1,7 ml d'élution propre fourni avec le kit.
- Ajouter 40 ul de tampon d'élution à l'axe de la colonne et la colonne à incuber chambre température pendant 1 min.
- Centrifuger la colonne à la vitesse maximale pendant 2 min (> 10 000 xg).
- Le tube d'élution contient votre produit PCR purifié prêt pour le séquençage. Jeter la colonne.
- Déterminer la concentration et de la qualité de l'ADN en utilisant un Nanodrop.
- Si aucun équipement dans la maison de séquençage sont disponibles, les produits de PCR purifiés peuvent être envoyés à un laboratoire de séquençage commercial à ce stade.
9. Séquençage Réactions
- Les produits de PCR sont séquences en utilisant le gros version kit de colorant terminateur 3,1 et 4 amorces pour chaque échantillon (deux avant et deux arrière). Les séquences des amorces sont présentées dans le tableau 2. Par conséquent, après l'exécution de séquençage, chaque échantillon aura quatre séquences à assembler dans un contig.
- Mettre en place des réactions de séquençage, comme indiqué dans le tableau 5 pour chacune des quatre amorces.
- Mélanger le tampon de séquençage et les amorces au vortex avant utilisation.
- Mélanger lade l'eau, un tampon et de l'amorce avant l'addition de la grande séquençage de colorant. Mélanger au vortex.
- Mélanger délicatement le mélange maître après l'ajout du grand séquençage mélange de colorant en inversant le tube ou le tapotant.
- Aliquote 9 ul du mélange maître dans une plaque optique 96 puits.
- Afin d'exécuter 24 échantillons / plaque, mettre en place la plaque comme indiqué ci-dessous la figure 3.
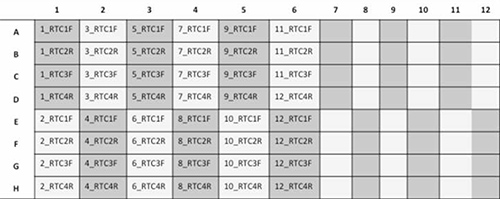
Figure 3. représentation de Schéma d'une plaque de 96 puits avec 12 échantillons de patients séquencés avec 4 amorces chacune (RTC1F, RTC2R, RTC3F, et RTC4R). Cliquez ici pour agrandir l'image.
- Ajouter 1,0 ml de l'échantillon d'ADN (~ 20-40 ng), couvrir la plaque avec unn adhésif couvercle en aluminium, puis mélanger délicatement il.
- Centrifuger à 3000 g pendant 1 min. Retirez le couvercle en aluminium et ajouter une natte d'étanchéité en caoutchouc.
- Placer la plaque sur le thermocycleur et lancer le programme de cyclisme suivante montre la figure 4.
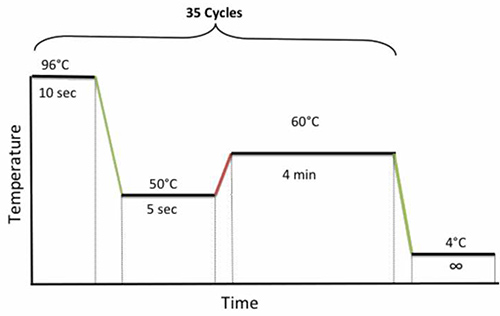
Figure 4. PCR conditions de cyclage pour le séquençage. Cliquez ici pour agrandir l'image.
- Lorsque la PCR terminée, nettoyer le produit de séquençage immédiatement.
10. Séquençage nettoyage
- Pour chaque réaction de séquençage, mélanger 50 ul d'éthanol absolu et 5 ul de 3 M d'acétate de sodium.
- En utilisant une pipette multicanaux, 55 &# 956; l de la solution d'acétate de sodium / EtOH à chaque puits.
- Sceller les puits avec couvercle adhésif en aluminium, veiller à ce que chaque puits est scellé correctement.
- Centrifuger à 3000 g pendant 20 min.
- Après 20 min, retirez le couvercle et retourner la plaque, dans un mouvement fluide, sur un tissu de laboratoire plié (NE PAS frapper pour se débarrasser de surnageant que ce sera déloger le culot!).
- Centrifuger la plaque retournée sur le même tissu à 150 g pendant 2 min.
- Immédiatement ajouter 150 ul froid EtOH à 70%. NE PAS retarder l'addition d'éthanol à cette étape.
- Sceller avec le même couvercle adhésif en aluminium et vortex.
- Centrifuger à 3000 g pendant 5 min.
- Inversez plaque sur un nouveau tissu plié et centrifugeuse inversé à 150 xg pendant 1 min.
- Après la centrifugation, placez découvert dans thermocycleur et le sécher à 50 ° C pendant 2 min.
- Une fois les plaques est sec, sceller avec de la colle couvre de feuilles, envelopper dans du papier et conserver à -20 ˚ C jusqu'au moment de procéder wie Les électrophorèse de séquençage.
- Lorsque vous êtes prêt à la séquence, dissoudre produits de séquençage nettoyés dans 10 ml Salut-Di formamide, dénaturer et la charge pour l'électrophorèse.
11. Bioinformatique
- Assemblée séquence
- Lancez le programme Geneious.
- Créez un dossier de travail pour stocker les séquences.
- Importez les fichiers ABI générés par la machine de séquençage dans le dossier de travail en utilisant l'outil d'importation. Geneious allouera pourcentage score de qualité pour chaque séquence importée.
- Séquences ouvertes avec des scores de qualité> 70% en double-cliquant sur eux.
- Chaque fichier doit s'ouvrir dans une nouvelle fenêtre. Le logiciel va indiquer la qualité à chaque position de nucleotide du chromatogramme de la qualité de la séquence en utilisant des barres bleu clair. Plus la barre est, meilleure est la qualité de l'appel de base.
- L'aide du curseur, sélectionnez la section du milieu de la séquence en laissant les extrémités, qui sont généralement de mauvaise qualité.
- Cliquez sur le bouton d'extraction pour extraire la région avec la séquence de bonne qualité.
- Sélectionnez les quatre séquences extraites pour chaque échantillon et les assembler contre une séquence de référence.
- Inspectez la séquence assemblée de s'assurer que vous êtes dans le cadre de lecture correct. Si vous êtes dans le cadre de lecture correct, le début de la protéase doit commencer par les acides aminés suivants: PQITLW. Le début de la RT va commencer avec PISPIE.
- Extrait de la région contiguë couvrant le début de relations publiques pour le 300e RT codon. Au cours de ce processus, consultez aussi pour des insertions ou des suppressions.
- Passez par la séquence consensus du contig extrait, identifier les ambiguïtés et vérifier les positions avec des bases mixtes en inspectant la qualité (symétrie, la hauteur, le fond et les épaules des régions adjacentes) des appels de base.
- Sélectionnez la séquence consensus et cliquez sur le bouton d'extraction pour créer un fichier distinct de la séquence consensus des quatre amorces et Labél de manière appropriée.
- Exporter la séquence dans un dossier de stockage de sauvegarde sur l'ordinateur ou un dossier réseau.
- Séquence évaluation de la qualité (HIVDB)
- Analyser la séquence en utilisant le programme HIVDB à http://hivdb.stanford.edu .
- Vérifier les suppressions et les insertions dans les données de synthèse et de s'assurer que la séquence couvre tous les 99 protéase (PR) codons et les 1er 300 codons RT.
- Vérifiez la présence souligné assurance de la qualité (AQ) des questions dans les deux régions PR et RT, tels que des codons stop, des décalages de cadre, des positions ambiguës et des résidus inhabituels.
- Contrôle de qualité séquençage
- Soufflez la nouvelle séquence contre une base de données de séquence locale de l'exécution précédente.
- Si la nouvelle séquence est> 97% semblable à une séquence dans la base de données, toutes les étapes du protocole devraient être examinés, à commencer par l'analyse de la séquence et revenir à l'extraction de l'ARN pour assue qu'il n'y avait pas les confusions (commutation de l'échantillon, les erreurs d'étiquetage) ou de contamination.
- Si aucun problème n'est constaté, répéter l'analyse de l'ancien et de nouveaux échantillons de la phase d'extraction d'ARN.
- Si les séquences sont toujours> 97% semblable, consulter l'historique du patient pour évaluer pour tout lien épidémiologique entre les individus.
- Analyse phylogénétique
- Aligner toutes les séquences de la base de données en utilisant le programme ClustalW dans Geneious.
- Vérifier manuellement l'alignement de séquences non alignées, des suppressions et des insertions et modifier en conséquence.
- Construire un arbre phylogénétique en utilisant PhyML, Geneious arbre constructeur ou d'autres constructeurs d'arbres dans Geneious.
- Examinez l'arbre pour les échantillons avec des longueurs de dérivation courts.
- Passez en revue les échantillons avec des longueurs de branches courtes de contamination possible.
12. REGA DB Informatique
- Séquence chargement
- Connectez-vous au RegAdB en utilisant un nom d'utilisateur et mot de passe.
- Dans le menu déroulant, sous Patient ID, sélectionnez "Commence par".
- Ajouter l'identification du patient et sélectionnez la personne dont le génotype est à télécharger.
- Dans le menu à gauche, sélectionnez "isolat viral".
- Dans les options sous isolat viral sélectionner "ajouter".
- Entrez la date de l'échantillon, ID échantillon, ID de séquence et la date de la séquence.
- Sélectionnez «Choisir le fichier», puis accédez au fichier fasta de la séquence à transférer.
- Après avoir sélectionné le fichier fasta à être transféré, cliquez sur téléchargement.
- Une fois la séquence téléchargé apparaît dans la boîte de nucléotides dans les identifie et les dates séquence, cliquez sur le bouton OK en bas à droite de la fenêtre.
- Vérifiez l'alignement de protéines PR et RT en cliquant sur la protéine et en sélectionnant soit PR ou RT.
- Vérifiez la mutation de résistance aux médicaments en cliquant sur le bouton de résistance. Cela donnevous les profils de résistance des trois algorithmes: ANRS, Stanford HIVDB et RegaDB.
- La génération de rapports à l'aide REGA
- Connectez-vous au RegaDB en utilisant votre nom d'utilisateur et mot de passe.
- Dans le menu déroulant, sous Patient ID, sélectionnez "Commence par".
- Ajouter l'identification du patient et sélectionnez la personne dont le rapport doit être généré.
- Dans le menu de droite, sélectionnez isolat viral.
- Dans les options sous isolat viral cliquez sur "voir".
- Double-cliquez sur l'isolat viral pour lequel vous souhaitez créer un rapport.
- Dans la fenêtre isolat viral, cliquez sur l'onglet virale rapport isolat.
- Sélectionnez les algorithmes pour l'interprétation du génotype du menu déroulant, puis sélectionnez modèle de rapport à utiliser.
- Une fois l'algorithme et le modèle sont sélectionnés, cliquez sur le bouton "Générer".
- Télécharger le document rtf généré.
- Ouvrez le rtf fairecument comme un document Word.
- Redimensionner le graphique de l'historique du traitement.
- Après le tableau, ajouter la section «tableau clinique et la résistance interprétation".
- En utilisant les données du tableau de la résistance et le tableau clinique, ajouter une description du profil de résistance du patient à partir de l'historique de traitement du patient, et les médicaments auxquels l'isolat viral est résistante. Ajouter également une description de la charge virale du patient et CD4 + profils de comptage cellulaire du tableau.
- Envoyer le rapport sur les maladies infectieuses (ID) spécialisés pour avis et des recommandations sur la gestion future du patient. Ce processus est également une étape d'assurance de la qualité très importante. Toute erreur dans le génotype ou incohérences dans l'histoire de traitement, profils virologiques et immunologiques peuvent être identifiées et examinées avant qu'un rapport final est envoyé, avec toutes les recommandations, au clinicien la gestion du patient.
Résultats
La méthode validée est une modification d'un procédé précédemment rapporté 20. Le procédé de génotypage ViroSeq, qui a été approuvé par la FDA, a été utilisé comme la méthode de référence dans la validation. Un panel d'échantillons d'essais d'aptitude obtenues auprès des Agences nationales françaises pour la recherche sur le sida et les hépatites virales (ANRS) a été utilisé dans la comparaison primaire entre les deux méthodes. Les deux méthodes de génotypage ont été de 100% dans l'identification concordante toutes les mutations associées à la résistance des médicaments cliniquement importants tel qu'il est interprété par le programme HIVDB pour les échantillons qui ont été amplifiés avec succès par les deux méthodes. Comme le montre le tableau 6, les séquences nucléotidiques des trois paires sont identiques à 99,5%. Les séquences d'acides aminés prédites sont identiques à 100%. Un échantillon de cinq n'a pas pu être amplifié avec succès par ViroSeq. En plus de l'échantillon non amplifié par ViroSeq, le procédé à l'interne n'a pas réussi à amplifier un second échantillon qui a été montréêtre un virus recombinant de circulation (CRF02_AG) par ViroSeq. Les trois échantillons qui ont amplifié avec les deux méthodes ont été sous-type B (deux échantillons) et le sous-type A (un échantillon).
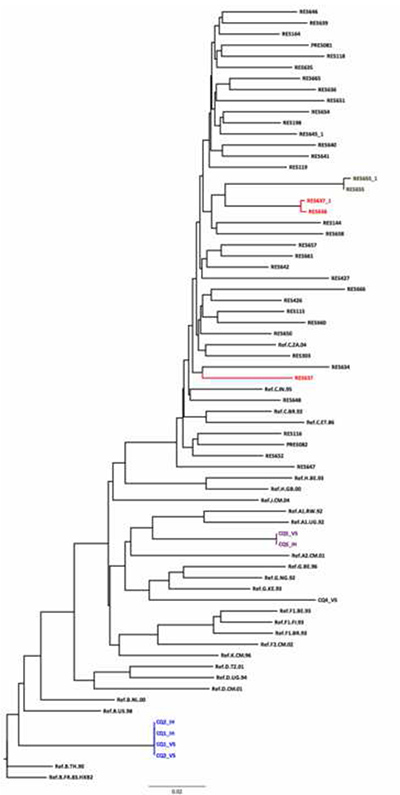
Figure 5. Utilisation d'un voisin de démarrage arbre HKY fait dans le cadre de l'assurance qualité de la séquence. Il ya quatre paires / groupes de séquence avec les distances génétiques très courts. La distance génétique entre RES655 et RES655_1 (mêmes échantillons séquencés sur plusieurs jours) est de 0,003. L'erreur est un potentiel de la paire de RES637_1/RES638 que leur distance génétique est trop courte (0,075) pour les échantillons provenant de différents individus non couplés sur le plan épidémiologique. Il existe une autre RES637 sur l'arbre avec une distance de 0,075 par rapport à RES638_1. La grappe CQ01/CQ02 suggère que les deux échantillonsà partir du panneau sont des copies de la même échantillon. Ils se regroupent avec la séquence de référence sous-type B confirme le sous-type attribué par l'outil REGA sous-typage. CQ05 CQ04 et regroupés avec les sous-types A et G, respectivement, alors que l'outil REGA de sous-typage entre eux dans la catégorie A et CRF02_AG respectivement. Un autre outil utile pour le sous-typage et la recombinaison du VIH est SCUEL, qui est disponible à http://www.datamonkey.org. Cliquez ici pour agrandir l'image.
Un panel de cinq échantillons a été utilisée pour évaluer la précision de la méthode en interne. Dix génotypes réplicats ont été générées pour chacun des cinq échantillons. Utilisation de l'analyseur génétique 16 capillaire 3130xl, 48 des 50 génotypes ont été générés à partir de 24 pistes, préparés le jour même. Pour tous les cinq échantillons, les séquences d'acides aminés prédites sont de 100% concordant entre les répétitions. Pour les séquences d'acide nucléique, eavant était> 99% de similarité par paires.
Au cours des deux premières années de l'utilisation de ce procédé, soixante échantillons ont été répétés de manière aléatoire à partir de l'extraction d'ARN à un séquençage. Il n'y avait pas de différences statistiquement significatives entre le score de qualité de la séquence et le nombre de bases mixtes entre les répétitions. Tant le nucléotidiques et d'acides aminés pour des comparaisons par paire des soixante paires sont supérieure à 99% identique. Ainsi, les mutations de résistance aux médicaments pour toutes les paires concordantes sont de 100%.
Réduction des coûts
Les volumes de réaction de RT, la PCR et le séquençage ont été réduites d'au moins la moitié, par rapport à la méthode d'origine 20, 32, sans compromis sur la qualité des séquences générées. Cela a permis une réduction de coût de 50% pour les étapes de RT et de PCR.
La nouvelle méthode a été initialement conçu pour fonctionner avec six amorce de séquençage de la séquence les 99 codons du gène de la protéase et les 300 premiers codons du gène de la transcriptase inverse 20, 32. Des méthodes similaires utilisent également six à huit amorces 33, 34. Certaines méthodes récemment publiées ont utilisé moins de six amorces, même si parfois, le séquençage de la protéase et des gènes RT seprately 35, 36. Nous avons cherché à réduire le nombre d'amorces de séquençage de six à quatre, (figure 6)
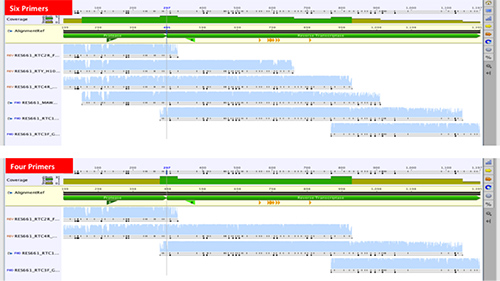
Figure 6. La comparaison des séquences contiguës à partir de six contre quatre amorces de séquençage pour la génération de la séquence de pol 1197 pb couvrant l'ensemble des 99 codons du VIH-1 de la protéase et les 300 premiers codons du gène de la transcriptase inverse.242/51242fig6highres.jpg "target =" _blank "> Cliquez ici pour agrandir l'image.
Des séquences provenant d'un ensemble de 17 échantillons générés à partir de six amorces ont été comparées à des séquences générées après exclusion des deux amorces (MAW46 et RTY). Les sous-types étaient 14 sous-type C, deux sous-type B, et un sous-type A. Il n'y avait pas de différences significatives dans les scores de qualité de la séquence. Là encore, l'identité par paires entre la moyenne des 17 paires d'acides nucléiques est de 99% et de 100% au niveau des acides aminés. Ainsi, en réduisant les amorces de séquençage de six à quatre ont donné lieu à une réduction du coût de mise en séquence de presque un tiers.
Le seul outil logiciel propriétaire utilisé dans ce protocole était Geneious pour l'assemblage de la séquence. Les outils résistance aux médicaments d'interprétation, ainsi que le rapport des outils génération sont tous des outils gratuits en libre accès. Cela réduit le coût en outre en éliminant les coûts associés à l'utilisation d'un logiciel propriétaire. En outre, collectivistee la négociation a permis aux réactifs de ce protocole à être emballés dans un kit pour faciliter l'accès de Life Technologies et est disponible comme les Technologies Saturne / Vie méthode 37 génotypage. En outre, les membres peuvent accéder à Saturne réactifs à un prix réduit.
Cadre clinique
Le protocole décrit a été mis en œuvre dans le suivi et la surveillance de la résistance aux médicaments dans une communauté rurale du KwaZulu-Natal. Un total de 604 génotypes ont été générées à partir d'échantillons cliniques entre Décembre 2010 et mai 2013 à un taux d'amplification de 95% pour les échantillons ayant une charge virale> 1000 copies d'ARN / ml. Cette étude clinique VIH de la résistance aux médicaments a été approuvé par le Comité de recherche d'éthique biomédicale de l'Université de KwaZulu-Natal (réf. BF052/10) et le Comité de recherche en santé du ministère de KwaZulu-Natal de la Santé (réf. HRKM 176/10). Les rapports individuels des patients ont été générés et envoyés vers les cliniquespour la gestion des patients.
Soixante-douze (72) génotypes ont été générées dans le cadre d'une surveillance de transmis étude de la résistance aux médicaments, imbriqués dans une grande étude prospective de surveillance du VIH dans la population. Les échantillons primaires sont tout aiguille piqûre sang recueilli dans des microtubes EDTA. Au génotypage il y avait un taux de 79% 19 d'amplification. Approbation éthique de la génotypage des échantillons de l'étude de surveillance a été obtenue à partir de l'Université du comité d'éthique de la recherche biomédicale KwaZulu-Natal (réf. BE066107).
| Nom Primer | Séquence | Longueur | Direction | Position HXB2 | |
| MAW-26 | TTGGAAATGTGGAAA GGAAGGAC | 23 | Avant | 2028-2050 | 1er tour PCR |
| RT-21 | CTGTATTTCAGCTATC AAGTCCTTTGATGGG | 31 | Inverse | 3539-3509 | 1er tour PCR |
| Pro-1 | TAGAGCCAACAGCCC cacca | 20 | Avant | 2147-2166 | 2e cycle de PCR |
| RT-20 | CTGCCAATTCTAATTC TGCTTC | 22 | Inverse | 3462-3441 | 2e cycle de PCR |
| RTC1F | ACCTACACCTGTCAA CATAATTG | 23 | Avant | 2486-2508 | Séquençage |
| RTC2R | TGTCAATGGCCATTG TTTAACCTTTGG | 27 | Inverse | 2630-2604 | Séquençage |
| RTC3F | CACCAGGGATTAGAT ATCAATATAATGTGC | 30 | Avant | 2956-2994 | Séquençage |
| RTC4R | CTAAATCAGATCCTAC ATACAAGTCATCC | 29 | Inverse | 3129-3101 | Séquençage |
| RT-y | GTGTCTCATTGTTTAT ACTAGG | 22 | Inverse | 2967-2946 | Séquençage |
| MAW-46 | TCCCTCAGATCACTC TTTGGCAACGAC | 27 | Avant | 2251-2277 | Séquençage |
Tableau 1. La transcription inverse, une PCR, des amorces et des produits personnalisés de séquençage utilisées dans la génération d'un fragment de 1197 pb de pol couvrant l'ensemble des 99 codons du VIH-1 de la protéase et les 300 premiers codons du gène de la trascriptase inverse.
| RT21 (5pmol/ml) | 0,5 | 0,2 |
| dNTP (10 mM) | 0,5 | 0,4 |
| Total | 1 |
Tableau 2. dNTP / mélange d'amorces pour la réaction de transcription inverse.
| Réactif | Volume (ml) / réaction | Concentration / réaction |
| First Strand Buffer (10x) | 1 | 1 |
| MgCl 2 (25 mM) | 2 | 4 |
| DTT (0,1 M) | 1 | 0,008 |
| RNaseOUT (40 U / ml) | 0,5 | 16 |
| Exposant III de la transcriptase inverse (200U/ml) | 0,5 | 8 |
| Total | 5 |
Tableau 3. Enzyme de mélange pour la réaction de transcription inverse.
| Réactif | Volume (ml) / réaction | Concentration finale / Réaction |
| Eau traitée au DEPC | 18.4 | - |
| PCR Buffer (10x) | 2.5 | 1 |
| MgCl 2 (50 mM) | 1 | 2 |
| dNTP mix (10 mM) | 0,5 | 0,2 |
| Amorce Froward (5 pmol / ml) | 0,25 | 0,05 |
| Amorce inverse (5 pmol / ml) | 0,25 | 0,05 |
| Platinum Taq polymerase (5 U / ml) | 0,1 | 0,02 |
| Total | 23 | - |
Tableau 4. Master mix pour la PCR nichée.
| Réactif | Volume (ml) / réaction | Concentration / réaction |
| Eau traitée au DEPC | 6.1 | |
| Séquençage tampon (5x) | 2 | 1 |
| Primer (3,2 pmol / ml) | 0,5 | 0,16 |
| Big Dye Terminator séquençage mélange | 0,4 | - |
| Total | 9 |
Tableau 5. Master mix pour les réactions de séquençage.
| ViroSeq | Interieur | NA% de similarité | |||||||
| ID échantillon | Sous | Le niveau de qualité | PR mutations | Mutations RT | Sous | Le niveau de qualité | mutations de relations publiques | RT mutations | |
| CQ01 | B | 99,9 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99,2 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ02 | B | 99,5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99,5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ03 | NA | NA | NA | NA | NA | NA | |||
| CQ04 | CRF02_AG | 98,4 | I54V, V82F, I84V | M41L, L74I, L210W, T215Y, V108I, Y181C | NA | NA | NA | NA | NA |
| CQ05 | A | 99,7 | K103N | A | 93 | K103N | 100 | ||
Tableau 6. Résul comparatifts d'une analyse parallèle entre la méthode de génotypage ViroSeq et la méthode en interne en utilisant un panel d'échantillons fournis par l'ANRS.
Discussion
Plusieurs faible coût des méthodes internes ont été décrits dans les efforts pour tenter de faire génotypage de la pharmacorésistance du VIH plus abordables 33, 34, 36. Il ne fait aucun doute de la nécessité d'intégrer les tests de résistance aux médicaments dans le continuum de soins pour les personnes sous traitement antirétroviral dans les pays à ressources limitées. Cependant, la plupart des méthodes déclarés se concentrent sur l'application de génotypage de la pharmacorésistance dans la surveillance de la résistance aux médicaments au niveau de la population. La méthode de génotypage Saturne / Life Technologies est un protocole entièrement intégrée pour la surveillance et le suivi de la résistance aux médicaments. Cette méthode a été conçue pour être un protocole abordable mise en œuvre de la plupart open source et open ressources accès bioinformatiques pour l'interprétation de la résistance aux médicaments et la production de rapports de gestion clinique.
Il a été démontré par la comparaison avec la méthode de génotypage ViroSeq approuvé par la FDA pour êtreprécis dans l'identification des mutations de résistance aux médicaments à partir d'un panel de l'ANRS échantillons d'essais d'aptitude, dans 100% des échantillons de panneaux de laboratoire qui ont été amplifiés avec succès. La précision a également été évaluée sur des échantillons cliniques de virus de sous-type C, le sous-type le plus dominant en Afrique australe. Le procédé était le plus précis sur des échantillons de sous-type C tel qu'il était sur le sous-type A et B. Cependant, si le procédé sera utilisé dans d'autres parties du monde où CRF02_AG est répandue, il existe un besoin pour la modification des amorces puisque le procédé pas amplifier un des échantillons du panel qui a été montré pour avoir CRF02_AG. En variante, un ensemble d'amorces sensibles à tous les virus du groupe M 33 dégénérée, 36 pourrait être utilisé dans les régions où le sous-type de la distribution est plus hétérogène 38.
La sensibilité de la transcription inverse et la PCR peut être augmentée par l'extraction de l'ARN à partir de l'augmentation des volumes de plasma, telles que 500 ml. Le plasma peut être centrifuged à 21 000 g pendant 90 min à concentrer les particules virales avant de poursuivre avec le protocole tel que décrit par le mini kit d'extraction d'ARN viral QIAamp.
Comme indiqué, la nouvelle méthode présente un avantage supplémentaire qu'il produit des rapports complets pour la gestion individuelle du patient. Ces rapports sont une consolidation du génotype, immunologiques et des données de surveillance virologique ainsi que l'histoire clinique et le traitement de RegaDB. Ceci est accompagné par une interprétation de laboratoire détaillée du profil de résistance suivie par un examen aussi détaillé de l'histoire clinique du patient ainsi que des recommandations de traitement. L'utilisation d'un spécialiste médecins d'examiner les rapports et faire des recommandations de traitement pour les patients fournit le mentorat bien nécessaire pour les infirmières praticiennes ainsi que des cliniciens expérimentés, qui sont de plus en plus offrent ART en Afrique du Sud dans le cadre des recommandations de l'OMS pour la délégation des tâches. Ces cliniquerapports ont été montrés comme des aides d'enseignement efficaces pour les cliniciens ayant peu ou pas d'expérience dans la gestion de la résistance aux médicaments. Du point de vue du patient, notre méthode réduit le besoin de se déplacer vers des sites centralisés pour accéder à des services spécialisés pour le VIH.
Ainsi, le protocole décrit dans son ensemble constitue une bonne plate-forme à travers laquelle la gestion de la résistance du VIH aux médicaments peut être intégré, à un coût abordable, dans le continuum de soins pour les personnes infectées par le VIH ART défaut. Les données générées peuvent être utilisés à des fins épidémiologiques pour évaluer l'évolution et la transmission de la résistance aux médicaments dans la communauté. La taille du fragment pol généré est assez bon pour l'analyse phylogénétique plus complexe qui va produire une meilleure compréhension de l'épidémie au niveau de la population.
Déclarations de divulgation
Ce travail a été soutenu par le Wellcome Trust (082384/Z/07/Z), Union européenne (2007 SANTE 147-790), le Centre américain pour le contrôle des maladies par CAPRISA (titre du projet: Renforcement des systèmes de santé et l'échec de traitement du VIH (VIH- TFC)), et le Programme commun de recherche suisse africain du Sud (SSJRP) subvention de recherche intitulé «Swiss Prot / Afrique du Sud: Protein Bioinformatics développement des ressources pour importants pathogènes liés à la santé". RL est soutenu par le Wellcome Trust (numéro de licence 090 999 / Z / 09 / Z). Les bailleurs de fonds n'ont joué aucun rôle dans la conception de l'étude, la collecte et l'analyse des données, la décision de publier, ou de la préparation du manuscrit. Les auteurs déclarent aucun intérêt financier concurrents.
Remerciements
Les auteurs tiennent à remercier tous les collègues qui ont rendu ce travail possible, en particulier Maya Balamane, Elizabeth Johnston Blanc, Sharon Sjoblom, Greg Ording Zakhona Gumede, Xolile Kineri, Phindile Mabaso, Lungisa Ndwandwe, James Garvey, Gavin Cobb, Senzo Maphanga, Terusha Chetty , Kevi Naidoo, Andrew Skingsley, Katharine Stott, et Lungani Ndwandwe. Les auteurs tiennent aussi à remercier tout le personnel du ministère de la Santé et le personnel du Centre Afrique qui travaillent le traitement du VIH Hlabisa et programme de soins.
matériels
| Name | Company | Catalog Number | Comments |
| Superscript III 1st strand Synthesis kit | Life Technologies | 18080051 | Reverse Transcription |
| SATURN/LiFE Technologies Custom Primers | Life Technologies | 4473517 | PCR |
| Platinum Taq | Life Technologies | 10966026 | PCR |
| PureLink QUICK PCR Purification Kit | Life Technologies | K310002 | PCR |
| Viroseq | ABBOTT | 4J94-20 | Reverse Transcription and PCR |
| Agarose Tablets (Dnase/Rnase free) | BIOLINE | BIO-41027 | PCR |
| TBE Buffer | MERCK | 1.06177.2500 | PCR |
| O'Range Ruler 200 bp DNA Ladder | Fermentas | FE SM0633 | PCR |
| Novel Juice | GeneDireX | LD001-1000 | PCR |
| MiniBis Bioimaging System | DNR Bioimaging Systems Ltd | Gel Documentation | |
| Power Pac 300 | BIORAD | Gel Electrophoresis | |
| Big Dye Terminator Kit version 3.1 | Life Technologies | 4337456 | Sequencing |
| Arrays | Life Technolgies | 4319899 | Sequencing |
| PoP | Life Technologies | 4363785 | Sequencing |
| 10x EDTA Buffer | Life Technologies | 402824 | Sequencing |
| Formamide | Life Technologies | 4311320 | Sequencing |
| 5x Sequencing Buffer | Life Tecgnologies | 4336697 | Sequencing |
| 3130 xl Genetic Analyzer | Life Technologies | Sequencing | |
| GeneAmp PCR System 9700 | Life Technologies | RT/PCR/Sequencing | |
| Centrifuge 5804 | EPPENDORF | Sample Processing | |
| Centrifuge 5415R | EPPENDORF | RNA Extraction | |
| Centrifuge 5415R | EPPENDORF | RT and PCR | |
| Centrifuge 5415D | EPPENDORF | PCR Product Clean up | |
| Centrifuge 5810 | EPPENDORF | Sequencing Clean up | |
| Picofuge | BIORAD | C1301-230V | RT and PCR |
| Vortex Genius 3 | IKA | RNA extraction and reagent preparation | |
| Vortex mixer | IKA | Sequencing Cleanup | |
| NanoDrop 2000 UV/VIS spectrophotometer | ThermoScientific | DNA quantification | |
| 3 M Sodium Acetate | MERCK | 567422 | Sequencing Clean up |
| Absolute Ethanol | MERCK | SAAR2233540LP | Sequencing Cleanup |
| 1.5 ml SARSTEDT Tubes | BIODEX | 72.692.005 | RNA Extraction |
| 2 ml SARSTEDT Tubes | BIODEX | 72.693.005 | RNA Extraction |
| 2 ml Collection tubes | SCIENTIFIC GROUP | MCT-200-NC/S | RNA Extraction |
| Optical MicroAmp 96-well reaction plates | Life Technologies | N8010560 | Sequencing |
| 200 µl 8 Strip StarPCR Tubes with attached flat caps | STAR Lab - supplied by CELTIC | A1402-3700 | RT and PCR |
| 200 µl PCR individual tubes | Scientific Group | CR/3745 | RT and PCR |
| Geneious | Biomatters | Sequence analysis | |
| Internet Access | Preferrably high speed | ||
| Web resources | |||
| hivdb.stanford.edu | Stanford University | Drug reistance analysis | |
| http://bioafrica.mrc.ac.za:8080/regadb-ui/RegaDB | SATuRN | database | |
| http://bioafrica.mrc.ac.za/tools/pppweb.html | SATuRN | Sequence quality tool | |
Références
- Shao, Y., Williamson, C. The HIV-1 epidemic: low- to middle-income countries. Cold Spring Harbor Persp. Med. 2, (2012).
- Mutevedzi, P. C., et al. Scale-up of a decentralized HIV treatment programme in rural KwaZulu-Natal, South Africa: does rapid expansion affect patient outcomes. Bull. World Health Organ. 88, 593-600 (2010).
- Houlihan, C. F., et al. Cohort profile: Hlabisa HIV treatment and care programme. Int. J. Epidemiol. 40, 318-326 (2011).
- Tanser, F., Barnighausen, T., Grapsa, E., Zaidi, J., Newell, M. L. High coverage of ART associated with decline in risk of HIV acquisition in rural KwaZulu-Natal. South Africa. Science. 339, 966-971 (2013).
- Bor, J., Herbst, A. J., Newell, M. L., Barnighausen, T. Increases in adult life expectancy in rural South Africa: valuing the scale-up of HIV treatment. Science. 339, 961-965 (2013).
- Montaner, J. S., et al. The case for expanding access to highly active antiretroviral therapy to curb the growth of the HIV epidemic. Lancet. 368, 531-536 (2006).
- Granich, R. M., Gilks, C. F., Dye, C., De Cock, K. M., Williams, B. G. Universal voluntary HIV testing with immediate antiretroviral therapy as a strategy for elimination of HIV transmission: a mathematical model. Lancet. 373, 48-57 (2009).
- Johnson, L. F., et al. Life expectancies of South african adults starting antiretroviral treatment: collaborative analysis of cohort studies. PLoS Med. 10, (2013).
- Blower, S., Ma, L., Farmer, P., Koenig, S. Predicting the impact of antiretrovirals in resource-poor settings: preventing HIV infections whilst controlling drug resistance. Curr. Drug Targets. 3, 345-353 (2003).
- Geretti, A. M. Epidemiology of antiretroviral drug resistance in drug-naive persons. Curr. Opin. Infect. Dis. 20, 22-32 (2007).
- Larder, B. A., Darby, G., Richman, D. D. HIV with reduced sensitivity to zidovudine (AZT) isolated during prolonged therapy. Science. 243, 1731-1734 (1989).
- Erice, A., et al. Brief report: primary infection with zidovudine-resistant human immunodeficiency virus type 1. N. Engl. J. Med. 328, 1163-1165 (1993).
- Williams, I., et al. British HIV Association guidelines for the treatment of HIV-1-positive adults with antiretroviral therapy. HIV Med. 13 Suppl 2, 1-85 (2012).
- DHHS, . US Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents. , (2012).
- Vandamme, A. M., et al. European recommendations for the clinical use of HIV drug resistance testing: 2011 update. AIDS Rev. 13, 77-108 (2011).
- DHHS, . US Panel on Antiretroviral Therapy and Medical Management of HIV-Infected Children. Guidelines for the Use of Antiretroviral Agents in Pediatric HIV Infection. , (2012).
- . Department of Health. Clinical guidelines for the management of HIV & AIDS in adults and adolescents. , (2010).
- Conradie, F., et al. The 2012 southern African ARV drug resistance testing guidelines. S. Afr. J.HIV Med. 13, 162-167 (2012).
- Manasa, J., et al. Primary Drug Resistance in South Africa: Data from 10 Years of Surveys. AIDS Res. Hum. Retroviruses. 28, 558-565 (2012).
- Tshabalala, M., et al. Surveillance of transmitted antiretroviral drug resistance among HIV-1 infected women attending antenatal clinics in Chitungwiza, Zimbabwe. PLoS ONE. 6, (2011).
- Bartolo, I., et al. Antiretroviral drug resistance surveillance among treatment-naive human immunodeficiency virus type 1-infected individuals in Angola: evidence for low level of transmitted drug resistance. Antimicrob. Agents Chemother. 53, 3156-3158 (2009).
- Bartolo, I., et al. HIV-1 genetic diversity and transmitted drug resistance in health care settings in Maputo, Mozambique. J. Acquir. Immune Defic. Syndr. 51, 323-331 (2009).
- Hamers, R. L., et al. HIV-1 drug resistance in antiretroviral-naive individuals in sub-Saharan Africa after rollout of antiretroviral therapy: a multicentre observational study. Lancet Infect. Dis. 11, 750-759 (2011).
- Hamers, R. L., et al. HIV-1 Drug Resistance Mutations Are Present in Six Percent of Persons Initiating Antiretroviral Therapy in Lusaka, Zambia. J. Acquir. Immune Defic. Syndr. , (2010).
- Nwobegahay, J., Selabe, G., Ndjeka, N. O., Manhaeve, C., Bessong, P. O. Low prevalence of transmitted genetic drug resistance in a cohort of HIV infected naive patients entering antiretroviral treatment programs at two sites in northern South Africa. J. Med. Virol. 84, 1839-1843 (2012).
- Iweriebor, B. C., et al. Molecular epidemiology of HIV in two highly endemic areas of northeastern South Africa. Arch. Virol. 157, 455-465 (2012).
- Parboosing, R., Naidoo, A., Gordon, M., Taylor, M., Vella, V. Resistance to antiretroviral drugs in newly diagnosed, young treatment-naive HIV-positive pregnant women in the province of KwaZulu-Natal South Africa. J. Med. Virol. 83, 1508-1513 (2011).
- Nwobegahay, J. M., et al. Prevalence of antiretroviral drug resistance mutations and HIV-I subtypes among newly-diagnosed drug-naive persons visiting a voluntary testing and counselling centre in northeastern South Africa. J. Health Popul. Nutr. 29, 303-309 (2011).
- Nwobegahay, J., et al. Prevalence of drug-resistant mutations in newly diagnosed drug-naive HIV-1-infected individuals in a treatment site in the waterberg district, limpopo province). S. Afr. Med. J. 101 (2011), 335-337 (2011).
- Rosen, S., Long, L., Sanne, I., Stevens, W. S., Fox, M. P. The net cost of incorporating resistance testing into HIV/AIDS treatment in South Africa: a Markov model with primary data. J. Int. AIDS Soc. 14, 24 (2011).
- Dalai, S. C., et al. Evolution and molecular epidemiology of subtype C HIV-1 in Zimbabwe. AIDS. 23, 2523-2532 (2009).
- Chen, J. H., et al. In-house human immunodeficiency virus-1 genotype resistance testing to determine highly active antiretroviral therapy resistance mutations in Hong Kong. Hong Kong Med. 18, 20-24 (2012).
- Lee, C. K., et al. An in-house HIV genotyping assay for the detection of drug resistance mutations in Southeast Asian patients infected with HIV-1. J. Med. Virol. 84, 394-401 (2012).
- Aitken, S. C., et al. A Pragmatic Approach to HIV-1 Drug Resistance Determination in Resource-Limited Settings by Use of a Novel Genotyping Assay Targeting the Reverse Transcriptase-Encoding Region Only. J. Clin. Microbiol. 51, 1757-1761 (2013).
- Zhou, Z., et al. Optimization of a low cost and broadly sensitive genotyping assay for HIV-1 drug resistance surveillance and monitoring in resource-limited settings. PLoS One. 6, (2011).
- , Life Technologies. Life Technologies and SATuRN Collaborate to Increase Access to HIV Testing in Africa. , (2012).
- Lihana, R. W., Ssemwanga, D., Abimiku, A., Ndembi, N. Update on HIV-1 diversity in Africa: a decade in review. AIDS Rev. 14, 83-100 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon