Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Haute résolution spatio-temporelle d'analyse de la dynamique des récepteurs par simple molécule microscopie de fluorescence
Dans cet article
Résumé
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
Résumé
Microscopie de molécule unique est en train de devenir une approche puissante pour analyser le comportement de molécules de signalisation, en particulier concernant les aspects (par exemple., La cinétique, la coexistence de différents états et des populations, des interactions transitoires), qui sont généralement cachés dans des mesures d'ensemble, tels que celles obtenues avec des procédés biochimiques ou microscopie standard. Ainsi, les événements dynamiques, tels que les interactions récepteur-récepteur, peuvent être suivies en temps réel dans une cellule vivante avec une résolution spatio-temporelle élevée. Ce protocole décrit une méthode basée sur l'étiquetage de petites et lumineuses fluorophores organiques et totale de fluorescence à réflexion interne (FRBR) de microscopie pour visualiser directement les récepteurs simples sur la surface de cellules vivantes. Cette approche permet de localiser précisément des récepteurs, de mesurer la taille de complexes de récepteurs, et capturer les événements dynamiques tels que les interactions récepteur-récepteur transitoire. Le protocole fournit une description détaillée de la façon de perform une expérience unique molécule, y compris la préparation de l'échantillon, l'acquisition de l'image et l'analyse d'image. A titre d'exemple, l'application de cette méthode pour analyser deux G-récepteurs couplés aux protéines, c'est à dire., Β 2-adrénergiques et γ-aminobutyrique de type acide B (GABA B) récepteur, est rapporté. Le protocole peut être adapté à d'autres protéines de membrane et des modèles de cellules différentes, les méthodes de transfection et les stratégies de marquage.
Introduction
Des récepteurs situés à la surface des cellules détectent l'environnement extracellulaire et de répondre à une variété de stimuli, tels que des agents odorants, des ions, des neurotransmetteurs petites et de grandes hormones protéiques. La nature du fluide des membranes cellulaires permet des mouvements de récepteurs et d'autres protéines membranaires. Ceci est essentiel pour la formation de complexes de protéines et la survenue d'interactions protéine-protéine transitoires, telles que celles utilisées par les récepteurs de s'assembler en des unités fonctionnelles et la transduction de signaux à l'intérieur de la cellule. Par exemple, G-récepteurs couplés aux protéines (RCPG), qui constituent la plus grande famille de récepteurs de surface cellulaire 1, ont été proposées pour former di-/oligomers, ce qui semble être impliquée dans la régulation de fine de la transduction du signal et pourrait avoir des conséquences physiologiques et pharmacologiques importantes 2-5.
Microscopie seule molécule a le grand potentiel de visualiser directement avec grande spatiotemprésolution par voie orale le comportement dynamique de récepteurs individuels situés sur la surface de cellules vivantes, y compris leur association pour former des dimères et des complexes moléculaires d'ordre supérieur 10.6. Cette offre plusieurs avantages par rapport aux méthodes biochimiques et de microscopie standard, qui relèvent habituellement le comportement moyen des milliers ou des millions de molécules.
étiquetage des protéines avec un fluorophore suffisamment lumineux et photostable est essentiel pour la microscopie de molécule unique. Ce protocole prend avantage de l'étiquette SNAP récemment introduit 11 à fixer de façon covalente les petites et les fluorophores organiques lumineuses à des récepteurs de surface cellulaire. SNAP est une étiquette 20 kD de la protéine dérivée de la réparation de l'ADN enzyme O 6-alkylguanine-ADN alkyltransférase humaine, qui peut être irréversiblement marqué avec un fluorophore conjugué benzylguanine des dérivés (fluorophore-BG). CLIP, un tag en outre conçu dérivé de SNAP, peut être à la place marquée avec un fluorophore cles dérivés de benzylcytosine onjugated 12.
Le protocole rapporté dans ce manuscrit explique comment transfecter et étiquette SNAP-étiqueté 11 récepteurs avec des petits fluorophores organiques et l'utilisation totale de fluorescence à réflexion interne (FRBR) de microscopie pour visualiser les récepteurs simples ou complexes de récepteurs à la surface des cellules vivantes 10. Les résultats de protocole signalés dans> 90% d'efficacité de l'étiquetage d'un SNAP-protéine marquée extracellulaire à la surface cellulaire 10. De plus amples informations sur la manière d'utiliser les données d'une seule molécule à analyser la taille et la mobilité des complexes de récepteurs, ainsi que pour capturer des interactions récepteur-récepteur transitoire, il est prévu. Un flux de travail schématique de l'ensemble du protocole est donné dans la figure 1. A titre d'exemple, la transfection de cellules ovariennes de hamster chinois (CHO) avec G-récepteurs couplés aux protéines SNAP-tag (RCPG) suivie par marquage avec un dérivé fluorophore BG que ainsi que son application à QUANTIFy et moniteur récepteur di-/oligomerization sont décrits. Ce protocole peut être étendu à d'autres protéines de surface cellulaire et des marqueurs fluorescents (par exemple., CLIP), ainsi que d'autres méthodes de transfection et d'étiquetage.
Protocole
1. Préparation de l'échantillon
- Nettoyage lamelle
REMARQUE: Travailler sous une hotte.- Utiliser des pinces propres à placer des lamelles de verre (diamètre 24 mm) dans un support de lamelle qui sépare les lamelles individuelles.
- Placer le support à lamelles dans un bécher, et ajouter le chloroforme jusqu'à ce que les lamelles sont couverts. Recouvrir de papier d'aluminium pour réduire l'évaporation et de traitement par ultrasons dans un bain sonique pendant 1 heure à température ambiante. Retirez le support de la lamelle sur le bécher et laisser les lamelles sec.
- Répétez l'étape 1.1.2 avec une solution de NaOH 5M au lieu de chloroforme.
- Retirez le support de la lamelle dans un nouveau récipient et laver trois fois avec de l'eau distillée. Mettre lamelles nettoyées dans une plaque de culture de cellules en verre rempli d'éthanol à 100%.
- Préparation d'échantillons d'étalonnage
- Dissoudre colorant fluorescent dans un solvant approprié.
- Préparer une dilution en série de 1:10 le colorant fluorescent allantà partir de 13 heures à 1 nM dans stérilisé par filtration (0,22 um) de l'eau.
- Prenez lamelles nettoyées stockées dans 100% d'éthanol et laver avec de l'eau stérilisée par filtration. Coin 20 ul de chaque dilution de colorant fluorescent sur une lamelle couvre-objet nettoyé séparément. Laissez sécher les lamelles sous une hotte stérile. Protéger les lamelles de la lumière et de la poussière jusqu'à utilisation. Utiliser ces échantillons pour estimer l'intensité de molécules fluorescentes à simple (voir l'étape 3).
- Transfection
- Culture de cellules CHO dans du milieu Eagle modifié 1:01 / mélange nutritif de Dulbecco F-12 (DMEM/F12) additionné de 10% de sérum bovin fœtal (FBS), 100 U / ml de pénicilline et 100 pg / ml de streptomycine à 37 ° C, dans . 5% de CO 2 NOTE: Utiliser les médias libres phénol rouge tout au long de l'expérience afin de minimiser autofluorescence.
- Prenez lamelles nettoyées de la solution d'éthanol à 100%, les laver avec une solution saline stérile tamponnée au phosphate (PBS), et placer une lamelle dans chaque puits d'une culture pl 6 cellules de bienmangé.
- Trypsiniser, et compter des cellules CHO de la graine à une densité de 3 x 10 5 cellules / puits dans la plaque de culture cellulaire à 6 puits contenant des lamelles couvre-objet. Laisser les cellules se développent dans un incubateur (37 ° C, 5% CO2) pendant 24 heures afin de parvenir à env. 80% de confluence, ce qui est de la densité cellulaire optimale pour la transfection.
- Pour chaque puits, diluer 2 pg de l'ADN de plasmide désiré (par ex., SNAP-marqué récepteur β 2-adrénergique), et 6 ul de Lipofectamine 2000 dans deux tubes séparés contenant 500 ul de milieu OptiMem. Incuber à température ambiante pendant 5 min.
- Combiner les solutions de l'étape 1.3.4 dans un tube et mélanger pour obtenir un mélange de transfection. Faire incuber le mélange de transfection à température ambiante pendant 20 min.
- Au cours de l'incubation (1.3.5), prendre les cellules CHO et laver deux fois avec préchauffé (37 ° C) PBS. Remplacer PBS avec 1 ml / puits de milieu de phénol DMEM/F12 dépourvu de rouge de supplémenté avec 10% de FBS, mais sans antibiotiques.
- Ajouter l'ensemble transfectmélange d'ions (1 ml) de l'étape 1.3.5 goutte à goutte à chaque puits, et doucement basculer la plaque arrière pour assurer un mélange complet.
- Incuber pendant 2 à 4 heures à 37 ° C, sous 5% de CO 2 et de passer immédiatement après à l'étape suivante. REMARQUE: Ces conditions de transfection ont été optimisés pour obtenir des densités des récepteurs <0,45 particule / um 2, qui sont adaptés pour une seule Imagerie molécule. Ajustements pourraient être nécessaires lors de l'utilisation de cellules différentes, des constructions ou des réactifs.
- l'étiquetage de protéines
- Diluer 1 ul de fluorophore-BG solution stock dans 1 ml de milieu DMEM/F12 supplémenté avec 10% de FBS pour obtenir une concentration finale de 1 uM. Prenez les cellules transfectées de l'incubateur et laver deux fois avec préchauffé (37 ° C) PBS. Remplacer PBS avec 1 ml de solution fluorophore BG-1 uM et incuber pendant 20 min à 37 ° C 5% de CO2 dans un incubateur.
- Après l'incubation, laver les cellules de troisfois avec du milieu DMEM/F12 additionné de 10% de FBS, à chaque fois suivies de 5 minutes d'incubation à 37 ° C. Prenez une lamelle (avec cellules marquées) avec des pincettes et le placer dans une chambre de l'imagerie.
- Laver deux fois avec 300 tampon d'imagerie ul. Ajouter 300 pi de tampon d'imagerie frais et procéder immédiatement à l'imagerie (Partie 2).
2. L'acquisition d'images
REMARQUE: Utilisez une réflexion interne totale fluorescence (FRBR) de microscope, équipé d'un objectif à immersion d'huile à grande ouverture numérique (par exemple, ouverture numérique magnification/1.46 100X.), Lasers appropriés (par exemple, 405 nm, 488 nm, 561 nm et 645 lasers à diode nm), un dispositif à couplage de charge de multiplication d'électrons (EMCCD) de la caméra, un incubateur et un contrôle de la température de visualiser des molécules fluorescentes simples.
- Réglez les paramètres du microscope souhaités, soit., Ligne de laser, l'angle de la FRBR (ce paramètre contrôle la pénétrationprofondeur tion du champ évanescent), temps d'exposition, le taux et le nombre d'images par film de 10 châssis. Gardez l'appareil de chauffage / incubateur et contrôle de la température toujours à éviter les dérives de température et l'humidité de condensation.
- Mettre une goutte d'huile d'immersion sur l'objectif du microscope 100X. Placer la chambre de formation d'image avec les cellules marquées sur le porte-échantillon du microscope, et d'amener les cellules à se concentrer en utilisant un éclairage en fond clair.
- Passez à l'éclairage de la FRBR. Maintenir la puissance aussi faible que possible pour permettre au laser de chercher la cellule souhaitée, mais en même temps de minimiser photoblanchiment.
- Sélectionnez la cellule désirée et bien la mise au point. Régler la puissance du laser à un niveau qui permet la visualisation des fluorophores uniques. Acquérir une séquence d'images et enregistrer le fichier brut de séquence d'image. Tiff.
3. Calibration (fluorophores simples sur monomères / Contrôle de récepteurs dimères verre et)
- Assemblez chaque sampl d'étalonnagee préparé comme décrit en 1.2 dans la chambre d'imagerie. Placez chaque échantillon sur le microscope et choisir l'échantillon contenant des taches de diffraction limitée bien séparées l'eau de Javel en une seule étape. REMARQUE: Ces points représentent des molécules simples de colorant fluorescent.
- Acquérir FRBR séquences d'images comme décrit à l'étape 2. Important: les mêmes paramètres d'imagerie doivent être utilisés pour toutes les expériences, y compris ceux pour le calibrage.
- Effectuer la détection et l'analyse de suivi tel que décrit dans 4.1 au 4.2. Extraire l'intensité de chaque particule comme décrit dans 4.2.6. A partir de ces données, calculer la moyenne (μ) et l'écart type (σ) de l'intensité des fluorophores uniques.
- Facultatif: effectuer la même analyse sur les cellules transfectées avec un récepteur de surface cellulaire monomère (. Par exemple, CD86), N-terminale étiqueté avec un ou deux exemplaires de SNAP 10 et étiqueté avec le dérivé fluorophore BG. Follow la procédure décrite ci-dessus pour le fluorophore unique sur le verre. Estimer l'efficacité de marquage tel que décrit dans Calebiro, D. et al. Dix
4. Analyse de l'image
- préparation de la séquence de l'image
- Utilisez un logiciel de traitement d'image (par exemple., ImageJ) à recadrer les images.
- Enregistrer des images individuelles séparées. Des images TIFF dans un nouveau dossier, en indiquant sur chaque image le numéro de châssis.
- Mesurer l'aire de surface de la cellule en établissant une région d'intérêt (ROI) le long du contour d'une cellule et en utilisant l'outil de mesure dans ImageJ ou un outil similaire dans un autre logiciel. Utiliser cette valeur pour calculer la densité des particules en divisant le nombre total de particules au début du film de la zone de surface de la cellule.
- détection de particules et de suivi
REMARQUE: Utilisez un logiciel non commercial tel que u-piste 13, travaillant dans Matlabenvironnement, de détecter automatiquement et de suivre les particules de récepteurs simples.
NOTE: L'algorithme u-piste est basée sur l'approche de suivi multi-hypothèse. Cette approche relie les particules entre les images par la construction des matrices de coûts, où les probabilités individuelles qu'une particule donnée dans un cadre correspond à une particule donnée dans la trame suivante, apparaît, disparaît ou se confond / divise avec / par les autres particules sont affectés. La solution qui minimise les coûts à l'échelle mondiale, à savoir., L'une avec la plus grande probabilité, est finalement sélectionné. Cela permet également de suivre une particule disparaître temporairement, un phénomène typique causé par fluorophore clignotant. La dernière version de u-piste (2.1.0) a des interfaces utilisateurs graphiques qui facilitent l'exécution de ces analyses.- De l'invite de commande Matlab, tapez "movieSelectorGUI" pour ouvrir l'interface de sélection de film. Suivez les instructions pour créer unnouvelle base de données de film à partir des images enregistrées précédemment distincts (voir 4.1.2).
- Fournir la taille de pixel en nm, l'intervalle de temps en secondes, ouverture numérique, la résolution de la caméra et la longueur d'onde d'émission du fluorophore, nécessaire pour la détection et le suivi des particules. Enregistrer la base de données de film.
- Dans l'interface de sélection de films, lancer l'analyse, le choix "simples particules" comme type d'objets. Une nouvelle fenêtre apparaît où les paramètres utilisés pour la détection de particules et le suivi peuvent être définis. Commencer avec les paramètres par défaut. Plus tard, ajuster ces paramètres si la qualité de la détection et / ou de suivi n'est pas satisfaisante (par exemple, certaines particules ne sont pas détectés ou titres sont fragmentés) en option:. Dans les paramètres de suivi, consultez "le suivi des résultats à l'exportation au format matrice" pour stocker les coordonnées et les amplitudes de toutes les particules dans une matrice unique (domaine dit "trackedFeaturedInfo»). Pour une description détaillée de ces paramètres, reportez-vous à la documentation u-piste.
- Exécuter l'algorithme de détection. Cet algorithme déterminer automatiquement l'emplacement et de l'intensité au-dessus de fond de chaque point à diffraction limitée (, complexes ie. Récepteurs uniques / récepteurs) de montage d'une fonction gaussienne bidimensionnelle avec un écart type égal à la fonction d'étalement de point du microscope autour des maxima d'intensité locale . Ensuite, exécutez l'algorithme de suivi. Stocker les résultats des analyses dans un fichier de tapis..
- Utilisez la routine "movieViewer", contenue dans le paquet u-piste, ou la coutume ceux semblables à visualiser les pistes et de vérifier la qualité de la détection et de suivi.
- Ouvrez le fichier de tapis. Pour voir la position et l'amplitude (c'est à dire., Intensité) des particules suivis à chaque trame. Les données générées à l'étape 4.2.4 sont contenues dans le domaine de l'ar tracksCoordAmpCG tracksFinalrayon et / ou dans trackedFeaturedInfo. A partir du nombre total de particules détectées calculer la densité des particules en divisant cette valeur par la surface de la cellule mesurée à 4.1.3.
- Facultatif: Utilisez coordonne la particule au cours du temps (voir 4.2.6) pour analyser le mouvement des particules de récepteurs. Calculer les déplacements moyennes carrés (TMS) et les coefficients de diffusion (D) à l'aide de Matlab ou un logiciel similaire. Pour chaque particule, et chaque intervalle de temps (Δ t) considéré, de calculer le déplacement quadratique moyen (MSD) à l'aide de la formule suivante:
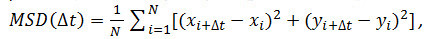
où Δ t est l'intervalle de temps dans des trames, N est le nombre de pas analysés, x et y sont de x et y les coordonnées de la particule à la trame indiquée par l'index. Utilisez le MSD sur des parcelles de temps pour évaluer le type de mouvement d'une particule donnée: relations linéairesindiquer diffusion libre (c'est à dire., mouvement brownien), une courbure positive (., la courbe ressemble à une parabole) suggère le mouvement dirigé, une courbure négative est indicative de mouvement limitée 14. Dans le cas de particules diffusant librement, de calculer le coefficient de diffusion (D) de chacune des particules en ajustant les données de TMS obtenues avec l'équation suivante: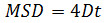
- Calcul de la taille des particules avec méthode d'ajustement gaussien
Remarque: Une fois que la distribution d'intensité d'échantillons d'étalonnage (fluorophores simples sur le verre et / ou des récepteurs monomères marqués par fluorescence), est connu, effectuer un ajustement gaussien mixte sur la distribution des intensités de particules au début d'une séquence d'image pour déterminer la taille des complexes de récepteurs (c.-à-., le nombre de récepteurs par particule) 10. Réaliser ces analyses sous Matlab ou slogiciel es mesures analogues.- Calculer l'intensité de chacune des particules en faisant la moyenne de l'intensité de la particule à partir de la première trame de la trame avant le premier changement d'intensité s'est produite (dans la plupart des cas en raison d'une diminution de photoblanchiment).
- De réaliser un ajustement gaussien mixte selon l'équation suivante:
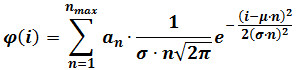
où φ (i) est la fréquence des particules ayant une intensité i, n est le nombre de composants, α n est un paramètre qui contribue à la hauteur de la composante n, μ et σ sont la moyenne et l'écart-type de l'intensité de référence fluorophores simples . (calculé tel que décrit à l'étape 3) NOTE: Déterminer til nombre maximal d'éléments (n max) pour chaque séquence d'image en augmentant progressivement n max jusqu'à ce que l'ajout d'un composant ne produit plus un meilleur ajustement statistique, tel que jugé par un F-test. - Facultatif: (. Par exemple, les 60 dernières images d'une séquence d'images 400) Effectuer un ajustement gaussien mixte sur la distribution d'intensité obtenu sur les dernières images du film. Remplacer μ et σ avec les valeurs obtenues après cette mise en place, qui fournissent des estimations plus précises de ces paramètres, et répétez l'étape 4.3.2.
- Calculer l'aire sous la courbe (AUC) de chaque composante de l'ajustement gaussien mixte. Calculer l'abondance relative de particules de récepteur de taille différente (par exemple, un monomère, dimère, trimère, etc) en divisant la valeur AUC de chaque composant par l'AUC de l'ensemble de distribution.
- Facultatif: Utiliser les données de différentes cellules et les densités de particules correspondantes (calculées comme décrit au point 4.2.6) pour générer des parcelles où la distribution des particules de taille différente est corrélée avec la densité de particules 10.
- Calcul de la taille des particules du corps avec l'étape procédé
REMARQUE: Utiliser une analyse montage étape comme une méthode alternative pour déterminer la taille de complexes de récepteurs 10. La base de cette analyse est que la destruction provoquée par la lumière (photo-blanchiment) d'un seul des résultats fluorophores dans sa disparition instantanée - ainsi des particules contenant n fluorophores devraient progressivement l'eau de Javel produire un profil d'intensité à n étapes.- Extrait profils d'intensité de chaque particule à partir du fichier de tapis. Généré par u-piste ou un logiciel de détection / suivi similaire (voir 4.2.6).
- Utiliser un algorithme d'ajustement étape, tel que celui présentédans la réf. 10, pour compter le nombre d'étapes de blanchiment pour chaque particule.
- En option: utiliser les résultats pour générer des distributions montrant l'abondance relative des particules de récepteurs de différentes tailles et les mettre en corrélation avec la densité de particules comme décrit précédemment pour les résultats de l'ajustement gaussien mixte (voir 4.3).
Résultats
Le protocole décrit peut être appliqué à une variété de différentes protéines membranaires. A titre d'exemple, des résultats représentatifs obtenus avec β 2-adrénergiques et GABA B sont rapportés 10. Comme les signaux fluorescents à partir de molécules simples sont faibles, la minimisation de la fluorescence de fond est la première étape clé de bons résultats. Ainsi, il est important d'utiliser des lamelles couvre largement nettoyées (Figure 2A),
Discussion
Le protocole décrit permet l'analyse de l'agencement spatial, la mobilité et la taille des complexes de récepteurs de surface cellulaire au niveau d'une seule molécule. Par rapport à l'utilisation de protéines fluorescentes, le marquage avec de petites fluorophores organiques, qui sont plus lumineux et plus photostable, a l'avantage de permettre une visualisation étendue de particules de récepteurs individuels. Etant donné que les niveaux d'expression obtenus sont extrêmement faibles (&...
Déclarations de divulgation
The authors declare that they have no competing financial interests.
Remerciements
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
matériels
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
Références
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon