Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection de biomarqueurs exosomales par déclenchement électrique induite par champ et évaluation (EFIRM)
Dans cet article
Erratum Notice
Résumé
Exosomes are microvesicular structures found within biofluids that potentially carry important disease discriminatory biomarkers. Here, a novel method is used to specifically extract exosomes and rapidly test the exosomal cargo for both RNA/protein targets following the disruption of exosomes using non-uniform electric cyclic square waves.
Résumé
Les exosomes sont des structures microvésiculaire qui jouent un rôle de médiateur dans la communication intercellulaire. Il est intéressant d'étudier la cargaison interne des exosomes pour déterminer si elles sont porteuses de maladies biomarqueurs discriminatoires. Pour effectuer une analyse de exosomal, il est nécessaire de mettre au point un procédé d'extraction et d'analyse des exosomes à partir de fluides biologiques cibles sans endommager le contenu interne.
Libération induite de champ électrique et de mesure (EFIRM) est une méthode pour extraire spécifiquement exosomes de biofluides, le déchargement de leur cargaison, et de tester leur contenu ARN / protéines interne. Utilisation d'un anticorps CD63 spécifique microparticule magnétique anti-humain, les exosomes sont tout d'abord précipités à partir de fluides biologiques. Après extraction, basse tension électriques des ondes carrées cycliques (CCF) sont appliqués à rompre la membrane vésiculaire et provoquer déchargement de marchandises. Le contenu de la exosomes est hybridée à des amorces d'ADN ou des anticorps immobilisés sur une surface d'électrode pour quantification du contenu moléculaire.
Procédé de EFIRM est avantageux pour l'extraction des exosomes et déchargement des marchandises pour l'analyse sans tampon de lyse. Ce procédé est capable de réaliser une détection spécifique de deux cibles d'ARN et de protéines biomarqueurs dans le exosomes. EFIRM extrait exosomes spécifiquement en fonction de leurs marqueurs de surface par opposition à des techniques basées sur la taille.
Microscopie électronique en transmission (TEM) et le dosage montrent la fonctionnalité du procédé de capture et d'analyse des exosomes. Procédé de EFIRM a été appliqué à exosomales analyse de 9 souris injectées avec des cellules H640 de cancer du poumon humain (une lignée cellulaire transfectée pour exprimer le marqueur d'exosomes CD63-GFP humain) afin de tester leur profil d'exosomes contre 11 souris recevant témoins de sérum physiologique. Des niveaux élevés de biomarqueurs exosomales (GAPDH du gène de référence et de la protéine de surface marqueurs humaine CD63-GFP) ont été trouvé pour le H640 souris injectées dans les deux échantillons de sérum et de salive. En outre, saliVA et des échantillons de sérum ont été démontré que la linéarité (R = 0,79). Ces résultats suggèrent pour la viabilité des biomarqueurs salivaires exosomes pour la détection de maladies distales.
Introduction
recherche Exosome est un domaine émergent de l'enquête qui examine microvésicules lipidiques qui portent une ARN, l'ADN 2, 3 et en protéines cargaison. Enquêtes précédentes de la biologie exosomes ont conduit à l'identification des exosomes dans biofluides tels que le sang 4, 5 urine, le lait maternel 6, 7 et la salive. Des études ont démontré que les exosomes jouent un rôle dans différentes voies cellulaires, méditant à distance communication entre les différents systèmes du corps 8. En raison du rôle exosomes jouent dans la communication intercellulaire, on suppose qu'elles peuvent conditionner biomolécules cibles (protéines, ARN et ADN) corrélés à des états pathologiques. 3 in vitro et de l'animal modèle 9 études semblent confirmer cette hypothèse. En recherchant la teneur exosomal pour la découverte de biomarqueurs, il est nécessaire de développer une méthodologie pour l'isolement de exosome sélective de fluides biologiques, expulsi induiteen charge des cargaisons d'exosomes, et la quantification de biomolécules d'exosomes. Dans la mesure de ce travail, les exosomes seront définis comme une structure ayant un diamètre d'environ 70 à 100 nm et possédant surface marqueur CD63.
Les chercheurs généralement abord purifier exosomes par ultracentrifugation 10 puis traiter le contenu exosomal grâce à l'utilisation de kits de tampon de lyse. Utilisation de méthodes de tampon de lyse nécessite des temps d'incubation allant de quelques minutes à quelques heures. Ce processus peut potentiellement nuire fret exosomes et conduire à la dégradation de l'échantillon. Par exemple, un ARN exosomes salivaire libéré par le tampon de lyse dans le milieu extracellulaire entourant possède une demi-vie de moins de 1 min, ce qui rend la mesure de l'ARN exosomal post-Lysis Buffer une tâche particulièrement difficile sans l'addition de réactifs de stabilisation 11. L'effet composé de l'ajout de divers réactifs pour la lyse et la stabilisation peut introduire des agents qui compliquent et interférer avec l'analysis du contenu de exosomal. Une autre approche peut être utile pour décharger rapidement le contenu de exosomal et la préservation de la cargaison en toute sécurité pour la caractérisation.
Dans ce travail, nous proposons l'utilisation d'un champ électrique non-uniforme pour la libération de contenu exosomal. Champs électriques ont été connus pour porter la capacité à polariser et perturber la bicouche lipidique qui forme les membranes cellulaires. Notre travail expérimental explore l'utilisation de non-uniformes ondes carrées cycliques (CSW) pour perturber la structure de microvésicule des exosomes et de libérer la cargaison transportée. Cette méthode utilise des tensions dans la gamme de plusieurs centaines de millivolts, ce qui signifie que la plupart des biomolécules ne seront pas perturbés. Nous démontrons que l'utilisation d'une onde cyclique carré est apte à actionner libération des exosomes salivaires contenu de l'ARNm dans l'environnement fluide environnant. Cette version de la teneur en exosomal est parfaitement intégré avec un système d'électrodes qui peut être utilisé pour quantifier les niveaux d'expression de biomarqueur 12,13. Cette méthode proposée permet sensibles, et le tampon de lyse analyse rapide, sans contenu de exosomes.

Figure 1. Vue d'ensemble de EFIRM Workflow.. La méthode de EFIRM est globalement divisé en trois grandes phases qui sont nécessaires pour la purification et l'analyse des exosomes.
Cette teneur en exosomal libération et l'analyse sur la base de la méthode CSW est utilisé en conjonction avec des microbilles magnétiques spécifiques à CD63 pour l'isolement d'exosomes. Ces billes permettent CD63 affinité pour l'isolement sélectif des exosomes à partir d'échantillons salivaires (et d'autres fluides biologiques). Après incubation et d'extraction des exosomes en utilisant les perles magnétisées, les perles sont migrés vers le système de capteur électrochimique pour la CSW de presse basée contenu et la partie de l'analyse de l'expérience. La figure 1 donne un aperçu du travailse écouler de la méthode de EFIRM.
Protocole
1. Extraction Exosome magnétique à base de perle
- Pipeter une solution bien mélangée de 5 ul de microparticules magnétiques revêtues de streptavidine dans 495 pl de tampon phosphate salin (PBS) tampon dans un tube de microcentrifugeuse pour remettre en suspension les billes. Lavez et remettre en suspension les perles avec 500 pi de PBS trois fois en utilisant une grille magnétique. Le rack est un ensemble d'aimants sur le côté d'une unité d'habitation qui peuvent maintenir les tubes échantillons à centrifuger.
- Pour chaque lavage, d'abord laisser les tubes se asseoir sur la grille pendant 1 min, puis utilisez une pointe de pipette pour enlever soigneusement le tampon de surnageant sans déranger les perles.
- Placer les tubes sur une grille régulière sans aimants sur le côté. Ajouter 500 pi de PBS dans les tubes, et utiliser la pipette pour mélanger la solution et de perles ensemble. Ensuite, mettre les tubes de retour sur la grille magnétique pour séparer à nouveau les perles de la solution.
- Effectuez cette élimination de tampon par aimantation et remise en suspension dans un PBStrois fois au total. Ceci effectue un lavage initiale des particules magnétiques.
- Remettre en suspension les billes dans 490 pl de tampon PBS, avec le tube placé sur la partie non magnétisée de la crémaillère magnétique. Pipeter 5 ul de l'anticorps CD63 anti-humain de souris biotinylé à 1,0 mg / ml concentration de matière dans le mélange de billes. Utilisez la pipette pour mélanger les perles et l'anticorps en solution.
- Placer les microtubes avec perles et mélange d'anticorps biotinylé sur un échantillon rotateurs. Régler les paramètres des rotateurs pour le dispositif de rotation de l'échantillon pour la rotation réciproque de basculement à 90 ° pendant 5 s et vibrant à 5 ° C pendant 1 sec. Faire tourner les tubes de mélange échantillon-bille à ces paramètres pendant 30 min à température ambiante.
- Retirer anticorps non lié après conjugaison.
- Après 30 min de rotation à la température ambiante, placer les tubes de retour dans le rack magnétique pendant 5 min.
- Effectuer trois lavages de perles en supprimant la phase liquide à l'aide d'une micropipette et laver avec 500 μ; L de PBS. Après la triple lavage, remettre en suspension les perles dans 490 ul de caséine-PBS et le lieu sur la partie non magnétisé de la crémaillère.
- extraction de Exosome en utilisant des billes revêtues d'anticorps.
- Étiqueter chaque tube avec ciblée ID de l'échantillon. Pipeter un échantillon de 10 ul de sérum ou de salive dans le tube de microcentrifugeuse. Utilisez la pipette pour mélanger l'échantillon et des billes magnétiques par pipetage à plusieurs reprises.
- Placer les tubes à échantillon et des billes anti-humains d'anticorps anti-CD63 sur la coiffe et faire tourner pendant 2 heures à température ambiante. Utiliser les mêmes paramètres des rotateurs comme décrit à l'étape 1.2.
- Suite à 2 h de l'échantillon tournant, effectuer un lavage triple en magnétisant à billes séparées de la solution, en éliminant la phase liquide avec la micropipette, et la remise en suspension des billes dans 500 pl de tampon Tris-HCl. Les perles résultantes sont désormais liés aux exosomes et sont prêts pour la libération du champ électrique et de mesure.
2. Champ électrique induite publié une Mesure de la teneur exosomales
- Revêtement préalable initiale de l'électrode avec Primer GADPH
- Appliquer une matière plastique et à un réseau d'électrodes afin d'éviter la contamination croisée des électrodes individuelles. Pour cette expérience, en utilisant un réseau d'électrodes 16 du capteur à chaque électrode de l'unité dans la matrice constituée d'un travail, le compteur, et l'électrode de référence en or nu.
- Préparer un mélange d'actions de 100 nM sonde d'ADN, 0,3 M de KCl, et 10 mM pyrrole par pipetage des réactifs d'achat d'actions dans un tube avec de l'eau distillée ultra pure. Bien mélanger au vortex.
NOTE: Pour cette étude, la sonde d'ADN choisie correspond au gène de référence de GAPDH, qui est connu pour exister dans exosomes. La séquence de la sonde utilisée est: 5'-biotine-AGGTCCACCACTGACACGTTG-3 '. Utilisez ce mélange sur toutes les électrodes. - Pipeter 60 ul du mélange de monomère d'ADN-sonde sur la surface de chaque électrode en or. Examinez les électrodes pour se assurer qu'il ya une couverture adéquate du travail, contre, et des électrodes de référence par le mélange liquide.
- Electropolymerize mélange monomère-sonde pour créer une couche de polymère conducteur sur la surface de l'électrode par application d'une onde carrée (CSW) le profil cyclique de champ électrique à la surface de l'électrode. Ce champ électrique consiste à appliquer 350 mV pour 9 sec et de commutation immédiatement 950 mV pour 1 sec. Appliquer ce profil cyclique onde carrée à l'électrode pendant 10 cycles, pour un total de 100 secondes de champ électrique appliqué.
- Rincer la surface du capteur 3 fois à l'eau distillée et on sèche avec de l'azote gazeux pour éliminer le liquide de la surface de l'électrode. Se assurer que le liquide est correctement supprimé de l'électrode.
- Exosome déchargement de la cargaison
- Charge 5 ul de 1 uM d'une sonde de détection dans 495 ul du mélange complexe de bourrelet exosomes et en utilisant une pipette pour mélanger.
REMARQUE: La sonde de détection est une séquence d'amorce d'ADN conjugué à une molécule de fluorescéine à l'extrémité 3 '. Le détecteur de probe séquence utilisée pour cette étude correspond à l'ARNm GAPDH trouvés dans exosomes. La séquence de la sonde de détection conjugué à la fluorescéine est la suivante: 5'-GCAGTGGGGACACGGAAGGCC-fluorescéine-3 '. - Introduire à la pipette 60 ul de la sonde et de perles exosomes mélange complexe sur la surface d'électrode en or avec un réseau d'aimants dessous. Ce réseau d'aimants est constitué de seize 2,54 mm diamètre des aimants en néodyme alignés pour correspondre aux électrodes de travail du capteur. La figure 2A illustre le placement des aimants et solution de bourrelet exosomes.
- Une fois que l'échantillon est chargé sur la surface de l'électrode, appliquer 20 cycles du champ électrique CSW avec neuf secondes à -300 mV et 1 s à 200 mV (200 du total sec). La cargaison de exosomal qui est libérée va se hybrider aux amorces sur la surface de l'électrode. Si des marqueurs de surface de la exosomes sont l'objet d'enquête, sauter cette partie de l'expérience. Figure 2B illustre ce processus.
- Lavez-off les analytes non liés à la surface de l'électrode par triple rinçage de la surface de l'électrode avec de l'eau distillée. Sécher l'électrode avec de l'azote gazeux.
- Charge 5 ul de 1 uM d'une sonde de détection dans 495 ul du mélange complexe de bourrelet exosomes et en utilisant une pipette pour mélanger.
- Reporter anticorps et Lecture
- Ajouter 60 ul de 150 unité / anticorps anti-fluorescéine ml conjugué à la peroxydase de raifort (HRP en 1: 1000 dilution) dilués dans caséine / PBS.
- Utilisez un champ électrique conjugaison conduit à HRP anti-fluorescéine complexe pour le sandwich de la sonde. Appliquer -200 mV pendant 1 s et 500 mV pendant 1 s à 5 cycles à la surface de l'électrode. La figure 2A montre les complexes de sonde de capture et de détection à la fois une protéine et d'acide nucléique système.
- Surface du capteur de lavage Triple utilisant de l'eau distillée et sécher avec de l'azote gazeux.
- Après le lavage-off de l'excès de l'anticorps anti-fluorescéine non lié, ajouter 60 ul de 3,3 ', 5,5'-tétraméthylbenzidine (TMB). Chargez ce substrat sur chaque surface de capteur à l'aide d'une pipette multicanaux.
- Effectuer une lecture ampérométrique du courant par mesure du courant d'électrode à -200 mV pendant 60 secondes en utilisant un potentiostat capable électrochimique de mesure simultanée de 16 canaux. La figure 2C est un exemple de profil de courant lors de la lecture.
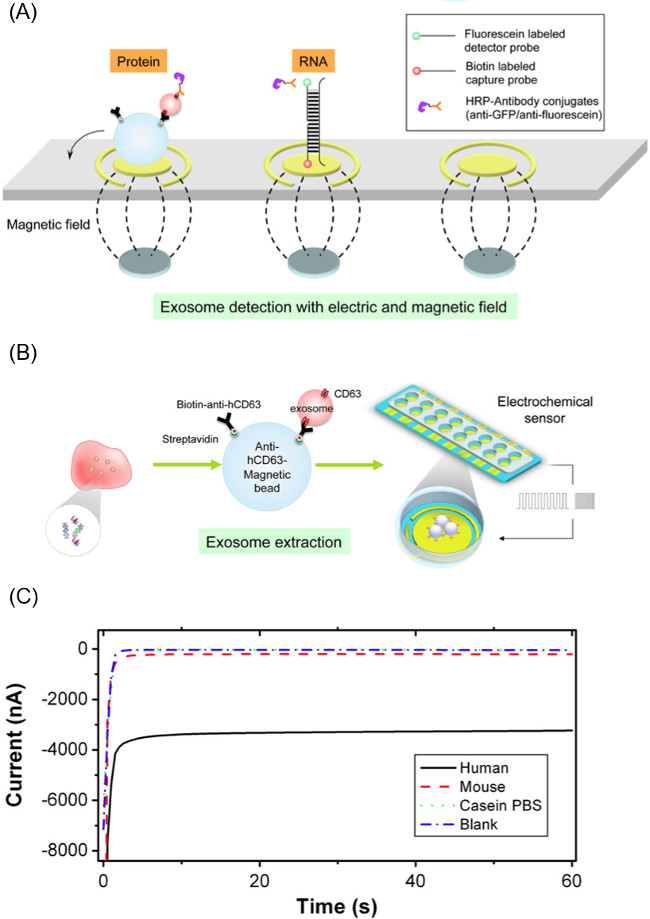
Figure 2. Composants de EFIRM méthode. (A) la méthode d'extraction exosomes de biofluide utilisant CD63 anti-humain revêtu microparticules magnétiques et déchargement de la cargaison de exosomes par ondes carrés cycliques appliquées au complexe de particules exosomes. (B) Schéma de l'électrode biocapteur utilisé pour détecter des cibles ARN / ADN / protéine de la exosomes libérés. (C) un exemple représentatif de lecture ampérométrique de la méthodologie de EFIRM, où plus grande ampleur actuelle correspond to niveaux plus élevés d'une biomolécule. Ce chiffre est de Wei et al. 14 Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
Validation de la capture des exosomes des Perles Utilisation TEM
L'isolement d'exosomes à partir de la salive à l'aide de billes magnétiques anti-CD63 humains a été validée en suivant le protocole d'extraction à l'aide de la microscopie électronique à transmission (TEM) d'images. TEM montre billes magnétiques avec 70 à 100 nm granules immédiatement adjacentes (voir la figure 3A, et 3B), compatibles avec le...
Discussion
Comme les résultats l'indiquent, CD63 anti-humain revêtu nanoparticules magnétiques sont capables de capturer spécifiquement les petites particules qui ont une taille allant de 70 à 100 nm. Cette particule capturée est cohérent avec le profil déjà observé des exosomes. En outre, l'utilisation de la CCF basse tension suivant la capture des particules est indiquée pour les retirer de la surface des billes et causer des profils de dégradation d'ADN similaires à celle d'une méthode traditionnel...
Déclarations de divulgation
David Wong est co-fondateur de RNAmeTRIX Inc., une société de diagnostic moléculaire. PeriRx LLC sous-licence de propriété intellectuelle se rapportant aux diagnostics moléculaires de RNAmeTRIX. David Wong est un consultant pour PeriRx.
Remerciements
Ce travail a été soutenu par le National Center for Research Resources et le Centre national pour l'avancement des sciences translationnelle, National Institutes of Health, par Grant UL1TR000124 (FW); Félix & Mildred Yip Doué chaire et le Fonds famille Barnes (à DTWW), l'Institut national de recherche dentaire et craniofaciale des Instituts nationaux de la santé en vertu Prix Nombre T90DE022734 (MT). Le contenu est de la seule responsabilité des auteurs et ne représentent pas nécessairement les vues officielles des National Institutes of Health.
matériels
| Name | Company | Catalog Number | Comments |
| Helios 16-Channel Reader System with Chip Interface | Genefluidics, USA | RS-1000-16 | |
| 16x Sensor Chip, Bare Gold, pack of 5 chips | Genefluidics, USA | SC1000-16X-B | |
| Biotinylated anti-human CD63 Antibody | Ancell, USA | 215-030 | |
| Dynabeads MyOne Streptavidin T1 | Invitrogen, USA | 65601 | |
| Neodynium Magnetics (1/10" dia. x 1/32" thick) | K&J Magnetics, USA | DH101 | |
| Ultrapure Distilled Water | Life Technologies, USA | 10977-023 | |
| Mettler Toldeo 3 M KCl Solution | Fisher Scientific, USA | 1911512 | |
| Pyrrole | Sigma-Aldrich, USA | W338605-100g | |
| Anti-Fluorescein-POD, Fab fragments | Roche, Germany | 11426346910 | |
| 3,3′,5,5′-tetramethylbenzidine substrate (TMB/H2O2, low activity) | Neogen, Usa | 330175 | |
| Phosphate Buffered Saline Solution | Life Technologies, USA | 10010023 | |
| Casein/PBS | Fisher Scientific, USA | 37532 |
Références
- Rabinowits, G., Gerçel-Taylor, C., Day, J. M., Taylor, D. D., Kloecker, G. H. Exosomal MicroRNA: A Diagnostic Marker for Lung Cancer. Clinical Lung Cancer. 10 (1), 42-46 (2009).
- Thakur, B. K., et al. Double-stranded DNA in exosomes: a novel biomarker in cancer detection. Cell Research. 24 (6), 766-769 (2014).
- Lau, C. S., Wong, D. T. W. Breast Cancer Exosome-like Microvesicles and Salivary Gland Cells Interplay Alters Salivary Gland Cell-Derived Exosome-like Microvesicles In. Vitro. PLoS ONE. 7 (3), e33037 (2012).
- Bala, S., et al. Circulating microRNAs in exosomes indicate hepatocyte injury and inflammation in alcoholic, drug-induced, and inflammatory liver diseases. Hepatolog. 56 (5), 1946-1957 (2012).
- Dear, J. W., Street, J. M., Bailey, M. A. Urinary exosomes: A reservoir for biomarker discovery and potential mediators of intrarenal signalling. Proteomics. 13 (10-11), 1572-1580 (2013).
- Lässer, C., et al. Human saliva, plasma and breast milk exosomes contain RNA: uptake by macrophages. Journal of Translational Medicine. 9 (1), 9 (2011).
- Palanisamy, V., et al. Nanostructural and Transcriptomic Analyses of Human Saliva Derived Exosomes. PLoS ONE. 5 (1), e8577 (2010).
- Camussi, G., et al. Exosomes/microvesicles as a mechanism of cell-to-cell communication. Kidney International. 78 (9), 838-848 (2010).
- Lau, C., et al. Role of pancreatic cancer-derived exosomes in salivary biomarker development. The Journal of Biological Chemistry. 288 (37), 26888-26897 (2013).
- Théry, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and Characterization of Exosomes from Cell Culture Supernatants and Biological Fluids. Current Protocols in Cell Biology. 3 (22), (2001).
- Park, N. J., Li, Y., Yu, T., Brinkman, B. M. N., Wong, D. T. Characterization of RNA in Saliva. Clinical Chemistry. 52 (6), 988-994 (2006).
- Wei, F., et al. Bio/Abiotic Interface Constructed from Nanoscale DNA Dendrimer and Conducting Polymer for Ultrasensitive Biomolecular Diagnosis. Small. 5 (15), 1784-1790 (2009).
- Wei, F., et al. Electrochemical Sensor for Multiplex Biomarkers Detection. Clinical Cancer Research. 15 (13), 4446-4452 (2009).
- Wei, F., Yang, J., Wong, D. T. W. Detection of exosomal biomarker by electric field-induced release and measurement (EFIRM). Biosensors and Bioelectronics. 44, 115-121 (2013).
Erratum
Formal Correction: Errata: Detection of Exosomal Biomarker by Electric Field-induced Release and Measurement (EFIRM)
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for Detection of Exosomal Biomarker by Electric Field-induced Release and Measurement (EFIRM). The disclosures were updated.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon