Method Article
PCR en temps réel basée sur une sonde Approches de mesure quantitative des microARN
Dans cet article
Résumé
MicroARN qui circulent ont récemment émergé comme biomarqueurs prometteurs et novateurs en vue de divers cancers et d'autres maladies. Le but de cet article est de discuter de trois plateformes de PCR différents basés sonde en temps réel et les méthodes qui sont disponibles pour quantifier et déterminer l'abondance des microARN circulation.
Résumé
PCR quantitative basée-Probe (qPCR) est une méthode privilégiée pour mesurer l'abondance des transcrits, puisque ce est l'une des méthodes de détection les plus sensibles qui fournit une analyse précise et reproductible. La chimie à base de sonde offre le moins de fluorescence d'arrière-plan par rapport aux autres chimies (à base de colorants). Actuellement, il existe plusieurs plates-formes disponibles que la chimie basée sur une sonde d'utilisation pour quantifier l'abondance des transcrits. qPCR dans une plaque de 96 puits est la méthode la plus couramment utilisée, mais seulement un maximum de 96 échantillons ou miARN peut être testée en un seul passage. Ce est fastidieux et pénible si un grand nombre d'échantillons / miRNAs sont à analyser. Plateformes sonde haut-débit tels que la microfluidique (par exemple TaqMan tableau Card) et des tableaux de nanofluidique (par exemple OpenArray) offre de facilité de détecter de façon reproductible et efficacement l'abondance de plusieurs microARN dans un grand nombre d'échantillons en peu de temps. Ici, nous démontrons le dispositif expérimental d'unee protocole pour la quantification de miARN échantillons de sérum ou plasma EDTA, en utilisant la chimie à base de sonde et de trois plates-formes différentes (plaque de 96 puits, la microfluidique et les tableaux de nanofluidique) offrant des niveaux croissants de débit.
Introduction
Les microARN (miARN) sont ~ 22 nucléotides non codante (NC) ARN, fonctionnant comme régulateurs de l'expression du gène 1-3. La plupart des miARN chez les animaux fonctionnent par appariement de bases spécifique de séquence avec un ARNm, ciblant l'extrémité 3 'UTR, ce qui conduit à une régulation négative de l'expression de gène 2-4. Cela se produit généralement par l'intermédiaire de l'inhibition de la traduction de l'ARNm ou en ribosomique de débarquement. miARN en circulation se sont révélés être de nouveaux biomarqueurs dans la recherche et les domaines cliniques pour une variété de maladies telles que le diabète, 7.5, 8 ovaire, de la prostate et cancer du sein 9 10,11, 12 l'hépatite B et d'autres 13 maladies auto-immunes. Des recherches ont été menées pour identifier les miRNAs abondantes dans différentes cellules ou des tissus, ainsi que dans la circulation du plasma humain et des échantillons de sérum, ce qui est plus accessible et moins invasive 9,11-15.
Différentes méthodes de quantification miARN ont été eréé en utilisant de multiples plates-formes, telles que la plate-forme de plaque standard 96 puits 4,12,16-18, la plate-forme de carte microfluidique 12,18-23 et la plate-forme de tableau 17,24 nanofluidique. Quantitative PCR en temps réel (qPCR) offre la possibilité de mesurer les nombres relatifs ou absolus de transcriptions utilisant multiple (teinturerie ou à base de sonde-chimiques). La chimie PCR en temps réel basée sur une sonde offre l'avantage d'une faible fluorescence de fond et une haute sensibilité pour détecter un seul exemplaire de la transcription. Il est relativement rentable, simple à utiliser et hautement reproductible, ce qui en fait une méthode privilégiée pour quantifier et déterminer miRNA expression 25. Méthode qPCR base-Probe implique généralement deux étapes: la transcription (RT) et qPCR 4,26,27 inverser. RT est l'endroit où l'amorce tige-boucle RT est hybridée à une molécule miARN mature ou primaire et convertie en (c) un ADN complémentaire. La quantification du produit de l'ADNc est ensuite effectuée en utilisant des amorces de PCR spécifiques miARN26-28. Le principe de la PCR quantitative à base de sonde est basée sur la détection de l'extension du brin complémentaire en temps réel, ce qui implique l'hydrolyse de la sonde marqués par fluorescence. Ces sondes sont conçues pour contenir un rapporteur fluorescent et un extincteur qui sont tout autre pour permettre FRET (de Fluorescence Resonance Energy Transfer). La détection de l'émission de la fluorescence rapporteur (émetteur) est masquée par la proximité de la molécule d'agent d'extinction. Lorsque la Taq polymerase (pol) se étend à partir de l'amorce amont et atteint une fourche (de l'extrémité 5 'de la sonde), l'activité pol d'exonucléase de la Taq hydrolyse de la sonde, ce qui conduit à une dissociation / séparation physique de l'émetteur fluorescent à partir de l'extincteur. Cette version d'une seule molécule de la fluorescence émetteur est enregistré par le détecteur et présenté comme une augmentation progressive du signal de fluorescence de ce puits / réaction. L'augmentation de la fluorescence est proportionnelle à la quantité de produit de PCR généré, ce qui permet une précision qu26,28 antification de la cible amplifiée.
Avec l'augmentation de la demande dans miRNA quantification, à moyen et à des technologies à haut débit ont été développés pour permettre à un plus grand nombre d'échantillons à traiter dans un court laps de temps. TaqMan Low Density Array (TLDA) est un débit moyen de conception innovante basée sur microfluidique à base de sonde-qPCR chimie offrant une augmentation du nombre de miARN analysés sur une plaque. TLDA impliquent l'utilisation d'un pool prédéfini de RT-amorces qui sont utilisées pour synthétiser l'ADNc. Ces ADNc sont ensuite filées en une carte personnalisée 384 et micro-fluidique pour déterminer l'expression de plusieurs miARN utilisant qPCR 22,26,29. Chaque puits de la carte contient séché amorces et des sondes pour amplifier miARN (s) spécifique, donc à 384 réactions peuvent être transformés dans la carte unique TLDA 26.
La matrice de nanofluidique est une plate-forme à haut débit qui est utilisé pour la détection de produits de transcription de gènes 24 en utilisant la même chimie du sonde. Il utilise une matrice exclusive offrant interactions hydrophobes hydrophile pour faciliter le chargement facile du mélange de réaction 33 en nanolitres en un tableau de 3072 trous traversants sur une lame en acier inoxydable 24. Cet article se concentre sur la démonstration de la façon dont ces méthodes pour quantifier miARN dans le sérum / plasma sont effectuées et les facteurs critiques qui doivent être considérés lors de l'exécution et l'interprétation de ces données. Prises pour compte, leurs avantages et leurs limites individuelles seront discutés dans cet article.
Protocole
L'ARN total peut être isolé à partir de sérum en utilisant un protocole établi dans notre laboratoire 30 ou en utilisant d'autres kits disponibles dans le commerce.
REMARQUE: tableurs interactifs supplémentaires pour le calcul des volumes de réaction dans chaque expérience (avec le volume en excès de 5% représentaient pipetage inclus) sont prévus.
1. base Probe-qPCR en temps réel en utilisant un standard de 96 puits Quai
- la synthèse d'ADNc (de transcription inverse) sur des échantillons d'ARN de sérum / plasma pour miRNA
- Calculer et sortir 10 ng d'ARN pour chaque réaction de synthèse d'ADNc. Ajouter de l'eau sans nuclease à amener le volume final de 10 ng d'ARN à 1,67 ul (pour une réaction 5 ul). Garder les échantillons sur la glace.
- Sélectionnez le miARN et dégeler leurs amorces de RT.
- Décongeler les RT composants de mélange réactif: RT Buffer (10X), dNTP (100 mM), RNase Inhibitor (20 U / pl). Ne gardez jamais lieu enzyme virus (recombinant Moloney de la leucémie murine(RMoMuLV)) (virus de Moloney de la leucémie murine recombinanat (rMoMuLV) la transcriptase inverse (50 U / ul) sur de la glace. La stocker à -20 ° C jusqu'à utilisation ou dans un bloc de congélation.
- Préparer RT mélange de réactifs sur la glace comme décrit dans le tableau 1A. Il est recommandé de préparer au moins 5% du volume en excès pour compenser une erreur de pipetage.
- Ajouter 2,33 pi du mélange de réactifs RT chaque dans un tube PCR 0,2 ml / plaque.
- Vortex les tubes d'amorces RT pour mixer, puis brièvement centrifuger à 10 000 g pendant 10 sec. Ajouter 1 ul de la RT miRNA amorce spécifique à leurs tubes respectifs de PCR ou plaque. Ajouter 1,67 pi de l'ARN dilué à leurs tubes ou puits de PCR respectives dans la plaque. Effectuer tous les ajouts sur la glace.
- Centrifuger le tube de réaction ou une plaque à 1950 g pendant 5 min à 4 ° C.
- Mettre en place le programme de synthèse de l'ADNc miRNA sur le thermocycleur avec les conditions de réglage suivantes: 16 ° C pendant 30 min, 42 ° C pendant 30 min, 85 °C pendant 5 min et 4 ° C en attente.
- Régler le volume de réaction à 10 pi. Charger les tubes de réaction ou plateau en thermocycleur. Démarrez la course RT. Conserver les réactions ADNc (RT) à -20 ° C, si le temps réel amplification PCR ne est pas immédiatement procédé.
- Probe-base en temps réel qPCR pour la détection de miARN matures
- Décongeler les tests choisis en fonction sonde-qPCR (20x) pour le produit de RT respective.
- Mélanger la seule solution de régularité de PCR en temps réel (qPCR mélange de réactifs) en agitant la bouteille.
- Préparer le mélange de réactifs qPCR pour chaque miARN à analyser. Pour préparer la qPCR obtenir un tube de 1,5 ml de microcentrifugation stérile pour chaque échantillon miARN et ajouter les composants dans chaque tube tel que décrit dans le tableau 1B. Il est recommandé de préparer au moins 5% du volume en excès pour compenser une erreur de pipetage.
- Ajouter 4,2 pi du mélange de réactifs qPCR respectif à chaque puits d'une plaque à 96 puits optique (ou tubes). Ajouter 0,8 ulde la réaction d'ADNc respective (synthétisé dans l'étape 1.1 pour chaque miARN) bien respective.
- Sceller la plaque avec couvercle optique approprié. Centrifuger la plaque (ou tubes) à 1950 g pendant 5 min à 4 ° C
- Allumez l'ordinateur, plaque de 96 puits / microfluidique lecteur de tableau et enfin lancer le logiciel. Se assurer que les machines sont correctement connectés et correcte bloc et couvercle chauffant (pour rapide plaque de 96 puits) sont en place.
- Sélectionnez expérience que 96 puits bloc rapide (0,1 ml), courbe standard, réactifs TaqMan et le mode rapide. Mettre en place qPCR sur le système PCR en temps réel en utilisant les conditions de cycle de programme suivant: 95 ° C pendant 20 sec, 50 cycles (95 ° C pendant 1 sec, 60 ° C pendant 30 s).
- Régler le volume de réaction à 10 pi. Charger le tube de réaction ou de la plaque dans l'appareil. Appuyez sur "Démarrer, Exécuter". Le programme prendra environ 1 heure pour terminer.
2. base-Probe microfluidique tableau Card (Cae A et B)
REMARQUE: base-Probe panneau miRNA vient comme un ensemble de deux cartes 384 puits microfluidiques (Array carte A et Array carte B). Chaque carte contient amorces séchées et sondes pour jusqu'à 380 miARN et des contrôles. produit d'ADNc (avec ou sans pré-amplification) spécifique à une carte ou une carte B est chargé sur le réseau respectif pour la PCR en temps réel.
- Transcription inverse (RT)
- Utilisez ce protocole pour une entrée d'ARN total de 1-1000 ng. Si l'entrée est entre 1-350 ng, effectuer une étape de pré-amplification (pré-ampli). Pour les entrées supérieures à 350 ng, charger ADNc directement sur les cartes du tableau sans pré-ampli.
NOTE: Pour miRNA sérique profilage, commencer avec 100 ng d'ARN total. Pour un profil complet miRNA, exécuter deux piscines d'ensembles prédéfinis RT-amorces de réactions RT (Groupe A et Pool B) par échantillon. - Décongeler les réactifs suivants sur la glace. RT Primaires (10x): Groupe A et Groupe B, dNTP avec dTTP (100 mM), Tampon RT (10x), MgCl 2 (25 mM), RNase Inhibitor (20 U / &# 181; l). Ne gardez pas l'enzyme (virus recombinant Moloney de la leucémie murine (rMoMuLV)) de la transcriptase inverse (50 U / ul) sur la glace. Conservez-la à -20 ° C jusqu'à ce que nécessaire.
- Vortexer doucement tous les réactifs, sauf l'enzyme, puis centrifuger brièvement les tubes à 10 000 g pendant 10 sec.
- Moissonneuse réactifs, comme décrit dans le tableau 2A, dans deux tubes; un tube pour le groupe A, l'autre pour la poule B. Chaque tube contient qu'une seule piscine prédéfini de RT ensemble d'amorces, soit à partir de la poule A ou de la poule B. Il est recommandé de préparer au moins 5% volume en excès pour compenser une erreur de pipetage .
- Mélanger par inversion, et brièvement, puis centrifuger à 10 000 g pendant 10 sec. Aliquote de 100 ng de chaque échantillon d'ARN dans un nouveau tube, puis ajouter un volume approprié d'eau sans nucléase pour faire un total de 3 pi.
- Aliquote 4,5 pi du mélange de réactifs RT appropriée dans des tubes respectifs. Mélanger par inversion, puis centrifuger brièvement à 10 000 g pendant 10 sec. Incuber sur de la glacependant 5 min.
- Placer les échantillons dans un thermocycleur et démarrer le RT en utilisant les conditions suivantes: 40 cycles (16 ° C pendant 2 min, 42 ° C pendant 1 min, 50 ° C pendant 1 s), 85 ° C pendant 5 min, maintien à 4 ° C. Magasin d'ADNc généré à l'aide de ceux-ci piscine prédéfini de RT-amorces à -15 à -25 ° C ou utilisé immédiatement.
- Utilisez ce protocole pour une entrée d'ARN total de 1-1000 ng. Si l'entrée est entre 1-350 ng, effectuer une étape de pré-amplification (pré-ampli). Pour les entrées supérieures à 350 ng, charger ADNc directement sur les cartes du tableau sans pré-ampli.
- Pré-amplification
- Décongeler la piscine prédéfini d'amorces pré-ampère sur la glace. Doucement amorces de vortex, puis centrifuger brièvement à 10.000 g pendant 10 sec. Swirl TaqMan PreAmp mélange de réactifs (2X) en tapotant doucement, pour mélanger.
- Moissonneuse réactifs, comme décrit dans le tableau 2B, en deux tubes; un tube pour le groupe A, l'autre pour la poule B.
NOTE: Comme décrit ci-dessus, Piscine amorces A pré-ampère iront à tube A et les amorces Piscine B devraient aller dans le tube B. Il est recommandé de préparer au moins 5% volume en excès pour compenser une erreur de pipetage. - Mélanger par inversion, puis centrifuger brièvement à 10 000 g pendant 10 sec.Aliquote 22,5 pi du mélange de réactif PreAmp approprié dans de nouveaux tubes. 2,5 ul d'aliquote de l'échantillon d'ADNc (préparé à l'étape RT) dans le tube respectif.
- Mélanger par inversion, puis centrifuger brièvement à 10 000 g pendant 10 sec. Incuber sur de la glace pendant 5 minutes. Placer les échantillons dans un thermocycleur et démarrer le cycle de pré-ampli. Les conditions de cyclage: 95 ° C pendant 10 min, 55 ° C pendant 2 min, 72 ° C pendant 2 min, 12 cycles (95 ° C pendant 15 sec, 60 ° C pendant 4 min), 99,9 ° C pendant 10 min, maintenir à 4 ° C.
- Inverser pré-amplifiée ADNc pour mélanger, puis centrifuger brièvement à 10 000 g pendant 10 sec. Ajouter 75 ul de tampon 0,1 x TE (pH 8,0) de l'ADNc de pré-amplifié (dilution 1: 4). Inversez échantillons de pré-ampère dilué de mélanger, puis centrifuger brièvement à 10 000 g pendant 10 sec. Magasin dilution, avant ADNc amplifié à -15 à -25 ° C pendant jusqu'à une semaine, ou utilisé immédiatement.
- Chargement des cartes microfluidiques et la réalisation qPCR
- Gardez la microfluidiquecartes miRNA l'extérieur pendant au moins une demi-heure pour atteindre la température ambiante. Décongeler dilué pré-ADNc amplifié sur la glace et mélanger en inversant les tubes puis brève rotation. Mélanger qPCR mélange de réactifs en agitant la bouteille.
- En nouveau tube ajouter 450 pi de mélange de réactifs qPCR à 9 pi de dilution ADNc pré-amplifiée. Pour la carte A, utiliser pré-amplifié produit préparé en utilisant l'amorce poule A. Ajouter 441 ul d'eau sans nucléase pour compenser le volume final à 900 ul. Inverser le tube pour mélanger, puis centrifuger brièvement à 10 000 g pendant 10 sec.
- Retirez la carte de son emballage TLDA (une fois qu'il atteint la température ambiante) et placez-le sur une surface propre avec le côté de la feuille vers le bas. Ajouter 100 ul de mélange réactionnel de PCR dans chacun des orifices de remplissage 8 de la carte. Il existe deux ports de chacun des réservoirs réservoir (8 au total) sur chaque carte.
REMARQUE: Le port de remplissage est le plus grand trou où le mélange réactionnel est ajouté, tandis que l'orifice d'évent est le plus petit trou. Chaque carte possède 8 miRNA TLDA réservoirs, chacun conduisant à 48 puits (24 puits dans une colonne de 2 x colonnes), en chargeant ainsi l'ensemble des puits 384 dans la carte avec le même mélange de réaction PCR. Veiller à ce que le groupe A et le groupe B de chaque échantillon sont chargés sur les cartes TLDA respectifs. Placez la carte de tableau dans les tableaux spécialisés microfluidique seaux titulaire de la carte dans la centrifugeuse. - Pour les cartes de réseau de microfluidique, utiliser une centrifugeuse appropriée (comme Heraeus Multifuge 3SR, 230V centrifugeuse) et seaux spécifiques (tels que les détenteurs de cartes "TaqMan Array»). Chaque godet peut contenir jusqu'à trois cartes (chargé / vide). Toujours se assurer que tous les trois fentes d'un seau sont occupés et le seau est équilibré en plaçant seau similaire (ce seau devrait également contenir trois cartes, soit vides ou pleines) dans la fente en face de la centrifugeuse. En plaçant la carte dans le porte-seau, assurez-vous que les huit réservoirs projettent vers le haut et puits de réaction face à la paroi extérieure de la centrifugeuse.
- Faites tourner les cartes à 331 g pendant 1 min à température ambiante. Aprèspremière rotation, ouvrir la centrifugeuse et se assurer visuellement le mélange de réaction a été distribué à travers les 384 puits. Répétez le spin au mêmes paramètres pour une fois de plus. Retirez la carte de seau et se assurer que le niveau de mélange réactionnel dans chacun des huit réservoirs est uniforme. Les incohérences dans les volumes de liquide dans les réservoirs gauche font la carte inapproprié d'utiliser davantage.
- des cartes de réseau de Microfluidique besoin d'un scellant spécialisé qui a un assemblage de précision de stylet (chariot) pour sceller les canaux de distribution de fluide de la matrice et également distribuer le mélange réactionnel dans tous les puits (volume total de réaction 1 pl / puits).
- Apportez le chariot vers la position de départ et insérez la carte chargée dans le scellant avec le côté de la feuille et la queue pour les broches de stylet sur le scellant. Dans une course lente et uniforme, stable et simple, pousser le chariot sur la carte jusqu'à ce qu'il atteigne le point de le scellant final. Retirez la carte de réseau fermé et puis coupez le réservoirs de la carte à l'aide de ciseaux.
- Se assurer que le bloc correct, le couvercle chauffé et l'échantillon support est installé dans le temps réel de la matrice de système microfluidique PCR / machine.
- Allumez l'ordinateur, puis le système de tableau de la microfluidique et enfin lancer le logiciel. Se assurer que les machines sont correctement connectés. Sélectionnez expérience que la carte de tableau, courbe standard, réactifs TaqMan et le mode standard. Importez les fichiers d'installation pour soit de la carte A et B. carte Enregistrez le fichier.
- Placez la carte scellée dans le plateau à instruments avec puits A1 dans le coin supérieur gauche et code à barres vers l'avant de l'instrument. Appuyez sur "Start Run". Le programme prendra environ deux heures à compléter.
Panneau Nanofluidique miARN humain basée-Probe 3.
- Transcription inverse (RT)
- Utilisez ce protocole pour une entrée d'ARN total de 50-200 ng cependant 100 ng est optimale pour la plupart des échantillons. Pour un profil complet de miARN, exécutez deux prédéfipiscines nies d'ensembles RT-amorces de réactions RT (Groupe A et Pool B) par échantillon.
- Décongeler les réactifs sur la glace: piscine prédéfini d'ensembles d'amorces RT (10x), dNTP avec dTTP (100 mM), Tampon RT (10x), MgCl 2, (25 mM), RNase Inhibitor (20 U / pl). Ne gardez pas l'enzyme (virus de Moloney recombinant de la leucémie murine (rMoMuLV) transcriptase inverse (50 U / ul) sur la glace. Conservez-la à -20 ° C jusqu'à ce que nécessaire. Vortex doucement tous les réactifs, sauf l'enzyme, et brièvement centrifuger eux à 10 000 xg pendant 10 sec.
- Moissonneuse réactifs, comme décrit dans le tableau 3A, en deux tubes; un tube pour le groupe A, l'autre pour la poule B. Chaque tube contient qu'une seule piscine ensemble prédéfini de RT-amorce, soit à partir de la poule A ou de la poule B. Il est recommandé de préparer au moins 5% volume en excès pour compenser pipetage erreur.
- Pipette pour mélanger et brièvement, puis centrifuger à 10 000 g pendant 10 sec. Aliquote de 100 ng de chaque échantillon d'ARN dans un nouveau tube, puis ajouter un volume appropriéd'eau sans nucléase pour faire un total de 3 pi. Aliquote 4,5 pi du mélange de réactifs RT approprié dans le tube respectif.
- Mélanger par inversion, puis centrifuger brièvement à 10 000 g pendant 10 sec. Incuber sur de la glace pendant 5 min. Placer les échantillons dans un thermocycleur et démarrer le programme RT. Les conditions de cyclage: 40 cycles (16 ° C pendant 2 min, 42 ° C pendant 1 min, 50 ° C pendant 1 s), 85 ° C pendant 5 min, maintien à 4 ° C. ADNc de magasin généré en utilisant ces piscine prédéfini de RT-amorce fixé à -15 à -25 ° C ou utiliser immédiatement.
- Pré-amplification (pré-ampli)
- Décongeler la piscine prédéfini de pré-ampli amorce mis sur la glace. Doucement amorces vortex, puis centrifuger brièvement à 10.000 g pendant 10 sec. Swirl TaqMan PreAmp mélange de réactifs (2X) pour mélanger.
- Moissonneuse réactifs, comme décrit dans le tableau 3B, en deux tubes; un tube pour le groupe A, l'autre pour la poule B. Chaque tube contient qu'une seule piscine prédéfini d'ensembles RT-amorces, soit from Piscine A ou de la poule B. Il est recommandé de préparer au moins 5% volume en excès pour compenser pipetage erreur.
- Pipette pour mélanger, puis centrifuger brièvement eux à 10 000 g pendant 10 sec. Aliquote 22,5 pi du mélange de réactif PreAmp approprié dans de nouveaux tubes. 2,5 ul d'aliquote de l'échantillon d'ADNc dans le tube respectif. Mélanger par inversion, puis centrifuger brièvement les tubes à 10 000 g pendant 10 sec. Incuber sur de la glace pendant 5 min.
- Placer les échantillons dans un thermocycleur et courent le pré-ampli. Régler le volume de réaction à 25 pi. Les conditions de cyclage: 95 ° C pendant 10 min, 55 ° C pendant 2 min, 72 ° C pendant 2 min, 12 cycles (95 ° C pendant 15 sec, 60 ° C pendant 4 min), 99,9 ° C pendant 10 min, maintenir à 4 ° C.
- Inverser pré-amplifiée ADNc pour mélanger, puis centrifuger brièvement à 10 000 g pendant 10 sec. Dans les nouveaux tubes ajouter 4 ul d'ADNc de pré-amplifiée à 156 pi de tampon TE pH 0,1 x 8,0 (dilution 1:40). Inversez échantillons de pré-ampère dilué pour mélanger, puis brièvement CEntrifuge à 10 000 xg pendant 10 sec. Magasin dilué et l'ADNc de pré-amplifiée non dilué à -15 à -25 ° C pendant jusqu'à une semaine, ou d'utiliser immédiatement.
- Chargement Nanofluidique tableaux et exécution qPCR
- Télécharger le fichier de la plaque correspondante (.tpf) sur le site 31 en utilisant le numéro de série de diapositives de tableau de nanofluidique. Celui-ci contient les informations de l'exécution de la diapositive de groupe spécifique de nanofluidique.
- Thaw dilué mélange de réactifs ADNc et TaqMan en temps réel qPCR pré-amplifiée (si vous utilisez pour la première fois) sur la glace. Mélanger qPCR mélange de réactifs en agitant la bouteille. Dans les nouveaux tubes ajouter 22,5 pi de mélange de réactifs qPCR à 22,5 ul de dilution, avant ADNc amplifié. Vortexer doucement pour mélanger, puis centrifuger brièvement à 10 000 g pendant 10 sec.
- Aliquote 5 pi de chaque échantillon dans huit puits (2 colonnes, 4 rangées) de la nanofluidique tableau flux de travail de 384 puits plaque d'échantillon. Assurez-vous que le groupe A et le groupe B de chaque échantillon sont dans des blocs de 8 puits adjacents (voir Figure 1 ou complémentaire feuille Excel pour la présentation).
- Chaque plaque de l'échantillon peut contenir jusqu'à huit diapositives valeur d'échantillons. Cependant, le système de nanofluidique tableau ne peut traiter quatre tableaux de nanofluidique en une seule opération. Si plus de quatre diapositives valeur d'échantillons doivent être chargés sur une plaque d'échantillon, se il vous plaît se assurer que les sections restantes sont scellés. Sceller avec OpenArray plaque échantillon scellant.
NOTE: Il est conseillé d'effectuer une pré-coupé le scellant dans les sections nécessaires, de sorte que les sections peut être scellé / descellé individuellement pour réduire l'évaporation. Alternativement, la plaque peut être scellé avec un scellant intacte, puis sections peut être coupé individuellement lorsque le chargement.
- Chaque plaque de l'échantillon peut contenir jusqu'à huit diapositives valeur d'échantillons. Cependant, le système de nanofluidique tableau ne peut traiter quatre tableaux de nanofluidique en une seule opération. Si plus de quatre diapositives valeur d'échantillons doivent être chargés sur une plaque d'échantillon, se il vous plaît se assurer que les sections restantes sont scellés. Sceller avec OpenArray plaque échantillon scellant.
- Centrifugeuse à assiettes à 490 g pendant 1 min à 4 ° C. Charger les lames de array nanofluidique à 1 h. En raison du temps limité accordé pour sceller les diapositives, se il vous plaît charger une seule lame à la fois. Retirer la lame nanofluidique de tableau du congélateur et laissez-le venir à la température ambianteambiante (~ 15 min).
- Assurez-vous que le bloc correct, couvercle chauffant et l'échantillon support est installé dans le système nanofluidique de tableau. Allumer l'ordinateur, et un système PCR en temps réel et le système de chargement. Accès au logiciel respectif et se assurer que les machines sont connectées. Retirez les consommables du système de chargement (Array couvercle coulissant, les plug et fluides d'immersion) de l'emballage.
- Tirez doucement sur le piston de la seringue de liquide d'immersion pour desserrer. Enlever le capuchon, placez astuce sur l'air et au ras de la pointe. Placez les pointes de système de chargement dans la machine et retirer le couvercle. Placez la plaque de l'échantillon dans le système PCR.
- Mettre des gants. Assurez-vous qu'ils sont bien ajusté afin de minimiser le risque de marquage accidentellement le couvercle coulissant. Ouvrir soigneusement l'emballage de diapositive. Basculer lentement glisser dans la main. Ne pas toucher le haut de la diapositive.
- Placer les lames dans le système PCR, avec le code à barres sur la gauche. Retirer de la partie de scellement de la plaque d'échantillon destiné à être chargé. Utilisez le système de chargementpour saisir le code à barres de slide, glisser la position, la position de l'échantillon et de la configuration de la pointe.
- Lorsque tous les contrôles nécessaires soient terminées, toboggan presse de charge. Bien que le système PCR chargement la diapositive, retirer le plastique transparent et rouge à partir du bas du couvercle coulissant. Lorsque vous avez terminé le chargement, retirez soigneusement et sceller la diapositive dans les 90 sec.
- Placer la lame à l'intérieur de la pince de plaque. Placez le couvercle de glisser sur la diapositive. Collier de serrage pour 30 sec. Se assurer que le couvercle est placé de sorte que code à barres se affiche correctement. Retirez l'ensemble de la fixation de la plaque.
- Position immersion fluide seringue dans la diapositive pour que la pointe est en appui contre le couvercle. Remplir lentement diapositive avec liquide d'immersion, se assurer que les pistes fluide le long du couvercle. Une fois plein, fermez la lame avec le bouchon, tourner la vis jusqu'à la poignée se détache.
- Retirez le couvercle en plastique sur le dessus du couvercle de la diapositive, puis placer soigneusement dans le support de diapositives du système PCR en temps réel. Assurez-vous qu'il est support sur le fond de la glissière comme il est abaissé, de sorte qu'il ne tombe pas brusquement et ne pas toucher la partie supérieure de la lame. Ce est OK pour toucher les côtés du système / cassette.Initialize la PCR de diapositives et démarrer le programme pour qPCR à 1 h.
- Sélectionnez "OpenArray" dans le logiciel PCR-système. Appuyez sur "Trouver ID de diapositives". Cela prendra quelques minutes. Si le logiciel ne peut pas trouver l'ID de la plaque, il demandera à être entré manuellement.
- Appuyez sur "Confirmer Centres de plaque". Encore une fois, cela va prendre quelques minutes. Vérifiez que le point rouge se trouve dans le centre et qu'il n'y a pas d'empreintes digitales / marques sur le haut de la diapositive. Chargez le fichier .tpf respective pour chaque diapositive et indiquez un nom de fichier de résultat et l'emplacement. Appuyez sur "Start Run". Le programme prendra environ deux heures à compléter.
Résultats
Le volume recommandé pour une réaction qPCR de dosage à base de sonde miARN est de 20 pi. REMARQUE: Nous avons confirmé que un volume réactionnel de 5 ul est capable de produire des résultats similaires à ceux obtenus en utilisant 20 ul de volume 4,7,30. L'abaissement du volume de réaction de 5 ul permet une diminution de 75% dans les coûts de réactifs sans perte appréciable de la sensibilité. Comme présenté sur la figure 2, les volumes de réaction de 20 ul et 5 ul montrent une corrélation forte jusqu'à 39 cycles (avec R 2 de 0,92, p = 0,0002).
réseau de microfluidique fournit un outil pour obtenir des données sur 754 miARN exprimés dans un échantillon d'environ 5 heures (pour carte A et B carte), qui est un moyen plus efficace d'analyser de multiples échantillons par rapport aux classiques 96 PCR de la plaque ainsi. Nous avons comparé miRNA cartes du tableau de la microfluidique pour le même échantillon (échantillon A et de l'échantillon A répétition) Figure 3A -. B montre un graphique de Bland-Altman ( 3A) et l'intrigue de corrélation (3B) pour tous les 754 miARN testés pour ces échantillons. Il ya trois miARN de contrôle différents (U6, RNU44 et RNU48) placés au hasard sur les deux cartes (carte A et B) dans de multiples endroits. Lorsque le seuil de cycle de U6 (CT) valeurs sont comparées entre les deux courses, nous ne avons pas observé de différences significatives entre les valeurs (tableau 4). Il est également important de noter ici que U6 est exprimé à plus grande abondance (valeur inférieure Ct) dans l'échantillon évalué. Nous avons ensuite comparé les miARN qui ont des valeurs Ct entre 0 à 19,99 dans les deux pistes (n = 150), qui avait expression similaire des miARN globale avec un coefficient de détermination de 98% (figure 3C-D). De tous les 277 miARN qui ont des valeurs Ct entre 20 et 29,99 dans les deux pistes, 16 Mirs différaient significativement entre les pistes originales et les reprises (Figure 3E - F) Le nombre de miARN avec différence significative betwee.n les pistes ont augmenté (89 sur 327) lorsque les valeurs de Ct ont été sélectionnés entre 30-40 pour les deux courses (Figure 3G - H).
La plate-forme nanofluidique tableau fournit des données pour 754 miARN de chaque échantillon sérum / plasma testées, comme représenté sur la figure 4A Il est important d'examiner ces courbes d'amplification -. Comme il est avec tous qPCR - pour se assurer que le résultat est révélateur d'une véritable amplification. Chacun des 48 sous-réseaux (Figure 1) contient également un test pour les trois "ménage" ncRNAs plus populaires:. U6, RNU44 et RNU48 figure 4B illustre un regroupement typique de U6 reproduit à partir d'un seul échantillon. Ces répétitions affichent faible déviation standard (SD <0,5) et sont donc un indicateur de la fiabilité. Alternativement, la figure 4C montre la variabilité accrue des répétitions de U6 (SD> 0,5) dans un second échantillon. Ce ne nie pas la validité of les essais restants, bien qu'il ne nécessite une critique plus approfondie. U6, comme avec la plupart miARN "d'entretien" dans les liquides biologiques, peut avoir une expression variable. Il convient de noter que l'un des échantillons, décrites dans la figure 4C, affiche quatre fois moins de contenu U6 que celle présentée dans la figure 4B. Puisque le niveau de U6 dans l'échantillon présenté dans 4C est 75% de moins pour commencer que celui présenté dans le panneau 4B, une plus grande variabilité technique est prévu en raison de la distribution de Poisson des transcriptions, qui est exacerbé par le faible volume de réaction 17.
Un autre outil utile est les images de contrôle de la qualité (CQ), disponibles à l'exportation une fois la course terminée. Une sélection de ceux-ci utilise la fluorescence du ROX, le colorant passif trouvé dans le mélange de réactifs qPCR, pour confirmer que chaque trou de passage a été chargé correctement (figure 5). Un trou de passage, voire une salle pneu sous-réseau, ne peut pas charger en raison du volume d'échantillon insuffisant, l'évaporation, des bulles présentes dans les puits de la plaque de l'échantillon de 384 puits, le défaut de supprimer complètement le joint de la plaque de l'échantillon, ou des défauts dans le système Accufill ou de ses conseils. Toute déchargés trous traversants doivent être identifiés afin d'éviter miARN d'étiquetage «indétectable», alors qu'en réalité, le test n'a jamais été chargé. Si ce problème est rencontré, confirmer que au moins 5 pi de l'échantillon / mastermix est chargé dans chaque puits de la plaque de l'échantillon de 384 puits, la plaque d'échantillon est correctement centrifugé avant le chargement, le papier d'aluminium est complètement enlevée, et l'OpenArray chargé diapositive est scellé et exécuté dans le délai imparti pour tous les essais successifs. Si des problèmes de chargement persistent, ils peuvent être plus susceptibles relatives à lot spécifique ou un lot des tableaux ou des consommables connexes et obtenir de l'aide devrait être recherchée par le fabricant.
"Src =" / files / ftp_upload / 52586 / 52586fig1.jpg "/>
Figure 1: Disposition des échantillons pour le tableau flux Nanofluidique: (A) Chaque plaque d'échantillon 384 puits peut contenir des échantillons jusqu'à huit tableaux de nanofluidique. (B) Dilué, ADNc pré-amplifiée est placé dans huit puits (2 colonnes par quatre lignes), avec piscine A et le groupe B en groupes de 8 puits adjacents. Chaque cercle représente un puits. (C) Chaque puits de la plaque de l'échantillon sera chargé dans un sous-réseau de la matrice de nanofluidique. Chaque petit carré représente une sous-tableau.
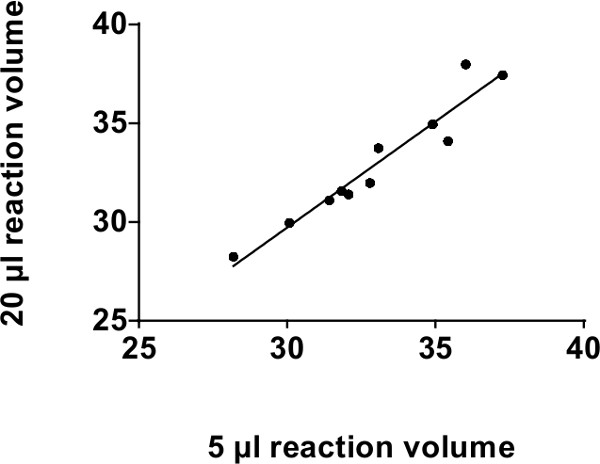
Figure 2: L'analyse des co-relation pour les plateformes classiques 96 puits PCR: Co-relation entre 20 pi et 5 volumes de réaction de pi sur TaqMan en temps réel qPCR utilisant une plate-forme de plaque standard 96 puits dans les valeurs CT (39 cycles). Nous avons comparé quatre différents microARN (miR-375, miR-30c, miR-30d et miR-7) dans quatre échantillons différents de sérum et de plasma humains. Seuls les 11 points de données sont tracés puisque les autres étaient indétectables. R 2 = 0,92, p = 0,0002.
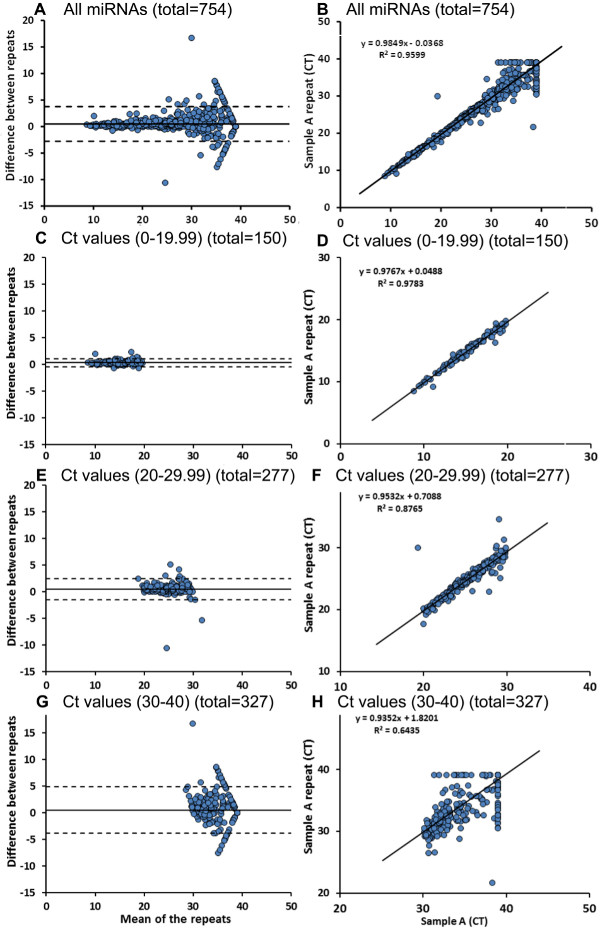
. Figure 3: circulation miRNA profilage à l'aide de cartes microfluidique tableau en utilisant deux cartes microfluidiques (carte-A et de la carte-B), un profil de 754 miARN est généré (A - B). Comme le montre ici, nous avons utilisé même échantillon pour deux tableau microfluidique exécute pour vérifier la reproductibilité des résultats de cartes de la microfluidique. Nous avons observé une expression similaire des miARN dans l'ensemble, avec des valeurs Ct entre 0 à 19,99 (C - D). Il ya peu de miARN (16 de 277) avec les valeurs de Ct entre 20 à 29,99 et significatives différences entre répétition pistes (E - F). Quatre-vingt neuf des 327microARN avec les valeurs de Ct plus élevés (30 à 40) présentaient des différences significatives entre les deux manches (G - H). Les données sont analysées en utilisant le test t apparié. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
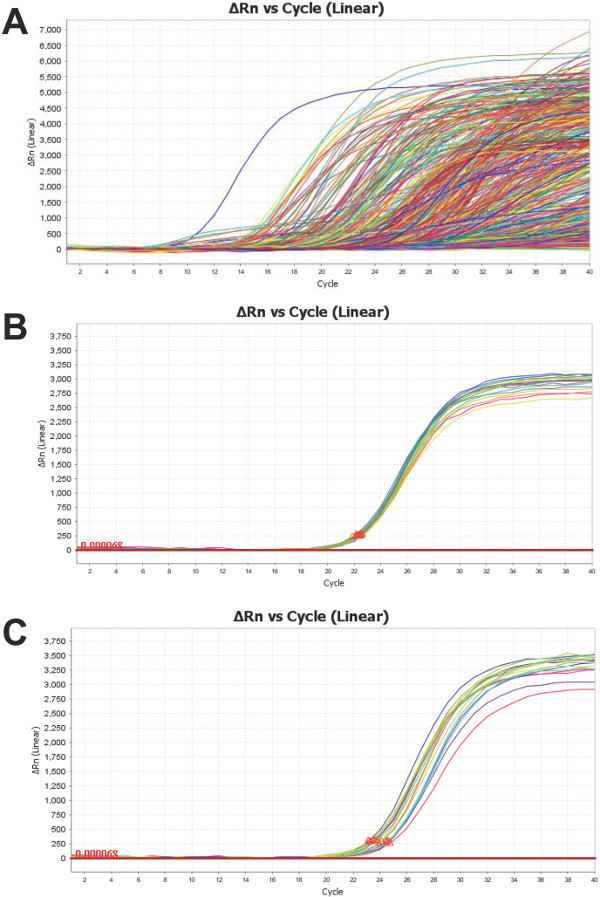
Figure 4: Profil des courbes représentant l'amplification par PCR produit: est une figure représentative des courbes (A) d'amplification combinés de toutes les cibles miARN pour un échantillon de plasma humain. Un dosage de U6 (un contrôle commun ARNnc) est placé dans chaque sous-réseau. L'échantillon en (B) présente une faible variabilité (SD <0,5), tandis que (C) montre écart-type élevé (SD> 0,5) dans U6 réplique. Les deux échantillons sont total ARN isolé à partir de plasma humain. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 5: Analyse des Nanofluidique QC tableaux: contrôle de la qualité (CQ) des images d'un tableau nanofluidique chargé correctement (à gauche) et un tableau nanofluidique mal chargé (à droite). Le colorant passif, ROX (présent dans le mélange de réactifs qPCR), émet une fluorescence à indiquer un trou traversant correctement chargé. Le tableau sur la droite dispose de plusieurs sous-réseaux / trous traversants qui ne sont pas chargées avec le mélange de réactifs qPCR et ceux-ci doivent être identifiés comme des réactions PCR faussement négatifs.
| Composants | Volume réactionnel par 5 ul (ul) |
| Tampon RT (10x) | 0,5 |
| dNTP (100 mM) | 0,05 |
| Inhibiteur RNase | 0,6 |
| L'eau sans nucléase | 1,39 |
| Transcriptase inverse | 0,33 |
| Le volume total | 2,33 |
Tableau 1A: RT composants de mélange réactif dans un 5 pi RT préparation de réaction pour TaqMan en temps réel qPCR utilisant une plate-forme de plaque de 96 puits standard.
| Composants | Volume réactionnel par 5 ul (ul) | Volume par réaction 20 ul (ul) |
| Mastermix PCR rapide (2x) | 2,5 | 10 |
| Dosage TaqMan PCR quantitative (20x) * | 0,25 | 1 |
| Sans nucléaseeau | 1,45 | 5,8 |
| Volume total: | 4.2 | 16,8 |
* Composant de dosage basé sur le miRNA choisi testé.
Tableau 1B: qPCR composants de mélange réactif dans un 5 ou 20 ul qPCR préparation de réaction pour TaqMan en temps réel qPCR utilisant une plate-forme de plaque de 96 puits standard.
| Composants | Volume par réaction (ul) |
| Megaplex RT Primaires (10x) | 0,8 |
| dNTP avec dTTP (100 mM) | 0,2 |
| MultiScribe la transcriptase inverse (50 U / ul) | 1,5 |
| 10X RT Buffer | 0,8 |
| MgCl 2 (25 mM) | 0,9 |
| RNase Inhibitor & # 160; (20 U / ul) | 0,1 |
| Eau sans nucléase | 0,2 |
| Total | 4.5 |
Tableau 2A: RT composants de mélange réactif dans un 5 pi RT préparation de réaction pour le panneau TLDA miARN.
| Composants | Volume par réaction (ul) |
| TaqMan PreAmp Mastermix (2x) | 12,5 |
| Megaplex PreAmp Primaires (10x) | 2,5 |
| L'eau sans nucléase | 7,5 |
| Total | 22,5 |
Tableau 2B: PreAmp composants de mélange réactif dans une préparation de réaction 25 pi pré-amplification pour le panneau TLDA miARN.
"Cellspacing =" 0 "> Composants Volume par réaction (ul) Megaplex RT Primaires (10x) 0,75 dNTP avec dTTP (100 mM) 0,15 MultiScribe la transcriptase inverse (50 U / ul) 1,5 10X RT Buffer 0,75 MgCl 2 (25 mM) 0,9 Inhibiteur de la RNase (20 U / ul) 0,09 Eau sans nucléase 0,35 Total 4.5Tableau 3A: RT composants de mélange réactif dans un 5 pi RT préparation de réaction pour le panneau nanofluidique miARN basé sonde.
| Composants | Volume par réaction (ul) |
| TaqMan PreAmp Mastermix (2x) | 12,5 |
| Megaplex PreAmp Primaires (10x) | 2,5 |
| L'eau sans nucléase | 7,5 |
| Total | 22,5 |
Tableau 3B:. PreAmp composants réactifs de mélange dans une préparation de réaction 25 pi pré-amplification pour le panneau nanofluidique miARN base de sonde REMARQUE: Le complémentaire interactive feuilles de calcul Excel fournies pour les trois plates-formes, représente déjà l'ajout de 5% afin de compenser pour le pipetage erreur.
| Échantillon A | Exemple Une répétition |
| 15,535 | 16,156 |
| 15,471 | 15,652 |
| 15.623 | 16,063 |
| 15,963 | 15,889 |
| 14,006 | 13,993 |
| 14,502 | 14,623 |
| 14,907 | 14,384 |
| 13,732 | 14,946 |
Tableau 4: circulation miRNA profilage U6 microfluidique carte de tableau. L'utilisation de deux cartes de réseau microfluidique (A et B). Les valeurs Ct de miARN de commande de U6 de l'échantillon A (total = 8). Contrôle de U6 Conformément miRNA abondance observée entre les pistes.
Discussion
Les étapes critiques dans les protocoles de qPCR basée sonde à obtenir des résultats précis et reproductibles pour se assurer 1) le même volume et la concentration de RT-produit est chargé dans chaque réaction qPCR, 2) les rapports et les volumes corrects des composants nécessaires pour réaction qPCR sont préparés et bien mélangé, 3) les volumes corrects et cohérents sont ajoutés à chaque réaction qPCR, et 4) la préparation et le chargement de chaque échantillon et mélange réactionnel est terminée dans le plus court laps de temps possible, tout en conscient de les étapes critiques ci-dessus mentionnés plus tôt.
Les cartes de réseau microfluidique besoin centrifugeuse appropriée et seaux spécifiques. Chaque godet peut contenir jusqu'à trois cartes (chargé / vide). Toujours se assurer que tous les trois fentes d'un seau sont occupés et le seau est équilibré en plaçant seau similaire (ce seau devrait également contenir trois cartes, soit vides ou pleines) dans la fente en face de la centrifugeuse. En plaçant la carte dans le Buckeporte-t, assurez-vous que les huit réservoirs projettent vers le haut et puits de réaction face à la paroi extérieure de la centrifugeuse. Faites tourner les cartes à 331 g pendant 1 min à température ambiante. Après la première centrifugation, ouvrir la centrifugeuse et se assurer visuellement le mélange de réaction a été distribué à travers les 384 puits. Répétez le spin au mêmes paramètres pour une fois de plus. Retirez la carte de seau et se assurer que le niveau de mélange réactionnel dans chacun des huit réservoirs est uniforme. Les incohérences dans les volumes de liquide dans les réservoirs gauche font la carte inapproprié d'utiliser davantage.
Pour avoir la même concentration de la RT-produit, même concentration d'entrée de l'ARN est ajoutée dans chaque réaction RT. La concentration d'ARN total est mesuré en utilisant un micro-volumespectrophotometer. Le ratio observé 260/280 peut être aussi faible que 1,3 pour l'ARN isolé à partir de plasma / sérum; cela ne semble pas avoir un effet sur le processus ou les données liées qPCR-aval généré 30. De même, le rapport de 260/280les échantillons d'ARN testés ici sont entre 1.3 à 1.7 sans effets anormaux dans le qPCR observés.
Lors de l'utilisation des échantillons à faible teneur d'ARN, tels que ceux de fluides biologiques, il peut être difficile de quantifier l'ARN avant le traitement. Nous recommandons l'utilisation du synthétique pic-in à l'isolement de l'ARN ainsi que les étapes de transcription inverse. Dans notre expérience, les candidats Arabidopsis thaliana (miARN miR-ath-159a et ath-miR-172a) sont préférés Caenorhabditis elegans miARN (tels que cel-miR-39 ou cel-miR-54), qui, dans notre expérience peut avoir plus homologie que ceux de A. thaliana. L'utilisation de tels spécifique du stade pic-in peut tenir compte de la normalisation des données miRNA sur plusieurs échantillons analysés à différents moments. L'utilisation d'un volume d'entrée fixe de l'ARN pour la réaction de synthèse d'ADNc est également recommandé 32,33.
Les trois protocoles basés sonde-miARN pour la quantification décrites ici nécessitent des quantités variablesde l'entrée de l'ARN total, différents flux de travail et les coûts. Chacun des flux de travail sont conçus pour répondre à différents débits basés sur le nombre de cibles miARN et le nombre d'échantillons à analyser. Avec l'augmentation de débit (96 RXN 384 RXN 3072 RXN), le coût par réaction diminue avec une augmentation de la quantité de données obtenues au cours d'une unité de temps. Étant donné que toutes ces plates-formes utilisent chimie TaqMan, la qualité des données obtenues peut se attendre à être similaires. TaqMan qPCR est une méthode bien établie pour identifier l'abondance des miARN dans les échantillons de sérum / plasma 9,11,27. Bien que les trois plates-formes discutées ici ont la même composition chimique, une diminution dans le volume de réaction conduit à une diminution de la gamme dynamique de détection de transcription (RJ Farr et al., Données non publiées). Le 96 puits plateforme qPCR est un débit plus faible, mais la plate-forme de haute sensibilité et à notre avis, le «gold standard» pour toutes les plateformes de PCR (ou colorant à base de) basée sonde. Cependant, cela peutne pas être la plate-forme la plus économique ni efficace si plusieurs centaines ou milliers d'échantillons sont analysés et pour plusieurs microARN. Microfluidique (TLDA) et nanofluidique (OA) plates-formes sont des plates-formes à haut débit / contenu conçus pour permettre l'acquisition de données plus grands dans un temps plus court. Bien que les différences de lots ont été observés dans les cartes TLDA, ce peut être minimisée en demandant TLDA cartes du même lot. Nous observons que la plate-forme TLDA (figure 3) montré une variation significative dans 17% des mi - miARN de faible abondance lorsqu'il est testé en utilisant le même échantillon sur différents lots de cartes TLDA. Nous vous recommandons donc d'utiliser les mêmes numéros de lot pour l'analyse en utilisant des cartes TLDA. Cette variation pourrait aussi être due à la variabilité technique, y compris des erreurs de chargement et de pipetage. Cependant, nous conseillons de commander / demandant le même lot de cartes TLDA. Aucune variation significative n'a été observée lot sur la plate-forme de l'arthrose. Malgré cela, TaqMan expéri basetal qPCR approches offrent facilité pour mesurer l'abondance des miARN dans les échantillons de plasma / sérum. Les approches de débit faible, moyenne et haute discutés ici offrent la flexibilité d'analyser une série d'échantillons et miARN l'aide d'un (faible bruit de fond) la chimie hautement efficace, reproductible et propre.
Déclarations de divulgation
Article coûts de transformation / de publication ont été couverts par Life Technologies, après acceptation de ce manuscrit pour publication.
Remerciements
Tous les auteurs reconnaissent le soutien du NHMRC CTC, Université de Sydney, la Fondation de recherche sur le diabète juvénile (FRDJ), l'Australie et la Fondation Rebecca Cooper, l'Australie infrastructure. Cette recherche a été financée par des subventions du Conseil australien de la recherche (FT110100254) et la FRDJ, Australie (CRN201314) pour AAH WW, RJF et MVJ effectuée tous laboratoire humide expérimentation, ASJ effectué l'analyse des données. WW a écrit la première ébauche. AAH prévu l'étude et analysé les données. Tous les auteurs ont lu et approuvé la version finale du manuscrit, des chiffres et des feuilles de travail soumis pour publication.
matériels
| Name | Company | Catalog Number | Comments |
| 96 well-platform | |||
| For Reverse transcription | |||
| TaqMan MicroRNA Assays INV SM | Applied Biosystems | 4427975 | https://www.lifetechnologies.com/au/en/home/life-science/pcr/real-time-pcr/real-time-pcr-assays/mirna-ncrna-taqman-assays/single-tube-mirna-taqman-assays.html?ICID=search-4427975 The assays comes as a pack of RT primers and PCR primer. |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366597 or 4366596 | https://www.lifetechnologies.com/order/catalog/product/4366597?ICID=search-4366597 or http://www.lifetechnologies.com/order/catalog/product/4366596?ICID=search-4366596 The TaqMan MicroRNA Reverse Transcription Kit is the same kit used for reverse transcription in all the threeTaqMan platform- 96-well platform, TLDA and OpenArray. |
| For qPCR run on a 96-well platform | |||
| 2x TaqMan Fast Universal PCR Master Mix, no AmpErase UNG | Applied Biosystems | 4367846 | https://www.lifetechnologies.com/order/catalog/product/4367846?ICID=search-4367846 The 2X TaqMan Fast Universal PCR Master Mix is used for qPCR on the two TaqMan platforms-96-well platform and TLDA. |
| MicroAmp Optical Adhesive Film | Applied Biosystems | 4311971 | https://www.lifetechnologies.com/order/catalog/product/4311971?ICID=search-product |
| MicroAmp Fast Optical 96-Well Reaction Plate, 0.1 ml | Applied Biosystems | 4346907 | https://www.lifetechnologies.com/order/catalog/product/4346907?ICID=search-product |
| Microfluidics platform (TLDA) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | https://www.lifetechnologies.com/order/catalog/product/4399966?ICID=search-4399966 The Megaplex RT Primers, Human Pool A v2.1 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex RT Primers, Human Pool B v3.0 | Applied Biosystems | 4444281 | https://www.lifetechnologies.com/order/catalog/product/4444281?ICID=search-4444281 The Megaplex RT Primers, Human Pool B v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| OR | OR | ||
| Megaplex Primer Pools, Human Pools Set v3.0 | Applied Biosystems | 4444750 | http://www.lifetechnologies.com/order/catalog/product/4444750?ICID=search-4444750 The Megaplex Primer Pools, Human Pools Set v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| For Pre-amplification | |||
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 or 4488593 | https://www.lifetechnologies.com/order/catalog/product/4391128?ICID=search-4391128 or http://www.lifetechnologies.com/order/catalog/product/4488593 The TaqMan PreAmp Master Mix is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool B v3.0 | Applied Biosystems | 4444303 | https://www.lifetechnologies.com/order/catalog/product/4444303?ICID=search-4444303 The Megaplex PreAmp Primers, Human Pool B v3.0 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399233 | https://www.lifetechnologies.com/order/catalog/product/4399233?ICID=search-4399233 The Megaplex PreAmp Primers, Human Pool A v2.1 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| 1x TE Buffer (100 ml) | Invitrogen | 12090-015 | https://www.lifetechnologies.com/order/catalog/product/12090015?ICID=search-product The 1x TE Buffer is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| To load and run the 384 well microfluidics (TLDA) card | |||
| TaqMan Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | https://www.lifetechnologies.com/order/catalog/product/4444913 |
| Nanofluidics platform (OpenArray) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| For Pre-amplification | |||
| Refer to TLDA pre-amplification reagents (as shown above) | |||
| To load and run the slides | |||
| TaqMan OpenArray Human MicroRNA Panel, QuantStudio 12K Flex (1 panel) | Applied Biosystems | 4470187 | https://www.lifetechnologies.com/order/catalog/product/4470187?ICID=search-4470187 |
| QuantStudio 12K Flex OpenArray Accessories Kit | Applied Biosystems | 4469576 | https://www.lifetechnologies.com/order/catalog/product/4469576?ICID=search-4469576 |
| OpenArray 384-well Sample Plates | Applied Biosystems | 4406947 | https://www.lifetechnologies.com/order/catalog/product/4406947?ICID=search-4406947 |
| TaqMan OpenArray Real-Time PCR Master Mix | Applied Biosystems | 4462159 | https://www.lifetechnologies.com/order/catalog/product/4462159?ICID=search-4462159 |
| OpenArray AccuFill System Tips | Applied Biosystems | 4457246 | https://www.lifetechnologies.com/order/catalog/product/4457246?ICID=search-4457246 |
| Others | |||
| Nuclease-Free Water | Qiagen | 129117 | http://www.qiagen.com/products/catalog/lab-essentials-and-accessories/nuclease-free-water |
Références
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Alvarez-Garcia, I., Miska, E. A. MicroRNA functions in animal development and human disease. Development. 132, 4653-4662 (2005).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-297 (2004).
- Joglekar, M. V., Wei, C., Hardikar, A. A. Quantitative estimation of multiple miRNAs and mRNAs from a single cell. Cold Spring Harb Protoc. 2010, pdb prot5478(2010).
- Joglekar, M. V., Parekh, V. S., Hardikar, A. A. Islet-specific microRNAs in pancreas development, regeneration and diabetes. Indian J Exp Biol. 49, 401-408 (2011).
- Farr, R. J., Joglekar, M. V., Taylor, C. J., Hardikar, A. A. Circulating non-coding RNAs as biomarkers of beta cell death in diabetes. Pediatr Endocrinol Rev. 11, 14-20 (2013).
- Joglekar, M. V., Joglekar, V. M., Hardikar, A. A. Expression of islet-specific microRNAs during human pancreatic development. Gene Expr Patterns. 9, 109-113 (2009).
- Iorio, M. V., Croce, C. M. MicroRNA profiling in ovarian cancer. Methods Mol Biol. 1049, 187-197 (2013).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. P Natl Acad Sci USA. 105, 10513-10518 (2008).
- Mattie, M. D., et al. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer. 5, 24(2006).
- Zhu, W., Qin, W., Atasoy, U., Sauter, E. R. Circulating microRNAs in breast cancer and healthy subjects. BMC Res Notes. 2, 89(2009).
- Zhu, H. T., et al. Identification of suitable reference genes for qRT-PCR analysis of circulating microRNAs in hepatitis B virus-infected patients. Mol Biotechnol. 50, 49-56 (2012).
- Yamada, H., Itoh, M., Hiratsuka, I., Hashimoto, S. Circulating microRNAs in autoimmune thyroid diseases. Clin Endocrinol (Oxf). , (2014).
- Liu, R., et al. Serum MicroRNA Expression Profile as a Biomarker in the Diagnosis and Prognosis of Pancreatic Cancer. Clinical Chemistry. 58, 610-618 (2012).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PLoS One. 3, e3148(2008).
- Joglekar, M. V., Parekh, V. S., Mehta, S., Bhonde, R. R., Hardikar, A. A. MicroRNA profiling of developing and regenerating pancreas reveal post-transcriptional regulation of neurogenin3. Dev Biol. 311, 603-612 (2007).
- Hardikar, A. A., Farr, R. J., Joglekar, M. V. Circulating microRNAs: understanding the limits for quantitative measurement by real-time PCR. J Am Heart Assoc. 3, e000792(2014).
- Wu, C., et al. Diagnostic and Prognostic Implications of a Serum miRNA Panel in Oesophageal Squamous Cell Carcinoma. PLoS One. 9, e92292(2014).
- McDonald, M. K., Capasso, K. E., Ajit, S. K. Purification and microRNA profiling of exosomes derived from blood and culture media. J Vis Exp. , e50294(2013).
- Joglekar, M. V., et al. The miR-30 family microRNAs confer epithelial phenotype to human pancreatic cells. Islets. 1, 137-147 (2009).
- Genda, Y., et al. microRNA changes in the dorsal horn of the spinal cord of rats with chronic constriction injury: A TaqMan(R) Low Density Array study. Int J Mol Med. 31, 129-137 (2013).
- Wang, B., et al. Systematic evaluation of three microRNA profiling platforms: microarray, beads array, and quantitative real-time PCR array. PLoS One. 6, e17167(2011).
- Cuk, K., et al. Circulating microRNAs in plasma as early detection markers for breast cancer. Int J Cancer. 132, 1602-1612 (2013).
- Morrison, T., et al. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34, e123(2006).
- Git, A., et al. Systematic comparison of microarray profiling, real-time PCR, and next-generation sequencing technologies for measuring differential microRNA expression. RNA. 16, 991-1006 (2010).
- Benes, V., Castoldi, M. Expression profiling of microRNA using real-time quantitative PCR, how to use it and what is available. Methods. 50, 244-249 (2010).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res. 33, e179(2005).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44, 31-38 (2008).
- Kodani, M., et al. Application of TaqMan low-density arrays for simultaneous detection of multiple respiratory pathogens. J Clin Microbiol. 49, 2175-2182 (2011).
- Taylor, C. J., Satoor, S. N., Ranjan, A. K., Pereirae Cotta,, V, M., Joglekar, M. V. A protocol for measurement of noncoding RNA in human serum. Exp Diabetes Res. 2012, 168368(2012).
- TPF & SPF File Download Options [Internet]. , Available from: http://www.appliedbiosystems.com/absite/us/en/home/products/tpf-spf-download.html (2014).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic Acids Res. 39, 7223-7233 (2011).
- Li, Y., Kowdley, K. V. Method for microRNA isolation from clinical serum samples. Anal Biochem. 431, 69-75 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon