Method Article
Corrélative confocale et microscopie électronique 3D d'une cellule sensorielle spécifique
Dans cet article
Résumé
Here, we introduce a method, cocem3D, to unveil the ultrastructure of a specific cell in its native tissue by bridging confocal and serial block-face scanning electron microscopy.
Résumé
Délimitation de l'ultrastructure d'une cellule est importante pour comprendre sa fonction. Cela peut être un projet de taille pour les types de cellules rares diffusés tout au long de tissus faits de divers types de cellules, comme les cellules entéro de l'épithélium intestinal. Ces capteurs gastro-intestinaux de l'alimentation et les bactéries ont été difficiles à étudier parce qu'elles sont dispersées parmi d'autres cellules épithéliales dans un rapport de 1: 1000. Récemment, des souris transgéniques rapporteurs ont été générés pour identifier des cellules entéro-endocrines au moyen de fluorescence. Un de ceux est le peptide YY-GFP souris. L'utilisation de cette souris, nous avons développé une méthode pour corréler confocale et microscopie électronique à balayage d'îlot de série. Nous avons nommé la méthode cocem3D et appliqués pour identifier une cellule entéroendocrine spécifique du tissu et de dévoiler la ultrastructure de la cellule en 3D. La résolution de cocem3D est suffisante pour identifier les organites aussi petit que vésicules de sécrétion et de distinguer les membranes cellulaires pour le volume rendering. Cocem3D peut être facilement adapté pour étudier l'ultrastructure 3D d'autres types de cellules spécifiques dans leur tissu natif.
Introduction
Vie à l'intérieur d'une cellule se déroule dans le temps et l'espace. Changements dans le temps sont souvent étudiés en utilisant la microscopie time-lapse combinée à des techniques d'imagerie de fluorescence, comme la microscopie de super-résolution. Espace, en particulier la disposition des organites à l'intérieur d'une cellule ou d'interactions de cellule à cellule, ne peut être obtenue par un compte rendu complet de la structure fine de la cellule. Un compte rendu complet de la structure fine d'une cellule peut aussi apporter de la clarté à la fonction génomique dans les cas où le génome est disponible, comme le C. elegans nématode 1 ou la Placozoaires tricoplax plat adhérentes 2. Microscopie électronique de sectionnement de série est maintenant reproductible, le temps efficace et moins coûteux tâche grâce au développement des technologies de microscopie électronique 3D automatisés, comme la microscopie électronique à balayage série d'îlot 3 (SBEM).
Le besoin d'information structurelle pour élucider la fonction est très évident dans certaines cell types où la fonction dépend des interactions physiques de cellule à cellule, comme les neurones, les cellules gliales, cellules épithéliales ou sensorielles. Nous sommes particulièrement intéressés à élucider comment les signaux sensoriels de nutriments dans le lumen de l'intestin sont transduits en un signal électrique qui module finalement comportements appétitives. Le circuit est complexe, mais commence à la paroi de l'intestin où les éléments nutritifs viennent en contact avec les cellules epitheliales sensorielles, appelées cellules entéro-endocrines. Contrairement à d'autres cellules epitheliales sensorielles, telles que des cellules gustatives, des cellules entéro-endocrines sont dispersées tout au long de l'épithélium intestinal dans un rapport de un à mille 7.4. En conséquence, ils ont été difficiles à identifier et étudier, et pour longtemps, ils ont été vu seulement comme une source d'hormones de l'intestin. Mais avec le développement de souris fluorescence rapporteurs spécifiques des cellules, la fonction sensorielle complexe de ces cellules est en train d'émerger. En utilisant une de ces souris rapporteurs, un peptide YY-GFP (PyyGFP) souris, nous avons trouvé que enteroendocrine cellules ont une queue cytoplasmique de premier plan que nous avons nommé neuropod. L'apparition de neuropods suggéré une fonction conservée dans la communication de cellule à cellule. Ainsi, nous avons pensé que, en documentant l'ultrastructure d'une cellule d'entéroendocrines, la fonction de neuropods pourrait être dérivée.
La nécessité de comprendre la structure d'un appendice dans une cellule dispersée qui est difficile à identifier était la raison principale pour développer une méthode pour combiner la microscopie confocale et SBEM. La cellule d'intérêt a été identifié en utilisant des souris rapporteurs spécifiques des cellules de PyyGFP. La méthode a permis de documenter l'ensemble ultrastructure d'une cellule entéroendocrine et son neuropod. Dans neuropods, nous avons trouvé les caractéristiques structurelles des axones neuronaux, et à l'extérieur neuropods, nous avons trouvé une relation physique à cellules gliales entériques 8. En effet, neuropods contiennent environ 70% de tous les vésicules sécrétoires qui suggère un rôle essentiel dans la fonction de sécrétion de ces cellules. Construire sur ledes données structurelles, et plus récemment, nous avons trouvé que, grâce à ces neuropods, des cellules entéro-endocrines et les neurones innervant l'intestin former un circuit neuroépithéliale, similaire à celle des cellules gustatives dans la langue 8,9.
Découvrir ces caractéristiques de mécanismes chimiosensoriels gastro-intestinaux refoulées à partir de données recueillies structurelles en utilisant cette méthode de microscopie corrélative. Nous croyons que cette méthode pourrait être utile dans d'autres domaines de la biologie cellulaire, en particulier lorsque les cellules sont dispersées dans les tissus à un taux très faible. Nous nous sommes référés à la méthode que confocale corrélative et la microscopie électronique à balayage face du bloc de série en 3D (Cocem3D). Le procédé comprend les étapes principales suivantes: dissection des tissus, la microscopie confocale, imagerie par SBEM, SBEM image confocale et la corrélation, et la segmentation manuelle. Comparé à d'autres méthodes de corrélation, le concept est assez simple parce que la corrélation est physique plutôt que chimique.
Protocole
Tous les soins des animaux et expériences ont été réalisées conformément à un protocole approuvé par le Comité institutionnel de protection et d'utilisation des animaux de l'Université Duke.
Cocem3D découle d'une combinaison de protocoles précédemment publiés 10,11 et a été appliqué ici pour étudier une cellule entéroendocrine spécifique en utilisant un journaliste souris PyyGFP 12. Ce procédé peut facilement être appliqué à d'autres cellules d'intérêt en utilisant des souris transgéniques rapporteuses disponibles dans le commerce. La méthode est décrite en trois sections: microscopie corrélative confocale, microscopie électronique à balayage bloc-face série (SBEM), et de données de rendu en 3D.
Corrélative microscopie confocale
L'objectif de cette section est de récolter un segment de tissu du côlon distal des dimensions qui permettent confocale corrélative et l'imagerie SBEM. Le protocole est le suivant:
1. Exploitation du Tissue
- Préparer10 ml de solution / ml d'héparine 50 ug dans du PBS.
- Préparer xylazine / kétamine anesthésique en mélangeant 10 ml de kétamine (100 mg / ml) et 1 ml de xylazine (20 mg / ml).
- Diluer xylazine / kétamine stock 1: 4 à 0,1 M PBS.
- Préparer une solution de 100 ml de fixatif contenant 4% de paraformaldehyde et 0,1% de glutaraldéhyde dans du PBS.
- Anesthésier une souris PyyGFP, âgés de 6 à 10 semaines, avec une dose létale de xylazine / kétamine anesthésique. Injecter l'anesthésique par voie intraperitoneale à 0,15 ml par 20 g de poids de la souris.
- Confirmez anesthésie appropriée de la souris en pinçant la queue ou des orteils, et d'utiliser la pommade sur les yeux pour prévenir la sécheresse.
- Utilisez un flux pompe péristaltique variable pour perfuser fixateur intracardiaque 10. Avec des ciseaux chirurgicaux (13 cm de longueur) coupe ouvrir la cavité abdominale pour exposer les intestins, le cœur et les poumons. Tenez coeur avec une pince étroite motif incurvé (12 cm de longueur) et insérer une aiguille à ailettes (19 G) de la pompe péristaltique iNto ventricule gauche. Immédiatement, couper l'oreillette droite avec des ciseaux droites de printemps (longueur 4 mm). Perfuser à un débit de 2 ml / min d'abord avec une solution d'héparine pendant 1 min, puis avec une solution de fixateur glacée pendant 15 minutes jusqu'à ce que la queue de la souris est tout à fait rigide.
Remarque: Il est très important pour perfuser à un rythme lent à empêcher l'éclatement des petits vaisseaux de la muqueuse intestinale. Débordant de navires va compromettre l'ultrastructure du tissu. Si la perfusion est adéquate, le foie doit pâlir de couleur dans 3-5 min. - Utilisation de petits ciseaux ouvrir la cavité abdominale et de l'accise de la totalité du côlon de la jonction avec le caecum au rectum distale. Placez le tissu dans du PBS glacé. Bien immergé dans PBS, couper ouverte avec de petits ciseaux de printemps du côlon long du mésentère.
2. Dissection et de fixation segments de tissu
- En utilisant un scalpel, couper environ six segments de tissu de petite taille (2 mm 2) dans le côlon distal. Pour ce faire, sur une feuille ocire dentaire f et immergé dans quelques gouttes de PBS.
- Post-fixer les segments de tissu dans la solution de fixation pendant 3 heures à 4 ° C.
3. blocs de tissus micro-dissection
- Préparer 5% bas point de fusion d'agarose dans du PBS et le garder dans un bain d'eau à 45 ° C.
- Incluez les segments de tissu dans 5% à faible point de fusion agarose en utilisant un petit récipient en plastique, comme la taille standard Tissue-Tek cryomoule.
- Montez les sections embarqués sur un microtome à lame vibrante et remplir le bac tampon avec PBS glacé.
- Couper 300 um bandes de tissu à 0,8 amplitude et 0,04 mm de vitesse / sec. Remarque: À ce stade, les bandes de tissus seront délogés de l'agarose.
- Re-intégrer 300 bandes de tissu de um dans l'agarose et les monter sur microtome perpendiculairement à la lame de l'vibratome.
- L'utilisation d'un vibratome, couper des bandes de tissu à une épaisseur de 50 um. Remarque: Les blocs de tissus définitifs devraient être d'environ 300 m de large x 50 um d'épaisseur. Ces dimensions sont optimisées à partir d'expériences pilote confocaux qui ont montré que l'épaisseur de la cellule entéroendocrine est comprise entre 15 à 20 um et une longueur de 30 à 70 nm de l'neuropod. Par conséquent, une cellule entière puisse être contenue dans un bloc de tissu de 300 um de largeur et 50 um d'épaisseur si l'orientation de la cellule est parallèle à la face du bloc de tissu. Ces dimensions varient selon le tissu et la cellule type d'intérêt.
- Stockez les blocs de tissus dans du PBS à 4 ° C.
4. imagerie confocale
- Préparer une solution de colorant nucléaire en diluant DAPI dans du PBS à 1: 4000.
- Incuber blocs de tissus dans DAPI colorant nucléaire pendant 5 min. Remarque: coloration DAPI facilite la distinction des cellules individuelles par leurs noyaux, ce qui est utile pour mettre en corrélation les images confocales et SBEM.
- Blocs de tissus de montage sur lame de verre chargée, ajouter quelques gouttes de PBS, et les couvrir avec une lamelle.
- En utilisant un microscope confocal, identifier blocs de villosités intact et cellules PyyGFP d'intérêt et de les imager pour obtenir z-piles sections optiques.
- Utilisez un objectif 20X / 0,8 plan Zeiss Apochromat pour obtenir z-piles de 1 um sections optiques.
- Pour le z-stack, utiliser les canaux de 405 nm (DAPI) pour déterminer la relation à d'autres cellules, 488 nm GFP endogène de localiser la cellule d'intérêt, et contraste d'interférence différentiel (DIC) pour déterminer l'emplacement de la cellule par rapport à la lumen.
- Utilisez une résolution d'image de 1024 pixels ou plus.
5. incorporation blocs dans Agarose
- Préparer 10 ml de fixatif contenant 4% de paraformaldehyde et 2,5% de glutaraldéhyde dans du PBS.
- Retirez les blocs de tissus provenant de la lame de verre, en ajoutant prudemment PBS dans les bords de la lamelle à glisser la lamelle loin de la lame de verre sans endommager le bloc de tissu. Ensuite, utilisez un pinceau de beaux-arts de transférer le bloc de tissu de la diapositive à un 10,5 ml de tube à centrifuger contenant du paraformaldéhyde 4% et 2,5% de glutaraldéhyde fixateur.
- Blocs de tissus post-fix O / N à 4 ° C.
- Transfert blocs de tissus à PBS. Ensuite, incorporer blocs plats dans 5% d'agarose bas point de fusion en les prenant en sandwich entre deux lames de verre.
Note: Intégrer les blocs dans une mince couche d'agarose facilite la manipulation postérieure pendant la coloration. - Couper le agarose dans un carré et de faire une encoche sur le côté supérieur de la main. Remarque: Cette étape est nécessaire pour maintenir l'orientation dans les étapes suivantes.
- blocs de magasins dans un tube de 1,5 ml avec du PBS à 4 ° C jusqu'à leur traitement ultérieur.
Serial Numérisation Bloc-face Electron Microscopy (SBEM)
Dans cette section, le bloc de tissu est préparé et imagée avec SBEM à faible grossissement. L'image de l'enquête de la face du bloc est ensuite corrélée avec les données confocale pour identifier la région contenant la cellule d'intérêt. Une fois la régionest identifié, le tissu est imagée avec une résolution de 7 nm / pixel et des tranches de 70 nm. Ce fut suffisant pour résoudre et de distinguer les grands-dense vésicules de sécrétion de base à partir d'autres organites. Dans les cellules entéro-endocrines, ce type de vésicules varie entre 100 et 150 nm de diamètre 13. Le protocole est le suivant:
6. Les articles de tissus coloration
- Retirer le tissu de PBS et rincer trois fois, cinq minutes à chaque fois, dans du tampon 0,1 M de cacodylate.
- blocs de taches pour l'acide tannique 1 h en 0,1% dissous dans un tampon cacodylate 0,1 M pour augmenter le contraste des membranes cellulaires 14.
- Procéder à la coloration et la déshydratation subséquente de tissu selon le protocole publié 11. Suite à Deerinck et al. protocole, de préparer ces solutions: 1) 1% (p / v) thiocarbonhydrazide (THC); 2) conduire solution aspartate; 3) l'acétate d'uranyle à 1%, et 4) le ferrocyanure tetraoxyde d'osmium / de potassium (OsO 4 / K ferrocyanure). Mélanger 4% OsO 4 et 2xK Ferrocyanide stocks [0,3 g K Ferrocyanide et 0,86 g Na cacodylate dans 10 ml H 2 O] au rapport 1: 1 de faire OsO 4 / K Ferrocyanide. Remarque: Avoir le tissu incorporé dans agarose facilite la manipulation au cours de la coloration. Retirez délicatement agarose pour l'infiltration subséquente de résine.
- Rincer les échantillons trois fois, 5 min chacune, dans un tampon cacodylate 0,1 M puis tache avec une solution OsO4 / K Ferrocyanide pendant 2 h à 4 ° C.
- Rincer à l'eau ultra pure trois fois, 5 minutes chacun, et la tache avec TCH pendant 30 min à 60 ° C.
- Rincer à l'eau ultra pure trois fois, 5 minutes chacun et tache avec 2% OsO 4 (pas K Ferrocyanide) pendant 60 min à température ambiante. Rincer à nouveau en eau ultra pure trois fois, 5 minutes chacun.
- Colorer avec 1% d'acétate d'uranyle O / N à 4 ° C. Rincer à l'eau ultra pure trois fois, 5 minutes chacun.
- Colorer avec aspartate plomb pendant 30 min à 60 ° C. Rincer à l'eau ultra pure trois fois, 5 minutes chacun.
- Déshydrater les tissus par rinçage dans solutions avec des concentrations croissantes d'éthanol. Rincer deux fois, cinq minutes à chaque fois, avec 50%, 75%, 85%, 95% et 100% d'éthanol absolu. A la fin, rinçage sections avec l'oxyde de propylene, deux fois 10 min chacun.
7. infiltrant and Embedding coupes de tissus dans Résine
- Infiltrer les blocs avec de la résine en utilisant le kit commercialement avaialble. Intégrer les tissus en résine en utilisant un kit d'encastrement epon. Préparer un mélange de résine époxy de 48%, 20% DDSA ml, 30 ml NMA%, et 2% DMP30. Agiter vigoureusement le mélange de résine pendant 5 minutes et ensuite placer mélange sous vide pendant 30 min pour permettre à des bulles montent à la surface.
- Mélanger la résine avec de l'oxyde de propylène au rapport 1: 1 et agiter vigoureusement. Retirer le rinçage de la déshydratation finale des tissus, et ajouter résine mélangée avec de l'oxyde de propylène. Lieu flacon avec des échantillons dans un rotateur O / N et laisser flacon non bouché. Le lendemain, ajouter résine EPON fraîchement préparé et mélanger pendant 90 min dans la coiffe.
- Intégrer blocs en résine aussi plat que possible en les prenant en sandwich dansentre des lames de verre qui ont été durcis avec un agent de libération de liquide pour les empêcher de coller les uns aux 15 autres.
- Une fois que les blocs sont intégrés plat, guérir les blocs pour une addition de 48 h à 60 ° C.
- Tirez diapositives à part pour libérer les blocs.
8. Montage et Recadrage de l'bloc de tissu pour l'imagerie
- Sous un microscope à dissection, correspondre à l'orientation des blocs de tissus inclus en résine avec celle des micrographies confocales pour faciliter l'identification des régions contenant les cellules d'intérêt avant de couper le bloc.
- Coupez le bloc de résine embarqué bas manuellement à un um îlot ~ 500 x 500.
- Monter le bloc à plat sur une broche contenant époxy et sec conducteur pendant 30 min. Ensuite, mettre le bloc à plat sur une surface sèche et O / N à 60 ° C.
- Enduire le bloc avec le liquide de l'argent colloïdal. Maintenir les sections de tissus plat afin de garantir le tranchage du bloc à angle droit pour faciliter la coopérationrrelating les micrographies série d'îlot et confocale.
9. SBEM
- L'image du bloc en utilisant un microscope électronique à balayage équipé d'un système de SBEM (e .g., 3view).
- Réglez l'épaisseur de coupe initiale à ~ 2 um incréments et coupé jusqu'à ce que le visage de tissu émerge du bloc.
- Acquérir une image de Surver de l'ensemble du visage de bloc.
- En utilisant le logiciel Fidji, prendre des mesures à la fois le bloc-face et confocale série micrographies pour générer un dénominateur et compte commun pour l'échantillon le déformer lors de la fixation et la coloration 16.
- Localisez la zone d'intérêt sur la face du bloc en multipliant le dénominateur pour les coordonnées dans les images confocales. Utilisez également d'autres caractéristiques structurelles, comme la position de microvillosités, cellules caliciformes ou lamina propria, comme une référence.
- région de l'image d'intérêts à un 2,25 kV et 7 nm / pixel (ou 15,147X grossissement) en mode à vide élevé. incréments de Slice devraient être fixésà 70 nm ou moins.
- Recueillir des données en format RAW SBEM .dm3 16-bit.
10. Optimisation SBEM Images pour la segmentation de surface
- Convertir des images de format SBEM .dm3 brut à 8 bits .tiff.
- Filtrez .tiff images en utilisant un filtre de 0,8 flou gaussien à Fidji.
- Réduire l'ensemble à 25% de la taille originale des données et l'enregistrer comme une pile .tiff pour minimiser la quantité de mémoire RAM nécessaire pour gérer l'ensemble.
- Alignez la pile d'images SBEM utilisant le plugin Fidji "d'alignement de la pile linéaire avec des EIPD», au mode de traduction et recadrée en utilisant le plugin "culture 3D". Note: Le rognage permet de réduire davantage la quantité de mémoire RAM nécessaire pour la segmentation et le rendu de volume.
Rendu des données en 3D
11. microscopie confocale
- Reconstruire z-piles en utilisant le mode automatique de l'outil "surfaces" surpassent. Figure 1D, montre une reconstruction de chaque canal en utilisant l'option lisse, le détail de la zone des surfaces à 0,126 um, et d'un seuil que l'intensité absolue.
12. série Bloc-face SEM
Remarque: Les données Manuel segmentation est une procédure très coûteuse en temps et en fonction des caractéristiques d'être rendus à la procédure peut prendre plusieurs semaines. Segmentation pour les vidéos et les chiffres présentés dans Bohórquez et al 2014 8. A été fait manuellement et a pris environ 500 heures de travail. Il est recommandé de donner la priorité aux caractéristiques essentielles nécessitant rendu avant de commencer le processus de segmentation. La procédure est la suivante:
- Utilisez le "surfaces" outil en mode de traction et l'option "contour" au mode de 50 ms dessin de segmenter manuellement et rendu de volume de la cellule d'intérêt.
- Tracer les contours sur chaque tranche de la cellule pour un rendu plus lisse ou toutes les 5 tranches pour un rendu plus rapide.
- Export im finaleâges en utilisant l'outil «instantané» à une résolution de 300 dpi ou plus.
Résultats
La méthode présentée ici a été utilisé pour étudier l'ultrastructure d'une cellule spécifique dans une couche de l'épithélium. La cellule d'intérêt dans ce cas est la cellule entéroendocrine insaisissable. Leur identification in situ n'a été possible que dans les dernières années avec le développement de souris fluorescence rapporteurs spécifiques des cellules. En 2011, nous avons développé une souris dans laquelle le promoteur de l'hormone de PYY entraîne l'expression de la protéine fluorescente verte 17. Dans ce PyyGFP souris, les cellules entéro de la partie distale de l'intestin grêle et du côlon sont facilement distingués sous lumière UV. Ce modèle de souris est une base pour identifier un bloc de tissu contenant une cellule entéroendocrine et le traiter pour SBEM. La méthode de corrélation est désigné ici comme cocem3D et a été construit sur les protocoles précédemment publiés 10,11.
L'optimisation des dimensions du bloc de tissu est critique pour la corrélation. Dans des expériences préliminaires,il a été déterminé que, dans un bloc de tissu de 300 um de long x 50 um d'épaisseur le nombre d'images SBEM est réduit à un minimum tout en couvrant l'ensemble du corps de la cellule d'intérêt. La figure 1A montre un Overlook de la dissection en utilisant une lame microtrome vibrant obtenir le bloc de tissu avec les bonnes dimensions. Au moins 100 blocs sont obtenus avant le tri à travers eux à choisir ceux avec des cellules intactes d'intérêt. Blocs sélectionnés sont imagées par microscopie confocale et d'image z empilements sont espacés optiquement chaque 1 pm (Figure 1B et C). La figure 1D montre un volume vue rendue d'une cellule entéroendocrine dans l'iléon de la souris. Son neuropod important se prolonge sous les cellules épithéliales de ~ 60 um.
La cellule d'intérêt se trouve en corrélant les z-piles confocale avec une image de SBEM de l'ensemble du visage de bloc. Un exemple est présenté ici d'un bloc de la partie distale du côlon d'un PyyGFPsouris. Après l'obtention confocale z-piles (figure 2A), le bloc est noyé dans une mince couche de bas point de fusion agarose (figure 2B). Cela est nécessaire pour la manipulation ultérieure du bloc. Il est important de maintenir l'orientation du bloc le plus plat possible de faire correspondre les tranches optiques avec les tranches de SBEM. Sur la figure 2C, une image représentative est affichée de la totalité de la face du bloc de tissu. Certaines parties de ce chiffre ont déjà été publiés dans Bohorquez et al., 2014 8. Ce fut ensuite corrélée avec l'image confocale en tenant compte des points de repère et des dimensions de tissus. Figure 2D montre une superposition du confocale et l'image SBEM du bloc, révélant la emplacement précis de notre cellule d'intérêt.
Une fois que la cellule a été identifié, l'imagerie par SBEM est fait à une résolution suffisamment élevée pour identifier les vésicules de sécrétion et d'autres organites cellulaires. L'ensemble de données, il a présentére a été imagée à 7 nm par pixel que les images du bloc ont été prises tous les 70 nm. L'ensemble des données contenues 643 images qui couvrent 45 um de la profondeur des tissus. A quelques microns sont rasés lors de l'imagerie initiale de l'ensemble du visage de bloc à faible grossissement. L'ensemble de données de SBEM brut a été acquis en format .dm3 et transformé en .tiff pour manipulation ultérieure. La pile de SBEM a été recadrée en utilisant le logiciel Fidji plugin "cultures (3D)" bloc pour contenir seulement la région d'intérêt. Cela réduit la quantité de mémoire RAM nécessaire pour le rendu de volume. La cellule a été identifié par sa position et les vésicules sécrétoires, et segmenté en utilisant le logiciel Imaris. La figure 3 contient un rendu en 3D de la cellule entéroendocrine.
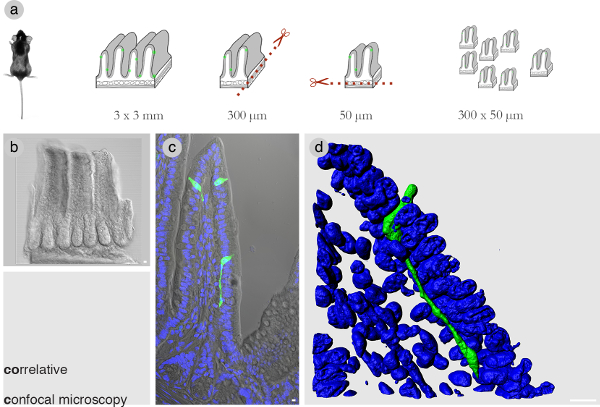
Figure 1:. Microscopie confocale corrélative (A) Workflow pour disséquer un morceau detissu intestinal dans un tissu bloquer 300 x 50 um d'épaisseur. (B) Un bloc de tissu représentant des bonnes dimensions. Notez qu'un bloc de tissu de ces dimensions de l'intestin grêle de souris étend sur trois villosités ou, dans le cas du côlon environ 8 cryptes. (C) Un prélèvement de villosités de l'intestin grêle qui contient une cellule d'intérêt. (D) Une pile confocale z-rendu en 3D en utilisant le logiciel Imaris représente une cellule entéroendocrine d'une course en avant neuropod sous l'épithélium intestinal. Bleu = DAPI colorant nucléaire. Bars = 10 um. S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
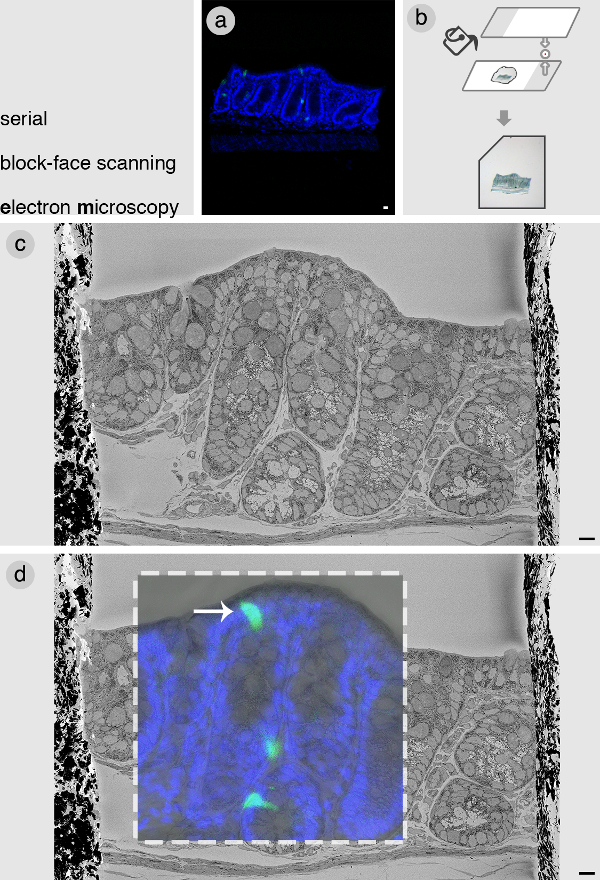
Figure 2: la microscopie électronique à balayage série d'îlot (A) Image confocale d'un sele.bloc de tissu DECT du côlon contenant les cellules de PyyGFP (vert) d'intérêt. (B) Une fois que le bloc est imagé au microscope confocal, il est ensuite incorporé à plat en bas point de fusion à l'aide d'agarose des lames de verre. Intégration de la bloc de tissu dans l'agarose facilite la manipulation ultérieure, et l'encoche dans le coin gauche permet de monter le bloc dans le SBEM dans la bonne orientation. (C) l'image SBEM de la face du bloc. (D) la corrélation des données confocale avec SBEM d'identifier cellule d'intérêt (flèche blanche). Images dans c et d de cette figure sont modifiés à partir Bohorquez et al., 2014 8. Bleu = DAPI colorant nucléaire. Bars = 10 um. S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
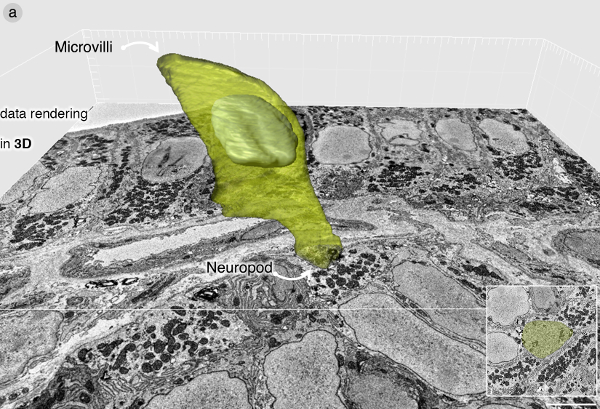
Figure 3:. Les données de rendu en 3D (A) Rendu de volume des cellules entéroendocrine utilisant un logiciel Imaris. La cellule contient un neuropod emballé avec des vésicules sécrétoires. Pour une description complète de l'ultrastructure d'une cellule entéroendocrine, s'il vous plaît se référer à Bohórquez et al., 2014 8. S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
Discussion
Chemosensation gastro-intestinal est en train de devenir un nouveau champ passionnant de la recherche biomédicale. Ceci est dû en grande partie à la découverte de récepteurs du goût fonctionnels dans les cellules entéro 18. Des études ultérieures ont montré que des cellules entéro-endocrines expriment des récepteurs spécifiques pour les substances nutritives, y compris des hydrates de carbone, des lipides, des acides aminés et des 5,6,19,20. Le facteur catalytique pour ces découvertes a été le développement de souris rapporteurs, dans lequel des cellules entéro-endocrines sont 20 marquées par fluorescence. Nous avons développé une de ces souris, la souris PyyGFP, pour étudier des cellules entéro-endocrines de l'intestin grêle et le côlon distal 17,21. Ces cellules sont d'intérêt parce qu'ils sécrètent PYY et du glucagon-like peptide 1, qui sont tous deux des inducteurs de satiété 22,23. À l'époque, un compte rendu complet ultra-structurelle de ces cellules était manquant, ce qui nous croyions était essentiel de comprendre leurs mécanismes de signalisation.
ontenu "> Ici, nous avons décrit une méthode visuelle pour combler microscopie confocale avec SBEM. La méthode est appelée en tant Cocem3D, qui nous a permis de documenter l'ultrastructure complète des cellules entéro. Nous avons signalé que ces cellules ont une neuropod qui contient trois quarts de tous les vésicules de sécrétion 8. Dans le neuropod, il ya neurofilaments et un peu comme axones neuronaux, neuropods sont nourries par des cellules gliales entériques 8. Plus important encore, il est bien que ces neuropods que entéroendocrine cellules connecter physiquement à des neurones innervant l'intestin et 9 côlon.Un des points forts de cocem3D est sa simplicité. La réduction de l'échantillon de tissu dans un bloc qui peut être imagé par SBEM confocale et facilite l'identification d'une cellule spécifique d'un tissu. Vésicules de sécrétion et d'autres organites peuvent être identifiés avec une facilité à une résolution de 7 nm / pixel. Parce que les sections en SBEM sont éliminés, d'autres procèschanter des tissus pour identifier les protéines spécifiques est pas une option à ce stade. Cependant, la mise au point de procédés tels que ATUM 24, dans lequel les sections du bloc de tissu sont conservés, sont susceptibles de permettre l'identification de protéines spécifiques dans la cellule. Détermination de l'emplacement spécifique des récepteurs sur les cellules chimiosensoriels entéroendocrines information est essentielle pour le développement de thérapies médicamenteuses pour l'obésité, car les cellules entéro sont une interface sensorielle entre les aliments dans l'intestin et de la satiété dans le cerveau.
Déclarations de divulgation
Dr Satish Medicetty est un employé de et reçoit 100% du salaire de Renovo Neural Inc; Toutefois, cela ne modifie pas l'adhésion de l'auteur de politiques JoVE sur les données et matériaux partage. Pas de conflit d'intérêts est déclaré pour les auteurs restants.
Remerciements
Our sincere appreciation is expressed to the following people: Drs. Sam Johnson and Benjamin Carlson of the Duke Light Microscopy Core Facility for their assistance with data visualization software, and Ms. Valerie Lapham and Dr. John M. Mackenzie, Jr. of the Center for Electron Microscopy at North Carolina State University for their advice on electron microscopy. We thank Dr. Elaine B. Bohórquez for her editorial assistance. Authors contributed in the following manner: DVB, SM, and RAL designed experiments and analyzed data. DVB performed experiments and FH performed manual rendering of data. SM is director of Renovo Neural, where SBEM data was acquired. DVB wrote the manuscript and all authors reviewed and edited the final manuscript. This work was supported by NIH grants R01DK091946 and Veterans Affairs grant I01BX002230 to RAL, and F32DK094704, to DVB.
matériels
| Name | Company | Catalog Number | Comments |
| Phosphate buffered saline | Life technologies | 10010023 | |
| Heparin sodium salt | Sigma | H4784 | |
| Paraformaldehyde | Sigma | 158127 | 4%, freshly made in PBS, final pH 7.4 |
| Glutaraldehyde | Sigma | G5882 | |
| Dental wax | Electron Microscopy Sciences | 72660 | |
| Low-melting agarose | Life technologies | 16520-100 | 5%, freshly made in PBS |
| Standard Tissue-Tek Cryomold | Electron Microscopy Sciences | 62534-25 | |
| DAPI nuclear stain | Life technologies | D1306 | |
| Postively charged glass slides and coverslips | |||
| Fine art paintbrush #1 | |||
| Cacodylate buffer | Electron Microscopy Sciences | 11650 | |
| Tannic acid | Electron Microscopy Sciences | 21700 | |
| EMbed 812 kit | Electron Microscopy Sciences | 14120 | |
| Liquid releasing agent | Electron Microscopy Sciences | 70880 | |
| Liquid silver colloidal | Electron Microscopy Sciences | 12630 | |
| CircuitWorks conductive epoxy | ITW Chemtronics | CW2400 | |
| Variable flow peristaltic pump | VWR | 70730-064 | |
| VT1200S Vibrating blade microtome | Leica | ||
| Zeiss 780i confocal microscope | Carl Zeiss | ||
| Sigma VP Scanning Electron Microscope | Carl Zeiss | ||
| 3view system | Gatan | ||
| Renovo Neural Inc (Cleveland, OH) | http://www.renovoneural.com | Renovo provides 3d EM services | |
| Fiji software | Open access software | ||
| Computer station with 16 GB of RAM or more | |||
| Data visualization software Imaris 7.5 | Bitplane |
Références
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological. 314, 1-340 (1986).
- Smith, C. L., et al. Novel cell types, neurosecretory cells, and body plan of the early-diverging metazoan Trichoplax adhaerens. Current biology : CB. 24, 1565-1572 (2014).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS biology. 2, e329 (2004).
- Engelstoft, M. S., et al. Seven transmembrane G protein-coupled receptor repertoire of gastric ghrelin cells. Molecular metabolism. 2, 376-392 (2013).
- Chandra, R., et al. Immunoglobulin-like domain containing receptor 1 mediates fat-stimulated cholecystokinin secretion. The Journal of clinical investigation. 123, 3343-3352 (2013).
- Wang, Y., et al. Amino acids stimulate cholecystokinin release through the Ca2+-sensing receptor. Am J Physiol Gastrointest Liver Physiol. 300, G528-G537 (2011).
- Rogers, G. J., et al. Electrical activity-triggered glucagon-like peptide-1 secretion from primary murine L-cells. The Journal of physiology. 589, 1081-1093 (2011).
- Bohórquez, D. V., et al. An enteroendocrine cell-enteric glia connection revealed by 3D electron microscopy. PloS one. 9, e89881 (2014).
- Bohórquez, D. V., et al. Neuroepithelial circuit formed by innervation of sensory enteroendocrine cells. The Journal of clinical investigation. , (2015).
- Knott, G. W., Holtmaat, A., Trachtenberg, J. T., Svoboda, K., Welker, E. A protocol for preparing GFP-labeled neurons previously imaged in vivo and in slice preparations for light and electron microscopic analysis. Nature protocols. 4, 1145-1156 (2009).
- Deerinck, T., Bushong, E., Thor, A., Ellisman, M. . NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. , (2010).
- Bohórquez, D. V., Chandra, R., Samsa, L. A., Vigna, S. R., Liddle, R. A. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. Journal of molecular histology. 42, 3-13 (2011).
- Nilsson, O., et al. Distribution and immunocytochemical colocalization of peptide YY and enteroglucagon in endocrine cells of the rabbit colon. Endocrinology. 129, 139-148 (1991).
- Mizuhira, V., Futaesaku, Y. New fixation method for biological membranes using tannic acids. Acta Histochem Cytochem. 5, 233-236 (1972).
- Reymond, O. L., Pickett-Heaps, J. D. A routine flat embedding method for electron microscopy of microorganisms allowing selection and precisely orientated sectioning of single cells by light microscopy. Journal of microscopy. 130, 79-84 (1983).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature. 9, 676-682 (2012).
- Bohórquez, D., Chandra, R., Samsa, L., Vigna, S., Liddle, R. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. J Mol Histol. 42, 3-13 (2011).
- Jang, H. J., et al. Gut-expressed gustducin and taste receptors regulate secretion of glucagon-like peptide-1. Proceedings of the National Academy of Sciences of the United States of America. 104, 15069-15074 (2007).
- Liou, A. P., et al. The G-protein-coupled receptor GPR40 directly mediates long-chain fatty acid-induced secretion of cholecystokinin. Gastroenterology. 140, 903-912 (2011).
- Reimann, F., et al. Glucose sensing in L cells: a primary cell study. Cell metabolism. 8, 532-539 (2008).
- Bohorquez, D. V., Liddle, R. A. Axon-like basal processes in enteroendocrine cells: characteristics and potential targets. Clinical and translational science. 4, 387-391 (2011).
- Batterham, R. L., et al. Gut hormone PYY(3-36) physiologically inhibits food intake. Nature. 418, 650-654 (2002).
- Flint, A., Raben, A., Astrup, A., Holst, J. J. Glucagon-like peptide 1 promotes satiety and suppresses energy intake in humans. The Journal of clinical investigation. 101, 515-520 (1998).
- Hayworth, K. J., et al. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Frontiers in neural circuits. 8, 68 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon