Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Planar Gradient système de diffusion d'enquêter Chimiotactisme dans une matrice de collagène 3D
Dans cet article
Résumé
Cell migration is an important part of human development and life. In order to understand the mechanisms that can alter cell migration, we present a planar gradient diffusion system to investigate chemotaxis in a 3D collagen matrix, which allows one to overcome modern diffusion chamber limitations of existing assays.
Résumé
The importance of cell migration can be seen through the development of human life. When cells migrate, they generate forces and transfer these forces to their surrounding area, leading to cell movement and migration. In order to understand the mechanisms that can alter and/or affect cell migration, one can study these forces. In theory, understanding the fundamental mechanisms and forces underlying cell migration holds the promise of effective approaches for treating diseases and promoting cellular transplantation. Unfortunately, modern chemotaxis chambers that have been developed are usually restricted to two dimensions (2D) and have complex diffusion gradients that make the experiment difficult to interpret. To this end, we have developed, and describe in this paper, a direct-viewing chamber for chemotaxis studies, which allows one to overcome modern chemotaxis chamber obstacles able to measure cell forces and specific concentration within the chamber in a 3D environment to study cell 3D migration. More compelling, this approach allows one to successfully model diffusion through 3D collagen matrices and calculate the coefficient of diffusion of a chemoattractant through multiple different concentrations of collagen, while keeping the system simple and user friendly for traction force microscopy (TFM) and digital volume correlation (DVC) analysis.
Introduction
Le mouvement préféré de cellules vers un gradient de concentration, connu comme le chimiotactisme, joue un rôle important dans les processus physiologiques et pathologiques dans le corps. De tels exemples sont la peau et les muqueuses une cicatrisation des plaies, la morphogenèse 2, 3 inflammation, et 4,5 de la croissance tumorale. Il a également été montré que les cellules cancéreuses peuvent migrer à travers les deux stratégies de migration de cellule 6 individuels et collectifs. En outre, des mécanismes d'instabilité diffusionnels peuvent induire la séparation des cellules individuelles ou groupées d'un corps / objet tumorale et peuvent ensuite immigrer vers une source de nutriments et donc envahir des zones plus vastes et les tissus 7.
En outre, il a été montré que divers mécanismes de migration peuvent être actifs en 2D et en 3D, en raison de différents rôles des molécules d'adhésion 8. Par conséquent, un passage à physiologiquement pertinente des essais in vitro pour étudier la motilité cellulaire dans un measureable et de façon simple est d'importance dans la compréhension de mouvement cellulaire phénomènes 9. Malheureusement, la difficulté à analyser la migration cellulaire, un test complet de chimiotactisme quantifiable nécessite habituellement une longue méthode laborieuse, fondée sur la mesure des modèles de la motilité cellulaire et les phénomènes de transport impartiaux.
Approches expérimentales passées pour enquêter sur la chimiotaxie de cellules comprennent la chambre de Boyden 10 et le titre d'essai agarose 11. Cependant, dans ces premiers essais, des expériences de migration cellulaire ne surveillaient pas le mouvement dans le temps. De plus, surtout, les gradients de concentration utilisées pour les expériences ne sont pas bien définies ou complètement compris, tandis que seulement le maintien de la signalisation de pas plus de quelques heures. En outre, chimiotaxie début tentatives de chambre restreinte migration cellulaire à deux dimensions et ne permettent de surveiller la cinétique de la migration 12. En regardant la chambre de Boyden, un test de point de terminaisonne permettrait pas au chercheur d'observer visuellement la migration et ne pouvait pas différencier directement chimiotaxie (mouvement directionnel) de chimiokinèse (mouvement aléatoire). En outre, plusieurs des variables-différences dans la taille des pores et l'épaisseur des membranes réalisés à la chambre très difficile à reproduire facilement dissimulés et la réaction des cellules migrant vers les chimiokines 13,14.
Avec la nouvelle compréhension de la microfluidique, de nouvelles chambres et micro-dispositifs ont été étudiés comme un instrument pour étudier la locomotion cellulaire dans des conditions d'écoulement interstitiels ou chimiotactisme 15,16. En vertu de ces nouveaux appareils, de nouvelles mesures de cellules ont été introduits et étudiés, comme l'effet de la contrainte de cisaillement sur une cellule 17,18. Malheureusement, les chambres de chimiotactisme microfluidiques précédents et actuels études de la migration des cellules limitées à des substrats 2D-un recul important puisque de nombreux processus biologiques, y compris l'invasion et la métastase de cellules tumorales, et imla migration cellulaire mune, implique la migration 3D.
Chambres, où d'observation directe d'une solution de chimioattractant est en contact avec un gel 3D contenant des cellules ont également été également rapporté 19,20. Ces chambres ont deux compartiments, l'un contenant un chimioattractive et l'un contenant des cellules, sont reliés côté de l'autre horizontalement 21 ou 22 anneaux concentriques. Ces systèmes sont pointés dans la bonne direction, mais ne gardent pas un système de chimiotactisme pour une période de temps prolongée.
En outre, les chercheurs ont également examiné la diffusivité à travers des membranes de collagène dans des cellules de dialyse, ainsi que la diffusion de molécules de traceur à travers des échantillons de collagène est soumis à une pression hydrostatique de 23 à 25. Des expériences de diffusion dans des gels de collagène reposent sur des modifications physiques et chimiques du gel à l'aide de champs magnétiques et incorporation chimique 26. Une méthode populaire pour la modélisation de diffusion dans collatissus Genous repose sur l'imagerie de fluorescence du point photoblanchiment continue. Cette méthode a révélé une anisotropie dans les coefficients de diffusion des macromolécules dans les tissus de collagène orientées. Cependant, photoblanchiment a été utilisé dans le cartilage articulaire et les matrices de collagène non. Bien que semblable, les expériences de modélisation nécessaires doivent être effectuées par la compréhension spécialement le coefficient de diffusion de gels de collagène. Plus important encore, les systèmes ne pas utiliser un procédé de mesure de génération de force de cellule.
Malheureusement, la plupart des systèmes semblent manquer un ou deux éléments clés pour un système idéal: l'permettant de suivi de la cellule, une compréhension de gradient de diffusion avec un facteur chimiotactique à travers la matrice, un ensemble relativement simple avec une facilité de reproductibilité, la minimisation de interactions cellule-cellule, et la capacité de mesurer les unités de mesure pour la quantification (c., la vitesse, la force, la concentration spécifique). Moghe et al. 27 proposé un système qui accomplit la plupart de ces exigences, dans lequel les cellules sont initialement dispersées dans le gel plutôt que concentrés sur la surface du filtre, mais il était difficile de mesurer les forces qui génère la cellule.
Pour cette fin, nous présentons un système de diffusion de gradient plane pour enquêter sur la chimiotaxie dans une matrice de collagène en 3D, qui permet de surmonter modernes limitations de la chambre de diffusion de tests existants, qui est basé sur la microscopie time-lapse, couplées avec des techniques d'analyse d'images pour mesurer cellulaire forces dans un environnement 3D. Ce protocole fournit un moyen simple, mais innovant de création d'une chambre de diffusion 3D simple qui peut être utilisé pour étudier la chimiotaxie 3D dans des cellules différentes.
Protocole
1. 3D Mold Design et pièces de rechange
- Moule
- Avant de travailler, obtenir une trousse d'élastomère de silicone, une chambre d'imagerie des cellules vivantes, une lamelle de verre de 22 mm, et un cube de métal aluminium usiné avec des dimensions de 10,07 mm x 3,95 mm x 5,99 mm. Préparer la chambre d'imagerie des cellules vivantes pour le moulage en plaçant la lamelle couvre-objet dans le support inférieur et l'assemblage du reste de la chambre comme indiqué par le fournisseur.
- Ensuite, en utilisant une pince, placez le cube en aluminium usiné de métal dans le milieu de la imagerie cellulaire logements en direct de la chambre et sur le dessus de la lamelle, puis mis de côté.
- Mélanger les solutions d'élastomère de silicone selon le protocole du fabricant à faire 5 ml d'élastomère.
- Utiliser un laboratoire spatule jetable, verser la solution d'élastomère de silicone dans la configuration en direct de la chambre de l'imagerie cellulaire et veiller à ne pas bouger placé usiné cube de métal d'aluminium. Placez le système sur le banc de laboratoire, dans un endroit sécuritaire O / N pour le durcissement.
- Le lendemain matin, déconstruirela chambre de l'imagerie de cellules vivantes, tel que recommandé par le fabricant et tirez moule en utilisant une pince. En utilisant des pinces, retirez délicatement le cube usinés en aluminium métallique de moule. Aller à l'évier et rincer le moule avec de l'eau déminéralisée. Placer le moule sur une serviette en papier pour sécher.
- Une fois sec, à l'aide d'un couteau de passe-temps, des fentes pratiquées à travers le moule, espacés de 2,34 mm de distance de chaque extrémité longitudinale, à l'intérieur du moule en silicone. Assurer les séjours de moisissures dans un endroit sûr et sec jusqu'à ce que vous êtes prêt à construire le système pour une expérience.
- Hydrophiles et hydrophobes Lamelles verre Préparation
- Pour permettre à la matrice de collagène à adhérer à une surface, de créer des lamelles hydrophiles:
- En utilisant une pipette jetable, mesurer 150 ul de 3-aminopropyl-triméthoxysilane et verser la solution dans un tube de 50 ml. Ajouter 30 ml d'éthanol à 100% sur le tube de 50 ml avec un deuxième pipette jetable et fermer le couvercle. Vortexer la solution pendant 2 minutes, assurant un mélange complet. Pnotre solution dans une boîte de Pétri en verre et mis de côté.
- Verser 15 ml d'alcool éthylique de 100% dans une seconde boîte de Pétri et mettre de côté (assurez-vous d'étiqueter plats).
- En utilisant une nouvelle pipette jetable, mesurez 30 ml d'eau déminéralisée et verser la solution dans un tube de 50 ml. Ensuite, en utilisant une nouvelle mesure de pipette jetable sur 1875 pi de glutaraldéhyde et verser la solution dans le même tube de 50 ml et fermer le couvercle. Vortexer la solution pendant 2 minutes, assurant un mélange complet. Verser le mélange dans la troisième boîte de Pétri, et mettre de côté.
- En utilisant des pinces, prendre un 22 mm lamelle de verre ronde et rincer les deux côtés avec le mélange d'éthanol à 100% à l'aide d'une pipette jetable.
- Placez rincé lamelle de verre en 3-aminopropyltriméthoxysilane avec 30 ml d'éthanol à 100% plat de solution et laisser reposer dans la solution pendant 5 min.
- Avec une pince, sortez lamelle de verre et rincer de nouveau avec une solution d'éthanol à 100% avec pipette jetable.
- Déposez rincé lamelle de verre en 1875pi de glutaraldéhyde et 30 ml de mélange d'eau déminéralisée et mettre de côté pendant 30 min.
- Après 30 min, retirez lamelle de verre avec une pince et rincer avec de l'eau déminéralisée et placer sur O de tissu sec / N à sécher à température ambiante.
- Répéter l'immersion de lamelle et émouvant pour autant lamelles au besoin avec les mêmes solutions générées.
- Assurez lamelles hydrophobes par le protocole donné.
- Ajouter 500 ul de tridécafluoro-1,1,2,2-tétrahydrooctyl, 100 ul d'acide acétique et 19,4 ml d'hexane dans un tube de 50 ml et vortex pendant 2 min. Verser la solution dans le verre boîte de Pétri et mettre de côté.
- En utilisant des pinces propres, prendre une lamelle de verre et déposez-les dans une solution mixte préparé pendant 2 min. Après 2 min ont passé, prendre lamelle de verre avec une pince et rincer avec de l'eau déminéralisée à l'aide d'une pipette et le lieu lamelle disponible sur O de tissu sec / N à sécher à température ambiante. lamelle de magasin dans les plats en plastique de Pétri jusqu'à utilisation.
- Répétez lamelle dippING et déplacer autant de lamelles selon les besoins avec les mêmes solutions générées.
- Pour permettre à la matrice de collagène à adhérer à une surface, de créer des lamelles hydrophiles:
2. Assemblée Mold
- Avant d'utiliser des moules, en utilisant une pipette jetable rincer le moule avec de l'alcool éthylique à 90% par rapport à un récipient de déchets chimiques. Ensuite, placez le moule dans une boîte de Pétri remplie d'eau déminéralisée et laisser reposer O / N.
- Prenez moule de la solution en utilisant une pince et placer sur une serviette sécher à l'air avant de commencer l'assemblage de moule.
- Alors que le moule est séchage à l'air, couper les lamelles hydrophiles carrés en deux rectangles légèrement plus grand que 3,95 mm x 5,99 mm en utilisant un outil de diamant traçage de haute précision. Glissez chaque lamelle de rectangle de coupe dans les fentes taillées dans le moule en silicone.
- Retourner le moule à l'envers et appliquer de la graisse à vide le long du fond du moule en silicone en utilisant une pipette jetable. Ensuite, retournez le moule arrière droite et appuyez sur le fond du moule sur les couvercles en verre hydrophiles circulaireslèvres pour créer un joint.
- Avec des gants jetables, ramasser le moule assemblé et le placer dans une boîte de Pétri jetables, puis dans un bio-capot. Allumez la lampe UV Bio-capot pendant 1 heure pour stériliser le moule avant l'expérimentation se produit.
3. Le collagène Mélange et 3D Matrix
- Avant de préparer le mélange de collagène, de la teinture et préparer des cellules pour former une image selon des protocoles standard 28.
- En utilisant des techniques et des protocoles 29,30 laboratoire standard, dans un bio-capot, mélangez jamais des supports cellulaires et chimioattractive dans un tube de 15 ml à la concentration chimioattractive souhaitée. Pipette 5 ml de solution de la concentration des médias-chimioattractive cellulaire spécifique dans un tube de 15 ml et déplacer la solution dans un bain d'eau chauffée.
- En utilisant une pipette, extraire 5 ml de milieu de cellules seulement dans un second tube de 15 ml et placer dans un bain d'eau chauffé pour chauffer la solution pour des expériences de microscopie.
- Dans un bio-aspirante, pipette 30 ul de 10x tampon phosphatesolution ed (PBS) en micro tube de centrifugeuse. Ajouter 6 ul de 1 N d'hydroxyde de sodium (NaOH) en même tube de centrifugation et micro vortex pendant 15 s.
- Obtenir une solution de collagène I de queue de rat dans le réfrigérateur et passer à la bio-hotte en utilisant des techniques et des protocoles 29 laboratoire standard. Vérifiez que le collagène est encore froid. Introduire à la pipette 168,3 pi de collagène dans un même tube de centrifugeuse micro.
- Ensuite, ajouter 18 pi de microsphères modifiées par carboxylate jaune-vert fluorescents à l'aide d'une pipette dans le même tube de centrifugeuse micro. Vortexer le tube de centrifugeuse micro avec toutes les solutions pendant 30 secondes.
- Ajouter 77,7 pi de mélange cellulaire médias cellule souhaitée (environ 2 x 10 6 cellules / ml de concentration) en micro tube de centrifugeuse et mélanger à l'aide pointe de la pipette pendant 20 sec. Le cas échéant, modifier la densité de la matrice en suivant le mélange de collagène personnalisé adapté pour l'addition de billes fluorescentes et une table de la viabilité des cellules (tableau 1).
- Pipette 300 & #181; l de mélange de collagène cellule dans le centre de bien (le puits n ° 2) de l'ensemble de moule préparé. Placer le moule sur une boîte de Petri à usage unique et de le déplacer dans un incubateur standard de 20 minutes à 37 ° C avec 5% de CO 2.
- Déposer le système de l'incubateur et remettez en bio-capot. En utilisant une pince, retirer les deux lamelles hydrophobes par serrage sur le dessus de chaque lamelle et tirant vers le haut et loin de la moule.
- Déplacer l'ensemble du système pour la microscopie et l'image souhaitée pour assurer côtés collagène de moule sont toujours rectiligne pour permettre un gradient de diffusion plane.
- Avec le système encore sous le microscope, en utilisant un seul canal pipette 100 pi, ajouter 100 ul de milieu de cellules dans le puits n ° 1. Ensuite, en utilisant un seul canal pipette 100 ul ajouter 100 pi de milieu cellulaire avec concentration chimioattractive souhaitée dans le puits n ° 3.
- Immédiatement après l'ajout des solutions dans les deux puits, commencera imagerie.
4. Imagerie et DiffusionModélisation
- Si l'on veut trouver le coefficient de diffusion de la densité de collagène spécifique pour calculer la concentration spécifique, suivre le protocole ci-dessous:
- Suivez le protocole donné ci-dessus intitulée «Mélange collagène et matricielle 3D," au lieu de générer les médias de la cellule avec une concentration chimioattractive désiré, faire 5 uM rhodamine dans un tube de 15 ml en utilisant des calculs et des protocoles 30 standard.
- Image en les matrices de collagène 3D avec un système confocal montés sur un microscope inversé en utilisant un laser à argon (488 nm) pour capter la fluorescence. Image toutes les 2 ou 3 secondes pour un maximum de 7 heures.
- Pour Diffusion Modélisation: à l'aide du modèle mathématique illustré ci-dessous, écrire un code de logiciel de calcul pour le traitement et l'analyse de ces étapes générales.
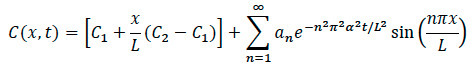
- Importer des images dans le logiciel de calcul pour mise à l'échelle et la normalisation par l'inten maximalesité. Convertir des images à une couleur gris-plan de l'image pour sélectionner le bord. Choix d'un axe le long du centre de l'image, à droite du centre et à la gauche du centre.
- En utilisant le code de logiciel informatique, tracer l'intensité normalisée, avec le vrai intensité et la différence entre les deux.
- Ensuite, entrez les paramètres nécessaires pour le montage (concentration, le temps, et de la longueur).
- Tracer la concentration normalisée en fonction du temps et le profil de concentration dans l'axe des x avec un ajustement polynomial pour les données.
- Calculer le coefficient de diffusion à l'aide du code de logiciel informatique.
5. Mesures expérimentales
- Revenant à l'équation de diffusion, réécrire l'équation pour trouver la concentration spécifique à tout emplacement dans le système, comme indiqué ci-dessous.
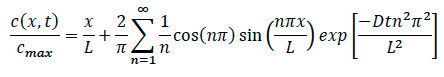
Où:
L = longueur de ee matrice de collagène
x = emplacement sous enquête
D: coefficient de diffusion
t = temps écoulé - Pendant les expériences de chimiotaxie, en sorte d'enregistrer le moment précis que le moment chimiokine a été ajouté au système. Une fois que le mouvement de migration cellulaire est trouvé, s'assurer d'enregistrer confocale enregistrement time-lapse, et notez le moment précis et le lieu à partir du bord où l'imagerie produite.
- Lors de l'analyse de données, branchez le noter le temps écoulé, l'emplacement et le coefficient de diffusion pour la cellule spécifique dans l'équation pour trouver la concentration spécifique normalisée à un point quelconque dans le collagène à la fois pour la diffusion et expériences sur les cellules.
6. Suivi de la migration cellulaire Utilisant TFM
- Pour suivre la migration des cellules en utilisant des techniques TFM / CVN suivent le protocole ci-dessous:
- Après l'addition de la concentration de facteur chimiotactique souhaitée dans le puits n ° 3, commencer l'imagerie des matrices de collagène 3D avec un système confocal monté sur un micr inverséoscope pour trouver une cellule d'enquêter.
- Une fois qu'une cellule se trouve en mouvement, placer la cellule dans le milieu du champ de vision et d'utiliser un moteur piézo-électrique (à l'image dans l'axe z), une image toutes les deux ou min.
- Pour le calcul de génération de force de la cellule et de la déformation, générer un code de calcul pour trouver les déplacements des perles de fluorescence suivant les protocoles TFM / CVN 31.
Résultats
La capacité de cet essai pour évaluer avec précision la migration de la cellule repose sur un bon réglage du système. Par conséquent, il est essentiel pour Assurez-vous de concevoir le moule du système de diffusion précise et prendre grand soin de placer des lamelles à la fois hydrophobes et hydrophiles, comme illustré sur la figure 1. Si le système est bien conçu et pendant la phase de modélisation de la diffusion assurer de trouver un très bonne ligne de départ linéaire, on est en mesu...
Discussion
Les étapes les plus critiques pour les expériences réussies de diffusion avec ou sans cellules sont: configurer correctement l'ensemble de moule; le développement de la dextérité manuelle nécessaire pour empêcher des dommages lors de l'extraction des lamelles hydrophobes; assurer de trouver une très bonne ligne de départ linéaire pour calculer correctement le coefficient de diffusion; corriger les calculs expérimentaux de collagène et chimioattractive; utiliser correctement de système d'imageri...
Déclarations de divulgation
The authors declare that they have no competing financial interests.
Remerciements
The authors would like to acknowledge Drs. Jonathan Reichner and Angle Byrd for cell experiment insight. The National Science Foundation Graduate Research Fellowship Program (GRFP) supported this work.
matériels
| Name | Company | Catalog Number | Comments |
| Silicone elastomer kit | Dow Corning Corp | 182 SIL ELAST KIT .5KG | a two-part misture with a 10:1 mix |
| Live cell imaging chamber | Live Cell Instrument | CM-B18-1 | CMB for 18 mm round coverslips |
| 22 mm Glass Coverslip | Fisher Scientific | NC0180281 | Neuvitro Corp. cover slip 22 mm 1.5 |
| Machined aluminum metal cube | |||
| Hobby utility knife | X-Acto | X3201 | |
| 3-(aminopropyl) trimethoxysilane | Sigma-Aldrich | 281778-5ML | |
| Glutaraldehyde | Polysciences, Inc | 00216A-10 | Glutaraldehyde, EM Grade, 8% |
| 50 ml tube | Fisher Scientific | 14-432-22 | Standard floor model and tabletop centrifuges |
| Glass Petri dish | Fisher Scientific | 08-747A | Reusable Petri Dishes: Complete (60 x 15 mm) |
| Forceps | Fisher Scientific | 22-327-379 | Fine Point Forceps |
| Cover glasses | Fisher Scientific | 12-518-105A | Rectangle; 30 x 22 mm; Thickness No. 1 |
| Tridecafluoro-1,1,2,2-tetrahydrooctyl | Gelest | SIT8174.0 | |
| Acetic acid | Sigma-Aldrich | 320099 | Acetic acid ACS reagent, ≥99.7% |
| Hexane | Sigma-Aldrich | 296090 | anhydrous, 95% |
| Ethyl alcohol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| High-precision diamond scribing tool | Lunzer | PV-081-3 | Straight extended tip scribe, .020" (.50 mm) diameter by .200" (5.0 mm) tip length |
| Vacuum grease | Dow Corning | 14-635-5C | High-Vacuum Grease |
| 15 ml tube | Fisher Scientific | 14-959-49D | 15 ml conical centrifuge tubes with hydrophobic, biologically inert surface |
| 10x phosphate buffered solution | Fisher Scientific | BP399-500 | 1.37 M Sodium Chloride, 0.027 M Potassium Chloride, and 0.119 M Phosphate Buffer |
| 1 N sodium hydroxide | Sigma-Aldrich | 38215 | Sodium hydroxide concentrate |
| Collagen I, rat tail | BD Biosciences | 354236 | Rat tail |
| Micro centrifuge tube | Fisher Scientific | 02-681-332 | Volume: 2.0 ml; O.D. x L: 13 x 40 mm; sterile; single-wrapped |
| [header] | |||
| Carboxylate-modified microspheres | Invitrogen | F-8813 | Carboxylate-modified microspheres, 0.5 µm, yellow-green fluorescent (505/515), 2% solids |
| Rhodamine | Sigma-Aldrich | 83689 | Rhodamine B for fluorescence |
Références
- Adzick, N. S. The Molecular and Cellular Biology of Wound Repair. Ann Surg. 225 (2), 236 (1997).
- Reddi, A. H. Bone morphogenetic proteins: an unconventional approach to isolation of first mammalian morphogens. Cytokine Growth F. R. 8 (1), 11-20 (1997).
- Coussens, , Werb, Z, L. M., Werb, Z. Inflammation and cancer. Nature. 420 (6917), 860-867 (2002).
- Vital-Lopez, F. G., Armaou, A., Hutnik, M., Maranas, C. D. Modeling the effect of chemotaxis on glioblastoma tumor progression. AIChE J. 57 (3), 778-792 (2011).
- Hughes-Alford, S. K., Lauffenburger, D. A. Quantitative analysis of gradient sensing: towards building predictive models of chemotaxis in cancer. Curr. Opin. Cell Biol. 24 (2), 284-291 (2012).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol Cell Bio. 10 (7), 445-457 (2009).
- Cristini, V. Morphologic Instability and Cancer Invasion. Clin. Cancer Res. 11 (19), 6772-6779 (2005).
- Lammermann, T., et al. Rapid leukocyte migration by integrin-independent flowing and squeezing. Nature. 453 (7191), 51-55 (2008).
- Parent, C. A., Devreotes, P. N. A cell's sense of direction. Science. 284 (5415), 765-770 (1999).
- Boyden, S. The chemotactic effect of mixtures of antibody and antigen on polymorphonuclear leucocytes. J. Exp. Med. 115, 453-466 (1962).
- Nelson, R. D., Quie, P. G., Simmons, R. L. Chemotaxis under agarose: a new and simple method for measuring chemotaxis and spontaneous migration of human polymorphonuclear leukocytes and monocytes. J. Immunol. 115 (6), 1650-1656 (1975).
- Rosoff, W. J., McAllister, R., Esrick, M. A., Goodhill, G. J., Urbach, J. S. Generating controlled molecular gradients in 3D gels. Biotechnol. Bioeng. 91 (6), 754-759 (2005).
- Wells, A., Kassis, J., Solava, J., Turner, T., Lauffenburger, D. A. Growth factor-induced cell motility in tumor invasion. Acta Oncol. 41 (2), 124-130 (2002).
- Wilkinson, P. C. Assays of leukocyte locomotion and chemotaxis. J. Immunol. Methods. 216 (1-2), 139-153 (1998).
- Bonvin, C., Overney, J., Shieh, A. C., Dixon, J. B., Swartz, M. A. A multichamber fluidic device for 3D cultures under interstitial flow with live imaging: development, characterization, and applications. Biotechnol. Bioeng. 105 (5), 982-991 (2010).
- Noo, L. J., et al. Neutrophil chemotaxis in linear and complex gradients of interleukin-8 formed in a microfabricated device. Nat. Biotechnol. 20 (8), 826-830 (2002).
- Shao, J., et al. Integrated microfluidic chip for endothelial cells culture and analysis exposed to a pulsatile and oscillatory shear stress. Lab Chip. 9 (21), 3118-3125 (2009).
- Haessler, U., Kalinin, Y., Swartz, M. A., Wu, M. An agarose-based microfluidic platform with a gradient buffer for 3D chemotaxis studies. Biomed. Microdevices. 11 (4), 827-835 (2009).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat. Rev. Cancer. 3 (5), 362-374 (2003).
- Sixt, M., Lammermann, T. In vitro analysis of chemotactic leukocyte migration in 3D environments. Methods Mol. Biol. 769, 149-165 (2011).
- Zigmond, S. H. Ability of polymorphonuclear leukocytes to orient in gradients of chemotactic factors. J. Cell Biol. 75 (2 Pt 1), 606-616 (1977).
- Zicha, D., Dunn, G. A., Brown, A. F. A new direct-viewing chemotaxis chamber. J. Cell. Sci. 99 (Pt 4), 769-775 (1991).
- Gilbert, D. L. Macromolecular diffusion through collagen membranes. Int. J. Pharm. 47 (1-3), 79-88 (1988).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys. J. 83 (3), 1650-1660 (2002).
- Weadock, K., Silver, F. H., Wolff, D. Diffusivity of 125I-calmodulin through collagen membranes: effect of source concentration and membrane swelling ratio. Biomaterials. 7 (4), 263-267 (1986).
- Erikson, A., Andersen, H. N., Naess, S. N., Sikorski, P., Davies, C. d. L. Physical and chemical modifications of collagen gels: impact on diffusion. Biopolymers. 89 (2), 135-143 (2008).
- Moghe, P. V., Nelson, R. D., Tranquillo, R. T. Cytokine-stimulated chemotaxis of human neutrophils in a 3-D conjoined fibrin gel assay. J. Immunol. Methods. 180 (2), 193-211 (1995).
- Sundd, P., et al. Live cell imaging of paxillin in rolling neutrophils by dual-color quantitative dynamic footprinting. Microcirculation. 18 (5), 361-372 (2011).
- Sanders, E. R. Aseptic Laboratory Techniques: Volume Transfers with Serological Pipettes and Micropipettors. J. Vis. Exp. (63), 2754 (2012).
- Stephenson, F. H. . Calculations for Molecular Biology and Biotechnology: A Guide to Mathematics in the Laboratory 2e. , (2010).
- Bar-Kochba, E., Toyjanova, J., Andrews, E., Kim, K., Franck, C. A Fast Iterative Digital Volume Correlation Algorithm for Large Deformations. Exp. Mech. In press, (2014).
- Shenoy, V., Rosenblatt, J. Diffusion of Macromolecules in Collagen and Hyaluronic Acid, Rigid-Rod-Flexible Polymer Composite Matrixes. Macromolecules. 28 (26), 8751-8758 (1995).
- Leddy, H., Guilak, F. Site-Specific Molecular Diffusion in Articular Cartilage Measured using Fluorescence Recovery after Photobleaching. Ann. Biomed. Eng. 31 (7), 753-760 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon