Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Haute résolution structurelle Imagerie par résonance magnétique de la sous-cortex humain
Dans cet article
Résumé
Here we present a protocol to determine the minimum number images that needed to be registered and averaged to resolve subcortical structures and test whether the individual layers of the LGN could be resolved in the absence of physiological noise.
Résumé
L'objectif de cette étude était de tester les limites de résolution de l'IRM structurelle d'un cerveau post-mortem par rapport à la vie des cerveaux humains. La résolution de l'IRM structurelle in vivo est finalement limitée par le bruit physiologique, y compris la pulsation, la respiration et le mouvement de la tête. Bien que le matériel d'imagerie continue à améliorer, il est encore difficile de résoudre des structures à l'échelle du millimètre. Par exemple, le premier visuel voies sensorielles synapse au corps genouillé latéral (CGL), un relais visuel et le noyau de contrôle dans le thalamus qui est normalement organisé en six couches de monoculaire entrelacés. Les études de neuroimagerie ont pas été en mesure de distinguer de façon fiable ces couches en raison de leur petite taille qui sont moins de 1 mm d'épaisseur.
La limite de résolution de l'IRM structurelle, dans un cerveau post-mortem a été testé à l'aide de plusieurs images en moyenne sur une longue durée (~ 24 h). Le but était de vérifier si il était possible de résoudre le l individuAyers du CGL en l'absence de bruit physiologique. Une densité de proton (PD) d'une séquence d'impulsions pondérée a été utilisée avec plus ou moins de résolution et d'autres paramètres pour déterminer le nombre minimum d'images nécessaires à l'enregistrement et à distinguer de manière fiable en moyenne le LGN et d'autres régions sous-corticales. Les résultats ont également été comparés à des images acquises en vivant les cerveaux humains. In vivo sujets ont été scannés afin de déterminer les effets supplémentaires de bruit physiologique sur le nombre minimum de scans de perfectionnement nécessaires pour différencier les structures sous-corticales, utiles dans les applications cliniques.
Introduction
Le but de cette recherche était de tester les limites de résolution de l'IRM structurelle en l'absence de bruit physiologique. Proton densité (PD) pondérés images ont été acquises dans un cerveau post-mortem sur une longue durée (deux ~ 24 séances de h) pour déterminer le nombre minimum d'images qui devaient être enregistrées et en moyenne pour résoudre les structures sous-corticales. A titre de comparaison, PD images pondérés ont également été acquises chez les humains vivant sur un certain nombre de séances. En particulier, l'objectif était de vérifier si il serait possible dans un meilleur scénario pour résoudre tous les six couches individuelles du LGN humaine, qui sont d'environ 1 mm d'épaisseur (Figure 1).
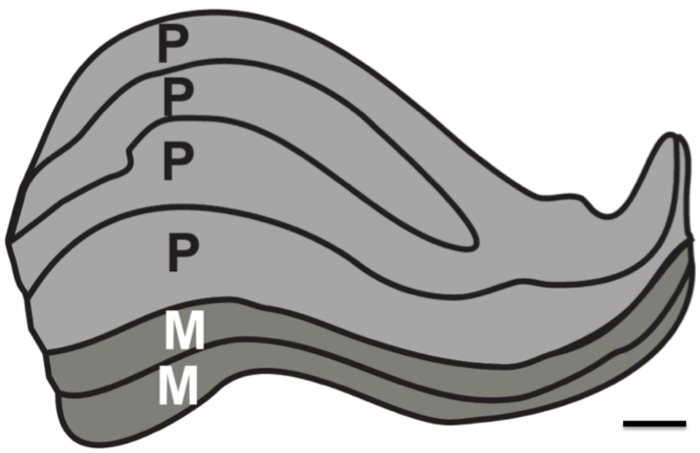
Figure 1. Human géniculé latéral couches noyau. Schématique de la structure laminaire des LGN. Magnocellulaire (M) couches sont constituées de plus grande neuronalela taille des cellules et de la densité de cellules plus petites qui sont responsables pour la résolution de mouvement et de cours contours (couches 1-2, représentée en gris foncé). Parvocellulaires couches (P) sont constitués de petite taille des cellules neuronales et la densité de cellule plus grande qui sont responsables pour la résolution fine forme et la couleur (couches 4-6, dépeint comme gris clair). Barre d'échelle 1 mm. Figure basé sur teinté LGN 12 humain.
La résolution spatiale est améliorée en IRM lorsque la taille de la matrice est augmenté, et quand de champ de vue (FOV) et l'épaisseur de coupe sont réduites. Cependant, l'augmentation de la résolution diminue le rapport signal sur bruit (SNR), qui est proportionnel au volume de voxel. SNR est aussi proportionnelle à la racine carrée du nombre de mesures. Chez l'homme vivant, bien que plusieurs images peuvent être acquises sur un certain nombre de séances d'imagerie distincts, la résolution finale est limitée par le bruit physiologique, tels que la respiration, pulsations circulatoires et mouvement de la tête.
Haut-Résolution (0,35 mm voxels dans le plan) PD analyses pondérées ont été acquis. Scans PD améliorer le contraste gris et blanc dans le thalamus 1, et se traduisent par des images qui minimisent T 1 et T 2 effets. Son image est fonction de la densité de protons sous forme d'eau et de macromolécules telles que des protéines et des matières grasses dans le volume d'imagerie. L'augmentation du nombre de protons dans un tissu résulte en un signal lumineux sur l'image due à la composante longitudinale supérieur de l'aimantation 2.
Scans PD-pondérés ont été recueillies depuis ils offrent un contraste plus élevé de structures sous-corticales avec le tissu environnant. Autres contrastes, comme T1 et images pondérées en T2 entraîner des difficultés dans la délimitation des structures sous-corticales comme le LGN en raison de petits ratios contraste-bruit, tel que déterminé ƒ 1,3.
De même, des études antérieures ont trouvé que les images de PD pondérée de formol fixés cerveaux post mortem resultej en plus élevés différences de contraste entre la matière grise et blanche par rapport à T1 et images pondérées en T2 qui avaient gris et blanc image importe intensités semblables 3,4. Les déterminants biophysiques sous-jacents peuvent expliquer ces différences. T1 (longitudinale) et T2 (transversal) temps de relaxation des protons de l'hydrogène dépendra de la façon l'eau se déplace à l'intérieur du tissu. Fixateurs tels que le travail de formol par des protéines de réticulation. Les différences entre la mobilité de l'eau sont réduites entre différents types de tissus utilisés lorsque fixateurs sont. Réduit le contraste des tissus T1 a été observé après fixation, tandis que les différences dans la densité de protons dans les tissus cérébraux augmente avec la fixation, permettant une meilleure différenciation de contraste 3, 4.
Des études précédentes ont identifié le CGL dans les analyses de Pd-pondérés en utilisant un T 5,6,7 1,5, et à 3 T scanner 8,9. Il est essentiel pour obtenir ces scans pour être en mesure de décrire précisément l'étendue dele CGL. Pour maintenir une couverture complète des noyaux sous-corticaux, 18 tranches de PD-pondérés ont été obtenus dans le thalamus. Chaque volume a été ré-échantillonné à deux fois la matrice résolution 1024, (0,15 mm taille dans le plan voxel), enchaîné, le mouvement corrigé et en moyenne pour produire une image 3D haute résolution de structures sous-corticales. Le nombre optimal d'images PD requis pour la prescription de la tranche suivante était de 5, ce qui réduit le temps de cycle à moins de 15 minutes chez l'homme vivant. Seulement 1 image PD a été nécessaire de délimiter clairement régions sous-corticales du cerveau post-mortem, réduisant le temps de cycle à moins de 3 min (Figure 2 et 3).
Un ensemble du cerveau post-mortem spécimen fixés au formol a été balayée d'une femme qui avait décédé d'un arrêt cardio-respiratoire à l'âge de 82 ans. Examen des dossiers médicaux a révélé qu'elle avait: maladie pulmonaire obstructive chronique, la chirurgie de poitrine, triple pontage 8 ans avant le décès, le cancer de l'utérus traitée avec une hystérectomie7 ans avant le décès, l'hyperlipidémie, le glaucome, et la chirurgie de la cataracte. Le cerveau spécimen post-mortem a été fixés par immersion dans 10% formol tamponné neutre pendant au moins 3 semaines à 4 ° C.Le post-mortem du cerveau a été scannée avec le même protocole d'imagerie ainsi que d'autres paramètres au cours de nombreuses heures pour les comparaisons de qualité d'image . Seuls les paramètres optimisés seront décrites pour le protocole.
Protocole
1. Participant et post-mortem du cerveau Set-Up
NOTE: Toutes les images ont été acquises en utilisant un scanner IRM 3 T avec une bobine de la tête à 32 canaux et tout l'IRM a été effectuée à la température ambiante, environ 20 ° C. Tous les participants étaient droitier et a donné un consentement éclairé écrit. Chaque participant était en bonne santé et sans antécédents de troubles neurologiques. Le protocole expérimental a été approuvé et suit les directives de participants de l'Université York humaines Comité d'examen.
- Demandez à chaque participant de remplir et signer un formulaire de consentement du patient qui détaille les directives de sécurité de l'IRM et le protocole neuro-imagerie.
- Pour chaque participant, placer des bouchons d'oreille dans chaque oreille et assurer leur tête avec pads pour minimiser le mouvement de la tête.
- Pour post-mortem imagerie cérébrale, assurez-vous que le cerveau est fixé avant la neuro-imagerie et est contenue dans un sac ou un contenant qui correspond dans la tête-bobine IRM. Placez le cerveau post-mortem dans la tête-bobine avec son axe z (supérieure à inferior) alignée avec l'alésage du scanner. Le tronc cérébral (postérieure) doit faire face vers le pied du lit du scanner.
- Placez les mains de coussin à vide autour du cerveau post-mortem pour un soutien supplémentaire.
2. Localiser et fixant la subcortex
Remarque: le thalamus est une structure à double lobe situé à proximité du centre du cerveau situé entre le mésencéphale et le cortex cérébral. Situé à l'intérieur du thalamus dorsal, le LGN humain est une petite structure sous-corticale qui se prolonge au maximum de ~ 10 mm.
- Pour enregistrer un nouveau participant, ouvrez le logiciel d'imagerie IRM et cliquez sur l'onglet du patient dans le coin supérieur gauche. Ensuite, cliquez sur Enregistrer.
- Remplissez les informations du patient approprié, puis cliquez sur l'onglet d'examen.
- Pour obtenir une analyse d'alignement de piste, cliquez sur l'onglet Explorateur de l'examen de créer un nouveau protocole. Observez la fenêtre set-up sur l'écran, cliquez sur l'onglet de routine, et entrez les paramètres suivants: acquisitiontemps 28 sec, matrice de l'acquisition de 160 × 160, 1 tranche, 1,6 mm d'épaisseur taille de voxel isotrope, FOV = 260 mm, la phase FoV = 100%, la résolution de la tranche = 69%, la phase et trancher la phase partielle Fourier = 6/8, TR = 3.15 ms, TE = 1,37 ms, Flip Angle = 8 °.
- Superposez la boîte de sélection de tranche utilisé pour l'acquisition des images PD plus de l'alignement de piste couvrant les noyaux sous-corticaux dans le thalamus ainsi que des structures environnantes (Figure 4).
3. Paramètres structurels haute résolution
- Créer un nouveau protocole pour obtenir des analyses de Pd-pondérée à haute résolution. Dans la fenêtre de configuration de l'écran, cliquez sur l'onglet de routine, et entrez les paramètres suivants dans l'orientation coronale: temps d'acquisition de 179 secondes, l'acquisition matrice 512 × 512, 0,3 × 0,3 × 1 mm 3 taille de voxel, TR = 3.25 sec , TE = 32 ms, angle de bascule = 120 °, l'acquisition de la tranche entrelacé, FoV lu = 160 mm, FoV la phase = 100%, l'imagerie parallèle (Grappa) avecun facteur d'accélération de 2.
- Utilisez une séquence Turbo Spin Echo, avec une longueur Echo train de 5. Le premier écho à 32 ms est l'écho efficace pour cette séquence. Réduire la bande passante (BW) à la mesure du possible, 40 Hz / pixel minimum, afin de maximiser le SNR. Pour réduire la durée du balayage, choisissez 18 tranches, chaque 1 mm d'épaisseur, avec un FOV = 160 mm. Cette dalle offre une couverture suffisante des régions sous-corticales d'intérêt.
NOTE: Pour une identification fiable des structures sous-corticales, acquérir 5 pistes avec les paramètres ci-dessus. La durée totale de l'analyse est seulement ~ 15 min (Figure 5). Fat-saturation n'a pas été employé.
- Utilisez une séquence Turbo Spin Echo, avec une longueur Echo train de 5. Le premier écho à 32 ms est l'écho efficace pour cette séquence. Réduire la bande passante (BW) à la mesure du possible, 40 Hz / pixel minimum, afin de maximiser le SNR. Pour réduire la durée du balayage, choisissez 18 tranches, chaque 1 mm d'épaisseur, avec un FOV = 160 mm. Cette dalle offre une couverture suffisante des régions sous-corticales d'intérêt.
- En post-mortem imagerie cérébrale, une identification fiable des structures sous-corticales peut être observée dans un seul scan avec la durée totale de seulement ~ 3 min suivant le même protocole de balayage comme dans 3.1 (figure 6).
4. Analyse d'image
REMARQUE: Pour analyser les données de l'IRM, utilisez la libre disposition de FMRIBBibliothèque de logiciels (FSL) forfait disponible pour téléchargement à l'adresse (https://www.fmrib.ox.ac.uk/fsl/).
- Ouvrez une fenêtre de terminal, et de convertir les fichiers DICOM premières à partir du scanner pour chaque volume de PD au format DICOM NIfTI avec un convertisseur NIfTI. Un certain nombre de qui sont librement disponibles pour le téléchargement (par ex., Https://www.nitrc.org/projects/mricron). Dans la ligne de commande, tapez dcm2nii suivi par le répertoire de chaque PD l'image pondérée terme.
- Dans une fenêtre de terminal obtenir les paramètres de la numérisation de PD originale. Tapez fslinfo dans la ligne de commande, suivi par le balayage de PD au format NIfTI.
- Créer une image vierge volume cible à haute résolution qui a deux fois la résolution et la moitié de la taille de voxel donné par les paramètres de fslinfo de l'analyse de PD originale. L'ordre des entrées de données pour cette commande sont les suivants:
fslcreatehd
REMARQUE: Par exemple, si l'analyse de PD originale avec les paramètres suivants tels que décrits au paragraphe 3.1 sont collectées (par exemple, 512 × 512 matrice, 18 tranche, 0,3 × 0,3 × 1 mm 3 taille de voxel, TR = 3,25 s), tapez la commande suivante dans la fenêtre de commande:
fslcreatehd 1024 1024 0,15 0,15 36 1 0,5 3,25 0 0 0 4 blankhr.nii.gz - Définir la transformation en utilisant une matrice d'identité. Tapez dans tout programme d'éditeur de texte dans un fichier texte enregistré en tant que 'identity.mat' qui ressemble à ceci:
0 0 0
1 0 0
0 1 0
0 0 1 - Utilisez la commande de flirt pour appliquer la transformation, échantillonnant chaque exécution pondérée PD originale de doubler la résolution totale d'une 512 à une matrice de 1024, et de réduire de moitié la taille de voxel dans chaque dimension résultant en une résolution de 0,15 × 0,15× 0,5 mm 3. Dans une fenêtre de terminal pour chaque volume de PD, tapez la commande suivante de flirt changer les noms originaux et de sortie par course:
flirter -interp sinc -in originalPD.nii.gz -ref blankhr.nii.gz -applyxfm -init identity.mat départ privé highresPD.nii.gz
NOTE: Où originalPD.nii.gz est le volume source, blankhr.nii.gz est la résolution de sortie souhaitée, et highresPD.nii.gz est le nom du volume de sortie. - Déplacez toutes les images à haute résolution pour un nouveau dossier, et s'y rendre dans une fenêtre de terminal.
- Pour chaque participant, concaténer toutes les images PD suréchantillonnées dans un fichier unique à l'aide de 4D fslmerge. Dans une fenêtre de terminal:
fslmerge -t concat_highresPD * .nii.gz
NOTE: Cela crée un fichier appelé 4D concat_highresPD.nii.gz. - Mouvement corriger le fichier concaténé utilisant mcflirt 10. Cet outil permet un enregistrement automatisé robuste pour linéaire (affine) des images inter et intermodales cerveau. Sélectionner unCorrection quatre étapes, qui utilise une interpolation sinc (interne) comme une nouvelle passe d'optimisation pour une meilleure précision. Dans une fenêtre de terminal:
mcflirt -in concat_highresPD départ privé mcf_concat_highresPD.nii.gz -stages 4 -plots
NOTE: Cela crée un fichier appelé 4D mcf_concat_highresPD.nii.gz. - Enfin, créer la 3D signifie image en utilisant fslmaths. Dans une fenêtre de terminal:
fslmaths mcf_concat_highresPD.nii.gz -Tmean mean_highresPD.nii.gz
NOTE: Cela crée un fichier 3D appelé mean_highresPD.nii.gz qui est de haute qualité - Visualiser l'image à haute résolution de résultat final 3D en utilisant la commande FSLView. Dans le répertoire de l'endroit où votre image est, tapez la commande suivante dans une fenêtre de terminal:
FSLView mean_highresPD.nii.gz. " - Inspectez profils d'intensité de Rois en question. Créer un ROI en utilisant FSLView (cela peut être une ligne verticale tracée à travers une région de l'LGN par exemple). Dans FSLView charger l'image de PD-haute résolution. Cliquez sur l'onglet Outils,puis cliquez sur l'onglet d'une seule image pour agrandir l'image pour dessiner ROI. Ensuite, cliquez sur l'onglet Fichier puis sur l'onglet Créer un masque. Tracez une ligne dans le ROI d'intérêt. Enregistrez le ROI en cliquant sur Fichier, puis Enregistrer sous. Répétez les masques de ligne pour de multiples domaines dans le retour sur investissement pour les comparaisons d'intensité et d'autres régions d'intérêt en question.
- Utilisez la commande 3dmaskdump de AFNI d'analyser l'intensité résultant de l'image. Dans l'annuaire des où les images sont, utilisez la commande suivante dans une fenêtre de terminal pour extraire les intensités et le placement d'image (donné comme result_mask.txt) de votre masque de ROI:
3dmaskdump -o result_mask.txt -noijk -xyz -mask ROI_linemask.nii.gz PDaverage_image.nii.gz
Résultats
Une fois que le sous-cortex est prescrit dans le thalamus, PD images pondérées sont recueillies dans la boîte de sélection de tranche (figure 4). Le SNR améliorée en augmentant le nombre de moyennes à la fois dans les analyses post-mortem et in vivo. Pour déterminer la qualité de l'image, le SNR à partir de différentes moyennes de balayage a été comparée en divisant le signal de la région cérébrale moyenne par l'écart type d'une certaine zone à l'ext?...
Discussion
Cette étude décrit un protocole optimisé dans l'acquisition et la technique de l'analyse afin d'obtenir haute résolution PD images pondérées des régions sous-corticales. Un certain nombre de paramètres d'analyse ont été testé et modifié avec les plus importantes celles relatives à la taille de la matrice, la taille de voxel, et la bande passante pour augmenter le SNR et de diminuer le nombre d'acquisitions, une étape cruciale de pouvoir déterminer les structures sous-corticales haute r...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
The authors acknowledge the following funding sources, the Natural Sciences and Engineering Research Council of Canada (NSERC), the Dorothy Pitts Research Fund (NG), and the Nicky and Thor Eaton Research Fund. The authors acknowledge Kevin DeSimone, and Aman Goyal and for their knowledge in MRI acquisition and analysis expertise.
matériels
| Name | Company | Catalog Number | Comments |
| Magnetom Trio 3T MRI | Siemens (Erlangen, Germany). | ||
| Vacuum cushion hand | Siemens | Mat No: 4765454 | Manufactured by: Johannes-Stark-Stk. 8 D-92224 Amberg |
Références
- Devlin, J. T., et al. Reliable identification of the auditory thalamus using multi-modal structural analyses. NeuroImage. 30 (4), 1112-1120 (2006).
- Fellner, F., et al. True proton density and T2-weighted turbo spin-echo sequences for routine MRI of the brain. Neuroradiology. 36 (8), 591-597 (1994).
- Schumann, C. M., Buonocore, M. H., Amaral, D. G. Magnetic resonance imaging of the post-mortem autistic brain. J Autism Dev Disord. 31 (6), 561-568 (2001).
- Tovi, M., Ericsson, A. Measurements of T1 and T2 over time in formalin-fixed human whole-brain specimens. Acta Radiol. 33 (5), 400-404 (1992).
- Fujita, N., et al. Lateral geniculate nucleus: anatomic and functional identification by use of MR imaging. Am J Neuroradiol. 22 (9), 1719-1726 (2001).
- Bridge, H., Thomas, O., Jbabdi, S., Cowey, A. Changes in connectivity after visual cortical brain damage underlie altered visual function. Brain. 131 (6), 1433-1444 (2008).
- Gupta, N., et al. Atrophy of the lateral geniculate nucleus in human glaucoma detected by magnetic resonance imaging. Br J Opthalmol. 93 (1), 56-60 (2009).
- Dai, H., et al. Assessment of lateral geniculate nucleus atrophy with 3T MR imaging and correlation with clinical stage of glaucoma. Am J Neuroradiol. 32 (7), 1347-1353 (2011).
- McKetton, L., Kelly, K. R., Schneider, K. A. Abnormal lateral geniculate nucleus and optic chiasm in human albinism. J Comp Neurol. 522 (11), 2680-2687 (2014).
- Jenkinson, M., Bannister, P., Brady, M., Smith, S. Improved optimization for the robust and accurate linear registration and motion correction of brain images. NeuroImage. 17 (2), 825-841 (2002).
- Dietrich, O., Raya, J. G., Reeder, S. B., Reiser, M. F., Schoenberg, S. O. Measurement of signal-to-noise ratios in MR images: influence of multichannel coils, parallel imaging, and reconstruction filters. J Magn Reson Imaging. 26 (2), 375-385 (2007).
- Andrews, T. J., Halpern, S. D., Purves, D. Correlated size variations in human visual cortex, lateral geniculate nucleus, and optic tract. J Neurosci. 17 (8), 2859-2868 (1997).
- Pfefferbaum, A., Sullivan, E. V., Adalsteinsson, E., Garrick, T., Harper, C. Postmortem MR imaging of formalin-fixed human brain. NeuroImage. 21 (4), 1585-1595 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon