Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
La cartographie du génome à l'échelle du médicament-ADN interactions dans les cellules avec COSMIC (réticulation de petites molécules pour isoler la chromatine)
Dans cet article
Résumé
Identifier les cibles directes de molécules du génome ciblage reste un défi majeur. Pour comprendre comment les molécules de liaison à l'ADN engagent le génome, nous avons développé une méthode qui repose sur la réticulation de petites molécules à isoler la chromatine (COSMIC).
Résumé
Le génome est la cible de certains agents chimiothérapeutiques les plus efficaces, mais la plupart de ces médicaments manquer de spécificité de séquence d'ADN, ce qui conduit à la dose-limitant la toxicité et d'effets secondaires indésirables. Le ciblage du génome de petites molécules spécifiques de séquence peut permettre molécules ayant un indice thérapeutique accru et moins d'effets hors cible. -methylpyrrole N / N -méthylimidazole polyamides sont des molécules qui peuvent être rationnellement conçus pour cibler des séquences d'ADN spécifiques avec précision exquise. Et contrairement à la plupart des facteurs de transcription naturels, les polyamides peuvent se lier à l'ADN méthylé et chromatinized sans perte d'affinité. La spécificité de séquence de polyamides a été largement étudié in vitro avec l'identification du site apparenté (CSI) et avec des approches biochimiques et biophysiques traditionnels, mais l'étude de polyamide se lier à des cibles génomiques dans les cellules reste insaisissable. Nous rapportons ici une méthode, la réticulation de petites molécules pour Isolate chromatine (COSMIC), qui identifie les sites de liaison de polyamide à travers le génome. Cosmique est similaire à immunoprécipitation de la chromatine (ChIP), mais diffère de deux manières importantes: (1) un photoréticulant est utilisé pour permettre sélectif capture, temporellement contrôlée par des événements de liaison de polyamide, et (2) la poignée d'affinité de la biotine est utilisée pour purifier le polyamide -ADN conjugués dans des conditions semi-dénaturantes pour diminuer l'ADN qui est lié de manière non covalente. Cosmique est une stratégie générale qui peut être utilisée pour faire apparaître les événements de liaison du génome entier de polyamides et d'autres agents chimiothérapeutiques du génome de ciblage.
Introduction
Les informations de rendre chaque cellule dans le corps humain est codé dans l'ADN. L'utilisation sélective de l'information régit le destin d'une cellule. Les facteurs de transcription (TF) sont des protéines qui se lient à des séquences spécifiques d'ADN pour exprimer un sous-ensemble particulier de gènes dans le génome, et le mauvais fonctionnement de TFS est liée à l'apparition d'un large éventail de maladies, y compris les défauts de développement, le cancer et le diabète . 1,2 Nous avons été intéressés par le développement de molécules qui peuvent se lier sélectivement à la génomique et de moduler les réseaux de régulation génétique.
Polyamides composés de N et N -méthylimidazole -methylpyrrole sont des molécules qui peuvent cibler l'ADN avec des spécificités et des affinités que les facteurs de transcription naturels rival. 3-6 Ces molécules se lient à des séquences spécifiques dans le sillon de l'ADN conçu rationnellement. 4,5,7 -11 polyamides ont été employées à la fois réprimer et activer l'expression de g spécifiqueEnes. 4,12-19 Ils ont aussi intéressants antiviraux et anticancéreux 20-24 12,13,25-30 propriétés. Une caractéristique intéressante de polyamides est leur capacité à accéder à des séquences d'ADN qui sont méthylés 31,32 et enroulé autour de protéines histones 9,10,33.
Pour mesurer les spécificités de liaison complets de molécules de liaison à l'ADN, notre laboratoire a créé la méthode apparentée identifiant du site (CSI). 34-39 L'apparition prévue de sites de liaison basées sur des spécificités (genomescapes) in vitro peuvent être affichées sur le génome, parce que la in vitro intensités de liaison sont directement proportionnelles à constantes d'association (K a). 34,35,37 Ces genomescapes donnent un aperçu de polyamide occupation à travers le génome, mais polyamide mesurant la liaison dans les cellules vivantes a été un défi. ADN est bien emballé dans le noyau, ce qui pourrait influer sur l'accessibilité des sites de liaison. Le Accessibilité de ces séquences d'ADN chromatinized à polyamides reste un mystère.
Récemment, de nombreuses méthodes pour étudier les interactions entre les petites molécules et les acides nucléiques ont émergé. 40-48 La capture d'affinité chimique et séquençage de l'ADN massivement parallèle (chem-SEQ) est une telle technique. Chem-seq utilise le formaldehyde pour réticuler petites molécules sur une cible génomique d'intérêt et un dérivé biotinylé d'une petite molécule d'intérêt à capter l'interaction ligand-cible. 48,49
Formaldéhyde réticulation conduit à des interactions indirectes qui peuvent produire des faux positifs. 50 Nous avons développé une nouvelle méthode, la réticulation de petites molécules d'isoler la chromatine (COSMIC), 51 avec un photoréticulant pour éliminer ces soi-disant pics "fantômes". 50 Pour commencer, nous avons conçu et synthétisé des dérivés trifonctionnels de polyamides. Ces molécules contenaient une pol de liaison à l'ADNyamide, un photoréticulant (psoralène), et une poignée d'affinité (biotine, figure 1). Avec polyamides trifonctionnels, on peut de manière covalente capturer interactions polyamide-ADN avec 365 nm irradiation UV, une longueur d'onde qui ne nuit pas à l'ADN ou l'induit non-psoralène à base de reticulation. 51 Ensuite, on fragmente le génome et purifier l'ADN capturé sous stricte, semi -denaturing conditions pour diminuer l'ADN qui est lié de manière non covalente. Ainsi, nous considérons cosmique comme une méthode liée à Chem-seq, mais avec une lecture plus directe de l'ADN de ciblage. Fait important, la faible (K 10 3 -10 4 M -1) affinité pour l'ADN de psoralène ne pas détectable incidence polyamide spécificité. 51,52 Les fragments d'ADN peuvent être analysés enrichies soit par polymérase quantitative réaction en chaîne 51 (COSMIC-qPCR) ou par séquençage de prochaine génération 53 (COSMIC-seq). Ces données permettent une conception impartiale, génome guidée de ligands qui entreagir avec leur loci génomiques désiré et minimiser les effets hors-cible.
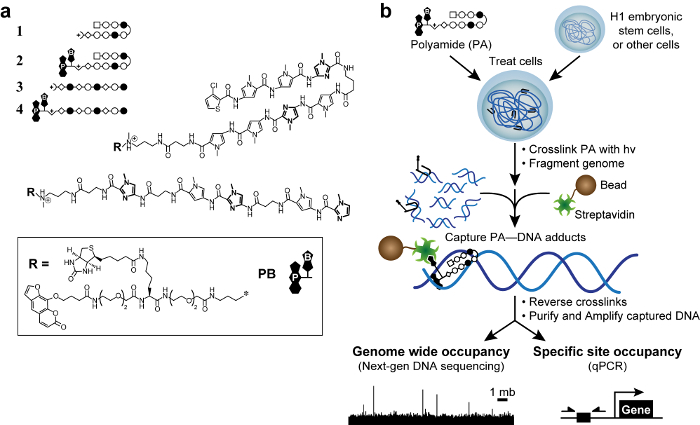
Figure 1. polyamides bioactifs et plan cosmique. Épingle à cheveux (A) les polyamides 1-2 cible la séquence d'ADN 5'-WACGTW-3 '. Polyamides linéaires 3 - 4 cibles 5'-AAGAAGAAG-3 '. Anneaux de N-méthylimidazole sont en gras pour plus de clarté. Les cercles vides et pleins représentent N et N -méthylimidazole -methylpyrrole, respectivement. Place représente 3-chlorothiophène, et les diamants représentent β-alanine. Psoralène et la biotine sont désignés par P et B, respectivement. (B) plan cosmique. Les cellules sont traitées avec les dérivés trifonctionnels de polyamides. Après réticulation par irradiation UV à 365 nm, les cellules sont lADN génomique et ysed est cisaillée. Des billes magnétiques revêtues de streptavidine sont ajoutées à saisir des produits d'addition polyamide-ADN. L'ADN est libéré et peut être analysé par PCR quantitative (qPCR) ou par séquençage de prochaine génération (NGS). S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
Protocole
1. La réticulation dans des cellules vivantes
- Commencer par 2,5x10 ~ 7 cellules H1, ou d'autres cellules en culture.
NOTE: Ce nombre de cellules H1 correspond à cinq plats de 10 cm (environ 40% de confluence). - Produire des cellules dans E8 médias sur des plats revêtus sur une surface qui soutient les cellules souches pluripotentes (voir list des matériaux) et les incuber à 37 ° C dans une atmosphère humidifiée de 5% de CO 2. Cellules de récolte enzymatique (voir liste matériaux). Remarque: Ne laissez pas les cellules de H1 à dépasser 90% de confluence; confluence induit la différenciation spontanée des cellules H1. Pour compter les cellules, de grandir un 10 cm extra plat de cellules, lever les cellules enzymatique tels que décrits dans les sections 1.8-1.10, et les compter avec un hémocytomètre.
- Préparer E8 milieux de culture comme décrit dans Chen et al. 54
- Avant d'ajouter polyamide, retirez le support de culture passé avec une pipette Pasteur attaché à un piège à vide. Ajouter des médias frais avec commepipette erological et la pipette distributeur (8 ml par 10 cm plat).
NOTE: Ajouter des médias sur le côté de la boîte afin d'éviter de perturber les cellules. De ce point en avant de protéger les cellules de la lumière pour éviter prématurée photo-réticulation. - Ajouter polyamide avec une pipette (8 ul de 400 uM dans du DMSO, polyamide 400 nM concentration finale) directement au milieu de culture de chaque plat. Agiter le plat pour disperser le polyamide uniformément dans les médias.
NOTE: La concentration peut être variée, mais veiller à ce qu'aucune toxicité cellulaire est observé pour le traitement choisi. - Incuber les cellules 24 h 37 ° C dans une atmosphère humidifiée de 5% de CO 2, et de veiller à ce qu'ils soient protégés de la lumière.
NOTE: Le temps d'incubation peut être modifiée pour mesurer la liaison à travers le temps polyamide. - Laver chaque plat de 10 cm avec 4 ml de PBS (1,05 mM de phosphate de potassium monobasique, le chlorure de sodium 155,17 mM, 2,97 mM de phosphate de sodium dibasique) en utilisant une pipette et pipette sérologique de disPenser. Aspirer le PBS et ajouter 3 ml E8 milieux de culture.
- Avec les lumières se sont éteintes, retirez le couvercle de 5 boîtes de culture et de placer les cellules sur une surface plane à l'extérieur de la hotte. Placez un filtre de verre au cours des 5 boîtes de culture pour filtrer la lumière avec λ <300 nm. Placer la source UV sur le dessus du filtre. Échantillons de reticulation 30 min à 365 nm irradiation UV (2,4 mW / cm 2).
REMARQUE: Le temps de réticulation doit être déterminée de manière empirique. - Transfert des boîtes de culture de retour à la hotte. Aspirer les médias avec une pipette Pasteur attaché à un piège à vide. Lavez chaque plat de 10 cm avec 4 ml de PBS. Aspirer le PBS. Ajouter 3 ml enzyme pour la dissociation cellulaire (voir liste matériaux) par 10 cm plat pour dissocier les cellules. Incuber 5 min à 37 ° C.
REMARQUE: Ne pas préchauffer l'enzyme. - Étancher l'enzyme avec 3 ml E8 médias par plat. Transfert dissocié cellules dans un tube conique de 15 ml.
NOTE: La place des cellules sur la glace partir de ce point. - CentriFuge dissocié cellules 5 min, 500 g à 4 ° C. Aspirer le surnageant pour éliminer l'enzyme et les médias.
NOTE: Mettre en pause le point. Les cellules peuvent être congelées instantanément dans de l'azote liquide, stocké à -80 ° C pour un traitement ultérieur à un moment ultérieur.
2. Isolement de la chromatine
- Ajouter 1,2 ml de tampon COSMIC (20 mM Tris-HCl [pH 8,1], EDTA 2 mM, NaCl 150 mM, 1% de Triton-X100, 0,1% de SDS) et la pipette de haut en bas plusieurs fois pour remettre en suspension le culot de cellules pour chaque échantillon dans 1,2 ml de tampon cosmique.
- Ajouter 133 ul de chacun des 100 mM de fluorure de phénylméthylsulfonyle (PMSF), 100 mM de benzamidine, pepstatine et 150 uM inhibiteurs de la protéase frais jusqu'à une concentration finale de 1 mM de PMSF et de benzamidine et 1,5 pM pour la pepstatine. Solution scission de lysat cellulaire en deux ambre 1,7 ml microtubes.
- Soniquer avec sonicateur 35 min (10 sec sur 10 sec éteint, 60% de la puissance) la fragmentation du génome entre 100 unend 500 pb.
- Maintenir le niveau de solution dans la chromatine microtube parallèle au niveau de l'eau dans le réservoir. Confirmez ce niveau par une inspection visuelle.
NOTE:. Vérifiez que l'ADN a été cisaillé avec un gel agarose à 1,5% 55 - Optimiser le temps de traitement par ultrasons de manière empirique. Utilisez une quantité minimale de la glace dans le réservoir pour refroidir les échantillons, et d'assurer la glace ne se mêle pas entre les échantillons et la corne de tasse.
- Maintenir le niveau de solution dans la chromatine microtube parallèle au niveau de l'eau dans le réservoir. Confirmez ce niveau par une inspection visuelle.
- Centrifugeuse l'échantillon de 12.000 xg 10 min. Enregistrer la solution aqueuse qui contient de la chromatine soluble en le transférant à un nouveau tube à centrifuger ambre avec une pipette. Jeter le culot.
- Transférer 110 pi (10%) de l'échantillon à un nouveau tube et l'étiqueter ADN entrée. Conserver à -80 ° C. Enregistrez le reste de l'échantillon de la chromatine sur la glace pour une utilisation dans l'étape 3.3.
3. La capture de ligand ADN Crosslinks
- Utiliser une pipette pour distribuer 60 pi streptavidin-billes magnétiques revêtues dans un tube de microcentrifugation. Ajouter 1 ml de tampon COSMIC et mélanger sur un min nutator 5 à température ambiante. Placez billes magnétiques sur la séparation magnétique en rack 2 min pour capturer perles. Retirer tampon cosmique avec une pipette.
NOTE: Ne pas laisser les perles se dessécher. Procéder immédiatement à l'étape suivante. - Ajouter échantillon chromatine (~ 1 ml) à des billes (60 pi) et remettre les perles. Incuber chromatine avec des billes magnétiques revêtues de streptavidine au moins 4 h sur une rotation, mélangeur à bascule à 4 ° C. Incuber les échantillons O / N, si désiré.
4. Isolement de l'ADN purifié par affinité
- Lavez perles avec 7 intervalle de minutes à température ambiante avec les tampons de lavage suivantes pour supprimer les interactions non spécifiques. Utilisez un rack de séparation magnétique pour capturer perles après chaque lavage. Remettre en suspension les perles après chaque changement de tampon de lavage.
- Préparer tampons de lavage à l'eau déminéralisée distillée et le filtre (0,2 um) avant utilisation. Tampons de lavage de magasin 1 et 2 à 4 ° C pour slusieurs mois. Préparer le tampon de lavage 3 frais chaque jour. Ajouter et supprimer des tampons de lavage de l'échantillon avec une pipette. Pour 2 et 4, ajouter 1 et 3 (5 mM), respectivement, dans les lavages. Les échantillons peuvent en outre être lavées deux fois avec du tampon COSMIC (une fois 12 heures, une fois 4 heures) avant les lavages ci-dessous.
- Laver une fois avec du tampon de lavage 1 (10 mM Tris-Cl [pH 8,0], EDTA 1 mM, 3% de SDS). Laver une fois avec du tampon de lavage 2 (10 mM Tris-Cl [pH 8,0], 250 mM de LiCl, 1 mM d'EDTA, 0,5% de NP40, du désoxycholate de sodium à 1%). Laver deux fois avec du tampon de lavage 3 (4 M d'urée, 10 mM de Tris-Cl [pH 7,5], EDTA 1 mM, 0,1% de NP-40). Laver deux fois avec du Tris EDTA (TE) du tampon (10 mM Tris-Cl [pH 8,0], EDTA 1 mM).
- Remettre en suspension les billes dans du tampon TE 200 pi avec une pipette. Cet échantillon de l'ADN capturé est appelé (AP) de l'ADN purifié par affinité.
- Compléter l'ADN d'entrée et AP 10x Crosslink Reprise tampon (100 mM Tris-Cl [pH 8,0], 1 M KOH, 4 mMEDTA) à 1x concentration finale. 56,57 incuber 30 min à 90 ° C.
REMARQUE: 254 nm irradiation UV peut également être utilisé pour inverser la reticulation psoralène, 58 mais cyclobutyle dimères de pyrimidine peuvent être formés à partir d'une irradiation à cette longueur d'onde et d'interférer avec le traitement en aval de l'ADN. - Pour AP ADN, placer le tube à centrifuger contenant l'échantillon sur la séparation magnétique en rack 2 min à température ambiante et en isolant liquide (ADN) avec une pipette. Transférer l'ADN AP (qui a été libéré par les perles) dans un nouveau tube à centrifuger ambre.
NOTE: L'ADN désiré est AP dans le liquide, plus attaché aux billes. - Neutraliser entrée AP et de l'ADN avec du HCl concentré à pH 7 par addition d'environ 1 ul de HCl 6 N par 100 ul de l'échantillon avec une pipette. Confirmer les échantillons sont neutralisés en ajoutant ~ 0,5 pi sur du papier pH avec une pipette. ATTENTION: HCl concentré est un acide corrosif forte. Poignée selon votre institution217; s lignes directrices pour l'équipement de protection individuelle approprié.
- Ajouter la RNase A (100 mg / ml) à 0,2 pg / pl de concentration finale et à la fois l'entrée AP de l'ADN. Incuber 1 heure à 37 ° C. Ajouter à la protéinase K (20 mg / ml) à 0,2 pg / pl de concentration finale et à la fois l'entrée AP de l'ADN, ajouter. Incuber 1 heure à 55 ° C.
- Purifier l'ADN avec un kit colonne d'ADN de nettoyage (voir la liste des matériaux). 55 Elute ADN dans 58 ul d'ADN de qualité H 2 O. Échantillons d'entrée de magasin et AP à -20 ou -80 ° C
- Analyser AP ADN par réaction quantitative en chaîne par polymerase (PCR quantitative) avec des amorces spécifiques de locus, et / ou par séquençage de nouvelle génération. Pour qPCR, en utilisant 2 ul ADN AP par locus avec les paramètres suivants: 1 cycle de 95 ° C 10 min et 40 cycles de 95 ° C 20 sec, 54 ° C ou 56 ° C 20 sec, 72 ° C 40 sec.
REMARQUE: La température de recuit doit être modifiée en fonction de la température de fusion des paires d'amorces utilisées. Sélectionnez une tempéra de recuitre qui minimise le cycle de quantification sans amplification non spécifique.
REMARQUE: Pour l'analyse par séquençage de prochaine génération, viser au moins 10 millions mappé lit. Le nombre de lectures mappé peut être augmentée en augmentant la quantité d'ADN AP et en combinant moins d'échantillons dans une course pour le séquençage. Plus lectures améliorer la sensibilité. Ensembles standard pour l'analyse ChIP-seq (par exemple, Homère, 59 MACS, 60 et 61 SPP) travaillent avec des données COSMIC-seq. Comme avec d'autres méthodes qui reposent sur le séquençage de prochaine génération, y compris le séquençage du génome et Chip-Seq, régions répétitives créer des ambiguïtés dans l'alignement et l'assemblage et restent donc un défi technique.
Résultats
Pour tenir compte des non-uniforme génome fragmentation et d'autres variables, l'ADN purifié doit toujours être normalisé contre une référence de l'ADN d'entrée. Des amorces spécifiques à un locus d'intérêt peuvent être utilisés. Il est utile d'analyser aussi un lieu où ne devrait pas se lier à la molécule, en tant que témoin négatif. Nous voyons une augmentation de> 100 fois en occupation polyamide lors de l'irradiation avec de la lumière de 365 nm (Figure 2).
Discussion
One of the primary challenges with conventional ChIP is the identification of suitable antibodies. ChIP depends heavily upon the quality of the antibody, and most commercial antibodies are unacceptable for ChIP. In fact, the Encyclopedia of DNA Elements (ENCODE) consortium found only 20% of commercial antibodies to be suitable for ChIP assays.50 With COSMIC, antibodies are replaced by streptavidin. Because polyamides are functionalized with biotin, streptavidin is used in place of an antibody to capture polyam...
Déclarations de divulgation
A.Z.A. is the sole proprietor of Vista Motif, LLC and WINStep Forward.
Remerciements
We thank members of the Ansari lab and Prof. Parameswaran Ramanathan for helpful discussions. This work was supported by NIH grants CA133508 and HL099773, the H. I. Romnes faculty fellowship, and the W. M. Keck Medical Research Award to A.Z.A. G.S.E. was supported by a Peterson Fellowship from the Department of Biochemistry and Molecular Biosciences Training Grant NIH T32 GM07215. A.E. was supported by the Morgridge Graduate Fellowship and the Stem Cell and Regenerative Medicine Center Fellowship, and D.B. was supported by the NSEC grant from NSF.
matériels
| Name | Company | Catalog Number | Comments |
| Phenylmethylsulfonyl fluoride (PMSF) | any source | ||
| Benzamidine | any source | ||
| Pepstatin | any source | ||
| Proteinase K | any source | ||
| Dynabeads MyOne Streptavidin C1 | Life Technologies | 65001 | |
| PBS, pH 7.4 | Life Technologies | 10010-023 | Other sources can be used |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | We have tried other manufacturers of DNA columns with success. |
| TruSeq ChIP Sample Prep Kit | Illumina | IP-202-1012 | This Kit can be used to prepare COSMIC DNA for next-generation sequencing |
| Matrigel Basement Membrane Matrix | BD Biosciences | 356231 | Used to coat plates in order to grow H1 ESCs |
| pH paper | any source | ||
| amber microcentrifuge tubes | any source | ||
| microcentrifuge tubes | any source | ||
| pyrex filter | any source | Pyrex baking dishes are suitable | |
| qPCR master mix | any source | ||
| RNase | any source | ||
| HCl (6 N) | any source | ||
| 10-cm tissue culture dishes | any source | ||
| Serological pipettes | any source | ||
| Pasteur pipettes | any source | ||
| Pipette tips | any source | ||
| 15-ml conical tubes | any source | ||
| centrifuge | any source | ||
| microcentrifuge | any source | ||
| nutator | any source | ||
| Magnetic separation rack | any source | ||
| UV source | CalSun | B001BH0A1A | Other UV sources can be used, but crosslinking time must be optimized empirically |
| Misonix Sonicator | Qsonica | S4000 with 431C1 cup horn | Other sonicators can be used, but sonication conditions must be optimized empirically |
| Humidified CO2 incubator | any source | ||
| Biological safety cabinet with vacuum outlet | any source |
Références
- Lee, T. I., Young, R. A. Transcriptional Regulation and Its Misregulation in Disease. Cell. 152, 1237-1251 (2013).
- Vaquerizas, J. M., Kummerfeld, S. K., Teichmann, S. A., Luscombe, N. M. A census of human transcription factors: function, expression and evolution. Nat Rev Genet. 10, 252-263 (2009).
- Meier, J. L., Yu, A. S., Korf, I., Segal, D. J., Dervan, P. B. Guiding the Design of Synthetic DNA-Binding Molecules with Massively Parallel Sequencing. J. Am. Chem. Soc. 134, 17814-17822 (2012).
- Dervan, P. B. Molecular recognition of DNA by small molecules. Bioorg. Med. Chem. 9, 2215-2235 (2001).
- Wemmer, D. E., Dervan, P. B. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 7, 355-361 (1997).
- Eguchi, A., Lee, G. O., Wan, F., Erwin, G. S., Ansari, A. Z. Controlling gene networks and cell fate with precision-targeted DNA-binding proteins and small-molecule-based genome readers. Biochem. J. 462, 397-413 (2014).
- Mrksich, M., et al. Antiparallel side-by-side dimeric motif for sequence-specific recognition in the minor groove of DNA by the designed peptide 1-methylimidazole-2-carboxamide netropsin. Proc Natl Acad Sci U S A. 89, 7586-7590 (1992).
- Edayathumangalam, R. S., Weyermann, P., Gottesfeld, J. M., Dervan, P. B., Luger, K. Molecular recognition of the nucleosomal "supergroove". Proc Natl Acad Sci U S A. 101, 6864-6869 (2004).
- Suto, R. K., et al. Crystal structures of nucleosome core particles in complex with minor groove DNA-binding ligands. J. Mol. Biol. 326, 371-380 (2003).
- Gottesfeld, J. M., et al. Sequence-specific Recognition of DNA in the Nucleosome by Pyrrole-Imidazole Polyamides. J. Mol. Biol. 309, 615-629 (2001).
- Chenoweth, D. M., Dervan, P. B. Structural Basis for Cyclic Py-Im Polyamide Allosteric Inhibition of Nuclear Receptor Binding. J. Am. Chem. Soc. 132, 14521-14529 (2010).
- Raskatov, J. A., et al. Modulation of NF-κB-dependent gene transcription using programmable DNA minor groove binders. Proc. Natl. Acad. Sci. U.S.A. 109, 1023-1028 (2012).
- Yang, F., et al. Antitumor activity of a pyrrole-imidazole polyamide. Proc. Natl. Acad. Sci. U.S.A. 110, 1863-1868 (2013).
- Mapp, A. K., Ansari, A. Z., Ptashne, M., Dervan, P. B. Activation of gene expression by small molecule transcription factors. Proc. Natl. Acad. Sci. U.S.A. 97, 3930-3935 (2000).
- Ansari, A. Z., Mapp, A. K., Nguyen, D. H., Dervan, P. B., Ptashne, M. Towards a minimal motif for artificial transcriptional activators. Chem. Biol. 8, 583-592 (2001).
- Arora, P. S., Ansari, A. Z., Best, T. P., Ptashne, M., Dervan, P. B. Design of artificial transcriptional activators with rigid poly-L-proline linkers. J. Am. Chem. Soc. 124, 13067-13071 (2002).
- Nickols, N. G., Jacobs, C. S., Farkas, M. E., Dervan, P. B. Modulating Hypoxia-Inducible Transcription by Disrupting the HIF-1–DNA Interface. ACS Chemical Biology. 2, 561-571 (2007).
- Pandian, G. N., et al. A synthetic small molecule for rapid induction of multiple pluripotency genes in mouse embryonic fibroblasts. Sci. Rep. 2, 544 (2012).
- Pandian, G. N., et al. Synthetic Small Molecules for Epigenetic Activation of Pluripotency Genes in Mouse Embryonic Fibroblasts. Chem Bio Chem. 12, 2822-2828 (2011).
- He, G., et al. Binding studies of a large antiviral polyamide to a natural HPV sequence. Biochimie. 102, 83-91 (2014).
- Edwards, T. G., Vidmar, T. J., Koeller, K., Bashkin, J. K., Fisher, C. DNA Damage Repair Genes Controlling Human Papillomavirus (HPV) Episome Levels under Conditions of Stability and Extreme Instability. PLoS ONE. 8, e75406 (2013).
- Edwards, T. G., Helmus, M. J., Koeller, K., Bashkin, J. K., Fisher, C. HPV Episome Stability is Reduced by Aphidicolin and Controlled by DNA Damage Response Pathways. Journal of Virology. , (2013).
- Edwards, T. G., et al. HPV episome levels are potently decreased by pyrrole-imidazole polyamides. Antiviral Res. 91, 177-186 (2011).
- Dickinson, L. A., et al. Inhibition of RNA polymerase II transcription in human cells by synthetic DNA-binding ligands. Proc. Natl. Acad. Sci. U.S.A. 95, 12890-12895 (1998).
- Dickinson, L. A., et al. Arresting Cancer Proliferation by Small-Molecule Gene Regulation. Chem. Biol. 11, 1583-1594 (2004).
- Nickols, N. G., et al. Activity of a Py–Im Polyamide Targeted to the Estrogen Response Element. Molecular Cancer Therapeutics. 12, 675-684 (2013).
- Raskatov, J. A., Puckett, J. W., Dervan, P. B. A C-14 labeled Py–Im polyamide localizes to a subcutaneous prostate cancer tumor. Bioorg. Med. Chem. 22, 4371-4375 (2014).
- Jespersen, C., et al. Chromatin structure determines accessibility of a hairpin polyamide–chlorambucil conjugate at histone H4 genes in pancreatic cancer cells. Bioorg. Med. Chem. Lett. 22, 4068-4071 (2012).
- Chou, C. J., et al. Small molecules targeting histone H4 as potential therapeutics for chronic myelogenous leukemia. Molecular Cancer Therapeutics. 7, 769-778 (2008).
- Nickols, N. G., Dervan, P. B. Suppression of androgen receptor-mediated gene expression by a sequence-specific DNA-binding polyamide. Proc. Natl. Acad. Sci. U.S.A. 104, 10418-10423 (2007).
- Minoshima, M., Bando, T., Sasaki, S., Fujimoto, J., Sugiyama, H. Pyrrole-imidazole hairpin polyamides with high affinity at 5CGCG3 DNA sequence; influence of cytosine methylation on binding. Nucleic Acids Res. 36, 2889-2894 (2008).
- Warren, C. L., et al. Fabrication of duplex DNA microarrays incorporating methyl-5-cytosine. Lab on a Chip. 12, 376-380 (2012).
- Dudouet, B., et al. Accessibility of nuclear chromatin by DNA binding polyamides. Chem. Biol. 10, 859-867 (2003).
- Carlson, C. D., et al. Specificity landscapes of DNA binding molecules elucidate biological function. Proc. Natl. Acad. Sci. U.S.A. 107, 4544-4549 (2010).
- Warren, C. L., et al. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. U.S.A. 103, 867-872 (2006).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z., Voigt, C. Chapter One - Sequence-Specificity and Energy Landscapes of DNA-Binding Molecules. Methods Enzymol. 497, 3-30 (2011).
- Puckett, J. W., et al. Quantitative microarray profiling of DNA-binding molecules. J. Am. Chem. Soc. 129, 12310-12319 (2007).
- Keles, S., Warren, C. L., Carlson, C. D., Ansari, A. Z. CSI-Tree: a regression tree approach for modeling binding properties of DNA-binding molecules based on cognate site identification (CSI) data. Nucleic Acids Res. 36, 3171-3184 (2008).
- Hauschild, K. E., Stover, J. S., Boger, D. L., Ansari, A. Z. CSI-FID: High throughput label-free detection of DNA binding molecules. Bioorg. Med. Chem. Lett. 19, 3779-3782 (2009).
- Lee, M., Roldan, M. C., Haskell, M. K., McAdam, S. R., Hartley, J. A. . In vitro Photoinduced Cytotoxicity and DNA Binding Properties of Psoralen and Coumarin Conjugates of Netropsin Analogs: DNA Sequence-Directed Alkylation and Cross-Link. 37, 1208-1213 (1994).
- Wurtz, N. R., Dervan, P. B. Sequence specific alkylation of DNA by hairpin pyrrole–imidazole polyamide conjugates. Chem. Biol. 7, 153-161 (2000).
- Tung, S. -. Y., Hong, J. -. Y., Walz, T., Moazed, D., Liou, G. -. G. Chromatin affinity-precipitation using a small metabolic molecule: its application to analysis of O-acetyl-ADP-ribose. Cell. Mol. Life Sci. 69, 641-650 (2012).
- Rodriguez, R., Miller, K. M. Unravelling the genomic targets of small molecules using high-throughput sequencing. Nat Rev Genet. 15, 783-796 (2014).
- Guan, L., Disney, M. D. Covalent Small-Molecule–RNA Complex Formation Enables Cellular Profiling of Small-Molecule–RNA Interactions. Angew. Chem. Int. Ed. 52, 10010-10013 (2013).
- White, J. D., et al. Picazoplatin, an Azide-Containing Platinum(II) Derivative for Target Analysis by Click Chemistry. J. Am. Chem. Soc. 135, 11680-11683 (2013).
- Rodriguez, R., et al. Small-molecule–induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol. 8, 301-310 (2012).
- Bando, T., Sugiyama, H. Synthesis and Biological Properties of Sequence-Specific DNA-Alkylating Pyrrole−Imidazole Polyamides. Acc. Chem. Res. 39, 935-944 (2006).
- Anders, L., et al. Genome-wide localization of small molecules. Nat. Biotechnol. 32, 92-96 (2014).
- Jin, C., et al. Chem-seq permits identification of genomic targets of drugs against androgen receptor regulation selected by functional phenotypic screens. Proc. Natl. Acad. Sci. U.S.A. 111, 9235-9240 (2014).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
- Erwin, G. S., Bhimsaria, D., Eguchi, A., Ansari, A. Z. Mapping Polyamide–DNA Interactions in Human Cells Reveals a New Design Strategy for Effective Targeting of Genomic Sites. Angew. Chem. Int. Ed. 53, 10124-10128 (2014).
- Hyde, J. E., Hearst, J. E. Binding of psoralen derivatives to DNA and chromatin: influence of the ionic environment on dark binding and photoreactivity. Biochemistry. 17, 1251-1257 (1978).
- Erwin, G. S., Bhimsaria, D., Rodríguez-Martínez, J. A., Grieshop, M. P., Ansari, A. Z. Genome-wide localization of polyamide-based genome readers reveals sequence-based binding to repressive heterochromatin. In preparation. , (2015).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Meth. 8, 424-429 (2011).
- Deliard, S., Zhao, J., Xia, Q., Grant, S. F. A. Generation of High Quality Chromatin Immunoprecipitation DNA Template for High-throughput Sequencing (ChIP-seq). J Vis Exp. (74), e50286 (2013).
- Shi, Y. B., Spielmann, H. P., Hearst, J. E. Base-catalyzed reversal of a psoralen-DNA cross-link. Biochemistry. 27, 5174-5178 (1988).
- Kumaresan, K. R., Hang, B., Lambert, M. W. Human Endonucleolytic Incision of DNA 3′ and 5′ to a Site-directed Psoralen Monoadduct and Interstrand. J. Biol. Chem. 270, 30709-30716 (1995).
- Cimino, G. D., Shi, Y. B., Hearst, J. E. Wavelength dependence for the photoreversal of a psoralen-DNA crosslink. Biochemistry. 25, 3013-3020 (1986).
- Heinz, S., et al. Simple Combinations of Lineage-Determining Transcription Factors Prime cis-Regulatory Elements Required for Macrophage and B Cell Identities. Mol. Cell. 38, 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9, R137 (2008).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nat Biotech. 26, 1351-1359 (2008).
- Diamandis, E. P., Christopoulos, T. K. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin. Chem. 37, 625-636 (1991).
- Martinson, H. G., True, R. J. On the mechanism of nucleosome unfolding. Biochemistry. 18, 1089-1094 (1979).
- Gloss, L. M., Placek, B. J. The Effect of Salts on the Stability of the H2A−H2B Histone Dimer. Biochemistry. 41, 14951-14959 (2002).
- Jackson, V. Formaldehyde Cross-Linking for Studying Nucleosomal Dynamics. Methods. 17, 125-139 (1999).
- Kasinathan, S., Orsi, G. A., Zentner, G. E., Ahmad, K., Henikoff, S. High-resolution mapping of transcription factor binding sites on native chromatin. Nat Meth. 11, 203-209 (2014).
- Teytelman, L., Thurtle, D. M., Rine, J., van Oudenaarden, A. Highly expressed loci are vulnerable to misleading ChIP localization of multiple unrelated proteins. Proc. Natl. Acad. Sci. U.S.A. 110, 18602-18607 (2013).
- . Phantompeakqualtools home page Available from: https://www.encodeproject.org/software/phantompeakqualtools/ (2010)
- Wang, D., Lippard, S. J. Cellular processing of platinum anticancer drugs. Nature Reviews Drug Discovery. 4, 307-320 (2005).
- Hurley, L. H. DNA and its associated processes as targets for cancer therapy. Nat. Rev. Cancer. 2, 188-200 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon