Method Article
Le séquençage profond impartiale de l'ARN virus à partir d'échantillons cliniques
Dans cet article
Résumé
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Résumé
Nous présentons ici un ARN protocole de séquençage de nouvelle génération qui permet de novo assemblées et intra-hôte variants appels de génomes viraux recueillies à partir de sources cliniques et biologiques. La méthode est impartiale et universelle; il utilise des amorces aléatoires pour la synthèse d'ADNc et ne nécessite aucune connaissance préalable du contenu de la séquence virale. Avant la construction de la banque, la digestion sélective à base de RNase H est utilisée pour appauvrir l'ARN non désiré - y compris le poly (rA) porteuse et l'ARN ribosomal - à partir de l'échantillon d'ARN viral. déplétion sélective améliore à la fois la qualité des données et le nombre de lit unique, dans des bibliothèques virales de séquençage de l'ARN. En outre, l'étape a 'tagmentation' à base de transposase est utilisée dans le protocole, car il réduit le temps global de la construction de la bibliothèque. Le protocole a permis le séquençage rapide et profonde de plus de 600 échantillons, y compris le virus Lassa et Ebola collections de sang et de tissus isolats et est largement applicable à d'autres études de génomique microbienne.
Introduction
séquençage de la prochaine génération de virus à partir de sources cliniques peut informer la transmission et l'épidémiologie des infections, ainsi que le soutien de l'aide nouvelle de diagnostic, les vaccins et le développement thérapeutique. synthèse d'ADNc en utilisant des amorces aléatoires a permis la détection et l' assemblage des génomes de divergent, co-infectant ou même de nouveaux virus 1,2. Comme avec d'autres méthodes impartiales, contaminants indésirables occupent beaucoup séquençage lit et un impact négatif sur les résultats de séquençage. Hôte et poly (rA) ARN porteur sont des contaminants présents dans de nombreuses collections virales existantes de l'échantillon.
Le protocole décrit un moyen efficace et rentable de profondes génomes de virus à ARN de séquençage basé sur l'ARN-seq impartiale totale. Le procédé utilise une RNase H déplétion sélective étape 3 pour supprimer ribosomal hôte indésirable et de l' ARN porteur. Déplétion sélective enrichit pour le contenu viral (figure 1) et améliore la qualité globale des données de séquençage(Figure 2) à partir d' échantillons cliniques. En outre, tagmentation est appliqué le protocole, car elle réduit considérablement le temps de construction de bibliothèques. Ces méthodes ont été utilisées pour générer rapidement de grands ensembles de données d'Ebola et Lassa virus génomes 2,4,5 et peuvent être utilisés pour étudier un large éventail de virus à ARN. Enfin, l'approche ne se limite pas à des échantillons humains; l'utilité de l' épuisement sélectif a été démontrée sur des échantillons de tissus prélevés à partir des rongeurs infectés par Lassa et des modèles de maladies de primate non humain 5,6.

Figure 1. L' ARN total du contenu Reflète Enrichissement de Lassa Virus Content Utilisation sélective Épuisement. À partir du contenu global (entrée d'ARN) et l' enrichissement des virus Lassa uniques (LASV) lit (contenu de la bibliothèque) sur ARNr déplétion de neuf isolats cliniques différents. Ce chiffre a été modifié depuis 6 . S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
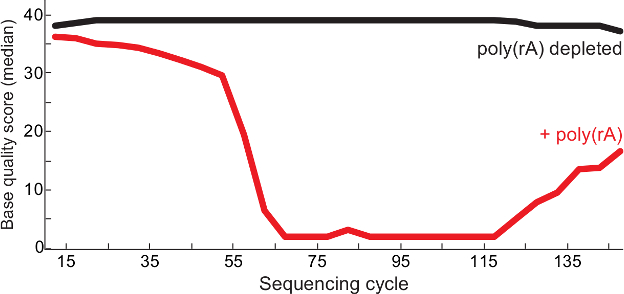
Figure 2. Qualité supérieure Séquençage Après l' ARN transporteur Épuisement. Les qualités médians de base par cycle de séquençage de poly (rA) bibliothèques de virus contaminés au Lassa (rouge) et le contrôle (aucun transporteur observé dans la bibliothèque, noir) du QC rapport 13. Les deux lu 1 et lu 2 de fin appariés lectures sont fusionnés dans le fichier de BAM de la bibliothèque et les scores de qualité sont présentés à chaque base. Ce chiffre a été modifié depuis 6. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
Le détails du protocole construction de bibliothèques d'ARN-seq virale directement à partir extraitL'ARN collecté à partir d'échantillons cliniques et biologiques. Pour assurer la sécurité personnelle, tous les échantillons viraux sérum, le plasma et les tissus doivent être inactivés dans des tampons appropriés avant l'extraction de l'ARN. Dans certains kits d'inactivation et d'extraction, le transporteur poly (rA) ARN est inclus; ce sera supprimée au cours de la RNase H étape initiale de déplétion sélective. Sur la base de la récupération complète, la concentration attendue de l'ARN porteur est de 100 ng / pl. Dans le protocole, 110 ng / ul oligo dT ARN (concentration de porteurs de 1,1x) est utilisé pour l'épuisement. Si le poly (rA) porteur ne sont pas présents dans l'échantillon, puis l'oligo (dT) ne doit pas être ajouté avant l'épuisement.
Le protocole suivant a été conçu pour 24 réactions dans le format de plaque PCR (jusqu'à un volume de 250 ul). Une version antérieure de ce protocole a été rapporté dans Matranga, et al. 6.
Protocole
déclaration éthique: les patients atteints de fièvre de Lassa ont été recrutés pour cette étude en utilisant des protocoles approuvés par les comités de sujets humains à l'Université Tulane, l'Université de Harvard, Broad Institute, Irrua Spécialiste CHU (ISTH), Hôpital gouvernement Kenema (HGK), l'Etat d'Oyo Ministère de la Santé, Ibadan , le Nigeria et la Sierra Leone Ministère de la Santé. Tous les patients ont été traités avec une norme de soins similaires et ont été offerts le médicament Ribavirin, si oui ou non ils ont décidé de participer à l'étude. Pour la fièvre Lassa (LF) patients, le traitement par Ribavirine a suivi les lignes directrices recommandées actuellement et a été généralement offerte dès LF a été fortement suspectée.
En raison de la grave épidémie d'Ebola virus de la maladie (EVD), les patients ne pouvaient pas être consenti par nos protocoles standards. Au lieu d'utiliser des échantillons cliniques en excès provenant de patients EVD a été évalué et approuvé par Institutional Review Board en Sierra Leone et à l'Université de Harvard. Le OffiBureau du Comité Sierra Leone éthique et d'examen scientifique, le Leone Ministère Sierra de la Santé et de l'assainissement, et le Comité de Harvard sur l'utilisation des sujets humains ont accordé une dispense du consentement à la séquence et de faire des séquences virales publiquement disponibles obtenues à partir d'échantillons de patients et de contact recueillies au cours de l'épidémie d'Ebola en Sierra Leone. Ces organes ont également l'autorisation d'utiliser les données cliniques et épidémiologiques pour les échantillons anonymisées recueillies auprès de tous les patients EVD suspects recevant des soins au cours de la réponse aux épidémies. La Sierra Leone Ministère de la Santé et de l'assainissement a également approuvé les envois d'échantillons non infectieux, non-biologiques de la Sierra Leone au Broad Institute et de l'Université de Harvard pour les études génomiques d'échantillons d'épidémie.
1. DNase-traitement de l'ARN de l'échantillon (jusqu'à 55 pi Extrait ARN total, ~ 4 h)
- Mettre en place la réaction de DNase dans une plaque à 96 puits PCR sur la glace dans une enceinte de sécurité biologique tel que décrit dans le tableau 1 , L'étape 1.1 (volume total, 70 pl / puits). Note: Un mélange maître peut être préparé.
- Vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min.
- Incuber à 37 ° C pendant 30 min.
- Nettoyage à l'aide d'ARN en phase solide réversible Immobilisation (SPRI) perles.
- des billes d'ARN réchauffer à température ambiante pendant 30 min.
- Secouer doucement perles d'ARN bouteille pour remettre en suspension les particules magnétiques qui ont pu se déposer. Ajouter le volume de 1,8x (126 ul) de billes d'ARN de l'ARN DNase traitée (70 pi), mélanger à la pipette 10 fois et incuber pendant 5 minutes à température ambiante (volume total dans le puits, 196 ul).
- Placez le mélange sur la station magnétique. Attendez que la solution pour effacer (5 - 10 min).
- Retirer solution compensés, sur la station par pipette et défausse. Alors que sur la station, laver les perles en couvrant culot avec 70% d'éthanol et incuber pendant 1 min. Retirer l'éthanol à la pipette et défausse. Répétez l'opération pour un total de deux lavages.
Remarque: L'utilisation précise de 70% fraîchement préparé etHanol est critique, car un pourcentage plus élevé se traduira par lavage inefficace des molécules de plus petite taille, tandis que <70% d' éthanol pourrait entraîner une perte de l' échantillon 7. - Gardez la plaque sur la station et de laisser ouverte sécher à l'air. Remarque: Assurez-vous de permettre aux billes de sécher complètement jusqu'à ce que les perles commencent à se fissurer.
- Ajouter 55 ul d'eau sans nucléase à la plaque pour éluer l'ARN. Retirer la plaque de la station de mélanger les perles et de l'eau par pipetage à fond. Remarque: Vous pouvez également utiliser moins d'eau (≤ 10 pi) afin de concentrer l'ARN total.
- Placez la plaque arrière de la station. Attendez jusqu'à ce que la solution autorise à transférer à la pipette dans un nouveau tube à bouchon à vis pour le stockage à long terme (-80 ° C). Placez 5 ARN ul dans la nouvelle-96 puits plaque PCR pour épuisement (étape. 2.4).
- Facultatif:. Enregistrer et diluer 1 ul dans 19 pi d' eau (1:20) pour qRT-PCR de l' ARNr (par exemple, 18S, 28S) (tableau 2) et les marqueurs viraux 5
2. déplétion sélective des ribosomique et Carrier ARN à partir d'ARN viral Sample (~ 4 h)
- Faire l' hybridation 5x et 10x RNase H tampons de réaction, et de l' eau sans nucléase avec linéaire porteuse d'acrylamide comme décrit dans le tableau 1.
- Mettre en place la réaction d'hybridation avec l' ARN en combinant les oligos ARNr de déplétion (tableau 3) et d' oligo (dT) sur de la glace dans une plaque à 96 puits PCR comme décrit dans le Tableau 1.
Note: Un mélange maître peut être préparé. 50 femtogrammes (fg) d'un ARN de synthèse uniques (ERCCs 8) peuvent être ajoutés pour le suivi à la fois le processus de séquençage viral et de l' indice de lire la contamination croisée.- Vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min.
Incuber à 95 ° C pendant 2 min, rampe lente à 45 ° C à -0,1 ° C par seconde. Pause thermocycleur à 45 ° C.
- Vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min.
- Mettre en place RNase H mélange réactionnel sur la glace comme décritd dans le tableau 1, puis le préchauffage à 45 ° C pendant 2 min. Note: Un mélange maître peut être préparé.
- Ajouter le pré-chauffé RNase H mélange pour la réaction d'hybridation en plaque tout en maintenant la plaque dans le thermocycleur à 45 ° C.
- Bien mélanger en douceur pipetage 6 - 8 fois. Incuber à 45 ° C pendant encore 30 min. Placer sur la glace.
- Mettre en place la DNase mélange réactionnel sur la glace comme décrit dans le tableau 1 . Note: Un mélange maître peut être préparé.
- Ajouter à la réaction RNase H dans la plaque, vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min. Incuber à 37 ° C pendant 30 min.
- Arrêter la réaction de DNase en ajoutant 5 ul d'EDTA 0,5 M. Vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min.
- Nettoyage en utilisant des billes d'ARN (voir l'étape 1.3) en utilisant un volume de 1,8x (144 pi) de perles. Eluer dans 11 pi d'eau sans nucléase. Remarque: Pour le stockage à froid sans risque, entreposer épuisé RNA échantillons à -80 ° C O / N.
3. Synthèse d'ADNc (~ 6 h)
- Mix rRNA / ARN porteur appauvri avec des amorces aléatoires sur la glace dans une plaque à 96 puits PCR comme décrit dans le tableau 1, vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min.
- Chauffer le mélange à 70 ° C pendant 10 minutes dans un thermocycleur. Immédiatement après dénaturation thermique, placez l'ARN sur la glace pendant 1 - 5 min. Ne laissez pas l'ARN de se tenir (même sur la glace) pendant plus de 5 minutes avant le premier brin réaction.
- Mettre en place du premier brin mélange de réaction de synthèse sur la glace comme décrit dans le tableau 1.
Remarque: Un maître-mélange peut être préparé.- Ajouter à ARN / mélange amorce aléatoire dans la plaque, vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min. On incube à 22-25 ° C pendant 10 min.
- Incuber à 55 ° C dans un incubateur à air pendant 60 min. Placer la plaque sur la glace pour terminer la réaction. ne pase: L'utilisation d'un incubateur à air est recommandé de créer le réchauffement progressif de la première brin réaction au cours de laquelle les amorces hybrident et le premier brin commence à allonger.
- Mettre en place du second brin mélange de réaction de synthèse sur la glace comme décrit dans le tableau 1.
Remarque: Un maître-mélange peut être préparé.- Ajouter au premier brin réaction de synthèse dans la plaque, vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min. Incuber pendant 2 heures à 16 ° C (maintenir le couvercle à 25 ° C). Ne pas laisser la température remonter au-dessus de 16 ° C.
- Placer la plaque sur la glace, puis inactiver réaction en ajoutant 5 ul d'EDTA 0,5 M, mélanger doucement et soigneusement, puis centrifuger à 280 xg à température ambiante pendant 1 min.
- Nettoyage avec des perles d'ADN (voir l'étape 1.3 pour le protocole) en utilisant le volume de 1,8x (153 pi) de perles. Éluer dans 9 ul de tampon d'élution (EB). Enregistrer 1 pl pour la quantification. Utilisez 1 ng d'ADNc pour subsequeles étapes NT. Si la concentration d'ADNc est trop faible pour détecter, utiliser 4 pi d'ADNc pour tagmentation (voir étape 4.1).
- Pour le stockage à froid sûr, le stockage d'ADNc double brin à 4 ° CO / N ou -20 ° C pour une conservation à long terme.
4. Bibliothèque Préparation - Bibliothèque ADN Construction (~ 4 h)
- Transfert 4 pi d'ADNc à une plaque de 96 puits et d'économiser l'ADNc restant pour une deuxième tentative si nécessaire.
- Mettre en place la réaction de tagmentation sur la glace comme décrit dans le tableau 1.
Remarque: Un maître-mélange peut être préparé. Afin de réduire le coût global de fond et, le volume total de la réaction de tagmentation est réduite de 20 à 10 pl. Que l' ADNc est le facteur limitant, la quantité d'ATM (c. -à- transposome) utilisé dans la réaction est également réduite pour diminuer le nombre de sites d'intégration.- Ajouter tagmentation mélange à l'ADNc dans la plaque, vortex doucement et en profondeur et centrifuger à 280 xg (à température ambiante) pendant 1 min.Incuber à 55 ° C pendant 5 min, maintenir à 10 ° C.
- Une fois à 10 ° C, ajouter immédiatement 2,5 ul de tampon Neutraliser Tagment (NT) pour terminer la réaction. Mélanger par pipetage de haut en bas, puis centrifuger à 280 xg (à température ambiante) pendant 1 min.
- Incubation à température ambiante pendant 5 min.
- Mettre en place réaction d'amplification PCR sur de la glace comme décrit dans le tableau 1.
- Vortex doucement et en profondeur, puis centrifuger à 280 xg à température ambiante pendant 1 min.
- Effectuer une PCR sur un thermocycleur en utilisant les conditions décrites dans le tableau 1.
Note: 12 cycles de PCR sont proposés pour 1 ng d'ADNc tagmented; Cependant, les échantillons cliniques viraux ont souvent des quantités indétectables d'ADNc. Pour de faibles quantités d'ADNc (<1 ng), utiliser jusqu'à 18 cycles de PCR pour créer suffisamment de bibliothèque pour le séquençage.
- préparation Library - le nettoyage et la mise en commun pour le séquençage
- Amener l'échantillon jusqu'à 50 pi avec EB.
- Nettoyage avecperles d'ADN (voir l'étape 1.3 pour le protocole) en utilisant le volume de 0.6x (30 pi) de perles. Éluer dans 15 ul EB.
- Déterminer la concentration de bibliothèque (figure 3) en procédant à l' analyse des régions (150 à 1000 pb) en utilisant le logiciel bioanalyser 9, à l' exclusion des dimères d'amorces (~ 120 pb) à partir de l' analyse de la région. Remarque: Alternativement, qPCR peut être utilisée pour quantifier les bibliothèques 10.
- bibliothèques de piscine à la concentration molaire plus faible de 1 nM ou plus. Si la bibliothèque est inférieure à 1 nM, ajouter un petit volume de la bibliothèque à la piscine (~ volume 1x d'autres bibliothèques) pour capturer des informations de séquence de ces bibliothèques.
- Piscine de nettoyage avec des perles d'ADN de 0.7x décrites ci-dessus (voir étape 2). Éluer dans 15 ul EB. Note: Le volume des perles dépendra du volume final de la piscine.
- Analyser la piscine 9. Déterminer la concentration molaire en procédant à l' analyse de la région (150 à 1000 pb) 9. Remarque: Alternativement, qPCR peut être utilisée pour quantifier la piscine bibliothèque 10 </ Sup>.
- Séquenceur de charge à une concentration de la bibliothèque de 10 pM pour générer 101 pb, appariés-end lit double avec code à barres lit 11.
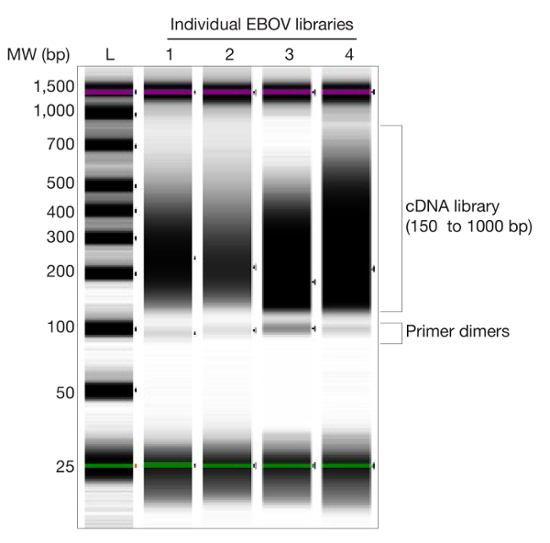
Figure 3. Les bibliothèques construites à partir d' Ebola Virus échantillons cliniques. Image Gel de 4 représentant le virus Ebola (EBOV) bibliothèques. Régions de bibliothèque et d' amorces dimères sont présentés. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
Le protocole décrit permet la génération de séquençage de haute qualité se lit de bas-entrée des échantillons d'ARN viral tout en enrichissant pour le contenu viral unique. Comme on le voit sur la figure 1, le protocole a enrichi le contenu unique du virus Lassa au moins cinq fois dans tous les échantillons ( par rapport aux témoins non appauvrie) avec au moins un million de copies d'ARNr 18S (~ 100 pg d'ARN total). De même, le succès de séquençage également en corrélation avec la quantité de virus dans un échantillon donné. QRT-PCR en utilisant comme substitut pour la quantité virale, des échantillons qui contenaient ~ 1000 ou plus virales copies du génome le plus souvent créé des ensembles complets (données non présentées). Par ailleurs, l' épuisement du poly (rA) porteuse réduit les séquences d'homopolymère de A et T dans les bibliothèques, ce qui entraîne des préparations plus propres et d' assurer une meilleure qualité de séquençage de lectures (figure 2). bibliothèques finales de faible entrée des échantillons cliniques viraux ont souvent une large longueur des fragments de 150 à 1000 pb(Figure 3).
Après le séquençage, pour réduire misidentification de l' échantillon et la diaphonie entre les bibliothèques dans un pool 12, seul indice lit avec un score de qualité de base de 25 (q25) et veiller à zéro inadéquations sont conservés pendant le processus de démultiplexage. Les génomes viraux sont assemblés à l' aide d' un pipeline de bio - informatique spécifique pour les virus divergentes 2,4-6. Ces outils sont disponibles à https://github.com/broadinstitute/viral-ngs ou par le biais des plates - formes de cloud commerciales 4.
| Etape 1.1: réaction de DNase | |
| Réactif | Volume par réaction (ul) |
| tampon 10x DNase | 7 |
| l'eau exempte de nucléase | 6 |
| ARN viral Extrait | 55 |
| DNase (2 U / & #181; l) | 2 |
| Volume total | 70 |
| Etape 2.1: tampon 5x Hybridation | |
| Réactif | Volume pour 1 ml (pi) |
| 5 M de NaCl | 200 |
| 1 M de Tris-HCl (pH 7,4) | 500 |
| l'eau exempte de nucléase | 300 |
| Volume total | 1000 |
| Etape 2.1: 10x tampon de réaction RNase H | |
| Réactif | Volume pour 1 ml (pi) |
| 5 M de NaCl | 200 |
| 1 M de Tris-HCl (pH 7,5) | 500 |
| 1 M de MgCl2 | 200 |
| l'eau exempte de nucléase | 500 |
| Volume total | 1000 |
| Etape 2.1: L' eau wacrylamide linéaire ith | |
| Réactif | Volume 1 ml de tampon (ul) |
| l'eau exempte de nucléase | 992 |
| acrylamide linéaire (5 mg / ml) | 8 |
| Volume total | 1000 |
| Etape 2.2: réaction d'hybridation pour déplétion sélective | |
| Réactif | Volume par réaction (ul) |
| 5x Hybridization Buffer | 2 |
| ARNr épuisement oligo- mélange (100 pM) | 1.22 |
| Oligo (d) T (550 ng / ul) | 1 |
| DNase traité l'ARN total | jusqu'à 5 |
| De Spike en ARN (Cette option est facultative) | 0,5 |
| Eau (avec linéaire acrylamide) | mettre à 10 au total |
| volu totalmoi | dix |
| Etape 2.3: RNase H réaction pour déplétion sélective | |
| Réactif | Volume par réaction (ul) |
| 10x RNase H Tampon de réaction | 2 |
| Eau (avec linéaire acrylamide) | 5 |
| Thermostable RNase H (5 U / pl) | 3 |
| Volume total | dix |
| Etape 2.4: DNase post-réaction déplétion sélective | |
| Réactif | Volume par réaction (ul) |
| 10x DNase Buffer | 7.5 |
| Eau (avec linéaire acrylamide) | 44,5 |
| inhibiteur de RNase (20 U / ul) | 1 |
| DNase RNase-I (2,72 U / ul) | 2 |
| Le volume total (avec la RNase H de réaction) | 75 |
| Etape 3.1: synthèse d'ADNc, amorce aléatoire hybridation | |
| Réactif | Volume par réaction (ul) |
| rRNA / ARN porteur appauvri | dix |
| 3 pg d'amorce aléatoire | 1 |
| Volume total | 11 |
| Etape 3.2: Premier volet réaction de synthèse d'ADNc | |
| Réactif | Volume (ul) |
| 5x First-Strand Reaction Buffer | 4 |
| 0,1 M DTT | 2 |
| 10 mM dNTP mix | 1 |
| inhibiteur de RNase (20 U / ul) | 1 |
| La transcriptase inverse (ajoutez dernier) | 1 |
| Le volume total (avec de l'ARN ci-dessus) | 20 |
| Etape 3.3: Deuxième volet réaction de synthèse d'ADNc | |
| Réactif | Volume (ul) |
| l'eau sans RNase | 43 |
| 10x Reaction Buffer Second-Strand | 8 |
| 10 mM dNTP mix | 3 |
| E. Coli DNA Ligase (10 U / pl) | 1 |
| E. coli DNA polymerase I (10 U / pl) | 4 |
| Coli RNase H (2 U / pl) | 1 |
| Le volume total (avec 1 er réaction de brin) | 80 |
| Etape 4.2: réaction de Tagmentation | |
| Réactif | Volume (ul) |
| Amplicon Tagment Mix (ATM) | 1 |
| Tampon ADN Tagment (TD) | 5 |
| Le volume total (avec de l'ADNc) | dix |
| Etape 4.3: réaction Library PCR | |
| Réactif | Volume (ul) |
| PCR Master Mix (NPM) | 7.5 |
| Indice 1 amorce (i7) | 2.5 |
| Indice 2 amorce (i5) | 2.5 |
| Le volume total (avec tagmented ADNc) | 25 |
| Étape 4.3.2: Les conditions de PCR Bibliothèque | |
| 72 ° C 3 min | |
| 95 ° C, 30 sec | |
| jusqu'à 18 cycles-10 sec à 95 ° C, 30 s à 55 ° C, 30 s à 72 ° C | |
| 72 ° C, 5 min | |
| 10 ° C, pour toujours | |
Tableau 1:. Réaction mise en place et les tampons étape par étape des tables avec des contenus de tous les tampons et les mélanges de réaction.
Tableau 2: qRT-PCR Amorces Sequences __gVirt_NP_NNS_NNPS<__ Amorces utilisées pour la mesure de l' hôte (18S) et virales (Ebola et Lassa) contenu.. 'KGH' Hospital Gouvernement Kenema en Sierra Leone, où les amorces d' Ebola ont été testés 2. 'Kulesh' est le chercheur qui a conçu le jeu d' amorces 14.
Tableau 3: ARN ribosomique (ARNr) Épuisement oligos 195 50 nucléotidiques de longues séquences complémentaires à l' ARNr humaine pour déplétion sélective étape 6.. S'il vous plaît cliquer ici pour télécharger ce fichier.
| Oligo Nom | Séquence (5 'à 3') |
| Ebola KGH FW | GTCGTTCCAACAATCGAGCG |
| Ebola KGH RV | CGTCCCGTAGCTTTRGCCAT |
| Ebola kulesh FW | TCTGACATGGATTACCACAAGATC |
| Ebola kulesh RV | GGATGACTCTTTGCCGAACAATC |
| Lassa SL FW | GTA AGC CCA GCD GYA AAB CC |
| Lassa SL RV | AAG CCA CAG AAA RCT GGS AGC A |
| 18S FW | TCCTTTAACGAGGATCCATTGG |
| 18S RV | CGAGCTTTTTAACTGCAGCAACT |
Discussion
L'approche décrite permet robuste, universel, séquençage rapide et a été utilisé pour le séquençage du virus Ebola au cours de la 2,4 2014 épidémie. Par couplage d'appauvrissement sélectif et la synthèse d'ADNc avec la construction de la bibliothèque de tagmentation, le temps de traitement global a été réduit par ~ 2 jours à partir des procédés de ligature adaptateur précédentes. Plus récemment, ce protocole a été employé par des collaborateurs internationaux et d' autres avec un grand succès 15,16 et sera déployé à des laboratoires en Afrique de l' Ouest pour soutenir les études et diagnostics 17 locales fondées sur la génomique recherche.
Le protocole décrit ici utilise des amorces aléatoires pour préparer l'ADNc pour les bibliothèques d'ARN-seq virales. Contrairement aux approches virales précédentes d'ARN-seq, il ne nécessite pas une connaissance a priori des données de séquence ou la conception d'amorce complexe et prend du temps pour un virus ou clade spécifique. Le procédé peut être appliqué à un échantillon d'ARN viral. Par exemple, il a été utilisé pour générer du contenu viral à la fois Ebolaet des échantillons de Lassa 6. Le protocole peut également être utilisé pour transcriptomique hôte, les projets de séquençage métagénomique et de découverte de l' agent pathogène 1.
Une étape critique du protocole est ciblé RNase H digestion, un haut-débit, méthode peu coûteuse pour éliminer porteuse parasite et de l'ARN hôte à partir d'échantillons viraux. L'étape de déplétion sélective du protocole utilise de nombreux composants et nécessite des compétences et de précision. temps et précautions supplémentaires doivent être prises lors de la configuration initiale.
Comme la plupart des échantillons de sérum et de plasma cliniques ont souvent très peu de matériel d'acide nucléique, la contamination et la perte de l'échantillon sont communs. Pour éviter ces problèmes, une attention particulière doit être prise lors de l'utilisation de ce protocole. Tout d'abord, l'ARN est très sensible à la dégradation; donc tous les domaines doivent être propres et exempts de nucléases. Deuxièmement, pour identifier les échantillons appropriés pour une utilisation dans ce protocole, les essais qRT-PCR pour l'ARN hôte et le virus devraient être utilisés pour la quantification 5,6 . Lorsque l'on compare l' entrée revient avec des résultats de séquençage du protocole, le succès de séquençage (c. -à- génération de données suffisantes pour l' assemblage viral complet) en corrélation avec les échantillons qui contenaient au moins 100 pg d' ARN total et 1.000 copies du virus. En troisième lieu, l'exposition à des sources d'acides nucléiques de l'environnement doit être évité. Le protocole décrit ici est fait dans une enceinte de sécurité biologique pour les précautions de sécurité et pour limiter les contaminants environnementaux. De plus, notre groupe et d' autres ont remarqué que les enzymes commerciales peuvent être une autre source d'acides nucléiques contaminants bactériens dans des échantillons d'entrée à basse 6,18. Utilisation d'un espace de travail propre (par exemple, hotte PCR, enceinte de sécurité biologique) et des contrôles négatifs (par exemple, l' eau ou du tampon) contribueront à atténuer et de suivre la contamination, respectivement. Pour les échantillons avec <100 pg d'ARN total, seul poly (rA) ARN porteur, non ARNr, devrait être épuisé pour garantir des résultats de séquençage de haute qualité tout en limitant la perte de matériel. Pour les trèsdes échantillons d'entrée faible, les méthodes d'amplification d' ADNc peut être plus approprié 19, bien que le poly (rA) support doit être éliminé avant la synthèse de l' ADNc.
L'épuisement de l' hôte ARNr pour enrichir le contenu viral dans les bibliothèques de séquençage et est applicable à la collecte d'échantillons différents , y compris le sérum ou le plasma, et de multiples types de tissus prélevés sur des rongeurs et des primates non humains 5,6. Dans les organismes non humains, les lectures alignant sur 28S est resté après l' épuisement, ce qui suggère 28S est moins conservée entre les humains et les autres espèces de 6,20. Lors de l' utilisation de cette méthode avec les isolats non humains, il peut être nécessaire de compléter avec des oligos d'ADN complémentaires aux séquences d' ARNr divergentes de la 3,21 hôte spécifique.
Étant donné que le protocole est impartiale, virale lit peut représenter seulement une petite fraction du contenu de la bibliothèque totale. Bien que l'ARNr est l'espèce la plus abondante de l'ARN hôte et seul un faible pourcentage des ARNr lit (0; 1%) se trouvent après l' épuisement sélectif, tous les autres ARN hôte (par exemple, ARNm) restera après l' épuisement et peut expliquer beaucoup de séquençage lit à partir de l'échantillon. Par conséquent , «suréchantillonnage» (c. -à- oversequencing) bibliothèques individuelles est nécessaire afin d'avoir une couverture suffisante pour montage et variantes appels viraux. Pour nos études, nous essayons de séquence ~ 20 millions lit par échantillon pour avoir assez de profondeur pour l' analyse des variantes génomiques et associée virale ainsi que le contenu métagénomique 2,5. Pour les études de découverte metagénomiques et d'agents pathogènes, il est important de noter que la contamination de l'ADN de l'hôte est éliminée par digestion par la DNase. Par conséquent, les virus et autres agents pathogènes qui contiennent des génomes d'ADN peuvent être perdues au cours du procédé, cependant intermédiaires d'ARN peuvent encore être séquencées.
Déclarations de divulgation
The authors have no competing financial interests.
Remerciements
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
matériels
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
Références
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon