Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Définition Substrat Spécificités pour lipase et phospholipase candidats
Dans cet article
Résumé
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Résumé
Microorganismes produisent un large spectre de (phospho) lipases qui sont sécrétées dans le but de faire des substrats extérieurs disponibles pour l'organisme. Alternativement, d'autres (phospho) lipases peuvent être associées physiquement l'organisme producteur provoquant un chiffre d'affaires de lipides intrinsèques et souvent donner lieu à un remodelage des membranes cellulaires. Bien que les potentiels (phospho) lipases peuvent être prédits avec un certain nombre d'algorithmes lorsque la séquence de gène / protéine est disponible, la preuve expérimentale de l'activité des enzymes, des spécificités de substrat et les fonctions physiologiques potentiels a souvent été obtenu. Ce manuscrit décrit l'optimisation des conditions d'essai pour les futurs (phospho) lipases avec des spécificités de substrat inconnus et comment employer ces conditions optimisées dans la recherche pour le substrat naturel d'un (phospho) lipase respective. En utilisant des substrats chromogènes artificiels, tels que les dérivés p - nitrophényle, peuvent aider à détecter un mineurl'activité enzymatique d'un (phospho) lipase prédite dans des conditions standard. Après avoir rencontré une telle activité enzymatique faible, les paramètres distincts d'un dosage enzymatique peut être modifiée afin d'obtenir une hydrolyse plus efficace du substrat artificiel. Après avoir déterminé les conditions dans lesquelles une enzyme fonctionne bien, une variété de substrats naturels potentiels doit être testé pour leur dégradation, un processus qui peut être suivie en utilisant des méthodes chromatographiques distinctes. La définition des spécificités de substrat pour de nouvelles enzymes, fournit souvent des hypothèses pour un rôle physiologique potentiel de ces enzymes, qui peuvent ensuite être testés expérimentalement. Suite à ces directives, nous avons été en mesure d'identifier une phospholipase C (SMc00171) qui dégrade la phosphatidylcholine à phosphocholine et diacylglycérol, dans une étape cruciale pour le remodelage des membranes dans la bactérie Sinorhizobium meliloti sur les conditions de phosphore limitation de la croissance. Pour deux prédit patatin-comme phospholipases (SMc00930 et SMc01003) du même organisme, nous pourrions redéfinir leurs spécificités de substrat et de préciser que SMc01003 est une lipase de diacylglycérol.
Introduction
Lipides à base de glycérol tels que triacylglycérols et (glycéro) phospholipides constituent importante et probablement les classes de lipides les plus connus 1. Triacylglycérols (TAG) sont des graisses ou des huiles, qui fonctionnent habituellement comme les lipides de stockage, et donc en tant que sources d'énergie et de carbone potentiels. TAGs peuvent être dégradés par les lipases, qui sont souvent sécrétées par l'organisme producteur à digérer TAGs externes et les rendre disponibles en tant que sources de carbone. En outre, les lipases ont été largement étudiés au cours des années en raison de leurs applications biotechnologiques importantes 2.
En raison de leur nature amphiphile et leur forme quasi cylindrique, les propriétés des membranes formant (glycéro) phospholipides présentent et constituent généralement les principaux composants lipidiques d'une membrane bicouche 3. Dans les micro - organismes simples, tels que la bactérie Escherichia coli, seules les trois principales variantes de groupe de tête, le phosphatidylglycérol (PG), cardiolipine (CL), et phosphatidylethanolamine (PE) sont rencontrées, mais il faut savoir que chacun d'entre eux pouvant être substitué par un grand nombre de différentes chaînes d'acyle gras en position sn -1 et sn -2 positions donnant lieu à un grand nombre d'espèces moléculaires différentes 4 . D'autres bactéries pourraient avoir d'autres phospholipides, en plus ou au lieu. Par exemple, Sinorhizobium meliloti, une bactérie du sol, qui est capable de former une racine nodule symbiose fixatrice d'azote avec la luzerne , des légumineuses (Medicago sativa), contient en outre au PE d' un second phospholipide zwitterionique, la phosphatidylcholine (PC) 5. En outre, les lipides ne contenant pas de phosphore ou de glycérol peuvent faire partie amphiphile et la forme de la membrane cellulaire. Par exemple, des conditions de croissance du phosphore limitatif, en S. meliloti (glycéro- phospholipides) sont en grande partie remplacés par des lipides membranaires qui ne contiennent pas de phosphore, à savoir, les sulfolipides, les lipides ornithine et diacylglyceryl trimethylhomoserine (dgts) 6. Chez les bactéries, dgts est formé à partir de diacylglycérol (DAG) dans une voie à deux étapes 7 , mais la source pour la génération DAG était pas claire. Expériences pulse-chase ont suggéré que PC pourrait être un précurseur pour dgts 8 et en utilisant la méthodologie décrite dans ce manuscrit nous avons pu identifier une phospholipase C (PLCP, SMc00171) qui est formé dans des conditions de phosphore limitatif et qui peut convertir PC en DAG et phosphocholine 8.
Dans une étude séparée, nous avons découvert que l'acyl-CoA synthétase (FADD) mutant déficient de S. meliloti ou d'Escherichia coli accumulée des acides gras libres lors de l' entrée en phase stationnaire de croissance 9. Bien que ces acides gras semblent être dérivés à partir des lipides membranaires, l'origine précise pour les acides gras libres ou de l'enzyme (s) en les libérant sont pas connus. Encore une fois, en utilisant la stratégie décrite dans ce manuscrit, deux 10 (phospho) lipases patatine-like (SMc00930 et SMc01003) qui a contribué à la formation d'acides gras libres dans S. meliloti 11 ont été prédit. De manière surprenante, SMc01003 DAG utilisé comme substrat le convertissant en monoacylglycérol et , enfin , du glycérol et des acides gras libres 11. Par conséquent, SMc01003 est une lipase DAG (DGLA).
Bien qu'un certain nombre d'algorithmes existent pour prédire le potentiel (phospho) lipases 12,13, leur fonction précise et rôle physiologique est généralement pas connue. Nous présentons ici un protocole, pour cloner et surexpriment (phospho) lipases prédites ou potentiels. Ce manuscrit explique comment les dosages enzymatiques peuvent être développées et optimisées pour le (phospho) lipase surexprimé en utilisant des substrats chromogènes artificiels. Nous fournissons des exemples comment avec un test enzymatique optimisé réel (phospho) substrat de lipase peut être rencontré et comment ces résultats pourraient enrichir notre compréhension de la physiologie microbienne.
Protocole
1. Clone et surexpriment gène de structure pour la lipase prédite
- En utilisant la réaction en chaîne par polymérase (PCR) 14 et des oligonucléotides spécifiques (Tableau 1) 15, amplifient le gène d'intérêt (smc01003, smc00930 ou smc00171), prévu pour coder pour une lipase ou phospholipase, de l'ADN génomique de l'organisme hôte (c. -à- S. meliloti).
- Introduire des sites de restriction spécifiques (avec la séquence des oligonucléotides conçus). Digérer le fragment d'ADN amplifié avec les enzymes de restriction correspondantes et le cloner dans un vecteur d'expression tel que des plasmides de la série pET 16.
- Après vérification de la séquence d'ADN pour le gène clone, de transformer le vecteur à une souche d'expression telle que Escherichia coli BL21 (DE3) pLysS 16.
- Préparer un pré-culture de nuit de l'hôte d'expression E. coli BL21 (DE3) pLETJ, hébergeant le vecteur pET respectif avec le gène clone ou le vecteur vide, dans 100 ml des flacons de culture contenant 20 ml de bouillon Luria Bertani (LB) 17 ainsi que les antibiotiques nécessaires. La culture des cellules à 30 ° C (ou à la température habituelle de croissance de la bactérie à partir de laquelle la lipase est originaire).
- En utilisant les pré-cultures de la nuit, inoculer 500 ml de milieu LB préchauffée (plus les antibiotiques nécessaires) dans 2 flacons L de culture pour obtenir une densité optique initiale à 620 nm (DO 620) = 0,05. Suivre la croissance des cultures et à une DO620 = 0,3, ajouter de l' isopropyl-β-D-thiogalactoside (IPTG) jusqu'à une concentration finale de 100 uM, et on incube sous agitation à 30 ° C pendant une période de 4 heures.
- A la fin de la période d'incubation, chaque culture transférer dans un tube à centrifuger de 500 ml et centrifuger à 5000 g à 4 ° C pendant 30 min. Remettre en suspension les culots de cellules bactériennes dans 5 ml de tampon de suspension (par exemple, SMc00930- et des cellules exprimant SMc01003 dans du Tris-HCl 50 mM pH 8,0 et les cellules exprimant SMc00171 dans 50 mM diéthanolamine-HCl, pH 9,8). Stocker les suspensions cellulaires à -80 ° C jusqu'à utilisation.
2. Préparer des extraits de protéines sans cellules et déterminer Protein Concentration
- Décongeler suspensions de cellules bactériennes et les stocker sur la glace. Passer des suspensions de cellules à trois reprises à travers une cellule sous pression à froid à 20.000 livres par pouce 2. Retirer les cellules intactes et les débris cellulaires par centrifugation à 5000 xg pendant 30 min à 4 ° C.
- Après centrifugation, on prépare des aliquotes de 100 et 500 ul du surnageant pour une analyse ultérieure et de les stocker à -80 ° C jusqu'à utilisation.
- Utiliser une fraction aliquote de 100 ul pour déterminer la concentration en protéines des extraits exempts de cellules distinctes par un procédé de choix , ou comme décrit 18.
3. Utiliser des substrats artificiels pour optimiser EnzymeActivités de (phospho) lipases
- Pour une couverture initiale des activités enzymatiques distinctes, en utilisant des substrats artificiels qui produisent un produit coloré lors de l' hydrolyse, tels que le p - nitrophénol (p -NP).
- Pour les dosages enzymatiques déjà optimisés avec des substrats d'ester p - nitrophényle artificiels (décrites pour la phospholipase C PLCP (SMc00171), ainsi que pour les phospholipases de patatine comme prédit SMc00930 et SMc01003), les systèmes de pipetage d'utilisation décrites dans le Tableau 2.
- Lors de l' exploration d' un nouveau potentiel (phospho) lipase, préparer un premier essai enzymatique standard contenant 50 mM de Tris-HCl, pH 8,5, NaCl 100 mM, 0,05% de Triton X-100, 0,5 mM de p composé contenant nitrophényle (p phosphate de nitrophényle , le phosphate de bis - p - nitrophényle, p - nitrophényle décanoate, ou p - nitrophényle palmitate), et l' extrait de protéine acellulaire (check 1, 3, 10, 30, 100, 300 et 1000 ug) dans un volume total de 1 ml dans 1 ml de Cuve en plastiquettes.
REMARQUE: Utiliser un pH alcalin (Figure 1) lorsque l'on suit l' hydrolyse de l' ester p - nitrophényle dans un dosage en continu. Vous pouvez également utiliser un seul des tests de temps de point pour une gamme de valeurs de pH, en ajoutant NaOH à la fin de la période d'incubation de mettre fin à la réaction enzymatique et de veiller à ce que tous les p -NP est présent sous forme de phénolate. - Suivre le cours du temps une augmentation de l' absorbance à 405 nm, due à la formation de p -NP, dans un spectrophotomètre à 30 ° C sur une période de 5 min. Quantifier la formation initialement linéaire de p -NP en déterminant la pente initiale de l' augmentation de l' absorbance par unité de temps.
- Calculer la variation de la concentration (Ac) pour p -NP utilisant la loi de Beer-Lambert (AA = ε Ac d) 1.
NOTE: AA est le changement linéaire d'absorbance déterminée, ε est le coefficient d'extinction molaire à la longueur d'onde respective (en unités de M -1 -1 cm), d est la longueur du trajet de lumière (1 cm) et Ac est la variation de concentration (en unités de M) à déterminer.- Considérant que le volume d'essai est de 1 ml, calculer la quantité de p -NP formé.
NOTE: Montant = concentration x volume. - Calculer l'activité enzymatique en divisant la quantité de p -NP formé par le temps dans lequel il est formé. Déterminer l'activité enzymatique spécifique en divisant l'activité enzymatique par la quantité de protéine (en mg) qui était responsable de la génération de cette activité.
- Considérant que le volume d'essai est de 1 ml, calculer la quantité de p -NP formé.
- Comparer les changements d'absorbance provoquées par des extraits de protéines dans lesquelles un gène candidat (smc00171, smc00930 ou smc01003) avait été exprimées avec des extraits qui abritent seulement un vecteur vide.
NOTE: Afin de poursuivre avec les étapes suivantes, les activités spécifiques, causées par des extraits de protéines dans lesquelles un gène candidat avait été exprimés, devraient être au moins deux fois ou mminerai que les valeurs obtenues pour les activités spécifiques causées par des extraits de protéines qui abritent seulement un vecteur vide. - Pour d' autres expériences, sélectionner les conditions dans lesquelles l' hydrolyse du composé p - nitrophényle contenant est minime avec des extraits exempts de cellules ( par exemple, vecteur vide) et pour lesquels la formation la plus prononcée de p -NP et l'anion p -nitrophenolate (Figure 1) peut être observé lorsque des extraits protéiques sont utilisés, dans lesquels un gène candidat a été exprimé.
- Après avoir déterminé l'activité enzymatique initiale de 3,1, d'optimiser les conditions de dosage pour l'enzyme concernée par le pH, le type de tampon variable, un tampon de force, des concentrations de NaCl, des détergents tels que le Triton X-100 et de l'absence ou en présence de divers cations bivalents.
- Pour différentes concentrations de chaque variable, déterminer l'activité enzymatique spécifique (voir 3.1.4.2) (le plus grand nombre obtenu définit la conditionde l'activité enzymatique maximale). Utiliser la combinaison des conditions optimales pour chacune des variables rencontrées pour définir un dosage enzymatique optimisé, dans lequel chaque variable est présent dans la concentration optimale.
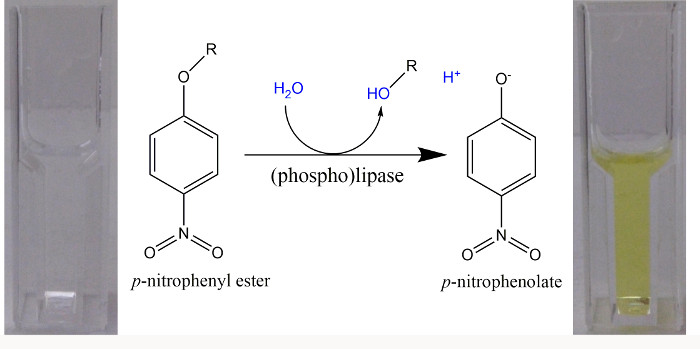
Figure 1. p esters de nitrophényle substrats artificiels pour (phospho) lipases dans un dosage spectrophotométrique. Lors de l' hydrolyse des esters p - nitrophényle, un acide (R-OH) et p - nitrophénol (p -NP) sont formés. En raison du pKa = 7,2 pour la dissociation du H + phénolique à partir de p -NP, à un pH> 9,2 , plus de 99% sont en forme jaune p -nitrophenolate lumineux et un coefficient d'extinction molaire de 18 000 M -1 cm - 1 peut être utilisé à une longueur d'onde de 405 nm pour la quantification de p libre 22 -nitrophenolate. Lorsque des tampons avec un pH de 8,5 ont été utilisés, l'absorbance a été déterminée à 400 nm et un coefficient d'extinction molaire de 14500 M -1 cm -1 a été utilisé 23. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
NOTE: Après avoir défini les conditions optimales pour l'activité de l'enzyme d'intérêt, se lancer dans la recherche pour le substrat réel / physiologique de cette lipase. En principe, prendre deux, souvent complémentaires, les approches pour atteindre cet objectif, une approche in vivo ou une approche in vitro.
4. In Vivo Identification de la Physiological Substrat d'une lipase
ove_content "> NOTE:. Dans une approche in vivo, exprimer la lipase d'intérêt dans un organisme hôte 8,11 afin de vous inscrire au fil du temps si l' expression de la lipase modifie le profil host's lipidique Dans une autre approche in vivo, générer un mutant déficient du gène d'intérêt 8,11 et étudier si son profil lipidique est distincte de la version de type sauvage 6,8,11. afin d'obtenir une évaluation quantitative du profil lipidique d'un organisme, une méthode simple consiste à radiomarquage composés cellulaires , l'extraction des lipides, en les séparant par chromatographie, et la quantification des lipides séparés marqués radioactivement.- Le marquage radioactif des lipides.
- Préparer un pré-culture de la nuit d'un organisme d'intérêt (E. coli ou S. meliloti) dans 5 ml de milieu de culture souhaitée (milieu complexe ou un milieu minimal défini) et de croître à 30 ° C.
- De la pré-culture, ensemencer dans 20 ml de same milieu frais dans une culture de 100 ml flacon pour obtenir une première DO 620 = 0,3 pour la culture.
- Prélever une partie aliquote (1 ml) de la culture dans des conditions stériles et transférer dans un tube à fond rond en polystyrène de 14 ml stérile.
- Ajouter 1 uCi de [1- 14C] acétate de méthyle (60 mCi par mmole) à la culture de 1 ml.
- Incuber la culture liquide, sous agitation à 30 ° C pendant une période de 24 heures.
- À la fin de la période d'incubation, transférer la culture dans un tube de microcentrifugation de 1,5 ml et centrifuger à 12 000 x g à température ambiante pendant 5 min.
- Remettre en suspension le culot dans 100 pl d'eau. À ce stade, stocker la suspension de cellules à -20 ° C ou immédiatement poursuivre l'extraction des lipides polaires (section 4.2).
- Extraction des lipides polaires.
NOTE: La méthode décrite ici essentiellement suit la procédure rapportée par Bligh et Dyer 19.- Pour les 100 pi de susp aqueuse de cellulesension, ajouter 375 ul de methanol: solution de chloroforme (2: 1, vol / vol).
- Vortex pendant 30 secondes et incuber pendant 5 minutes à température ambiante.
- Centrifuger 5 minutes à 12 000 x g à température ambiante.
- Transférer le surnageant dans un nouveau tube de 1,5 ml microcentrifugeuse.
- Ajouter 125 ul de chloroforme et 125 pi d'eau, vortex 30 sec.
- Centrifuger 1 min à 12 000 x g à température ambiante.
- Transférer la phase de chloroforme inférieure à un tube frais et sec avec un courant d'azote gazeux.
- Dissoudre les lipides séchés dans 100 ul de chloroforme: une solution de methanol (1: 1, vol / vol).
REMARQUE: À ce stade, une partie aliquote de 5 ul de la solution lipidique peut être quantifiée par comptage par scintillation liquide. - Pour la Chromatographie sur couche mince (CCM), on sèche jusqu'à 95 pl restants avec un courant d'azote gazeux et redissoudre les lipides séchés dans 20 ul de chloroforme et une solution de methanol (1: 1, vol / vol). Utiliser une fraction aliquote de 3 ulpour l'analyse par CCM.
- La séparation des lipides polaires par chromatographie sur couche mince (CCM).
NOTE: En fonction des classes de lipides à analyser, différentes combinaisons de phases solides et mobiles peuvent être utilisés pour la séparation. Voici une séparation typique des lipides chargés polaires et une autre, plus adaptés pour les lipides polaires neutres, en utilisant haute performance chromatographie sur couche mince (HPTLC) feuilles de gel de silice en aluminium comme phase solide, sont décrites.- La séparation des lipides polaires chargés par CCM à deux dimensions (2D-CCM).
- Appliquer une aliquote de 3 ul d'échantillon de lipide dans un coin d'une feuille CCMHP de gel de silice et d'aluminium (10 x 10 cm), à 2 cm du bord de la plaque.
- Préparer et mélanger la phase mobile (140 ml de chloroforme, 60 ml de methanol et 10 ml d'eau) pour la séparation dans la première dimension.
- Manteau une chambre de développement CCM en interne avec du papier de chromatographie.
Remarque: il est de faire en sorte que la phase gazeuse de la chambresera saturé rapidement (en 30 minutes) après la phase mobile pour la première dimension a été ajoutée à la chambre et la chambre a été fermée avec une plaque de verre. - Préparer et mélanger la phase mobile (130 ml de chloroforme, 50 ml de méthanol et 20 ml d'acide acétique glacial) pour la séparation dans la deuxième dimension et le transfert à une deuxième chambre de développement CCM revêtues intérieurement avec du papier de chromatographie et de laisser le saturer de chambre.
- Transférer soigneusement la feuille d'aluminium du gel de silice HPTLC avec l'échantillon de lipide séché à la première chambre et le développement ( par exemple, effectuer la Chromatographie) , la plaque pendant 60 minutes dans la chambre fermée dans la première dimension 5.
- Retirer la plaque de la chambre et de laisser les solvants sèchent dans une hotte à flux pendant 30 min.
- Après avoir tourné la plaque de 90 degrés par rapport à la chromatographie précédente, transférer la feuille d'aluminium et de gel de silice HPTLC, sur laquelle les lipides ont été séparés dans une dimension, à la secondechambre de d , et développer la plaque pendant 60 minutes dans la deuxième dimension 5.
- Retirez la feuille de la chambre et laisser les solvants sèchent dans une hotte à flux pendant au moins 2 h.
- Séparation de lipides polaires neutres.
- Appliquer 3 aliquotes d'échantillons de lipides sur une feuille de gel d'aluminium CCMHP de silice à partir de 2 cm des bords de la plaque. Si plusieurs échantillons sont analysés en chromatographie unidimensionnel, à une distance d'au moins 1,5 cm entre les différents points d'application de l'échantillon.
- Préparer et mélanger la phase mobile (140 ml d'hexane, 60 ml diéthyléther, et 8 ml d'acide acétique) et le transfert à une chambre de développement CCM revêtues intérieurement avec du papier de chromatographie et recouverts d'une plaque de verre pour laisser le saturer de chambre (30 min).
- Transférer la feuille d'aluminium de gel de silice avec CCMHP des échantillons de lipide séché à la chambre et à développer la plaque pendant 30 minutes dans la chambre fermée.
- Retirer la plaque de la chambreet laisser les solvants sèchent dans une hotte à flux pendant 2 heures.
- La séparation des lipides polaires chargés par CCM à deux dimensions (2D-CCM).
- Quantification et la visualisation des lipides polaires séparés.
- Une fois la feuille TLC développé est sec, incuber avec un écran luminescence photostimulable (PSL) dans une cassette fermée pendant 3 jours.
- Exposer l'écran incubé à un scanner de PSL et d'acquérir une image virtuelle des lipides radiomarqués séparés.
- Effectuer la quantification en utilisant un logiciel de PSL 20.
- Visualisation et l'isolement des différentes classes de lipides polaires.
- Incuber feuille CCM développée pendant 10 minutes dans une chambre de chromatographie en présence de 1 g de cristaux d'iode.
NOTE: Séparé des composés lipidiques vont dissoudre l'iode et apparaissent comme des taches brunâtres. - Encerclez les taches avec un crayon, les comparer à la mobilité relative (R f) de composés standards (ie, le 1,2-dipalmitoyl- sn - glycérol, dipalmitoyl-L-α-phosphatidylcholine, DL-α-monopalmitine, ou l'acide palmitique), et d'identifier à laquelle lipidique classe ils pourraient appartenir.
- Dans une hotte, laissez l'iode évaporer de la feuille TLC.
- Avec l'aide d'une spatule, gratter le gel de silice contenant le composé d'intérêt à partir de la feuille, et en extraire le composé à partir du gel de silice avec un mélange de 100 ul d'eau et 375 pi de methanol: solution de chloroforme (2: 1; en volume / vol).
- Continuer avec extraction selon Bligh et Dyer comme indiqué (4.2.2 et suivantes).
- Magasin purifié classe de lipides dans 100 ul de chloroforme et une solution de methanol (1: 1; volume / volume) à -20 ° C jusqu'à utilisation.
- Incuber feuille CCM développée pendant 10 minutes dans une chambre de chromatographie en présence de 1 g de cristaux d'iode.
5. In Vitro Identification de la Physiological substrat d'une lipase
NOTE: Dans une approche in vitro, étudier si la lipase d'intérêt peut convertir un mélange de lipides isolés ou de lipides purs individuels à l'hydrol correspondantproduits YSE dans les conditions définies comme optimale en 3.2.
- Utilisez les systèmes de pipetage pour les dosages enzymatiques selon le tableau 3 pour phospholipase spécifiques PC C SMc00171 (voir 5.2), phospholipase A (voir 5.3), et DAG lipase SMc01003 (voir 5.4) activité.
- Détermination de l' activité phospholipase C spécifique au PC (tableau 3).
- Pour un tube de 1,5 ml, ajouter 5.000 coups par minute (cpm) du total 14 C marqué PC et une solution de Triton X-100.
- Mélanger et sécher sous un courant d'azote.
- Ajouter diéthanolamine-HCl, pH 9,8 tampon, ainsi que NaCl et MnCl2 solutions et de l' eau bidistillée pour obtenir un volume final de 99,5 pi. Vortex pendant 5 sec.
- Ajouter 0,5 ul d'enzyme (5 pg de protéine) (soit un extrait exempt de cellule dans laquelle surexprimé SMc00171 est présent) pour amorcer la réaction. Mélanger brièvement.
- Incuber à 30 ° C pendant 4 heures.
- Arrêter la réaction par l'addition de 250 ul de methanol et 125 ul de chloroforme.
- Extraire les lipides comme décrit précédemment (voir 4.2).
- lipides séparés par une dimension (1D) -TLC (voir 4.3.2 et 4.4), et les analyser par imagerie par PSL.
- Détermination de l' activité phospholipase (tableau 3).
- Un tube de 1,5 ml, ajouter 5 000 cpm d' un total de 14 phospholipides marqué au C et une solution de Triton X-100.
- Mélanger et sécher sous un courant d'azote.
- Pour un dosage final de 100 ul, ajouter du Tris-HCl, pH 8,5, un tampon, une solution de NaCl et de l'eau. Vortex pendant 5 sec.
- Ajouter 5 pi d'enzyme (50 pg de protéines) (soit un extrait exempt de cellule dans laquelle surexprimé SMc00930 ou SMc01003 est présent).
- Incuber à 30 ° C pendant 5 heures.
- Arrêter la réaction par addition de 250 ul de methanol et 125 ul de chloroforme.
- Extraire les lipides comme décrit précédemment (voir 4.2), séparéselles par 1D-CCM en utilisant 130 ml de chloroforme, 50 ml de methanol et d'acide acétique glacial 20 ml en tant que phase mobile, et de les analyser par imagerie PSL.
- Détermination de diacylglycérol (DAG) activité de la lipase.
- Préparation de 14 C marqué par DAG.
- Radiomarqueur S. cultures meliloti (voir 4.1) et extraire les lipides polaires (voir 4.2) comme décrit. Séparée S. meliloti totaux extraits lipidiques par 1D-CCM dans un mélange chloroforme: methanol: acide acétique (130: 50: 20, volume / volume) en utilisant les conditions décrites pour la séparation dans la deuxième dimension dans le 4.3.1.
- Visualisez PC par coloration d'iode et d'utiliser un crayon pour marquer la localisation de phosphatidylcholine (PC).
- Isoler PC radiomarqué comme décrit en 4.5.
- Quantifier PC extraite par comptage par scintillation.
NOTE: A propos de 320.000 cpm PC est attendue. - Traiter le PC (250 000 cpm) , avec 0,1 U de la phospholipase C de Clostridium perfringens dans 50 mM de Tris-HCl, pH 7,2, 0,5% De Triton X-100 et 10 mM de CaCl2 pendant 2 heures dans un volume total de 100 ul et d' arrêter la réaction en ajoutant 250 pi de methanol et 125 ul de chloroforme.
- Extraire les lipides comme décrit précédemment et séparés par eux par 1D-CCM (voir 4.3.2).
- Diacylglycérol isolât de la plaque de silice et la quantification par comptage par scintillation (comme décrit en 4.2)
- Dosage de la lipase diacylglycérol (tableau 3).
- Un tube de 1,5 ml, ajouter 5 000 cpm de 14C-DAG marqué et une solution de Triton X-100.
- Mélanger et sécher sous un courant d'azote.
- Pour un dosage final de 100 ul, ajouter du Tris-HCl (pH 9,0) tampon, une solution de NaCl et de l'eau bidistillée. Vortex pendant 5 sec.
- Initier la réaction en ajoutant 5 ul d'enzyme (50 ug de protéine d'extrait acellulaire).
- Incuber à 30 ° C pendant 4 heures.
- Arrêter la réaction par addition de 250 ul de methanol, und 125 ul de chloroforme et extrait les lipides comme décrit précédemment (voir 4.2).
- Analyser les lipides polaires neutres par 1D-CCM (voir 4.1.3.2) et l'imagerie PSL ultérieure.
- Préparation de 14 C marqué par DAG.
Résultats
L' activité de la phospholipase C spécifique au PC avec SMc00171 Bis- p - nitrophényle phosphate
Extraits exempts de cellules obtenues à partir de E. coli BL21 (DE3) pLysS x, qui ont exprimé smc00171, ont été étudiés pour leur capacité à hydrolyser p bis- esters de phosphate de nitrophényle, en utilisant un d...
Discussion
Au cours des 20 dernières années, les génomes de nombreux organismes ont été séquencés et même si un grand nombre de données de séquence du génome a été générée, l'interprétation fonctionnelle est à la traîne et entrave donc notre compréhension du fonctionnement du génome. fonctions de gènes dans les génomes sont souvent attribués en fonction de similarité avec des gènes de la fonction ou l'apparition de motifs conservés connu. Cependant, la fonction précise d'un gène donné est ...
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Ce travail a été soutenu par des subventions de Consejo Nacional de Ciencias y Tecnología-México (CONACyT-Mexique) (82614, 153998, 253549 et 178359 dans Investigación Científica Básica ainsi que 118 à Investigación en Fronteras de la Ciencia) et de Dirección General de Asuntos de Personal Académico-Universidad Nacional Autónoma de México (UNAM DGAPA-; PAPIIT IN202616, IN203612).
matériels
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
Références
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon