Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Cytométrie en flux Analyse des cellules tueuses naturelles lytique Activité dans le sang humain
Dans cet article
Résumé
This work describes an advanced workflow for the accurate and fast determination of NK (Natural Killer) cell count and NK cell cytotoxicity in human blood samples.
Résumé
Cytotoxicité des cellules NK est une mesure largement utilisée pour déterminer l'effet d'une intervention extérieure sur la fonction des cellules NK. Cependant, la précision et la reproductibilité de cet essai peut être considéré comme instable, soit à cause des erreurs de l'utilisateur ou du fait de la sensibilité des cellules NK à une manipulation expérimentale. Pour éliminer ces problèmes, un flux de travail qui les réduit à un minimum a été créé et est présenté ici. Pour illustrer, nous avons obtenu des échantillons de sang, à divers moments, des coureurs (n = 4) qui ont été soumis à un combat intense d'exercice. Tout d'abord, les cellules NK sont simultanément identifiés et isolés par CD56 de marquage et de tri magnétique à base, directement à partir de sang total et d'aussi peu que un millilitre. Les cellules NK triées sont retirées de tous les anticorps de réactifs ou de coiffage. Ils peuvent être comptés afin d'établir un décompte précis des cellules NK par millilitre de sang. Deuxièmement, les cellules NK (cellules triées effecteurs ou E) peuvent être mélangés avec 3,3'-Diotadecyloxacarbocyanine perchlorate (DIO) marqué des cellules K562 (cellules cibles ou T) à un 1 dosage optimal: 5 T: rapport E et analysé à l'aide d'un flux-cytomètre d'imagerie qui permet la visualisation de chaque événement et l'élimination de tout faux positif ou de faux négatifs (tels que des doublets ou des cellules effectrices). Ce flux de travail peut être complété en environ 4 h, et permet des résultats très stables, même lorsque l'on travaille avec des échantillons humains. Lorsqu'elles sont disponibles, les équipes de recherche peuvent tester plusieurs interventions expérimentales chez des sujets humains, et de comparer les mesures à travers plusieurs points de temps sans compromettre l'intégrité des données.
Introduction
Les cellules tueuses naturelles sont un élément essentiel du système immunitaire inné. Alors qu'ils sont très réglementés, ils ont la capacité de reconnaître et d' éliminer les cellules anormales par contact de cellule à cellule et sans activation préalable 1. En tant que tel, ils forment une ligne rapide de défense contre les infections. L' exercice, surtout intense a été montré pour déprimer le système immunitaire transitoirement 2, 3, 4, 5. Les cellules NK sont particulièrement sujettes à cet effet , 4, 6, 7, créant ainsi une fenêtre d' une sensibilité accrue aux maladies. Par conséquent, l'étude des interventions avant, pendant ou après un exercice intense dans le but de réduire son impact sur la fonction des cellules NK est d'un intérêt particulier pour le bien-être des athlètes post-concours.
Toutefois, l'étude de ces interventions est compliquée par de nombreux facteurs: 1) les cellules NK sont rares 8, à environ 1% du blanc compartiment des cellules sanguines; 2) Les cellules NK sont très sensibles au stress et reposent sur une exposition constante à des conditions physiologiques pour rester viable et stable au cours de l'expérimentation; et 3) des tests standard pour déterminer la cytotoxicité des cellules NK, tels que les gradients de Ficoll 9 et libèrent des essais 10, ne sont pas fiables et incohérentes. La variabilité inhérente des échantillons humains ne font qu'aggraver ces problèmes. échantillons humains frais prélevés lors d'interventions sont assez réglementé et difficile à se procurer, au moins par rapport à des échantillons d'animaux ou de lignées cellulaires immortalisées. Cela réduit les possibilités de répéter des expériences ou ajouter des participants à la cohorte de l'étude pour atteindre les seuils statistiques significatives. Collectivement, ces questions prennent en charge la nécessité d'un protocole simplifié qui permet for à la fois à haut débit et une analyse de haute fiabilité de NK activité lytique des cellules dans des échantillons humains.
Nous avons établi un flux de travail qui réduit le temps nécessaire pour identifier, isoler et les cellules NK de test à partir de sang total humain, tout en minimisant l'exposition à des facteurs externes. La méthode optimise l'utilisation des deux instruments, un trieur magnétique à base de cellules et d'un flux d'imagerie cytomètre et un spécifique de test, T optimisé: rapport E pour permettre la détection d'une diminution ou une augmentation de cytotoxicité des cellules NK.
Protocole
NOTE: Toutes les procédures de collecte de sang ont été effectuées en conformité avec les lignes directrices énoncées par l'Appalachian State University Board Review (ASU) Institutionnel (CISR).
1. Toute la collection de sang
- Avoir un phlébotomiste certifié prélever du sang selon les directives de l'Organisation mondiale de la Santé.
- Prélever le sang dans un tube de 4 ml de collecte de sang contenant de l' acide Dipotassique éthylènediaminetétraacétique (EDTA K 2). Invertir le tube de prélèvement de sang selon les instructions du fabricant. Gardez le tube de collecte de sang à la température ambiante sur une bascule paillasse jusqu'à la séparation.
2. Préparation des cellules cibles marquées DiO
- Cultiver des cellules K562 dans son Complete Iscove modifié Médias Dulbecco (IMDM) additionné de 10% de sérum de veau fœtal (FBS) et 1% de pénicilline streptomycine pendant plusieurs semaines avant l'essai pour assurer la santé des cellules. Réglez le concentration des cellules à 3 x 10 5 cellules / mL par jour grâce à la réalisation d'une cellule de comptage et le passage ultérieur des cellules. Effectuer un changement de support complet tous les 2 à 3 jours.
- Le jour de l'essai, effectuer une numération cellulaire en utilisant un mélange 1: 1 hémocytomètre.
- Retirer 10 ul du flacon K562 et le placer dans le tube 1,5 ml.
- Ajouter 10 pl de colorant bleu trypan dans le même tube de 1,5 ml pour un facteur de dilution de 1: 1.
- Doucement le tube flick pour mélanger des cellules K562 et colorant bleu trypan.
- Laisser K562 colorant bleu cellule trypan à incuber pendant 1 min à température ambiante.
- Retirer 15 ul de cellules K562 colorées du tube.
- Pipette sur hémocytomètre pour le nombre de cellules.
- Compter les cellules dans les quatre cases de coins, ainsi que la place centrale pour un total de cinq places.
- Calculer le nombre de cellules en utilisant l'équation suivante:
cellules totales / ml = Total cellules comptées X (facteur de dilution / Nombre de places) X 10000 / mL
- Remettre en suspension les cellules cibles K562 à une densité finale de 1 x 10 6 cellules / ml dans un milieu de culture IMDM exempt de sérum.
- Pour les cellules cibles K562 non marquées, ajouter 10 ml de cellules cibles K562 à une 15 tubes ml à une densité de 1 x 10 6 cellules / ml pour un total de 10 x 10 6 cellules K562. Placer dans un incubateur à 37 ° avec 5% de CO 2 jusqu'à ce que prêt à l' emploi.
- Pour les cellules cibles DiO teinté K562, ajouter 10 ml de K562 cible les cellules à un 15 tube de ml à une densité de 1 x 10 6 cellules / ml pour un total de 10 x 10 6 cellules K562.
- Ajouter 1 pl de DiO solution cellulaire étiquetage par ml de suspension cellulaire dans 15 ml tube désigné pour DiO coloration et doucement vortex. Par exemple, ajouter 10 ul de solution DiO cellule marquage à 10 x 10 6 cellules K562 / ml à un volume de 1 x 10 6 cellules / ml.
- Incuber solution K562-DIO pendant 20 min à 37 ° C avec 5% de CO 2 dans un tube de 15 ml.
- Suivreing incubation, ajouter 3 ml de solution saline température ambiante tamponnée au phosphate (PBS) à la solution K562-DiO contenant 15 ml tube.
- Centrifuger pendant 10 min à 135 xg à la température ambiante.
- Retirez délicatement le surnageant sans perturber le culot cellulaire avec un volume réglable pipette 1000 pi.
- Ajouter 10 ml de IMDM frais à culot cellulaire contenant 15 ml tube.
- Doucement tube vortex pour remettre en suspension les cellules.
- Répétez les étapes 2.7 à 2.10 deux fois plus.
- Stocker les piles dans un incubateur à 37 ° avec 5% de CO 2 jusqu'à ce que prêt à l' emploi.
Note: Les cellules peuvent être stockées dans l'incubateur pendant au moins 24 h, mais il est préférable de les utiliser sur le même jour.
3. Préparation des commandes
- Transférer le suivant dans 1,5 ml tubes séparés et étiquetés de manière appropriée:
- Ajouter 500 pi de IMDM frais contenant remis en suspension DiO marqué des cellules K562 dans le "Double positif" étiqueté tube de 1,5 ml.
- Ajouter 500 pi de IMDM frais contenant remis en suspension les cellules K562 DiO marquées dans le "DiO seulement" tube de 1,5 ml étiqueté.
- Ajouter 500 pi de IMDM frais contenant des cellules K562 non marquées remises en suspension dans le "iodure de propidium (PI) que" tube de 1,5 ml étiqueté.
- Placer le positif Double et PI seulement tubes dans un bain d'eau à 55 ° C pendant 5 min.
- Après 5 min écoulé, retirer les tubes et essuyez avec 70% d'éthanol.
- Ajouter 10 L de PI à la Positive Double et PI seulement 1,5 ml tubes.
- Placer tous les trois contrôles K562 de cellules cibles dans l'incubateur à 37 ° C pendant 30 min.
- Après 30 minutes d'incubation est écoulée, centrifuger les trois K562 contrôles de cellules cibles pendant 2 min à 163 x g.
- Retirez délicatement le surnageant sans perturber le culot cellulaire.
- Resuspendre chaque contrôle avec 20 ul de milieu frais de culture cellulaire IMDM, et laisser dans le 37 ° C incubateur avec 5% de CO 2 pendant au moins30 min pour le signal de DiO optimal.
REMARQUE: Les contrôles sont maintenant prêts à être exécutés à travers le flux d'imagerie cytomètre.
4. cellules NK Séparation automatique
- Allumez séparateur de cellules et laisser le cycle de démarrage pour terminer.
- Veiller à ce que toutes les bouteilles enluminures fluides sont verts et que la bouteille de déchets est vide.
- Obtenir un rack tube mL température 15 ambiante.
NOTE: La sélection est basée sur des volumes d'échantillons. Par exemple, pour un volume inférieur à 3 ml d'utiliser un support de tube de 15 ml et un volume de plus de 3 ml d'utiliser un support de tube de 50 ml. - tubes d'échantillons d'étiquettes en conséquence (Répéter par échantillon / participant): Participant 1 échantillon de sang; Participant 1 fraction négative; Participant 1 fraction positive.
- pipette doucement 1,5 ml de sang total de l'étape 1.2 dans "échantillon de sang total" 15 ml tube.
- Placez correctement étiquetés 15 ml "échantillon de sang total" tube de l'étape 4.5 et marqué 15 mL "Fraction négative" ettubes "Fraction positive" de l'étape 4.4 dans le rack du tube. Utilisez rack d'échantillons thefollowing set-up: Row (R1) A: échantillon de sang total, Rangée (R2) B: Négatif, fraction non marquée, Rangée (R4) C: Positif, fraction magnétiquement marqué.
- Insérez le séparateur en rack sur le MiniSampler pour autolabeling.
- Sélectionnez "réactif" dans le menu et mettre en évidence la position où le flacon sera placé dans le rack de séparation.
- Sélectionnez "Lire réactif" pour activer le lecteur de code 2D.
- Placer le flacon de réactif approprié devant le lecteur de code 2D entre 0,5-2,5 cm du couvercle du lecteur de code.
REMARQUE: par exemple, le réactif nécessaire pour la séparation des cellules NK CD56. - Tenir flacon de réactif à un angle en face du lecteur de code 2D pour une lecture optimale.
- Placer le flacon de réactif dans la position correcte de la grille de séparation.
- Mettre en évidence les positions souhaitées sous l'onglet de séparation sur l'écran.
- Dans le sous-menu d'étiquetage, d'attribuer une fille auprogramme de tolabeling.
- Attribuer la cellule de séparation microbilles réactif CD56 à accumuler les positions 1, 2, 3 et 4.
- Sélectionnez le programme de séparation "de posselwb".
- Sélectionnez le "rinçage" programme de lavage.
- Insérez un volume d'échantillon de 1500 pi dans le "Volume" sous-menu à l'aide du pavé numérique.
- Sélectionnez l'option "Enter" sur le clavier.
- Sélectionnez "Exécuter" pour commencer la séparation des cellules.
- Sélectionnez "OK" pour confirmer que le tampon assez est disponible pour le traitement de tous les échantillons.
5. NK Cell Count Après séparation cellulaire
- Immédiatement après la séparation des cellules avec le séparateur de cellules, de récupérer la fraction positive. Laisser à température ambiante. Cette fraction contient la population de cellules NK pur souhaitée.
- Pour chaque échantillon, effectuer un comptage des cellules en utilisant un hémocytomètre selon l' étape 2.2.
- Après les calculs, enregistrer le nombre de cellules.
6. Essai de cytotoxicité Préparation d'échantillons
- Préparer et étiqueter les tubes de 1,5 mL pour chaque échantillon / participant en conséquence.
- Pipette désiré rapport des cellules NK et les cellules K562 DiO-labled dans chaque tube.
REMARQUE: par exemple, le rapport désiré de cellules cibles K562 et des cellules effectrices NK est de 1: 5. - Centrifuger pendant 5 min à 135 x g.
- Retirez délicatement le surnageant sans perturber le culot cellulaire.
- Remettre en suspension NK-DIO marqué mélange de cellules K562 dans 500 ul de milieu de cellules NK sans interleukine-2 (IL-2) et 2-mercaptoéthanol (2-ME) (NK incomplète des milieux de culture cellulaire).
NOTE: Le NK milieux de culture cellulaire incomplète est du milieu essentiel minimum d'Eagle avec du bicarbonate de sodium, sans L-glutamine, ribonucléosides et désoxyribonucléosides. - Ajouter 5 ul de PI à chaque tube.
- Centrifugeuse pendant 2 min à 163 x g.
- Incuber les cellules à 37 ° C pendant 2 h.
- Après 2 heures d'incubation, centrifuge pendant 2 min à 163 x g.
- Retirez délicatement le surnageant sans perturber le culot cellulaire.
- Resuspendre les cellules dans 25 ul incomplet NK milieux de culture cellulaire.
7. Préparation de spontanée ( «S») Echantillon
- Pipette 500 pi de cellules K562 DiO marqués (concentration de 1 x 10 6 cellules / mL) en tube de 1,5 ml.
- Ajouter 10 pi de PI à chaque tube.
- Tube à centrifuger pendant 2 min à 163 x g.
- Incuber les cellules à 37 ° C pendant 2 h.
- Après 2 heures d'incubation, centrifuger 2 min à 163 x g.
- Retirez délicatement le surnageant sans perturber le culot cellulaire.
- Resuspendre les cellules dans 25 ul incomplète Essential Medium (α-MEM) des milieux de culture de cellules alpha-minimum.
8. Acquisition de données avec imagerie cytomètre de flux
- Appuyez sur le bouton vert à l'intérieur de la porte d'entrée de l'imagerie cytomètre en flux pour allumer l'instrument.
- Allumez all ordinateurs associés à l'imagerie du cytomètre en flux.
- Lancer le flux d'imagerie cytomètre logiciel.
- Cliquez sur le bouton "Démarrage" pour purger le système et préparer la ligne d'échantillon.
- Une fois que le "démarrage" est terminée, fermez la fenêtre "étalonnages".
- Attribuer canaux: sur le côté supérieur gauche, cliquez sur chaque canal afin de les affecter.
- Du côté de la main droite, cliquez sur le bouton de nuage de points pour créer 4 diagrammes de dispersion: Raw Max Pixel _MC_6 vs Area_M06, Raw Max Pixel _MC_2 vs Area_M02, Raw Max Pixel _MC_5 vs Area_M05 et FieldArea_M01 vs AspectRatio_M01.
- Commencez l' analyse d' échantillons en utilisant d' abord la commande "Double Positive".
- Cliquez sur "Load".
- Placer le tube de 1,5 ml avec l'échantillon "Double Positive" de Steps 3.4 à 3.9 dans l'échantillon chargeur.
- Sélectionnez l'objectif 40X sous l'onglet «Grossissement».
- Allumez le mW 405 etDes lasers à 642 mW.
- Tourner sur le canal "Brightfield".
- Cliquez sur "Sélectionner l'intensité."
- Sur la base de l'échantillon de contrôle "Double positif", déterminer l'intensité souhaitée pour le laser de 405 mW de sorte que le détecteur ne soit pas surchargé.
Remarque: Par exemple, l'intensité optimale pour cette expérience a été fixée à 11 mW.
- Après la mise en place souhaitée est atteinte, l' acquisition de données.
- Sous l'onglet "Fichier Acquisition", entrez dans un texte personnalisé de nom de fichier. Sélectionnez un dossier pour enregistrer le fichier (s) de données.
- Entrez le nombre de cellules d'acquérir à côté de "Collect". Généralement, ce nombre varie entre 1.000 à 10.000.
- Cliquez sur "Acquire."
NOTE: Une fois que le nombre souhaité de cellules est acquise, le fichier de données est automatiquement enregistrée dans le dossier sélectionné précédemment.
- Après l' acquisition terminée, charger le prochain échantillon de contrôle - DiO seul contrôle.
- Cliquez sur "Load".
- Placer le tube de 1,5 ml avec le "DiO seulement" échantillon dans le chargeur.
- Laissez l'objectif 40X sous l'onglet «Grossissement» sélectionné.
- Laisser le laser de 405 mW allumé.
- Eteignez le laser 642 mW et le canal "Brightfield".
NOTE: Maintenant que le set-up souhaité a été atteint, les données peuvent être acquises. - Sous l'onglet "Fichier Acquisition", entrez dans un texte personnalisé de nom de fichier et sélectionnez un dossier pour enregistrer le fichier (s) de données. Entrez le nombre de cellules d'acquérir à côté de "Collect.". Généralement, ce nombre est de 1000.
- Cliquez sur "Acquire."
NOTE: Une fois que le nombre souhaité de cellules est acquise le fichier de données est automatiquement enregistrée dans le dossier sélectionné précédemment.
- Répétez l'étape 8.10 pour le "PI seulement" échantillon de contrôle. Les échantillons expérimentaux sont maintenant prêts à être collectés.
- Manipuler les remaining échantillons expérimentaux, y compris les "échantillons" S "spontanées" comme suit:
- Laissez l'objectif 40X sous l'onglet «Grossissement» sélectionné.
- Allumez le 405 mW et 642 mW lasers.
- Allumez le canal "Brightfield".
- Cliquez sur "Définir l'intensité."
- Sous l'onglet "Fichier Acquisition", entrez dans un texte personnalisé de nom de fichier et sélectionnez un dossier pour enregistrer le fichier (s) de données. Entrez dans un texte personnalisé de nom de fichier.
- Entrez le nombre de cellules d'acquérir à côté de "Collect."
- Cliquez sur le bouton "Acquire".
NOTE: Une fois que le nombre souhaité de cellules est acquise le fichier de données est automatiquement enregistrée dans le dossier sélectionné précédemment.
- Répétez l'étape 8.12 pour tous les échantillons expérimentaux.
- Après toutes les données expérimentales et les fichiers ont été collectés, cliquez sur le bouton "Arrêter" pour stériliser le système.
9. Imaging cytomètre de flux Analyse d'échantillon
- Ouvrir le flux d'imagerie cytomètre application logicielle d'analyse.
- Sous "Fichier", ouvrir un fichier .rif expérimental.
- Construire une nouvelle matrice en utilisant les fichiers .rif de couleur unique (DIO uniquement contrôler et PI-seulement le contrôle, créé au cours des étapes 8.10 et 8.11) en sélectionnant «Créer un nouveau Matrix" sous l'onglet Compensation dans le flux d'imagerie cytomètre logiciel.
REMARQUE: Le logiciel vous demandera la sélection des fichiers d'une seule couleur et de les fusionner pour créer un fichier de matrice (extension de fichier .ctm), qui doit être sélectionné pour appliquer une compensation de canal. - Créer des parcelles de points en utilisant la fonction "blocs de construction" du logiciel.
- Créer un point-plot (BrightFieldGradient_RMS vs fréquence) à la porte des cellules ciblées. Appelez la porte "Focus" (figure 1A).
- Utilisation de la porte "Focus", créez un tracé de points de Bright Zone Champ vs brillant Champ Aspect Ratio à g mangé les cellules singlet. Appelez la porte "Single" (figure 1B).
- Utilisation de la porte "Single", créer un terrain dot de Intensity_MC_Ch02 vs Intensity_MC_Ch5. Utilisez ce terrain pour identifier et porte des cellules doubles positives (cibles, morts) DiO positif seules les cellules (cibles, vivant) et PI-DiO (figure 1C).
NOTE: Toutes les parcelles décrites dans les étapes 9.4.1, 9.4.2 et 9.4.3 peuvent être créés en utilisant la fonction "blocs de construction" du logiciel.
- Cliquez sur la fonction statistique du tracé de points pour accéder aux numéros de cellules de chaque porte.
- Calculer le pourcentage de cibles mortes dans l'échantillon spontanée et des échantillons expérimentaux utilisant la formule suivante:
% des cibles mortes dans l'échantillon = (cibles de #dead x 100) / (# vivent cibles + #dead cibles) - Calculer la cytotoxicité en utilisant la formule suivante:
% Cytotoxicité = [(expérimentale mort spontanée morts) / (100 morts-spontanée)] x100
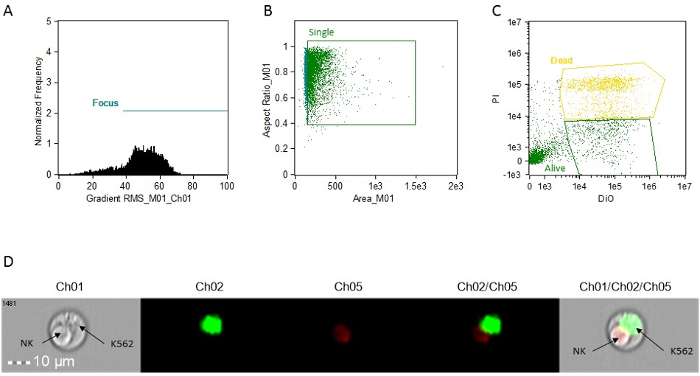
Figure 1: histogrammes représentatifs, des diagrammes de dispersion et des images pour l' analyse de l' activité cytotoxique. Analyse (A) de la cellule de mise au point. (B) seule analyse cellulaire. (C) Analyse de coloration de la cellule cible. Toutes les déterminations sont faites en utilisant l'image jointe à chaque événement. Cela peut être consulté dans le logiciel d'analyse en cliquant simplement sur l'événement sur les graphiques. (D) d'image représentative d'un événement de doublet, montrant une cellule apoptotique NK et une cible de K562 en direct. Ch01, Brightfield. Ch02, DiO. CH05, PI. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
Détermination de NK numération cellulaire
L'effet de la course lourde sur NK numération des cellules dans le sang total a été mesurée en utilisant le protocole d'exercice décrit à la figure 2. Des échantillons de sang ont été prélevés avant l'exercice, immédiatement après l'exercice, 1,5 h après l'exercice, et enfin 24 et 48 h après le prélèvement sanguin initial. La concentration des cellules NK par ml de sang total a été ...
Discussion
La méthode décrite dans cette étude mesure directement l'activité spécifique des cellules NK d'un individu en réponse à des stimuli (dans ce cas particulier, l'exercice prolongé). Typiquement, les cellules NK sont isolées à partir de sang en utilisant un gradient de densité ou de tri cellulaire en utilisant une combinaison de marqueurs. Bien que ces méthodes sont largement utilisées, elles présentent de nombreux inconvénients: ils prennent beaucoup de temps, impliquent de multiples manipulatio...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
This project was supported by Agriculture and Food Research Initiative Competitive Grant no. 2100-68003-30395 from the USDA National Institute of Food and Agriculture.
matériels
| Name | Company | Catalog Number | Comments |
| K-562 lymphoblasts | ATCC | CCL-243 | |
| Iscove's Modified Dulbecco's Media | ATCC | 30-2005 | High glucose, with L-Glutamine, with HEPES, Sterile-filtered |
| Alpha Minimum Essential medium | ATCC | CRL-2407 | Without ribonucleosides and deoxyribonucleosides but with 2 mM L-glutamine and 1.5 g/L sodium bicarbonate |
| Trypan Blue Solution 0.4% | Amresco | K940-100ML | Tissue culture grade |
| Propridium Iodide Staining Solution | BD Pharmingen | 51-66211E | |
| Vybranto DiO cell-labeling solution | Vybranto | V-22886 | |
| autoMACS Pro Separator | Miltenyi Biotec | 130-092-545 | |
| autoMACS Running Buffer | Miltenyi Biotec | 130-091-221 | |
| autoMACS Washing Buffer | Miltenyi Biotec | 130-092-987 | |
| autoMACS Columns | Miltenyi Biotec | 130-021-101 | |
| Whole Blood CD56 MicroBeads, human | Miltenyi Biotec | 130-090-875 | |
| ImageStream X Mark II Imaging Flow Cytometer | EMD Millipore | ||
| Speedbeads | Amnis Corporation | 400030 | |
| 0.4-0.7% Hypochlorite (Sterilizer) | VWR | JT9416-1 | |
| Coulter Clenz | Beckman Coulter | 8546929 | |
| 70% Isopropanol (Debubbler) | EMD Millipore | 1.3704 | |
| D-PBS (Sheath fluid) | EMD Millipore | BSS-1006-B (1X) | No calcium or magnesium |
| INSPIRE Software | EMD Millipore | Version Mark II, September 2013 | |
| Ideas Application Software | EMD Millipore | Version 6.1, July 2014 |
Références
- Cerwenka, A., Lanier, L. L. Natural killer cells, viruses and cancer. Nat Rev Immunol. 1 (1), 41-49 (2001).
- Nieman, D. C. Exercise, infection, and immunity. Int J Sports Med. 15, 131-141 (1994).
- Romeo, J., Warnberg, J., Pozo, T., Marcos, A. Physical activity, immunity and infection. Proc Nutr Soc. 69 (3), 390-399 (2010).
- Nieman, D. C. Marathon training and immune function. Sports Med. 37 (4-5), 412-415 (2007).
- Simpson, R. J., Kunz, H., Agha, N., Graff, R. Exercise and the Regulation of Immune Functions. Prog Mol Biol Transl Sci. 135, 355-380 (2015).
- Walsh, N. P., et al. Position statement. Part one: Immune function and exercise. Exerc Immunol Rev. 17, 6-63 (2011).
- Timmons, B. W., Cieslak, T. Human natural killer cell subsets and acute exercise: a brief review. Exerc Immunol Rev. 14, 8-23 (2008).
- Westermann, J., Pabst, R. Distribution of lymphocyte subsets and natural killer cells in the human body. Clin Investig. 70 (7), 539-544 (1992).
- Boyum, A. Isolation of leucocytes from human blood. Further observations. Methylcellulose, dextran, and ficoll as erythrocyteaggregating agents. Scand J Clin Lab Invest Suppl. 97, 31-50 (1968).
- McMillan, R., Scott, J. L. Leukocyte labeling with 51-Chromium. I. Technic and results in normal subjects. Blood. 32 (5), 738-754 (1968).
- Berk, L. S., et al. The effect of long endurance running on natural killer cells in marathoners. Med Sci Sports Exerc. 22 (2), 207-212 (1990).
- McAnulty, L. S., et al. Effect of blueberry ingestion on natural killer cell counts, oxidative stress, and inflammation prior to and after 2.5 h of running. Appl Physiol Nutr Metab. 36 (6), 976-984 (2011).
- Millard, A. L., et al. Brief Exercise Increases Peripheral Blood NK Cell Counts without Immediate Functional Changes, but Impairs their Responses to ex vivo Stimulation. Front Immunol. 4, 125 (2013).
- Janeway, C. A., Travers, P., Walport, M., Shlomchik, M. J. . Immunobiology: The Immune System in Health and Disease. 5th edn. , (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon