Method Article
Évaluation de la Contamination d’ADN dans les échantillons d’ARN issu de l’ADN ribosomique
Dans cet article
Résumé
Nous présentons ici un protocole pour le suivi des échantillons d’ARN génomique contamination ADN (génomique). La méthode présentée utilise des amorces spécifiques pour la région de l’espaceur interne transcrit (ITS) des gènes ribosomiques d’ADN (ADNr). La méthode est adaptée pour une détection fiable et sensible d’ADN contaminant dans la plupart des eucaryotes et des procaryotes.
Résumé
Une méthode largement utilisée pour la quantification des modifications de l’expression génique et abondance de la transcription est quantitative PCR en temps réel de transcription inverse (RT-qPCR). Il fournit des résultats précis, sensibles, fiables et reproductibles. Plusieurs facteurs peuvent affecter la sensibilité et la spécificité de la RT-qPCR. L’ADN génomique résiduelle (ADNg) contamination des échantillons d’ARN est l’un d'entre eux. Dans l’analyse de l’expression génique, amplification non spécifique en raison de la contamination de l’ADNg surestime l’abondance des niveaux de transcription et peut affecter les résultats de la RT-qPCR. Généralement, l’ADNg est détecté par qRT-PCR utilisant des amorces paires recuit aux régions intergéniques ou un intron du gène d’intérêt. Malheureusement, les annotations intron/exon ne sont pas encore connues de tous les gènes de vertébrés, bactéries, protistes, champignons, plantes et espèces de métazoaires invertébrés.
Nous présentons ici un protocole pour des échantillons de détection de la contamination de la génomique dans l’ARN à l’aide de l’ADN ribosomique (ADNr)-basé des amorces. La méthode est basée sur les caractéristiques uniques de l’ADNr : leur nature multigénique, séquences hautement conservées et haute fréquence dans le génome. Également comme étude de cas, un ensemble unique d’amorces ont été conçus basés sur la région conservée de l’ADN ribosomique (ADNr) de la famille des Poaceae. L’universalité de ces paires d’amorces a été testée par courbe fonte analyse et agarose électrophorèse sur gel. Bien que notre méthode explique comment ADNr amorces peuvent être appliquées pour le dosage de contamination ADNg dans la famille des Poaceae, il pourrait être facilement utilisé à d’autres espèces procaryote et eucaryote
Introduction
Explorant la régulation transcriptionnelle d’intéressant des ensembles de gènes ou de réseaux de signalisation est indispensable pour comprendre les mécanismes moléculaires complexes impliqués dans les événements biologiques1. Actuellement, l’analyse qPCR est le plus couramment approche pour gene expression études pouvant cibler (le génome) d’ADN ou ARN (le transcriptome) permettant l’analyse du transcriptome et de méthylome, respectivement. La transcription inverse (RT) suivie de qPCR est largement utilisée pour l’analyse du transcriptome qui mesurent les niveaux d’expression de gène dans divers domaines de la recherche biologique2. Par rapport à d’autres méthodes telles que l’hybridation traditionnelle du Nord, de la détection spécifique des tissus via in situ hybridation, essais de protection de la ribonucléase (RPA) et semi-RT-PCR, la précision, commodité, vitesse et une gamme dynamique de qPCR analyses sont hautement remarquable3,4. Il y a plusieurs facteurs importants qui doivent être considérés pour une quantification fiable des ARN messager (ARNm), y compris la qualité et la quantité d’ARN à partir de matériel. En outre, amplification non spécifique, l’efficacité de la RT-qPCR et l’efficacité de la PCR sont à considérer5,6.
La présence d’ADNg est un problème inhérent au cours de l’extraction de l’ARN, en partie, aux propriétés physiques et chimiques semblables de l’ADN et l’ARN7. En raison de l’identité de séquence génomique et l’ADN complémentaire (ADNc) dérivé des échantillons d’ADN messagère, amplification non spécifiques peut se produire, qui influeront sur l’exactitude des résultats de la RT-qPCR. L’ADNg restant conduira à une surestimation de l’abondance de cibler des ARNm dans gene expression analyse8.
Fondamentalement, l’amplicon non spécifiques pour la plupart découle formation de dimères amorce ou amplification de fond non spécifique en raison de la génomique, qui peuvent être évaluées en utilisant des échantillons de contrôle approprié. Ces échantillons sont aucun contrôle de modèle (NTC), aucun contrôle de la transcriptase inverse (NRT), respectivement. Étant donné que les niveaux de contamination de la génomique dans les échantillons étudiés sont différents et la sensibilité envers ADNg diffère grandement entre les gènes analysés, les contrôles NRT sont requis pour chaque paire d’échantillon/test. Bien que cela augmente considérablement le coût et le travail dans les études de profilage RT-qPCR, ces contrôles sont obligatoire,7,9.
Méthodes alternatives traitant ADNg contamination comprennent l’utilisation de paires d’amorces recuit aux régions intergéniques ou un intron du gène d’intérêt10et l’utilisation des amorces qui encadrent un grand intron ou couvrent une jonction exon-exon, c'est-à-dire les sites de recuit sont absents dans l’ARNm mature de la séquence1,4. Toutefois, les annotations intron/exon pour tous les gènes de nombreux vertébrés, bactéries, protistes, champignons, végétaux et métazoaires invertébrés sont encore connues. En outre, de nombreux organismes eucaryotes ont pseudogènes dérivés des évènements de duplication. En outre, conception d’amorce dans les introns ne garantit pas non-amplification de l’ADNg. Comme la chromatine accessibilité des régions génomiques de la DNase I varie, il est recommandé de concevoir les différentes amorces ciblant différents chromosomes10.
Les génomes des organismes eucaryotes peuvent englober jusqu'à un millier d’exemplaires de l’ADNr de gènes codant pour les sous-unités ribosomiques nécessaires pour la formation des ribosomes. Ces gènes ADNr sont souvent organisées en tableaux unique ou répété en tandem11. Polycistronique rRNA (Figure 1), y compris la grande sous-unité (LSU) et la petite sous-unité (SSU) est transcrits par l’ARN polymérase I (ARN j’ai). Le pre-rRNA qui en résultent est transformés en éliminant les deux régions de l’espaceur interne transcrit ITS1 et ITS2. Comme les produits finaux, trois matures rRNA, 17-18 s (SSU), 5. 8 s et 25-28 s ARNr (LSU) sont généré12. rADN est des représentants typiques d’une famille multigénique avec séquences hautement conservées. Ils se produisent avec une fréquence élevée dans le génome et sont potentiellement présents à plus d’une localisation chromosomique13. Le traitement de l’ARNr et la dégradation des entretoises transcrits sont un processus rapide dans le nucléole. En raison du degré élevé de répétition, le ratio du nombre de copies génomiques et détectables premolecules de RNA non transformées est inférieur par rapport aux séquences d’intron de faibles copies et précurseurs unspliced. Ces caractéristiques font de rADN, bien adapté pour une détection fiable et hautement sensible des ADNg contamination dans la plupart des eucaryotes et des procaryotes3.
Une nouvelle procédure de détection ADNg contamination dans les échantillons d’ARN est décrit ici. On présente un ensemble d’amorces universelles basé sur la séquence de l’ADNr conservée pour des analyses de génomique chez plusieurs espèces de graminées. La spécificité et l’universalité des amorces proposées ont été testés par l’analyse des courbes de fusion utilisant l’ADN comme gabarit. Notre protocole n’est pas seulement applicable aux Poaceae, mais pourrait facilement être adapté à d’autres espèces eucaryotes et procaryotes.
Protocole
Remarque : N’importe quel tissu peut être utilisé.
1. l’acide nucléique Extraction
- Mettre 100 mg d’échantillons de tissus dans un tube de 2,0 mL, ajouter deux perles de 5 mm en acier inoxydable et homogénéiser les tissus à 25-30 Hz pendant 30 s (durée de l’homogénéisation et la fréquence selon le type de tissu) de l’ARN et l’ADN.
- Isoler l’ARN total selon les instructions du fabricant.
- Isoler l’ADN total selon les instructions du fabricant.
- Contrôler la pureté et la quantité des échantillons RNA en mesurant l’absorbance à 260 et 280 nm.
- Contrôler la pureté et la quantité des échantillons ADN de mesurer l’absorbance à 260 et 280 nm.
NOTE : Bien que les acides nucléiques absorbent la lumière avec une longueur d’onde 260 nm (A260), l’absorbance de la lumière à la longueur d’onde de 280 (A280) peut être utilisé pour quantifier la quantité de protéines et phénols présent dans l’échantillon. Par conséquent, le ratio de A260/A280 nm peut être utilisé pour évaluer la pureté de l’ADN et l’ARN extrait d’un échantillon. A260/280 valeurs dans la plage de ≥1.8 et > 2.0 sont généralement considérés comme « pure » d’ADN et d’ARN, respectivement. Des valeurs plus faibles de A260/280 peuvent indiquer la contamination par des protéines ou de produits chimiques organiques. - Tester la qualité de l’ADN en exécutant une électrophorèse sur gel d’agarose de 0,7 %. Préparer le gel et exécuter dans 1 x tampon TRIS-borique EDTA (TBE : 89 mM Tris, d’acide borique 89 mM et 2 mM EDTA) à 100 V pour 30 min. haute qualité ADNg apparaît comme un sharp, high-molecular-weight (HMW) bande avec aucun frottis dans la gamme des molécules de faible poids moléculaire (HBPM).
- Vérifier les ARN pour la quantité, la pureté et l’intégrité dans des conditions dénaturantes par une électrophorèse en gel d’agarose thiocyanate (GTC) guanidine ou puce d’électrophorèse capillaire, selon les instructions du fabricant.
- Préparer le gel de la GTC en ajoutant 5 mM GTC à un gel d’agarose à 1 % des x TBE 1 standard après refroidissement la gélose à 60 ° C.
NOTE : GTC est toxique, donc la dispensent dans une hotte aspirante et porter un équipement de protection individuelle approprié. - Préparer l’ARN dénaturant tampon de charge : 95 % formamide, 10 mM EDTA pH 8.0, bleu de bromophénol 0,1 % 0,1 % xylène cyanole et du bromure d’éthidium 10 µL.
NOTE : Formamide, le bromure d’éthidium sont toxique et il devrait être dispensé sous une hotte. - Charge 1 à 5 µg d’ARN total dans l’ARN dénaturant tampon de charge, chaleur du mélange pendant 5 min à 70 ° C, placez-le sur la glace avant de le charger sur un gel et puis se séparent RNA sur gel GTC à 100 V pour 45 min. charge ADN ou ARN de marqueur de poids moléculaire comme une norme aux côtés l’échantillon de RNA.
- Tacher les gels au bromure d’éthidium et visualiser les bandes à l’aide de systèmes de capture d’image sous lumière ultraviolette. Chez les eucaryotes, l’ARN total intact à des conditions dénaturantes montrera au moins deux claire et nette ARNr bandes (28 s et 18 s) avec un ratio d’intensité de 2:1.
- Préparer le gel de la GTC en ajoutant 5 mM GTC à un gel d’agarose à 1 % des x TBE 1 standard après refroidissement la gélose à 60 ° C.
- Éliminer les traces d’ADNg par traitement à la DNase (DNase I RNase-libre). Ajouter dans un tube exempt de RNase dans le volume total de 10 µL : 0,1 - 1 µg d’ARN total, une unité de la DNase I et 1 µL de réaction 10 x tampon avec MgCl2. Incuber le mélange pendant 30 min à 37 ° C. Mettre fin à la réaction en ajoutant 1 µL de 50 mM EDTA et incubation à 65 ° C pendant 10 min.
- Éliminer les traces de l’ARN par gDNA extraits à l’aide de DNase gratuit RNase A, selon le protocole du fabricant. Ajouter 5 µL de RNase 10 mg/mL à l’ADN total et incuber à 37 ° C pendant 1 h. Banque d’ARN et ADN extraits à-80 ° C.
2. amorces d’ADNr région pour ADNg Assay
Remarque : La séquence de l’ADNr pleine longueur comporte deux régions (ITS1 et ITS2), qui sont éliminées dans la molécule d’ARNr matures par une série de clivages endonucléolytique et ensuite dégradent (Figure 1).
- Récupérer la séquence de nucléotides ADNr de NCBI (http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?) pour les espèces d’intérêt. Le meilleur mot clé pour rechercher la base de données est « espaceur interne transcrit. »
- L’entrée de la séquence nucléotidique de cible dans une recherche BLASTn pour trouver des régions de l’espaceur interne transcrit (SPI), SSU et LSU conservé des régions.
- Sélectionnez les amorces que soit flanquer une séquence de SPI ou qu’amplifier les séquences de secrétariats professionnels internationaux qui ne sont pas présents dans l’ARNr matures.
- Conception d’amorces flanquant les séquences ITS1 ou ITS2 : aligner les régions conservées de diverses espèces de ClustalW. Design des amorces spécifiques pour sa région flanquante après analyse spécifique/hétérospécifique taxons avec le logiciel AlleleID. L’amorce de deux paires d’amplification SSU-5. 8 s et 5,8 s-LSU amplicons peut être conçu basé sur les régions flanquantes de ITS1 et ITS2, respectivement. Parce que ces amplicons sont s’étendant sur toute la région de STI, la longueur de l’amplicon augmentera d’au moins 300 PB chez amplicons d’ADNg. Cette augmentation réduit la sensibilité.
- Flanquant ITS1 : Sélectionnez SSU et 5,8 s séquence de l’ARNr. Amorces sélectionnées pour les Poaceae sont : SSU, SF : CGTAACAAGGTTTCCGTAGGTG, r : GGTTCACGGGATTCTGCAAT. Cette paire d’amorces (SF : marche avant et arrière de gauche à droite :) amplifie la région partielle de SSU, la pleine longueur de ITS1 et la région partielle de 5,8 s ADNr.
- Flanquant ITS2 : Choisissez 5. 8 s et les séquences LSU. Amorces sélectionnées pour les Poaceae sont : la séquence consensus F: ATTGCAGAATCCCGTGAACC LSU, LR : TGCTTAAAYTCAGCGGGTAGYC. Cette paire d’amorces amplifie la région partielle de 5. 8 s, la pleine longueur de ITS2 et dans la région partielle de LSU (Figure 1).
Remarque : Dans le cas de la conception d’amorce issue des ITSs flanquant la région, les domaines hautement conservées de SSU, 5. 8 s et LSU ont été identifiés. Les amorces et inverses de 5,8 s ARNr a conçu un motif conservé dans les plantes à fleurs,14. On ont conçu les amorces et inverses sur la SSU et LSU conservent régions Poaceae, respectivement. La divergence des amorces SSU et LSU pour chaque espèce est donnée dans le tableau 1.
- Amorces d’amplification d’une séquence de SPI : dans ce protocole, des amorces de conception ITS1 basé sur Aeluropus sa séquence (numéro taxid NCBI : 110873). Pour les amorces, utiliser : avant : GGTATGGCGTCAAGGAACACT, inverse : ATAGCATCGCTGCAAGAGGT. Selon les amplicons générés par l’apprêt paires in silico, la taille doit être comprise entre 60 à 200 bp. C’est aussi la taille recommandée pour l’analyse de qPCR.
- Conception d’amorces flanquant les séquences ITS1 ou ITS2 : aligner les régions conservées de diverses espèces de ClustalW. Design des amorces spécifiques pour sa région flanquante après analyse spécifique/hétérospécifique taxons avec le logiciel AlleleID. L’amorce de deux paires d’amplification SSU-5. 8 s et 5,8 s-LSU amplicons peut être conçu basé sur les régions flanquantes de ITS1 et ITS2, respectivement. Parce que ces amplicons sont s’étendant sur toute la région de STI, la longueur de l’amplicon augmentera d’au moins 300 PB chez amplicons d’ADNg. Cette augmentation réduit la sensibilité.
- Prendre l’amorces tout en tenant compte de ces recommandations : contenu en GC : 40 à 60 %, de la longueur de l’amorce : longueur de base, produit PCR de 18 à 23 : bp 60-160 (spécialement pour son amorce), température de fusion (Tm) : 60 ° C, le Tm final pour les deux amorces ne diffèrent pas de plus de 5 ° C et la prim ERS ne sont pas complémentaires pour eux-mêmes ou en partenariat des amorces.
- Vérifier la spécificité et copie le numéro de l’apprêt. Effectuer des analyse in silico de la séquence de l’apprêt sélectionné par programme d’amorce-blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/).
- Ouvrez la page de présentation de Primer-BLAST. Entrez les deux séquences d’amorces dans la section paramètres de l’apprêt du formulaire. La spécificité de paire de primer vérifiant la section paramètres, entrez un nom de l’organisme (ou le nom de groupe d’organisme) et sélectionnez la base de données du génome. Ces paramètres donnent les spécificité des informations sur les paramètres de séquence et apprêt cibles notamment produit durée, position sur le chromosome et nombre de copies.
3. Effectuez qPCR étape pour la Validation des amorces ADNr avec matrices d’ADN
Remarque : La fonctionnalité des amorces conçues doit être validée en effectuant qPCR ADNg en utilisant comme modèle. Pour exécuter plusieurs réactions parallèles et réduire les erreurs de pipetage, la préparation d’un mélange maître est recommandée. Pour un mélange maître, préparer un volume équivalent au nombre total de mélange réactionnel plus environ 10 %.
- Préparer un mélange maître en mélangeant tous les éléments de réaction sauf la matrice d’ADN dans un tube de réaction PCR. Haut de gamme au besoin d’une réaction de préparer le Master mix : 5 µL SYBR green (SYBR) master mix (2 x), 0,3 apprêt µL (0,3 µM chacun d’apprêt avant et arrière) et ajuster le volume final à 10 µL d’eau exempte de RNase. Utiliser environ ≤200 ng 1 µL de modèle ADNg pour analyse.
NOTE : Décongeler, assembler et maintenir tous les réactifs, les composants et les mélanges de réaction sur la glace. - Aliquoter le mélange réactionnel dans une optique plaque 96 puits. Distribuer 1 µL d’ADNg dans chaque puits et puis couvrez-la avec plaque optique film d’étanchéité. Spin et placer dans le cycleur.
- Exécutez le test de qPCR sur un thermocycleur en temps réel dans les conditions suivantes : 10 min à 95 ° C suivie de 40 cycles de 95 ° C pour l’étape 15 s et 60 ° C pendant 1 min. effectuer d’opération d’acquisition de données à le recuit/extension de 60 ° C.
- Après l’intervention de l’amplification, sous réserve de toutes les réactions de PCR à une analyse des courbes de fusion avec la mesure de la fluorescence continue de 55 ° C à 95 ° C. En général, recueillir un point de données chaque cycle par une augmentation progressive de la température de 0,5 ° C par cycle.
NOTE : Inclure au moins 2 contrôles sans modèle (NTC) pour chaque mélange maître paire de primer. Effectuer tous les essais au moins trois répétitions. - Confirmer la spécificité de l’amorce par l’intermédiaire de l’analyse de courbe de fusion. Analyser les courbes avec le cycle de seuil simple et la méthode d’ajustement de la courbe soustraites.
Remarque : L’apparition d’un pic individuel indique un amplicon individuel uniform. Produits de dimère d’amorce peuvent apparaissent comme des pics individuels à des températures plus basses. - Valider la taille de chaque amplicon par électrophorèse sur gel d’agarose.
- Préparer un gel d’agarose de 3 % en mélangeant 3 g d’agarose avec 100 mL de tampon TBE (TBE : 89 mM Tris, d’acide borique 89 mM et 2 mM EDTA).
- Mélanger 5 à 10 µL du produit PCR avec ADN et 1-2 µL de 6 x tampon de charge. Charger le produit de la PCR à côté d’une échelle d’ADN sur gel d’agarose à 3 %. Effectuez une séparation par électrophorèse dans 1 x TRIS-borique EDTA-tampon à 100 V pendant 45 min.
- Tacher les gels avec du bromure d’éthidium ou tout autre agent intercalant et visualiser les bandes à l’aide de systèmes de capture d’image sous lumière ultraviolette.
Remarque : Les agents intercalants de l’ADN (par exemple, le bromure d’éthidium) sont cancérogènes et doivent être manipulés avec soin et distribués séparément. L’apparition d’une bande forte unique (en ce qui concerne la taille et sans apprêt-dimère ou amplification de fond artificiel) confirme la spécificité de l’amplicon.
4. ADNg Contamination MODE OPERATOIRE avec les modèles de l’ARN
Remarque : Après le traitement avec la DNase, l’ARN purifié échantillon est testé par des amorces spécifiques ADNr. En raison de la transformation de l’intron-like caractéristique des secrétariats professionnels internationaux lorsque ces régions sont utilisées pour l’amplification, aucun signal amplification ne devrait être détecté dans les échantillons d’ARN sans ADN. Sur cette base, si un signal amplification en qPCR est détecté ou une bande dans le gel d’agarose observée avec la taille attendue (estimée par une analyse in silico ), que cela doit être due à la contamination de l’ADNg. Les étapes effectuées dans cette section, sont similaires à l’article 3, sauf que cDNA de tous les échantillons sert de modèle au lieu de l’ADNg.
- Préparer un mélange maître en mélangeant tous les éléments de réaction sauf descripteur d’ARN dans un tube de réaction PCR. Maîtriser la combinaison de mélange pour une réaction : 5 µL SYBR master mix (2 x), 0,3 apprêt µL (0,3 µM chaque mélange primer avant et arrière) et ajuster le volume final à 10 µL d’eau exempte de RNase. Utiliser environ 500 ng modèle RNA dans 1 volume µL pour analyse.
- Aliquote master mix dans une optique plaque 96 puits. Pipetter 1 RNA µL à chaque puits et puis couvrez-la de plaque optique film d’étanchéité. Centrifugeuse et place dans cycleur.
NOTE : Inclure au moins deux contrôles NTC et deux contrôles positifs ADNg pour chaque dosage. Effectuer tous les essais en trois répétitions techniques. - Exécutez le test de qPCR sur un thermocycleur en temps réel dans les conditions suivantes : 10 min à 95 ° C suivie de 40 cycles de 95 ° C pour l’étape 15 s et 60 ° C pendant 1 min. effectuer d’opération d’acquisition de données à le recuit/extension de 60 ° C.
- Après l’intervention de l’amplification, sous réserve de toutes les réactions de PCR à une analyse des courbes de fusion avec la mesure de la fluorescence continue de 55 ° C à 95 ° C. Habituellement, recueillir un point de données chaque cycle par une augmentation progressive de la température de 0,5 ° C par cycle.
- Vérifier l’électrophorèse sur gel de tous les produits PCR en exécutant sur agarose à 3 %.
Remarque : L’apparition d’une bande ou pic dans la réaction de NTC est probablement liée à la formation de dimères apprêt que l'on voit habituellement à des températures basses dans la courbe de fusion, tandis que la présence d’une bande ou un pic dans les échantillons d’ARN est le résultat de la contamination de l’ADNg. Il est recommandé de tester d’abord tous les échantillons d’ARN par ADNr amorces, et puis non-ADN des échantillons contaminés sont utilisés pour des applications en aval comme la synthèse de cDNA, analyse de l’expression génique, etc..
5. RT-PCR étape pour la synthèse de cDNA et qPCR analyse
- Décongeler la DNase-traités des ARN et les réactifs de la synthèse de cDNA à température ambiante. Après décongélation, tournez en bas les réactifs. Ajouter 1 µg d’ARN et de 1 µL d’Oligo (dT) 18 amorce dans un tube exempte de nucléase. Ajuster le volume total de 12 µL d’eau exempte de RNase, mélanger doucement et ensuite stocker sur la glace.
- Faire fondre les structures secondaires de descripteur d’ARN en incubant la réaction à 65 ° C pendant 5 min. Spin down et refroidir le flacon sur la glace.
- Préparer le mélange maître de réaction (volume final 20 µL de chaque réaction) comme suit : 1 µL de la transcriptase inverse (200 U/µL), 4 µL de tampon de réaction (x 5), 1 µL d’inhibiteur de RNase (20 U/µL) et 2 µL de dNTP Mix (10 mM). Mélanger doucement et laisser refroidir le flacon sur la glace. Ajouter 19 µL dans le tube de préparation contenant de l’ARN.
- Incubez la réaction pendant 60 min à 42 ° C et puis incuber à 70 ° C pendant 5 min pour mettre fin à l’activité transcriptase inverse. Placer les réactions de RT sur la glace et passez à analyse de l’expression génique par procédure systématique de qPCR (comme expliqué dans la section 3 et 4).
Résultats
Nous proposons l’utilisation des amorces ADNr pour valider l’absence de contamination ADNg dans des échantillons d’ARN des tissus foliaires. L’organigramme de qPCR analyse et ADNg test de contamination est montré dans la Figure 2. Le protocole présenté, deux stratégies complémentaires ont été utilisés pour la conception d’amorce ADNr : amorces 1) propres à chaque espèce ont été sélectionnés dans des séquences de SPI et amorces 2) universelles ont été sélectionnés parmi les ITSs les régions flanquantes. Pour preuve de concept, nous avons conçu des amorces spécifiques pour Aeluropus littoralis et amorces universelles basées sur les espèces de Poacées, telle qu’elle figure dans le protocole. Les 5, 8 s amorces avant et arrière ont été sélectionnés, fondée sur un motif conservé de 14 paires de bases (bp) qui montre la similitude entre les plantes à fleurs, bryophytes et plusieurs ordres d’algues et champignons14. Les caractéristiques des amorces sont donnés dans le tableau 2. L’universalité du SSU, 5. 8 s et l’amorce de l’USJ ont été vérifiés par BLASTn, et amorce une homologie résultats sont présentés dans la Figure 3 comme un logo motif. La liste des espèces incluses dans l’analyse d’homologie, mais aussi les amorces divergentes pour chaque espèce sont donnés Table 1. Spécificité de l’apprêt a été check de Primer-BLAST. Pour les espèces pour lesquelles la séquence du génome entier est disponible, on estimait la localisation chromosomique des gènes de l’ADNr. Par exemple, dans l’ADNr Oryza sativa et l’Arabidopsis thaliana, les gènes sont situés sur deux chromosomes différents et Zea mays sur trois chromosomes différents.
validation de qPCR d’amorces ADNr a été effectuée avec l’analyse de la courbe des amplicons ITS1 et ITS2-flanc en utilisant l’ADN comme un modèle de fusion. Telle que présentée dans Figure 4 et Figure 5, spécificité apprêt a été confirmée expérimentalement par l’observation d’un seul pic pointu avec aucune formation de dimères amorce chez différentes espèces de Poacées, notamment Triticum aestivum, Hordeum vulgare, Oryza sativaet dans les dicotylédones, Medicago sativa, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia fabaet Arabidopsis thaliana. Le nouvel essai des produits d’amplification par la séparation électrophorétique taille montre une bande unique. Comme prévu, les bandes provenant des échantillons des différentes espèces variées en taille (Figure 6 a et 6 b). Fait intéressant, l’utilisation des amorces universelles spécialement conçu pour les trois espèces de graminées ne sont pas seulement utiles pour d’autres espèces de graminées, mais aussi pour d’autres espèces végétales telles qu’a. thaliana et pour un champignon endophyte viz. Piriformospora indica.
La validité de l’amorce spécifique conçu (ITS1) a également été confirmée par qPCR dans a. littoralis utilisant ADNg comme gabarit. Un seul pic avec aucune formation de dimères apprêt a été observé. Étonnamment, l’apprêt a. littoralis ITS1 (comme l’amorce spécifique) a généré une seule bande forte non seulement en a. littoralis, mais aussi pour toutes les autres espèces étudiées à l’exception de Nicotiana tabacum et Trifolium alexandrinum, qui produit des deux groupes (Figure 6). Le test de contamination ADNg a été réalisé par les amorces STI ou STI-accompagnement dans tous les échantillons d’ARN. Une représentation schématique de la plaque d’amplification dans le test de contamination ADNg et l’interprétation des résultats sont présenté à la Figure 7.
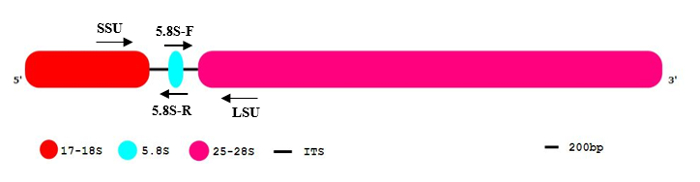
Figure 1 : Le schéma général d’organisation de séquence des ADNr eucaryotes.
Le segment de l’ADNr eucaryotes contient 17-18 (rouge), 5. 8 s (bleu) et 25-28 s ARNr (rose). Les espaceurs internes transcrits (ITS) sont indiquées par des lignes noires. 5´and 3´ indiquer l’orientation de la molécule d’ADN. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
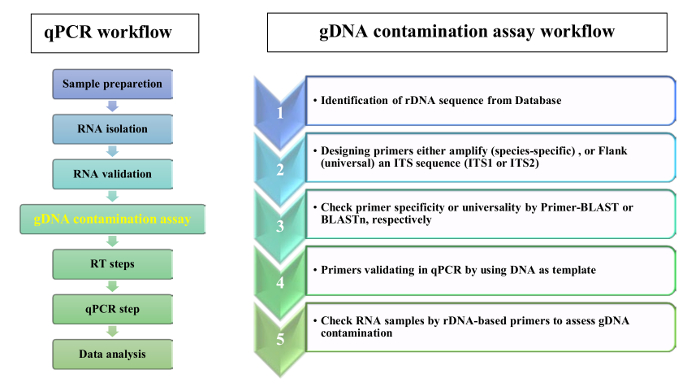
Figure 2 : "Workflow" pour un test de contamination RT-qPCR et ADNg. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
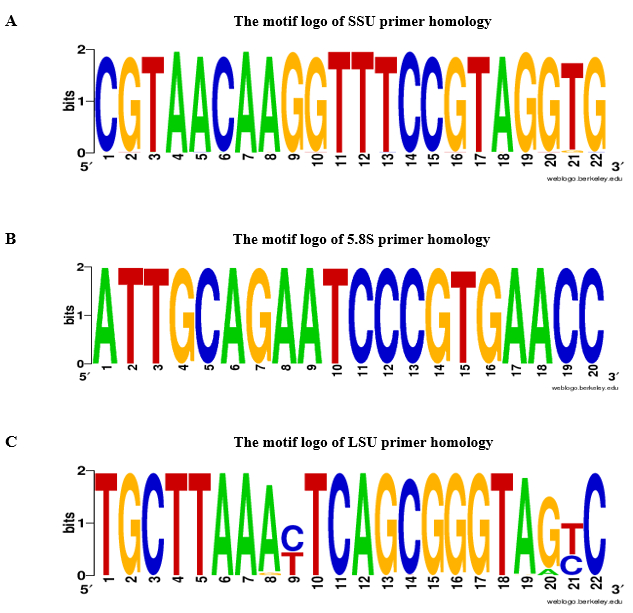
Figure 3 : logo Motif de SSU A., B. 5. 8 s et C. LSU apprêt homologie. Pour SSU, 5, 8 s, et amorces LSU, le logo motif a été construit par BLASTn basé sur 2 000 enregistrements de plante verte (numéro taxid NCBI : 33090) avec un cut-off e-valeur ≤ 10-10. A-adénine, Thymine-T, G-Guanine, Cytosine-C. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
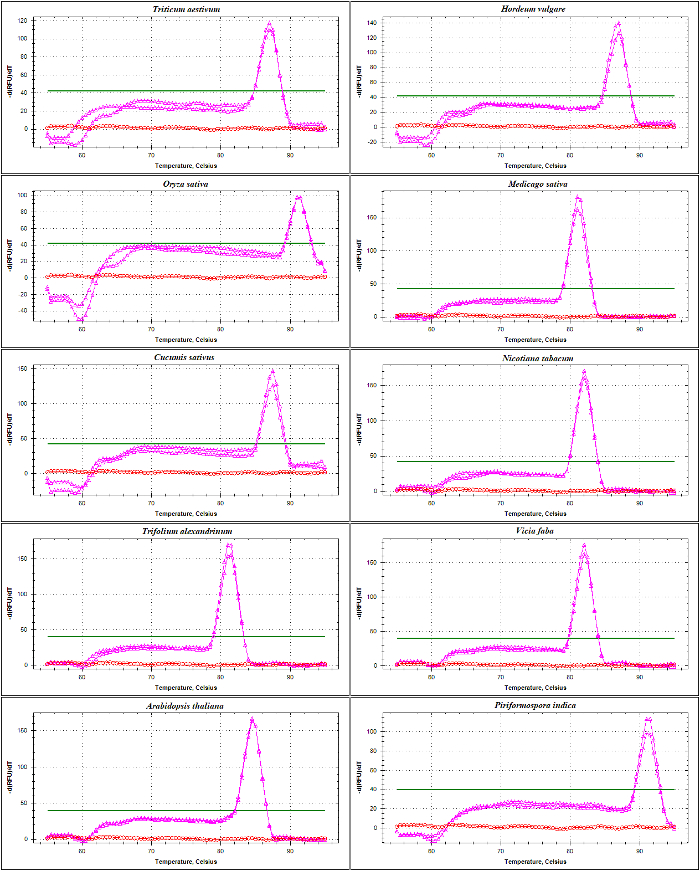
Figure 4 : L’analyse de courbe de fusion d’amplicon flanquante ITS1 chez différentes espèces.
Cette amplicon, amplifié par la SSU et 5,8 s-amorces R, contient une partie de la séquence de la région de codage de 17-18, la séquence entière de ITS1 et séquence partielle de 5. 8 s. Sont les courbes de fusion des amplicons générés (rose) et NTC (rouge) de Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia faba, Arabidopsis thaliana et Piriformospora indica. La ligne plate "BOLD" indique le seuil de référence. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
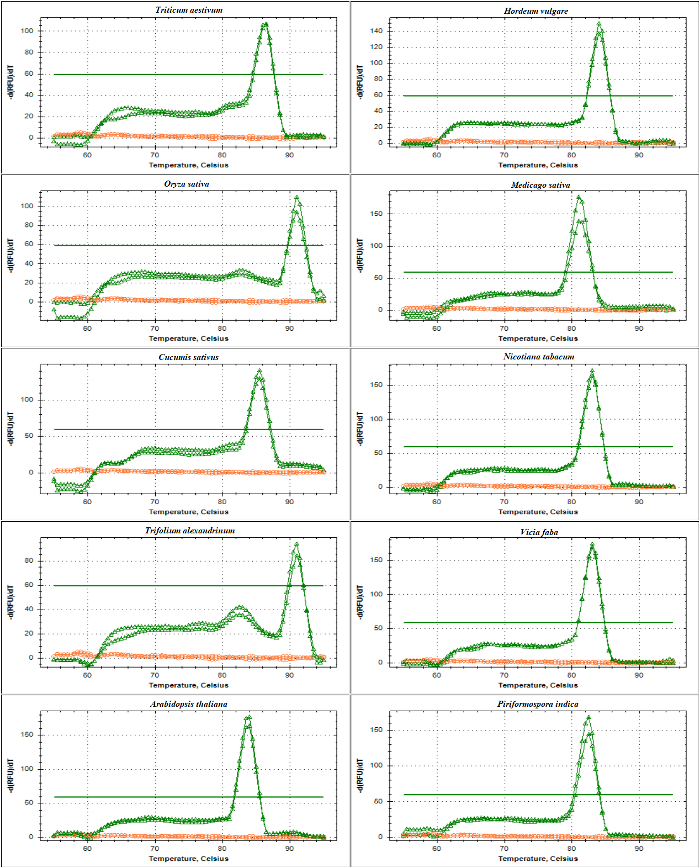
Figure 5 : L’analyse de courbe de fusion d’amplicon flanquante ITS2 chez différentes espèces.
Cette amplicon est générée par l’utilisation de 5. 8 s-F et LSU amorces. L’amplicon décrite contient des séquences d’une partie de 5,8 S, la séquence entière de ITS2 et une séquence partielle de 25-28 s. Sont les courbes de fusion des amplicons (vert) et NTC (rouge) générée à partir de Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia faba, Arabidopsis thaliana et Piriformospora indica. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
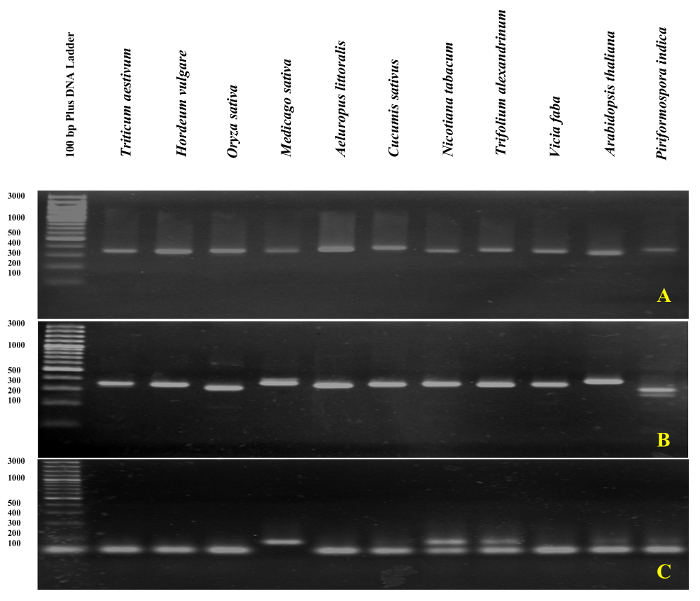
Figure 6 : Analyse de gel d’Agarose du produit PCR ADNr.
L’amplicon de ITS1-flancs (A), ITS2-flancs (B) et ITS1 (C) ont été exécutés sur gel d’agarose à 3 %. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 7 : Caractéristiques semblables à Intron de SPI sont envisageables pour concevoir des amorces qui peuvent détecter la contamination ADNg.
Une crête ou la bande avec la taille attendue en qPCR analyse indiquent ADNg contamination de l’échantillon de RNA. UNK : échantillon inconnu, pos : contrôle positif, NTC : commande sans modèle. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Apprêt | Genre | Taxid ID | Espèces | Apprêt divergente | ||
| SSU | Arabidopsis | 3701 | kamchatica, thaliana et lyrata | - | ||
| Vicia | 3904 | villosa, americana, unijuga, amoenane, amurensis, craccamal, pseudo-orobus, multicaulis, japonica, ramuliflora et faba | ||||
| Trifolium | 3898 | alexandrinum, montanum, resupinatum et repens | - | |||
| Nicotiana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, clevelandii, nesophila, solanifolia, cordifolia, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, petunioides, obtusifolia, miersii, pauciflora, atténuer, acuminés, linearis, alata, sylvestris, rustica et suaveolens | - | |||
| Cucumis | 3655 | anguria, melo et sativus | CGTAACAAGGTTTCCGTAGGKG | |||
| Aeluropus | 110873 | - | Aucun apprêt trouvé | |||
| Medicago | 3877 | sativa, lupulina, pamphylica, lunata, rostrées, plicata et truncatula | - | |||
| Oryza | 4597 | sativa, glumipatula, rufipogon barthii glaberrima ponctuée, longistaminata, meridionalis, nivara, meridionalis et longistaminata | - | |||
| Triticum | 4564 | aestivum, urartu et monococcum | - | |||
| Hordeum | 4512 | vulgare, bulbosum, marinum, brevisubulatum et bogdanii | - | |||
| LSU | Arabidopsis | 3701 | petraea, thaliana et lyrata | TGCTTAAACTCAGCGGGTAATC | ||
| Vicia | 3904 | sylvatica, tetrasperma, sativa, hirsute, sepium, parviflora, cracca, lathyroides, orobus, orobus, bithynica et faba | TGCTTAAATTCAGCGGGTAGCC | |||
| Trifolium | 3898 | prétexte, nigrescens, resupinatum occidentale, subterraneum, strictum, ochroleucon, glomeratum, squamosum, ornithopodioides et repens | TGCTTAAATTCAGCGGGTAGCC | |||
| Nicotiana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, clevelandii, nesophila, solanifolia, cordifolia, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, petunioides, obtusifolia, miersii, pauciflora, atténuer, acuminés, linearis, alata, sylvestris et suaveolens | TGCTTAAACTCAGCGGGTAGTC | |||
| Cucumis | 3655 | Melo, ritchiei et javanica | TGCTTAAACTCAGCGGGTAGTC | |||
| Aeluropus | 110873 | lagopoide, pungens et littoralis | TGCTTAAATTCAGCGGGTAATC | |||
| Medicago | 3877 | ruthenica, sativa, lupulina, arabica polymorpha et minima | TGCTTAAATTCAGCGGGTAGCC | |||
| pamphylica, lunata, rostrée et plicata | TGCTTAAACTCAGCGGGTAGTC | |||||
| Oryza | 4597 | sativa, glumipatula, rufipogon, barthiial, glaberrima, Australiensis, officinalis, australiensis, ridleyi, malampuzhaensis, alta, nivara, rufipogon, meridionalis et longistaminata | TGCTTAAACTCAGCGGGTAGTC | |||
| Triticum | 4564 | aestivum, spelta, turgidum, dicoccoides, petropavlovskyi, urartu et monococcum | TGCTTAAACTCAGCGGGTAGTC | |||
| Hordeum | 4512 | vulgare, bulbosum, murinum, secalinum, brevisubulatum et bogdanii | TGCTTAAACTCAGCGGGTAGTC | |||
| Une amorce dégénérée est définie comme système IUPAC pour la nomenclature des nucléotides | ||||||
Tableau 1 : La liste des espèces considérées pour la cueillette des amorces ADNr.
Site de liaison du SSU en comparaison de site de fixation de LSU homologie de séquence supérieur sur donné genre.
| Longueur de l’amplicon | Zone d’amplification | Séquence | Nom de l’apprêt | Amplicon | |
| 332 - 405 bp | Séquence partielle de SSU, toute séquence de ITS1 et séquence partielle de 5. 8 s | CGTAACAAGGTTTCCGTAGGTG | SSU | ITS1-flancs | |
| GGTTCACGGGATTCTGCAAT | 5. 8 S-R | ||||
| 318 - 361 bp | Séquence partielle de 5. 8 s, une séquence complète de ITS2 et séquence partielle de LSU | ATTGCAGAATCCCGTGAACC | 5. 8 S-F | ITS2-flancs | |
| TGCTTAAAYTCAGCGGGTAGYC | LSU | ||||
| 100 - 200 bp | ITS1 | GGTATGGCGTCAAGGAACACT | ITS1-F | ITS1 | |
| ATAGCATCGCTGCAAGAGGT | ITS1-R | ||||
Tableau 2 : Séquences d’amorce.
Discussion
Analyse de l’expression génique par PCR quantitative a été largement appliquée dans ces dernières années. Le principal avantage de cette méthode rapide, rentable et automatisé est son résultat précis et exact. Cependant, gagner des avantages optimaux de ces avantages exige une compréhension claire de la configuration des paramètres utilisés pour l’expérience de qPCR. Pour obtenir un résultat fiable en qPCR analyse d’expression de gène, il est nécessaire éviter l’amplification non spécifique qui résulte de la contamination-dimère d’amorce ou ADNg dans l’échantillon des RNA3,15. Il est prévu que les niveaux de transcription d’ARN vont être surestimés l’ADNg contamination8. Ici, les caractéristiques uniques d’un gène d’ADNr a été jugé pour un dosage de contamination ADNg dans des échantillons d’ARN.
Des propriétés de base de l’ADNr utilisés dans le présent protocole : Les gènes ribosomiques se composent de deux secrétariats professionnels internationaux, à savoir ITS1 et ITS2 et les trois gènes codant ARNr, 17-18, 5. 8 s et 25-28 s sous-unité12. Deux régions ITS ne font pas partie de la séquence codante des sous-unités ribosomiques. Ils sont éliminés par au moins trois activités enzymatiques pour traiter le précurseur à mûrir ARNr : une activité exonucléase endonucléase et hélicase. Comme l’ARN ribosomique est transcrit comme une transcription polycistronique, un produit primaire contenant le SPI est certainement présent. Le traitement est très rapide et se déroule dans le nucléole, et la quantité de molécules précurseurs détectables contenant l’EVI est inférieure au seuil de détection de la méthode de qPCR. Par conséquent, lorsqu’ITS1 ou ITS2 sont amplifiées par ITS flanquant les amorces, aucune amplification ne peut être détectée dans des échantillons d’ARN sauf ADNg contamination est présente. Le nombre des ADNr gènes dans le génome d’organismes eucaryotes était estimé à inclure jusqu'à un millier d’exemplaires, qui est disposé dans des tableaux simples ou tandem sur les chromosomes11. Dans ce protocole, nous proposons une alternative, au lieu de NRT, pour détecter la contamination ADNg, qui est utilisée dans chaque réaction/essai.
Avantages et les limites en ce qui concerne les méthodes existantes : NRT est généralement utilisée pour tester si l’échantillon de RNA préparé est propre ou contaminé par ADNg. Depuis ADNg contamination n’est pas uniformément répartie entre les différents échantillons d’ARN, et la sensibilité de réaction de l’ADNg est considérablement affectée par les gènes analysés, NRT contrôles sont nécessaires pour chaque échantillon/dosage paire7,15. Ceci ajoutera considérablement le coût et le travail lors de la manipulation de plusieurs échantillons simultanément3,9. Autres méthodes alternatives dans la littérature comprennent l’utilisation des amorces spécifiques pour la détection de l’ADNg intron, ou des amorces de conception qui flanquent un intron ou couvrent une jonction exon-exon. Les limitations de ces méthodes proviennent de l’indisponibilité des informations de séquence intron, annotation incomplet de la structure de l’intron/exon et l’absence d’introns dans les gènes ou les pseudogènes d’intérêt1,4,10 . En raison de l’évolution, rADN existe en tant que familles de gènes multigénique et hautement conservée. Elles sont très abondantes dans le génome et présent sur différents chromosomes13. Comparées aux autres gènes codant ou nonconding, le rADN montre la meilleure solution pour la détection de la contamination de l’ADNg. Dans les analyses transcriptomiques comparative, la normalisation de qPCR données par calibrateur ARNr n’est pas recommandée pour certaines questions, telles que des différences de cDNA préparation (polyA amorçage vs hexamère aléatoire d’amorçage), de grandes différences dans l’abondance des ARNm et ARNr , et biogenèse différente qui peut-être générer trompeuse résultats10,16. Cependant, les problèmes que nous venons de parler sont un avantage pour le test de contamination ADNg. Par exemple, en ce qui concerne la plus grande abondance de site ciblage dans le génome et localisation sur des chromosomes différents, amorces ADNr améliorent considérablement la sensibilité de détection de l’ADNg en comparaison avec les méthodes existantes.
Polyvalence des ADNr basée sur d’autres organismes : rADN est une famille de gènes bien étudiés dans la plupart des organismes. La méthode proposée d’ADNr représente un système simple, économique et très sensible pour les tests de contamination ADNg qui peut être facilement adaptée à d’autres organismes eucaryotes et procaryotes (Protocole N° 2 - 5). Une étude de cas, nous avons démontré ici l’utilité de cette méthode dans certaines espèces de Poceae (Figure 4 et Figure 5). Les amorces utilisées montrent un taux élevé de transférabilité à d’autres espèces de Poceae en raison de la structure hautement conservée de sous-unités ADNr entre les espèces. Cette question devient encore plus importante lorsque suffisamment d’informations séquence génomique n’est pas disponible pour la conception d’amorce. Ainsi, ITS flanquante amorces d’une espèce peuvent être utilisés dans une espèce apparentée. En outre, les 5, 8 s-amorces de F/R ont été cueillies fondée sur un motif conservé qui montre la similitude élevée dans la plupart de plantes à fleurs14. Bien que les techniques de séquençage à haut débit en permanence augmentent le nombre de génomes connus, l’annotation de l’exon-intron de la plupart des organismes n’est pas terminée, et donc il n’est souvent pas possible de concevoir des amorces pour traverser une frontière exon-exon. Notre méthode explique comment axée sur l’ADNr amorces peuvent être appliquées pour le dosage de contamination ADNg qPCR analyse des procaryotes et des eucaryotes dans le but d’éliminer chers contrôles NRT dans chaque combinaison d’essai/amorces.
Déclarations de divulgation
Les auteurs n’ont aucun intérêt financier concurrentes.
Remerciements
Cette recherche a été financée par la génétique et l’Institut de biotechnologie agricole de Tabaristan (GABIT) Sari agricoles et l’Université des ressources naturelles (SANRU). Le groupe de recherche junior Abiotic Stress génomique a été financé par IZN (Centre interdisciplinaire de recherche sur les cultures végétales, Halle (Saale), Allemagne. Nous remercions Rhonda Meyer pour une lecture critique du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| Maxima SYBR Green / ROX qPCR Master Mix (2X) | Thermo Scientific | K0221 | |
| TissueLyser II | QIAGEN | 85300 | |
| RevertAid H Minus First Strand cDNA Synthesis Kit | Thermo Scientific | K1631 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| 96 well WHT/CLR | Bio-Rad | HSP9601 | |
| Microseal B film | Bio-Rad | MJ-0558 | |
| Low tube strip CLR | Bio-Rad | TLS0801 | |
| Flat cap strips | Bio-Rad | TCS0803 | |
| NanoDrop 2000 | Peqlab | ND-2000 | |
| RNaseZAP | Ambion | 9780 | |
| Centrifuge | Eppendorf | 5810 R | |
| Agilent RNA 6000 Nano Kit | Agilent Technologies | 5067-1511 | |
| 2100 Electrophoresis Bioanalyzer | Agilent Technologies | G2939AA | |
| RNase A, DNase and Protease-free | Thermo Scientific | EN0531 | |
| DNase I, RNase-free | Thermo Scientific | EN0523 | |
| TRIZOL Reagent | Ambion | 15596026 | |
| CFX96 Touch Real-Time PCR Detection System | BIO RAD | 1855195 | |
| PCR tube, 0.2 mL, RNase-free | Stratagene | Z376426 | |
| Guanidine thiocyanate for molecular biology | Sigma-Aldrich | G9277 | |
| Agarose - Nucleic Acid Electrophoresis | Sigma-Aldrich | A9414 | |
| Boric Acid for molecular biology | AppliChem | A2940 | |
| bromophenol blue | AppliChem | A2331 | |
| ethidium bromide | AppliChem | A1151 | |
| Gel documentation system | BIO RAD | Gel Doc 2000 |
Références
- Bustin, S. A., Nolan, T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction. J Biomol Tech. 15 (3), 155-166 (2004).
- Gutierrez, L., Mauriat, M., Pelloux, J., Bellini, C., Van Wuytswinkel, O. Towards a systematic validation of references in real-time RT-PCR. Plant Cell. 20 (7), 1734-1735 (2008).
- Hashemi, S. H., Nematzadeh, G., Ahmadian, G., Yamchi, A., Kuhlmann, M. Identification and validation of Aeluropus littoralis reference genes for Quantitative Real-Time PCR Normalization. J Biol Res (Thessalon). 23 (1), 18 (2016).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J. Mol. Endocrinol. 25 (2), 169-193 (2000).
- Andersen, C. L., Jensen, J. L., Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64 (15), 5245-5250 (2004).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40 (7), e51 (2012).
- Galiveti, C. R., Rozhdestvensky, T. S., Brosius, J., Lehrach, H., Konthur, Z. Application of housekeeping npcRNAs for quantitative expression analysis of human transcriptome by real-time PCR. RNA. 16 (2), 450-461 (2010).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40, e51 (2012).
- Caldana, C., Scheible, W. R., Mueller-Roeber, B., Ruzicic, S. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3 (1), 7 (2007).
- Lawrence, R. J., Pikaard, C. S. Perspectives Chromatin Turn Ons and Turn Offs of Ribosomal RNA Genes. Cell Cycle. 3 (7), 880 (2004).
- Boisvert, F. M., van Koningsbruggen, S., Navascues, J., Lamond, A. I. The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8 (7), 574-585 (2007).
- Alvarez, I., Wendel, J. F. Ribosomal ITS sequences and plant phylogenetic inference. Mol Phylogenet Evol. 29 (3), 417-434 (2003).
- Jobes, D. V., Thien, L. B. A conserved motif in the 5.8 S ribosomal RNA (rRNA) gene is a useful diagnostic marker for plant internal transcribed spacer (ITS) sequences. Plant Mol Biol Report. 15 (4), 326-334 (1997).
- Padhi, B. K., Singh, M., Huang, N., Pelletier, G. A PCR-based approach to assess genomic DNA contamination in RNA: Application to rat RNA samples. Anal Biochem. 494, 49-51 (2016).
- Dheda, K., et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques. 37 (1), 112-114 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon