Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Actionneur à base de cellules musculaires cardiaques et Biorobot auto-stabilisant - Partie 2
Dans cet article
Résumé
Dans cette étude, un actionneur biologique et un biorobot de stabilisation auto-stabilisable avec des bras en porte-à-faux élastomères fonctionnalisés sont ensemencés avec des cardiomyocytes, cultivés et caractérisés pour leurs propriétés biochimiques et biomécaniques au fil du temps.
Résumé
Au cours des dernières années, des dispositifs hybrides constitués d'une cellule vivante ou d'un composant tissulaire intégré à un squelette mécanique synthétique ont été développés. Ces appareils, appelés biorobots, sont alimentés uniquement par la force générée par l'activité contractile du composant vivant et, en raison de leurs nombreux avantages inhérents, pourrait être une alternative aux robots conventionnels entièrement artificiels. Ici, nous décrivons les méthodes de semence et de caractérisation d'un actionneur biologique et d'un biorobot qui a été conçu, fabriqué et fonctionnalisé dans la première partie de cet article en deux parties. L'actionneur biologique fabriqué et les dispositifs de biorobot composés d'une base de polydiméthylsiloxane (PDMS) et d'un cantilever à film mince ont été fonctionnalisés pour la fixation de cellules avec la fibronectine. Après la fonctionnalisation, les cardiomyocytes néonatals de rat ont été ensemencés sur le bras en porte-à-faux PDMS à haute densité, ce qui a donné lieu à une feuille cellulaire confluente. Les appareils ont été imagés tous les jours et le mouvement du cantiLes bras de levier ont été analysés. Le deuxième jour après l'ensemencement, nous avons observé la flexion des bras en porte-à-faux en raison des forces exercées par les cellules pendant les contractions spontanées. Lors de l'analyse quantitative de la flexion en porte-à-faux, une augmentation graduelle du stress superficiel exercée par les cellules au fur et à mesure de leur maturation au cours du temps a été observée. De même, nous avons observé le mouvement du biorobot en raison de l'actionnement du bras en porte-à-faux PDMS, qui a agi comme une aileron. Lors de la quantification des profils de natation des dispositifs, on a observé différents modes de propulsion qui ont été influencés par l'angle de repos de la nageoire. La direction du mouvement et la fréquence de battement ont également été déterminées par l'angle de repos de l'aileron et une vitesse de nage maximale de 142 μm / s a été observée. Dans ce manuscrit, nous décrivons la procédure pour peupler les dispositifs fabriqués avec des cardiomyocytes, ainsi que pour l'évaluation de l'actionneur biologique et de l'activité de biorobot.
Introduction
Les Biorobots sont des dispositifs basés sur des cellules vivantes qui sont incorporées dans un squelette mécanique qui est habituellement composé de matériaux doux et élastiques tels que PDMS ou hydrogels 1 . Les cellules subissent des contractions rythmiques, soit spontanément, soit en réponse à des stimuli, et fonctionnent ainsi comme actionneur. La puissance générée par la contraction des cellules entraîne différents biorobots. Les cellules cardiaques de mammifères (cardiomyocytes) et les cellules musculaires squelettiques sont souvent utilisées pour l'actionnement de biorobot en raison de leurs propriétés contractiles. Outre les cellules des cardiomyocytes et des muscles squelettiques, d'autres types de cellules, comme les tissus musculaires 2 et les tissus musculaires explantés 3 , ont été utilisés. Les tissus musculaires des insectes permettent le fonctionnement des actionneurs biologiques à température ambiante.
La fonction et la performance d'un biorobot sont principalement déterminées par la force et la cohérence de l'actionneur biologique ( c.-à-d.. Cellules musculaires), tandis que la structure du squelette mécanique détermine principalement les mécanismes de la locomotion, de la stabilité et de la puissance. Étant donné que ces dispositifs sont uniquement alimentés par des forces générées par les cellules, il n'y a pas de polluants chimiques ou de bruits d'exploitation. Par conséquent, ils forment une alternative économe en énergie à d'autres robots classiques. Plusieurs sources de littérature ont discuté des différentes méthodes pour intégrer les cellules vivantes et les tissus dans les biorobots 1 , 4 , 5 . Des progrès dans les techniques de microfabrication et de génie tissulaire ont permis le développement de biorobots qui peuvent marcher, s'accrocher, nager ou pomper 5 , 6 . En général, les cellules sont cultivées directement sur le squelette mécanique (polymère) en tant que feuille de cellule confluente ou elles sont moulées dans des structures d'actionnement tridimensionnelles dans des échafaudages tels que des anneaux et des bandes. Le plus souvent, les biorobots sontFabriqués à l'aide de feuilles de cardiomyocytes 6 , 7 , car ces cellules ont une capacité innée à présenter une contraction spontanée sans stimuli externe. D'autre part, les rapports sur les feuilles de cellules musculaires squelettiques sont limités en raison de leur besoin de stimuli pour initier des contractions in vitro afin d'initier une dépolarisation de la membrane 8 .
Ce protocole décrit d'abord comment semer des cardiomyocytes sur un actionneur biologique fonctionnalisé constitué d'un mince PDMS en porte-à-faux. Il décrit ensuite en détail l'ensemencement et l'analyse des profils de natation. Le cantilever est fonctionnalisé avec une protéine adhésive cellulaire telle que la fibronectine et est ensemencé confluent avec les cardiomyocytes. En tant que cellules ensemencées sur le contrat de l'appareil, elles font que le porte-à-faux se plie et agit comme actionneur. Au fil du temps, à mesure que les cellules arrivent à maturité, nous analysons les changements de stress superficiel sur l'appareil en analysant des vidéos de laFlexion en porte-à-faux. L'actionneur biologique développé ici peut être utilisé pour déterminer les propriétés contractiles de n'importe quel type de cellule, comme les fibroblastes ou les cellules de tiges pluripotentes induites, puisqu'elles subissent une différenciation.
Une grande partie de la recherche antérieure sur les biorobots a été axée sur le développement d'actionneurs biologiques, tandis que l'optimisation de l'architecture et des capacités fonctionnelles de Biorobot a été largement négligée. Récemment, quelques études ont démontré la mise en place de modes de natation dans les biorobots qui s'inspirent de la nature. Par exemple, les biorobots de natation avec le mouvement à base de flagelles 6 , la propulsion des méduses 9 et les rayons bio-hybrides 4 ont été conçus. Contrairement à d'autres travaux dans la littérature, nous nous concentrons ici sur la variation des propriétés du squelette mécanique pour créer une structure auto-stabilisante. Le biorobot développé dans cette étude est capable de maintenir un ton, un roulement constant et unProfondeur de morsion pendant qu'il nage. Ces paramètres peuvent être modifiés en faisant varier l'épaisseur de chaque composite de base. Les étapes de fabrication impliquées dans le développement de l'actionneur PDMS, du biorobot immergé et de la fonctionnalisation du dispositif sont décrites en détail dans la partie 1 de cet article en deux parties, ainsi que dans notre travail récent 7. La technique développée ici peut ouvrir la Mode de développement de biorobots nouveaux et hautement efficaces pour diverses applications, telles que la livraison de fret.
Le processus d'isolement suivi dans cette étude est similaire au processus décrit dans un travail antérieur 10 , ainsi que dans les travaux récemment publiés 7 . Les méthodes de microfabrication utilisées pour fabriquer les actionneurs PDMS et les dispositifs biorobot sont décrites en détail dans la partie 1 de ce manuscrit en deux parties. La section de protocole de ce manuscrit décrit les étapes de l'implantation de cardiomyocytes sur le PDMS fabriqué aCtuator et le biorobot après leur fonctionnalisation avec des protéines adhésives cellulaires.
Protocole
Toutes les procédures décrites ici ont été menées à l'aide d'un protocole approuvé et conformément aux règlements du Comité institutionnel pour les soins et l'utilisation des animaux de l'Université de Notre Dame.
1. Semence cellulaire et culture
- Avant de commencer, préparez les éléments requis: un petit entonnoir, des pipettes et un milieu Eagle Eagle modifié par Dulbecco complété par 10% de sérum bovin fœtal (FBS) et 1% d'antibiotique pénicilline (DMEM complet).
- Prenez les flacons T-25 avec le dispositif fonctionnalisé (actionneur biologique ou biorobot). Reportez-vous à la section 4 de la partie 1 de ce manuscrit en deux parties pour plus de détails concernant la préparation, la fonctionnalisation et le stockage de l'appareil avant l'ensemencement cellulaire.
- Préparez un entonnoir, qui peut être fabriqué en roulant une petite feuille de plastique carrée. Placez-le sur l'actionneur biologique ou le biorobot à l'intérieur du ballon T-25. Ajustez le diamètre de l'extrémité plus large pour s'adapter à l'ensemble du dispositifEt la hauteur afin qu'il soit bien ajusté lorsque le haut de la fiole est serré.
- Pour les biorobots, utilisez un aimant pour maintenir le dispositif en place au bas de la fiole tout au long du processus d'ensemencement.
Note: Ici, un seul aimant de disque de néodyme (1,26 "de diamètre) a été utilisé, mais n'importe quel aimant de taille et de résistance similaires peut également être utilisé pour maintenir le biorobot avec la base composite magnétique nickel-PDMS. - UV stérilisez la feuille de plastique pendant au moins 30 minutes avant utilisation.
- Pour les biorobots, utilisez un aimant pour maintenir le dispositif en place au bas de la fiole tout au long du processus d'ensemencement.
- Assurez-vous qu'il n'y a pas un grand écart entre la base de l'entonnoir et le flacon.
- Resuspendre les cardiomyocytes dans le DMEM complet à une densité de 1,6 x 10 7 cellules / ml et déposer lentement 400 μL de suspension sur l'appareil à travers l'entonnoir. Utilisez un hémocytomètre ou tout autre compteur de cellules pour déterminer le nombre de cellules obtenues.
- Retournez lentement le système dans l'incubateur sans déranger l'appareil et l'entonnoirHin. Culture pendant 24 h à 37 ° C.
- Après la période d'incubation, retirez lentement l'entonnoir, lavez doucement l'échantillon avec du PBS et rallumez le ballon avec 10 mL de DMEM frais.
Remarque: Pour les biorobots, retirez l'aimant afin que l'appareil soit à flot.
2. Caractérisation biochimique
- Dosage du flux de calcium
Remarque: Le dosage du flux de calcium est effectué pour évaluer l'interconnectivité cellulaire. La procédure de chargement des cellules avec le colorant fluorescent, spécifique à l'ion calcium, suit le processus décrit dans un protocole 11 établi précédemment.- Tout d'abord, préparer les matériaux requis, l'ester fluo-4-acétoéthylique (AM), le polyol tensioactif non ionique (voir la Table des matériaux ) et la solution salée de Tyrode.
- À l'aide de longues pinces, transférez doucement le dispositif du ballon de culture à une boîte de Petri de 35 mm avec 2 ml de solution de sel de Tyrode Tion.
- Dans un tube de centrifugation séparé, prendre 1 mL de solution de Tyrode chaude (chauffée à 37 ° C) et ajouter 3-5 μL de colorant de calcium fluo-4 AM (concentration de travail: 3-5 μM) et une partie égale de tensioactif non ionique Polyol (concentration de travail: 0,2%). Remplacer la solution d'échantillon par une solution de Tyrode chaude complétée par le colorant indicateur de calcium, fluo-4 et 0,2% d'agent tensioactif non ionique. Incuber pendant 25 à 30 min à 37 ° C.
- Retirez la solution de colorant et lavez doucement l'échantillon avec une solution fraîche de Tyrode. Réincuber l'échantillon dans 2 mL de DMEM frais complet pendant 30 minutes supplémentaires à 37 ° C avant l'imagerie.
Remarque: Les résultats de cet essai et les vidéos connexes sont fournis dans le travail précédemment publié 7 .
- Immunofluorescence
Note: La double immunocoloration de tous les échantillons a été réalisée selon un protocole établi précédemment> 12.- Préparez d'abord 10% de sérum de chèvre (GS) dans une solution saline tamponnée au phosphate (PBS), 4% de paraformaldehyde (PFA) dans du diH 2 O, 0,1% de détergent cellulaire (voir la Table des matières ) dans du PBS, des anticorps primaires (anti- - conjuguer des anticorps monoclonaux à la troponine-I et des anticorps monoclonaux anti-lapins-43), des anticorps secondaires (conjugué Alexa 594 et conjugué Alexa 488 d'IgG de chèvre anti-souris (H + L) et DAPI.
Attention : le paraformaldéhyde est cancérigène. - Retirez l'échantillon d'intérêt du ballon et laissez-le doucement deux fois avec du PBS. Reportez-vous à la section 4 de la partie 1 de ce manuscrit en deux parties pour plus de détails concernant la préparation et la fonctionnalisation des échantillons.
- Ajouter une gouttelette de PBS sur une petite lamelle (diamètre: 12 mm ou 15 mm). Tenir doucement la base de l'appareil avec une pince à épiler et couper les minces bras PDMS (en porte-à-faux, figure 1 ) en utilisant des ciseaux à partir de l'extrémité où il se connecte au sommet du basE. Transférez les bras en porte-à-faux sur la gouttelettes avec le côté collé vers le haut vers le haut. La goutte PBS empêchera les cellules de se sécher.
- Réparez les échantillons avec 4% de PFA et effectuez une double immunocoloration des échantillons, comme décrit précédemment 12 .
- Après l'immunocoloration, montez les échantillons sur une lame de verre propre en utilisant un réactif de montage anti-fade et mis de côté, sans perturbation, dans l'obscurité pendant 24 h.
- Répétez la procédure pour tous les échantillons.
Note: Les résultats de cet essai et les images connexes sont discutés en profondeur dans le travail précédemment publié 7 .
- Préparez d'abord 10% de sérum de chèvre (GS) dans une solution saline tamponnée au phosphate (PBS), 4% de paraformaldehyde (PFA) dans du diH 2 O, 0,1% de détergent cellulaire (voir la Table des matières ) dans du PBS, des anticorps primaires (anti- - conjuguer des anticorps monoclonaux à la troponine-I et des anticorps monoclonaux anti-lapins-43), des anticorps secondaires (conjugué Alexa 594 et conjugué Alexa 488 d'IgG de chèvre anti-souris (H + L) et DAPI.
3. Imagerie
- Placez le ballon T-25 droit dans un incubateur de CO 2 et prépare le système d'imagerie à l'intérieur de l'incubateur. Enregistrez l'appareil à l'aide d'une caméra (voir la table des matières ) avec un zoom (voir la table des matières ). Pour une source lumineuse,Utilisez une bande de lumières LED.
Remarque: Une bande de LED à lumière blanche a été utilisée ici, mais toutes les LED normales fonctionneront également. - Connectez l'appareil photo à un système d'exploitation et ouvrez le logiciel spécifique à la caméra (voir la table des matières ). Cliquez sur l'image de la caméra sous l'onglet "Fichier", dans le panneau supérieur, pour ouvrir toutes les options de la caméra et choisissez la caméra correcte.
- Choisissez "en direct" dans la liste des onglets du panneau supérieur du logiciel.
- Mettre l'image manuellement dans la mise au point en ajustant la molette de l'objectif. Sélectionnez "Recadrer vers la région d'intérêt (ROI)" dans le panneau supérieur. Ensuite, dessinez manuellement un rectangle dans le cadre vidéo, en joignant le dispositif d'actionnement biologique et les bras en porte-à-faux pour marquer le ROI.
Remarque: Choisir un ROI approprié minimise la taille des fichiers image.- Dans le cas des biorobots, capturez l'écran entier afin d'enregistrer le mouvement de natation de l'appareil.
NOTE: Il n'est pas nécessaire de générer un ROI pour les biorobots
- Dans le cas des biorobots, capturez l'écran entier afin d'enregistrer le mouvement de natation de l'appareil.
- Avant de commencer l'enregistrement, sélectionnez «Réglages de la caméra» à partir de l'un des onglets du panneau supérieur de l'écran. Réglez la fréquence d'images en ajustant l'exposition et le rapport de pixel de l'image en direct en faisant glisser la barre pour chacune ou en saisissant manuellement les valeurs. Réglez les taux d'image à environ 30 ± 2 fps.
Remarque: La modification de l'exposition et du rapport de pixels modifie la luminosité et le contraste de l'image en direct. - Cliquez sur le bouton "Enregistrer" dans le panneau supérieur du logiciel pour commencer à enregistrer les vidéos des actionneurs avec une résolution de 1000 x 1000 pixels pour une précision de 30 s. Répétez le processus pour tous les échantillons.
4. Analyse d'image des actionneurs biologiques sur une base stationnaire
- Analyser les images en utilisant un logiciel de programmation ( par exemple, Matlab) exécutant le script personnalisé. Voir la table des matières Rong > et le fichier supplémentaire pour plus de détails.
Remarque: Le script affiche chaque image des vidéos enregistrées, reçoit l'entrée de la souris de l'utilisateur pour enregistrer la coordination des points du porte-à-faux sur les images, calcule le diamètre et le centre du cercle qui passe les points entrés par l'intermédiaire d'un ajustement carré et Exporte toutes les données entrées et calculées pour une utilisation ultérieure.- Ouvrez le logiciel de programmation en cliquant sur l'icône. Cliquez sur "Fichier" -> "Ouvrir" dans la barre de menus en haut et sélectionnez le fichier script .m pour l'analyse d'image. Assurez-vous que les images TIFF enregistrées sont dans le même dossier que le fichier .m. Cliquez sur "Exécuter" pour exécuter le script.
Remarque: Un affichage interactif apparaîtra pour modifier. - Appuyez sur "play" pour lancer le programme actuel. Cliquez sur le bouton "Ouvrir" et localisez le fichier TIFF qui va être analysé.
- Cliquez sur "base & #34; , Puis cliquez sur le point où le porte-à-faux s'attache à la base en haut; Appuyez sur Entrée. Cela placera un marqueur carré sur l'image pour chaque image pour désigner l'emplacement de la base en porte-à-faux.
- Cliquez sur le bouton «Scale» puis cliquez sur un bord du cordon de verre. Apportez le pointeur de la souris sur le côté opposé du cordon de verre et appuyez sur "Entrée".
Remarque: Cela devrait tracer une ligne qui mesure le diamètre du cordon de verre. Étant donné que le cordon de verre a un diamètre de 3 mm, cela correspond à 3 mm aux pixels affichés. - Cliquez sur le bouton "Analyser". Cliquez le long du cantilever à une courte distance du premier marqueur carré qui représente la base en porte-à-faux.
- Cliquez sur le bouton "Analyser". Ensuite, cliquez le long du cantilever à une courte distance du premier marqueur carré qui représente la base en porte-à-faux. Continuez à cliquer le long du porte-à-faux, y compris la pointe, et appuyez sur Entrée lorsqueterminé. Cela placera un "x" sur chaque point cliqué sur le porte-à-faux.
Remarque: En fonction de la coordination du marqueur carré et des marqueurs x, le centre et le diamètre d'un cercle seront calculés à l'aide d'une fonction d'ajustement carré minimal (voir le fichier supplémentaire attaché pour le script utilisé). Le cercle, qui passe les marqueurs x et le créateur carré, sera superposé à l'image automatiquement. - Vérifiez si le cercle superposé trace correctement le profil en porte-à-faux.
Remarque: Lorsque le porte-à-faux est très plat, il est difficile de juger si le profil en porte-à-faux est correctement tracé. Reportez-vous à la Figure 3 . - Cliquez sur le bouton "image suivante". Cela passera à l'image suivante dans le fichier TIFF. La base et l'échelle sont déjà définies à partir de l'étape précédente.
- Répétez les étapes 4.1.5 à 4.1.7 jusqu'à ce que toutes les images du fichier TIFF aient été complétées. Une fois que tous les cadres ont été préparésSsed, cliquez sur le bouton "Exporter".
Remarque: Cela produira un fichier de tableur avec le nom de fichier TIFF pour le porte-à-faux qui vient d'être analysé. Modifiez le nom du fichier pour inclure quel côté (gauche ou droite) du porte-à-faux a été analysé.
- Ouvrez le logiciel de programmation en cliquant sur l'icône. Cliquez sur "Fichier" -> "Ouvrir" dans la barre de menus en haut et sélectionnez le fichier script .m pour l'analyse d'image. Assurez-vous que les images TIFF enregistrées sont dans le même dossier que le fichier .m. Cliquez sur "Exécuter" pour exécuter le script.
- Calcul du stress dans la feuille de calcul.
- Calculez le stress de surface, "σ", sur le porte-à-faux en utilisant l'équation suivante:
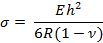
Où E, R, v et h sont le module de Young, le rayon de courbure, le rapport de Poisson et l'épaisseur en porte-à-faux, respectivement.
Remarque: L'épaisseur du porte-à-faux peut être modifiée pour modifier la sensibilité. Dans cette étude, les valeurs étaient les suivantes: E = 750 kPa, v = 0,49 et h = 25 μm 13 , 14 . - Calculez le stress de surface en utilisant l'équation à l'étape 4.2.1. Ouvrez le.Xls tableur. Notez que la sortie a plusieurs colonnes montrant d'abord les coordonnées x et y de la base et du cercle et ensuite le rayon de courbure. Calculez les coordonnées x et y de chaque point en cliquant sur celles-ci.
Remarque: Tracer les contraintes sur les cadres d'images temporelles montre des changements dans la force exercée sur le porte-à-faux dans le temps. Les creux montrent le stress sur le cantilever pendant la relaxation des cardiomyocytes ou le stress statique exercé sur le porte-à-faux en raison des forces de traction cellulaire. Les pics montrent le stress dynamique sur le cantilever, qui a été exercé par le battement des cardiomyocytes. Cette valeur correspond à la quantité maximale de force générée par la contraction des cardiomyocytes.
- Calculez le stress de surface, "σ", sur le porte-à-faux en utilisant l'équation suivante:
5. Analyse des Biorobots de natation
- Enregistrez la position du biorobot en utilisant un logiciel d'analyse d'image.
Remarque: consultez la liste des matériaux pour le logiciel utilisé dans thEst une section.- Ouvrez le logiciel d'analyse d'image ( p. Ex., ImageJ). Appuyez sur "Fichier" et "Ouvrir" et sélectionnez le fichier vidéo de natation Biorobot. Cliquez sur "OK" et laissez le programme charger le fichier. Ouvrez le logiciel.
- Dans la vidéo biorobot chargée, recherchez une référence de dimensions connues ( par exemple, le cordon de verre de 3 mm de diamètre qui a été incorporé dans l'actionneur biologique).
Remarque: Tout objet ayant une dimension connue fonctionnera. Cela déterminera les rapports pixel-à-longueur dans chaque vidéo. - Utilisez l'outil "Straight" pour tracer une ligne sur le cordon de verre. Cliquez sur "Analyser" et sélectionnez "Définir l'échelle". Réglez le champ "Distance connue" à "3 000 μm" et cliquez sur "OK".
Remarque: cela définira les coordonnées x et y en micromètres. - Choisissez un point sur le périphérique qui ne diminue pas entre les images pour servir de marqueur.
Remarque:Il est recommandé de choisir un coin de la base. - Pointez le point sélectionné dans 5.1.4 sur la première image. Enregistrez les coordonnées x et y sur la feuille de calcul.
- Retournez à la fenêtre du logiciel d'analyse d'image et appuyez sur la flèche vers la droite pour passer à la prochaine image. Pointez sur le marqueur à nouveau (à partir de l'étape 5.1.4) et enregistrez les coordonnées x et y sur la feuille de calcul.
- Répétez l'étape 5.1.6 pour toutes les images.
- Calculez les paramètres de natation du biorobot en utilisant la feuille de calcul des coordonnées 7 .
- Calculez la période entre les images à partir de la fréquence d'images connue de chaque vidéo.
- Calculez le changement dans les coordonnées x et y entre les images pour trouver la distance déplacée, y compris la distance totale.
- Calculez l'amplitude de contraction à partir du changement maximal le long de l'axe des y. Déterminer la fréquence de battement pour chaque biorobot de l'inverse de la période entre deux contractions.
- CAugmentez la vitesse de baignade de chaque appareil à partir du temps total et de la distance déplacée dans la direction x.
- Répétez l'étape 5.2 pour chaque vidéo de Biorobot qui a été analysée.
- Normaliser chaque paramètre mesuré.
Remarque: normalisez toutes les valeurs pour mieux visualiser les différences. Ce protocole démontre une normalisation par rapport à un biorobot de mode horizontal avec des contractions à basse fréquence (LF horizontale) ( Figure 4 ) 7 .
6. Analyse de l'expression des protéines
Note: Les échantillons montés préparés aux étapes 2.2.4 et 2.2.5 ont été imagés à l'aide d'un microscope confocal. Les images ont été acquises séquentiellement à 20X, 40X et 60X en trois canaux simultanément: 460 nm, 488 nm et 594 nm. Un ensemble de 5 images a été capturé à un grossissement de 40X, à partir de différentes positions pour chaque échantillon, et chaque canal a été enregistré en tant qu'individu .TIFFfichier. Le réglage de l'exposition a été déterminé par le grossissement de l'objectif utilisé et a été mis en constante pour toutes les captures à ce grossissement.
- Ouvrez le logiciel d'analyse d'image et sélectionnez "Fichier" -> "Ouvrir" pour charger les images.
- Dessinez un polygone rectangulaire sur le cadre d'image pour marquer le ROI. Sélectionnez "Analyser" -> "Mesurer" pour mesurer l'intensité fluorescente moyenne.
- Répétez l'étape 6.2 pour collecter les mesures d'intensité de tous les échantillons et calculez l'intensité moyenne respective pour chaque condition.
Note: Ici, une condition différente se réfère à différents points de temps, comme dans le jour 1, le jour 2 et le jour 6. - Exportez les résultats sur une feuille de calcul pour une analyse statistique supplémentaire et pour la production de parcelles de données.
Résultats
L'actionneur biologique constitué d'un mince PDMS en porte-à-faux (25 μm d'épaisseur) et de cardiomyocytes constitue le noyau de la natation biorobot, comme le montrent les schéma et la capture d'écran des dispositifs de la figure 1 . Les cellules commencent à présenter des contractions après 24 h en culture et la flexion des bras en porte-à-faux a été observée au jour 2. Le profil latéral de l'appareil a été enregistré tous les jours...
Discussion
La procédure décrite ici décrit une méthode d'ensemencement réussie pour les actionneurs et les biorobots à base de PDMS, ce qui facilite la fixation de cardiomyocytes. En outre, le processus d'acquisition d'image et l'analyse ultérieure qui caractérise le comportement des cellules et la performance des appareils ont été décrits.
Nous avons observé une contraction spontanée des cellules sur les bras en porte-à-faux après 24 h; L'intensité des contractions ...
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
MT Holley est soutenu par le programme Graduate Fellows du Louisiana Board of Regents, et C. Danielson est soutenu par le Howard Hughes Medical Institute Professors Program. Cette étude est soutenue par NSF Grant No: 1530884.
matériels
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Cardiomyocytes (primary cardiac cells) | Charles River | NA | Isolated from 2-day old neonatal Sprague Dawley rats |
| Dulbecco’s modified eagle’s media (DMEM) | Hyclone Laboratories | 16750-074 | with 4500 mg/L glucose, 4.0 mM L-glutamine, and 110 mg/L sodium pyruvate |
| Fetalclone III serum | Hyclone industries, GE | 16777-240 | Fetal bovin serum (FBS) |
| Dulbecco’s phosphate buffer (PBS) | Sigma-Aldrich | D1408-100ML | |
| Penicillin-G sodium salt | Sigma-Aldrich | P3032 | |
| Goat serum | Sigma-Aldrich | G9023 | |
| 4,6-diamidino-2-phenylindole dihydrocholride powder (DAPI) | Sigma-Aldrich | D9542 | |
| Fibronectin from bovine plasma | Sigma-Aldrich | F1141 | Solution (1 mg/ml) |
| Calcein-AM and ethidium homodimer-1 kit (Live/Dead Assay) | Molecular Probes | L3224 | |
| Calcium Fluo-4, AM | Molecular Probes | F14217 | calcium indicator dye |
| Tyrodes salt solution | Sigma-Aldrich | T2397 | buffer solution |
| Pluronic F-127 | Molecular Probes | P3000MP | nonionic surfactant-20 % solution in Dimethylsiloxane (DMSO) |
| 16% Parafomaldehyde | Electron microscopy | 15710 | Caution: Irritant and combustible |
| Triton x-100 | Sigma-Aldrich | X-100 100 mL | cell lyses detergent, (4-(1,1,3,3-Tetramethylbutyl)phenyl-polyethylene glycol, t-Octylphenoxypolyethoxyethanol, Polyethylene glycol tert-octylphenyl ether) |
| ProLong gold antifade reagent | Molecular Probes | P10144 | Mounting agent |
| Alexa Fluor 594 Phalloidin | Molecular Probes | A12381 | Actin filament marker |
| Goat anti-rabbit IgG (H+L) secondary antibody, Alexa Fluor 594 conjugate | Molecular Probes | A-11012 | |
| pha | Molecular Probes | A-11001 | |
| Anti-connexin 43 antibody | Abcam | ab11370 | Gap junction marker |
| Anti-cardiac troponin I antibody | Abcam | ab10231 | Contractile protein |
| 16% EM grade paraformaldehyde solution | Electron microscopy | 100503-916 | |
| Polydimethylsiloxane (PDMS) | Elsevier | Sylgard 184 | |
| Materials and Equipment | |||
| Camera | Thor Labs | DCC1545M | |
| LED light strip | NA | NA | Any white LED without spectrum emission |
| Confocal microscope | Nikkon C2 | NA | Confocal microscope with three filter set. |
| Zooming lens | Infinity | Model# 252120 | |
| Software | |||
| Matlab | Mathworks | NA | Used in Section 4) for biological actuator analysis. |
| Image J | National Institute of Health | NA | Java-based image processing software. Used in Section 5) for biorobot analysis. Free Image Processing and Analysis software in java. (https://imagej.nih.gov/ij/) |
| Thor Cam | Thor Labs | NA | Camera operating software |
Références
- Feinberg, A. W. Biological Soft Robotics. Annu. Rev. Biomed. Eng. 17, 243-265 (2015).
- Akiyama, Y., et al. Room Temperature Operable Autonomously Moving Bio-Microrobot Powered by Insect Dorsal Vessel Tissue. PLOS ONE. 7, 38274 (2012).
- Herr, H., Dennis, R. G. A swimming robot actuated by living muscle tissue. J. NeuroEng Rehabil. 1, 6 (2004).
- Park, S., et al. Phototactic guidance of a tissue-engineered soft-robotic ray. Science. 353 (6295), 158-162 (2016).
- Cvetkovic, C., et al. Three-dimensionally printed biological machines powered by skeletal muscle. Proc. Natl. Acad. Sci. 111, 10125-10130 (2014).
- Williams, B. J., Anand, S. V., Rajagopalan, J., Saif, M. T. A. A self-propelled biohybrid swimmer at low Reynolds number. Nat. Commun. 5, (2014).
- Holley, M. T., Nagarajan, N., Danielson, C., Zorlutuna, P., Park, K. Development and characterization of muscle-based actuators for self-stabilizing swimming biorobots. Lab. Chip. 16, 3473-3484 (2016).
- Hopkins, P. M. Skeletal muscle physiology. Contin Educ Anaesth Crit Care Pain. 6, 1-6 (2006).
- Nawroth, J., et al. A tissue-engineered jellyfish with biomimetic propulsion. Nat Biotechnol. 30 (8), 729-797 (2012).
- Ehler, E., Moore-Morris, T., Lange, S. Isolation and Culture of Neonatal Mouse Cardiomyocytes. J. Vis. Exp. JoVE. (79), e50154 (2013).
- Bers, D. M. Calcium Fluxes Involved in Control of Cardiac Myocyte Contraction. Circ. Res. 87, 275-281 (2000).
- Shin, S. R., et al. Carbon-Nanotube-Embedded Hydrogel Sheets for Engineering Cardiac Constructs and Biological actuators. ACS Nano. 7, 2369-2380 (2013).
- Park, J., et al. Real-Time Measurement of the Contractile Forces of Self-Organized Cardiomyocytes on Hybrid Biopolymer Microcantilevers. Anal. Chem. 77, 6571-6580 (2005).
- Tamayo, J., et al. Quantification of the surface stress in microcantilever biosensors: revisiting Stoney's equation. Nanotechnology. 23, 475702 (2012).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat. Methods. 10, 781-787 (2013).
- Louch, W. E., Sheehan, K. A., Wolska, B. M. Methods in Cardiomyocyte Isolation, Culture, and Gene Transfer. J. Mol. Cell. Cardiol. 51, 288-298 (2011).
- Alford, P. W., Feinberg, A. W., Sheehy, S. P., Parker, K. K. Biohybrid thin films for measuring contractility in engineered cardiovascular muscle. Biomaterials. 31, 3613-3621 (2010).
- Sfakiotakis, M., Lane, D. M., Davies, J. B. C. Review of fish swimming modes for aquatic locomotion. IEEE J. Ocean. Eng. 24, 237-252 (1999).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon