Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Optique pH Quantification d’intracellulaire dans l’épithélium des tubules Malpighian Drosophila melanogaster ayant un pH fluorescents génétiquement codé indicateur
Dans cet article
Résumé
Transport ionique cellulaire peut souvent être évaluée en contrôlant le pH intracellulaire (pHj’ai). Génétiquement Encoded pH-indicateurs (GEpHIs) fournissent une quantification optique du pH intracellulaire dans les cellules intactes. Ce protocole détaille la quantification du pH intracellulaire par cellulaires ex vivo live-imagerie de Malpighi de Drosophila melanogaster avec pHerry, une pseudo-ratiométrique codé génétiquement-indicateur de pH.
Résumé
Transport épithélial ionique est vitale pour l’homéostasie ionique systémique ainsi que la maintenance des gradients électrochimiques cellulaires essentiels. PH intracellulaire (pHi) est influencée par de nombreux transporteurs ioniques et donc suivi pHj’ai est un outil utile pour évaluer l’activité de transporteur. PH de codé génétiquement moderne-indicateurs (GEpHIs) fournit des optique quantification de pHj’ai dans les cellules intactes à l’échelle cellulaire et subcellulaire. Ce protocole décrit la quantification en temps réel des cellulairesj’ai régulation du pH dans les tubes de Malpighi (MTs) de Drosophila melanogaster par ex vivo live-image de pHerry, un GEpHI pseudo-ratiométrique avec un pKun bien adapté pour suivre les changements de pH dans le cytosol. Mouche adulte extraite MTs sont composés de sections morphologiquement et fonctionnellement distinctes de l’épithélium de la couche de cellules individuelles et peuvent servir comme un modèle d’étude du transport épithélial accessible et génétiquement tractable. GEpHIs offre plusieurs avantages par rapport aux colorants fluorescents conventionnels sensibles au pH et électrodes sélectives. GEpHIs pouvez étiqueter les populations cellulaires distincts autant éléments promoteurs appropriées sont disponibles. Cet étiquetage est particulièrement utile dans les ex vivo, in vivoet in situ préparations, qui sont par nature hétérogènes. GEpHIs permettent aussi de quantification de pHi dans les tissus intactes au fil du temps sans avoir besoin d’externalisation de traitement ou d’un tissu colorant répétées. Le principal inconvénient du GEpHIs actuel est la tendance à agréger dans les inclusions cytosoliques en réponse aux dommages de tissu et de construire la surexpression. Ces lacunes, leurs solutions et les avantages inhérents au GEpHIs sont illustrés dans ce protocole à travers l’évaluation de basolatérale transport de protons (H+) dans les cellules principales et stellaires fonctionnellement distinctes de mouche extraite MTs. Les techniques et les analyses décrites sont facilement adaptables à une grande variété de préparations de vertébrés et d’invertébrés, et la sophistication du dosage peut passer de laboratoires aux complexe détermination du flux ionique par l’intermédiaire de transporteurs spécifiques d’enseignement.
Introduction
Ce protocole vise à décrire la quantification du pH intracellulaire (pHi) à l’aide d’un indicateur pH codé génétiquement (GEpHI) et démontrer comment cette méthode peut être utilisée pour évaluer le transport H+ basolatérale chez un insecte modèle (D. melanogaster) structure rénale, les tubules Malpighian (MT). MTs servent les organes excréteurs de la mouche à fruit et sont fonctionnellement similaires aux mammifères néphron à plusieurs égards essentiels1. MTs sont disposées sous forme de 2 paires de tubules (antérieures et postérieures) dans le thorax et l’abdomen de la mouche. Le tube épithélial unicellulaire de chaque marqueur est composé de cellules métaboliquement actives principales avec distinct apicales (luminal) et polarité basolatérale (hémocoele) ainsi que des cellules étoilées intercalées. MTs antérieures sont composés de 3, morphologiquement, fonctionnellement, et débats développemental distincts, notamment la première dilatés segment segment transitoire et sécrétoire segment principal, qui relie à l' uretère2. À l’échelle cellulaire transport trans épithélial ionique dans la lumière s’effectue par une V-ATPase de la membrane plasmique apicale3 et un échangeur d’alcali-métal/H+ comme un basolatérale++de Na -K-ATPase4, vers l’intérieur-redresseur K+ canaux5, Na+-driven Cl−/HCO3− échangeur (NDAE1)6et++de Na -K-2 Cl− cotransporteur (NKCC ; Ncc69)7, tandis que les cellules étoilées médient Cl– et8,9de transport de l’eau. Ce système physiologique complex mais accessible offre d’excellentes possibilités pour l’étude des mécanismes de transport des ions endogènes lorsqu’il est combiné avec les divers ensembles d’outils génétiques et comportementaux de la drosophile.
La justification de ce protocole était de décrire un système génétiquement malléable pour étudier le transport épithélial ionique avec un potentiel pour l’intégration de la cellule au comportement et à l’exportation d’outils à d’autres systèmes de modèle. Expression de pHerry10, un GEpHI dérivé d’une fusion de vert écliptique super sensibles au pH pHluorin11,12 (EERH) et rouge mCherry insensible à pH13, dans MTs permet quantification des transports H+ cellules MT uniques à travers la haute K+/nigericin calibration technique14. Alors que de nombreux transporteurs ioniques équivalents H+ , quantification du pH intracellulaireje sert une représentation fonctionnelle du mouvement ionique via une variété de transporteurs. Le système de modèle drosophile MT aussi offre de puissants outils génétiques en tissu-spécifique transgène15 et RNA interférence (ARNi)16 expression qui peut être combinée avec l’imagerie cellulaire et dosages de l’ensemble-orgue17 , 18 , 19 de fonction tubule pour créer un ensemble d’outils robuste grâce à l’intégration verticale des molécules au comportement. Cela contraste avec les nombreux autres protocoles pour évaluer biologie épithéliale, comme historiquement ces mesures sont sont appuyés sur complexe et intimidant des électrodes sélectives micro-dissection et sophistiqué20,21, et sensibles au pH cher colorants22 avec exigences restrictives chargement et mauvaise spécificité cellulaire en tissus hétérogènes. GEpHIs ont été utilisés intensivement mesurer pHj’ai dans une variété de types de cellule23. Les premiers travaux exploités la pH-sensibilité inhérente de Green Fluorescent Protein (GFP) pour surveiller pHj’ai dans les cellules épithéliales en culture24 , mais les deux dernières décennies ont vu GEpHIs utilisé dans les neurones25, glia26, champignons27 , et28des cellules végétales. La combinaison de la possibilité d’un ciblage cellulaire des constructions génétiques par le biais de l’expression des GAL4/UAS système15 et l’accessibilité physiologique de la drosophile MT cela fait une préparation idéale pour les enquêtes de pHj’ai règlement et transport épithélial ionique.
régulation du pHj’ai a été étudiée pendant des décennies et est essentielle à la vie. La préparation de MT vous propose un modèle robuste pour enseigner la physiologie de la régulation du pHj’ai mais aussi effectuer un sophistiqué enquêtes de pHi règlement ex vivo et in vivo. Ce protocole décrit la quantification du mouvement H+ à travers la membrane basolatérale des cellules épithéliales de la drosophile MT à l’aide de la NH4Cl l’acide impulsion technique21de chargement, mais comme l’indicateur de pH est génétiquement codé, ces méthodes et leur cadre théorique peuvent être appliqués toute préparation se prêtent à la transgénèse et création d’images en direct.
Protocole
Toutes les étapes dans ce protocole conforme aux lignes directrices Mayo Clinic (Rochester, MN) l’utilisation des animaux.
1. voler élevage
- Augmenter les mouches et ensemble traverse selon élevage standard29.
Remarque : L’expression du Rapporteur Fluorescent par le système de GAL4/UAS est proportionnelle à la température et donc élever la température peut être ajustée pour modifier le niveau d’expression. Tandis que les niveaux d’expression élevée conduisent souvent à un meilleur rapport signal sur bruit cette condition est également associée à des agrégats cytosoliques et organites accrues lors de l’utilisation de GFP pour fusion de protéine fluorescente rouge (DP) construit comme pHerry10, 30,31. Si l’agrégation est inévitable, la quantification est encore possible en effectuant point étalonnages dans chaque expérience et normaliser les données, telles qu’un rapport de fluorescence de 1,0 correspond à pHj’ai 7.0 (Voir l’étape 7.4 Remarque sur l’étalonnage ci-dessous). - Valeur des croisements des homozygotes PACCR-GAL432 mâles homozygotes SAMU-pHerry10 femelles vierges et homozygotes c724-GAL42 mâles homozygotes SAMU-pHerry femelles vierges permettant l’imagerie de pHj’ai dans les cellules principales et des cellules étoilées de la MT, respectivement. 6 place SAMU-pHerry femelles 3 mâles GAL4 dans les flacons de frais d’aliments et de la laisser s’accoupler à 28 ° C.
Remarque : Les larves doivent être évidents au bout de 4 jours et adultes commenceront à eclose autour du jour 10. - Recueillir les mouches femelles sur eclosion et mis de côté à l’âge de 10 jours à 28 ° C.
NOTE : Le calendrier de l’expérimentation peut être réglé pour correspondre à toute épreuves comportementales restrictives (par exemple les Ramsay sécrétion dosage17,19) qui vont être corrélées à l’imagerie intracellulaire pH en temps réel. Mouches mâles peuvent être utilisés, mais les tubules des femelles sont souvent plus gros et plus robuste.
2. préparation des lames de la Poly-L-Lysine.
- Dessiner une bordure de 40 x 20 mm avec un stylo PAP hydrophobe autour du sommet de diapositives standard 75 x 25 mm et mettre de côté à sécher pendant 15 min à RT. utilisation grandes lamelles, si les lames ne sont pas compatibles avec l’imagerie optique.
- Transférer 2 mL de solution mère de Poly-L-Lysine (PLL) de 0,01 % sur chaque diapositive et mis de côté pendant 1 h à température ambiante.
- Retirez l’excès PLL avec une pipette. Enregistrez la solution dans une fiole conique de 50 mL pour une utilisation future. Conserver à 4 ° C.
- Aspirer toute solution restante avec une ligne vide. Exécutez la ligne vide au-dessus de la surface de la diapositive entière pour s’assurer qu’aucune solution ne reste sur les diapositives.
- Définir les diapositives côté pendant 1 h supplémentaire à avant l’utilisation de RT. Stocker les lames sèches à ta jusqu'à 1 mois dans un livre de diapositive standard.
3. préparation du plat et baguettes de verre de dissection
- Ajouter 0,5 mL d’adjuvant de salaison élastomère à 4,5 mL d’élastomère de base dans un polystyrène de 35 x 10 mm boîte de Pétri à RT pour produire une profondeur de 5 mm Mix avec une pointe de pipette à usage unique. Permettre à élastomère guérir O/N à température ambiante.
NOTE : Élastomère doit être claires et sans bulles. Compensation de bulles peut être facilitée en gardant les plaques élastomère dans un bocal vide pendant 10-15 min après coulée. - Tenir une baguette de verre de 5 mm de diamètre entre les mains et faire fondre le centre de la tige au-dessus d’un brûleur Bunsen éclairé tout en tirant sur les extrémités en dehors. Comme le verre fondu tirer plus rapidement pour produire un mince (0,1 mm) et l’arbre conique (Figure 1).
Remarque : Un angle de 45 ° à la tige est souvent utile dans le traitement des tubules. Ceci peut être réalisé en abaissant une main comme la tige est tirée (voir Figure 1). - Briser le corps mince au milieu avec le côté brutal d’une lame de rasoir en acier au carbone seul tranchant. Inspecter l’extrémité de la tige sous une dissection étendue pour assurer que la coupure est propre.
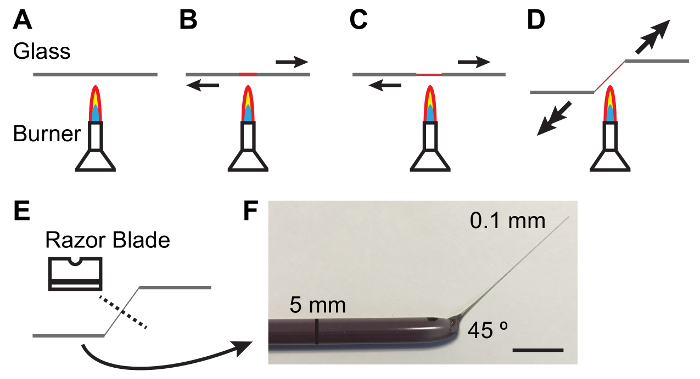
Figure 1 : Fabrication de baguettes de verre pour la manipulation de tubes de Malpighi.
A - E. Processus de chauffage et en tirant une baguette de verre pour produire une conicité et angle approprié pour le traitement des MTS. flèches indiquent la direction et l’amplitude de la force à appliquer. F. photographie d’un outil de verre fabriqué de manière appropriée. Echelle = 10 mm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
4. préparation des Solutions et système de Perfusion
Remarque : Les systèmes de Perfusion diffèrent par le fabricant. Ce protocole est basé autour d’un réservoir alimenté par gravité 8 canaux ouvert avec un régulateur de vitesse de flux d’entrée et une sortie axée sur le vide, mais la méthode de montage MTs comme décrit ici peuvent être adaptés pour fonctionner avec n’importe quel système de perfusion.
-
Préparer les solutions suivantes :
- D’aliquote Schneider moyen (40 mL dans des fioles coniques de 50 mL) et conserver à 4 ° C.
- Préparer des solutions (c.-à-d. insecte Phosphate-Buffered Saline (consolidation de la paix) et la consolidation de la paix avec NH4Cl) à ta si nécessaire selon le tableau 1). Solutions chaudes RT avant utilisation le jour de l’expérience.
Remarque : consolidation de la paix et de consolidation de la paix avec 40 mM NH4Cl peut être préparée en gros volumes (1 L ou plus) et stockées à 4 ° C. - Préparer des solutions d’étalonnage 8 volumes de 500 mL à un pH = 5.0, 6.0, 6.5, 7.0, 7.3, 7.6, 8.0 et 9.0, comme indiqué dans le tableau 1 et conserver à 4 ° C. Ajuster le pH de chaque solution par titrage avec N-méthyl-D-glucamine (NMDG) et HCl.
- Le jour des expériences, chauffer 5 ml de solutions d’étalonnage pour RT et ajouter nigéricine stock solution (20 mM dans le diméthylsulfoxyde (DMSO)) pour obtenir une concentration finale de 10 µM.
ATTENTION : Poignée nigéricine avec des gants. Traiter tout le matériel qui entre en contact avec nigéricine comme un produit jetable. Nigéricine reste sur le verre et le plastique et compromettra les préparations biologiques si le matériel est réutilisé.
-
Système de perfusion :
- Amorcez le système de perfusion en remplissant tous les réservoirs avec FD2O (Figure 2). Ouvrir les voies un à la fois pour permettre à toutes les lignes proximale pour le régulateur de débit taux à remplir.
Remarque : Il peut être nécessaire d’enlever l’air dans les canalisations en ouvrant le canal au point mort et à l’aide d’un plongeur au lecteur flux provenant du réservoir. - Ouvrir 2 canaux et laisser les ddH2O s’égoutter. Une fois que les réservoirs sont presque vides, remplir le réservoir de première consolidation de la paix et le deuxième réservoir avec NH4Cl-pulsé consolidation de la paix. Régler le débit au maximum avec le régulateur de vitesse de débit et laissez chaque solution s’écouler pendant 1 min remplir les lignes distales, puis arrêter le débit (Figure 2).
- Position 2 ensembles de « helping hands » pinces à souder sur la platine du microscope d’imagerie. Placer une pince de chaque côté de la plate-forme d’imagerie.
- Réchauffer soigneusement les 0,5 pouces distales d’un morceau de verre capillaire (diamètre intérieur 1. 5 mm, diamètre extérieur 0,86 mm, longueur 100 mm) au-dessus d’un brûleur Bunsen. Créer un coude de 45 ° en permettant à l’extrémité distale se plier par gravité et retirer le verre à la flamme une fois l’angle désiré est atteint. Répétez ce processus avec un deuxième morceau de verre capillaire.
- Insérer les capillaires de verre courbé dans le flux en ligne et la ligne de sortie connecté à vide, respectivement et monter dans les « helping hands » afin qu’ils cadrent avec l’étape d’imagerie du microscope (Figure 3).
- Amorcez le système de perfusion en remplissant tous les réservoirs avec FD2O (Figure 2). Ouvrir les voies un à la fois pour permettre à toutes les lignes proximale pour le régulateur de débit taux à remplir.

Figure 2 : Système de Perfusion et d’imagerie de Configuration.
Composants nécessaires pour l’évaluation physiologique de la fonction de transport basolatérale MT par simultanée en direct change de solution rapide et d’imagerie par fluorescence. Les conduites de gaz indiqués sont facultatifs et permettent l’expansion d’expériences pour l’évaluation du HCO3– transport. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
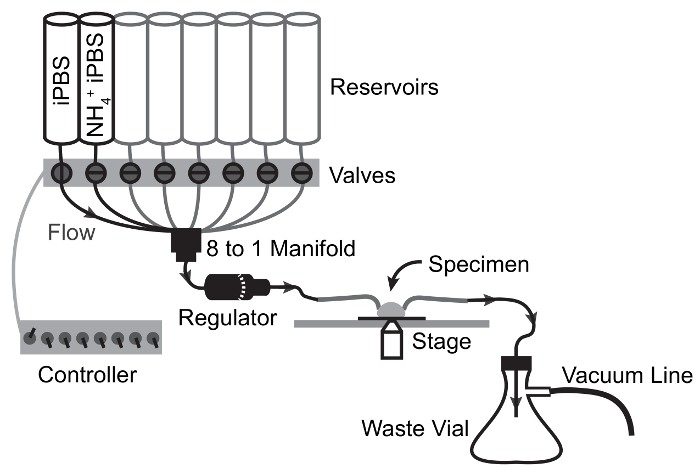
Figure 3 : Schéma de flux d’appareil de Perfusion pour NH4Cl Pulse expérimente.
Flèches représentent chemin d’écoulement et vanne points de commutation. Solution se déplace du réservoir au spécimen par écoulement gravitaire et provient de la chambre du spécimen dans le ballon de déchets par aspiration sous vide. Veuillez cliquer ici pour visionner une version agrandie de cette figure.
5. dissection de Malpighi antérieure adulte drosophile .
- Recueillir le plat de dissection et tiré une diapositive PLL-enduit de l’article 2, un diviseur de perfusion-puits adhésif, graisse sous vide, tige de verre du chapitre 3, une 4 x 2" bande de cachetage de film, 2 paires de #5 fines pinces et 40 ml de Schneider glacee moyen et RT consolidation de la paix.
- Étendre les graisse sous vide sur le ruban adhésif, puis appuyez sur le diviseur de perfusion-puits adhésif sur la cassette pour recouvrir le fond avec de la graisse. Décollez l’adhésif diviseur de perfusion-puits et placez-le graisse incliné sur le dessus une glissière enduite de PLL. Supprimer le séparateur de perfusion-puits pour laisser chaque spécimen puits tracées en graisse hydrophobe.
- Place 200 µL de RT consolidation de la paix dans la graisse-encerclé ainsi sur la glissière enduite de PLL et déplacer la diapositive sous le stéréoscope.
- Place SAMU-pHerry/PACCR-GAL4 mouches dans une mouche vide flacon et anesthésier leur sur la glace pendant 10 min.
Remarque : Cette méthode d’anesthésie, à la différence de CO2, veille à ce que les mouches ne pas déshydratent. - Versez le milieu de Schneider glacée dans le plat de dissection et pince fine permet de transférer une seule mouche femelle anesthésiée dans le plat dans un stéréoscope dissection.
- Tenez la volée par le thorax avec un ensemble de pinces et l’autre permet de fixer la partie postérieure de l’abdomen en douceur. Ouvrir l’extrémité postérieure de la volée en utilisant la pince en bref, des mouvements délibérés. Une fois que l’intestin est visible, saisir l’extrémité distale et libérer l’intestin et la MTs des trachéoles sous-jacent en tirant sur l’intestin postérieur du corps par le biais de remorqueurs répétitives, brefs.
Remarque : Le système commercial multilatéral antérieur et postérieur sera visible où ils rencontrent la jonction de l’intestin moyen et l’intestin par l’intermédiaire de l’uretère. La première paire de MTs pour être libre sera probablement être les tubules postérieurs qu’ils encerclent l’intestin postérieur. Ceux-ci peuvent être ignorés (Figure 4 a). - Pinch off les MTs antérieures à l’uretère avec une pince fine, une fois que le deuxième groupe de MTs est libre de l’abdomen. Cela séparer les MTs antérieures provenant de l’intestin et fermer l’uretère.
- Ramasser les gratuit MTs antérieures avec l’agitateur en verre tiré en faisant glisser la tige dans l’uretère, telles que les tubules tombent de part et d’autre. Soulevez le système commercial multilatéral tout droit hors de la solution.
- Tourner la tige de verre tel que la MTs et l’uretère sont adhérés à la face inférieure de la tige et abaissez l’uretère tout droit sur la diapositive. Apposer l’uretère et sceller les extrémités distales du système commercial multilatéral en enfonçant l’uretère sur la lame de verre (Figure 4 b). Ne pas manipuler le système commercial multilatéral plus que nécessaire. Le système commercial multilatéral devrait flotter vers le haut dans la solution avec l’uretère ancré à la diapositive.
- Utiliser l’extrémité fine de la baguette de verre pour balayer doucement chaque tubule sur toute la surface de glissement. Renfort de la tige contre la diapositive pour ne pas écraser le tubule et glissez la tige sur le dessus du tubule, déplacement distal vers proximal, d’attacher toute la longueur de chaque tubule à la surface de la lame de PLL-enduit (Figure 4).
- Replacez le diviseur de perfusion-puits adhésif sur la diapositive pour former une petite remplie de liquide bien sur le tubule monté.
- Placez l’échantillon sur la platine du microscope. Positionner l’entrée et sortie des capillaires sur l’entrée et l’orifice de sortie de la perfusion bien, respectivement.
Remarque : Le séparateur bien peut être laissé si vous souhaitez une chambre de perfusion ouvert. Dans ce cas les capillaires d’entrée et de sortie peuvent être alignés aux côtés opposés d’une imagerie bien.
6. validation de l’imagerie de protocole et la santé de Tubule
Remarque : Ce protocole est effectué sur un microscope à épifluorescence de grand champ inversé avec GFP (EERH) et DP (mCherry) jeux de filtres (excitation de 470/40 nm (ex), émission de longpass 515 nm (em), 500 nm nm dichroïque et 546/10 ex em de longpass 590 nm, 565 nm dichroïque), 10 X / 0,45 air objectif, une caméra monochrome pour la capture de l’image en direct et des logiciels d’imagerie. Le protocole peut être adapté pour n’importe quel droit ou microscope inversé avec filtre automatique de commutation entre GFP et DP optique et image logiciel d’acquisition, bien que les durées d’exposition optimale, intensité lumineuse et binning paramètres varient. Dans toutes les analyses, l’intensité de fluorescence doit être analysée comme intensité moyenne pixel dans la région d’intérêt (ROI), après que soustraction du fond dans chaque canal à l’aide d’un retour sur investissement avec ne contient aucune fluorescence adjacent au signal de retour sur investissement.
- Allumez le microscope, source lumineuse et système d’imagerie.
- Logiciels d’imagerie associée ouvert.
- Regardez dans l’oculaire et de régler manuellement la mise au point jusqu'à ce que la lumière de la MT est clairement visible sous la lumière transmise.
- Cliquez sur l’onglet « Acquisition » dans le logiciel d’analyse image et sélectionnez « 2 x 2 » dans le menu déroulant « Binning » dans la section « Mode d’Acquisition ».
- Insérez un filtre de densité neutre de 5 % dans le trajet optique pour réduire l’éclairage et de minimiser le photoblanchiment.
- Cliquez sur le canal de la GFP (EERH) dans le menu « Channels », puis cliquez sur « Live » pour observer le signal fluorescent grâce à la caméra.
- Ajustez le curseur de « Time » pour définir la durée d’exposition, telles que les valeurs des pixels les plus brillants de l’histogramme de l’intensité sont environ 40 % de la valeur maximale, puis cliquez sur « Stop » pour arrêter l’éclairage.
- Répétez les étapes 6,6-6,7 dans la DP (mCherry) canal et confirment la présence du segment initial du marqueur antérieur dilatée et l’absence de mCherry cytosolique agrégats (indicatif de lésions tissulaires ou surexpression) (Figure 4).
NOTE : Le segment dilaté devrait être clairement évident, comme c’est le segment plus proximal du tube et le diamètre de la lumière interne de ce segment est supérieure à celle du segment adjacent transitoire de ~ 20 µm. 2 x 2 pixels binning suffit souvent mais peut être augmentée pour réduire l’intensité de l’éclairage nécessaire. Temps d’exposition typiques sont entre 150 et 800 ms/canal. Utiliser comme peu de lumière que possible afin de minimiser le photoblanchiment. Minimisant le photoblanchiment est vital pour l’utilisation d’indicateurs de double-fluorophore comme pHerry car les deux fluorophores peut blanchir indépendamment, invalidant ainsi tout étalonnage de ratio. - Activer un Time-lapse protocole d’imagerie en cliquant sur la case à cocher « Time Series ».
- Ajuster la « durée » dans le menu déroulant dans la section « Time Series » à 10 min et le curseur « Intervalle » à 0 pour régler l’heure de capture totale avec une fréquence d’acquisition maximale de l’image. Une fréquence d’acquisition total de 0,2 Hz est souvent suffisante.
- Cochez les deux cases de DP (mCherry) dans la section « Channels » et la GFP (EERH).
- Ouvrez la ligne de consolidation du système de perfusion en activant le contrôleur soupape appropriée et de commencer le protocole d’imagerie en cliquant sur « Start expérience. » Après 1 min, passer à NH4Cl impulsion solution pour 20 s en ouvrant la soupape appropriée et fermant la ligne de consolidation de la paix, puis revenez à la consolidation de la paix par le NH4Cl ligne de fermeture et réouverture de la vanne de la consolidation de la paix. Autoriser le protocole d’imagerie complet à remplir avant d’arrêter le système de perfusion.
Remarque : Le Time-lapse analyse devrait révéler un signal stable mCherry et un signal de l’EERH qui augmente en présence de NH4Cl, étanche après lavage et récupère progressivement. - Effectuer un étalonnage de 2 points.
- Enlever le séparateur bien de peeling il loin de la diapositive sous-jacent et retirez les capillaires de perfusion et les colliers de l’imagerie bien.
- Appliquer 200 µL d’étalonnage consolidation de la paix (pH 7,4, 10 µM nigéricine) à l’imagerie bien avec une pipette de 200 µL. Supprimer la solution de l’imagerie bien avec la pipette, puis remplacez par une autre de 200 µL de solution d’étalonnage. Répétez cette opération 4 fois afin d’assurer l’échange de la solution complète.
- Incuber la préparation en solution d’étalonnage pendant 30 min avant l’imagerie. Répéter le protocole d’imagerie utilisant les mêmes paramètres déterminés en étapes 6,6-6.11, avec la modification de seulement 1 min d’image capture.
Remarque : Le système de perfusion et les capillaires ne sont pas nécessaires dans cette étape et ne doit pas être fixés à l’imagerie bien pour éviter d’exposer les capillaires à nigéricine. - Ajouter 200 µL d’étalonnage consolidation de la paix (pH 9.0, 10 µM nigéricine) à l’imagerie bien avec une pipette de 200 µL. Supprimer la solution de l’imagerie bien avec la pipette, puis remplacez par une autre de 200 µL de solution d’étalonnage. Répétez cette opération 4 fois afin d’assurer l’échange de la solution complète.
- Incuber la préparation à la deuxième solution d’étalonnage pendant 10 min avant l’imagerie. Répétez le protocole d’imagerie comme à l’étape 6.13.3.
- Examiner la pile de l’image capturée dans le logiciel d’analyse image pour confirmer qu’aucun pixel dans des canaux n’est saturés en cliquant sur « ROI signifie » défilement si la pile de l’image avec le curseur « Frame » tout en observant qu’aucune valeurs ne rapportées dans l’histogramme de l’intensité atteindre la valeur maximale détectable. Si tous les blocs contiennent des pixels qui atteignent l’intensité maximale détectable, réduire l’intensité de temps ou éclairage exposition et de la répéter section 6.
Remarque : Une fois établie ne changent pas de paramètres d’imagerie entre expériences ou étalonnage à moins que le point des étalonnages doivent être utilisées dans chaque préparation (voir étape 8.3).
- Analyser la pile d’image pour tracer l’intensité de fluorescence et rapport de fluorescence (EERH/mCherry) en fonction du temps.
- Cliquez sur « Signifie ROI » et sélectionnez l’outil forme libre. Maintenez le clic gauche pour tracer un ~ 50 µm de longueur de Mt. Faites un clic droit pour finir le retour sur investissement de dessin, puis répéter dans une zone adjacente à la MT pour définir un arrière-plan ROI (Figure 5 a).
- Cliquez sur « Intensité moyenne » sous « Mesure ». Créer une table des valeurs d’intensité en cliquant sur « Exporter > Table de données > créer. »
- Cliquez sur l’icône de configuration à crémaillère et désélectionnez l’option tous les paramètres sauf « Time » et « Intensité moyenne ». Cliquez sur l’onglet correspondant à la table de données nouvellement créée, sélectionnez « Enregistrer sous » et exporter les données dans un fichier .csv.
Remarque : Des mesures semblables sont également à l’aide de logiciels libres tels que ImageJ. - Ouvrir une table de feuille de calcul et importer la table de données en sélectionnant l’onglet « Données » suivi « Du texte ».
- Utiliser des fonctions dans le tableur pour soustraire l’EERH fond intensité de l’intensité du Signal EERH à chaque instant. Répétez ce processus pour le signal de mCherry.
- Tracer chaque intensité du canal en fonction du temps en sélectionnant les colonnes contenant les temps et les données d’intensité de référence-corrigée et puis en cliquant sur « Insérer > Scatter (graphiques) > Scatter avec lignes droites » (Figure 5 b).
- Utilisez les fonctions de feuille de calcul pour calculer le ratio de fluorescence SEpH/mCherry à chaque instant.
- Rapport de fluorescence de parcelle en fonction du temps en sélectionnant les colonnes contenant les données temps et ratio et puis en cliquant sur « Insérer > Scatter (graphiques) > disperser avec des lignes droites » (Figure 5).
7. la pleine étalonnage de pHerry dans les tubes de Malpighi Ex Vivo.
- Disséquer et monter une nouvelle série de MTs antérieures comme décrit à l’article 5.
- Échange de consolidation de la paix pour la consolidation de la paix d’étalonnage (pH 7,4, nigéricine 10 µM) tel que décrit à l’étape 6.13.2. Incuber pendant 30 minutes.
- Localiser le système commercial multilatéral et rassembler les paires d’images SEpH/mCherry comme décrit aux points 6.1-6.11. Remplacer la solution avec un autre stock d’étalonnage, consolidation de la paix tel que décrit à l’étape 6.13.4, attendre 10 min et encore une fois l’image. Répétez ce processus jusqu'à ce que le ratio de l’EERH/mCherry a été photographié sur toutes les solutions. Obtenir le pH 9,0 images dernière alors que le spécimen remet rarement d’un pH élevé.
- Tracer rapport de fluorescence de l’EERH pour mCherry d’étalonnages dans huit spécimens en fonction du pH imposéj’ai tel que décrit à l’étape 6.14.9. Adapter les données d’étalonnage avec une courbe de Boltzmann pour obtenir la fonction de calibrage complet selon l’équation 1 (Figure 5). Si les données sont incohérentes, tracer des ensembles de calibration de chaque spécimen normalisé de sorte qu’un rapport de fluorescence de 1,0 correspond à pHi 7.0 et réanalyser (Figure 5E).
Remarque : Si ce dernier procédé est nécessaire expériences individuelles devra leur propre point interne étalonnages33 (voir quantification procédure ci-dessous (étape 8.3)). -
Équation 1
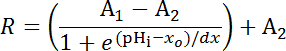
Où R = ratio SEpH/mCherry et un1, un2 xoet dx est courbe des paramètres d’ajustement représentant ratio minimum fluorescence, rapport de fluorescence maximale, pKuneet la largeur de la fonction respectivement. xo = apparent pKun de pHerry, qui peut varier entre 7,1 et 7,4 selon le type de cellule et les conditions de calibrage exact.
8. quantification des acides basolatérale Extrusion de l’épithélium des tubules Malpighian Ex Vivo .
- Image pHerry exprimant des cellules étoilées et exprimant le pHerry principal des cellules en même temps.
- Disséquer les MTs antérieures d’une mouche de SAMU-pHerry/PACCR-GAL4 tel que décrit à l’article 5, mais ne cèdent pas MTs du milieu de la dissection Schneider à l’imagerie bien.
- Disséquer MTs antérieures d’une mouche de SAMU-pHerry/c724-GAL4 dans le même plat de dissection à l’aide de la procédure décrite à l’article 5.
- Transférer les 2 séries de MTs dans l’imagerie même bien comme décrit aux étapes 5,8-5.11.
Remarque : Lorsque balayant les bras du système commercial multilatéral à la diapositive, placer le système commercial multilatéral de l' UAS-pHerry/c724-GAL4 et les tubules de SAMU-pHerry/PACCR-GAL4 près de l’autre afin que les cellules principales et stellaires exprimant pHerry peuvent être visualisées dans la même (champ Figure 6 a).
- Appliquer le NH4Cl préimpulsion tel que décrit à l’étape 6.12.
NOTE : Si l’étalonnage cohérente (Figure 4 b) ne pouvait pas être atteint, effectuer un étalonnage de point en affectant à pHi 7.0 à la fin de chaque expérience avec la consolidation de la paix d’étalonnage (pH 7.0, 10 µM nigéricine, 30 min d’incubation) après l’adhésif diviseur de perfusion-puits ainsi que le matériel de perfusion ont été supprimées. - Étalonner les traces des cellules étoilées et principales des différents segments de MT (en utilisant le ratio normalisé ou absolu selon le cas) avec l’équation 2 et d’analyser la phase de récupération après NH4Cl retrait en appliquant à l’aide des fonctions de décroissance exponentielle logiciel d’analyse statistique ainsi que la constante de désintégration (τ) (Figure 6 b).
Équation 2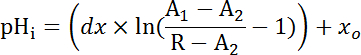
Où R = ratio SEpH/mCherry et un1, un2 xoet dx est courbe des paramètres d’ajustement déterminées par étalonnage à l’étape 7,4 (équation 1).- Calculer taux d’expulsion acide (JH +, voir équation 3) en fonction du pHj’ai pour tenir compte des variations de repos pHj’ai et charge entre préparations34acide. Fonctions exponentielles dérivées à l’étape 8.3 permet de calculer la dérivée de pHj’ai en ce qui concerne les temps de chaque intervalle.
Équation 3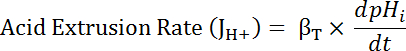
- Calculer la capacité intrinsèque de tampon (βi; Équation 4) du cytosol au pHj’ai dès le début de chaque intervalle à l’étape 8.3.1 basé sur la littérature antérieure (voir équation 4).
Remarque : Chez la drosophile, la caractérisation plus approfondie des βje vient de larves nerf moteur bornes35 et ces données peuvent être censées pour tenir pour les cellules MT en l’absence d’autres données disponibles.
Équation 4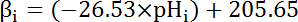
- Calculer le produit de βT (de l’étape 8.3.2)et dpHj’ai/dt (de l’étape 8.3.1) pour déterminer JH + (équation 3).
Remarque : Dans des solutions nominalement sans bicarbonate telles que celles décrites dans le présent protocole, pouvoir tampon bicarbonate de dérivés (βb) est supposé pour être ~ 0 mM. Total pouvoir tampon (βT) est la somme des β j’etβbet donc βi = βT en l’absence de HCO3–/co236. - Terrain JH + en fonction de la pHj’ai au début de chaque intervalle de temps, comme indiqué dans l’étape 6.14.9.
- Appliquer les fonctions de décroissance exponentielle de la part de tous les ensembles de données qui se chevauchent dans le pHj’ai à l’aide du logiciel d’analyse statistique. Comparez les prix des changement dans les fonctions qui en résulte pour comparer les taux d’expulsion acide entre les cellules et les segments de MT (Figure 6).
Remarque : La fonction plus appropriée utilisée pour l’ajustement de courbe peut être pas toujours un seul exponentiels. Autres fonctions peuvent être remplacées si elles améliorent la qualité de l’ajustement. - Calculer l’acide en fonction du pHj’ai pour tenir compte des variations dans la taille des cellules et la forme de flux (voir équation 5).
Équation 5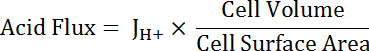
NOTE : Dimensions de la maille peuvent être mesurées directement en images ou approximées. Cellules principales peuvent être représentés comme deux moitiés d’un tube creux avec les dimensions suivantes : diamètre intérieur 24 µm ; diamètre extérieur 48 µm ; hauteur 50 µm. transition cellules étoilées sont variables mais peuvent être représentés à peu près comme les bouteilles avec des hauteurs de 50 µm et d’un diamètre de 10 µm. Voir le paragraphe final du Représentant résultats ci-dessous. - Appliquer les fonctions de décroissance exponentielle de la part de tous les ensembles de données qui se chevauchent dans pHj’ai à l’aide du logiciel d’analyse statistique. Comparez les prix des changement dans les fonctions qui en résulte pour comparer les flux acides entre les cellules et les segments de MT (Figure 6).
- Calculer taux d’expulsion acide (JH +, voir équation 3) en fonction du pHj’ai pour tenir compte des variations de repos pHj’ai et charge entre préparations34acide. Fonctions exponentielles dérivées à l’étape 8.3 permet de calculer la dérivée de pHj’ai en ce qui concerne les temps de chaque intervalle.
Résultats
Tissus sains et une pièce d’identité de MTs antérieurs sont essentiels à la réussite du présent protocole. Au cours de la dissection, il faut pas directement toucher le système commercial multilatéral et à poignée unique par l’uretère en saisissant le système commercial multilatéral directement conduiront à la rupture (Figure 4 a– B). Lorsque MTs sont balayés à plat sur le toboggan, les tubules doivent ...
Discussion
Le succès de dosage de pHj’ai in Drosophila MTs dépend entièrement de la santé des extraits MTs et la qualité de montage et de dissection (Figure A – C). Ainsi, la manipulation soigneuse de tissu comme décrit est impérative. Diapositives fraîchement enduits dans PLL substantiellement aide MT montage car ils ont tendance à être beaucoup plus adhésif que glisse qui ont déjà été exposés à la solution. Un montage soigneux aideront ?...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par les NIH DK092408 et DK100227 à MFR. AJR a été pris en charge par T32-DK007013. Les auteurs tiennent à remercier le Dr Julian A.T. Dow pour le PACCR-GAL4 et c724-GAL4 stocks de drosophile . Nous remercions également Jacob B. Anderson pour assistance, maintien des croisements expérimentaux de mouche.
matériels
| Name | Company | Catalog Number | Comments |
| Poly-L-Lysine (PLL) Solution | Sigma-Aldrich | P4832 | Store at 4 °C, can be reused. |
| Nigericin Sodium Salt | Sigma-Aldrich | N7143 | CAUTION: Handle with gloves. Store as aliquots of 20 mM stock solution in DMSO at 4 °C. |
| Adhesive Perfusion Chamber Covers, adhesive size 1 mm, chamber diameter × thickness 9 mm × 0.9 mm, ports diameter 1.5 mm | Sigma-Aldrich | GBL622105 | Can be substituted as needed to match perfusion system. |
| Sylgard 184 Silicone Elastomer Kit | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Available from multiple vendors. |
| Helping Hands Soldering Stands | Harbor Freight Tools | 60501 | Available from multiple vendors. |
| Open Gravity-fed Perfusion System with Valve Controller, 8 to 1 Manifold and Reserviors | Bioscience Tools | PS-8S | Any comparable perfusion system can be used. |
| Flow Regulator | Warner Instruments | 64-0221 | Can be substituted as needed to match perfusion system. |
| Schneider's Medium | Fisher Scientific | 21720024 | Store at 4 °C in sterile aliquots. |
| #5 Inox Steel Forceps | Fine Science Tools | 11252-20 | Can be substituted based on experimenter comfort. |
| 35 x 10 mm polystyrene Petri dish | Corning Life Sciences | Fisher Scientific 08-757-100A | Exact brand and size are unimportant. |
| 75 x 25 mm Microscope Slides | Corning Life Sciences | 2949-75X25 | Exact brand and size can vary as long as perfusion wells are compatible. |
| Filimented Borosilicate Capillary Glass, ID 1.5 mm, OD 0.86 mm, thickness 0.32 mm | Warner Instruments | 64-0796 | Filiment not necessary, glass can be substituted to match perfusion tubing and perfusion wells. |
| Tygon Tubing, ID 1/16", OD 1/8", thickness 1/32" | Fisher Scientific | 14-171-129 | Available from multiple vendors, can be substituted to match perfusion system. |
| Vacuum Silicone Grease | Sigma-Aldrich | Z273554 | Available from multiple vendors. |
| Plastic Flow Control Clamp | Fisher Scientific | 05-869 | Available from multiple vendors, sterility not required |
| Glass rods, 5 mm diameter | delphiglass.com | 9198 | Exact size is personal preference, multiple vendors available |
| PAP Hydrophobic Pen | Sigma-Aldrich | Z377821 | Available from multiple vendors. |
| Sealing Film | Sigma-Aldrich | P7668 | Available from multiple vendors. |
| 15 mL Falcon tube | BD Falcon | 352096 | Available from multiple vendors. |
| 50 mL Falcon tube | BD Falcon | 352070 | Available from multiple vendors. |
| HEPES; 4-(2-Hydroxyethyl)piperazine-1-ethanesulfonic acid | Sigma-Aldrich | H3375 | Available from multiple vendors. |
| MES; 4-Morpholineethanesulfonic acid monohydrate | Sigma-Aldrich | 69892 | Available from multiple vendors. |
| TAPS; N-[Tris(hydroxymethyl)methyl]-3-aminopropanesulfonic acid | Sigma-Aldrich | T5130 | Available from multiple vendors. |
| 10X/0.45 Air Objective | Zeiss | 000000-1063-139 | Comparable objectives can be substituted. 40X objectives can be used for single cell imaging. |
| Dissecting Stereoscope | Zeiss | Discovery.V8 | Any dissecting stereoscope can be used. |
| UAS-pHerry transgenic Drosophila melagnogaster | Available from Romero Lab | First published: Citation 10 | |
| capaR-GAL4 driver line Drosophila melagnogaster | Available from Romero Lab | First published: Citation 32 | |
| c724-GAL4 driver line Drosophila melagnogaster | Available from Romero Lab | First published: Citation 2 | |
| Monochromatic High Sensitivity Digital Camera | Zeiss | Axiocam 506 mono | Exact brand and model can vary, can be replaced with any monochromatic high-sensitivity camera suited to live cellular imaging. |
| GFP/FITC filter set, 470/40 nm ex., 515 nm longpass em., 500 nm dichroic | Chroma | CZ909 | Any GFP/FITC filer set can be substituted. |
| RFP/TRITC filter set, 546/10 nm ex., 590 nm longpass em., 565 nm dichroic | Chroma | CZ915 | Any GFP/FITC filer set can be substituted. |
| Inverted Epifluoescent Microscope | Zeiss | Axio Observer Z.1 | Any comparable microscope with motorized filter switching can be used. Upright microscopes can be used with open perfusion baths and water-immersion objectives. |
| Statistical Analysis Software | Microcal | Origin 6.0 | Any software with comparable functionality can be substituted |
| Image Analysis Software | National Institutes of Health | ImageJ 1.50i | Any software with comparable functionality can be substituted |
| Image Acquisition Software | Zeiss | Zen 1.1.2.0 | Any software with comparable functionality can be substituted |
| Single-edged Carbon Steel Razor Blade | Electron Microscopy Sciences | 71960 | Available from multiple vendors. |
| Microscopy Slide Folder | Fisher Scientific | 16-04 | Available from multiple vendors. |
| Bunsen Burner | Fisher Scientific | 50-110-1231 | Available from multiple vendors. |
| Polystrene Drosophila Rearing Vials with Flugs | Genesee Scientific | 32-109BF | Comparable items can be substituted. |
| 2.5 L Laboratory Ice Bucket | Fisher Scientific | 07-210-129 | Available from multiple vendors. |
| NMDG; N-Methyl-D-glucamine | Sigma-Aldrich | M2004 | Available from multiple vendors. |
| 200 uL barrier pipette tips | MidSci | AV200 | Available from multiple vendors. |
| 200 μL variable volume pipette | Gilson Incorporated | PIPETMAN P200 | Available from multiple vendors. |
Références
- Dow, J. A. T., Romero, M. F. Drosophila provides rapid modeling of renal development, function, and disease. Am J Physiol Renal Physiol. 299 (6), F1237-F1244 (2010).
- Sozen, M. A., Armstrong, J. D., Yang, M., Kaiser, K., Dow, J. A. Functional domains are specified to single-cell resolution in a Drosophila epithelium. P Natl Acad Sci USA. 94 (10), 5207-5212 (1997).
- Davies, S. A., et al. Analysis and inactivation of vha55, the gene encoding the vacuolar ATPase B-subunit in Drosophila melanogaster reveals a larval lethal phenotype. J Biol Chem. 271 (48), 30677-30684 (1996).
- Torrie, L. S., et al. Resolution of the insect ouabain paradox. P Natl Acad Sci USA. 101 (37), 13689-13693 (2004).
- Evans, J. M., Allan, A. K., Davies, S. A., Dow, J. A. Sulphonylurea sensitivity and enriched expression implicate inward rectifier K+ channels in Drosophila melanogaster renal function. J Exp Biol. 208 (Pt 19), 3771-3783 (2005).
- Sciortino, C. M., Shrode, L. D., Fletcher, B. R., Harte, P. J., Romero, M. F. Localization of endogenous and recombinant Na(+)-driven anion exchanger protein NDAE1 from Drosophila melanogaster. Am J Physiol Cell Physiol. 281 (2), C449-C463 (2001).
- Ianowski, J. P., O'Donnell, M. J. Basolateral ion transport mechanisms during fluid secretion by Drosophila Malpighian tubules: Na+ recycling, Na+:K+:2Cl- cotransport and Cl- conductance. J Exp Biol. 207 (Pt 15), 2599-2609 (2004).
- O'Donnell, M. J., et al. Hormonally controlled chloride movement across Drosophila tubules is via ion channels in stellate cells. Am J Physiol. 274 (4 Pt 2), R1039-R1049 (1998).
- Cabrero, P., et al. Chloride channels in stellate cells are essential for uniquely high secretion rates in neuropeptide-stimulated Drosophila diuresis. P Natl Acad Sci USA. 111 (39), 14301-14306 (2014).
- Rossano, A. J., Kato, A., Minard, K. I., Romero, M. F., Macleod, G. T. Na+ /H+ -exchange via the Drosophila vesicular glutamate transporter (DVGLUT) mediates activity-induced acid efflux from presynaptic terminals. J Physiol. 595 (3), 805-824 (2017).
- Sankaranarayanan, S., De Angelis, D., Rothman, J. E., Ryan, T. A. The use of pHluorins for optical measurements of presynaptic activity. Biophys J. 79 (4), 2199-2208 (2000).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat biotechnol. 22 (12), 1567-1572 (2004).
- Thomas, J. A., Buchsbaum, R. N., Zimniak, A., Racker, E. Intracellular pH measurements in Ehrlich ascites tumor cells utilizing spectroscopic probes generated in situ. Biochemistry. 18 (11), 2210-2218 (1979).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448 (7150), 151 (2007).
- Dow, J. A., et al. The malpighian tubules of Drosophila melanogaster: a novel phenotype for studies of fluid secretion and its control. J Exp Biol. 197, 421-428 (1994).
- Hirata, T., et al. In vivo Drosophilia genetic model for calcium oxalate nephrolithiasis. Am J Physiol Renal Physiol. 303 (11), F1555-F1562 (2012).
- Schellinger, J. N., Rodan, A. R. Use of the Ramsay Assay to Measure Fluid Secretion and Ion Flux Rates in the Drosophila melanogaster Malpighian Tubule. J Vis Exp. (105), (2015).
- Caldwell, P. C. An investigation of the intracellular pH of crab muscle fibres by means of micro-glass and micro-tungsten electrodes. J Physiol. 126 (1), 169-180 (1954).
- Boron, W. F., De Weer, P. Intracellular pH transients in squid giant axons caused by CO2, NH3, and metabolic inhibitors. J Gen Physiol. 67 (1), 91-112 (1976).
- Rink, T. J., Tsien, R. Y., Pozzan, T. Cytoplasmic pH and free Mg2+ in lymphocytes. J Cell Biol. 95 (1), 189-196 (1982).
- Bizzarri, R., Serresi, M., Luin, S., Beltram, F. Green fluorescent protein based pH indicators for in vivo use: a review. Anal Bioanal Chem. 393 (4), 1107-1122 (2009).
- Kneen, M., Farinas, J., Li, Y., Verkman, A. S. Green fluorescent protein as a noninvasive intracellular pH indicator. Biophys J. 74 (3), 1591-1599 (1998).
- Raimondo, J. V., Irkle, A., Wefelmeyer, W., Newey, S. E., Akerman, C. J. Genetically encoded proton sensors reveal activity-dependent pH changes in neurons. Front Mol Neurosci. 5, 68 (2012).
- Raimondo, J. V., et al. Tight Coupling of Astrocyte pH Dynamics to Epileptiform Activity Revealed by Genetically Encoded pH Sensors. J Neurosci. 36 (26), 7002-7013 (2016).
- Bagar, T., Altenbach, K., Read, N. D., Bencina, M. Live-Cell imaging and measurement of intracellular pH in filamentous fungi using a genetically encoded ratiometric probe. Eukaryot Cell. 8 (5), 703-712 (2009).
- Gjetting, K. S., Ytting, C. K., Schulz, A., Fuglsang, A. T. Live imaging of intra- and extracellular pH in plants using pHusion, a novel genetically encoded biosensor. J Exp Bot. 63 (8), 3207-3218 (2012).
- Greenspan, R. J. . Fly pushing: the theory and practice of Drosophila genetics. , (2004).
- Raimondo, J. V., et al. A genetically-encoded chloride and pH sensor for dissociating ion dynamics in the nervous system. Front Cell Neurosci. 7, 202 (2013).
- Koivusalo, M., et al. Amiloride inhibits macropinocytosis by lowering submembranous pH and preventing Rac1 and Cdc42 signaling. J Cell Biol. 188 (4), 547-563 (2010).
- Terhzaz, S., et al. Mechanism and function of Drosophila capa GPCR: a desiccation stress-responsive receptor with functional homology to human neuromedinU receptor. PloS one. 7 (1), e29897 (2012).
- Boyarsky, G., Ganz, M. B., Sterzel, R. B., Boron, W. F. pH regulation in single glomerular mesangial cells. I. Acid extrusion in absence and presence of HCO3. Am J Physiol. 255 (6 Pt 1), C844-C856 (1988).
- Chesler, M. The regulation and modulation of pH in the nervous system. Prog Neurobiol. 34 (5), 401-427 (1990).
- Rossano, A. J., Chouhan, A. K., Macleod, G. T. Genetically encoded pH-indicators reveal activity-dependent cytosolic acidification of Drosophila motor nerve termini in vivo. J Physiol. 591 (7), 1691-1706 (2013).
- Roos, A., Boron, W. F. Intracellular pH. Physiol Rev. 61 (2), 296-434 (1981).
- Vaughan-Jones, R. D., Wu, M. L. pH dependence of intrinsic H+ buffering power in the sheep cardiac Purkinje fibre. J Physiol. 425, 429-448 (1990).
- Buckler, K. J., Vaughan-Jones, R. D., Peers, C., Nye, P. C. Intracellular pH and its regulation in isolated type I carotid body cells of the neonatal rat. J Physiol. 436, 107-129 (1991).
- Bevensee, M. O., Schwiening, C. J., Boron, W. F. Use of BCECF and propidium iodide to assess membrane integrity of acutely isolated CA1 neurons from rat hippocampus. J Neurosci Methods. 58 (1-2), 61-75 (1995).
- Arosio, D., et al. Simultaneous intracellular chloride and pH measurements using a GFP-based sensor. Nat Methods. 7 (7), 516-518 (2010).
- Wu, Y., Baum, M., Huang, C. L., Rodan, A. R. Two inwardly rectifying potassium channels, Irk1 and Irk2, play redundant roles in Drosophila renal tubule function. Am J Physiol Regul Integr Comp Physiol. 309 (7), R747-R756 (2015).
- Schulte, A., Lorenzen, I., Bottcher, M., Plieth, C. A novel fluorescent pH probe for expression in plants. Plant Methods. 2, 7 (2006).
- Shen, Y., Rosendale, M., Campbell, R. E., Perrais, D. pHuji, a pH-sensitive red fluorescent protein for imaging of exo- and endocytosis. J Cell Biol. 207 (3), 419-432 (2014).
- Johnson, D. E., et al. Red fluorescent protein pH biosensor to detect concentrative nucleoside transport. J Biol Chem. 284 (31), 20499-20511 (2009).
- Mahon, M. J. pHluorin2: an enhanced, ratiometric, pH-sensitive green florescent protein. Adv Biosci Biotechnol. 2 (3), 132-137 (2011).
- Li, Y., Tsien, R. W. pHTomato, a red, genetically encoded indicator that enables multiplex interrogation of synaptic activity. Nat Neurosci. 15 (7), 1047-1053 (2012).
- Tantama, M., Hung, Y. P., Yellen, G. Imaging intracellular pH in live cells with a genetically encoded red fluorescent protein sensor. J Am Chem Soc. 133 (26), 10034-10037 (2011).
- Matlashov, M. E., et al. Fluorescent ratiometric pH indicator SypHer2: Applications in neuroscience and regenerative biology. Biochimica et biophysica acta. 1850 (11), 2318-2328 (2015).
- Kogure, T., et al. A fluorescent variant of a protein from the stony coral Montipora facilitates dual-color single-laser fluorescence cross-correlation spectroscopy. Nat biotechnol. 24 (5), 577-581 (2006).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. P Natl Acad Sci USA. 95 (12), 6803-6808 (1998).
- Poburko, D., Santo-Domingo, J., Demaurex, N. Dynamic regulation of the mitochondrial proton gradient during cytosolic calcium elevations. J Biol Chem. 286 (13), 11672-11684 (2011).
- Stornaiuolo, M., et al. KDEL and KKXX retrieval signals appended to the same reporter protein determine different trafficking between endoplasmic reticulum, intermediate compartment, and Golgi complex. Mol Biol Cell. 14 (3), 889-902 (2003).
- Makkerh, J. P., Dingwall, C., Laskey, R. A. Comparative mutagenesis of nuclear localization signals reveals the importance of neutral and acidic amino acids. Curr Biol. 6 (8), 1025-1027 (1996).
- Zacharias, D. A., Violin, J. D., Newton, A. C., Tsien, R. Y. Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells. Science. 296 (5569), 913-916 (2002).
- McGuire, R. M., Silberg, J. J., Pereira, F. A., Raphael, R. M. Selective cell-surface labeling of the molecular motor protein prestin. Biochem Biophys Res Comm. 410 (1), 134-139 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon