Method Article
Microscopie épiscopique à haute résolution (HREM) - Protocoles simples et robustes pour le traitement et la visualisation des matières organiques
Dans cet article
Résumé
Nous fournissons des protocoles simples et robustes pour le traitement de biopsies de diverses espèces, des embryons d'organismes modèles biomédicaux et des échantillons d'autres tissus organiques afin de permettre la génération de données numériques de volume avec la méthode de microscopie épiscopique à haute résolution.
Résumé
Nous fournissons des protocoles simples pour générer des données numériques de volume avec la méthode de microscopie épiscopique haute résolution (HREM). HREM est capable d'imager des matériaux organiques avec des volumes jusqu'à 5 x 5 x 7 mm 3 dans des résolutions numériques typiques entre 1 x 1 x 1 et 5 x 5 x 5 μm 3 . Les spécimens sont incorporés dans la résine de méthacrylate et sectionnés sur un microtome. Après chaque section, une image de la surface du bloc est capturée avec une caméra vidéo numérique qui se trouve sur le phototube connecté à la tête de microscope composée. L'axe optique traverse un cube filtrant de protéine fluorescente verte (GFP) et est aligné avec une position à laquelle le bras du support de la batterie repose après chaque section. De cette façon, une série d'images numériques alignées de façon inhérente, qui présente des surfaces de blocs ultérieures, est produite. Le chargement d'une telle série d'images dans un logiciel de visualisation tridimensionnel (3D) facilite la conversion immédiate en données de volume numériques, ce qui permet des virtualitésDans divers plans orthogonaux et obliques et la création de modèles informatiques volumineux et de surface. Nous présentons trois protocoles simples et spécifiques aux tissus pour le traitement de divers groupes de spécimens organiques, y compris les embryons de souris, de poussins, de cailles, de grenouilles et de zèbres, de biopsie humaine, de papier non revêtu et de matériel de remplacement de la peau.
Introduction
L'analyse structurale des matériaux organiques et anorganiques est la première étape dans la compréhension de leurs propriétés physiques et de leur fonction. La base de cette analyse est souvent l'information bidimensionnelle (2D) obtenue grâce à une observation minutieuse des sections histologiques, avec une variété de méthodes d'imagerie simples et sophistiquées qui extraient les détails de l'architecture tissulaire, la morphologie cellulaire et la topologie, la composition moléculaire et les propriétés biomécaniques 1 , 2 , 3 . Cependant, les informations 2D ne conviennent pas pour rechercher des arrangements spatialement complexes. Par conséquent, un nombre croissant de méthodes in vivo et ex vivo qui permettent la génération de données de volume numérique ont été établies au cours des dernières décennies 4 et beaucoup d'autres sont en cours de développement.
Le principe méthodique de la plupart des méthodes de génération de données de volume est la génération de piles virtuellesD'images numériques affichant des sections obtenues par sectionnement virtuel ou physique d'un objet. Si les images de la section sont alignées correctement, cela crée un volume, qui peut être recoupé dans des plans de section virtuels, ou utilisé pour créer des modèles 3D en surface et en volume. Les techniques populaires pour visualiser les humains et les spécimens biologiques plus importants sont la tomographie par résonance magnétique (MRT), la tomodensitométrie (CT), la tomographie par émission de positons (PET) et la tomodensitométrie par émission de photons (SPECT). Les petits échantillons sont habituellement visualisés en utilisant l'imagerie par résonance magnétique (μMRI), la tomographie par projection optique (OPT), la tomographie par cohérence optique (OCT), la tomographie photoacoustique (PAT), les méthodes de sectionnement histologique, la microscopie confocale et la tomographie électronique 5 , 6 , 7 , 8 , 9 , 10 , 11 , 12 , 13 , 14 , 15 , 16 , 17 .
Une technique de génération de données de volume relativement nouvelle, qui produit des données numériques de petits spécimens et d'échantillons de tissus histologiques, est la méthode HREM, qui a été développée en étroite collaboration avec Tim Mohun 18 , 19 . C'est une simple technique basée sur le microscope, qui génère des données de volume numériques à partir de matériaux intégrés en résine qui sont sectionnés sur un microtome. Les données facilitent l'analyse détaillée de l'architecture des tissus et des distributions cellulaires ainsi que l'analyse métrique de petites caractéristiques sur un niveau microscopique optique intermédiaire.
HREM produit des piles d'images numériques alignées de manière inhérente qui semblent capturées à partir d'eSections histologiques colorées aux osines. Le contraste des tissus et la résolution des données par rapport au champ de vision dépassent ceux des données produites avec μCT, μMRI et OPT, mais sont inférieures à celles réalisables avec la fibre confocal, la lumière et la microscopie électronique 20 . Cependant, contrairement à ce dernier, HREM est capable de visualiser des échantillons avec des volumes relativement importants allant jusqu'à 5 x 5 x 7 mm 3 en qualité histologique. Un certain nombre d'études récentes fournissent des caractérisations et des comparaisons détaillées des avantages et des inconvénients des techniques d'imagerie unique et, dans un souci d'objectivité, nous nous référons à ceux pour plus d'informations concernant leurs limites et champs d'application potentiels 4 , 21 , 22 , 23 , 24 .
Cette étude met l'accent sur la méthode d'imagerie HREM et vise à fournirDes protocoles très simples pour générer des données HREM d'un large éventail de matériaux organiques, ainsi que des exemples de leur application. Le flux de travail pour créer des données HREM est simple et s'applique à tous les matériaux qui peuvent être intégrés dans la résine de méthacrylate ( figure 1 ). Pourtant, il existe des différences spécifiques de tissus dans la préparation des échantillons, qui doivent être prises en considération. Nous proposons donc trois protocoles standard pour préparer différents échantillons. L'intégration et les étapes de protocole de génération de données sont identiques pour toutes.
Protocole
Toutes les procédures ont été effectuées conformément aux directives éthiques de l'Université médicale de Vienne.
1. Préparation de l'échantillon
- Préparation des embryons et des tissus embryonnaires (jusqu'à 5 x 5 x 5 mm 3 )
- Fixer les embryons ou les parties des embryons dans 4% de PFA / PBS ou de fixateur de Bouin à 4 ° C pendant au moins 16 à 24 h sous un basculement constant.
REMARQUE: On peut utiliser des embryons de plusieurs espèces et de diverses étapes de développement, comme le poisson zèbre 48 h, le poussin et la caille de 19 h, ainsi que des embryons prénataux de souris, qui ont été utilisés ici (voir Résultats représentatifs ). Les échantillons ont été récoltés et transférés dans du PBS à température ambiante pour la mise en scène avant leur mise en fixation. Si nécessaire, couper les éprouvettes avec des microcisres ou des scalpels avant la fixation pour s'adapter aux moules d'encastrement. - Retirer le fixateur et laver le tissu embryonnaire dans du PBS à 4 ° C sous un balancement constantG pendant 24 h (2-3 changements).
- Déshydratez les échantillons dans 70%, 80%, 90%, 96% d'éthanol à 4 ° C pendant 2 à 3 h chacun. Placer les tubes contenant les échantillons sur un rotateur.
REMARQUE: pour les petits échantillons (<2 x 2 x 2 mm 3 ), le temps pendant lequel un échantillon repose dans chaque éthanol peut être réduit à 60 min. Pour les gros échantillons (> 5 x 5 x 5 mm 3 ), il peut être prolongé jusqu'à 4 h. - Effectuez les étapes d'infiltration suivantes sous une hotte portant des gants de protection.
- Préparer la solution d'infiltration en ajoutant 1,25 g de peroxyde de benzoyle (catalyseur plastifié) et 0,4 g d'éosine à 100 ml de solution A (pour plus de détails, voir Tableau des matériaux ). Mettez ceci dans un bécher sur une plaque d'agitation magnétique à 4 ° C jusqu'à ce que l'éosine et le catalyseur soient complètement dissous (2 à 3 h).
- Placez les échantillons pendant 12 à 24 h dans la solution d'infiltration. Gardez-le à 4 ° C sous un basculement continu ou une rotation. Remplacer la solution d'infiltration avec une solution fraîche après 3 et 12H.
REMARQUE: Étendre le temps d'infiltration à 48 h pour les grands embryons (> 1 cm de couronne / longueur de croupe).
- Fixer les embryons ou les parties des embryons dans 4% de PFA / PBS ou de fixateur de Bouin à 4 ° C pendant au moins 16 à 24 h sous un basculement constant.
- Préparation des échantillons de tissus adultes (jusqu'à 5 x 5 x 7 mm 3 )
- Fixer les tissus dans 2% de PFA / PBS contenant 4% d'acide carbolique à 4 ° C pendant au moins 3 jours.
REMARQUE: L'échantillon de tissu provenant de la peau, du foie, du pancréas, des reins, de la thyroïde, du cœur, du muscle strié, du cerveau, des nerfs et des modèles de tumeur de plusieurs humains néonatals et adultes, des rongeurs, des porcs, des mouches des fruits et des poissons zèbres peut être résolu dans cette solution Puis traité pour l'imagerie HREM (nous démontrons la fixation des échantillons, voir les résultats représentatifs ). Exciser les échantillons de tissus avec des scalpels, des ciseaux ou des punaises de biopsie et les transférer directement en fixateur. Pour la fixation de l'utilisation du tissu nerveux, 4% de PFA / PBS. - Retirez le fixateur et lavez le tissu sous l'eau courante du robinet pendant 3-6 h.
- Déshydratez les échantillons par la suiteEn 70%, 80%, 90%, 96% d'éthanol, mélangé avec 0,4 g d'éosine par 100 mL (2 à 3 h chacun). Conservez les solutions avec des échantillons à 4 ° C sous un basculement constant ou sur un rotateur.
REMARQUE: pour les petits échantillons (<2 x 2 x 2 mm 3 ), le temps pendant lequel un échantillon repose dans chaque éthanol peut être réduit à 60 min. Pour les gros échantillons (> 5 x 5 x 5 mm 3 ), il peut être prolongé jusqu'à 4-8 h. - Effectuez toutes les étapes d'infiltration suivantes sous une hotte munie de gants de protection.
- Préparez la solution d'infiltration en ajoutant 1,25 g de peroxyde de benzoyle (catalyseur plastifié) et 0,4 g d'éosine à 100 ml de solution A (pour plus de détails, voir Tableau des matériaux ). Incorporer un bécher sur une plaque d'agitation magnétique à 4 ° C jusqu'à ce que l'éosine et le catalyseur soient complètement dissous (2 à 3 h).
- Placez les échantillons dans la solution d'infiltration pendant 24 à 36 h. Gardez-le à 4 ° C sous un basculement continu ou une rotation. Remplacer la solution d'infiltration avec une solution fraîche après 3 et12 h.
REMARQUE: pour les gros échantillons (> 5 x 5 x 5 mm 3 ), prolonger le temps d'infiltration à 48 h.
- Fixer les tissus dans 2% de PFA / PBS contenant 4% d'acide carbolique à 4 ° C pendant au moins 3 jours.
- Préparation d'autres matières organiques
REMARQUE: Coupez les substituts de la peau et le papier à la taille souhaitée avec des ciseaux ordinaires. Aucune fixation n'est nécessaire.- Étapes de fixation, de lavage et de déshydratation du saut.
- Effectuez toutes les étapes d'infiltration suivantes sous une hotte munie de gants de protection.
- Préparez la solution d'infiltration en ajoutant 1,25 g de peroxyde de benzoyle (catalyseur plastifié) et 0,4 g d'éosine à 100 ml de solution A (pour plus de détails, voir Tableau des matériaux ). Incorporer un bécher à 4 ° C sur une plaque d'agitation magnétique jusqu'à ce que l'éosine et le catalyseur soient complètement dissous (2 à 3 h).
- Placez les échantillons dans la solution d'infiltration pendant 3 à 12 h. Gardez-le à 4 ° C sous un basculement continu ou une rotation. Remplacer la solution d'infiltration avec une solution fraîche après 1 et 2 - 4 h.
2. Incorporation
REMARQUE: Effectuez toutes les étapes sous une hotte avec des gants de protection.
- Préparez une solution d'infiltration fraîche telle que décrite ci-dessus (étape 1.1.4.1). Ajouter 4 mL de solution B (pour plus de détails, voir le tableau des matériaux ) à une solution d'infiltration de 100 mL pour préparer la solution d'encastrement.
REMARQUE: si une orientation précise de l'échantillon dans les moules d'encastrement est nécessaire et pour permettre l'encastrement de plusieurs échantillons en parallèle, ralentir la polymérisation de la solution mère en mettant le bécher dans un bain de glace. - Utilisez des plateaux de moulage (pour plus de détails, voir Table des matériaux ) avec des moules ayant un volume de routine de 6 x 8 x 5 mm 3 ou 13 x 19 x 5 mm 3 , ou des moules personnalisés avec des volumes jusqu'à 8 x 10 x 15 mm 3 ( figure 2 ).
- Remplissez les moules avec une solution d'encastrement et transférez l'échantillon dans le moule en utilisantune cuillère. Pendant le transfert, s'assurer que l'échantillon est entièrement recouvert d'une solution d'encastrement pour éviter le piégeage d'air.
REMARQUE: ceci peut être effectué à température ambiante, si démarré immédiatement après la collecte des échantillons et la solution d'incorporation du réfrigérateur.- Orientez l'échantillon dans le moule en utilisant des aiguilles ou des pinces.
- Dès que la solution d'encastrement commence à devenir visqueuse, placez un support de bloc (voir la Table des matériaux ) sur le dessus du moule et ajoutez une solution d'encastrement à travers le trou central du support de bloc jusqu'à ce que la base du support de bloc soit recouverte de 1 à 2 Mm de solution d'encastrement.
- Sceller le plateau de moulage en le recouvrant avec de la paraffine liquide et attendre jusqu'à ce qu'il soit durci. Alternativement, placez un deuxième plateau de moulage à l'envers sur le plateau de moulage avec des échantillons et scellez-le avec du ruban adhésif à l'air.
- Permettre aux blocs de terminer la polymérisation en stockant les plateaux de moulage scellés pour 1 -2 jours à température ambiante.
- Pour le traitement post-polymérisation, placez les plateaux de moulage avec les blocs polymérisés dans un four de laboratoire standard et faites cuire à une température de 70 à 80 ° C pendant au moins 1 à 2 jours jusqu'à ce qu'ils deviennent de couleur rouge foncé. Recueillir les plateaux du four et retirer les blocs du moule.
REMARQUE: La cuisson peut se faire dans des conditions environnementales normales. Le plateau de moulage de revêtement, qui assure une étanchéité à l'air dans la première phase après l'encastrement, peut être retiré pour permettre de vérifier la couleur de la résine après 10 h. Immédiatement après la cuisson, la résine est douce et les blocs peuvent être facilement retirés du plateau de moulage. Dans les 2 à 3 heures, ils sont durs et peuvent être conservés à température ambiante pendant plusieurs mois.
3. Génération de données
REMARQUE: Le prototype HREM 18 , 25 utilisé ici comprend les éléments suivants: (i) Microtome rotatif avec un support de blocQui s'arrête après chaque coupe à son point de virage supérieur. (Ii) Couteau à microtome standard, non jetable, métal dur, profil D (pour plus de détails, voir Tableau des matériaux ). (Iii) Tête de microscope à fluorescence avec revolver objectif et un cube de filtre GFP (excitation 470/40; filtre d'émission 525/50) dans son axe optique. L'axe optique est disposé perpendiculairement à la surface fraîchement découpée d'un bloc monté sur le microtome et est maintenu par un dispositif qui permet des mouvements ascendants et descendants. (Iv) table transversale motorisée portant le microtome. La table peut être déplacée dans la direction de l'axe optique et latéralement. (V) Caméra vidéo numérique attachée au microscope à cristaux liquides. (Vi) Source de lumière monochrome (470 nm). (Vii) Ordinateur connecté à la caméra, avec logiciel de génération de données (pour plus de détails, voir Table des matières ).
- Procédure
- Indiquez la région d'intérêt.
- Utiliser un laboratoire standardGht source (voir Table of Materials ) pour diriger la lumière blanche obliquement sur la surface du bloc.
- Identifiez les contours du matériau embarqué en projetant la surface du bloc. Indiquez la région d'intérêt et le dernier champ de vision sur la surface du bloc en utilisant un stylo ou une lame de rasoir.
REMARQUE: cette étape est obligatoire pour définir l'étendue du champ de vision avant la section.
- Définissez le champ de vision.
- Monter le bloc de résine avec un échantillon sur le microtome.
- Déplacez le support de bloc à sa position d'arrêt (point de virage supérieur de l'excursion du porte-bloc).
- Démarrez l'appareil photo numérique et le logiciel de génération de données, et obtenez une image en direct.
- Choisissez un objectif, avec un grossissement approprié pour couvrir la région d'intérêt indiquée sur la surface du bloc.
REMARQUE: les objectifs avec des agrandissements 2.5X, 5X, 10X, 20X et des ouvertures numériques entre 0.07 et 0.40 sont couramment utilisés. - Déplacez l'optique vers le haut et vers le bas et le microtome latéralement en utilisant la table transversale motorisée jusqu'à ce que la région d'intérêt correspond au champ de vision affiché sur l'écran de l'ordinateur.
- Concentrer.
- Effectuez une coupe manuelle étape par étape jusqu'à ce que les premières structures de l'échantillon deviennent visibles.
- Disposez la surface du bloc dans le plan de focalisation de l'optique, en déplaçant le microtome dans la direction de l'axe optique.
- Choisissez l'épaisseur de la section; Des épaisseurs de section de 0,5 à 5 μm sont recommandées.
- Indiquez la région d'intérêt.
- Production et traitement de données
- Démarrez la routine du logiciel qui organise la section et la capture d'image. Suivez les instructions documentées du logiciel de capture d'image.
- Arrêtez la routine du logiciel et placez une diapositive de mise à l'échelle devant la surface du bloc dès que l'échantillon est complètement sectionné.
- Capturez une image de manière interactive pour un étalonnage ultérieurTion.
REMARQUE: ceci doit être fait chaque fois qu'un objectif est utilisé dans une configuration nouvellement organisée. - Convertissez la série d'images en images à l'échelle de gris de 8 bits au format jpg.
- Chargez des séries d'images dans un logiciel de visualisation 3D et ajustez les contrastes.
REMARQUE: la conversion d'image et la visualisation 3D peuvent être effectuées avec différents progiciels standard disponibles dans le commerce ou en libre-service, en suivant les instructions du logiciel concerné.
Résultats
HREM génère des séries d'images numériques alignées de façon inhérente avec des contrastes similaires aux images des coupes histologiques colorées à l'hématoxyline / éosine. Contrairement aux images de la section 2D, les piles d'images HREM permettent une visualisation et une analyse de l'architecture des tissus, de la morphologie et de la topologie d'une grande variété de matériaux organiques en 3D. Le contraste élevé facilite souvent la visualisation 3D rapide et simple des modèles informatiques volumineux et des résultats de contour semi-automatique pour la génération de modèles rendus en surface.
La taille des données HREM varie selon la taille de la cible de la caméra, le mode de capture d'image (8, 12, 16 bits, échelle de gris, couleur) et le nombre d'images de face à bloc unique. Les plus petits ensembles de données de 1000 images 8-bit.jpg en échelle de gris de 2,048 pixels x 2,048 pixels ont une taille d'environ 900 Mo. Plus grands ensembles de données de 3 000 images à l'échelle de gris de 8 000.jpg de 4,096 pIxel x 4,096 pixels ont des volumes d'environ 20 Go.
Les protocoles fournis sont simples et robustes, et au cours de la dernière décennie, ils ont été employés pour générer des données HREM de nombreux spécimens différents. La section de protocole 1.1 a été optimisée pour traiter des embryons entiers d'organismes modèles biomédicaux avec une longueur de jusqu'à 1 cm et des échantillons de tissu embryonnaire d'une dimension allant jusqu'à 5 x 5 x 5 mm 3 ; Nous avons utilisé cette méthode pour produire des données HREM d'embryons des espèces suivantes: souris ( Mus musculus) , poussin ( Gallus gallus ), poisson zèbre ( Danio rerio ), cailles ( Coturnix coturnix) , grenouille africaine ( Xenopus laevis ), cheval ( Equus ferus caballus ), un insecte d'amidon ( Oncopeltus fasciatus ), crocodile ( Crocodylia ) et pieuvre ( Octopus vulgaris ). Les données de toutes les espèces étaient d'excellente qualité ( p . Ex. , Figure 3 )
La section de protocole 1.2 a été optimisée pour le traitement d'échantillons de tissus juvéniles et adultes avec des dimensions allant jusqu'à 5 x 5 x 7 mm 3 et utilisé pour visualiser des échantillons de tissu de la La peau, le foie, le pancréas, le rein, la thyroïde, le cœur, les muscles striés, le cerveau, les nerfs et les modèles de tumeurs récoltés chez les humains ( Homo sapiens ), les souris ( Mus musculus ), les rats ( Rattus norvegicus ), les porcs ( Sus scrofa domestica ) le furet ( Mustela Furo ), la mouche des fruits ( Drosophila melanogaster ) et le poisson zèbre ( Danio rerio ). Bien que les résultats soient excellents avec la plupart des spécimens ( p . Ex. , Figure 4 , Animation 1 ), les parties centrales de la peau (avec l'épiderme) et les échantillons de cerveau supérieurs à 3 x 3 x 3 mm 3 sont souvent restés non colorés en raison de la pénétration insuffisante de l'éosine à travers Ces tissus.
Protocole sLa section 1.3 a été optimisée pour le traitement de la matière organique fibreuse et utilisée pour visualiser l'architecture des fibres de papier couché, du papier non revêtu, du matériau de substitution dermique natif et du matériau de substitution dermique semé par les cellules souches. Ces échantillons ont été faciles et rapides à traiter. Les données du papier non revêtu et de la plupart des substituts de la peau étaient de bonne qualité ( Figure 5 , Animation 2 ). Des problèmes se sont produits dans le traitement du papier couché, lorsque le matériau anorganique entravait la pénétration de l'éosine. Un autre problème s'est produit lors du traitement des substituts de la peau sur la base de l'agar car ils ont été partiellement digérés par la solution d'infiltration.
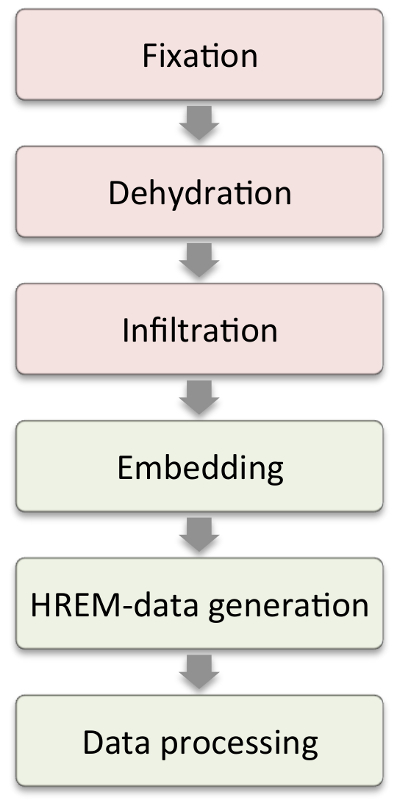
Figure 1: flux de travail. Les étapes indiquées dans les boîtes rouges nécessitent des modifications selon les caractéristiques de l'échantillon. Les étapes dans les cases vertes sont celles quiSont similaires dans tous les échantillons. Cliquez ici pour voir une version plus grande de ce chiffre.

Figure 2: Moules personnalisés. Les moules peuvent être adaptés en coupant un trou dans le moule d'origine, en insérant l'ampoule d'une pipette Pasteur et en l'étanchant avec des matériaux de modélisation. Cliquez ici pour voir une version plus grande de ce chiffre.
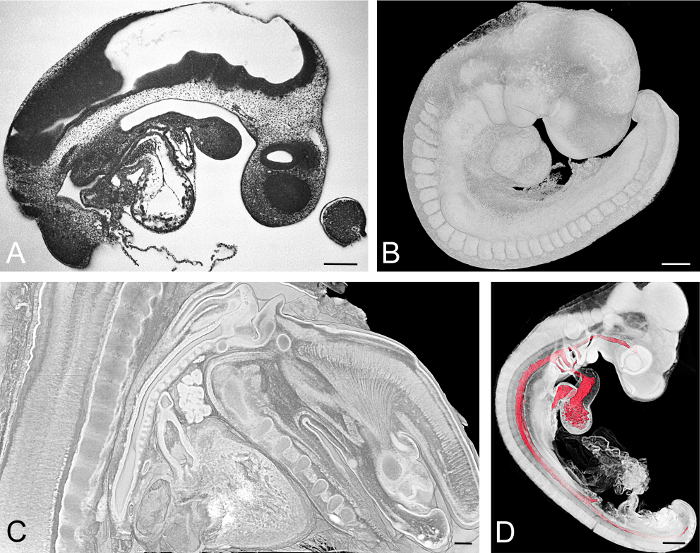
Figure 3: Embrayages illustrés de manière exemplaire. (A, B) embryon de souris récolté au jour embryonnaire, E = 9,5. L'image de la section HREM est affichée dans (A) . Le modèle 3D représenté en volume montrant la surface est affiché en (B) . (C) Section sagittale virtuelle à travers un modèle volumétrique du cou d'un embryon de souris E15.5. (D) embryon de poulet au stade de développement Hamburger Hamilton (HH) étape 18. Modèle de surface de la luminaire des composants cardiovasculaires combiné avec le rendu du volume de tous les tissus embryonnaires. Barres d'échelle = 200 μm. Cliquez ici pour voir une version plus grande de ce chiffre.
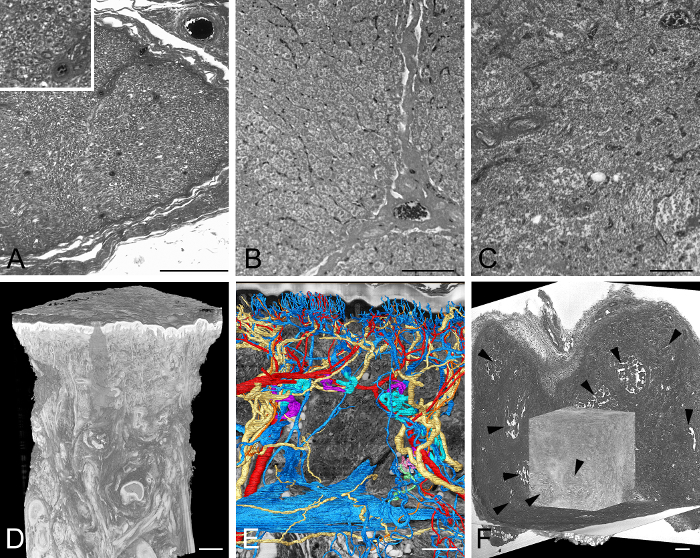
Figure 4: Échantillons de tissus adultes illustrés de manière exemplaire. (A) Partie d'une image de section HREM à travers un nerf humain. Inlay montre une partie de l'image plus en détail. (B) Partie d'une image de la section HREM à travers le foie porcin. Notez le nUclei. (C) Partie d'une image en coupe HREM du tissu lymphatique humain. (D, E) Peau épaisse d'un pouce humain. Modèle 3D volumineux de la totalité de la biopsie. (D) Modèles rendus en surface des artères, des veines et des nerfs devant une résection virtuelle à travers les données HREM (E) . (F) Tumeur expérimentale dans un tissu de souris adulte. Le modèle 3D converti en volume devant trois sections virtuelles à travers les données HREM. Notez les parties nécrotiques (pointes de flèche). Barres d'échelle = 200 μm. Cliquez ici pour voir une version plus grande de ce chiffre.

Figure 5: matériau fibreux illustré de manière exemplaire. (A, B) Image de la section HREM de papier brun non revêtu. (B) montre une section de (A) . Notez les fibres et leur lumière. (C) section HREM du papier couché. Notez que les fibres restent non colorées. (D) Modèle de volume de matériau substitutif dermique natif. Notez le calibre différent et la forme des fibres. Barres d'échelle = 100 μm. Cliquez ici pour voir une version plus grande de ce chiffre.
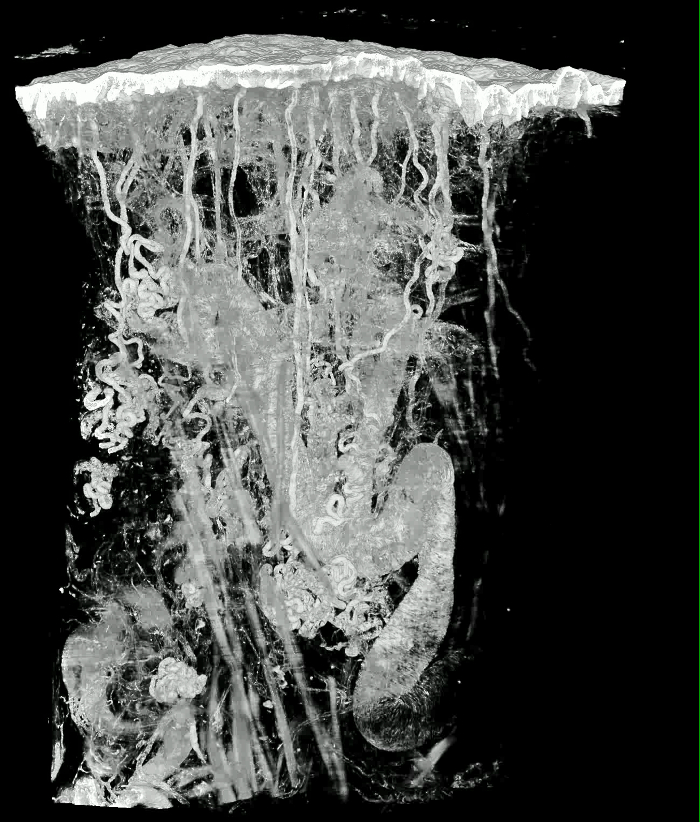
Animation 1: Modèle sous forme de volume de la peau épaisse d'un coussin humain. La taille du bloc de tissu est d'environ 4,2 x 2,7 x 2,7 mm 3 . La taille de Voxel est de 1,07 x 1,07 x 2 μm 3 . Cliquez ici pour voir cette vidéo. (Cliquez avec le bouton droit de la souris pour télécharger.)
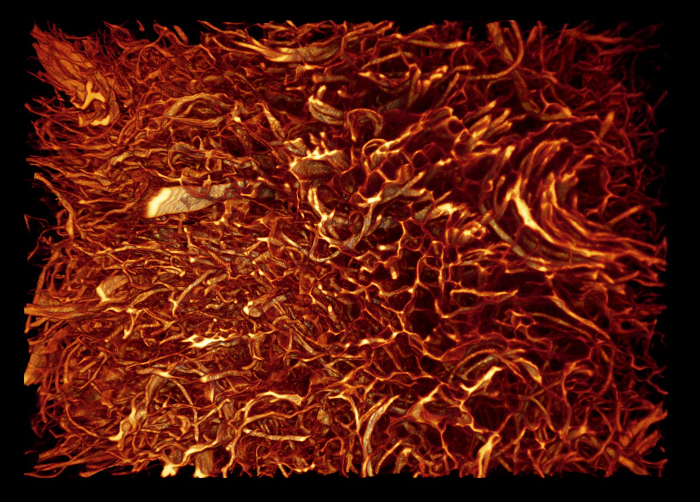
Animation 2: Modèle sous forme de volume du matériel de remplacement cutané natif. La taille de l'échantillon est d'environ 1,2 x 0,85 x 0,4 mm 3 . La taille de Voxel est de 0,54 x 0,54 x 2 μm 3 . Cliquez ici pour voir cette vidéo. (Cliquez avec le bouton droit de la souris pour télécharger.)
Discussion
HREM est une méthode microscopique hautement robuste, idéale pour visualiser un large éventail de matériaux organiques utilisés dans la biomédecine et l'industrie 18 , 21 , 26 , 27 , 28 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 , 38 , 39 , 40 . Il peut être utilisé comme une modalité d'imagerie exclusive, actuellement utilisée par le programme Mécanismes de troubles du développement (DMDD) 41 , 42 , 43 , 44 , ou comme une partie intégrante des pipelines d'imagerie multimodale 45 .
Un appareil de génération de données HREM entièrement opérationnel peut être assemblé à partir de composants de laboratoire classiques et comprend un microtome motorisé, un microscope, une table transversale motorisée et un ordinateur avec un logiciel approprié 25 . Il est essentiel d'utiliser un microtome équipé d'un support de bloc qui s'arrête de manière reproductible après chaque section à une position définie et des cubes de filtre GFP à l'intérieur de la voie optique. Cependant, des solutions tout compris entièrement fonctionnelles peuvent être achetées auprès d'entreprises telles que Indigo Scientific.
HREM fait face aux mêmes limites que toutes les techniques histologiques, sauf qu'aucun artefact n'est introduit pendant la section ou le montage en coupe. Cependant, il existe des limites qui résultent de la nécessité de tacher les spécimens avant la section etDes caractéristiques du matériau d'encastrement. La pénétration de l'éosine dans l'ensemble de l'échantillon est requise pour obtenir suffisamment de contrastes tissulaires; Un matériau très dense, des tissus adipeux et des substances anorganiques entrave efficacement la pénétration de l'éosine et cela entraîne des tissus non colorés au centre des objets. L'utilisation de fixateurs spéciaux aide à tacher les spécimens de peau, mais il n'existe toujours aucune méthode appropriée pour résoudre complètement le problème. Une autre limitation est que les résines qui bloquent plus de 2 cm ont tendance à se casser pendant la section. Cela peut être partiellement évité en coupant séparément les spécimens et les pièces de traitement.
Le positionnement correct de petits échantillons ou d'échantillons avec des surfaces irrégulières dans les moules lors de l'encastrement est souvent problématique. Couvrir les échantillons avec de l'agarose et traiter les blocs d'agarose comme décrit dans le protocole règle habituellement ce problème 19 . Une approche alternative, qui contribue également à la rupture des blocs pendant la SectiOn doit enlever le bloc déjà durci de son support et l'intégrer à nouveau, en suivant la procédure d'encastrement décrite.
Un ensemble de données HREM typique comprend 500 à 3 000 images simples. Sa résolution numérique est déterminée par la distance entre les images successives ( c'est-à - dire par épaisseur de section), la caractéristique de la cible de caméra et les propriétés de l'optique utilisée. Nous avons utilisé des épaisseurs de section comprises entre 1 μm et 5 μm et avons obtenu de bons résultats, bien que les protocoles présentés n'éliminent pas complètement le brillant des artefacts 20 , 46 . Ces artefacts sont causés par des tissus intensément colorés situés à l'intérieur du bloc, ce qui entraîne un flou des informations sur les tissus sur les surfaces des blocs.
Les caméras avaient des dimensions cibles de 2 560 x 1,920 pixels 2 , 2 048 x 2,048 pixels 2 et 4,096 x 4,096 pixels 2 et étaient combinésNed avec des lentilles d'objectif 1.25X, 2.5X, 5X, 10X et 20X. Il en résulte des tailles de pixels numériques comprises entre 0,18 x 0,18 μm 2 et 5,92 x 5,92 μm 2 , ce qui s'est révélé suffisant pour l'analyse 3D de l'architecture tissulaire et des formes cellulaires, et même pour la visualisation des noyaux. Compte tenu de la résolution numérique élevée, d'autres organites cellulaires devraient également être visibles. Des contrastes insuffisants dus à la coloration simple de l'éosine et les propriétés optiques des objectifs réduisent considérablement la possibilité de discriminer les structures. La résolution spatiale maximale maximale des données HREM, qui prend en compte l'ouverture numérique, est d'environ 1 x 1 x 1 μm 3 et ne permet donc qu'une discrimination efficace des structures supérieures à environ 3 x 3 x 3 μm 3 .
Un problème commun à toutes les techniques d'imagerie numérique est le compromis entre la taille du champ de vision, qui définit la partie du spécimen qui peut être affichéeD sur la cible de l'appareil photo, et la résolution numérique de l'image. Plus le champ de vision est grand, plus la résolution numérique maximale possible est inférieure à 46 . La configuration HREM utilisée ici permet la génération de données HREM avec un champ de vision entre 0,74 x 0,74 mm 2 (objectif 20X) affiché dans une résolution numérique de 0,18 x 0,18 μm 2 et 12,12 x 12,12 mm 2 (objectif 1,25X) affiché dans Une résolution numérique de 2,96 x 2,96 μm 2 . Des configurations alternatives et commerciales peuvent fournir de plus grands champs de vues, mais au prix d'une résolution réelle. Néanmoins, ils fournissent d'excellents résultats, comme le montrent les données affichées sur la page d'accueil du programme DMDD 47 .
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Les auteurs remercient Tim Mohun pour ses contributions inestimables dans le développement de HREM et Petra Heffeter pour la fourniture d'échantillons.
matériels
| Name | Company | Catalog Number | Comments |
| JB-4 Plus Embedding Kit | Polysciences Europe GmbH | 18570-1 | includes Benzoyl Peroxide, Plasticized (Catalyst) and Solution A+B |
| Polyethylene Molding Cup Trays, 6 x 8 x 5 mm hexagon (9 cavities) | Polysciences Europe GmbH | 17177A-3 | |
| Polyethylene Molding Cup Trays, 13 x 19 x 5 mm (9 cavities) | Polysciences Europe GmbH | 17177C-3 | |
| JB-4 Plastic Block Holders | Polysciences Europe GmbH | 15899-50 | |
| Eosin | Waldeck GmbH & Co. KG, Division Chroma | 1A-196 | |
| Microtec CUT 4060E | rotary microtome | ||
| Leica DM LM, fluorescence compound microscope | Leica Mikrosysteme Handelges.m.b.H | ||
| GFP filter set | Leica Mikrosysteme Handelges.m.b.H | 11090937180000 | |
| Motorised cross table | Walter Uhl, technische Mikroskopie GmbH & CO. KG | KT5-LSMA | |
| Digital video camera SPOT-FLEX | Visitron Systems GmbH. | ||
| precisExcite High-Power LED | Visitron Systems GmbH. | light source | |
| VisiView 2.1.4 | Visitron Systems GmbH. | Image capturing software | |
| Hard metal knife (tungsten carbide), profile D | Leica Mikrosysteme Handelges.m.b.H | ||
| KL 2500 LCD | Schott AG | light source |
Références
- Murray, K. K., Seneviratne, C. A., Ghorai, S. High resolution laser mass spectrometry bioimaging. Methods. 104, 118-126 (2016).
- Holzlechner, M., et al. In Situ Characterization of Tissue-Resident Immune Cells by MALDI Mass Spectrometry Imaging. J Proteome Res. 16 (1), 65-76 (2017).
- Elsayad, K., et al. Spectrally coded optical nanosectioning (SpecON) with biocompatible metal-dielectric-coated substrates. Proc Natl Acad Sci U S A. 110 (50), 20069-20074 (2013).
- Norris, F. C., et al. A coming of age: advanced imaging technologies for characterising the developing mouse. Trends Genet. 29 (12), 700-711 (2013).
- Schneider, J. E., et al. High-resolution imaging of normal anatomy, and neural and adrenal malformations in mouse embryos using magnetic resonance microscopy. J Anat. 202 (2), 239-247 (2003).
- Sharpe, J. Optical projection tomography as a new tool for studying embryo anatomy. J Anat. 202 (2), 175-181 (2003).
- Metscher, B. D. MicroCT for developmental biology: A versatile tool for high-contrast 3D imaging at histological resolutions. Dev Dyn. 238 (3), 632-640 (2009).
- Syed, S. H., Larin, K. V., Dickinson, M. E., Larina, I. V. Optical coherence tomography for high-resolution imaging of mouse development in utero. J Biomed Opt. 16 (4), 046004 (2011).
- Zhang, E. Z., et al. Multimodal photoacoustic and optical coherence tomography scanner using an all optical detection scheme for 3D morphological skin imaging. Biomed Opt Express. 2 (8), 2202-2215 (2011).
- Singh, M., et al. Applicability, usability, and limitations of murine embryonic imaging with optical coherence tomography and optical projection tomography. Biomed Opt Express. 7 (6), 2295-2310 (2016).
- Weninger, W. J., Meng, S., Streicher, J., Müller, G. B. A new episcopic method for rapid 3-D reconstruction: applications in anatomy and embryology. Anat Embryol (Berl). 197 (5), 341-348 (1998).
- Weninger, W. J., Mohun, T. Phenotyping transgenic embryos: a rapid 3-D screening method based on episcopic fluorescence image capturing. Nat Genet. 30 (1), 59-65 (2002).
- Khairy, K., Keller, P. J. Reconstructing embryonic development. Genesis. 49 (7), 488-513 (2011).
- Medalia, O., et al. Macromolecular architecture in eukaryotic cells visualized by cryoelectron tomography. Science. 298 (5596), 1209-1213 (2002).
- Berrios-Otero, C. A., Wadghiri, Y. Z., Nieman, B. J., Joyner, A. L., Turnbull, D. H. Three-dimensional micro-MRI analysis of cerebral artery development in mouse embryos. Magn Reson Med. 62 (6), 1431-1439 (2009).
- Liu, M., et al. Dual modality optical coherence and whole-body photoacoustic tomography imaging of chick embryos in multiple development stages. Biomed Opt Express. 5 (9), 3150-3159 (2014).
- Mohun, T. J., Leong, L. M., Weninger, W. J., Sparrow, D. B. The morphology of heart development in Xenopus laevis. Dev Biol. 218 (1), 74-88 (2000).
- Weninger, W. J., et al. High-resolution episcopic microscopy: a rapid technique for high detailed 3D analysis of gene activity in the context of tissue architecture and morphology. Anat Embryol. 211 (3), 213-221 (2006).
- Mohun, T. J., Weninger, W. J. Embedding Embryos for High-Resolution Episcopic Microscopy (HREM). Cold Spring Harb Protoc. 2012 (6), 678-680 (2012).
- Geyer, S. H., Mohun, T. J., Weninger, W. J. Visualizing vertebrate embryos with episcopic 3D imaging techniques. ScientificWorldJournal. 9, 1423-1437 (2009).
- Bruneel, B., et al. Imaging the zebrafish dentition: from traditional approaches to emerging technologies. Zebrafish. 12 (1), 1-10 (2015).
- Adams, D., et al. Bloomsbury report on mouse embryo phenotyping: recommendations from the IMPC workshop on embryonic lethal screening. Dis Model Mech. 6 (3), 571-579 (2013).
- Heddleston, J. M., Chew, T. L. Light sheet microscopes: Novel imaging toolbox for visualizing life's processes. Int J Biochem Cell Biol. 80, 119-123 (2016).
- Powell, K. A., Wilson, D. 3-dimensional imaging modalities for phenotyping genetically engineered mice. Vet Pathol. 49 (1), 106-115 (2012).
- Geyer, S. H., Mohun, T. J., Kamolz, L. P., Weninger, W. J. High resolution episcopic microscopy - current applications. Current Biotechnology. 1 (4), 281-286 (2012).
- Weninger, W. J., Maurer, B., Zendron, B., Dorfmeister, K., Geyer, S. H. Measurements of the diameters of the great arteries and semi-lunar valves of chick and mouse embryos. J Microsc. 234 (2), 173-190 (2009).
- Geyer, S. H., Maurer, B., Dirnbacher, K., Weninger, W. J. Dimensions of the great intrathoracic arteries of early mouse fetuses of the C57BL/6J strain. The Open Anatomy Journal. 4 (1), 1-6 (2012).
- Geyer, S. H., Maurer, B., Pötz, L., Singh, J., Weninger, W. J. High-Resolution Episcopic Microscopy Data-Based Measurements of the Arteries of Mouse Embryos: Evaluation of Significance and Reproducibility under Routine Conditions. Cells Tissues Organs. 195 (6), 524-534 (2012).
- Geyer, S. H., Weninger, W. J. Some Mice Feature 5th Pharyngeal Arch Arteries and Double-Lumen Aortic Arch Malformations. Cells Tissues Organs. 196 (1), 90-98 (2012).
- Kee, H. J., et al. Ret finger protein inhibits muscle differentiation by modulating serum response factor and enhancer of polycomb1. Cell Death Differ. 19 (1), 121-131 (2012).
- Geyer, S. H., Nohammer, M. M., Tinhofer, I. E., Weninger, W. J. The dermal arteries of the human thumb pad. J Anat. 223 (6), 603-609 (2013).
- Geyer, S. H., Weninger, W. J. Metric characterization of the aortic arch of early mouse fetuses and of a fetus featuring a double lumen aortic arch malformation. Ann Anat. 195 (2), 175-182 (2013).
- Maurer, B., Geyer, S. H., Weninger, W. J. A Chick Embryo With a yet Unclassified Type of Cephalothoracopagus Malformation and a Hypothesis for Explaining its Genesis. Anat Histol Embryol. 42 (3), 191-200 (2013).
- Geyer, S. H., et al. High-Resolution Episcopic Microscopy (HREM): A Tool for Visualizing Skin Biopsies. Microsc Microanal. 20 (5), 1356-1364 (2014).
- Weninger, W. J., et al. Phenotyping structural abnormalities in mouse embryos using high-resolution episcopic microscopy. Dis Model Mech. 7 (10), 1143-1152 (2014).
- Geyer, S. H., et al. High-resolution episcopic microscopy (HREM): A useful technique for research in wound care. Ann Anat. 197, 3-10 (2015).
- Captur, G., et al. Morphogenesis of myocardial trabeculae in the mouse embryo. J Anat. 229 (2), 314-325 (2016).
- Wong, R., Geyer, S., Weninger, W., Guimberteau, J. C., Wong, J. K. The dynamic anatomy and patterning of skin. Exp Dermatol. 25 (2), 92-98 (2016).
- Jenner, F., et al. The embryogenesis of the equine femorotibial joint: The equine interzone. Equine Vet J. 47 (5), 620-622 (2015).
- Wiedner, M., et al. Simultaneous dermal matrix and autologous split-thickness skin graft transplantation in a porcine wound model: a three-dimensional histological analysis of revascularization. Wound Repair Regen. 22 (6), 749-754 (2014).
- Mohun, T., et al. Deciphering the Mechanisms of Developmental Disorders (DMDD): a new programme for phenotyping embryonic lethal mice. Dis Model Mech. 6 (3), 562-566 (2013).
- Wilson, R., et al. Highly variable penetrance of abnormal phenotypes in embryonic lethal knockout mice. Wellcome Open Res. 1, 1 (2016).
- Wilson, R., McGuire, C., Mohun, T. Deciphering the mechanisms of developmental disorders: phenotype analysis of embryos from mutant mouse lines. Nucleic Acids Res. 44 (D1), D855-D861 (2016).
- Dickinson, M. E., et al. High-throughput discovery of novel developmental phenotypes. Nature. 537 (7621), 508-514 (2016).
- Pieles, G., et al. microMRI-HREM pipeline for high-throughput, high-resolution phenotyping of murine embryos. J Anat. 211 (1), 132-137 (2007).
- Weninger, W. J., Geyer, S. H. Episcopic 3D Imaging Methods: Tools for Researching Gene Function. Curr Genomics. 9, 282-289 (2008).
- . Deciphering the Mechanisms of Developmental Disorders: DMDD Available from: https://dmdd.org.uk/ (2017)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon