Method Article
Visualisation des cellules multiciliées dans le poisson-zèbre à travers un protocole combiné de toute monture Fluorescent In Situ hybridation et Immunofluorescence
Dans cet article
Résumé
Développement de cils est vitale pour l’organogenèse appropriée. Ce protocole décrit une méthode optimisée pour étiqueter et de visualiser les cellules ciliées du poisson-zèbre.
Résumé
Ces dernières années, l’embryon de poisson zèbre est devenue un modèle populaire pour étudier la biologie du développement en raison de caractéristiques telles que l’ex utero le développement de l’embryon et de la transparence optique. En particulier, l’embryon de poisson zèbre est devenu un organisme important d’étudier l’organogenèse rein vertébrés ainsi que le développement de cellules multiciliées (MCC). Pour visualiser les tableaux dans le rein de l’embryon de poisson zèbre, nous avons développé un protocole combiné de l’ensemble-mount fluorescent in situ hybridation (poisson) et monter ensemble immunofluorescence (IF) qui permet l’imagerie haute résolution. Ce manuscrit décrit notre technique pour localiser conjointement les transcriptions d’ARN et de protéine comme un outil pour mieux comprendre la réglementation des programmes de développement par le biais de l’expression des divers facteurs de la lignée.
Introduction
Depuis les dernières décennies, le poisson zèbre (Danio rerio) est devenu un organisme modèle privilégié pour étudier la biologie du développement. Les embryons se développent en dehors de la mère et sont optiquement transparents. En outre, la formation des organes vitaux tels que le œil, des reins et du cerveau se produit rapidement, avec des structures formées par seulement 24 heures après la fécondation (hpf). Ce qui est important, le génome du poisson-zèbre est très conservé avec mammifères1,2,3. En outre, poisson-zèbre et organes de mammifères ont similaires l’anatomie et la physiologie. Le poisson-zèbre rein embryonnaire ou pronéphros, démontre la valeur du système modèle pour étudier la fonction du gène pendant néphrogénèse précoce et de la détermination du sort des populations de cellules épithéliales conservée du néphron vertébrés4, 5 , 6 , 7 , 8 , 9 , 10. de même, l’embryon de poisson zèbre est devenu de plus en plus important dans l’examen de l’ontogénie du MCCs11,12,13,14,15, 16 , 17.
Comme leur nom l’indique, le CCM est des cellules épithéliales, caractérisés par un faisceau de cils mobiles situées sur la surface apicale17. Dans le poisson-zèbre, MCCs fonctionnent dans les écoulements de fluides et sont dispersées dans un « poivre » comme la mode dans le milieu de chaque néphron du pronéphros par 24 hpf11,12,13,14, 15 , 16 , 17. bien qu’ils n’ont été notés dans une poignée de rein humain maladie cas18,19,20,21, CCM est fréquents dans les autres tissus comme le cerveau des mammifères et trachée22,23,24, qui pose une multitude de défis pour la conception expérimentale. Des études élégants dans divers modèles vertébrés, y compris le poisson-zèbre ont démontré une voie conservée du destin MCC, avec l’encoche voie de signalisation comme inhibiteur de la MCC développement12,17,25 ,26,27. Par conséquent, le poisson-zèbre pronéphros fournit un modèle facilement accessible afin d’étudier les mécanismes génétiques de MCC development in vivo11,12,13,14, 15 , 16 , 17.
Transparence tant facile manipulation génétique des embryons de poissons zèbres se sont avérés pour être des traits précieux lorsque l'on étudie les voies génétiques et moléculaires qui régulent le sort de la cellule, la croissance des tissus et le développement de l' embryon précoce1,2 ,3. Comme telles, techniques traditionnelles pour visualiser les transcriptions de protéines et de gènes, tels que l’hybridation in situ et toute monture si, ont été appliqués aux et optimisé pour le poisson-zèbre16,28,29, 30,31,32,33,34,35,36,37,38. En combinant les protocoles populaires pour les poissons et si, il est possible d’étiqueter et d’analyser les CCM en vivo28,16,37,38.
Protocole
Le protocole suivant utilise des poissons zèbres adultes maintenus et pris en charge par le Centre pour la recherche de poisson-zèbre à l’Université de Notre Dame. Toutes les méthodes pour travailler avec des embryons et des poissons zèbres adultes ont été approuvées par le Comité de l’emploi et d’institutionnels animalier.
1. Fixation de l’embryon
- Recueillir des embryons de poisson-zèbre à l’aide des méthodes décrites précédemment39,40. À 24 hpf, ajouter 500 µL de solution de mélange de protéase non spécifique (50 mg/mL) à l’E3 dans un embryon plat incuber à température ambiante (RT).
- Une fois les embryons commencent à sortir du chorion, évacuer tout le liquide de la capsule de l’embryon.
- Lavez les embryons 2 - 3 fois avec frais E3 en ajoutant environ 20 mL d’E3, agitant doucement l’E3 dans le plat et puis transvaser le liquide dans un réceptacle de déchets liquid.
- Pour euthanasier les embryons avant fixation, ajouter 2 mL de tricaïne 0,2 % pour l’E3.
- Transférer les embryons dans un flacon en verre de 5 mL à l’aide d’une pipette Pasteur de plastique. Enlever la plus grande partie de la solution de tricaïne/E3 à l’aide de la pipette, après que les embryons sont sont installés au fond du flacon.
- Difficulté 24 embryons hpf avec 5 mL de paraformaldéhyde à 4 % (PFA) pour 2-4 h à ta, sans agitation.
ATTENTION : PFA est toxique et des solutions PFA doivent être manipulées sous une hotte chimique, tandis que le chercheur porte des lunettes de protection, des gants et une blouse de laboratoire. Préparation du fixateur PFA de la poudre PFA granulé doit être effectuée avec des soins exceptionnels, comme la PFA granulé tend à se disperser dans une charge statique.
Remarque : Les embryons peuvent fixer dans 4 % PFA pour la nuit à 4 ° C. Préparer 4 % PFA en chauffant 1 x tamponné phosphate salin (PBS) à ébullition en remuant sans cesse la solution pour dissoudre complètement la PFA granulé. Une fois que la solution est refroidie vers le bas à RT, les 4 % PFA est filtre stérilisé en le faisant passer à travers un système jetable de 0,45 µm pour filtration sous vide, puis et aliquotés en portions de 15 mL ou 50 mL pour le stockage à-20 ° C. - Enlevez les 4 % PFA et embryons de laver deux fois avec 1 tamponné de phosphate X saline avec 0,1 % Tween-20 (PBST) à température ambiante. Lavez les embryons une fois avec 5 mL de méthanol (MeOH) à 100 % à RT. Ajouter 5 mL de frais 100 % MeOH pour les embryons et incuber à-20 ° C pendant au moins 20 min.
Remarque : Les embryons peuvent être stockés dans 100 % MeOH à-20 ° C jusqu’au moment de la préparation de l’embryon.
2. hybridation et préparation de l’embryon
- Réhydrater les embryons par un lavage une fois dans 5 mL de 50 % MeOH dans 1 x PBST pendant 5 min à continuer RT. réhydratation en lavant les embryons dans 5 mL de 30 % MeOH dans 1 x PBST pendant 5 min à température ambiante. Lavez les embryons dans 5 mL de 1 x PBST deux fois pour 5 min chacun, à RT.
- Permeabilize les embryons en incubant eux dans 5 mL d’une solution de 1 : 1 000 protéinase K (pK) (10 mg/mL) à 1 x PBST pendant 2 min à température ambiante.
Remarque : Les embryons âgés peuvent être incubés plus longtemps que 2 min de la concentration de pK indiquée ci-dessus, mais une concentration de 5 mg/mL et incubation temps moins de 2 min est suggérée pour les embryons âgés de moins de 24 hpf. - Enlever la solution de pK et laver immédiatement en 5 mL 1 x PBST deux fois à température ambiante. fixer les embryons dans 5 mL de 4 % PFA à ta pendant au moins 20 min.
Remarque : Les embryons peuvent fixer dans 4 % PFA pour la nuit à 4 ° C. - Supprimer les PFA et laver deux fois dans 5 mL de 1 x PBST à température ambiante.
- Transfert des embryons dans 5 mL de 1 x PBST dans des tubes de microcentrifuge de fond plat placé debout dans un panier de microcentrifuge RT pour faciliter les interactions entre chaque embryon et la solution d’hybridation ultérieure.
- Lavez les embryons deux fois en 1,5 mL de solution d’hybridation (Hyb +) à RT. Ajouter 1,5 mL de frais Hyb + et incuber les embryons à 70 ° C dans un four à hybridation pour 4 à 6 h.
- Remplacer les 1,5 mL de Hyb + avec un volume de solution de sonde (10 µL RNA sonde/500 µL Hyb +) suffisant pour couvrir entièrement les embryons.
Remarque : Une synthèse antisens sonde ARN utilisant précédemment décrit des méthodes39. - Hybrider la sonde pendant la nuit dans le four de l’hybridation à 70 ° C avec le poteau de tubes individuels placés dans le panier de microcentrifuge, sans agitation.
3 chaudes lavages et blocage
NOTE : Hot lavages sont effectuées en mettant la solution appropriée sur les embryons et puis incubation dans le four de l’hybridation à 70 ° C. Pour conserver les solutions de lavage à 70 ° C, placer les tubes de 50 mL de chaque solution dans le four à hybridation lorsque la sonde / Hyb + mélange est ajouté.
- Retirer la sonde. Lavez les embryons deux fois à 70 ° C à 1,5 mL de 50 % formamide/2 x tampon citrate de solution saline-sodium (SSC) pendant 20-30 min chaque.
Remarque : La sonde peut être stockée dans Hyb + à-20 ° C et ré-utilisée à une date ultérieure. - Laver une fois les embryons à 70 ° C à 1,5 mL de 2 x SSC pendant 15 min. Lavez les embryons deux fois à 70 ° C à 1,5 mL de 0,2 X SSC de 20-30 min chacun. X SSC remplacer 0,2 à 1,5 mL de réactif de blocage et incuber une nuit à 4 ° C.
Remarque : Il est possible d’incuber blocage réactif à ta pendant 4 h au lieu de la nuit.
4. anticorps d’Incubation et de lavages de tampon acide maléique
NOTE : Garder les embryons abri de la lumière ambiante.
- Remplacer le réactif de blocage avec 0,2 mL d’anti-DIG-POD en bloquant réactif à un ratio de 1 : 1 000 et incuber pendant 3 heures sous une feuille ou d’une autre couverture occultants à température ambiante.
Remarque : Par ailleurs, les anticorps peuvent incuber pendant la nuit à 4 ° C. - Après 3 h, lavez les embryons dans 1,5 mL de tampon de l’acide maléique 2 - 4 fois pour 10-15 min chacun, à RT. Incuber les embryons pendant la nuit à 4 ° C à 1,5 mL de tampon de l’acide maléique fraîches.
Remarque : Il n’est pas nécessaire d’incuber pendant la nuit dans le tampon de l’acide maléique, mais cela diminue donc fond de sonde.
5. sonde de détection et la suppression de la peroxydase
NOTE : Garder les embryons abri de la lumière ambiante.
- Retirer le tampon de l’acide maléique et remplacez par 1,5 mL de PBS 1 x. Lavez les embryons deux fois dans 1,5 mL de PBS 1 x de 5 min chacun à RT. Incuber les embryons dans 0,2 mL de Cy3 fluorescent coloration solution pendant 60 min à température ambiante.
Remarque : Le temps d’Incubation peut varier pour différentes sondes. - Lavez les embryons avec 1,5 mL de la MeOH pourcentage suivant en 1 X PBS pendant 10 min à RT : 30 % MeOH, 50 % MeOH, 75 % MeOH et 100 % MeOH.
- Incuber les embryons avec 1,5 mL de 1 % H2O2 dans MeOH pendant 30 min à RT. Wash les embryons avec 1,5 mL de MeOH solutions suivantes dans du PBS 1 x pendant 10 min à RT : 75 % MeOH, 50 % MeOH et 30 % MeOH. Lavez les embryons deux fois plus de 5 min chacun à ta dans 1,5 mL de PBS 1 x.
Remarque : Les embryons peuvent être stockés dans du PBS pendant la nuit à 4° C.
6. immunofluorescence
NOTE : Garder les embryons abri de la lumière ambiante.
- Lavez les embryons dans 1,5 mL de ddH2O pendant 5 min à RT. Wash les embryons dans 1,5 mL d’acétone préalablement réfrigérée (conservé à-20 ° C) à 7 min à température ambiante. Wash les embryons une fois dans 1,5 mL de ddH2O pendant 5 min à RT. Wash les embryons une fois à 1.5 mL de 1 x PBST avec 1 % de DMSO (PBDT) pendant 5 min à température ambiante.
- Incuber les embryons dans 1,5 mL de PBDT + 10 % sérum fœtal (SVF) sur un rocker à ta pendant 2 h.
- Supprimer PBDT + FBS et remplacer avec 1,5 mL d’anticorps primaires, tubuline anti-acétylé (produit à partir de souris) et anti-γ-tubuline (issus de lapin), dilué ensemble à 1 : 400 PBDT + 1 % FBS. Incuber les embryons pendant une nuit à 4 ° C.
Remarque : Le temps d’Incubation peuvent varier selon l’anticorps primaire. Essais de différents intervalles de temps peuvent être nécessaires pour optimiser l’étiquetage.
7. secondaires anticorps
NOTE : Garder les embryons abri de la lumière ambiante.
- Pour éliminer les anticorps non lié excès des embryons, laver dans 1,5 mL de PBDT + 1 % FBS + 0,1 M NaCl, pendant 1 min à ta sur un rocker.
- Lavez les embryons 5 fois dans 1,5 mL de PBDT + 1 % FBS + 0.1 M de NaCl de 30 min chacun sur un rocker à température ambiante. Lavez les embryons dans 1,5 mL de PBDT + 1 % FBS pendant 30 min à ta sur un rocker.
- Incuber les embryons pendant la nuit à 4 ° C à 200 µL de l’anticorps secondaires, Alexa 488 chèvre anti-souris IgG et Alexa 647 chèvre anti-lapin IgG, dilué ensemble dans PBDT au 1/500.
8. la coloration DAPI
NOTE : Garder les embryons abri de la lumière ambiante.
- Lavez les embryons rapidement à ta dans 1,5 mL de PBDT deux fois. Remplacer les PBDT avec 1,5 mL de DAPI, dilué 1:15000 dans 1 X PBST et incuber 15 min à ta sur un rocker.
- Lavez les embryons dans 1,5 mL de PDBT + 1 % FBS + 0,1 M NaCl trois fois à ta pendant 15-20 min chaque. Conservation des embryons en 1,5 mL de PBDT à 4 ° C jusqu’au moment de monter et de l’image.
9. montage et d’imagerie pour le poisson-zèbre pronéphros
- Embryon laïcs latérale sur la lame de verre dans un petit volume, environ 10 µL, de PBDT.
- Presser l’embryon derrière les yeux avec une paire de pinces fines pour retirer la tête et certains de la balle jaune.
- Utilisez la pince fine pour gratter doucement la boule jaune d’oeuf restant du corps de l’embryon. Retirez le jaune dissocié et le liquide supplémentaire de la diapositive avec un tissu mince.
- Ajouter une goutte de montage multimédia à la queue restante de l’embryon et placer pour que la queue est disposée latéralement. Placez un lamelle couvre-objet sur le dessus de la queue pour aplatir l’échantillon, puis placer dans une boîte couverte de diapositive.
- Cils de rein au grossissement 60 X sur un microscope confocal, via les canaux suivants d’image : DAPI en bleu (laser ensemble à 408.0 nm), FITC en vert (laser ensemble à 488,0 nm), Cy3 marqués au colorant en rouge (laser ensemble à 561.0 nm) et Alexa 680 en blanc (laser ensemble à 637,0 nm).
Résultats
Des embryons de poisson-zèbre sauvage ont été fixés à 24 hpf et immédiatement préparé comme indiqué ci-dessus. La figure 1 illustre un flux de travail expérimental ainsi que les étapes illustrées sélectionnées. Le flux de travail décrite aux étapes 1 à 8 englobe les processus d’approvisionnement de l’échantillon, la fixation et manipulation du tissu fixe d’étiqueter les transcriptions endogènes avec cavitation antisense suivie par immunofluorescence à étiquette protéines d’intérêt. Étape 9 dans le flux de travail désigne spécifiquement conçus pour optimiser la visualisation du poisson-zèbre tronc embryonnaire de techniques d’imagerie et le montage. Dans les dessins qui accompagnent l’étape 9, nous illustrons la méthode de manipulation permettant de positionner le tissu sur une lame de verre. Dans cette étape, la boule de tête et le jaune de œuf embryon sont retirés, laissant la queue pour être positionnés latéralement entre lame et lamelle. Enlèvement de la boule jaune d’oeuf et la tête est suggérée pour la meilleure image de la population résidente de la MCC dans le pronéphros, comme la boule jaune auto-fluorescente et entrave le processus de montage.
Les images représentatives obtenues à l’aide d’un microscope confocal figurent dans la Figure 2, qui montre le même embryon sauvage au grossissement 60 X et un zoom numérique au même grossissement. Les meilleurs panneaux fournissent des images de chaque canal individuellement, tandis que les deux panneaux de fond sont la superposition composite de ces données d’imagerie. La case blanche (à l’image de la colonne de gauche) décrit la zone qui est au cœur du zoom numérique (l’image de la colonne de droite). Mis en évidence dans la dernière case du zoom numérique, noyaux ont été marquées au DAPI et CCM ont été détectés, basé sur un ribosonde antisens conçu pour reconnaître les odf3b, où les anticorps anti-γ-tubuline désigne les corpuscules basaux et α-tubuline indique les cils. Dans le zoom numérique de l’incrustation en composite, le cercle en pointillés jaune indique le périmètre d’un MCC, qui a été identifié en raison de la possession des transcriptions de odf3b , plusieurs corpuscules basaux et cils. La cellule adjacente mono-ciliées, étiquetée avec un cercle en pointillé jaune, a été identifiée basées sur le phénotype de posséder un seul corps basal et un seul cil.
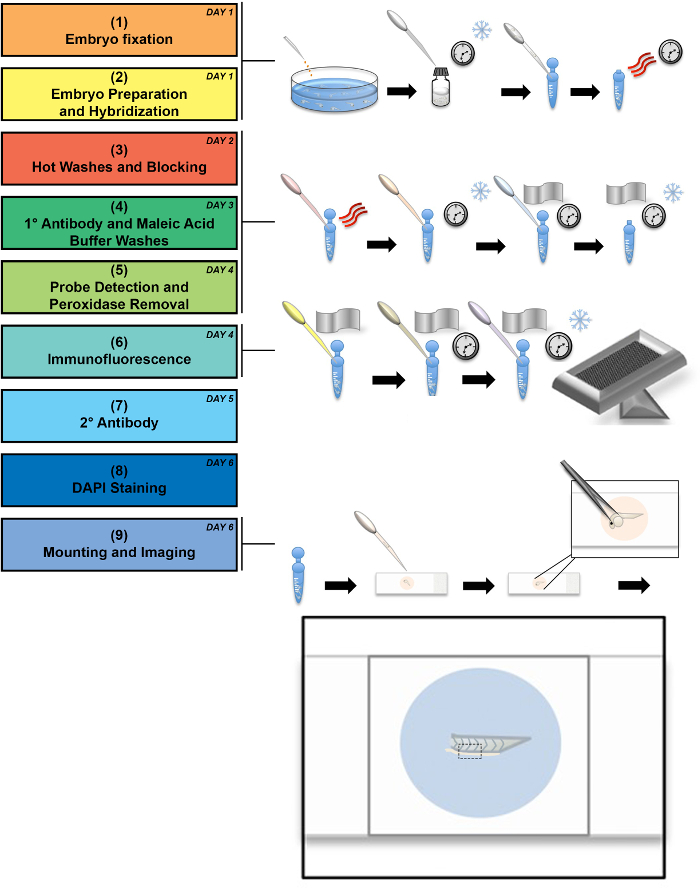
Figure 1 : schéma de diagramme expérimental. Cette organigramme montre un flux de travail expérimental, accompagnée d’illustrations des étapes critiques, au cours duquel le flocon indique incubation froide, les trois lignes courbes représentent la chaleur et l’horloge représente le temps d’incubation longue. Le flux de travail peut être effectuée dans un minimum de 6 jours (voir le numéro du jour dans le coin supérieur droit de chaque étape du processus), même si certaines mesures peuvent être effectuées sur de longues périodes de temps, tel qu’indiqué dans le protocole. Une description détaillée du processus de montage est dessinée, la queue de l’embryon montée finale entre la lame et lamelle. Une boîte noire pointillée indique la zone qui est imagée au grossissement 60 X. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : représentant les résultats de visualisation MCCs du pronéphros zebrafish. Projections de maximale de l’image d’un embryon de poisson zèbre sauvage hpf 24 à 60 X grossissement, mais aussi un zoom numérique sur la confocale à grossissement 60 X de la même embryon. Les cases blanches indiquent la région a été consacrée pour le zoom. Taches individuelles de DAPI (noyaux), odf3b (CCM), γ-tubuline (corpuscules basaux) et α-tubuline (cils) sont étiquetés et ensuite fusionnées dans les panneaux de fond deux. Dans le zoom numérique, nous fournissons des approximations de l’emplacement de la cellule comme suit : un tableau est délimité par le cercle en pointillés de jaune, et le cercle en pointillé jaune indique une cellule mono-ciliées. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Le protocole décrit ci-dessus est optimisé pour marquage CCM dans le pronéphros avec transcriptions odf3b et α-tubuline dans des embryons de poisson-zèbre 24 hpf. Pour de meilleurs résultats, il est recommandé d’utiliser des embryons fraîchement fixes et préparés. Les embryons qui ont été fixés et stockés dans MeOH ou Hyb + à-20 ° C pendant plus d’une semaine peuvent être utilisés, mais la probabilité de fond indésirables augmente avec le temps que les embryons sont conservés dans le stockage de coloration.
Modifications et dépannage de cette technique sont nécessaires pour adapter cette méthode pour les autres suites de marqueurs particulières, par exemple les autres anticorps et autres cavitation antisense. Plusieurs méthodes permettant d’ajuster les paramètres des étapes du processus d’hybridation, support entier in situ ont été documentés précédemment par notre groupe et d’autres31,34,36,38, 39. De même, chaque protéine anticorps nécessitera quelques dépannage en termes de temps de coloration et gammes approximatives de dilutions et temps d’incubation pour chaque anticorps primaire sont mieux estimés par évaluation des résultats documentés28, 35. pour les embryons âgés de moins de 24 hpf, pK traitement devrait être inférieur à 2 min, où des embryons plus vieux que 24 hpf devrait être traitées avec pK pendant plus de 2 min. En outre, des embryons plus vieux que 24 hpf devrait être blanchi de pigment avant traitement pK.
Après coloration avec la solution de coloration fluorescente, il est essentiel d’enlever toute tache, anticorps et peroxydases restants en accomplissant la série de lavages de méthanol et d’eau oxygénée. Les lavages de PBS immédiatement après sont vitales pour enlever l’excès de méthanol des embryons. Ce qui est important, avant de poursuivre avec les anticorps de l’IF, nous avons trouvé que l’acétone et eau déionisée lavages faire les embryons moins susceptibles d’adhérer à un autre et/ou les tubes à centrifuger. Dans notre expérience, les anticorps pour les facteurs de transcription spécifiques et d’autres gènes, bien qu’ils peuvent travailler efficacement taches occidentales, ne fonctionnent pas bien, car si dans le poisson-zèbre. Toutefois, des protéines hautement conservées et abondantes, comme la tubuline α et β-caténine, fonctionnent bien dans le poisson-zèbre pour IF16,37.
Avec du poisson, il est possible de visualiser des ARN transcrits des gènes qui n’ont pas encore d’anticorps spécifiques dans le poisson-zèbre. En combinant les poissons avec IF, comme en témoigne le présent protocole, co-localisation des transcriptions d’ARN et de protéine peut être vu en vivo (Figure 2). La souplesse du protocole permet un dépannage rapide pour la visualisation de diverses transcriptions d’ARN et de protéines dans de nombreux tissus et points dans le temps. Lorsqu’il est combiné avec les traits déjà respectés des embryons de poisson-zèbre, ce protocole de poisson + si fournit un autre outil pour étudier l’expression des gènes et des protéines importantes du règlement de la voie du développement.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu en partie par la subvention R01DK100237 à RAW et la National Science Foundation Graduate Research Fellowship no DGE-1313583 à A.N.M. Nous remercions également le Collège de Science Summer Undergraduate Research Fellowship Program pour soutien à M.U. Nous tenons à remercier le Centre pour la recherche de poisson-zèbre à l’Université de Notre Dame pour leurs soins dévoués de notre poisson-zèbre. Nous tenons également à remercier le département des Sciences biologiques ainsi que les membres de notre laboratoire pour l’ensemble de leur soutien et leur compréhension valable.
matériels
| Name | Company | Catalog Number | Comments |
| non-specific protease mixture solution | Roche | 11459643001, pronase from Streptomyces griseus | Dilute in E3 without methylene blue to make 50mg/mL stock solution; store at -20°C |
| E3 solution | Dilute 50X E3 (250 mM NaCl, 8.5 mM KCl, 16.5 mM CaCl2, 16.5 mM MgSO4 in distilled water) in distilled water; add 1 x 10-5 M methylene blue (sigma M9140) to inhibit contamination | ||
| tricaine (ethyl 3-aminobenzoate methanesulfonate) | Fluka | A5040-250G | To make a 0.2% stock solution, dissolve 1 g of tricaine powder in 10 mL Tris, pH 9.5 and distilled water up to 500 mL |
| embryo dishes | VWR | 351029 | |
| 5 mL glass vials | Wheaton | 225012 | |
| disposable plastic Pasteur pippettes | VWR | 414004-004 | |
| 4% paraformaldehyde (PFA) solution | Electron Microscopy Services | 19210 | Dissolve 4% PFA in 1X PBS and bring to a boil under a fume hood. Cool and aliquot, then store at -20°C. Do not refreeze once thawed for use. |
| 10X PBS | American Bioanalytical | AB11072 | Dilute in distilled water to make a 1X stock |
| Tween-20 stock | American Bioanalytical | AB02038 | Make a 0.1% Tween-20 stock by diluting in distilled water. |
| 1X phosphate buffered saline with 0.1% Tween 20 (PBST) | Sigma | P9416 | 0.1% Tween-20 in 1X PBS |
| methanol (MeOH) | Sigma | 34860-4L | |
| proteinase K | Roche | 3115879001 | Dissolve in distilled water to make a 10 mg/mL stock; aliquot and store at -20°C |
| flat bottom microcentrifuge tubes | VWR | 87003-300; 87003-298 | |
| formamide | American Bioanalytical | AB00600 | store at -20°C |
| hybridization solution (HYB+) | 50% formamide, 5X SSC, 0.1% Tween-20, 5 mg/mL yeast torula RNA, 50 μg/μL heparin; store at -20°C | ||
| hybridization oven | Fisher Scientific | 13-247-10Q | |
| 20X saline-sodium citrate (SSC) buffer solution | American Bioanalytical | AB13156 | dilute in distilled water to make 2X and 0.2X stocks |
| blocking reagent solution | Roche | 11096176001 | dilute in maleic acid buffer to make a 10% stock solution; store at 4°C |
| maleic acid buffer solution | Sigma | M0375 and S7653 | 150mM maleic acid, 100mM NaCl (pH 7.5) |
| anti-Digoxigenin-POD, Fab fragments | Roche | 11207739910 | store at 4°C |
| Cy3 fluorescent staining solution | PerkinElmer, Inc. | NEL744001KT, TSA Plus Cyanin3 system | store at 4°C; prepare staining solution fresh by making a 1:50 dilution of TSA reagent (dissolved in 60 uL of DMSO) in the kit buffer |
| Hydrogen peroxide (H2O2) | Sigma | H1009-500mL | store at 4°C |
| molecular grade distilled water (ddH2O) | Mediatech | 25-055-CM | |
| acetone | American Bioanalytical | AB00636-01000 | store an aliquot at -20°C |
| DMSO | American Bioanalytical | AB00435-01000 | |
| fetal bovine serum (FBS) | Gibco | 10438-034 | aliquot and store at -20°C |
| monoclonal anti-acetylated tubulin clone 6-11B-1 | Sigma-Aldrich | T6793 | aliquot and store at -20°C |
| anti-γ-tubulin anitbody produced in rabbit | Sigma-Aldrich | T5192 | aliquot and store at -20°C |
| odf3b cDNA clone MGC:63985 | OpenBiosystems | IMAGE:6792478 | store bacterial glycerol stock at -80°C |
| NaCL | American Bioanalytical | AB01915-05000 | |
| Alexa flour 647 goat anti-rabbit IgG | Life Technologies | A21245 | store at 4°C; protect from light |
| Alexa fluor 488 goat anti-mouse IgG | Life Technologies | A11029 | store at 4°C; protect from light |
| DAPI | Life Technologies | D1306 | aliquot and store at -20°C |
| glass slide | Thermo-Fisher | 4445 | |
| glass coverslip | Thermo-Fisher | 12-540A | 18 x 18 mm |
| fine forceps | Roboz | RS-1050 | Dumont Tweezers Pattern #55 |
| mounting media | Polysciences, Inc. | 18606, Aqua-Poly/Mount | store at 4°C |
| confocal microscope and associated software | We use a Nikon C2plus Confocal Microscope with NIS-elements AR software | ||
| rocker | Bio-Rad | 1660710EDU |
Références
- Lieschke, G. J., Currie, P. D. Animal models of human disease: zebrafish swim into view. Nat Rev Genet. 8, 353-367 (2007).
- Goldsmith, J. R., Jobin, C. Think small: zebrafish as a model system of human pathology. J Biomed Biotechnol. 2012, 817341(2012).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496, 498-503 (2013).
- Drummond, I. A. Kidney development and disease in the zebrafish. J Am Soc Nephrol. 16, 299-304 (2005).
- Wingert, R. A., et al. The cdx genes and retinoic acid control the positioning and segmentation of the zebrafish pronephros. PLoS Genet. 3, 1922-1938 (2007).
- Wingert, R. A., Davidson, A. J. The zebrafish pronephros: a model to study segmentation. Kindey Int. 73 (10), 1120-1127 (2008).
- Wingert, R. A., Davidson, A. J. Zebrafish nephrogenesis involves dynamic spatiotemporal expression changes in renal progenitors and essential signals from retinoic acid and irx3b. Dev Dyn. 240, 2011-2027 (2011).
- Chambers, B. E., Wingert, R. A. Renal progenitors: roles in kidney disease and regeneration. World J Stem Cells. 8 (11), 367-375 (2016).
- Drummond, B. E., Wingert, R. A. Insights into kidney stem cell development and regeneration using zebrafish. World J Stem Cells. 8 (2), 22-31 (2016).
- Poureetezadi, S. J., Wingert, R. A. Little fish, big catch: zebrafish as a model for kidney disease. Kidney Int. 89 (6), 1204-1210 (2016).
- Kramer-Zucker, A. G., Olale, F., Haycraft, C. J., Yoder, B. K., Schier, A. F., Drummond, I. A. Cilia-driven fluid flow in the zebrafish pronephros, brain and Kupffer's vesicle is required for normal organogenesis. Development. 132, 1907-1921 (2005).
- Liu, Y., Narendra, P., Kramer-Zucker, A., Drummond, I. A. Notch signaling controls the differentiation of transporting epithlelia and multiciliated cells in the zebrafish pronephros. Development. 134, 1111-1122 (2007).
- Ma, M., Jiang, Y. J. Jagged2a-Notch signaling mediates cell fate choice in the zebrafish pronephric duct. PLoS Genetics. 3, e18(2007).
- Li, Y., Cheng, C. N., Verdun, V. A., Wingert, R. A. Zebrafish nephrogenesis is regulated by interactions between retinoic acid, mecom, and Notch signaling. Dev Biol. 386 (1), 111-122 (2014).
- Wang, L., et al. miR-34b regulates multiciliogenesis during organ formation in zebrafish. Development. 140, 2755-2764 (2013).
- Marra, A. N., Wingert, R. A. Epithelial cell fate in the nephron tubule is mediated by the ETS transcription factors etv5a and etv4 during zebrafish development. Dev Biol. 411 (2), 231-245 (2016).
- Marra, A. N., Li, Y., Wingert, R. A. Antennas of organ morphogenesis: the roles of cilia in vertebrate kidney development. Genesis. 54 (9), 457-469 (2016).
- Duffy, J. L., Suzuki, Y. Ciliated human renal proximal tubular cells. Observations in three cases of hypercalcemia. Am J Pathol. 53, 609-616 (1968).
- Katz, S. M., Morgan, J. J. Cilia in the human kidney. Ultrastruct Pathol. 6, 285-294 (1984).
- Hassan, M. O., Subramanyan, S. Ciliated renal tubular cells in crescentic glomerulonephritis. Ultrastruct Pathol. 19, 201-203 (1995).
- Ong, A. C., Wagner, B. Detection of proximal tubular motile cilia in a patient with renal sarcoidosis associated with hypercalcemia. Am J Kidney Dis. 45, 1096-1099 (2005).
- Worthington, W. C., Cathcart, R. S. III Ependymal cilia: distribution and activity in the adult human brain. Science. 139, 221-222 (1963).
- Cowan, M. J., Galdwin, M. T., Shelhamer, J. H. Disorders of ciliary motility. Am J Med Sci. 321, 3-10 (2001).
- Jain, R., et al. Temporal relationship between primary and motile ciliogenesis in airway epithelial cells. Am J Respir Cell Mol Biol. 43, 731-739 (2010).
- Stubbs, J. L., Vladar, E. K., Axlerod, J. D., Kitner, C. Multicilin promotes centriole assembly and ciliogenesis during multiciliate cell differentiation. Nat Cell Biol. 14, 140-147 (2012).
- Tan, F. E., et al. Myb promotes centriole amplification and later steps of the multiciliogenesis program. Development. 140, 4277-4286 (2013).
- Zhou, F., Narasimhan, V., Shboul, M., Chong, Y. L., Reversade, B., Roy, S. Gmnc is a master regulator of the multiciliated cell differentiation program. Curr Biol. 25, 3267-3273 (2015).
- Jowett, T. Analysis of Protein and gene expression. Methods Cell Biol. 59, 63-85 (1999).
- Schulte-Merker, S. Chapter 2: Looking at Embryos. Zebrafish: A practical approach. 261, 39-58 (2002).
- Brend, T., Holley, S. A. Zebrafish whole mount high-resolution double fluorescent in situ hybridization. J Vis Exp. (25), (2009).
- Lauter, G., Soll, I., Hauptmann, G. Two-color fluorescent in situ hybridization in the embryonic zebrafish brain using differential detection systems. BMC Dev Biol. 11, (2011).
- Drummond, B. E., Li, Y., Marra, A. N., Cheng, C. N., Wingert, R. A. The tbx2a/b transcription factors direct pronephros segmentation and corpuscle of Stannius formation in zebrafish. Dev Biol. 421 (1), 52-66 (2017).
- Jaffe, K. M., Thiberge, S. Y., Bisher, M. E., Burdine, R. D. Imaging cilia in zebrafish. Methods Cell Biol. 97, 415-435 (2010).
- Lauter, G., Soll, I., Hauptmann, G. Multicolor fluorescent in situ hybridization to define abutting and overlapping gene expression in the embryonic zebrafish brain. Neural Dev. 6, (2011).
- Sorrells, S., Toruno, C., Stewart, R. A., Jette, C. Analysis of apoptosis in zebrafish embryos by whole-mount immunofluorescence to detect activated caspase 3. J Vis Exp. (82), e51060(2013).
- Schumacher, J. A., Zhao, E. J., Kofron, M. J., Sumanas, S. Two-color fluorescent in situ hybridization using chromogenic substrates in zebrafish. Bio Techniques. 57, 254-256 (2014).
- Julich, D., et al. beamter/deltaC and the role of Notch ligands in the zebrafish somite segmentation, hindbrain neurogenesis and hypochord differentiation. Dev Biol. 286, 391-404 (2005).
- Gross-Thebing, T., Paksa, A., Raz, E. Simultaneous high-resolution detection of multiple transcripts combined with localization of proteins in whole-mount embryos. BMC Biology. 12, (2014).
- Cheng, C. N., Li, Y., Marra, A. N., Verdun, V., Wingert, R. A. Flat Mount Preparation for Observation and Analysis of Zebrafish Embryo Specimens Stained by Whole Mount In situ Hybridization. J Vis Exp. (89), e51604(2014).
- Poureetezadi, S. J., Donahue, E. K., Wingert, R. A. A Manual Small Molecule Screen Approaching High-throughput Using Zebrafish Embryos. J Vis Exp. (93), e52063(2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon