Method Article
Vivre l’imagerie suivie d’une cellule unique de suivi pour surveiller la biologie cellulaire et la Progression de la lignée de plusieurs Populations de neurones
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un protocole robuste à surveiller les populations neuronales en Time-lapse vidéo-microscopie suivie de post-traitement logicielle est décrite. Cette méthode représente un outil puissant pour identifier les événements biologiques dans une population sélectionnée au cours d’expériences d’imagerie live.
Résumé
Comprendre les mécanismes qui contrôlent les événements biologiques critiques des populations de cellules neurales, tels que la prolifération, la différenciation ou décisions de cellule de sort, seront crucial pour concevoir des stratégies thérapeutiques pour de nombreuses maladies affectant le système nerveux. Les méthodes actuelles de suivre les populations de cellules comptent sur leurs résultats finaux en images fixes et en général, ils ne parviennent pas à fournir une résolution temporelle suffisante pour identifier les caractéristiques comportementales dans des cellules individuelles. En outre, des variations dans la mort cellulaire, hétérogénéité comportementale au sein d’une population cellulaire, dilution, diffusion ou la faible efficacité des marqueurs utilisé pour analyser des cellules sont tous les handicaps importants qui mèneront à des afficheurs incomplètes ou incorrectes des résultats. À l’inverse, spectacle live d’imagerie et de la cellule unique de suivi dans des conditions appropriées représente un outil puissant pour surveiller chacun de ces événements. Un Time-lapse protocole vidéo-microscopie, suivi de post-traitement, est décrit ici, pour assurer le suivi des populations neuronales avec résolution unicellulaire, employant des logiciels spécifiques. Les méthodes décrites permettent aux chercheurs de répondre à des questions essentielles concernant la progression de biologie et de la lignée cellulaire des populations neuronales distinctes.
Introduction
Afin de développer des stratégies thérapeutiques nouvelles et plus efficaces pour régénérer les populations neuronales, nous devons d’abord comprendre les mécanismes de base qui maintiennent les cellules avec un potentiel de régénération neuronal. Poursuivant cet objectif exige une connaissance approfondie des facteurs qui régulent l’équilibre entre la quiescence, prolifération/différenciation, le mode et le moment de la durée du cycle cellulaire, capacités migratoires, viabilité, division, etc.. Même si c’est une approche technique qui a travaillé pendant de nombreuses années1, imagerie en direct et observation directe restent encore la meilleure option pour surveiller les événements énumérés ci-dessus. Contrairement à nombreuses autres approches centrées sur les afficheurs de point final, en direct d’imagerie et unicellulaire suivi de fournir des informations sur toute la longueur d’une expérience de2,3,4,5, 6. ainsi, l’ajout de résolution temporelle permet la mort cellulaire, comportement de cellules hétérogènes ou décisions destin cellulaire, ainsi que plusieurs autres événements critiques d’identifier qui pourraient autrement passer inaperçus. Idéalement, ces caractéristiques de cellules devraient mieux être surveillés à la cellule unique niveau in vivo, où les deux intrinsèque (cellule autonome) et extrinsèques cues (niche de la cellule) sont pris en compte.
Cependant, bien que dans in vitro situation se produisent dans un environnement qui ne reproduit pas le milieu naturel, les conditions de culture faible densité généralement utilisées dans ces protocoles sont plus adaptées révéler des caractéristiques intrinsèques de la cellules. Par ailleurs, un contrôle plus simpliste du milieu environnant, en modifiant simplement le milieu de croissance, peut constituer un outil précieux pour étudier le rôle individuel de chaque facteur extrinsèque qui définit la niche neurale, ainsi que les facteurs environnementaux qui peuvent être induit dans les scénarios pathologiques7,8,9,10,11,12,13. Par conséquent, lorsqu’il est correctement configuré, comme dans le protocole proposé ici, imagerie live fournit une solution réalisable en vitro pour répondre à la plupart des questions énumérées précédemment.
En bref, ce protocole décrit le matériel, de logiciels, les conditions de culture et les principales étapes nécessaires pour réaliser avec succès une expérience d’imagerie live suivie d’une cellule unique de suivi. Cette approche offre des renseignements précieux qui permettant de révéler des aspects fondamentaux de la biologie et de la progression de la lignée, de plusieurs populations de neurones.
Protocole
Les sections suivantes décrivent les étapes nécessaires pour son imagerie live suivie suivi de cellule unique de plusieurs populations de neurones (Figure 1). Toutes les procédures impliquant des animaux décrits dans le présent protocole doivent être effectués conformément aux directives du Conseil International pour Laboratory Animal Science (ICLAS).
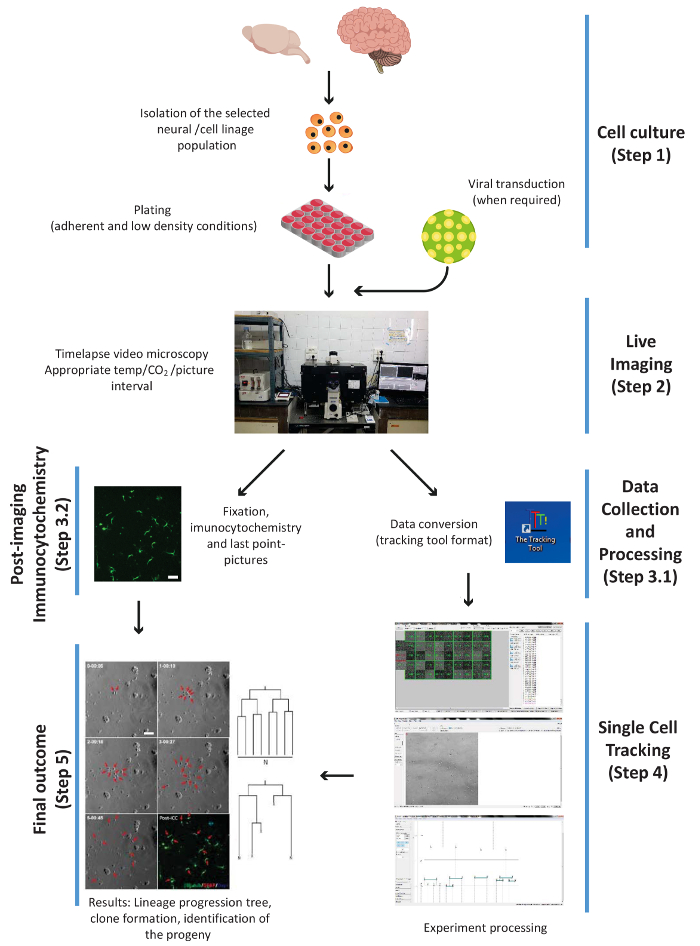
Figure 1. Schéma illustrant les principales étapes expérimentales de la procédure, c'est-à-dire: cell culture, imagerie live, PICC et collecte de données, seule cellule de suivi et le résultat final. Les étapes sont numérotées en fonction du flux de travail du protocole. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
1. Culture cellulaire : Isolation et plaquage de sélectionné Population neurale ou une lignée de cellules
NOTE : En conjonction avec ce protocole, des exemples de son application à des populations cellulaires distincts sont donnés pour valider son utilité pour analyser la biologie de cellules nerveuses. Ceux-ci incluent : les cellules souches neurales adultes (aNSCs) dérivé de la souris subépendymaire Zone (ZES) (pour un isolement détaillé protocole voir14) ; Astrocytes corticaux postnatales pour étudier une reprogrammation neuronale (pour une isolation détaillé protocole voir15) ; Astrocytes cérébelleux postnatals (pour une isolation détaillée méthode Voir16) ; et ligne de cellules de neuroblastome souris Neuro-2 a (N2a).
- Les semences les cellules directement sur les plaques 24 puits enduits de poly-D-lysine. Utiliser 1 mL de milieu de culture par puits. Incuber les plaques à 37 ° C et 8 % CO2 pendant aNSCs, ou à 37 ° C et 5 % de CO2 pour la lignée d’astrocytes/cellulaire pendant 2 h avant l’imagerie live. Évitez d’utiliser des lamelles couvre-objet pour empêcher tout déplacement intempestif comme la platine du microscope motorisé ne soit déplacée qui en fait la seule cellule suivi infaisable.
Remarque : La densité des cellules et des milieux de culture utilisés dans les expériences sont : 30-40 000 cellules/puits pour aNSCs en Dulbecco de l’aigle modifié (moyenne de DMEM:F12 mélange de nutriments) ; 20 000 cellules/puits pour les cellules dans un milieu DMEM hyperglycémie N2a, 80 000 cellules/puits pour les astrocytes cérébelleux dans un milieu DMEM hyperglycémie ; et les 55-65 000 cellules/puits pour les astrocytes postnatals dans un milieu DMEM:F12 mélange de nutriments. - Normaliser le protocole de culture en ajustant la densité cellulaire de la culture pour le plus petit nombre de cellules possibles. Néanmoins, la densité cellulaire doit être suffisamment élevée pour maintenir la viabilité de la culture.
Remarque : Si la densité cellulaire est trop élevée, l’excès de débris ou de pauvre dissociation (touffes) susceptibles de gêner suivi des cellules individuelles.
2. vivre d’imagerie en Time-lapse vidéo-microscopie
- Allumer les systèmes microscope, caméra, matériel et l’incubation. Régler la température et la pression à 37 ° C et 8 % de CO2 pour aNSCs, ou à 37 ° C et 5 % de CO2 pour la lignée astrocytes/cellulaire. Laisser la température et les niveaux de CO2 se stabiliser pendant 1 à 2 h.
Remarque : Matériel spécifique est nécessaire pour effectuer une analyse vidéo Time-lapse, y compris : microscopes contraste/fluorescence champ lumineux et en phase avec les composants motorisés ; dispositifs d’incubation qui contrôlent la température, de CO2 et de l’humidité. et enfin, fiable et suffisamment puissant matériel et logiciels capables d’acquérir et de traiter le volume de photos obtient lors des expériences d’imagerie direct (s’il vous plaît vérifier la Table des matières). - Une fois que les cellules soient solidement fixés à la plaque (2 h après le placage), utilisez un stylo marqueur permanent pour faire une petite marque sur le fond d’un puits qui ne servira pas pour le suivi, c'est-à-dire un bien qui ne contient-elle pas de cellules.
Remarque : Cette marque servira comme référence à zéro les coordonnées xyz, et il peut être utilisé à tout moment pendant ou après l’expérience ou entre les changements de milieu, pour revenir à la position zéro. - Placer la plaque à l’intérieur de la chambre d’incubation du microscope et fixez solidement la plaque sur la scène pour éviter tout mouvement intempestif pendant le déplacement de la platine motorisée du microscope.
- Laisser la température du milieu de culture cellulaire s’équilibrer dans la chambre pendant environ 20 min. Cette étape permettra d’éviter une perte de focus lors de l’enregistrement en raison de la dilatation des composants.
- Lancez le logiciel de création d’images en direct et sélectionnez le module Time-lapse pour entamer l’expérience.
- Définissez la durée totale de l’expérience et les cycles d’acquisition image dans le menu « onglet Calendrier ». En raison de la phototoxicité inhérente de la transmission ou la lumière de fluorescence utilisée, définir un intervalle suffisant à l’équilibre entre la résolution temporelle de l’analyse et la mort cellulaire potentiels.
NOTE : par exemple, un total de 120 h a été sélectionné pour les cultures aNSC, acquérir des images de fond clair chaque 5 min. prendre en considération que l’acquisition de 120 h d’un seul film dans cette configuration nécessite 120-150 gigaoctets d’espace de stockage gratuit dans le périphérique de l’ordinateur. - Sélectionner les positions d’image définies par x et y coordonnées et la distance focale (la coordonnée z) dans le « menu points xyz tab ». Inclure le point de référence (coordonnées xyz zéro) comme la position initiale afin de récupérer les coordonnées à tout moment.
- Sélectionnez le type d’acquisition dans la « longueur d’onde onglet menu de sélection », fond clair, seuls ou en combinaison avec épifluorescence excitation lorsque requis. Sélectionnez la durée d’exposition. Gardez à l’esprit que la surexposition aux transmis et éclairage fluorescent en particulier, peut compromettre la viabilité de cellules (comme indiqué ci-dessus).
- Pour aNSCs, cérébelleuses astrocytes et les cellules N2a, sélectionnez fond clair (temps d’exposition de 10 à 50 ms).
- Pour les astrocytes corticaux transduits sélectionnez fond clair (temps d’exposition de 10 à 50 ms) en combinaison avec fluorescence rouge/vert, selon le journaliste utilisé pour l’expérience (longueur d’onde rouge d’excitation : temps d’exposition 550 nm et 400 ms ; excitation verte longueur d’onde : 460-500 nm et durée d’exposition de 100 ms).
- Définir le nom de l’expérience et le dossier où seront stockées les images. Enregistrer la liste des postes pour recharger l’expérience à tout moment et une fois que toutes les conditions ont été fixées, exécuter l’expérience en cliquant sur le bouton « Exécuter maintenant ».
- L’expérience de mettre en pause et réajuster les conditions de mise au point en cliquant sur le bouton z « écraser » une fois par jour jusqu'à ce que l’expérience est terminée. Si des changements dans le milieu sont nécessaires durant l’imagerie en direct, mettre en pause l’expérience et récupérer la plaque de la chambre de laps de temps.
Remarque : Les modifications du pH du milieu en raison de la mort cellulaire ou prolifération excessive, ainsi que les variations de température de la pièce, susceptibles d’affecter la mise au point correcte du microscope sur les cellules. Pour les cultures sensibles (par exemple, aNSCs), nous recommandons l’utilisation du milieu supplémenté avec 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acide (HEPES) (concentration finale : 1 mM).
3. l’imagerie immunocytochimie (PICC), la collecte de données et traitement
- Une fois l’expérience est terminée, mettre en pause le logiciel et récupérer la plaque de fixation et PICC, tel que décrit dans les étapes suivantes.
- Effectuer la fixation cellulaire : laver les cellules une fois avec une solution saline tamponnée au phosphate de 1 mL (PBS) et ajouter 500 µL de paraformaldéhyde (PFA) (4 % dans du PBS), incuber 10 min à température ambiante (RT).
ATTENTION : Paraformaldéhyde est un fixateur fort et doit être manipulé avec soin pour éviter le contact avec la peau ou les yeux. Il doit être manipulé qu’à l’intérieur d’une hotte aspirante. - Laver les cellules trois fois avec 1 mL de PBS et ajouter 500 µL de la solution de saturation (PBS contenant 2 % (wt/vol) de l’albumine sérique bovine (BSA) et de 0,2 % (vol/vol) d’un agent tensioactif non ionique). Incuber 1 h à température ambiante.
- Enlever la solution de blocage et ajouter 250-400 µL de la solution d’anticorps primaires. Incuber pendant 2 heures à température ambiante. La solution d’anticorps primaires contient des anticorps primaires dilués dans du PBS contenant 2 % (wt/vol) de BSA et de 0,2 % (vol/vol) d’un agent tensioactif non ionique. Les anticorps utilisés dans les expériences décrites ici : GFAP (1/500), βIII-tubuline (1:1, 000) et α-tubuline (1:1, 000). Comme cela se fait directement dans le puits, les plus grands volumes des solutions sont nécessaires (250-400 µL) pour couvrir toutes les cellules.
- Laver trois fois avec 1 mL de PBS et ajouter 250-400 µL de la solution d’anticorps secondaires (diluée comme indiqué au point 3.4). Anticorps secondaires utilisés dans les expériences décrites ici : anti-souris fluorescéine (FITC) (1:800), anti-lapin Cy3 (1/500). Incuber 1 h à RT dans l’obscurité.
- Laver trois fois dans 1 mL de PBS. Garder les cellules dans 1 mL de PBS pour les étapes ultérieures du protocole.
- Replacez la plaque sur la platine du microscope et l’attacher fermement sur scène pour éviter les mouvement intempestif pendant le déplacement de la platine du microscope motorisé.
- Récupérer le xyz position zéro à l’aide de la marque effectuée à l’étape 2.2 et re-définir les positions à ce point de référence en appuyant sur le bouton « Compenser tous les X, Y, Z ». Réglez la distance focale pour chaque poste.
- Acquérir une dernière série d’images, configurer les conditions nécessaires à l’émission de fluorescence dans le menu « longueur d’onde sélection onglet » afin de détecter les antigènes ciblés auparavant dans la PICC.
- En bref, en plus du fond clair, activer FITC (Excitation : 495 nm) et Cy3 (Excitation : 550 nm) options d’acquisition du logiciel. 10-50 ms pour l’exposition de 400 ms fond clair permet de détecter les fluorophores et appuyez sur la touche « 1 boucle temporelle », en vue d’acquérir une dernière série de photos.
Remarque : L’intensité de la fluorescence peut différer en fonction des résultats PICC. Régler le temps d’exposition afin d’obtenir la qualité d’image optimale.
- En bref, en plus du fond clair, activer FITC (Excitation : 495 nm) et Cy3 (Excitation : 550 nm) options d’acquisition du logiciel. 10-50 ms pour l’exposition de 400 ms fond clair permet de détecter les fluorophores et appuyez sur la touche « 1 boucle temporelle », en vue d’acquérir une dernière série de photos.
- Sélectionnez l’option fichier/exporter du logiciel et d’exporter les photos Tagged Image File Format (Tiff) ou format Joint Photographic Experts Group (Jpeg) dans un dossier de destination prédéfini.
- Convertir les images exportées au format requis par le logiciel de suivi : l’outil de suivi17 (tTt). Pour ce faire, définir l’entrée et sortie de dossier dans le « outil de conversion de tTt » fenêtre, ainsi que les marqueurs utilisés pour les postes (xy) et canaux (c)-points dans le temps (t) et appuyez sur le bouton « convertir des images ».
Remarque : Les images doivent être renommées selon les réglages spécifiques et ils doivent être stockés dans des dossiers individuels pour chaque poste utilisé dans l’expérience. Instructions pour l’installation, exigences, renommage des postes/images et l’utilisation de l’outil de suivi sont disponibles en téléchargement sur : https://www.bsse.ethz.ch/csd/software/tTt-and-qtfy.html.
4. cellule suivi
- Après avoir renommé les données, exécutez le logiciel de tTt. Sélectionnez un dossier de travail utilisateur nom et tTt.
Remarque : Le tracking tool travail dossier contiendra toutes les données analysées et les résultats exportés. Le dossier de travail doit être nommé tTtexport, contenant des sous-dossiers nommés « AVIexport », « Configs », « TreeExport » et « tTtfiles ». - Choisir l’expérience d’être chargé dans la « fenêtre de dossier sélectionnez experiment », indiquant le chemin du dossier où l’expérience est stockée, et puis cliquez sur le bouton « Charger l’expérience ».
- Exécuter le convertisseur de fichier journal pour transformer les images chargées dans un format qui peut être lu par le logiciel de suivi (pour les expériences chargées pour la première fois, cela sera demandé automatiquement par le logiciel).
- Sélectionnez un poste pour le suivi en cliquant sur son icône (après que conversion, un aperçu des positions enregistrées au cours des expériences s’affichera dans la fenêtre « position du schéma »). Chaque poste sera représentée par un symbole composé dans une photo de la poste et son numéro correspondant (voir Figure 1).
- Une fois que la position a été sélectionnée et une liste des images disponibles est affichée sur la droite de la fenêtre « position du schéma », sélectionnez-les et cliquez sur le bouton « Charger les images ».
- Une fois que le chargement est terminé et la « fenêtre de l’éditeur de cellule » s’affiche, sélectionner les longueurs d’onde et l’intervalle d’image à suivre dans la « fenêtre d’éditeur de cellule ». Longueur d’onde 0 correspond à fond clair, 1 au FITC et 2 à Cy3 et 3 au DAPI. Dans les expériences décrites ici, intervalle de 1 a été utilisée, c.-à-d., toutes les images chargées. Pour clarifier, intervalle 2 signifie chargement de chaque seconde de l’image.
- Une fois que les images ont été chargées, revenir à la fenêtre « position du schéma » et double-cliquez sur l’icône qui représente la position précédemment chargée. La fenêtre « film » apparaîtra qui permet le suivi de la cellule unique à exécuter.
- Suivant les instructions de l’outil de suivi, effectuer le suivi. Sélectionnez le canal 0 (correspondant à fond clair) et ajuster la luminosité et le contraste (« gamma bouton d’ajustement »). Démarrez le suivi en appuyant sur la touche F2.
Remarque : Lors du suivi, la cellule de suivi sera suivie en plaçant le pointeur de la souris dessus et en appuyant sur la touche « 0 ». La division cellulaire, l’apoptose des cellules et la cellule perdu boutons sont disponibles pour surveiller ces événements spécifiques de cellules.
5. issue
- Une fois que la cellule de suivi a été achevée et la progéniture identifiée, sauver l’expérience (onglet Éditeur de cellule fenêtre/fichier / enregistrer l’arborescence actuelle comme) et continuer à exporter les résultats.
- Exporter les arbres de la lignée et les données des cellules dans le menu « Export », situé dans la « fenêtre d’éditeur de cellule ». De même, exporter les images de la cellule et les films via le menu « Export » accessible par le biais de la fenêtre « film ». Les images, les arbres de la lignée, données et films seront exportés dans le dossier de travail de tTt.
Résultats
La méthode décrite permet aux questions cruciales concernant la biologie de cellules de plusieurs populations de neurones doivent être résolus. Par exemple, il a été possible de surveiller la progression de la lignée neurogène et oligodendrogliogenic d’aNSCs7,8,14,18. Par le suivi unique aNSCs et leur progéniture (Figure 2 a, B), il a été possible de démontrer qu’aNSCs isolées in vitro conservent leur nature neurogène, produisant principalement des neuroblastes, et qu’ils suivent une séquence proposée dans vivo19 mais n’a pas été démontrée au niveau de la cellule unique. En outre, ce système de culture a permis des divisions cellulaires asymétriques à visualiser pour la première fois dans la lignée d’aNSC de la ZES (Figure 2 b), offrant un modèle unique pour étudier la NSC autorenouvellement8,14. De même et quelle que soit la lignée analysés, il était possible d’obtenir des données précieuses au sujet de la croissance des cellules, des séries de division, la viabilité des cellules ou longueur du cycle cellulaire.
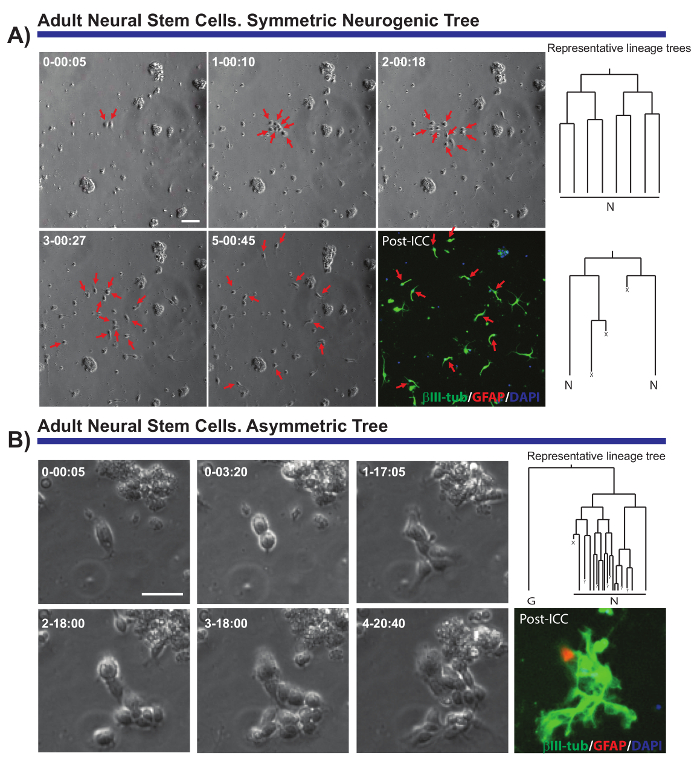
Figure 2. Exemple d’aNSCs isolées de la ZES et analysée par imagerie direct et unique cellule de suivi. Images de contraste de phase dépeignent la progression du clone à différents moments (jour-h: min). L’image finale et correspond à l’immunocytochimie imagerie post (PICC) protéine fibrillaire acide gliale (GFAP, rouge), βIII-tubuline (vert) 4', 6-diamidino-2-phénylindole (DAPI, bleu). (A) analyse des arbres neurogène symétrique grâce à différentes séries amplifier les divisions pour générer des neuroblastes post-mitotiques. Flèches rouges indiquent les cellules incluses dans les arbres symétriques. Sur la droite, les arbres de la lignée correspondant aux clones et générés par le logiciel de tTt sont affichés. (B) exemple d’un progéniteur générant un arbre neurogène asymétrique, avec une branche subissant des divisions amplification pour produire des neuroblastes tandis que l’autre donne lieu à quiescentes cellules positives GFAP via un événement autorenouvellement potentiel. Sur la droite, s’affiche l’arborescence de la lignée généré par le logiciel de tTt. Dans tous les arbres de la lignée : « N » représente des neuroblastes post-mitotiques ; « G », les cellules quiescentes GFAP-positive ; « X », la mort cellulaire ; et « ? » une cellule perdue. Barre d’échelle représente 50 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Imagerie en direct et seule cellule suivi analyse également fournit un affichage précis des capacités migratoires d’une population de neurones. Ces renseignements proviennent de postnatals astrocytes cérébelleux, soumis à une plaie zéro dosage20, générant des informations concernant la distance moyenne parcourue par les astrocytes, lors de la fermeture de la plaie (Figure 3). En outre, il était possible de voir que certains d'entre les astrocytes divisée pendant le processus de guérison, alors que d’autres restent inchangés tout au long de l’expérience. Étonnamment, ceux qui divisait semblent des comportements migratoires plus prolifique que leurs homologues non Division (voyageant deux fois comme beaucoup en moyenne). Ce phénomène suggère une hétérogénéité très intéressante dans la capacité d’astrocytes de former une cicatrice sur la blessure, ce qui serait ont été dilué dans la lecture d’une expérience d’analyse classique point final.
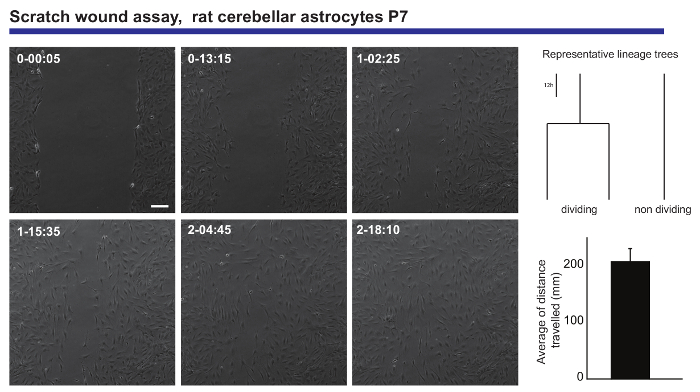
Figure 3. Analyse du comportement migratoire des postnatals astrocytes cérébelleux dans une égratignure plaie dosage. Images de contraste de phase dépeignent la plaie à différents moments (jour-h: min). Arbres de lignage, générés par le logiciel de tTt, illustrent le comportement représentatif, en termes de division cellulaire, des astrocytes en refermant la plaie. Histogramme montre la distance moyenne parcourue par les astrocytes analysées par la cellule de suivi (moyenne ± S.E.M.). Barre d’échelle représente 50 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Une autre caractéristique intéressante des expériences vidéo-microscopie Time-lapse est la capacité à comparer la prolifération et la différenciation dans une population de cellules. Nous avons testé les cellules N2a plaqués dans des conditions qui favorisent la prolifération (en présence de 10 % sérum bovin fœtal (SVF)) ou différenciation (en présence de 0,5 % BF + acide arachidonique 10 µM). Il était possible de suivre la progression de la lignée de ces cellules dans des conditions prolifératives (Figure 4 a), tandis que la différenciation des cellules ne prolifèrent pas et elles forment des neurites (Figure 4 b). Remarquablement, seule cellule de suivi a permis de colonies avec différents prolifération capacités à distinguer et élongation des neurites (et rétraction) à évaluer, fournir des données précises et quantitatives qui peuvent ensuite être exportées.
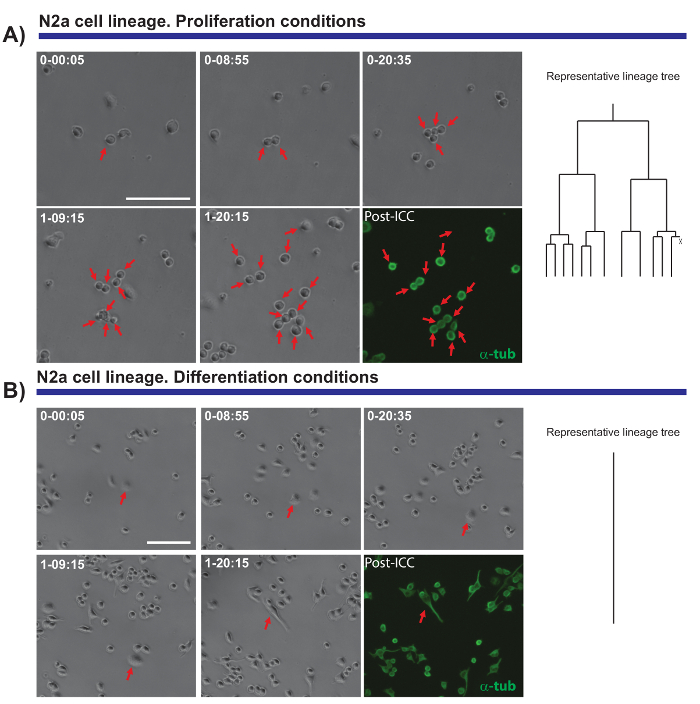
Figure 4. Le suivi de la biologie cellulaire N2a prolifération (A) ou les conditions de la différenciation (B). Montrant la progression du clone à différents moments (jour-h: min) des images à contraste de phase. L’image finale correspond à l’immunocytochimie imagerie post (PICC) pour α-tubuline (vert). (A) seule cellule suivi permet des séries de division à surveiller, ainsi que l’hétérogénéité dans la réponse proliférative des cellules différentes. Sur la droite, l’arborescence de la lignée généré par le logiciel de tTt illustre le comportement prolifératif des cellules N2a. (B) des cellules dans des conditions de la différenciation quitter le cycle cellulaire et génèrent des neurites, un processus qui peut être effectivement mesuré par imagerie après analyse. Unique, suivi de la cellule, représentée par l’arborescence de la lignée à droite, illustre comment les cellules N2a sortie cycle cellulaire et arrêter la division cellulaire dans des conditions de la différenciation. Barre d’échelle représente 50 µm.S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Vivre enfin, imagerie et cellule de suivi est extrêmement utile pour surveiller les changements morphologiques et moléculaires lorsque les cellules sont soumises à la reprogrammation. Imagerie Live des astrocytes postnatals transduites avec homologue Achaete-scute 1 (Ascl1) fournit des données précieuses concernant les changements morphologiques qui se produisent au cours de reprogrammation ou le blocage de la division cellulaire lorsque des astrocytes sont reprogrammé (voir Figure 5). En outre, Ascl1 transduction est associé à la transduction d’un construct codant pour la protéine de fluorescence verte (GFP) sous le contrôle du promoteur Double Cortin (DCX), il est possible de définir le point de temps précis lorsque neuronale des marqueurs spécifiques commencent à être exprimée dans les cellules reprogrammés (Figure 5). Time-lapse vidéo-microscopie permet également le nombre de cellules qui complètent avec succès la reprogrammation pour être quantifié et comparées aux cellules qui meurent au cours de ce processus. Surveillance des événements conduit à l’identification des critiques « checkpoints » dans les cellules qui ont été avec succès reprogrammé9.
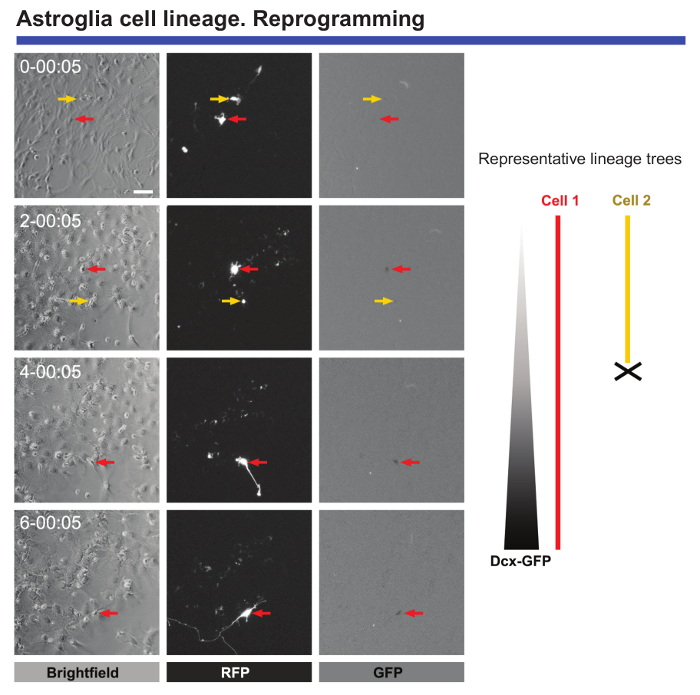
Figure 5. Analyse des astrocytes corticaux postnatals, soumis à une reprogrammation neuronale. La reprogrammation a été induite par transduction avec des vecteurs de protéine (DP) de Fluorescence rouge-Ascl1 pro-neurogène. Conversion neuronale a été suivie par transduction Co avec un vecteur codant la GFP sous le contrôle d’un promoteur DCX. Images de contraste de phase montrent la progression de reprogrammation à différents moments (jour-h: min). Images de fluorescence d’expression DP et GFP, respectivement. Imagerie en direct et unique cellule de suivi a permis des événements cruciaux à suivre, comme les changements morphologiques, l’absence de division cellulaire lors de la reprogrammation, la mort cellulaire, et l’heure exacte lorsque reprogrammé les cellules commencent à exprimer les marqueurs neurones peut être définis . Barre d’échelle représente 80 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Une des valeurs plus importantes de l’imagerie live est la possibilité d’effectuer le traçage précis de lignée, élucider les aspects cruciaux de la progression de la lignée dans une population de neurones. Traçage de la lignée est défini comme l’identification et surveillance de tous les descendants d’un ancêtre unique, du fondateur du clone pour le clone subséquent formé21. Remarquablement, les méthodes alternatives de lignée tracing (p. ex., transduction virale ou journaliste multicolor constructions21) présentent un inconvénient critique, auquel cas le résultat final est basé sur des images fixes et il ne le fait pas nécessairement constituent la séquence entière. Cela signifie que la mort cellulaire, hétérogénéité dans le comportement de la population cellulaire, dilution, épandage ou mauvaise efficacité des marqueurs, ainsi que d’autres handicaps importants, conduire à des afficheurs incomplètes ou incorrectes des résultats2. En outre, imagerie live permet au chercheur d’analyser les caractéristiques importantes de la biologie des populations de neurones, tels que la mode et le moment de la division cellulaire, la croissance cellulaire, migration, prolifération et différenciation, longueur du cycle cellulaire, neurite formation, de complexité et de longueur, cellule sort sélection (différenciation), ou la conversion (reprogrammation).
En outre, l’imagerie live peut être facilement complété par toute autre analyse destinée à obtenir des données de cellules uniques tels que, par exemple, le séquençage de RNA. Toutefois, pour réaliser des prestations combinées de l’imagerie live et d’autres techniques exige que ces cellules précédemment contrôlés dans les films sont plus tard réidentifiés et individuellement prélevés pour l’analyse secondaire. Ceci peut être réalisé à l’aide de microscopes qui incluent les coordonnées positionnelles, en appliquant des reporters fluorescentes pour des cellules spécifiques ou analyse de la répartition des groupes de cellules comme références. En effet, la combinaison du profil transcriptome et le comportement des cellules individuelles peuvent représenter une voie puissante pour élucider les nouveaux signaux moléculaires impliqués dans la biologie des cellules.
L’un des principaux problèmes qui peuvent compromettre une expérience d’imagerie live est une densité de culture cellulaire insuffisante. Comme indiqué précédemment, à haute densité l’excès de débris ou de dissociation pauvre (formation de touffe) peuvent affecter la qualité et la résolution spatiale des images, faisant seule cellule suivi infaisable. Par conséquent, les conditions des populations cellulaires distincts à l’étude devraient être positionnées sur le plus petit nombre de cellules possibles sans compromettre la viabilité de la culture cellulaire.
La fréquence d’acquisition d’images est également cruciale et doit être ajustée avec soin, surtout lorsque l’éclairage de fluorescence est utilisé. Une surexposition aux transmis et surtout lumière de fluorescence peut compromettre la viabilité des cellules. Par ailleurs, une lenteur excessive entre la capture des images peut interférer avec la résolution temporelle de l’analyse.
Une autre étape cruciale lors de l’expérience d’imagerie live est l’ajustement périodique de mise au point. Échec dans le bon réglage/re-setting de la distance focale d’entraver suivi de cellule unique. En outre, il est nécessaire de bien vérifier que la chambre d’incubation conserve la température adéquate, l’humidité et niveaux de CO2 , modifiant des variations indésirables que peuvent induire la mort cellulaire.
Enfin, une fois que le PICC a été effectuée, il est important de récupérer correctement la position xyz zéro avant la dernière série d’acquisition d’images. Mauvais re-réglage de le xyz position zéro rendra difficile de faire correspondre les images à contraste de phase et d’immunofluorescence, empêchant l’identification de la descendance de la cellule.
Bien que cette approche présente de multiples aspects positifs, certaines limitations à l’imagerie direct des populations neuronales persistent. Par exemple, la densité cellulaire faible requise pour effectuer le suivi des succès monocellulaire d’aNSCs rend impossible d’employer des épreuves biochimiques, tels que Western blotting14. En outre, surveillance divisant rapidement les populations comme les astrocytes cérébelleux ou cellules N2a est temporellement limité comme c’est souvent trop difficile de suivre les cellules comme les cultures près de confluence. En outre, plusieurs méthodes de culture, ainsi que les restrictions biologiques inhérentes liées à l’isolement de cellules, souvent compromettent la viabilité des cellules sur de longues périodes, limitant la durée des expériences d’imagerie live. Enfin, isoler les cellules de leur environnement naturel a des effets positifs et négatifs. Cellules isolées de leur niche physiologique peuvent ne pas recevoir les signaux importants qui modulent leur comportement, tandis que dans le même temps, il représente un moyen puissant pour tester l’effet de ces signaux individuellement dans la progression de la lignée des neurones spécifiques populations.
Étant donné les limitations décrites ci-dessus, il est clair que le parfait scénario méthodologique serait à réaliser direct d’imagerie et unique cellule suivi expériences sous des conditions physiologiques normales in vivo. Cependant, les techniques actuelles sont incapables de suivre les cellules individuelles pendant de longues périodes de temps dans les régions profondes du cerveau2. Par conséquent, l’avenir de l’imagerie live devrait se concentrer sur surmonter cette limitation, visant à analyser entièrement la biologie cellulaire des cellules individuelles en vivo avec le plus petit possible interférences de l' environnement physiologique3.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Beatriz Gascon pour son travail d’assistance et de l’art à la Figure 1. Nous remercions également m. C. Norris pour son aide. Le travail présenté ici a été appuyé par les subventions de recherche, « Red de excelencia Consolider-Ingenio espagnol Ion Channel Initiative » (BFU2015-70067REDC), MEC (BFU2014-53654-P), BRADE-CM (S2013/ICE-2958), Université Complutense de Madrid-Santander (PR26/16-18 b-3) et Fundación Ramon Programme de Areces Grant (PR2018/16-02). Felipe Ortega reconnaît le Ramon y Cajal programme du ministère espagnol de l’Economie et compétitivité (MEC : RYC-2013-13290).
matériels
| Name | Company | Catalog Number | Comments |
| Poly-D-lysine | Sigma | P0899 | Working solution 0.02 mg mL-1 |
| 24 wells plate | Falcon | 352047 | |
| Dulbecco’s modified Eagle’s medium (DMEM): F12 Nutrient Mixture medium (-L Glutamine) | Invitrogen | 21331-020 | |
| DMEM High Glucose medium | Sigma | D6546 | |
| Bovine Serum Albumin | Sigma | A6003 | |
| Triton X-100 | Merck | 11869 | non-ionic surfactant |
| Mouse anti-β III Tubulin | Sigma | T8660 | |
| Rabbit anti-GFAP | DakoCytomation | Z0334 | |
| Mouse anti-α Tubulin | Sigma | T5168 | |
| Anti-Mouse FITC | Jackson Laboratories | 715-095-150 | |
| Anti-Rabbit Cy3 | Jackson Laboratories | 711-165-152 | |
| Brightfield/Phase contrast/fluoresence microscope | Nikon | TE-2000-E | |
| CFI PLAN FLUOR DLLL 10X objetives | Nikon | Ref 280MRH10101 | |
| CFI SUPER PLAN FLUOR ELWD AMD 20X objetives | Nikon | Ref 280MRH48230 | |
| pE-300 LED fluorescence | Cool LED | Ref Number 1981 | |
| 310M-201 Incubation system (temperature) | OKO-Lab | Serial Nº VOF007307 | |
| Pro-ScanII Motorized stage system | Prior | Serial Nº 60018 | |
| High precision microscope camera version 4.2 | ANDOR Zyla | VSC-03650 | |
| Specifc software for live imaging with timelapse module: NIS-Elements AR4.5 | Nikon | NIS-Elements AR4.5 -Hasp ID: 13CE819E | |
| OKO touch Incubation system (CO2) | OKO-lab | Serial number 1716 | |
| Murine Neuro-2a Neuroblastoma Cell line | ATCC | ATCCCCL131 | |
| HEPES buffer solution 1 M | Invitrogen | 15630-056 |
Références
- Conklin, E. G. The Mutation Theory From the Standpoint of Cytology. Science. 21 (536), 525-529 (1905).
- Ortega, F., Costa, M. R. Live Imaging of Adult Neural Stem Cells in Rodents. Front Neurosci. 10, 78 (2016).
- Skylaki, S., Hilsenbeck, O., Schroeder, T. Challenges in long-term imaging and quantification of single-cell dynamics. Nat Biotechnol. 34 (11), 1137-1144 (2016).
- Schroeder, T. Tracking hematopoiesis at the single cell level. Ann N Y Acad Sci. 1044, 201-209 (2005).
- Schroeder, T. Imaging stem-cell-driven regeneration in mammals. Nature. 453 (7193), 345-351 (2008).
- Etzrodt, M., Endele, M., Schroeder, T. Quantitative single-cell approaches to stem cell research. Cell Stem Cell. 15 (5), 546-558 (2014).
- Ortega, F., et al. Oligodendrogliogenic and neurogenic adult subependymal zone neural stem cells constitute distinct lineages and exhibit differential responsiveness to Wnt signalling. Nat Cell Biol. 15 (6), 602-613 (2013).
- Costa, M. R., et al. Continuous live imaging of adult neural stem cell division and lineage progression in vitro. Development. 138 (6), 1057-1068 (2011).
- Gascon, S., et al. Identification and Successful Negotiation of a Metabolic Checkpoint in Direct Neuronal Reprogramming. Cell Stem Cell. 18 (3), 396-409 (2016).
- Karow, M., et al. Reprogramming of pericyte-derived cells of the adult human brain into induced neuronal cells. Cell Stem Cell. 11 (4), 471-476 (2012).
- Kleiderman, S., et al. Conversion of Nonproliferating Astrocytes into Neurogenic Neural Stem Cells: Control by FGF2 and Interferon-gamma. Stem Cells. 34 (12), 2861-2874 (2016).
- Bunk, E. C., et al. Prox1 Is Required for Oligodendrocyte Cell Identity in Adult Neural Stem Cells of the Subventricular Zone. Stem Cells. 34 (8), 2115-2129 (2016).
- Aravantinou-Fatorou, K., et al. CEND1 and NEUROGENIN2 Reprogram Mouse Astrocytes and Embryonic Fibroblasts to Induced Neural Precursors and Differentiated Neurons. Stem Cell Reports. 5 (3), 405-418 (2015).
- Ortega, F., et al. Using an adherent cell culture of the mouse subependymal zone to study the behavior of adult neural stem cells on a single-cell level. Nat Protoc. 6 (12), 1847-1859 (2011).
- Heinrich, C., et al. Generation of subtype-specific neurons from postnatal astroglia of the mouse cerebral cortex. Nat Protoc. 6 (2), 214-228 (2011).
- Jimenez, A. I., et al. Potentiation of ATP calcium responses by A2B receptor stimulation and other signals coupled to Gs proteins in type-1 cerebellar astrocytes. Glia. 26 (2), 119-128 (1999).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotechnol. 34 (7), 703-706 (2016).
- Ortega, F., Berninger, B., Costa, M. R. Primary culture and live imaging of adult neural stem cells and their progeny. Methods Mol Biol. 1052, 1-11 (2013).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annu Rev Neurosci. 32, 149-184 (2009).
- Yu, A. C., Lee, Y. L., Eng, L. F. Astrogliosis in culture: I. The model and the effect of antisense oligonucleotides on glial fibrillary acidic protein synthesis. J Neurosci Res. 34 (3), 295-303 (1993).
- Kretzschmar, K., Watt, F. M. Lineage tracing. Cell. 148 (1-2), 33-45 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon