Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Haut débit séquençage parallèle à la mesure de remise en forme de Leptospira interrogans Transposon Insertion Mutants durant Golden syrien Hamster Infection
Dans cet article
Résumé
Nous décrivons ici une technique qui associe une mutagenèse par transposon séquençage haut-débit pour identifier et quantifier des mutants de leptospires transposon dans les tissus après une contestation des hamsters. Ce protocole peut être utilisé à des mutants de l’écran de survie et de la diffusion chez les animaux et peut également être appliqué à des études in vitro .
Résumé
Dans ce manuscrit, les auteurs décrivent un transposon séquençage (Tn-Seq) technique pour identifier et quantifier des mutants de Leptospira interrogans altérés de remise en forme au cours de l’infection de hamsters syriens dorés. TN-Seq combine la mutagénèse aléatoire transposon avec la puissance de la technologie de séquençage haut-débit. Animaux est mis au défi avec une piscine de mutants transposon (entrée piscine), suivie de la récolte du sang et des tissus quelques jours plus tard afin d’identifier et de quantifier le nombre de mutants dans chaque organe (sortie piscines). Les piscines de sortie sont comparés à la piscine d’entrée afin d’évaluer l’aptitude en vivo de chaque mutant. Cette approche permet le dépistage d’une grande piscine de mutants dans un nombre limité d’animaux. Avec des modifications mineures, ce protocole peut être effectué avec n’importe quel modèle animal de la leptospirose, modèles hôte réservoir comme les rats et les infections aiguës comme les hamsters, ainsi que des études in vitro . TN-Seq fournit un outil puissant à l’écran pour des mutants présentant des défauts de remise en forme in vivo et in vitro .
Introduction
Identification des gènes de virulence pour certaines bactéries, comme Leptospira spp., est difficile en raison du nombre limité d’outils génétiques disponibles. Une approche couramment utilisée est la création d’une collection de mutants par mutagenèse aléatoire transposon, suivie de l’identification du site d’insertion dans chaque mutant et test de virulence des mutants de transposon individuels dans un modèle animal. Cette approche est longue et coûteuse et nécessite un grand nombre d’animaux.
Lorsque la mutagénèse aléatoire a été d’abord développé pour le pathogène Leptospira interrogans, gènes impliqués dans la virulence ont été identifiés en testant différents mutants dans un modèle animal1. Mutants ont été sélectionnés selon des critères comme leurs rôles potentiels dans la signalisation ou la motilité ou leur prédit la membrane externe ou surface emplacement. Comme la majorité des leptospires gènes codent pour des protéines hypothétiques de fonction inconnue2, sélection des mutants basé sur ces limites de critères la capacité à découvrir des gènes de virulence de leptospires roman.
Plus récemment, piscines de mutants de transposon L. interrogans ont été examinés pour l’agent infectieux dans les modèles3hamster et souris. Chaque animal a été contestée avec une piscine de 10 mutants. Infectiosité d’un mutant a été marquée comme étant positif s’il a été détecté par PCR des cultures obtenues à partir de sang et les reins. Test de PCR a été laborieux car il exigeait une réaction de PCR individuelle pour chaque mutant dans la piscine. Parce que la fréquence de chaque mutant dans les cultures n’avait pas été évaluée, l’approche était biaisée vers l’identification des mutants très atténués.
Nous décrivons un transposon séquençage (Tn-Seq) technique, comme une stratégie pour dépister plus efficacement les gènes de virulence. TN-seq consistant en la création d’une bibliothèque de mutants par mutagénèse du transposon suivie par séquençage parallèle massive4,5,6. Brièvement, transposon mutants sont mis en commun, inoculés dans animaux et par la suite récupérés de différents organes (sortie piscines). L’ADN de la piscine de sortie est extraite et digéré par les enzymes de restriction ou cisaillée par sonication. Deux cycles de PCR ciblant les jonctions des sites d’insertion de transposon sont effectuées. Cette étape permet l’ajout de l’adaptateur nécessaire pour le séquençage. Les produits PCR obtenus sont analysés par séquençage haut-débit pour identifier le site d’insertion de transposon de chaque mutant de la piscine ainsi que de leur abondance relative, qui est comparé à la composition initiale du bassin de mutant.
Le principal avantage de cette approche est la capacité à l’écran simultanément un grand nombre de mutants avec un petit nombre d’animaux. TN-Seq n’exige pas la connaissance préalable des sites d’insertion de transposon qui augmente les chances de découvrir des nouveaux Leptospira-certains gènes impliqués dans la virulence avec moins de temps et une plus grande efficacité. Parce que le fardeau de leptospires dans les tissus est relativement élevée en rongeur modèles susceptibles d’être mortelles infections (typiquement 104 à 108 bactéries/g de tissu)7,8,9 , ainsi que dans des hôtes réservoirs 10,11, les tissus peuvent être analysés directement sans avoir à la culture, réduire les biais en raison de la croissance in vitro .
Dans les études de Tn-Seq avec la plupart des bactéries pathogènes décrites à ce jour, la haute fréquence de mutagenèse insertionnelle permis infection avec grandes piscines contenant des mutants ayant collectivement plusieurs insertions transposon rapprochés au sein de chaque gène4 ,12,13,14. TN-Seq a également été développé pour une bactérie dont la fréquence de la mutagénèse est beaucoup inférieur6. Avec Leptospira, une bibliothèque de mutants de transposons peut être générée en introduisant le transposon sur un plasmide mobilisable par conjugaison tel que décrit par Slamti et al.15. Toutefois, la fréquence de la mutagénèse transposon de L. interrogans est faible. Lorsque le transposon Himar1 a été introduit dans un plasmide conjugatif, la fréquence de transconjuguant a été signalée comme seuls 8,5 x 10-8 par destinataire de cellules souche de L. interrogans16 Lai et est susceptible d’être de même pauvre avec la plupart des autres souches de L. interrogans. Le protocole décrit ici est en partie basé sur conçu pour Borrelia burgdorferi, dont la fréquence de la mutagénèse insertionnelle transposon est également faible6.
Pour notre expérience pilote avec le protocole17, nous avons mené mutagénèse du transposon avec L. interrogans serovar Manilae souche L495 en raison du succès des autres groupes à isoler des mutants d’insertion de transposon dans la souche ainsi que sa faible DL50 (dose létale) pour virulence1. Nous avons projeté 42 mutants par Tn-Seq et identifié plusieurs candidats mutants défectueux dans la virulence, dont deux avec insertions dans un gène de l’adénylate cyclase du candidat. Une évaluation individuelle des deux mutants chez les hamsters ont confirmé qu’ils étaient déficients en virulence17.
Protocole
ATTENTION : Des souches pathogènes de Leptospira spp doivent être manipulés sous procédures de confinement de niveau de biosécurité 2 (BSL-2). Portez un équipement de protection individuelle (EPI) approprié doit être porté. Une armoire de biosécurité classe II doit être utilisée pour toutes les manipulations des pathogènes Leptospira spp.
1. création du Transposon bibliothèque mutante15
-
Transfert du transposon dans Leptospira spp. par conjugaison (Figure 1)
- Ensemencer un volume de la culture de la phase exponentielle de pathogènes Leptospira spp., correspondant à 10 cellules de7 à 10 mL de Ellinghausen-McCullough-Johnson-Harris moyen (EMJH)18,19. Incuber à 30 ° C à 150 tr/min, secouant jusqu'à ce que la densité atteigne 2-8 x 108 cellules/mL.
NOTE : Le temps de doublement des leptospires pathogènes est de 12 à 24 h, selon les souches. - Ensemencer à 50 µL du donneur Escherichia coli souche β216320 portant le plasmide (pCjTKS2) de transposon mobilisable16 dans 5 mL de Luria bouillon (LB) additionné de 0,3 mM de 2, 6-diaminopimelicacid (DAP), 50 µg/mL de kanamycine (Km) et 50 µg/mL de spectinomycine (Spc) et le lieu du jour au lendemain dans un incubateur à 37 ° C à 255 tr/min.
- Ensemencer 60 µL de cellules d’Escherichia coli dans 3 mL de EMJH additionné de 0,3 mM de DAP (EMJH + DAP). Incuber à 37 ° C à agitation 255 tr/min pendant 3-4 h jusqu'à une OD600nm≈ 0,3.
- Pour monter l’unité de filtration (Figure 1), placer la base sur une tige latérale 125 mL fiole d’Erlenmeyer, placez un filtre avec la cellulose acétate (taille des pores 0,1 mm ; diamètre 25 mm) sur la base et pince l’entonnoir sur le socle. Connectez l’appareil de filtration à un système de vide.
- Ajouter 5 mL de Leptospira spp. culture et 0,5 mL de culture d’e. coli dans l’entonnoir. Aspirer le liquide à travers le filtre.
- Transférer le filtre avec la surface des bactéries vers le haut sur un EMJH + DAPplate. Incuber à 30 ° C pendant la nuit avec le filtre vers le haut.
- Placer le filtre dans un tube de 15 mL contenant 1 mL de EMJH et vortexer pendant 10 s pour libérer les bactéries dans les médias. Répandre 200 µL de la suspension sur 5 EMJH platescontaining 50 µg/mL de Km à l’aide de perles de verre stériles de 1 mm 10-15 ou un épandeur jetable stérile. Enveloppez les plaques avec du parafilm et les incuber à l’envers à 30 ° C pendant 3 à 4 semaines jusqu'à ce que les colonies sont visibles.
- Transférer des colonies individuellement dans 3 mL de EMJH contenant 50 µg/mL de Km (EMJH + Km) à 30° C sous agitation à 150 tr/min pour 7 à 10 jours jusqu'à ce que la culture atteint une densité de ≈10/8ml.
Remarque : Les Cultures peuvent être stockées à-80 ° C ou dans l’azote liquide (avec 4 % de glycérol).
- Ensemencer un volume de la culture de la phase exponentielle de pathogènes Leptospira spp., correspondant à 10 cellules de7 à 10 mL de Ellinghausen-McCullough-Johnson-Harris moyen (EMJH)18,19. Incuber à 30 ° C à 150 tr/min, secouant jusqu'à ce que la densité atteigne 2-8 x 108 cellules/mL.
-
Identification du site d’insertion transposon par PCR nichée (Figure 2)
- Lyse 50 µL de chaque mutant transposon dans des tubes PCR par incubation à 95 ° C pendant 15 min.
Remarque : L’ADN peut être purifié au lieu de cela, à l’aide d’un kit d’extraction d’ADN. - Préparer le mélange PCR avec des amorces Deg1 et Tnk1 (tableau 3) selon le tableau 1. Transférer 23,7 µL du mélange dans chaque tube PCR et ajouter 1,3 µL de cellules lysées. Exécutez le programme : 95° C pendant 5 min ; 40 cycles : 95 ° C pendant 15 s, 40 ° C pendant 1 min, 72 ° C pendant 2 min ; 72 ° C pendant 10 min.
- Faire le mélange PCR avec des amorces Tag et TnkN1 (tableau 3) selon le tableau 2. Transférer 24,2 µL du mélange dans chaque tube PCR et ajouter 0,8 µL de réaction PCR #1. Exécutez le programme : 95 ° C pendant 5 min ; 35 cycles : 95 ° C pendant 15 s, 55 ° C pendant 30 s, 72 ° C pendant 2 min ; 72 ° C pendant 10 min.
- Exécuter 3 µL des produits PCR sur un gel d’agarose à 1 % avec un 1 tampon X Tris-acétate-EDTA (TAE) à 10-15 V/cm (Figure 2 b).
- Purifier les produits PCR des échantillons positifs à l’aide d’un kit de purification de PCR. Éluer ADN avec le volume le plus bas permis par le kit afin d’optimiser la concentration de l’ADN récupéré.
- Envoyer des produits PCR purifiés pour Sanger sequencing utilisant l’amorce de TnkN1 (tableau 3).
- Identifier les sites d’insertion en comparant la séquence obtenue avec la séquence du génome de la souche parentale par analyse BLASTN (http://blast.ncbi.nlm.nih.gov/) ou en utilisant la base de données (http://www.genoscope.cns.fr/agc/mage) de SpiroScope21.
- Confirmer le site d’insertion du transposon de PCR avec des amorces recuit aux séquences flanquantes hôte.
Remarque : Le transposon augmente la taille de la séquence de type sauvage par ≈ 2 Ko.
- Lyse 50 µL de chaque mutant transposon dans des tubes PCR par incubation à 95 ° C pendant 15 min.
2. animal Experiment (Figure 3)
-
Culture des mutants Leptospira
- Se développer individuellement chaque mutant transposon sélectionnés dans 10 mL de EMJH + Km à 30 ° C à l’agitation de 150 tr/min pour une densité de7-10 108 leptospires/mL.
- Compter les leptospires par microscopie à fond noir avec un compteur Petroff-Hausser ou décrites par Miller,23.
- Diluer chaque culture en EMJH à la même densité, par exemple 106 cellules/mL.
- Pour assembler l’entrée piscine, mélanger les cultures dilués en volumes égaux.
NOTE : Inclure des contrôles dans la piscine d’entrée en ajoutant des mutants présentant des défauts de conditionnement physique connue comme loa2217,24 et mutants avec fitness inaltéré comme ligB17,25, respectivement. Piscines de mutants peuvent être stockés avec 4 % de glycérol à-80 ° C ou dans l’azote liquide.
-
Défi
Remarque : Les méthodes décrites ici ont été approuvées par le Comité de l’utilisation (protocole #09018-14) et de vétérans affaires grand Los Angeles institutionnels animalier.- Injecter par voie intrapéritonéale de 1 mL de la piscine d’entrée pour chaque animal avec une seringue à insuline U-100 avec aiguille 26 x ½".
Remarque : Dans l’expérience pilote, 8 animaux ont été contestées avec 1 mL de la piscine d’entrée, c.-à-d. 106 bactéries totales17. L’infection a été autorisée à aller de l’avant pendant 4 jours avant l’euthanasie. - Collecter 10 mL de la piscine d’entrée et actionner pour 20 min à 3 220 x g. Retirer délicatement le surnageant sans déranger le culot. Stocker le culot cellulaire à - 80 ˚c jusqu'à utilisation (étape 3.1.3).
- Surveiller les animaux tous les jours jusqu'à ce qu’ils sont terminent au point de terminaison pré-déterminé. Peser les hamsters quotidiennement et recherchez les critères du point de terminaison : perte d’appétit, démarche ou respiration difficile, prostration, fourrure hérissée, ou > 10 % du poids maximal atteint.
- Injecter par voie intrapéritonéale de 1 mL de la piscine d’entrée pour chaque animal avec une seringue à insuline U-100 avec aiguille 26 x ½".
-
In vitro de l’expérience
- Le jour du défi, ensemencer 3 Fioles jaugées de 25 mL d’EMJH + Km avec 5 mL de la piscine d’entrée. Cultiver des cultures à 30 ° C sous agitation 150 tr/min.
- Compter les leptospires quotidiennement en utilisant une des méthodes de la section 2.1.2. Lorsque la densité atteint ≈ 1 x 108/ml, tourner vers le bas de chaque suspension pendant 20 min à 3 220 x g.
- Stocker les pellets de cellule à-80 ° C jusqu'à l’utilisation.
-
Récolte et stockage des tissus
- Euthanasier les animaux par inhalation isoflurane suivie par thoracotomie bilatérale26.
- Immédiatement percevoir 1 à 2 mL de sang par ponction cardiaque avec une seringue de 3 mL et une aiguille de 25 x 5/8". Transférer le sang dans le tube contenant de l’EDTA. Mélanger par retournement, 5 à 6 fois.
- Recueillir un rein et ⅓ à la moitié du lobe médian ventral du foie dans des cryotubes.
- Conserver les tissus à-80 ° C jusqu'à l’utilisation.
3. construction de banques génomiques pour le séquençage haut-débit (Figure 4)
-
Extraction d’ADN
- Extraction de l’ADN du sang.
- Transférer 100 µL de sang du tube EDTA dans un tube de microcentrifuge.
- Purifier l’ADN à l’aide d’un kit d’extraction d’ADN. Suivez les instructions du fabricant.
- Extraction de l’ADN provenant de tissus.
- Avec des ciseaux et scalpels, dés entre 50 à 80 mg de chaque organe en petits morceaux (1 x 1 mm) et de les transférer dans un tube sec stérile un bouchon qui visse. Mesurer le poids du tissu avec une balance de précision.
- Ajouter 500 µL de PBS stérile dans le tube.
- Homogénéiser les échantillons en utilisant le disruptor PDT 1 min à 5 mouvements par seconde.
- Calculer le volume correspondant à 25 mg de tissu à l’aide de l’équation suivante :
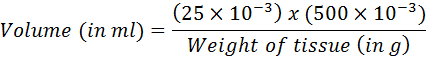
- Transvaser le volume calculé dans le kit d’extraction d’ADN. Procéder à la purification d’ADN suivant les instructions du fabricant.
- Extraction de l’ADN des cultures d’entrée piscine et in vitro .
- Décongelez les granulés bactériennes à température ambiante pendant 5-10 min.
- Procéder à l’extraction d’ADN suivant les instructions qui accompagnent le kit.
- ADN de magasin à-80 ° C jusqu'à l’utilisation.
- Extraction de l’ADN du sang.
-
Tonte l’ADN (Figure 5)
- Transférer 50 µL d’extrait d’ADN sur un tube de microtubes de 1,5 mL.
- Placer les tubes dans le panier de la corne de coupe sonicateur rempli d’eau froide (4 ° C).
- Exécutez le sonicateur pendant 3 min à 80 % d’intensité avec 10 s sur pulse et 5 s au large de l’impulsion.
ATTENTION : Porter des coquilles antibruit ou bouchons d’oreilles pour protéger l’audition. - Courir 2,5 µL de la cisaillé ADN sur un gel d’agarose à 2 % pour confirmer que la majeure partie de l’ADN est < 600 bp en taille.
-
Ajout de la C-queue
- Mesurer la concentration d’ADN avec un spectrophotomètre de petit volume.
- Calculer le volume correspondant à 500 ng d’ADN en utilisant l’équation suivante :
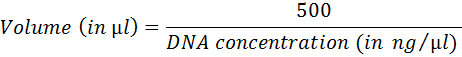
- Préparer les résidus de réaction (tableau 4). Incuber 1 h à 37 ° C ; inactiver à 75 ° C pendant 20 min.
Remarque : Pour les échantillons nécessitant un réglage pour un volume supérieur à 14,5 µL, augmenter le volume final de la réaction de 40 µL et intensifier les composants restants en conséquence. - Nettoyer les échantillons avec un kit de purification de PCR. Éluer l’ADN avec 12 µL de tampon d’élution.
-
PCR nichée
- PCR #1
- Préparer des mélanges PCR selon les tableaux 3 et 5. Transférer 22 µL du mélange bibliothèque dans chaque tube PCR et ajouter 3 µL d’ADN purifié. Procéder de même avec le mélange de contrôle.
NOTE : Primer TnkN317 est spécifique au transposon et apprêt olj3766 est spécifique à la queue-de-C. Le mélange de contrôle fait défaut à l’amorce de TnkN3, qui vise spécifiquement le transposon. - Exécutez le programme suivant : 95 ° C pendant 2 min ; 24 cycles : 95 ° C pendant 30 s, 60 ° C pendant 30 s, 72 ° C pendant 2 min ; 72 ° C pendant 2 min.
- Préparer des mélanges PCR selon les tableaux 3 et 5. Transférer 22 µL du mélange bibliothèque dans chaque tube PCR et ajouter 3 µL d’ADN purifié. Procéder de même avec le mélange de contrôle.
- PCR #2.
- Préparer les mélanges PCR deuxième selon les tableaux 3 et 6. Transférer 49 µL du mélange bibliothèque dans chaque tube PCR et ajouter 1 µL de la réaction de PCR #1 bibliothèque. Transférer 24,5 µL du mélange contrôle dans chaque tube PCR et ajouter 0,5 µL de réaction PCR #1 du contrôle.
Remarque : PMargent2 Primer est spécifique au transposon, et l’IP amorces6 contiennent six-paires code à barres et des séquences spécifiques reconnus par la plate-forme de séquençage de prochaine génération. - Exécutez le programme suivant : 95 ° C pendant 2 min ; 18 cycles : 95 ° C pendant 30 s, 60 ° C pendant 30 s, 72 ° C pendant 2 min ; 72 ° C pendant 2 min.
- Préparer les mélanges PCR deuxième selon les tableaux 3 et 6. Transférer 49 µL du mélange bibliothèque dans chaque tube PCR et ajouter 1 µL de la réaction de PCR #1 bibliothèque. Transférer 24,5 µL du mélange contrôle dans chaque tube PCR et ajouter 0,5 µL de réaction PCR #1 du contrôle.
- Exécuter 3 µL sur un gel d’agarose de 2 %. La bibliothèque devrait montrer un frottis avec la majorité du signal entre 200 et 600 bp (Figure 6) et pas d’amplification de la réaction de contrôle.
- PCR #1
-
Purification des produits de PCR.
NOTE : Nettoyer les banques génomiques avec un kit de purification de PCR suivant les instructions du fabricant. Éluer l’ADN avec 30 µL de tampon d’élution. - Mesurer la concentration d’ADN à l’aide d’un fluorimètre. Moyenne des 2 ou 3 lectures.
- Calculer le volume de chaque bibliothèque équivalente à 15 ou 20 ng à l’aide de l’équation suivante :
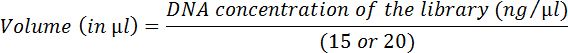
-
Mélanger toutes les bibliothèques ensemble après les précédents calculs. Déterminer la concentration molaire de l’ADN avec l’équation du site suivant :
http://www.molbiol.edu.ru/eng/scripts/01_07.html
ATTENTION : Conditions de concentration et volume ADN dépendent de la plate-forme de séquençage.
4. high-throughput séquençage et l’analyse des données
- Séquençage
- Bibliothèques de séquence que les lectures de single-end bp 64 utilisant le séquençage personnalisé apprêt pMargent3 et l’amorce de séquençage commercial standard. La plate-forme de séquençage vous fournira les fichiers FastQ avec toutes les lectures de séquençage.
- Analyse avec le logiciel Galaxy
- Télécharger le fichier de séquence du génome
- Dans la page d’accueil de la base de données SpiroScope (Voir l’étape 1.2.7 pour lien), sélectionnez l’organisme utilisé pour l’expérience et de cliquez sur « LOAD dans GENOME BROWSER ».
- Dans la barre d’outils (vers le haut de la page d’accueil), sélectionnez « Rechercher/Export > Télécharger data », dans la ligne « Séquence (fasta) », cliquez sur « Génome » pour télécharger la séquence.
- Ouvrez le fichier avec le bloc-notes (PC) ou TextEdit (Mac) et renommez le chromosome (par exemple « ChrI »). Conserver le format fasta.
- Suivez les mêmes étapes pour télécharger la séquence du chromosome II. Combiner les deux séquences du chromosome dans un fichier .txt unique en copiant et en collant.
Remarque : Des séquences du Chromosome aussi peuvent être téléchargés depuis le site NCBI (https://www.ncbi.nlm.nih.gov) et regroupées dans un fichier .txt unique.
- Télécharger des fichiers sur le serveur de la galaxie
NOTE : La galaxie est un open source, plateforme web pour la gestion des données bioinformatique intensive des workflows27,28,29,30 et peut être consulté à https://usegalaxy.org/.- Dans le menu Outils, sélectionnez « obtenir des données > Télécharger le fichier de votre ordinateur ». Glissez l’ou les fichiers .fastq généré par la plate-forme de séquençage dans la fenêtre et cliquez sur « Démarrer ».
- Suivez les mêmes étapes pour télécharger le fichier Leptospira genome sequence .txt.
- Lit Groom
- Sélectionnez « NGS : QC et manipulation > toiletteur FASTQ » dans le menu outils.
- En regard de fichier de toiletter, sélectionnez les bibliothèques téléchargés à l’étape 4.2.2. Dans type de scores de qualité FASTQ d’entrée, sélectionnez le système de séquençage approprié. Pour les Options avancées, laissez Hide Advanced Office sélectionné.
- Cliquez sur « Exécuter ».
- Supprimer les artefacts de séquençage
- Sélectionnez « NGS : QC et manipulation > supprimer les artefacts de séquençage ». À côté de la bibliothèque pour filtrer, sélectionnez les fichiers damées générés à l’étape 4.2.3. Cliquez sur « Exécuter ».
- Supprimer les séquences C-queue
Remarque : Répétez les étapes suivantes une ou deux fois pour s’assurer que tous les C-queues sont supprimés.- Sélectionnez « NGS : QC et manipulation > Clip des séquences de l’adaptateur ».
- Sélectionnez ou entrez la commande suivante :
Bibliothèque de clip : sélectionnez les fichiers générés à l’étape 4.2.4.
Longueur de la séquence minimale : 15
Source : entrez une séquence personnalisée
Entrez la séquence de découpage personnalisé : CCCCCCC
Entrez la valeur non nulle pour garder les séquences de l’adaptateur et les bases de x qui le suivent : 0
Jeter des séquences avec des bases inconnues (N) : Oui
Options de sortie : sortie des séquences coupées et non découpé - Cliquez sur « Exécuter ».
- Supprimer les séquences de l’adaptateur
- Sélectionnez « NGS : QC et manipulation > Clip des séquences de l’adaptateur ».
- Sélectionnez ou entrez la commande suivante :
- Bibliothèque de clip : sélectionnez les fichiers générés à l’étape 4.2.5.
- Longueur de la séquence minimale : 15
- Source : entrez une séquence personnalisée
- Entrez la séquence de découpage personnalisé : CGTATGCCGTCTTCTGCTTG
- Entrez la valeur non nulle pour garder les séquences de l’adaptateur et les bases de x qui le suivent : 0
- Jeter des séquences avec des bases inconnues (N) : Oui
- Options de sortie : sortie des séquences coupées et non découpé
- Cliquez sur « Exécuter ».
- Lectures de filtre basées sur leur qualité
- Sélectionnez « NGS : QC et manipulation > filtre de qualité ».
Remarque : Cet outil sélectionne lectures basés sur les scores de qualité. - Sélectionnez le texte suivant :
Bibliothèque pour filtrer : sélectionner les fichiers générés à l’étape 4.2.6.
Valeur de seuil de qualité : 20
Pour cent de bases dans l’ordre que doit avoir la qualité égale à/supérieur à la valeur seuil : 95 - Cliquez sur « Exécuter ».
Remarque : Avec ces paramètres, lectures plus courts que 20 nucléotides ou avec un score de qualité de 20 ou moins pour 95 % des cycles sont ignorées. Adapter les valeurs de la rigueur à votre expérience.
- Sélectionnez « NGS : QC et manipulation > filtre de qualité ».
- Carte lit32
- Sélectionnez « NGS : cartographie > Bowtie2 »32.
- Les champs dans la fenêtre principale, sélectionnez ce qui suit :
Est cette bibliothèque simple ou jumelée : single-fin
Fichier FASTQ : sélectionnez la bibliothèque filtrée pour la qualité de l’étape 4.2.7.
Écrire des lectures non alignés (au format fastq) pour séparer les fichier (s) : aucun
Écrire lectures alignés (au format fastq) pour séparer les fichier (s) : aucun
Allez-vous choisir un génome de référence de votre histoire ou utiliser un index intégré ? : utiliser un génome de l’histoire et construire l’index
Sélectionnez le génome de référence : sélectionnez le fichier de genome.txt Leptospira téléchargé à l’étape 4.2.2.
Jeu de lire les informations de groupes ? : ne définissez pas
Mode d’analyse de sélection : 1 : réglage par défaut uniquement
Voulez-vous utiliser les préréglages ? : non, il suffit d’utiliser par défaut
Enregistrer les statistiques de mappage bowtie2 dans l’histoire : No
Paramètres de ressources d’emploi : utiliser les paramètres de ressource de travail par défaut - Cliquez sur « Exécuter » pour aligner les lectures au génome.
- Convertir des fichiers
- Sélectionnez « NGS : SAMtools > BAM-à-SAM convertir BAM à SAM ».
- Sélectionnez le texte suivant :
Fichier de BAM pour convertir : Select mappé bibliothèque étape 4.2.8.
Des options d’en-tête : inclure l’en-tête dans la sortie de SAM (-h) - Cliquez sur « Exécuter ».
- Convertir des fichiers
- Sélectionnez « NGS : SAMtools > convertir SAM à intervalle ».
- Sélectionnez le texte suivant :
Sélectionnez Groupe de données à convertir : sélectionnez le fichier de bibliothèque mappé SAM généré à l’étape 4.2.9.
Imprimer tous ? : Oui - Cliquez sur « Exécuter ».
- Lectures de tri
- Sélectionnez « filtrer et trier > Trier les données en ordre croissant ou décroissant ».
- Sélectionnez le texte suivant :
Sorte dataset : sélectionnez l’intervalle fichier généré à l’étape 4.2.10.
colonne : 2
saveur : l’ordre numérique
tout dans : par ordre croissant - Cliquez sur « Exécuter ».
- Sélectionnez lectures appariement chromosomique j’ai
- Sélectionnez « filtrer et trier > sélectionner les lignes qui correspondent à une expression ».
- Sélectionnez le texte suivant :
Sélectionnez les lignes de : sélectionnez le fichier avec des lectures triés généré à l’étape 4.2.11.
qui : correspondance
le modèle : entrez le nom du chromosome I comme déterminé à l’étape 4.2.1.3 (p. ex., « ChrI »). - Cliquez sur « Exécuter ».
- Sélectionnez lectures correspondant du chromosome II
Aller de l’avant après l’étape 4.2.12. - Groupe lit selon les sites d’insertions dans le chromosome j’ai
- Sélectionnez « Join, soustraire et Group > grouper des données par une colonne et d’effectuer une opération globale sur les autres colonnes ».
- Sélectionnez le texte suivant :
Sélectionner des données : sélectionnez le fichier résultant de l’étape 4.2.12.
Groupe de colonne : 2
Ignorer la casse tout groupement ? : non
Ignorer les lignes commençant par ces caractères : Ø
Fonctionnement > + opération d’insertion
Type : comte
Colonne : 2
Arrondir le résultat au nombre entier le plus proche : NO - Cliquez sur « Exécuter ».
- Groupe lit selon les sites d’insertion dans le chromosome II
Aller de l’avant après l’étape 4.2.14. - Sites d’insertion de la sorte sur le chromosome j’ai
- Sélectionnez « filtrer et trier > Trier les données en ordre croissant ou décroissant ».
- Sélectionnez le texte suivant :
Sorte de Dataset : Sélectionnez le fichier de l’étape 4.2.14.
sur colonne : 1
saveur : l’ordre numérique
tout dans : par ordre croissant - Cliquez sur « Exécuter ».
- Trier les sites d’insertion sur le chromosome II
Aller de l’avant après l’étape 4.2.16.
- Télécharger le fichier de séquence du génome
- Analyse statistique
- Transférer les données de la galaxie dans des fichiers de feuille de calcul en copiant et collant des deux colonnes des étapes 4.2.16. et 4.2.17. dans un fichier Excel.
Remarque : La première colonne est la coordonnée de nucléotide de l’emplacement d’insertion de transposon, et la deuxième colonne indique le nombre de lectures à chaque site d’insertion. - Identifier le gène hébergeant le transposon à l’aide de la coordonnée de nucléotides figurant dans le tableau.
NOTE : par exemple, un transposon inséré au nucléotide #30718 du chromosome I est situé dans le gène LIC10024 , qui s’étend sur les nucléotides 29263-31539 chromosome en I. - Calculer les fréquences relatives (F) pour chaque mutant dans chaque tissu et dans la piscine d’entrée suivant l’équation ci-dessous :
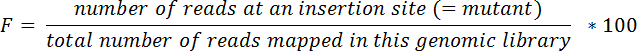
- Calculer les rapports d’entrée/sortie (R) pour chaque mutant et les tissus à l’aide de l’équation suivante :
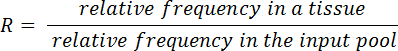
- Test de Wilcoxon signé-rangs
- Normaliser tous les rapports d’entrée/sortie en définissant le ratio médian pour chaque tissu dans chaque animal à 1.0. Un ratio de 1.0 est neutre, > 1.0 est désavantageux, et < 1,0 est avantageuse33.
- Comparer les ratios d’entrée/sortie à 1.0 (remise en forme neutre) en utilisant le test de Wilcoxon avec les valeurs de P < 0,05 considérés comme statistiquement significatifs.
- Transférer les données de la galaxie dans des fichiers de feuille de calcul en copiant et collant des deux colonnes des étapes 4.2.16. et 4.2.17. dans un fichier Excel.
Résultats
Création d’une bibliothèque de mutants de transposon dans L. interrogans par conjugaison nécessite une unité de filtration, comme illustré à la Figure 1. Nous avons récupéré des transconjugants 100-200 de chaque croisement.
Le site d’insertion de transposon est identifié dans chaque mutant par séquençage du produit PCR généré par PCR semi aléatoire qui cible à la fin du...
Discussion
Bien que les résultats de notre expérience pilote pour hamster contestée par voie intrapéritonéale avec 42 L. interrogans mutants sont présentés17, gageons que plus grandes piscines de mutants peuvent être projetés par Tn-suiv. Parce que la fréquence des transconjugants est faible (100-200 transconjugants/accouplement), plusieurs accouplements sont nécessaires pour générer un nombre suffisant de mutants pour les grandes expériences de Tn-Seq. Maintenir un grand nombre de mut...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par un prix de mérite d’affaires des anciens combattants (à D.A.H.) et un Institut National de la santé accorde le R01 AI 034431 (à D.A.H.).
matériels
| Name | Company | Catalog Number | Comments |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K4000 | |
| 2,6-diaminopimelic acid | Sigma-Aldrich | D1377 | |
| Spectinomycin dihydrochloride pentahydrate | Sigma-Aldrich | S4014 | |
| Axio Lab A1 microscope with a darkfieldcondenser | Zeiss | 490950-001-000 | |
| DNeasy blood and tissue kit | Qiagen | 69504/69506 | |
| MinElute PCR Purification | Qiagen | 28004/28006 | |
| QIAquick PCR purification kit | Qiagen | 28104/28106 | |
| Model 505 Sonic Dismembrator | Fisher Scientific | FB-505 | |
| 2.5" Cup horn | Fisher Scientific | FB-4625 | |
| Bead Ruptor 24 | Omni International | 19-010 | Step 3.1.2.4 |
| Terminal deoxynucleotidyl transferase | Promega | M828C | |
| Master mix Phusion | Thermo Scientific | F531 | Preparation of genomic libraries, step 3.4. |
| DreamTaq Master Mix | Thermo Scientific | K9011/K9012 | Identification of the transposon insertion site, step 1.2. |
| dCTP | Thermo Scientific | R0151 | |
| ddCTP | Affymetrix/ USBProducts | 77112 | |
| T100 Thermal cycler | BioRad | 1861096 | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | step 3.6. |
| Qubit dsDNA HS assay kit | Invitrogen | Q32851/Q32854 | step 3.6. |
| Qubit assay tubes | life technologies | Q32856 | step 3.6. |
| PBS pH 7.2 (1X) | Gibco | 20012-027 20012-050 | |
| Disposable scalpel No10 | Feather | 2975#10 | |
| Plastic K2 EDTA 2 ml tubes | BD vacutainer | 367841 | |
| syringe U-100 with 26G x ½” needle | BD vacutainer | 329652 | IP challenge, step 2.2.1. |
| 3 mL Luer-Lok tip syringe | BD vacutainer | 309657 | Cardiac puncture, step 2.4.2. |
| 25G X 5/8” needle | BD vacutainer | 305901 | Cardiac puncture, step 2.4.2. |
| 25 mm fritted glass base with stopper | EMD Millipore | XX1002502 | Filtration unit system, step 1.1.7. |
| 25 mm aluminum spring clamp | EMD Millipore | XX1002503 | Filtration unit system, step 1.1.7. |
| 15 ml borosilcate glass funnel | EMD Millipore | XX1002514 | Filtration unit system, step 1.1.7. |
| 125 ml side-arm Erlenmeyer flask | EMD Millipore | XX1002505 | Filtration unit system, step 1.1.7. |
| Acetate-cellulose filter VVPP (pore size 0.1 mm; diameter 25 mm) | EMD Millipore | VVLP02500 |
Références
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 77 (2), 810-816 (2009).
- Adler, B., Lo, M., Seemann, T., Murray, G. L. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 153 (1-2), 73-81 (2011).
- Marcsisin, R. A., et al. Use of a high-throughput screen to identify Leptospira mutants unable to colonize the carrier host or cause disease in the acute model of infection. J Med Microbiol. 62, 1601-1608 (2013).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat Methods. 6 (10), 767-772 (2009).
- van Opijnen, T., Lazinski, D. W., Camilli, A. Genome-wide fitness and genetic interactions determined by Tn-seq, a high-throughput massively parallel sequencing method for microorganisms. Curr Protoc Microbiol. 36, 1E.3.1-1E.3.24 (2015).
- Troy, E. B., et al. Understanding barriers to Borrelia burgdorferi dissemination during infection using massively parallel sequencing. Infect Immun. 81 (7), 2347-2357 (2013).
- Wunder, E. A., et al. Real-time PCR reveals rapid dissemination of Leptospira interrogans after intraperitoneal and conjunctival inoculation of hamsters. Infect Immun. 84 (7), 2105-2115 (2016).
- Lourdault, K., Aviat, F., Picardeau, M. Use of quantitative real-time PCR for studying the dissemination of Leptospira interrogans in the guinea pig infection model of leptospirosis. J Med Microbiol. 58, 648-655 (2009).
- Coutinho, M. L., et al. Kinetics of Leptospira interrogans infection in hamsters after intradermal and subcutaneous challenge. PLoS Negl Trop Dis. 8 (11), e3307 (2014).
- Athanazio, D. A., et al. Rattus norvegicus as a model for persistent renal colonization by pathogenic Leptospira interrogans. Acta Trop. 105 (2), 176-180 (2008).
- Ratet, G., et al. Live imaging of bioluminescent Leptospira interrogans in mice reveals renal colonization as a stealth escape from the blood defenses and antibiotics. PLoS Negl Trop Dis. 8 (12), e3359 (2014).
- Gutierrez, M. G., Yoder-Himes, D. R., Warawa, J. M. Comprehensive identification of virulence factors required for respiratory melioidosis using Tn-seq mutagenesis. Front Cell Infect Microbiol. 5 (78), (2015).
- Gawronski, J. D., Wong, S. M., Giannoukos, G., Ward, D. V., Akerley, B. J. Tracking insertion mutants within libraries by deep sequencing and a genome-wide screen for Haemophilus genes required in the lung. Proc Natl Acad Sci U S A. 106 (38), 16422-16427 (2009).
- Gallagher, L. A., Shendure, J., Manoil, C. Genome-scale identification of resistance functions in Pseudomonas aeruginosa using Tn-seq. MBio. 2 (1), (2011).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods Mol Biol. 859, (2012).
- Picardeau, M. Conjugative transfer between Escherichia coli and Leptospira spp. as a new genetic tool. Appl Environ Microbiol. 74 (1), 319-322 (2008).
- Lourdault, K., Matsunaga, J., Haake, D. A. High-throughput parallel sequencing to measure fitness of Leptospira interrogans transposon insertion mutants during acute infection. PLoS Negl Trop Dis. 10 (11), e0005117 (2016).
- Ellinghausen, H. C., McCullough, W. G. Nutrition of Leptospirapomona and growth of 13 other serotypes: Fractionation of oleic albumin complex and a medium of bovine albumin and polysorbate 80. Am J Vet Res. 26, 45-51 (1965).
- Johnson, R. C., Harris, V. G. Differentiation of pathogenic and saprophytic letospires. I. Growth at low temperatures. J Bacteriol. 94 (1), 27-31 (1967).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPa) conjugative machineries and their cognate Escherichia coli host strains. Res Microbiol. 156 (2), 245-255 (2005).
- Vallenet, D., et al. MicroScope: a platform for microbial genome annotation and comparative genomics. Database (Oxford). , bap021 (2009).
- Goodman, A. L., Wu, M., Gordon, J. I. Identifying microbial fitness determinants by insertion sequencing using genome-wide transposon mutant libraries. Nat. Protocols. 6 (12), 1969-1980 (2011).
- Miller, J. . Spirochetes in body fluids and tissues manual of investigative methods. , 22-23 (1971).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathog. 3 (7), (2007).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infect Immun. 76 (12), 5826-5833 (2008).
- Haake, D. A. Hamster model of leptospirosis. Curr Protoc Microbiol. 12, 2 (2006).
- Giardine, B., et al. Galaxy: a platform for interactive large-scale genome analysis. Genome Res. 15 (10), 1451-1455 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Blankenberg, D., et al. Galaxy: a web-based genome analysis tool for experimentalists. Curr Protoc Mol Biol. (Chapter 19), 11-21 (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Res. 8 (44), (2016).
- McCoy, K. M., Antonio, M. L., van Opijnen, T. MAGenTA; a Galaxy implemented tool for complete Tn-Seq analysis and data visualization. Bioinformatics. , 1367 (2017).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Kamp, H. D., Patimalla-Dipali, B., Lazinski, D. W., Wallace-Gadsden, F., Camilli, A. Gene fitness landscapes of Vibrio cholerae at important stages of its life cycle. PLoS Pathog. 9 (12), e1003800 (2013).
- Poggi, D., Oliveira de Giuseppe, P., Picardeau, M. Antibiotic resistance markers for genetic manipulations of Leptospira spp. Appl Environ Microbiol. 76 (14), 4882-4885 (2010).
- van Opijnen, T., Camilli, A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms. Nat Rev Microbiol. 11 (7), 435-442 (2013).
- Pappas, C. J., Picardeau, M. Control of gene expression in Leptospira spp. by Transcription activator-like effectors demonstrates a potential role for LigA and LigB in Leptospira interrogans virulence. Appl Environ Microbiol. 81 (22), 7888-7892 (2015).
- Koskiniemi, S., Sun, S., Berg, O. G., Andersson, D. I. Selection-driven gene loss in bacteria. PLoS Genet. 8 (6), e1002787 (2012).
- Troy, E. B., et al. Global Tn-seq analysis of carbohydrate utilization and vertebrate infectivity of Borrelia burgdorferi. Mol Microbiol. 101 (6), 1003-1023 (2016).
- Ramsey, M. E., et al. A high-throughput genetic screen identifies previously uncharacterized Borrelia burgdorferi genes important for resistance against reactive oxygen and nitrogen species. PLoS Negl Trop Dis. 13 (2), e1006225 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon