Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Titrage ELISA comme une méthode pour déterminer la constante de Dissociation de l’Interaction Ligand du récepteur
Dans cet article
Résumé
Un protocole détaillé pour effectuer un titrage ELISA est décrite. En outre, un nouvel algorithme est présenté afin d’évaluer le titrage des tests ELISA et d’obtenir une constante de dissociation de liaison d’un ligand soluble à un récepteur immobilisée plaque de microtitration.
Résumé
La constante de dissociation décrit l’interaction entre les deux partenaires dans l’équilibre de liaison et est une mesure de leur affinité. C’est un paramètre crucial pour comparer différents ligands, par exemple, des inhibiteurs compétitifs, isoformes protéiques et mutants, pour leur force de la liaison à un partenaire de liaison. Constantes de dissociation sont déterminés en fonction des concentrations de lié contre ligand libre comme les courbes de liaison. En revanche, les courbes de titration, dans lequel un signal qui est proportionnel à la concentration de ligand lié est comploté contre la concentration totale de ligand ajouté, sont beaucoup plus faciles à enregistrer. Le signal peut être détecté par spectroscopie et par dosage immunoenzymatique (ELISA). Ceci est illustré dans un protocole pour un titrage ELISA qui mesure la liaison de la rhodocetin dérivé de venin de serpent à son domaine cible immobilisé de l’intégrine α2β1. Titrage des tests ELISA sont polyvalents et largement utilisé. N’importe quelle paire de protéines qui interagissent utilisable comme récepteur immobilisé et ligand soluble, pourvu que les deux protéines sont purs, et leurs concentrations sont connues. Jusqu'à présent, la difficulté a été de déterminer la constante de dissociation d’une courbe de titrage. Dans cette étude, une fonction mathématique qui sous-tend les courbes de titrage est introduite. Sans aucune estimation Erreurs graphique d’un rendement de saturation, cet algorithme permet le traitement des données brutes (intensité de signal à différentes concentrations du ligand ajouté) directement par l’évaluation mathématique par régression non linéaire. Ainsi, plusieurs courbes de titration peuvent être enregistrés simultanément et transformés en un ensemble de paramètres caractéristiques, dont la constante de dissociation et la concentration de liaison active du récepteur, et elles peuvent être évaluées statistiquement. Lorsqu’il est combiné avec cet algorithme, titrage ELISA bénéficier de l’avantage de présenter directement la constante de dissociation. Donc, ils peuvent être utilisés plus efficacement dans l’avenir.
Introduction
La constante de dissociation K est un paramètre clé pour décrire l’affinité d’un récepteur (R) pour son ligand (L). Se fondant sur la Loi d’action de masse, K est défini pour l’équilibre, dans lequel la RL complexe récepteur-ligand dissocie le récepteur R et le ligand l :
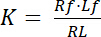 Équation 1
Équation 1
avec les indices f indiquant l’État libre/non consolidé de récepteurs et ligands. La concentration du complexe récepteur-ligand, RL, est identique à la concentration de ligand lié aux récepteurs Lb. Comme la concentration totale de récepteur Rt est la somme du récepteur libre Rf et liaison ligand du récepteur Rb = Lb, la constante de dissociation peut également s’écrire :
 Équation 2
Équation 2
Par conséquent, la saturation rendement Y, définie comme la fraction de ligand lié Lb en fonction de la concentration totale de récepteur Rt,
 Équation 3
Équation 3
dépend de la concentration de ligand libre Lf:
 Équation 4
Équation 4
Cette relation hyperbolique décrit la courbe de liaison d’une interaction ligand-récepteur et son intrigue montre la concentration de ligand lié Lb en fonction de la concentration de ligand libre Lf. De la courbe de liaison, la constante de dissociation K peut être dérivée comme la concentration de ligand libre au rendement de saturation maximale. En outre, différents algorithmes de linéariser les courbes de liaison ont été établis, tels que le tracé à double réciprocité par Klotz1,2, ou des transformations selon Scatchard ou Hanes (évaluée par Bisswanger3). Cependant, tous les algorithmes souffrent du problème que le rendement de saturation, qui l'on approche asymptotiquement à des concentrations élevées de ligand libre dans la courbe de liaison, la valeur maximale doit être estimée dans une pré-évaluation graphique et est donc sujette aux erreurs.
En outre, la détermination d’une courbe de liaison nécessite la quantification du ligand libre et liée au cours de l’équilibre de liaison. À cette fin, le ligand libre doit être séparé du ligand lié aux récepteurs et quantifiée. Par conséquent, le ligand et le récepteur doivent se distinguent par leurs propriétés, comme un ligand non protéique par opposition à un récepteur de protéine. Si les deux partenaires de liaison sont des protéines, ils doivent être distingués dans leurs tailles, frais ou autres caractéristiques moléculaires. Néanmoins, la quantification des concentrations de ligand dans les approches contraignantes à petite échelle est une tâche difficile. Marquage radioactif du ligand a souvent été nécessaire pour détecter la faible concentration de ligand lié, surtout si des quantités substantielles de récepteurs n’étaient pas disponible ou accessible. En outre, le ligand lié aux récepteurs peut dissocier pendant et après l’isolement d’une manière non négligeable. Par conséquent, les méthodes complexes, tels que l’équilibre gel filtration4, électrophorèse capillaire5et impulsion protéolyse6, sont tenus de quantifier lié aux récepteurs ligand et séparer de ligand libre.
Contrairement à ces essais de liaison, les expériences de titrage ne nécessitent pas la séparation quantitative du ligand lié et libre. À cette fin, un récepteur à une concentration constante est titré avec différentes concentrations de ligand ajouté. En se liant au récepteur, le ligand lié a une propriété biophysique qui distingue le ligand libre et celles-ci sont mesurables selon, par exemple, photométrie, fluorométrie ou détection d’anticorps. Ainsi, un signal S, qui est proportionnelle à la saturation rendement Y et par conséquent aussi à la concentration de ligand lié aux récepteurs (Lb), est détecté en fonction de la concentration totale de ligand ajouté (L,t). Les deux paramètres, le signal S et la concentration totale de ligand ajouté sont quantifiées de manière directe et plus facile que les concentrations du ligand lié et libre. Surtout, la détection du ligand lié aux récepteurs par dosage immunoenzymatique (ELISA) a permis la réduction du volume des échantillons à au-dessous de 100 µL, ainsi que des mesures parallèles de plusieurs concentrations de ligand en microplaques multipuits. Dans un titrage ELISA, un récepteur est physiquement adsorbé sur une plaque de microtitrage à la même concentration et titré par ligand soluble. Le récepteur est immobilisé à la surface en plastique essentiellement par adsorption hydrophobe. La concentration de surface du récepteur immobilisé corrélats avec la concentration de revêtement du récepteur dans une relation non linéaire, probable selon Langmuir´s adsorption isotherme7. Outre le nombre total de molécules adsorbées du récepteur, leur état d’activité est un autre paramètre important pour des dosages de titrage. Seulement immobilisé les récepteurs qui ont conservé l’activité de liaison ligand, sont pertinentes pour la détermination du titrage et éventuellement contribuent à la concentration totale de récepteurs actifs Rt de l’essai de titrage, qui ne peut être déterminée directement.
Sites sur la surface en plastique, qui ne sont pas couverts par le récepteur immobilisé ont tendance à s’adsorber les autres protéines, tels que le ligand. L’adsorption physique du ligand vers ces sites de surface en plastique se traduirait en un signal analogue comme le ligand lié aux récepteurs, mais de façon non spécifique. Pour réduire ce signal non spécifique, les sites de surface en plastique des plaques microtitration qui n’ont pas été enduits de protéine mais seront bloqués avec l’albumine sérique bovine (BSA). Toutefois, pour certains tests de titrage de récepteur-ligand, les signaux de fond non spécifiques peuvent être observées. Ensuite, les autres agents de blocage, comme une solution de gélatine de 0,2 % ou de 0,04 % Tween 20, sont recommandés.
Après liaison au récepteur, le ligand libre est éliminé par deux étapes de lavage. Ligand lié reste avec le récepteur, qui est immobilisé sur la surface en plastique du puits microplaques et éventuellement renforcé par fixation chimique. Pour la suivante Croix-liaison covalente du ligand lié et récepteur immobilisé au glutaraldéhyde, la substance tampon TRIS est remplacée pour HEPES, sans aucune modification de liaison du ligand. HEPES, contrairement aux TRIS, ne pas inactiver glutaraldéhyde. La Croix-liaison covalente au glutaraldéhyde corrige le ligand lié avec son récepteur et empêche sa dissociation au cours des étapes ultérieures de lavage et d’incubation. Ainsi, l’interaction de récepteur-ligand est chimiquement gelée et garantit une courbe de titrage, qui n’est pas affectée par les étapes suivantes de lavage et d’incubation. Cependant, la fixation de glutaraldéhyde peut modifier chimiquement le ligand et le récepteur de telle sorte que leur interaction est réduite ou supprimée. En outre, modification des épitopes dans le ligand peut changer l’affinité de l’anticorps de détection, surtout si un anticorps monoclonal est utilisé pour quantifier le ligand lié. Bien qu’aucun de ces effets indésirables de la fixation de glutaraldéhyde se produit dans ce titrage ELISA, la sensibilité du test vers le glutaraldéhyde seront essayée lors de chaque interaction ligand-récepteur avant l’expérience de titrage. Après fixation, glutaraldéhyde excès est éliminé en trois étapes de lavage avec le tampon TRIS contenant. TRIS inactive groupes aldéhyde restants, qui pourraient réagir de façon non spécifique avec détection d’anticorps dans l’étape ultérieure.
Doser le ligand lié avec des anticorps-enzymatique, qui fournissent un signal photométrique de ELISA S. C’est tracé par rapport à la concentration L de ligand totalt ajouté à chaque puits. Malgré son acquisition plus facile, la courbe de titrage n’est pas une fonction hyperbolique, contrairement à la courbe de liaison. En outre, on sait pas comment calculer la constante de dissociation K d’une courbe de titrage. Bien que les algorithmes de linéariser des courbes de titrage par spectroscopie acquis ont été rapportés indépendamment de Stockell8 et Heyn et Weischet9, ils sont tombés courts en raison de leur incertitude de l’estimation du signal maximum valeur que le rendement de saturation s’approche à des concentrations élevées de ligand ajouté.
Ici, un titrage ELISA et un algorithme de régression non linéaire sont décrits pour calculer la constante de dissociation K d’interaction ligand du récepteur d’une courbe de titrage. Ce protocole est illustré pour l’interaction de l’A-domaine de collagène-fixation de l’intégrine α2β1 avec un inhibiteur de dérivés de venin de serpent. Les intégrines sont des molécules d’adhérence cellulaire, qui négocient l’ancrage des cellules de la matrice extracellulaire environnante ou les membrane basale sous-jacente10,11. Par ailleurs, les intégrines transmettent des signaux importants entre les cellules et la matrice extracellulaire en recrutant d’autres molécules de signalisation et formant de nouveaux organites de la cellule, adhesomes, sur la cellule-matrice interaction12,13, 14. collagène, le ligand de l’intégrine α2β1, est la protéine la plus abondante du corps humain et est une composante cruciale d’échafaudage du tissu conjonctif15. L’interaction entre l’intégrine α2β1 et collagène est médiée par l’A-domaine de la sous-unité α2 de l’intégrine. L’intégrine α2A-domaine contienne un cation divalent, qui est requise pour la liaison de collagène et stabilise sa structure. La forme sauvage, mais aussi les mutants du domaine α2A, telle que celle dans laquelle les résidus de surface exposée Y216 avaient été remplacé pour une glycine, peuvent être facilement produits inoculation dans un système d’expression bactérienne et isolés par l’intermédiaire de leurs oligo-son-tags avec un NiNTA colonne de SuperFlow avec une dialyse ultérieur contre tampon TRIS salin (SCT, 50 mM TRIS/HCl, pH 7,4, NaCl 150 mM), contenant 2 mM MgCl216. Leurs concentrations ont été mesurées avec le dosage d’acide bicinchoninic (BCA) et leur pureté est testées par SDS-PAGE conventionnelle et colorées au bleu de Coomassie-Brilliant Blue R250.
L’interaction entre l’intégrine α2β1 et collagène est bloquée par la liaison de la composante de venin de serpent, rhodocetin, de la Malayan pit viper (Calloselasma rhodostoma)16,17. Utilisé comme un ligand soluble dans ce titrage, ELISA, rhodocetin a été purifiée à partir du pétrole brut venin comme décrit plus haut16. Il est dissous dans une solution saline tamponnée HEPES (HBS ; 10 mM HEPES/NaOH, pH 7,4, NaCl 150 mM) et est conservé congelés à-20 ° C. Sa concentration a été déterminée par BCA et sa pureté a été prouvée par SDS-PAGE. Comme antagoniste, rhodocetin bloque non seulement collagène liant à l’intégrine α2β1 A-domaine, mais aussi se stabilise la conformation inactive de l’intégrine, empêchant ainsi toute signalisation de collagène dans les cellules ou les plaquettes sanguines18. Il est d’une grande importance biomedical pour déterminer la constante de dissociation de rhodocetin avec son objectif de récepteur et ainsi éclaircir son mécanisme moléculaire et pharmaceutiques potentiels par exemple, un agent antithrombotique. À cette fin, un titrage ELISA est décrite, y compris son évaluation, qui s’applique à presque n’importe quelle interaction de récepteur-ligand avec une stoechiométrie d’interaction 1:1.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. stocks Solutions
- Pour préparer 100 mL de 10 x TBS solution pH 7,4, dissoudre 6,06 g de TRIS et 8,77 g de NaCl dans 90 mL d’eau désionisée, ajuster le pH à 7,4 avec 37 % HCl solution, remplir le volume à 100 mL d’eau désionisée et filtrer la solution.
- Préparer 100 mL de 1 M HEPES/NaOH, solution à pH 7,4, dissoudre 23,83 g de HEPES dans 90 mL d’eau désionisée, ajuster le pH à 7,4 avec 1,5 M NaOH, remplir le volume à 100 mL d’eau désionisée et filtrer la solution.
- Pour la préparation de 100 mL de solution de NaCl à 5 M, dissoudre 29,2 g de NaCl dans 90 mL d’eau désionisée. Remplissez le volume à 100 mL d’eau désionisée et filtrer la solution.
- Pour préparer 100 mL d’une solution 1 M MgCl2 , dissoudre 20,33 g de MgCl2 · 6H2O dans 90 mL d’eau désionisée. Remplissez le volume à 100 mL d’eau désionisée et filtrer la solution.
- Pour préparer 5 % inactivés par la chaleur de la BSA dans l’eau, pèse 2,5 g de BSA (fraction V, pH 7,0) dans un tube de 50 mL, dissoudre dans 45 mL d’eau désionisée. Remplir la solution à 50 mL d’eau désionisée et chauffer la solution dans un bain d’eau à 68 ° C pour 45 min. refroidir dans un bain de glace et conserver à-20 ° C.
- Préparer la solution aqueuse de glutaraldéhyde 25 %.
ATTENTION : Glutaraldéhyde est nocif si avalé, toxique par inhalation et provoque des brûlures. Portez des vêtements protecteurs, gants et de la protection des yeux/visage. - Soulever l’antisérum de lapin comme décrit précédemment19. Le titre de l’antisérum a été déterminé conformément aux protocoles standard20.
- Préparer les immunoglobulines-anticorps de anti-lapin de chèvre conjugué à la phosphatase alcaline.
- Pour préparer 100 mL d’une solution 0,1 M glycine, dissoudre 0,75 g de glycine dans 90 mL d’eau désionisée. Ajuster le pH à 10.4 avec une solution de NaOH de 1,5 M, remplissez le volume à 100 mL d’eau désionisée et filtrer la solution.
- Préparer 100 mL de 0,5 M Zn (II)-solution d’acétate, dissoudre 10,98 g de Zn (II)-acétate · 2H2O dans 90 mL d’eau désionisée. Remplissez le volume à 100 mL d’eau désionisée et filtrer la solution.
- Pour la préparation de 100 mL de solution de NaOH à 1,5 M, dissoudre 6,0 g de NaOH dans 90 mL d’eau désionisée. Remplissez le volume à 100 mL d’eau désionisée et filtrer la solution.
2. Préparez les tampons et Solutions de travail
- Diluer 5 mL de 10 x TBS pH 7,4, avec 45 mL d’eau désionisée et ajouter 100 µL d’une solution 1 M MgCl2 . Gardez-le à température ambiante. Le SCT, pH 7,4, 2 mM MgCl2 solution est pour l’immobilisation du receveur et laver la plaque de microtitration.
- Diluer 10 mL de 5 % de BSA inactivés par la chaleur dans l’eau et 5 mL de 10 x TBS, pH 7,4 avec de l’eau désionisée jusqu'à 50 mL. Ajouter 100 µL de solution mère de 1 M du MgCl2 et bien mélanger la solution. Gardez-le sur glace et le stocker pour d’autres expériences à-20 ° C. Noter que 2,5 mL de 1 % de BSA au SCT, pH 7,4, 2 mM MgCl2 sont requis pour chaque courbe de titrage.
- Diluer 2,5 mL de 1 M HEPES/NaOH, solution de NaCl 5 M pH 7.4 et 1,5 mL à 50 mL avec de l’eau désionisée, ajouter 100 µL de 1 M du MgCl2 solution et mélanger la solution pour préparer 50 mL de HBS, pH 7,4 : 50 mM HEPES/NaOH , NaCl 150 mM.
- Ajouter 5 µL de la solution stock MgCl2 1M et 2 µL 0,5 M Zn (II)-solution mère d’acétate de 5 mL de solution de glycine 0,1 M, pH 10.4 pour préparer le tampon de la phosphatase alcaline (PA) (solution de glycine de 0,1 M, pH 10.4, 1 mM MgCl2, 0. 2 mM Zn(II)-acetate).
3. immobilisation du récepteur (intégrine α2A-domaine) sur une plaque de microtitration
- Diluer la solution mère d’intégrine α2A-domaine au SCT, pH 7,4, 2 mM MgCl2 pour une concentration finale de 5 µg/mL.
Remarque : Le volume de la solution de revêtement est 650 µL pour une courbe de titrage comprenant un puits 12-ligne d’une moitié de zone-microplaque. - Remplir chaque cupule d’une ligne sur une moitié de zone-microplaque avec 50 µL/puits de la solution de revêtement 5 µg/mL d’intégrine α2A-domaine (sauvage ou mutant). Effectuer tous les rangs par titrage au moins en double (dans cet exemple comme quadruplés ; Voir présentation de la plaque de microtitration à la Figure 1).
- Sceller la plaque d’aluminium ou le fermer avec un couvercle. Laisser la plaque à 4 ° C durant la nuit.
4. lavage des puits recouverts de la plaque deux fois avec le SCT, pH 7,4, 2 mM MgCl2
- Pour supprimer le récepteur soluble molécules qui n’ont pas été immobilisées à la surface en plastique par adsorption physique, enlever la solution de revêtement et remplissent chaque bien avec 50 µL de TBS, pH 7,4, 2 mM MgCl2.
- Enlever la solution de lavage. S’assurer que les puits ne deviennent pas secs. Par conséquent, ne pas taper la plaque de microtitration sur un chiffon de tissu pour éliminer le liquide résiduel. Utiliser une pipette plusieurs étapes ou une pipette multi-canaux pour remplir les puits rapidement.
- Répéter une fois cette étape de lavage.
5. bloquer les Sites de fixation non spécifique
- Ajouter 50 µL de solution à 1 % BSA au SCT, pH 7,4, 2 mM MgCl2 dans chaque puits.
- Refermer les puits avec du papier ou fermer avec un couvercle.
- Incuber les puits pendant 1 h à température ambiante.
Remarque : Aucune étape d’incubation du présent protocole peut être effectuée sur une plate-forme bascule ou secouer. Toutefois, cela n’est pas nécessaire et n’altère pas le résultat de l’expérience.
6. préparation d’une ligne série de Dilution du Ligand, Rhodocetin
- Varier la concentration de départ de rhodocetin et le facteur de dilution de la dilution en série, pour obtenir une gamme appropriée de concentrations du ligand et d’enregistrer une courbe de titrage complet avec un signal minimum et maximum. Dans cette expérience, emploient une concentration de début de 243 rhodocetin nM et un facteur de dilution de 2.3. Diluer la solution mère de rhodocetin à la plus forte concentration de ligand de la ligne de dilution en série. Pour chaque courbe de titrage avec un facteur de dilution de la 2.3, préparer 115 µL de solution de rhodocetin 243 nM (c.-à-d., 15,2 µg/mL) chez 1 % BSA/SCT, pH 7,4, 2 mM MgCl2 en tube à essai #1.
- Remplir 65 µL de solution à 1 % BSA au SCT, pH 7,4, 2 mM MgCl2 de 10 tubes à essai, étiqueté #2 à #11.
- Transférer 50 µL de la dilution de rhodocetin du tube à essai #1 pour tube à essai #2, mélanger les deux solutions (volume total : 115 µL ; facteur de dilution : 1 : 2. 3) par la trituration et ensuite transférer 50 µL de ce mélange pour tube à essai #3, etc.
- Continuer cette dilution en série jusqu’au tube à essai #11.
Remarque : Les volumes indiqués en étapes 6.1-6.4 suffisent pour une courbe de titrage. Ces volumes il faut multiplier par le nombre de répétitions. Dans ce cas, effectuer huit courbes de titrage (quadruplés de deux formes α2A-domaine) et préparer les volumes suivants : 920 µL de solution de rhodocetin à la concentration maximale en tube à essai #1 ; 520 µL de 1 % de BSA au SCT, pH 7,4, 2 mM MgCl2 à remplir dans chacun des tubes de test #2 à #11 ; et 400 µL du volume de transfert d’un tube à la suivante.
7. fixation du Ligand (Rhodocetin) à différentes Concentrations d’immobilisé du récepteur (intégrine α2A-domaine)
- Enlever la solution de blocage provenant des puits de plaque de microtitration par une ligne vide.
- Immédiatement ajouter 50 µL de la solution de rhodocetin dans 1 % BSA/SCT, solution à pH 7,4/MgCl2 de tube à essai #1 dans les puits de la colonne 1, solution de tube à essai #2 à Herbert George wells de la colonne 2,etc. ajouter 50 µL de 1 % de BSA au SCT, pH 7,4 , 2 mM MgCl2 (tampon de blocage et de la dilution) comme un témoin sans ligand aux puits de colonne 12 (voir présentation de la plaque de microtitration, Figure 1).
- Refermer les puits avec du papier ou fermer avec un couvercle.
- Incuber les puits pendant 1,5 h à température ambiante (environ 20-22 ° C).
8. Lavez puits de la plaque deux fois avec HBS, pH 7,4, 2 mM MgCl2
- Pour supprimer des molécules non liées au ligand, enlever la solution de liaison et remplir chaque bien avec 50 µL de HBS, pH 7,4, 2 mM MgCl2. Ensuite, enlever la solution de lavage.
- Veiller à ce que les puits ne deviennent pas secs. Par conséquent, ne pas taper la plaque de microtitration sur un chiffon de tissu pour éliminer le liquide résiduel. Utiliser une pipette plusieurs étapes ou une pipette multi-canaux pour remplir les puits rapidement.
- Répéter une fois cette étape de lavage.
9. fixer le Ligand lié aux récepteurs avec 2,5 % glutaraldéhyde à HBS, pH 7,4, 2 mM MgCl2
- Préparer une solution de glutaraldéhyde 2,5 % frais en mélangeant 1 partie de la solution de glutaraldéhyde 25 % et de 9 parties de HBS, pH 7,4, 2 mM MgCl2.
- Remplir chaque puits de la microplaque avec 50 µL de la solution de glutaraldéhyde à 2,5 % en HBS, pH 7,4, 2 mM MgCl2. Incuber la microplaque pendant 10 min à température ambiante.
10. laver le puits de la plaque trois fois avec 50 µL/puits du SCT, pH 7,4, 2 mM MgCl2
- Pour supprimer et inactiver les excès glutaraldéhyde, enlever la solution de fixation et remplir chaque bien avec 50 µL de TBS, pH 7,4, 2 mM MgCl2. Retirez ensuite la solution de lavage.
NOTE : Versez la solution contenant du glutaraldéhyde de fixation de la plaque de microtitrage dans un plat et jeter la solution de fixation après que elle a été inactivée par un volume égal de TBS, pH 7,4. - Veiller à ce que les puits ne deviennent pas secs. Par conséquent, ne pas taper la plaque de microtitration sur un chiffon de tissu pour éliminer le liquide résiduel. Utiliser une pipette plusieurs étapes ou une pipette multi-canaux pour remplir les puits rapidement.
- Répétez cette étape de laver deux fois.
11. quantification du Ligand lié aux récepteurs par ELISA
- Ajouter 50 µL/puits de la solution d’anticorps primaire 1 % BSA au SCT, pH 7,4, 2 mM MgCl2. La solution d’anticorps primaire est un antisérum de lapin contre rhodocetin19, dilué 1:2,000 1 % BSA au SCT, pH 7,4, 2 mM MgCl2.
- Incuber les plaques pour 75-90 min à température ambiante. Laver tous les puits de la plaque trois fois avec 50 µL/puits du SCT, pH 7,4, 2 mM MgCl2. Au cours des trois étapes de lavage, taraudage de la plaque de microtitration sur un tissu tissu n’est pas nécessaire.
- Ajouter 50 µL/puits de la solution d’anticorps secondaire 1 % BSA au SCT, pH 7,4, 2 mM MgCl2. À cette fin, diluer l’anticorps secondaires, anticorps de chèvre-ciblant les immunoglobulines lapin conjugués à la phosphatase alcaline, à 1:2,000 1 % BSA au SCT, pH 7,4, 2 mM MgCl2. Incuber les plaques pour 75-90 min à température ambiante.
- Préparer la solution AP-détection de dissoudre un comprimé de 5 mg contenant 4-nitrophényl phosphate disodique sel hexahydrate (substrat de phosphatase) dans 5 mL de tampon de AP (solution de glycine de 0,1 M, pH 10.4, contenant 1 mM MgCl2 et 0,2 mM Zn(II)-acetate).
- Laver tous les puits de la plaque trois fois avec 50 µL/puits du SCT, pH 7,4, 2 mM MgCl2, immédiatement avant de passer à l’étape suivante. Appuyez sur la plaque de microtitration sur un chiffon de tissu après la dernière étape de lavage pour éliminer toute trace de liquide.
- Ajouter 50 µL/puits de la solution de détection des AP dans les puits de la plaque de microtitration. Ajouter la solution AP-détecter rapidement dans toutes les loges pour démarrer la conversion enzymatique aussi simultanément que possible. Par conséquent, utiliser une pipette multi-canaux.
- Incuber les plaques à température ambiante jusqu'à ce que la solution dans les puits ayant la plus forte concentration de ligand jaunissent.
Remarque : Le temps d’incubation peut varier entre 5 min et 1 h selon l’intensité du signal. - Arrêter la conversion du substrat phosphatase en ajoutant 50 µL/puits de la solution de NaOH à 1,5 M. Laissez le reposer plaque pendant plusieurs minutes afin d’assurer sans stries mélange des deux solutions. Pour justifier la même période de l’incubation dans tous les puits, utiliser une pipette multi-canaux et ajoute les 1,5 M de NaOH dans le même ordre ajouté aux puits que celle du substrat AP-détectant à l’étape 11,8.
- Mesurer la densité optique (do) à 405 nm de chaque puits par un lecteur ELISA.
12. l’évaluation des signaux titrage
- Ouvrez la table de données brutes, les valeurs de405nm OD, avec Excel. Ces valeurs de signal des courbes de titrage sont lire en lignes, transposer les valeurs d’une colonne et une étiquette avec les concentrations du ligand ajouté dans une autre colonne.
- Ouvrez le Graphpad Prism 5 (Version 5.0). Ouvrez un nouveau fichier de projet dans le menu principal. Choisissez le format XY graph& nouvelles données. Choisissez l’option entrée et tracé un point unique pour chaque valeur de l’axe y.
- Copiez les deux colonnes, la concentration de ligand ajouté et du signal (valeurs OD405nm ) de l’excellent dossier et collez-les dans la fiche de données de GraphPad Prism comme valeurs X et Y, respectivement.
- Ouvrez le programme sous analyse de GraphPad Prism 5 et choisissez l’option régression non-linéaire sous XY-analyse. Choisissez l’équation définie par l’utilisateur et appuyez sur le bouton nouveau pour créer une nouvelle équation.
- Entrez l’équation de la courbe de titrage, sous la forme : Y =(Smax-Smin)*((X+R+K)-sqrt((X+R+K)^2-4*R*X)) /(2*R) + Smin + B * X dans la feuille de gabarit nouvellement ouvert, avec Y étant la valeur du signal S, X étant la concentration de ligand ajouté L , R étant la concentration d’immobilisé récepteur, K étant la constante de dissociation, et B en arrière-plan de pente. Définir les contraintes appropriées, telles que K > 0 et R > 0.
Remarque : Cette équation est la même équation de l' équation 9 sous une forme différente. - Analyser les valeurs de la feuille de données en choisissant l’équation définie par l’utilisateur qui a été nouvellement créée. Ouvrez la table avec les valeurs calculées de rapprochement (K, R,t, Smax, Sminet B) qui sont présentés sous la rubrique résultat sur le côté gauche de l’écran du logiciel.
Remarque : Le logiciel détermine les 5 paramètres en ajustant itératif de la régression non linéaire seulement si la courbe de titrage se compose de données au moins 5 points. - Évaluer les paramètres K, R,t, Smax, Sminet B pour chaque groupe de courbes de titrage statistiquement et établir une corrélation entre les paramètres grâce à la fonction spécifique du groupe (ligand muté ou modifiés chimiquement ou récepteur).
Access restricted. Please log in or start a trial to view this content.
Résultats
Après le test ELISA a été mis au point, la couleur jaune du substrat phosphatase alcaline converti, para- nitrophénolate, indique que le montant du ligand lié rhodocetin diminue avec la baisse des concentrations de rhodocetin supplémentaire de colonnes 1 à 11 (Figure 1). Les puits incolores dans les puits rhodocetin-libre, dans la colonne 12 montrent un signal de faible bruit de fond.
Access restricted. Please log in or start a trial to view this content.
Discussion
Le titrage ELISA est un système polyvalent de test afin de déterminer la dissociation d’une interaction de récepteur-ligand. Comme le titrage ELISA contourne la nécessité de distinguer nettement les ligands libres et liées et d’analyser leurs concentrations quantitativement, sensiblement plus d’études et de publications ont employé le titrage ELISA au lieu d’enregistrer des courbes de liaison . En outre, titrage ELISA sont faciles à effectuer et ont besoin de quantités relativement basses des récepteur...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
L’auteur n’a rien à divulguer.
Remerciements
Le protocole et l’algorithme ont été développés au sein d’un projet financé par la Deutsche Forschungsgemeinschaft (DFG grant SFB1009 A09 et EB177/13-1). L’auteur remercie Barbara Schedding et Felix Schmalbein pour le support technique et Dr Niland pour la lecture critique du manuscrit.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| TRIS | neoFrox | 1125KG001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| NaCl | Applichem | 1,316,591,214 | |
| MgCl2 | Merck | 172571 | |
| integrin a2A, wild-type and mutant, recombinant | isolated in author's lab | ||
| NiNTA superflow column | Qiagen, Germany | 30821 | |
| Coomassie-Brilliant Blue R250 | Serva | 35050 | |
| bicinchoninic acid assay (BCA), protein concentration determination kit | Fisher Scientific | 23225 | |
| bovine serum albumine (BSA), fraction V | Applichem | A1391 | |
| 25 % solution of glutaraldehyde | Merck | 354400 | |
| anti-rabbit immunglobulin-antibodies from goat, conjugated with alkaline phosphatase | Sigma-Aldrich | A9919 | |
| Glycine | Applichem | A1377 | |
| Zn(II)-acetate | Applichem | A4324 | |
| NaOH | Applichem | A1551 | |
| Alkaline phosphatase substrate tablet (5 mg) | Sigma-Aldrich | S0942 | |
| Costar half-area microtiter plate | Thermo Scientific | Corning 3690 | |
| micro reaction tubes | Eppendorf | 30120086 | |
| Microplate ELISA reader | BioTek | Synergy HT |
Références
- Klotz, I. M. The application of the law of mass action to binding by proteins; interactions with calcium. Arch Biochem. 9, 109-117 (1946).
- Klotz, I. M. Ligand-receptor complexes: origin and development of the concept. J Biol Chem. 279 (1), 1-12 (2004).
- Bisswanger, H. Ch. 1: Multiple Equilibria, Principles and Derivations. Enzyme Kinetics: Principles and Methods. , Wiley VCH Verlag GmbH. 1-26 (2017).
- Shimura, K., Kasai, K. Affinity gel titration: quantitative analysis of the binding equilibrium between immobilized protein and free ligand by a continuous titration procedure. Anal Biochem. 149 (2), 369-378 (1985).
- Gong, M., Nikcevic, I., Wehmeyer, K. R., Limbach, P. A., Heineman, W. R. Protein-aptamer binding studies using microchip affinity capillary electrophoresis. Electrophoresis. 29 (7), 1415-1422 (2008).
- Hanes, M. S., Ratcliff, K., Marqusee, S., Handel, T. M. Protein-protein binding affinities by pulse proteolysis: application to TEM-1/BLIP protein complexes. Protein Sci. 19 (10), 1996-2000 (2010).
- Latour, R. A. The Langmuir isotherm: a commonly applied but misleading approach for the analysis of protein adsorption behavior. J Biomed Mater Res A. 103 (3), 949-958 (2015).
- Stockell, A. The binding of diphosphopyridine nucleotide by yeast glyceraldehyde-3-phosphate dehydrogenase. J Biol Chem. 234 (5), 1286-1292 (1959).
- Heyn, M. P., Weischet, W. O. Circular dichroism and fluorescence studies on the binding of ligands to the α subunit of tryptophan synthase. Biochem. 14 (13), 2962-2968 (1975).
- Campbell, I. D., Humphries, M. J. Integrin structure, activation, and interactions. Cold Spring Harb Perspect Biol. 3 (3), (2011).
- Zeltz, C., Gullberg, D. The integrin-collagen connection--a glue for tissue repair? J Cell Sci. 129 (4), 653-664 (2016).
- Kanchanawong, P., et al. Nanoscale architecture of integrin-based cell adhesions. Nature. 468 (7323), 580-584 (2010).
- Luo, B. H., Carman, C. V., Springer, T. A. Structural basis of integrin regulation and signaling. Annu Rev Immunol. 25, 619-647 (2007).
- Luo, B. H., Springer, T. A. Integrin structures and conformational signaling. Curr Opin Cell Biol. 18 (5), 579-586 (2006).
- Ricard-Blum, S. The collagen family. Cold Spring Harb Perspect Biol. 3 (1), a004978(2011).
- Eble, J. A., Tuckwell, D. S. The α2β1 integrin inhibitor rhodocetin binds to the A-domain of the integrin α2 subunit proximal to the collagen-binding site. Biochem J. 376 (Pt 1), 77-85 (2003).
- Eble, J. A., et al. The α2β1 integrin-specific antagonist rhodocetin is a cruciform, heterotetrameric molecule. FASEB J. 23 (9), 2917-2927 (2009).
- Eble, J. A., et al. Dramatic and concerted conformational changes enable rhodocetin to block α2β1 integrin selectively. PLoS Biol. 15 (7), e2001492(2017).
- Bracht, T., Figueiredo de Rezende, F., Stetefeld, J., Sorokin, L. M., Eble, J. A. Monoclonal antibodies reveal the alteration of the rhodocetin structure upon α2β1 integrin binding. Biochem J. 440 (1), 1-11 (2011).
- Harlow, E., Lane, D. Chapter 5: Immunizations. Antibodies, a laboratory manual. 5, Cold Spring Harbor Laboratory. 53-138 (1988).
- Lu, D. Analyzing interactions between SSB and proteins by the use of fluorescence anisotropy. Methods Mol Biol. 922, 155-159 (2012).
- Fielding, L. NMR methods for the determination of protein-ligand dissociation constants. Curr Top Med Chem. 3 (1), 39-53 (2003).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr Opin Struct Biol. 11 (5), 560-566 (2001).
- McDonnell, J. M. Surface plasmon resonance: towards an understanding of the mechanisms of biological molecular recognition. Curr Opin Chem Biol. 5 (5), 572-577 (2001).
- Rich, R. L., Myszka, D. G. Advances in surface plasmon resonance biosensor analysis. Curr Opin Biotechnol. 11 (1), 54-61 (2000).
- Pesquero, N. C., et al. Real-time monitoring and kinetic parameter estimation of the affinity interaction of jArtinM and rArtinM with peroxidase glycoprotein by the electrogravimetric technique. Biosens Bioelectron. 26 (1), 36-42 (2010).
- Scheuermann, T. H., Padrick, S. B., Gardner, K. H., Brautigam, C. A. On the acquisition and analysis of microscale thermophoresis data. Anal Biochem. 496, 79-93 (2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon