Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Sélection de plusieurs sous-ensembles de biomarqueur de même Classification binaire efficace performances
Dans cet article
Résumé
Les algorithmes existants génèrent une seule solution pour un dataset de détection des biomarqueurs. Ce protocole ne démontre l’existence de plusieurs solutions de même efficaces et présente un logiciel convivial pour aider les chercheurs biomédicaux à enquêter sur leurs ensembles de données pour le défi proposé. Informaticiens peuvent également fournir cette fonctionnalité dans leur biomarqueur des algorithmes de détection.
Résumé
Détection des biomarqueurs est l’une des plus importantes questions biomédicales pour les chercheurs de haut-débit « omiques », et presque tous les algorithmes de détection des biomarqueurs existants génèrent un sous-ensemble de biomarqueurs à la mesure des performances optimisées pour un dataset donné . Cependant, une récente étude a démontré l’existence de plusieurs sous-ensembles de biomarqueurs avec des représentations de la classification de la même façon efficace ou même identiques. Ce protocole présente une méthodologie simple et directe pour détecter des sous-ensembles de biomarqueurs avec des performances de classification binaire, mieux qu’un seuil défini par l’utilisateur. Le protocole se compose de préparation des données et chargement, synthèse d’information base, paramètre tuning, dépistage de biomarqueurs, visualisation du résultat et interprétation, biomarqueur gène annotations et exportation de résultat et de la visualisation à qualité de publication. Le projet biomarqueur stratégie de dépistage est intuitif et illustre une règle générale pour le développement d’algorithmes de détection des biomarqueurs. Une facile à utiliser interface graphique (GUI) a été développée en utilisant le langage de programmation Python, permettant ainsi aux chercheurs biomédicaux d’avoir un accès direct à leurs résultats. Le manuel de kSolutionVis et le code source peuvent être téléchargés de http://www.healthinformaticslab.org/supp/resources.php.
Introduction
La classification binaire, un des plus couramment étudiée et exploration des problèmes dans le domaine biomédical, de données difficiles est utilisé pour générer un modèle de classification formé sur deux groupes d’échantillons avec la plus exacte la discrimination puissance1, 2 , 3 , 4 , 5 , 6 , 7. Toutefois, les grandes données générées dans le domaine biomédical ont l’inhérente « grand p petit n » paradigme, avec le nombre de fonctions généralement beaucoup plus grandes que le nombre d’échantillons de8,6,9. Par conséquent, chercheurs biomédicaux doivent réduire la dimension de l’élément avant d’utiliser les algorithmes de classement afin d’éviter le surapprentissage problème8,9. Biomarqueurs de diagnostic sont définies comme un sous-ensemble de fonctionnalités détectées séparant les patients d’une maladie donnée de contrôle sain échantillons10,11. Les patients sont généralement définis comme les échantillons positifs, et les contrôles sains sont définis comme les échantillons négatifs12.
Des études récentes ont suggéré qu’il existe plus d’une solution avec des performances identiques ou de même efficace de classification d’un ensemble de données biomédicales5. Presque tous les algorithmes de sélection de fonctionnalité sont les algorithmes déterministes, ne produisant qu’une seule solution pour le même groupe de données. Algorithmes génétiques peuvent générer simultanément des solutions multiples avec des performances similaires, mais ils essaient toujours de choisir une solution avec la meilleure fonction de remise en forme comme sortie pour un dataset donné13,14.
Algorithmes de sélection de fonction peuvent être regroupés à peu près comme les filtres ou les wrappers12. Un algorithme de filtrage choisit lesk caractéristiques de haut - rang par leur importante association particulière avec les étiquettes de classe binaire fondée sur l’hypothèse que les caractéristiques sont indépendants des uns des autres15,16,17 . Bien que cette hypothèse ne tient-elle pas vraie pour presque tous les ensembles de données réelles, la règle de filtrage heuristique effectue bien dans de nombreux cas, par exemple, l’algorithme de Lagarde (redondance Minimum et Maximum de pertinence), le test basé de Wilcoxon fonctionnalité filtrage (WRank) l’algorithme et l’intrigue ROC (Receiver fonctionnement characteristic) basent algorithme de filtrage (ROCRank). Lagarde, est un algorithme de filtrage efficace parce qu’il rapproche du problème combinatoire d’estimation avec une série de problèmes beaucoup plus petites, en comparant à l’algorithme de sélection de fonctionnalité maximum et de dépendance, dont chacun comporte seulement deux variables, et utilise donc des probabilités mixte par paires qui sont plus robustes18,19. Cependant, mRMR peut-être sous-estimer l’utilité de certaines fonctions, comme il ne mesure pas les interactions entre les éléments qui peuvent augmenter la pertinence et donc ne manque certaines combinaisons de fonctionnalités qui sont individuellement inutile mais sont utiles uniquement lorsqu’il est combiné. L’algorithme de WRank calcule un score non paramétrique de comment discriminatoire une caractéristique se situe entre deux catégories d’échantillons et est connue pour sa robustesse pour les valeurs aberrantes20,21. En outre, l’algorithme ROCRank évalue quelle est l’importance de la zone sous le ROC courbe (AUC) d’une fonctionnalité particulière est pour la classification binaire enquête performance22,23.
En revanche, un wrapper évalue le rendement du classifieur prédéfinis d’un sous-ensemble de la fonctionnalité donnée, itérativement généré par une règle heuristique et crée le sous-ensemble de la fonctionnalité avec le meilleur rendement mesure24. Généralement, un wrapper surpasse un filtre dans l’exercice de classification mais s’exécute plus lentement25. Par exemple, l’algorithme de27 26,forêt aléatoire régularisé (RRF) utilise une règle gourmande, en évaluant les fonctionnalités sur un sous-ensemble des données d’entraînement à chaque nœud de la forêt au hasard, dont scores d’importance caractéristique sont évalués par l’indice de Gini . Le choix d’une nouvelle fonctionnalité sera pénalisé si son gain d’information ne s’améliore pas que des caractéristiques choisies. En outre, l’analyse de prédiction pour Microarrays (PAM)28,29 algorithme, aussi un algorithme de wrapper, calcule un centroïde pour chacune des étiquettes de classe et sélectionne ensuite les fonctions à rétrécir les centroïdes de gène vers l’ensemble barycentre de la classe. PAM est robuste pour les fonctionnalités communes.
Des solutions multiples avec la performance de classification supérieure peuvent être nécessaires pour n’importe quel dataset donné. Tout d’abord, l’objectif d’optimisation d’un algorithme déterministe est définie par une formule mathématique, par exemple, erreur minimum taux30, qui n’est pas forcément idéal pour les échantillons biologiques. Deuxièmement, un groupe de données peut avoir des solutions sensiblement différentes, multiple, avec des performances similaires efficaces ou même identiques. Presque tous les algorithmes de sélection de fonctionnalité existante sélectionnera au hasard une de ces solutions comme la sortie31.
Cette étude mettra en place un protocole analytique informatique pour générer des solutions multiples de sélection de fonctionnalité avec des performances similaires pour n’importe quel dataset donné classification binaire. Considérant que les chercheurs biomédicaux plus ne sont pas familiers avec les techniques de l’informatiques ou le codage informatique, une facile à utiliser interface graphique (GUI) a été développée pour faciliter l’analyse rapide des ensembles de données biomédicales classification binaire. Le protocole analytique se compose de chargement des données et résumant, le réglage de paramètre, exécution du pipeline et interprétations du résultat. D’un simple clic, le chercheur est capable de générer des sous-ensembles de biomarqueurs et visualisation de qualité publication parcelles. Le protocole a été testé en utilisant les transcriptions des deux datasets classification binaire des leucémie lymphoblastique aiguë (ALL), c'est-à-dire, APE1 et les212. Les ensembles de données d’APE1 et les2 ont été téléchargés à partir du Broad Institute du génome Data Analysis Center, disponible à http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi. APE1 contient 128 échantillons avec 12 625 caractéristiques. Ces échantillons, 95 sont des lymphocytes B tous et 33 sont des lymphocytes T tous. Les2 comprend 100 échantillons avec 12 625 caractéristiques ainsi. Parmi ces échantillons, il y a 65 patients qui ont souffert de rechute et 35 patients qui n’ont pas. APE1 était un dataset simple classification binaire, avec une précision de moins de quatre filtres et quatre enveloppes étant 96,7 % et 6 les algorithmes de sélection de 8 fonctionnalité réalisation 100 %12. Alors que les2 était un ensemble de données plus difficile, avec les algorithmes de sélection de 8 fonctionnalité ci-dessus réalisation pas mieux que 83,7 % précision12. Cette meilleure précision a été réalisée avec 56 caractéristiques détectés par l’algorithme de wrapper, sélection de fonctionnalité axée sur la corrélation (CFS).
Protocole
Remarque : Le protocole suivant décrit les détails de la procédure analytique de l’informatique et des codes des modules principaux. Le système d’analyse automatique a été développé à l’aide de Python version 3.6.0 et les pandas de modules Python, abc, numpy, scipy, sklearn, sys, PyQt5, sys, Lagarde, math et matplotlib. Les matériaux utilisés dans cette étude sont énumérés dans la Table des matières.
1. préparer la matrice de données et étiquettes de classe
- Préparer le fichier de matrice de données comme un fichier délimité par des tabulations ou des virgules de matrice, tel qu’illustré dans la Figure 1 a.
Remarque : Chaque ligne possède toutes les valeurs d’une fonction, et le premier élément est le nom de la fonction. Une caractéristique est un ID de probeset pour le dataset transcriptome microarray-basé ou peut-être une autre valeur ID comme un résidu de cystéine avec sa valeur de méthylation d’un ensemble de données méthylomiques. Chaque colonne donne les valeurs de la fonction d’un échantillon donné, avec le premier élément étant le nom de l’échantillon. Une ligne est divisée en colonnes par une tabulation (Figure 1 b) ou une virgule (Figure 1). Un fichier délimité par des tabulations de matrice est reconnu par le fichier extension .tsv, et un fichier délimité par des virgules de matrice a l’extension .csv. Ce fichier peut être généré en enregistrant une matrice soit comme le format .tsv ou .csv de logiciels tels que Microsoft Excel. La matrice de données peut-être également être générée par ordinateur de codage. - Préparer le fichier de libellés de classe comme un délimité par des tabulations ou des virgules fichier de matrice (Figure 1), similaire au fichier matrice de données.
Remarque : La première colonne donne les noms de l’échantillon, et l’étiquette de la classe de chaque échantillon est indiquée dans la colonne intitulée classe. Compatibilité maximale est considérée dans le processus de codage afin que les colonnes supplémentaires peuvent être ajoutées. Le fichier d’étiquette de classe peut être formaté comme un fichier .tsv ou .csv. Les noms dans la colonne de classe peuvent être des termes, et il peut y avoir plus de deux classes d’échantillons. L’utilisateur peut choisir deux des classes pour l’analyse qui suit.
2. Chargez la matrice de données et étiquettes de classe
- Chargez les étiquettes de matrice et de la classe de données dans le logiciel. Cliquez sur le bouton de la matrice de données charge de choisir le fichier de matrice de données spécifiée par l’utilisateur. Cliquez sur le bouton étiquettes de classe charge de choisir le fichier de libellés de classe correspondante.
Remarque : Après que les deux fichiers sont chargés, kSolutionVis procédera à un écran de routine de la compatibilité entre les deux fichiers. - Résumer les caractéristiques et les échantillons prélevés dans le fichier de matrice de données. Estimer la taille du fichier matrice de données.
- Résumer les échantillons et les classes à partir du fichier d’étiquette de classe. Estimer la taille de l’étiquette du fichier de classe.
- Vérifier si chaque échantillon de la matrice de données possède une étiquette de classe. Résumer les numéros des échantillons avec les étiquettes de classe.
3. résumer et afficher les statistiques de base de l’objet Dataset
- Cliquez sur le bouton résumer, sans n’importe quel mot clé spécifié d’entrée, et le logiciel affichera 20 fonctions indexées et les noms de fonctionnalités correspondantes.
Remarque : Les utilisateurs doivent spécifier le nom de la fonction qu’ils souhaitent trouver pour voir ses statistiques de base et de la répartition de la valeur correspondante parmi tous les échantillons d’entrée. - Fournir un mot clé, par exemple « 1000_at », dans la zone de texte fonction de trouver une caractéristique spécifique à synthétiser. Cliquez sur le bouton résumer pour obtenir les statistiques de base pour cette fonctionnalité donnée.
Remarque : Le mot clé peut apparaître n’importe où dans les noms de fonction cible, faciliter le processus de recherche des utilisateurs. - Cliquez sur le bouton résumer pour trouver plus d’une fonction avec le mot clé donné et puis spécifiez l’ID de fonction unique pour procéder à l’étape précédente de résumer une caractéristique particulière.
4. déterminer les étiquettes de classe et le nombre de fonctions de classement
- Choisit les noms de positif (« P (33) ») et les classes de négatif (« N (95) ») dans les listes déroulantes Classe positifs et Négatifs de classe, comme illustré à la Figure 2 (celle du milieu).
Remarque : Il est suggéré de choisir qu'un dataset classification binaire équilibré, c'est-à-direla différence entre le nombre d’échantillons positifs et négatifs est minime. Le nombre d’échantillons est aussi donné entre parenthèses après le nom de chaque étiquette de classe dans les deux listes déroulantes. - Choisissez 10 comme le nombre de fonctions de classement (paramètre pTopX) dans la zone de liste déroulante Top_X ( ?) pour un écran complet du sous-ensemble de la fonctionnalité.
Remarque : Le logiciel classe automatiquement toutes les caractéristiques de la P-valeur calculée par un test t de chaque fonctionnalité en comparant les catégories positifs et négatifs. Une fonction avec un petit P-valeur a un fort pouvoir mieux discriminant entre les deux classes d’échantillons. Le module de projection globale est par le calcul intensif. Le paramètre pTopX est de 10 par défaut. Les utilisateurs peuvent modifier ce paramètre dans l’ordre de 10 à 50, jusqu'à ce qu’ils trouvent satisfaisant disposent de sous-ensembles avec des performances de bon classement.
5. accordez les paramètres système pour des Performances différentes
- Choisir la mesure du rendement (pMeasurement), à la précision (Acc) dans la liste déroulante Acc/bAcc ( ?) pour le classificateur s├⌐lectionn├⌐ extrême d’apprentissage Machine (ELM). Une autre option de ce paramètre est la mesure de précision équilibrée (bAcc).
Remarque : Laissez TP, FN, TN, et FP le nombre de vrais positifs, faux négatifs, effectivement négatifs et des positifs fausses, respectivement. La mesure de l’Acc est définie comme (TP+TN)/(TP+FN+TN+FP), qui fonctionne le mieux sur un dataset équilibrée6. Mais un classifieur optimisé pour Acc tend à affecter tous les échantillons à la classe négative si le nombre des échantillons négatifs est beaucoup plus grand que celle des critères positifs. Le bAcc est définie comme (Sn + Sp) / 2, où Sn = TP/(TP+FN) et Sp = TN/(TN+FP) sont les tarifs correctement prédits pour la borne positive et négative des échantillons, respectivement. Par conséquent, bAcc normalise les performances de prévision sur les deux classes et peut conduire à une représentation équilibrée de prédiction plus deux classes déséquilibrées. Le CAC est le choix par défaut de pMeasurement. Le logiciel utilise le classificateur ELM par défaut pour calculer les performances de la classification. L’utilisateur peut aussi choisir un classifieur SVM (Support Vector Machine), KNN (plus proche voisin k), arbre de décision ou Naïve Bayes. - Choisissez la valeur seuil 0,70 (paramètre pCutoff) pour la mesure de performances spécifiées dans la boîte d’entrée pCutoff :.
Remarque : Les Acc et bAcc comprise entre 0 et 1, et l’utilisateur peut spécifier une valeur pCutoff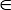 [0, 1] sous le seuil de décision pour afficher les solutions correspondantes. Le logiciel effectue un sous-ensemble-fonctionnalité global de dépistage, et un choix convenable de pCutoff fera la visualisation 3D plus intuitive et plus explicite. La valeur par défaut pour pCutoff est de 0,70.
[0, 1] sous le seuil de décision pour afficher les solutions correspondantes. Le logiciel effectue un sous-ensemble-fonctionnalité global de dépistage, et un choix convenable de pCutoff fera la visualisation 3D plus intuitive et plus explicite. La valeur par défaut pour pCutoff est de 0,70.
6. Exécutez le Pipeline et produire les résultats visualisés INTERACTIVE
- Cliquez sur le bouton Analyze pour exécuter le pipeline et générer les parcelles de visualisation, comme illustré à la Figure 2 (en bas).
Remarque : La table de gauche donne tous les sous-ensembles de fonctionnalité et de leur pMeasurement calculé par la stratégie de validation croisée 10 fois du classifieur ELM, comme décrit plus haut5. Deux parcelles de nuages de points 3D et deux lignes sont générées pour la procédure de présélection du sous-ensemble de la fonctionnalité avec les réglages de paramètre actuels. - Choisissez 0,70 comme la valeur par défaut de la coupure de pMeasurement (paramètre piCutoff, zone de saisie valeur) et 10 comme valeur par défaut du nombre des meilleurs sous-ensembles de fonctionnalité (paramètre piFSNum).
Remarque : Le pipeline est exécuté à l’aide des paramètres pTopX, pMeasurement et pCutoff. La fonctionnalité détectée des sous-ensembles peuvent être encore projeté à l’aide de la coupure piCutoff, cependant piCutoff ne peut pas être inférieure à pCutoff. Par conséquent, piCutoff est initialisé en tant que pCutoff et uniquement les sous-ensembles de fonctionnalité avec la performance mesure ≥ piCutoff vont être visualisés. La valeur par défaut de piCutoff est pCutoff. Parfois kSolutionVis détecte plusieurs solutions et seulement le meilleur piFSNum (par défaut : 10) des sous-ensembles de fonctionnalité vont être visualisés. Si le nombre de sous-ensembles de fonctionnalité détecté par le logiciel est inférieur à piFSNum, tous les sous-ensembles de fonctionnalité vont être visualisés. - Recueillir et interpréter les éléments détectés par le logiciel, tel qu’illustré à la Figure 3.
Remarque : Le tableau dans la boîte de gauche montre les sous-ensembles de fonctionnalité détectés et leur mesure du rendement. Les noms des trois premières colonnes sont « F1 », « F2 » et « F3 ». Les trois caractéristiques à chaque sous-ensemble de la fonctionnalité sont donnés dans leur ordre de classement dans une rangée (F1 < F2 < F3). La dernière colonne donne la mesure du rendement (CAC ou bAcc) de chaque sous-ensemble de la fonctionnalité, et son nom de la colonne (CAC ou bAcc) est la valeur de pMeasurement.
7. interpréter les parcelles du nuage de points 3D-visualiser et interpréter les sous-ensembles de fonctionnalité avec des Performances de Classification binaire de même effectif à l’aide de diagrammes 3D
- Cliquez sur le bouton Analyze pour générer le diagramme 3D des sous-ensembles 10 fonctionnalité albums avec les meilleures performances de classification (CAC ou bAcc) détecté par le logiciel, comme illustré à la Figure 3 (boîte moyenne). Trier les trois caractéristiques d’un sous-ensemble de la fonctionnalité par ordre croissant de leurs rangs et d’utiliser les rangs des trois caractéristiques comme les axes de F1/F2/F3, c'est-à-direF1 < F2 < F3.
Remarque : La couleur d’un point représente le rendement de la classification binaire du sous-ensemble caractéristique correspondante. Un dataset peut avoir plusieurs sous-ensembles de fonctionnalité avec la même manière les mesures de rendement efficace. Un diagramme de dispersion interactive et simplifiée est donc nécessaire. - Remplacez la valeur 0,70 dans la zone de saisie pCutoff : et cliquez sur le bouton Analyze pour générer le nuage de points 3D des sous-ensembles caractéristique avec la performance mesure ≥ piCutoff, comme on le voit à la Figure 3 (case de droite). Cliquez sur le bouton 3D tuning pour ouvrir une nouvelle fenêtre pour ajuster manuellement les angles de vision du nuage de points 3D.
Remarque : Chaque sous-ensemble de la fonctionnalité est représenté par un point de la même manière que ci-dessus. Le nuage de points 3D a été générée en l’angle par défaut. Pour faciliter la visualisation 3D et tuning, une nouvelle fenêtre s’ouvrira en cliquant sur le bouton 3D tuning. - Cliquez sur le bouton réduire pour réduire la redondance des sous-ensembles caractéristique détecté.
Remarque : Si les utilisateurs souhaitent plus choisir les triplets de fonctionnalité et minimiser la redondance des sous-ensembles caractéristique, le logiciel fournit également cette fonction à l’aide de l’algorithme de sélection de fonctionnalité mRMR. Après avoir cliqué sur le bouton réduire , kSolutionVis va supprimer ces fonctionnalités redondantes dans les triplets de fonctionnalité et régénérer la table et les deux diffusent emplacements mentionnés ci-dessus. Les fonctionnalités enlevées des triplets de fonctionnalité seront remplacées par le mot clé dans la table. Les valeurs None dans l’axe de F1/F2/F3 seront établies comme valeur de piFSNum (la plage de la valeur normale de F1/F2/F3 est [1, top_x]). Par conséquent, les points qui incluent une valeur None semble être parcelles points « aberrantes » dans la 3D. On trouvera les parcelles 3D réglables manuellement dans « Réglage manuel des parcelles 3D dot » dans les documents supplémentaires.
8. trouver des Annotations de gène et de leurs Associations avec des maladies humaines
Remarque : Les étapes 8 à 10 illustrera comment annoter un gène au niveau de la séquence de l’ADN et de protéines. Tout d’abord, le symbole de gène de chaque ID de biomarqueurs de l’étapes ci-dessus est récupéré de la base de DAVID32, et puis deux serveurs web représentatif seront utilisés pour analyser ce symbole de gène par rapport aux niveaux de l’ADN et de protéines, respectivement. Le serveur GeneCard fournit une annotation fonctionnelle globale d’un symbole de gène donné et l’hérédité mendélienne en ligne dans la base de données de l’homme (OMIM) fournit la curation plus complet des associations de gène de la maladie. Le serveur UniProtKB est l’un de la plus vaste base de données de protéine, et le serveur du système de prévision par groupe (GPS) prédit la signalisation phosphorylation pour une très grande liste de kinases.
- Copiez et collez le lien web de la base de DAVID dans un navigateur web et ouvrez la page web de cette base de données. Cliquez sur le lien La Conversion génique ID vu dans la Figure 4 a et entrez la fonction IDs 38319_at/38147_at/33238_at du premier sous-ensemble biomarqueur du dataset APE1 (Figure 4 b). Cliquez sur le lien Liste de gène et cliquez sur Soumettre la liste comme illustré dans la Figure 4 b. Récupérer les annotations d’intérêt, puis cliquez sur Afficher la liste de gène (Figure 4). Obtenir la liste des symboles de gène (Figure 4).
NOTE : Les symboles de gène récupérées ici servira pour les autres annotations fonctionnelles dans les prochaines étapes. - Copiez et collez le lien web de la base de données des cartes de gène dans un navigateur web et ouvrez la page web de cette base de données. Rechercher le nom d’un gène CD3D dans la zone d’entrée de requête de base de données et trouver les annotations de ce gène de Gene cartes33,34, comme indiqué dans le tableau 1 et Figure 5 a.
Remarque : Cartes de gène est un gène complet knowledgebase, fournissant nomenclature, génomique, protéomique, localisation sous-cellulaire et voies impliquées et autres modules fonctionnels. Il fournit également des liens externes à diverses autres bases de données biomédicales comme APB/PDB_REDO35, Entrez Gene36, OMIM37et UniProtKB38. Si le nom de la fonction n’est pas un symbole de gène standard, utiliser la base de données ENSEMBL du pour convertir en39. CD3d est le nom du gène T-Cell Receptor T3 Delta Chain. - Copiez et collez le lien web de la base de données OMIM dans un navigateur web et ouvrez la page web de cette base de données. Rechercher le nom d’un gène CD3D et trouver les annotations de ce gène de la base de données OMIM37, comme indiqué dans le tableau 1 et Figure 5 b.
NOTE : OMIM sert maintenant comme une des sources plus complètes et faisant autorités des connexions de gènes humains atteints de maladies héréditaires. OMIM a été initié par le Dr Victor A. McKusick à cataloguer les mutations génétiques associés à la maladie40. OMIM couvre maintenant plus de 15 000 gènes humains et des phénotypes plus 8 500, as of December 1st 2017.
9. annoter les protéines et les Modifications post-traductionnelles
- Copiez et collez le lien web de la base de données UniProtKB dans un navigateur web et ouvrez la page web de cette base de données. Rechercher le nom d’un gène CD3D dans la zone d’entrée de la requête de UniProtKB et de trouver les annotations de ce gène de la base de données38, comme indiqué dans le tableau 1 et Figure 5.
Remarque : UniProtKB rassemble une riche source d’annotations pour les protéines, y compris des informations fonctionnelles et nomenclature. Cette base de données fournit également des liens externes vers d’autres bases de données largement utilisées, y compris APB/PDB_REDO35, OMIM37et Pfam41. - Copiez et collez le lien web du serveur web GPS dans un navigateur web et ouvrez la page web de ce serveur web. Récupérer la séquence de la protéine codée par le gène biomarqueur CD3D de la base de données de UniProtKB38 et prédire les résidus de modification post-traductionnelle (PTM) de la protéine à l’aide de l’outil en ligne de GPS, comme le montre le tableau 1 et Figure 5.
NOTE : Un système biologique est dynamique et complexe, et les bases de données existantes recueillent seulement les renseignements connus. Par conséquent, les outils en ligne de prédiction biomédicale ainsi que des programmes en mode hors connexion peuvent fournir une preuve utile pour compléter un mécanisme hypothétique. GPS a été développé et amélioré pour plus de 12 ans7,42 et peut être utilisé pour prédire les résidus de PTM une protéine sur un peptide donné ordre43,44. Des outils sont également disponibles pour les divers sujets de recherche, y compris la prédiction d’une protéine localisation subcellulaire45 et transcription factor binding motifs 46 parmi d’autres.
10. annoter des Interactions protéine-protéine et leurs Modules fonctionnels enrichis
- Copiez et collez le lien web du serveur web String dans un navigateur web et ouvrez la page web de ce serveur web. Recherchez dans la liste pour les gènes CD3D et P53 et trouver leurs propriétés orchestrées à l’aide de la base de données chaîne47. La même procédure peut effectuer à l’aide d’un autre serveur web, DAVID32.
Remarque : Outre les annotations susmentionnées de gènes individuels, il y a beaucoup d’outils informatique à grande échelle disponible pour étudier les propriétés d’un groupe de gènes. Une étude récente a démontré que les gènes marqueurs individuellement mauvais pourraient constituer un set de gène grandement améliorée5. Par conséquent, il convient des coûts informatiques pour dépister les biomarqueurs plus compliqués. La base de données String peut visualiser les connexions des interactions connues ou prévues, et le serveur David peut détecter les modules fonctionnels avec un phénotype-associations significatives dans les gènes interrogé47,32. Plusieurs autres outils d’analyse informatique à grande échelle sont également disponibles.
11. exporter les sous-ensembles de biomarqueurs généré et les parcelles de visualisation
- Exporter les sous-ensembles de biomarqueurs détecté comme un fichier texte .tsv ou .csv pour une analyse plus approfondie. Cliquez sur le bouton Exporter la Table sous la table de tous les sous-ensembles de biomarqueurs détectés et choisir quel format de texte à enregistrer sous.
- Exporter les parcelles de visualisation sous forme de fichier image. Cliquez sur le bouton Enregistrer sous chaque parcelle et choisir quel format d’image à enregistrer en tant que.
Remarque : Le logiciel supporte le pixel format .png et le vecteur format .svg. Les images de pixels sont bonnes pour l’affichage sur l’écran de l’ordinateur, tandis que les images vectorielles peuvent être converties à toute résolution nécessaire aux fins de publication journal.
Résultats
L’objectif de ce flux de travail (Figure 6) est de détecter des sous-ensembles de biomarqueurs multiples avec une efficacité similaire pour un dataset de classification binaire. L’ensemble du processus est illustré par deux ensembles de données exemple APE1 et les2 extraites d’une détection des biomarqueurs publié récemment étudier12,48. Un utilisateur peut installer kSolutionVis en suiv...
Discussion
Cette étude présente un protocole de détection et la caractérisation de facile-à-suivre la multi-solution biomarqueurs pour un dataset de classification binaire spécifié par l’utilisateur. Le logiciel met l’accent sur la convivialité et les interfaces souples import/export pour différents formats de fichiers, permettant à un chercheur en sciences biomédicales d’enquêter sur l’ensemble de leur données facilement à l’aide de l’interface graphique du logiciel. Cette étude souligne également la n?...
Déclarations de divulgation
Nous n’avons aucun conflit d’intérêt concernant ce rapport.
Remerciements
Ce travail a été soutenu par le programme de recherche stratégique prioritaire de l’Académie chinoise des Sciences (XDB13040400) et la subvention de démarrage de l’Université de Jilin. Évaluateurs anonymes et les utilisateurs de tests biomédicaux ont été appréciés pour leurs commentaires constructifs sur l’amélioration de la convivialité et la fonctionnalité de kSolutionVis.
matériels
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
Références
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon