Method Article
Dissection de la fonction de Enhancer utilisant le Multiplex Enhancer CRISPR axée sur les interférences dans les lignées cellulaires
Dans cet article
Résumé
Ce protocole décrit les étapes nécessaires pour concevoir et effectuer le ciblage multiplexé des rehausseurs avec la protéine de fusion désactivant SID4X-dCas9-KRAB, également connu sous le nom des interférences enhancer (Enhancer-i). Ce protocole permet l’identification des activateurs qui régulent l’expression des gènes et facilite la dissection des relations entre les exhausteurs de réglementer un gène cible commun.
Résumé
Exhausteurs de multiples souvent réglementent un gène donné, mais pour la plupart des gènes, on ne sait quels exhausteurs sont nécessaires à l’expression des gènes, et comment ces exhausteurs se combinent pour produire une réponse transcriptionnelle. Comme des millions de rehausseurs ont été identifiées, des outils de haut-débit sont nécessaires pour déterminer la fonction rehausseur sur une échelle de tout le génome. Les méthodes actuelles pour l’étude de fonction rehausseur comprennent destructions génétiques à l’aide de Cas9 nucléase-compétent, mais il est difficile d’étudier les effets combinatoires d’exhausteurs de multiples à l’aide de cette technique, comme plusieurs lignées clonales successifs doivent être générée. Nous présentons ici Enhancer-i, une méthode axée sur les interférences CRISPR permettant des interrogatoires fonctionnelle d’exhausteurs de multiples simultanément à leurs locus endogènes. Enhancer-i se sert de deux domaines répressifs fusionnées à nucléase-deficient Cas9, SID et KRAB, pour atteindre désactivation enhancer via la désacétylation des histones au locus ciblées. Ce protocole utilise une transfection transitoire du guide ARN pour permettre une inactivation transitoire des régions ciblées et est particulièrement efficace pour blocage inductibles transcriptionnelles réponses aux stimuli dans les milieux de culture de tissus. Enhancer-i est hautement spécifique tant dans le ciblage de génomique et de ses effets sur l’expression génique global. Résultats obtenus par ce protocole aident à comprendre si un renforceur contribue à l’expression des gènes, l’ampleur de la contribution, et comment la contribution est affectée par autres exhausteurs de proximité.
Introduction
À grande échelle, séquençage des projets tels que ENCODE1et2de la feuille de route épigénomique FANTOM3 ont identifié des millions d’exhausteurs putatifs dans le génome humain sur des centaines de types de cellules. On estime que chaque porteur de projet, associé à une moyenne de 4,9 exhausteurs et chaque enhancer contacte en moyenne 2,4 gènes3, ce qui suggère que l’expression des gènes est souvent le résultat de l’intégration des multiples interactions réglementaires distribuées. Un défi de taille restant est de définir non seulement comment les exhausteurs de contribuent à l’expression des gènes, mais comment ils se combinent pour affecter l’expression. Approches génétiques sont couramment utilisés pour identifier les relations entre les rehausseurs en organismes modèles de drosophile4 à5de la souris. Toutefois, ces expériences sont chronophages et faible débit pour l’étude des exhausteurs de plusieurs à plusieurs gènes.
Une approche pour l’étude de fonction rehausseur à grande échelle consiste à reporter massivement parallèle des essais. Ces tests permettent la projection simultanée de milliers de séquences d’ADN pour leur capacité à conduire l’expression d’un gène de journaliste6. Bien que ces essais ont montré que des séquence d’ADN peut suffire seul à transmettre le gène règlement informations7, ils viennent avec les mises en garde d’être exécutée en dehors du contexte de la chromatine native et un promoteur hétérologue. En outre, la taille de la séquence d’ADN analysé lors d’essais de reporter massivement parallèle est généralement inférieure à 200 basepairs, ce qui pourrait exclure la séquence environnante pertinente. Ce qui est important, comme journaliste dosages ne mesurent l’activité d’une séquence à la fois, ils ne tiennent pas compte les relations complexes qui peuvent exister entre les rehausseurs. Ainsi, tout journaliste massivement parallèle des essais peuvent être instructifs sur l’activité intrinsèque d’une séquence d’ADN, ils ne pas forcément nous informer de la fonction de cette séquence d’ADN dans le cadre du génome.
Récemment développés CRISPR/Cas9 outils8 ont facilité l’étude de la régulation des gènes, car ils permettent la suppression des enrichisseurs du locus endogène. Cependant, suppression des exhausteurs de multiples simultanément peut conduire à l’instabilité génomique, et c’est beaucoup de temps pour générer des destructions successives enhancer dans une lignée cellulaire unique. En outre, nouvelle séquence génomique est créé sur le site de la suppression après réparation, et cette séquence peut gagner fonction régulatrice. Une version alternative de Cas9 a été développée spécifiquement pour moduler l’expression des gènes, en s’appuyant sur les fusions d’activation9,10 ou réprimer les domaines12 11,à la forme de nucléase déficients de Cas9 (dCas9). Ces protéines de fusion sont idéales pour l’étude de plusieurs loci simultanément comme ils le ne font pas physiquement modifier la séquence d’ADN et au contraire moduler épigénétique afin d’interroger une région régulatrice. La fusion répressive plus largement utilisée est KRAB, qui recrute le complexe de répresseur co KAP1, promotion de la déposition de l’histone H3 lysine 9 triméthylation (H3K9me3)13associée à la répression. dCas9-KRAB, également connu sous le nom CRISPR interférence14, a été utilisé pour cible et écran exhausteurs individuels pour leurs contributions à l’expression de gène15,16; Cependant, il n’a pas été optimisée pour cibler des régions multiples simultanément. Une seule version de multiplex interférence CRISPR enrichisseurs, mosaïque-seq17, utilise seule cellule RNA-seq comme une lecture, mais cette technologie est coûteux et convient uniquement pour l’étude des gènes fortement exprimés en raison de la faible sensibilité de la cellule unique RNA-Seq.
Nous avons cherché à élaborer une méthode axée sur les interférences de CRISPR pour disséquer la fonction rehausseur combinatoire dans le cadre d’une réponse transcriptionnelle à l’oestrogène. Environ la moitié des gènes sensibles aux oestrogènes contiennent 2 ou plusieurs exhausteurs liés par œstrogènes alpha du récepteur (ER) est proche de18, suggérant que les exhausteurs de multiples peuvent être participant à la réponse de l’oestrogène et comprendre que la logique de réglementation exigerait ciblage des exhausteurs de multiples simultanément. Comme les études initiales à l’aide de brouillage CRISPR aux promoteurs suggèrent que pas tous les promoteurs sont également sensibles aux répression médiée par KRAB19, nous avons pensé que l’ajout d’un domaine répressif distinct à dCas9 peut faciliter la désactivation de exhausteurs de diverses. Nous avons choisi le Sin3a interagissant domaine de Mad1 (SID)20 qui mène au recrutement des histones désacétylases21, qui éliminent les groupes d’acétyle sur les histones qui sont associés à l’activité transcriptionnelle. Important, le domaine SID était efficace pour réduire l’expression de gène lorsqu’elle est fondue à dCas922 et23de la contes et Sin3a s’est avéré être un co-facteur répressif puissant dans une variété de renforceur séquence contextes24. Nous avons utilisé des SID4x-dCas9-KRAB (Enhancer-i) afin de cibler les 10 exhausteurs différents liés par l’ER et identifier les sites de fixation de ER (ERBS) qui sont nécessaires à la réponse transcriptionnelle oestrogène à 4 gènes18. Aussi, nous avons ciblé les combinaisons des rehausseurs pour identifier les sites qui coopèrent dans la production de la réponse transcriptionnelle de l’oestrogène. Nous avons constaté que jusqu'à 50 sites peuvent potentiellement être ciblés en même temps avec les modifications de l’expression génique détectable. À l’aide de ChIP-seq et RNA-seq, nous avons démontré que Enhancer-i est une technique très spécifique pour l’étude des exhausteurs de multiples simultanément.
Dans ce protocole, nous décrivons les étapes impliquées dans l’exécution de Enhancer-i, une technique flexible qui permet l’étude fonctionnelle des exhausteurs de multiples simultanément dans un milieu de culture de tissus. Enhancer-i est fortement corrélé avec délétion génétique mais fournit désactivation transitoire qui est tributaire des histones désacétylases (HDAC). En livrant guide ARN par transfection transitoire, par opposition à l’intégration stable par l’intermédiaire de vecteurs viraux, ce protocole évite les dépôts et la propagation potentielle de H3K9me3. Ce modèle de guide RNA de détails de protocole et le clonage par assemblage de Gibson, la transfection de guideront RNAs utilisant lipofection et l’analyse de l’expression génique qui en résulte transforme par qPCR. Nous incluons également les méthodes d’évaluation de la spécificité de Enhancer-i ciblage au niveau du génome et du transcriptome. Tandis que cette technique a été développée afin d’étudier la régulation génique par ER lié exhausteurs en lignées cellulaires cancéreuses humaines, elle s’applique à la dissection de n’importe quel mammifère enhancer.
Protocole
1. génération de cellule lignes exprimant de manière stable SID4X-dCas9-KRAB
Remarque : Les conditions de transfection et les concentrations de médicament présentées ici ont été optimisées pour les cellules Ishikawa, une lignée de cellules de cancer de l’endomètre, cultivées dans un milieu RPMI 1640 additionné de 10 % SVF et 1 % la pénicilline/streptomycine (RPMI complet). Autres lignées cellulaires peuvent exiger des conditions différentes de transfection et concentrations du médicament. Aussi les utilisateurs peuvent effectuer des expériences de transfection transitoire dans des cellules de type sauvage, au lieu de générer une lignée cellulaire stable, avec un plasmide exprimant SID4X-dCas9-KRAB avec guide RNA exprimant des plasmides ; Cependant, les résultats de transfections transitoires peuvent être difficiles à reproduire comme SID4X-dCas9-KRAB niveaux peuvent varier par transfection.
- Plaque de cellules Ishikawa au moins 2 puits d’une plaque de 6 puits au confluent de 30 à 50 % (environ 300 000 cellules Ishikawa) dans 3 mL de milieu RPMI complet.
- Aspirer les médias des cellules. Laver les cellules une fois avec du PBS 1 x (pH 7,4). Aspirer le PBS et ajouter la trypsine (4 mL pour un plat de 10 cm ou 5 mL pour un flacon de T-75).
- Incuber les cellules pendant ~ 5 min à 37 ° C, vérifiant toutes les 2 min pour les cellules individuelles et secouant doucement le navire.
- Une fois que les cellules ont détaché, pipette de trypsinisées cellules de haut en bas plusieurs fois et pipette doucement sur le côté du navire pour libérer les cellules attachées.
- Transférer les cellules dans un tube conique de 15 mL et faire tourner les cellules vers le bas pendant 5 min à 250 g.
- Aspirer la trypsine et remettre en suspension les cellules de 5 à 10 mL de médias. Utiliser une pipette P1000 de dissocier les amas de cellules si nécessaire.
- Compter les cellules et déterminer le volume nécessaire pour plaque ~ 300 000 cellules / puits dans un volume total de 3 mL. Ajouter des cellules à 2 puits distincts d’une plaque de 6 puits. Remplir chaque bien à 3 mL avec RPMI complet.
- Secouez légèrement la plaque toutes les 5 min dans les 15 premières minutes après l’électrodéposition pour faire en sorte que les cellules sont réparties uniformément sur la plaque. Utiliser un microscope pour s’assurer que les cellules ont dispersé au milieu du puits.
- Dans les 24h de l’électrodéposition, effectuer les suivantes transfections, utilisant un réactif de transfection appropriée pour la lignée de cellules d’intérêt. Pour les cellules Ishikawa, utilisez la procédure décrite ci-dessous.
Remarque : Ce protocole suppose l’utilisation de réactifs de transfection d’axée sur les liposomes cationiques. Électroporation fournit une méthode alternative pour les types de cellules qui sont très sensibles à ces réactifs ou qui présentent des efficacité de transfection faible avec lipofection. Conditions de transfection doivent être optimisées pour la lignée cellulaire d’intérêt avant de tenter des expériences de Enhancer-i.- Dans un tube Eppendorf de 1,7 mL, diluer 2,5 μg de plasmide SID4X-dCas9-KRAB et 800 ng de plasmide exprimant une protéine fluorescente dans un milieu sans sérum, tel que le volume final du tube soit 155 µL et la concentration finale de plasmide est 0,020 µg/µL.
- Dans un autre tube, diluer 3,3 µg d’un plasmide qui ne contient pas une cassette de résistance de néomycine, tels que CMVp-GFP, dans un milieu sans sérum de sorte que le dernier volume du tube est 155 µL et la concentration finale de plasmide est 0,020 µg/µL.
- Vortex chaque tube brièvement et filer vers le bas à l’aide d’un microtube.
- Ajouter 9,9 µL de réactif de la transfection (Table des matières) dans chaque tube. Mélanger au vortex brièvement à basse vitesse. Faites tourner les tubes vers le bas avec un microtube.
- Incuber les tubes à température ambiante pendant au moins 5 min, mais pas plus de 20 min.
- Dans la prévention des risques biotechnologiques armoire, ajouter 150 µL du mélange préparé ADN : réactif goutte à un puits sur la plaque de 6 puits. Répétez pour l’autre tube de mélange de l’ADN : réactif préparé. Mélanger les plaques en agitant doucement et remettez la plaque dans l’incubateur.
- Transfection de jour 2 de post, changer les médias et compléter avec G418 à une concentration finale de 600 ng/μl. Cette concentration doive être optimisé pour le type de cellule.
- Modifier les médias RPMI et compléter avec G418 tous les deux jours pendant 2 à 4 semaines jusqu'à ce que les cellules contrôle transfectée sont morts et les puits contenant des SID4X-dCas9-KRAB deviennent confluentes. Le montant exact du temps nécessaire pour récupérer des cellules dépendra le temps de doublement des cellules.
- Lorsque les cellules devenus confluentes, passage à un navire T-25 ou T-75, en remplissez RPMI avec une dose plus faible de G418 (300 ng/μl de cellules Ishikawa). Lors de ce passage, que 2 parties aliquotes de ~ 100 000 cellules chaque (environ 1/10ème d’une plaque de 6 puits) dans 2 tubes Eppendorf de distinct 1,7 mL pour isoler l’ARN et ADN, respectivement. Tourner ces tubes vers le bas (5 min, 250 g de x), de supprimer la trypsine en pipettant également et geler les tubes à-20 ° C pour une utilisation future.
- Isoler l’ADN génomique à l’aide de kits disponibles dans le commerce et effectuer PCR à l’aide de la « pAC95_PCR » ou « SID4X_PCR » amorces (tableau 1) pour vérifier la présence de la protéine de fusion au sein de la lignée cellulaire. Utiliser l’ADN génomique extrait de la lignée parentale comme témoin négatif et l’ADN de plasmide SID4x-dCas9-KRAB comme témoin positif. Utilisez un mélange maître polymérase haute fidélité avec 50 à 100 ng d’ADN génomique et les conditions suivantes de cyclisme : 98 ° C pendant 30 s, 25 cycles de (98 ° C pendant 10 s, 58 ° C pendant 30 s, 72 ° C pendant 2 min), 72 ° C pendant 5 min , tenir à 4 ° C.
- Pour vérifier que l’expression de la protéine de fusion au niveau du RNA, effectuez qPCR avec de l’ARN extrait de la lignée cellulaire utilisant des kits disponibles dans le commerce. Utilisez les amorces de « dCas9_qPCR » (tableau 1) et le protocole de qPCR one-step fourni à l’étape 6.3 du présent protocole.
- Pour vérifier que l’expression de la protéine au niveau de la fusion, effectuez un Western blot sur des lysats de la lignée cellulaire. Utilisez Anti-Flag ou anti-HA anticorps pour détecter la protéine de fusion.
2. guide RNA Design
Remarque : Ce protocole est conçu pour une utilisation avec l’ARN guide de U6 clonage vector créé par le laboratoire de l’église et disponible sur Addgene (Addgene 41824). Pour créer une version de ce vecteur contenant résistance puromycine qui permettait à la même stratégie de clonage comme 41824, nous avons déménagé le site multiple de clonage de ce vecteur dans le vecteur pGL3-U6-sgRNA-PGK-puromycine (Addgene 51133). Addgene 41824 ou notre version avec puromycine (Addgene 106404) sont compatibles avec la stratégie de clonage décrite ci-dessous.
- Obtenir 600-900 basepairs de séquence d’ADN pour chaque région régulatrice d’intérêt. Utiliser les sites de fixation de facteurs de transcription et/ou accessibilité de la chromatine d’orientation pour savoir où définir la zone d’intérêt (Figure 2 a).
Remarque : Alors que l’exemple de la Figure 2 a dispose d’exhausteurs en amont et en aval, il est également possible de cibler les éléments régulateurs agrave introns. - Placez toutes les séquences obtenues dans un fichier texte simple au format FASTA.
- Indiquez au moins une région de contrôle négatif qui ne devrait pas changer au fil des conditions expérimentales, telles qu’un promoteur d’un gène qui n’est pas exprimé dans la lignée de cellules d’intérêt. Obtenir la séquence d’ADN pour cette région et l’ajouter au fichier texte au format FASTA.
Remarque : Nous utilisons guide RNAs ciblant les IL1RN promoteur25 comme témoin négatif pour toutes les régions que nous ciblons. Aussi, les utilisateurs peuvent sélectionner des séquences intergéniques près de la région d’intérêt qui ne contient-elle pas de sites de fixation de facteurs de transcription comme témoin négatif. Toutefois, si plusieurs loci est ciblés en même temps, une région unique témoin négatif simplifie la conception expérimentale et l’interprétation des résultats. Si l’amplificateur ciblée est intronic, il peut être utile pour cibler une région intronic au même locus qui ne contient pas un élément régulateur présumé comme un contrôle négatif supplémentaire, comme la fusion de dCas9 peut-être interférer avec la transcription. - Identifier les régions de contrôle positif, tels que les promoteurs qui sont les cibles présumées des régions d’intérêt réglementaires ou promoteurs de gènes qui sont transcrits très dans la lignée de cellules d’intérêt. Obtenir la séquence d’ADN pour ces régions et ajoutez-le au fichier texte au format FASTA.
- Utilisez un programme tel que e-crisp26 (http://www.e-crisp.org/E-CRISP/) sur les séquences d’ADN générés pour trouver le guide ARN avec faibles hors-cibles (idéalement 0-3). ARN guide se composent de 20 nucléotides en amont d’un motif adjacent de protospacer (PAM), qui prend la forme « NGG » pour le dCas9 de S. pyogenes.
- Sur le site e-crisp, sélectionnez l’organisme d’intérêt en utilisant le menu déroulant. L’Assemblée de génome apparaît à droite du nom de l’espèce.
- Sélectionnez la case d’entrée est la séquence FASTA . Copiez les séquences FASTA d’en haut et les coller dans la boîte de dialogue. Veiller à ce qu’un en-tête FASTA est inclus pour chaque séquence.
Remarque : Jusqu'à 50 séquences peuvent être interrogées en même temps. - Dans le menu déroulant, sélectionnez la case d’option moyen et la conception unique .
- Cliquez sur le bouton lancer la recherche de sgRNA. Ouvrira un nouvel onglet du navigateur, et les résultats seront affichés. Télécharger les séquences de candidat en cliquant sur le bouton Télécharger un rapport sous forme de tableau Excel au format pour toutes les séquences de requête ensemble.
- Ouvrez le rapport sous forme de tableau à l’aide d’Excel ou un programme d’édition de texte.
- Utilisez le navigateur de génome UCSC BLAT candidat gRNA pleine longueur des séquences (23 basepairs) au génome.
- Dans un navigateur, accédez au site UCSC genome browser (http://genome.ucsc.edu). Dans la section nos outils, recherchez le mot BLAT et cliquez dessus. L’outil de recherche BLAT s’ouvrira.
- Utilisez les menus déroulants situés sous le texte BLAT recherche génome pour sélectionner l’organisme et de génome d’intérêt.
- Copiez les séquences d’ARN guide depuis le rapport tabulaire généré par e-croquants et les coller dans la boîte de dialogue. Veiller à ce que chaque séquence possède un en-tête FASTA unique, puis cliquez sur soumettre au bas de la boîte de dialogue.
Remarque : Jusqu'à 25 séquences peuvent être examinés en même temps. Sur la page de Résultats de recherche BLAT , alignements de chaque guide séquence d’ARN apparaîtra, avec chaque ligne représentant un alignement. Idéalement, il faudrait un alignement pour chaque guide RNA, indiquant le caractère unique de ce guide RNA. - Éviter les guides qui correspondent à plusieurs endroits dans le génome si possible.
- Pour examiner le guide RNA localization et la distribution dans la région d’intérêt, cliquez sur le lien de navigateur en vertu de la section ACTIONS pour l’un de l’ARN guide interrogé. Le navigateur de génome apparaîtra et sera centré sur le guide sélectionné RNA. Utilisez les boutons de Zoom arrière en haut de la page pour visualiser la distribution des autre guide RNAs identifiés par e-crisp au sein de la région d’intérêt.
- Sélectionnez 4 préférence non-cumul guide séquences d’ARN qui sont distribués dans toute la région d’intérêt (Figure 2 b). Si la région d’intérêt dépasse 600 bp, pensez à ajouter 1 à 2 guides supplémentaires. Évitez les guide ARN avec des étirements de homopolymeric et de contenu en GC extrême, car ces fonctionnalités peuvent entraver l’ARN guide processus de clonage et réduire le guide RNA, ciblant l’efficacité.
- Une fois que les guides ont été sélectionnés, créez un fichier contenant la séquence d’ARN (23 nucléotides) le guide complet pour chaque guide désiré et puis retirez le nucléotide 5' ainsi que le PAM (NGG) de l’extrémité 3'. Cette étape facilite oligo vous passez votre commande.
- Ajouter la séquence suivante à l’extrémité 5' de la séquence : GTGGAAAGGACGAAACACCG.
- Ajouter la séquence suivante à l’extrémité 3' de la séquence : GTTTTAGAGCTAGAAATAGC.
Remarque : La séquence finale devrait être 59 nucléotides longs et regardez comme ceci : GTGGAAAGGACGAAACACCG-cible (19 nt)-GTTTTAGAGCTAGAAATAGC. - Veiller à ce que chaque élément réglementaire ciblé avec Enhancer-i possède au moins 4 oligonucléotides uniques conçus pour lui. Commander ces séquences ainsi que les amorces de « U6_internal » figurant au tableau 1.
3. guide RNA clonage
Remarque : Guide RNA clonage via l’Assemblée Gibson s’est avéré très efficace dans nos mains, ce qui donne des centaines de colonies par boîte, avec peu ou pas de colonies présentes dans le contrôle de vecteur seul. Cette efficacité est essentielle pour le maintien de complexité au cours de la mise en commun de clonage. Un autre avantage de l’Assemblée de Gibson clonage est que les utilisateurs n’ont pas à s’inquiéter de la présence d’une enzyme de restriction coupée le site dans le guide RNA ils essaient d’insérer dans le vecteur de clonage U6. Néanmoins, le présent protocole peut être adapté pour traditionnel enzyme de restriction fondé clonage si vous le souhaitez.
- Reconstituer le guide RNA oligos à une concentration finale de 100 μM de l’eau ultrapure (RNase-libre, sans DNase). Il devrait y avoir au moins 4 guide séparé RNA oligos pour chacune des régions d’intérêt.
- Pour chaque région régulatrice d’intérêt, créer un bassin de tous les oligos correspondant à la zone d’intérêt. Dans un tube Eppendorf, mélanger 5 μL de chaque guide reconstitué individuels RNA oligo pour chaque région. La piscine bien mélanger en Vortex, puis enlever 1 μL et diluer cette 1 : 200 aliquote dans l’eau ultrapure.
Remarque : Si vous le souhaitez, ces piscines ciblant les régions régulatrices individuelles peuvent être combinés pour produire une complexe piscine cibler des régions multiples. Jusqu'à 50 régions régulatrices peuvent viser simultanément dans un pool unique (Figure 3). - Effectuer une courte PCR avec les amorces U6 pour joindre les régions d’homologie aux oligos avant le montage de Gibson. Environ 40 bases seront ajoutés à chaque oligo, ce qui donne un produit bp 100 ~ qui contient suffisamment d’homologie avec le vecteur U6 aux deux extrémités.
- Pour chaque guide piscine RNA, mis en place un 20 μL PCR avec un mélange maître polymérase de haute fidélité et les composants suivants : 1 μL de piscine oligo dilué de l’étape 3.2, 1 μL d’apprêt avant de U6 (10 μM), 1 μL de U6 reverse primer (10 μM) et l’eau jusqu'à 20 μL.
- Incuber dans un thermocycleur dans les conditions suivantes : 98 ° C pendant 30 s, 10 cycles de (98 ° C pendant 10 s, 55 ° C pendant 30 s, 72 ° C pendant 2 min), 72 ° C pendant 5 min et maintenez à 4 ° C.
- Exécuter 5 μL de la réaction sur un gel d’agarose de 1 à 2 % avec une échelle de faible poids moléculaire. Le produit final devrait être basepairs ~ 100 (Figure 2).
- Nettoyer la réaction d’extension avec un kit de purification de l’ADN basée sur les colonnes et éluer à 20 μL de tampon d’élution fourni dans le kit.
Remarque : Comme le produit est court, évitez d’utiliser axée sur la perle nettoyages, qui visent à exclure les petits fragments de moins de 100 bp. - Quantifier l’ADN purifié à l’aide d’un fluorimètre ou spectrophotomètre (attendu le rendement est de 10-20 ng/μL). Guide RNA insertions peuvent être conservées à-20 ° C, ou peuvent être utilisées immédiatement dans l’Assemblée de Gibson avec un vecteur linéarisé de U6.
- Pour préparer le destinataire U6 vecteur de clonage pour l’assemblage de Gibson, mis en place un "Digest" de l’enzyme de restriction. Si de nombreuses réactions de l’Assemblée de Gibson sont à effectuer, mis en place plusieurs recueils pour assurer le rendement suffisant du vecteur coupé.
- Utilisez 20 unités de AflII enzyme et 1 μg de plasmide dans une réaction de 20 μL avec le tampon de l’enzyme de restriction appropriées. Incuber à 37 ° C pendant 1 à 2 h.
- Nettoyer le condensé de perles ou un kit basée sur les colonnes pour la purification de l’ADN et éluer à 20 μL de tampon d’élution. Quantifier l’ADN purifié à l’aide d’un fluorimètre ou un spectrophotomètre. Les échantillons peuvent être congelés à-20 ° C pour une utilisation ultérieure.
- Effectuer l’Assemblée Gibson sur le vecteur préparé et insérer.
- Mettre en place les réactions de l’Assemblée de Gibson sur la glace. Utilisation 50 ng du vecteur et 7 ng de l’insert dans une réaction de 20 μL. Diluer les insertions 01:10 dans de l’eau ultrapure pour faciliter le pipetage. Mettre en place une vecteur seul Gibson réaction Assemblée, à l’aide de 50 ng du vecteur et de remplacement de l’insert avec de l’eau.
- Incuber les réactions de l’Assemblée de Gibson pendant 15 min à 50 ° C, suivie d’une prise à 4 ° C.
- Transférer les produits assemblés à la glace. Diluer les produits assemblés 1 / 4 dans l’eau ultrapure sur la glace. Par exemple, ajouter 5 μl de produit de l’Assemblée de Gibson à 15 μL d’eau ultrapure.
- Transformer les produits d’assemblage Gibson dilués.
- Décongeler les cellules compétentes à haut rendement sur la glace et faire 25 μL parties aliquotes pour chaque transformation. Si vous désirez une piscine complexe ciblant plusieurs sites, décongeler assez de cellules dans des tubes différents pour effectuer des transformations multiples indépendantes du même pool gRNA complexes.
- Pour chaque produit dilué, ajouter 1 μL de cette dilution dans un 1,7 mL tube Eppendorf contenant 25 μL de cellules compétentes. Mélanger en effleurant brièvement le tube. Incuber les tubes sur la glace pendant 30 min.
- Chaleur de choc les cellules pendant 30 s à 42 ° C, puis transférer immédiatement à la glace pendant 2 min.
- Ajouter des éléments multimédias SOC 300 μL (tryptone de 2 %, 0,5 % d’extrait de levure, 10 mM NaCl, KCl, 2,5 mM 10 mM MgCl2, 20 mM et 10 mM MgSO4glucose) et laisser les cellules récupérer pendant 1 h à 37 ° C sous agitation (300 tr/min). Pendant ce temps, chauffer les plaques de gélose avec ampicilline/carbénicilline à 37 ° C dans un incubateur. Utiliser une assiette pour chaque transformation.
- Plaque 50 μL de cellules et placer les plaques dans un incubateur à 37 ° C pendant la nuit. Pour les piscines ciblant les sites individuels, directement dans les 3 à 5 mL de bouillon LB (Table des matières) contenant l’ampicilline/carbénicilline (1 mg/mL) et incuber pendant la nuit avec la secousse à 250 tr/min à 37 ° C pendant minipreps.
- Récolter les cellules et isoler l’ADN.
- Pour les bibliothèques de RNA grand guide ciblant plusieurs sites, utilisez un grattoir plaque pour recueillir toutes les colonies de chaque plaque individuelle dans un maxiprep (culture liquide 150 mL). Cela peut être facilité par verser environ 5 mL de LB avec l’antibiotique approprié dans un tube falcon de 50 mL et en raclant les colonies dans le tube. Pour les bibliothèques ciblant les sites individuels, racler les plaques dans un miniprep (culture liquide de 3 à 5 mL).
- Incuber ces cultures avec l’antibiotique approprié pendant 3-5 h à 37 ° C sous agitation à 250 tr/min.
- Effectuer l’extraction d’ADN à l’aide d’un kit qui se traduit par preps exempte d’endotoxine.
- Quantifier l’ADN à l’aide d’un fluorimètre ou un spectrophotomètre. Plasmides peuvent être utilisés immédiatement dans la transfection ou stockés à-20 ° C pour une utilisation future.
- Pour confirmer la présence d’une séquence d’ARN guide dans le vecteur U6 pour petites piscines ciblant des sites simples, utilisez Sanger séquençage sur la miniprep préparé avec l’amorce de la « U6_PCR_R » figurant au tableau 1. En raison de la mise en commun du guide RNAs, la séquence cible de 19 régions gRNA procurera les bases mixtes, mais le promoteur U6 et l’échafaudage de RNA guide entourant cette séquence doivent être intacts.
4. la transfection de Enhancer-i
Remarque : Pour le blocus réussi d’une réponse d’oestrogène avec Enhancer-i dans les cellules Ishikawa, il est nécessaire de priver les cellules de l’oestrogène pour 5-7 jours avant la transfection par leur maintien dans le rouge de phénol RPMI gratuit avec 10 % charbon-dépouillé SVF et 1 % la pénicilline/streptomycine. Cellules doivent être cultivées dans ce média pendant et après la transfection si essaie de bloquer une réponse de l’oestrogène. Nous recommandons l’utilisation de rouge de phénol trypsine libre pour passage des cellules dans le rouge de phénol complet gratuitement RPMI.
- La veille de la transfection, plaque les cellules (sauvage ou exprimant de manière stable SID4X-dCas9-KRAB) dans une plaque 24 puits à la confluence de 30 à 50 % (~ 60 000 cellules / puits pour cellules Ishikawa). Plaque de suffisamment de cellules telles que transfections peuvent être effectuées en double et comprennent des puits à transfecter avec guide de contrôle RNAs. Veiller à ce que les cellules sont uniformément répartis dans le puits en le secouant doucement la plaque après le placage de la cellule comme au point 1.1.7.
Remarque : Ce protocole suppose l’utilisation de réactifs de transfection d’axée sur les liposomes cationiques. Électroporation fournit une méthode alternative pour les types de cellules qui sont très sensibles à ces réactifs. Conditions de transfection doivent être optimisées pour la lignée cellulaire d’intérêt avant de tenter des expériences de Enhancer-i. - Le lendemain, préparer des transfections selon les instructions du réactif transfection de choix. Pour les cellules Ishikawa, utilisation 550 ng de plasmide total pour chaque puits d’une plaque 24 puits. Diluer les plasmides à une concentration finale de 0,020 μg/μL dans un milieu sans sérum (1,1 μg d’ADN dans le 52 μL de volume total de transfection 2 puits). Utiliser 3 μl de réactif de transfection pour chaque 1 μg d’ADN, vortex et incuber tel que décrit à l’étape 1.2. Ajouter 25 μl du mélange final dans chaque puits.
Remarque : À des combinaisons de cible des sites, utilisez le même poids de plasmide pour chaque site et remplissez le poids reste avec un plasmide contrôle (ARN guide vide clonage vecteur ou guide RNAs visant une région témoin négatif comme IL1RN promoteur). Pour transfections transitoires, utiliser un ratio de plasmide de RNA guide : fusion 3:2 Cas9. Plasmides contenant des reporters fluorescents peuvent être ajoutés pour surveiller l’efficacité de transfection. - À la transfection de 36 h de post, changer les médias à l’aide de rouge de phénol et la fourniture de puromycine gratuite RPMI avec 10 % charbon-dépouillé SVF et 1 % la pénicilline/streptomycine (pour les cellules Ishikawa) (concentration finale : 1 μg/mL) et la néomycine (concentration finale : 300 ng/mL). Si les cellules sont sensibles au réactif de transfection, les médias peuvent être modifiés plus tôt, mais antibiotiques devraient être ajoutés pas avant la transfection post 24h.
Remarque : Attendez au moins 24 heures après l’ajout d’antibiotique avant la récolte des cellules. Modifications de l’expression en raison de Enhancer-i peuvent être détectées tôt que 48 h après transfection et jusqu'à 5 jours après la transfection. Si vous travaillez avec cellules Ishikawa qui ont été privés d’oestrogène, effectuer une induction 10 nM 17β-estradiol (E2) de 8 h le lendemain de l’antibiothérapie et puis récolter les cellules immédiatement.
5. récolte et Extraction de l’ARN des cellules
- Préparer le tampon de lyse avec 1 % de β-mercaptoéthanol (BME). Veiller à ce qu’il y a assez mélange de lyse-BME (300 μL de chaque bien être récoltées).
- Aspirer les médias à l’aide d’un aspirateur sous vide.
- Laver les cellules une fois avec un volume égal de PBS 1 x (500 μL) et aspirer pour retirer autant de PBS que possible.
- Ajouter 300 μl de solution de lyse-BME dans chaque puits à l’aide d’une pipette multicanaux. La solution de lyse, déposer vers le haut et vers le bas de 8 - 10 fois et le transfert d’une plaque de fond de puits ou 1,7 mL Eppendorf tubes sur la glace. L’ARN peut être extrait immédiatement, ou lysats peuvent être congelés à-80 ° C pour le futur traitement.
- Pour extraire les RNA de lysats, utiliser un kit disponible dans le commerce qui inclut un traitement de DNase. Éluer dans le plus petit volume conseillé de l’eau ultrapure (RNase-libre, sans DNase) ou élution tampon et quantifier des RNA. Pour un petit nombre d’échantillons, utiliser un fluorimètre ou un spectrophotomètre. Pour un grand nombre d’échantillons, utiliser une sonde fluorescente qui détecte l’ARN et mesure sur un lecteur de plaque. Les échantillons peuvent être congelés à-80 ° C avant ou après la quantification.
6. quantification des modifications de l’Expression génique en utilisant une étape qPCR et RNA-seq
- Obtenir des amorces de qPCR pour les gènes d’intérêt et gène de ménage au moins un qui est exprimé près du niveau des gènes ciblés et ne change pas dans des conditions expérimentales. Idéalement, ces amorces seront étendra sur une jonction exon-exon pour éviter l’amplification de l’ADN génomique.
- Tester ces amorces d’ARN provenant de la lignée cellulaire d’intérêt. Utilisation de fonte analyse des courbes de vérifier la production d’un produit unique. Si un seul produit n’est pas produit, tester des paires d’amorces supplémentaires.
- Pour chaque guide RNA traité échantillon Enhancer-i et de contrôle, indiquer combien de gènes doivent être dosés dans cet échantillon. Cet ensemble de gènes devrait inclure des gènes domestiques, tels que le FCCT ou GAPDH.
- Mettre en place des réactions de qPCR.
- Diluer les échantillons à la même concentration dans l’eau, tels que 50 ng d’ARN total est facilement éjectant et il est suffisamment dilué RNA pour chaque réaction. Par exemple, diluer RNA à ~16.6 ng/μL et utiliser 3 μL d’ARN dans chaque réaction. Gardez RNA sur glace tout en créant des mélanges maîtres.
- Préparer des mélanges maîtres distincts pour chaque gène à mesurer à l’aide de kits de qPCR commercialement disponible en une seule étape. Utiliser un volume de réaction 20 μL avec 1 μL de chaque amorce (solution mère de 10 μM). Mettre en place ces réactions sur la glace.
- Dans une plaque de réaction qui est appropriée pour le thermocycleur, ajoutez des échantillons d’ARN suivies de mélanges maîtres. Sceller avec un film adhésif et mélanger doucement à l’agitation ou de pipetage. Centrifuger brièvement la plaque (140 x g pendant 60 s) pour s’assurer que le liquide est au fond des puits.
- Incuber la plaque dans un thermocycleur comme suit (ou en kit instruit) : 48 ° C pendant 30 min, 95 ° C pendant 10 min, 40 cycles de (95 ° C pendant 15 s, 60 ° C pendant 1 min).
- Obtenir les valeurs de Ct pour chaque gène mesurées dans chaque échantillon. Utilisez la méthode comparative de Ct pour identifier les modifications dans l’expression des gènes.
- Soustraire le Ct de la gène de ménage de la Ct de chaque gène d’intérêt pour chaque échantillon pour générer des valeurs normalisées de Ct.
- Pour les échantillons de contrôle traité, prendre une moyenne des valeurs Ct normalisées pour chaque gène. Log base 2-échelle pli répression peut ensuite être calculée en soustrayant l’échantillon normalisée de Enhancer-i traité Ct pour chaque gène provenant de la même valeur pour l’échantillon de contrôle traité pour le gène correspondant.
- Pour déterminer les changements globaux dans l’expression des gènes après traitement Enhancer-i, préparer les échantillons pour le séquençage de RNA en utilisant un kit disponible dans le commerce compatible avec la technologie de séquençage de l’utilisateur. Utilisation ~ 500 ng d’ARN pour le matériel de départ et de préparer des bibliothèques au moins 2 réplicats biologiques.
7. la vérification d’un ciblage spécifique génomiques par à l’aide de la SID4X-dCas9-KRAB ChIP-seq
Remarque : La protéine de fusion SID4X-dCas9-KRAB contient une balise épitope de drapeau et une balise d’épitope HA, mais les meilleurs résultats pour ChIP-seq ont été obtenus avec des anticorps anti-drapeau. Si désiré, l’utilisateur peut effectuer des expériences additionnelles de ChIP-seq pour des facteurs de transcription potentiellement touchés par Enhancer-i, ou H3K27ac, une marque d’activité enhancer qui est diminuée de Enhancer-i. Toutefois, chaque expérience de ChIP-seq nécessite 10 x 106 cellules, donc plan en conséquence.
- Transfecter cellules avec piscines Enhancer-i.
- Plaque de 10 x 106 cellules dans un plat de culture de tissu de 15 cm. Chaque plat représente 1 expérience de ChIP-seq pour 1 facteur d’intérêt.
- Le lendemain, transfecter les cellules à l’aide de 20 μg d’ADN total par plat. Pour transfections dans des lignées de cellules exprimant de manière stable SID4X-dCas9-KRAB, l’ADN doit être un pool de plasmide du guide RNAs ciblant tous les sites d’intérêt et éventuellement un plasmide exprimant la protéine fluorescente. Pour transfections transitoires, utiliser un ratio de 3:2 dCas9 pool de RNA de fusion protéine : guide. Pour ChIP-seq d’autres TFs ou modifications d’histone, effectuez au moins une transfection de guide RNA un contrôle supplémentaire sur un autre plat.
- Traiter les plats avec la puromycine (1 μg/mL) et la néomycine (300 ng/mL) à la transfection post 24-48 h. Attendre au moins 24h avant la récolte de la chromatine.
- Récolter la chromatine de vaisselle.
Remarque : Pour étudier les effets de Enhancer-i sur la liaison génomique ER dans les cellules Ishikawa, effectuer un traitement de nM E2 1 h 10 sur la vaisselle transfectées avec contrôle guide RNAs et prieur de Enhancer-i à la récolte. Pour étudier les effets de Enhancer-i sur H3K27ac, effectuez un 8 h 10 nM E2 induction sur plats transfectées avec contrôle guide RNAs et prieur de Enhancer-i à la récolte.- Appliquer 500 μl de 37 % de formaldéhyde à chaque plat (concentration finale de 1 %). Agiter les plaques brièvement. Laisser les plaques à température ambiante pendant 10 min.
- Ajouter 1 mL de glycine de 2,5 M (concentration finale de 125 mM). Agiter les plaques brièvement.
- Décanter les médias avec le formaldéhyde et glycine. Ajouter un volume égal (environ 20 mL) de PBS 1 x contre le rhume.
- Décanter le PBS. Aspirer avec un aspirateur vide pour enlever autant de PBS que possible. Placer les plats sur la glace.
- Ajouter 3 à 5 mL de froid 1 x tampon de lyse PBS ou Farnham (pH de tuyaux de 5mM 8,0, 85 mM KCl, 0,5 % NP-40) avec 1 x inhibiteur de la protéase (ajouté juste avant utilisation) pour chaque plaque. Grattez le plat avec un grattoir plaque et transvaser la solution dans un tube conique 15 mL sur la glace.
- La chromatine de granule de filer vers le bas des tubes dans une centrifugeuse pendant 5 min à 4 ° C à 1000 x g. jeter le surnageant et stocker les pellets à-80 ° C pour une utilisation future ou procéder à du protocole ChIP-seq de choix à l’aide d’un anticorps anti-drapeau ou anticorps ciblant l’autre facteurs de transcription ou des modifications d’histone d’intérêt (H3K27ac, H3K9me3).
Résultats
La figure 1 présente un schéma du flux de travail décrit dans le protocole. Pour déterminer les contributions d’ER-lié aux exhausteurs près du gène oestrogène-régis MMP17, qui dispose de 3 sites de liaison à proximité telles que définies par ChIP-seq (Figure 2 a), guident les ARN ont été conçus pour chaque région. Pour concevoir le guide RNAs, une fenêtre de bp 600-900 de séquence entourant chaque ER accepteur d’intérêt a été sélectionné et mis dans un programme de conception de RNA guide. Ce qui guide RNA sequences avec 0-2 prévues éloignent sites étaient alignés sur le génome humain à l’aide de BLAT. Non-cumul quatre guide ARN qui s’étend sur la zone définie par ChIP-seq et DNase i hypersensibilité ont été choisis pour le ciblage (Figure 2 b). Une séquence additionnelle (tableau 1) a été ajoutée à chaque extrémité pour faciliter le clonage en aval et le résultant 59 fragments de nucléotides ont été commandés. À l’arrivée, guide ARN ont été dilués et regroupées par site et une courte PCR a été effectuée pour ajouter des régions d’homologie avant le montage de Gibson. Figure 2 montre le produit de RNA guide attendu après une courte PCR utilisant les amorces de « U6_internal » (tableau 1), ce qui ajouteront 20 basepairs de séquence à chaque extrémité de la régions 59 guide fragment de RNA, résultant en une séquence de ~ 100 régions. Suite à l’Assemblée de Gibson, ces piscines guide RNA ont été transformées en bactéries et plasmide minipreps ont été préparés le jour suivant. Figure 2D montre des résultats à partir d’une expérience de dissection enhancer, où plusieurs exhausteurs est proche de MMP17 sont ciblés seul ou en combinaison avec Enhancer-i. Sites ciblés par Enhancer-i sont indiquées par un hexagone noir. Guide RNA plasmides ciblant les sites indiqués ont été transfectés dans une lignée de cellules de Ishikawa oestrogène privés exprimant de manière stable SID4X-dCas9-KRAB. Deux jours plus tard, les médias a été changé et la puromycine a été ajoutée pour enrichir pour les cellules transfectées. Le lendemain, les cellules ont été récoltées après un traitement à l’oestradiol 8 h 10 nM. L’ARN a été isolé et un qPCR en une seule étape a été réalisée. Dans cet exemple, les sites 1 et 2 sont nécessaires pour une réponse complète et oestrogénique de MMP17, tandis que site 3 ne contribue pas dans ces conditions (Figure 2D, voies ii-iv). Lorsque les sites uniquement 2 ou 3 sont actifs (vi et vii), la réponse de l’oestrogène est semblable à où aucun site n’est actif (viii), ce qui suggère que ces sites ne peuvent pas contribuer indépendamment. Site 1 peut contribuer quelque expression en elle-même (v), mais la plus grande activité est vu lorsque les sites 1 et 2 sont actives (iv).
Pour manipuler les 10 exhausteurs de près de 4 différents gènes simultanément (Figure 3 a), complexes piscines de guident ARN ont été générés contenant 42 guides enhancer et 16 guides de promoteur. Guide RNA oligos ont été groupés avant le guide initial extension RNA PCR (étape 3.3), et des produits PCR obtenus ont été purifiés et combinés avec le vecteur de clonage U6 puromycine vide à l’aide de l’Assemblée de Gibson. Suite à l’Assemblée de Gibson, plusieurs transformations indépendantes a eu lieu et plaquées. Les plaques ont été grattées dans LB et autorisés à pousser pendant 2-4 h avant maxiprep. Figure 3 b montre des réductions représentante dans l’expression des gènes de qPCR lorsque ces pools de RNA guide ont été transfectés dans une lignée de cellules de Ishikawa oestrogène privés exprimant de manière stable SID4X-dCas9-KRAB et traités comme décrit ci-dessus (Figure 2D). Réductions de Enhancer-i sont semblables à ceux obtenus en ciblant le promoteur du gène cible putative. La figure 3 montre les effets de la dilution du guide RNAs sur la réduction de la réponse d’oestrogène avec Enhancer-i. A 01:50 dilution d’un pool de guide RNA ciblant l’enrichisseur près G0S2 encore conduit à une réduction significative dans l’expression des gènes, ce qui suggère que Enhancer-i peut être utilisé pour cibler jusqu'à 50 sites à la fois. Toutefois, la désactivation peut être diluée, ce qui indique que des centaines de sites ne peut pas être ciblés en même temps à moins que les méthodes de détection plus sensibles sont employés.

Figure 1. Protocole de schéma pour la dissection enhancer multiplex à l’aide de Enhancer-i. ARN Guide (rouge et bleu) est conçus à l’aide de e-crisp et sélectionné en utilisant le navigateur de génome UCSC. Guide de quatre RNAs sont choisis qui enjambent les régions d’intérêt (transcription sites de liaison tel que défini par ChIP-seq). Des oligonucléotides de RNA guide qui ont été mis en commun de la région d’intérêt (rouge et bleu) subissent une PCR pour ajouter des régions d’homologie (orange) avant d’assembler de Gibson et transformation. Piscines de plasmide qui en résultent sont transfectées par lipofection dans des lignées de cellules exprimant de manière stable SID4X-dCas9-KRAB ou dans des cellules de type sauvage en conjonction avec le plasmide SID4X-dCas9-KRAB. Guide RNA plasmide piscines peuvent être transfectées individuellement afin de cibler un site à la fois, ou en combinaison pour cibler plusieurs sites simultanément. Les cellules transfectées sont traités avec des antibiotiques pour enrichir des cellules contenant le guide d’ARN. À la transfection post ~ 72 h, les cellules sont récoltées. Les acides nucléiques peuvent être extraites pour qPCR, RNA-seq ou ChIP-Seq. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
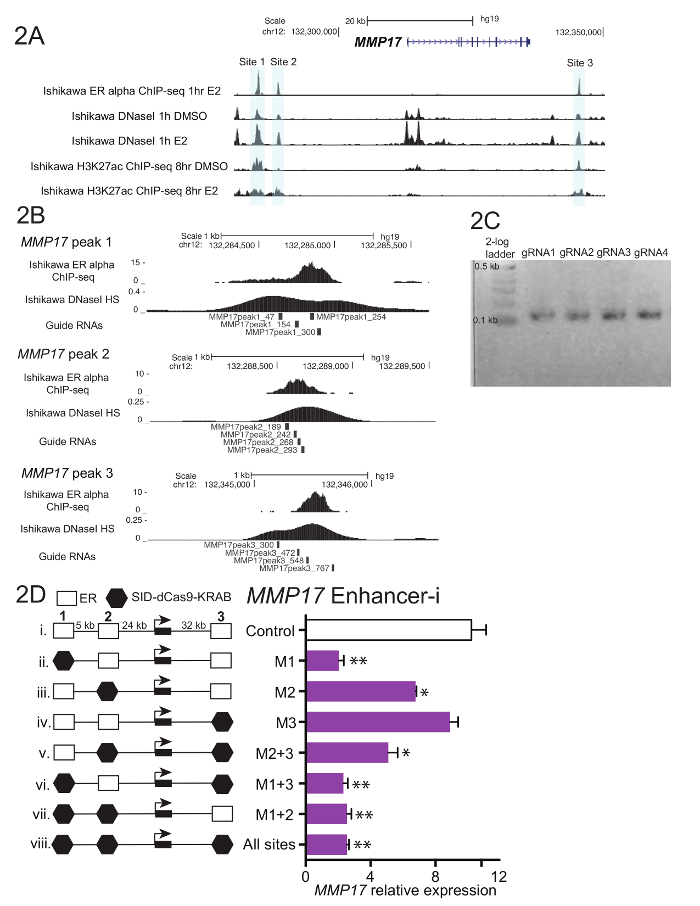
La figure 2. Guide RNA conception et exhausteur de dissection pour MMP17. (A) génome navigateur capture d’écran des exhausteurs d’alpha-bondissent ER (gris) d’être la cible près de MMP17. Ce chiffre a été modifié par Carleton, et al. 18. (B) l’ARN Guide conçoit pour les 3 binding sites18. Le site de liaison pour ER tel que défini par ChIP-seq est la cible et la tuile de RNAs 4 guide dans toute cette région. Le signal de sensibilité de DNase i, qui s’étend sur le site de fixation, permet également de définir la séquence cible pour guide de conception de RNA. ChIP-seq tant les données de la DNase i HS proviennent de cellules Ishikawa traitées avec 10 estradiol nM pour 1 h. (C) guide représentant séquences d’ARN qui sont prêtes pour l’assemblage de Gibson, ayant subi une courte PCR pour ajouter des régions de l’homologie. (D) l’expression Relative de MMP17 mesuré à qPCR après ciblage des régions spécifiques avec Enhancer-i et un traitement à l’oestradiol nM 8-h10. Expression est par rapport au niveau FCCT et l’expression de MMP17 dans les cellules non traitées avec l’estradiol. Guide de contrôle ARN cible le promoteur de la IL1RN. Toutes les barres d’erreur représentent SEM, double astérisque indique p < 0,01 et unique astérisques indiquent p < 0,05 dans un test t apparié. Ce chiffre a été modifié par Carleton, et al. 18. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
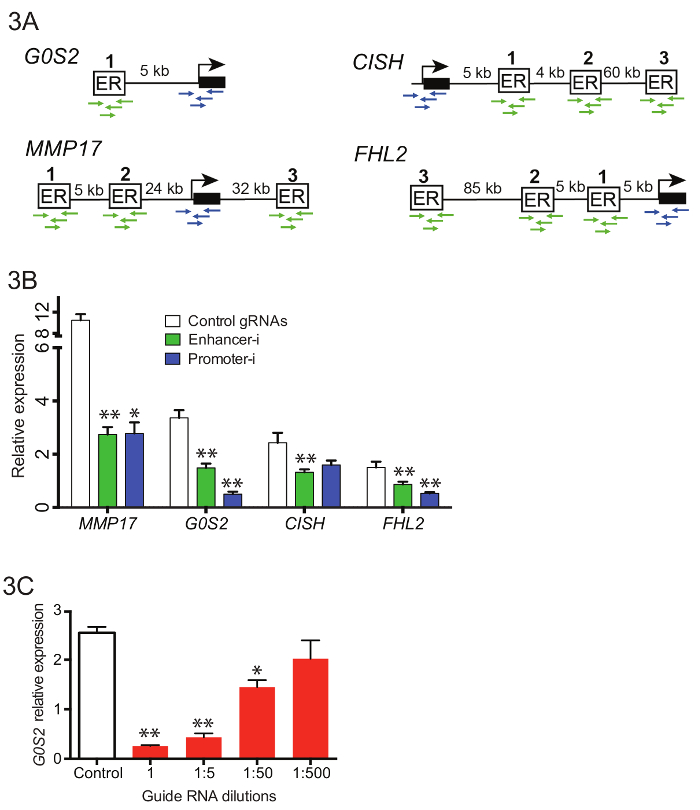
Figure 3. Ciblant plusieurs exhausteurs près de différents gènes simultanément avec mises en commun Enhancer-i. (A) schéma des sites de fixation et promoteurs à viser en commun Enhancer-i. (B) les effets sur l’expression telle que mesurée par qPCR après que traitement de E2 sur Ishikawa cellules transfectées avec piscine de plasmide Enhancer-i (vert), la piscine de plasmide de promoteur-i (bleu) ou contrôle gRNAs (blanc)18. On observe une réduction significative à tous les gènes avec Enhancer-i. Ce chiffre a été modifié de Carleton, et al. 18. (C) les effets sur les niveaux d’expression de G0S2 après le traitement de l’E2 sur Ishikawa cellules transfectées avec différentes quantités de guide RNAs ciblant les G0S2. Une réduction significative peut être vu même avec de petites quantités de guide RNA (01:50 dilution), suggérant que jusqu'à 50 peut ciblé des sites simultanément. Toutes les barres d’erreur représentent SEM, double astérisque indique p < 0,01 et unique astérisques indiquent p < 0,05 dans un test t apparié. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Nom | Séquence |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
Le tableau 1. Amorces utilisées pour guide RNA extension et séquençage, qPCR et détection de la protéine de fusion.
Discussion
Ce protocole décrit une méthode simple et flexible pour disséquer la fonction rehausseur au locus génomique endogène sans altérer physiquement la séquence d’ADN. Bien que similaire au concept des protocoles de brouillage CRISPR précédemment publiés en utilisant dCas9-KRAB27, Enhancer-i diffère de ces protocoles de 3 façons. Tout d’abord, Enhancer-i utilise le domaine interaction SIN3A MAD120 pour atteindre la désactivation de l’enrichisseur. Désactivation de Enhancer peut être sauvée en utilisant des inhibiteurs d’HDAC, suggérant que le principal mécanisme de désactivation est HDAC dépendant. À la différence des interférences CRISPR avec dCas9-KRAB, Enhancer-i ne conduit pas à la déposition de H3K9me3. Cela est probablement dû au fait que Enhancer-i repose sur l’introduction transitoire du guide RNAs, avec les cellules étant récoltées à 3 jours après la transfection. Interférence CRISPR, il est observé une augmentation H3K9me3 à 7 jours post transduction12. Enfin, le protocole Enhancer-i fournit une stratégie permettant de cibler plusieurs sites simultanément et surveiller l’efficacité du ciblage. Mosaïque-seq17, dCas9-KRAB est utilisé pour cibler les exhausteurs de multiples simultanément, mais cette technique repose sur le séquençage de RNA monocellulaires pour identifier les modifications de l’expression, et de nombreux gènes (tels que des gènes sensibles aux oestrogènes) passent inaperçus en raison de la faible sensibilité des unicellulaires RNA-Seq. Enhancer-i fournit une méthode fiable pour étudier les enrichisseurs individuellement ou en combinaison pour n’importe quel gène.
L’étape la plus critique de Enhancer-i est la transfection, qui doit être optimisée pour la lignée de cellules d’intérêt. Ce protocole repose sur le traitement de la puromycine enrichir pour les cellules transfectées, mais il est possible que guide co-transfection ARN avec une protéine fluorescente et le tri des cellules fluorescentes cytométrie peut s’avérer une meilleure méthode d’enrichissement pour quelques cellules types. Il est recommandé de surveiller le niveau d’expression du guide RNAs et SID4x-dCas9-KRAB de qPCR de dépanner et de confirmer la transfection. Si les niveaux indicatifs RNA sont faibles (cycle seuil > 30), les utilisateurs peuvent également envisager alternative guide stratégies de production de RNA tels que in vitro de transcription28. Il est également possible que, malgré un niveau élevé gRNA, guide RNA ciblage de la protéine de SID4x-dCas9-KRAB est inefficace, auquel cas en sélectionnant différents guide de séquences d’ARN peut être nécessaire. En effectuant des ChIP-seq sur la protéine de fusion avec la chromatine des cellules Enhancer-i traité, l’efficacité du ciblage peut être surveillée. Si il y a un signal de SID4x-dCas9-KRAB à la région d’intérêt et aucune modification de l’expression dans son gène cible putatifs n’est détectées, puis la région probablement ne contribue pas à l’expression de ce gène dans les conditions étudiées.
Une des limites possibles de Enhancer-i, c’est que les effets hors cible peuvent s’accumuler si trop de sites sont ciblés en même temps. Néanmoins, stratégies de brouillage CRISPR knockdown ont moins hors effets de la cible que l’ARNi29, particulièrement lorsqu’une lignée de cellules polyclonaux exprimant dCas9-KRAB est utilisée. Alors que nous avons vu hors cible liaison génomique du SID4X-dCas9-KRAB lors du ciblage de 10 sites en même temps, nous n’avons pas identifié modifications de l’expression génique à la suite de ces événements de liaison. Comme certains exhausteurs peuvent communiquer avec plusieurs promoteurs et/ou autres exhausteurs, il est possible que plusieurs gènes peuvent changer expression sur le ciblage d’un renforceur unique, même s’il est difficile de savoir si cette forme de régulation génique est fréquente. Pour confirmer que les modifications de l’expression observées sont dues à cibler un rehausseur spécifique et non hors-cible des effets, les utilisateurs peuvent effectuer Enhancer-i avec deux ensembles distincts de non-cumul guide RNAs ciblant la même région. En outre, la suppression génétique de la région à l’aide de Cas9 nucléase-compétente peut confirmer ses effets sur l’expression génique.
En tant que fonctions Enhancer-i par le biais de désacétylation des histones, il est possible que ses capacités de neutralisation sont limitées aux rehausseurs qui ont des niveaux appréciables d’acétylation des histones. Il y a une variété d’autres fusions répressives qui peut être plus efficace au ciblage spécifiques exhausteurs. Fusions d’ADN méthyltransférase à dCas9 peuvent être utilisées pour réduire l’expression de gène lorsque destiné aux enrichisseurs distale30, mais cette répression n’est pas souvent transitoire. Une autre fusion répressive utilise le domaine de l’ami de GATA1 (FOG1), qui mène de l’histone H3 lysine 27 triméthylation et réprime l’expression des gènes à des niveaux comparables à dCas9-KRAB dans une variété de cellule promoteurs et lignes31. Fait intéressant, ajoutant plus de copies de FOG1 à dCas9 réduit le potentiel répressif aux promoteurs, ce qui suggère qu’une seule copie du domaine SID peut fournir désactivation d’enhancer plus que les 4 copies utilisées actuellement Enhancer-i. Il est possible que certains loci peut-être bénéficier du double ciblage par différentes combinaisons des fusions dCas9 ci-dessus. Par exemple, la répression à long terme stable est possible par transduction simultanée de dCas9-DNMT3a et dCas9-KRAB32. La plupart de ces fusions répressives seulement ont été ciblée à un locus unique à la fois, et on ne sait toujours pas qui est le plus efficace à la manipulation des exhausteurs de multiples simultanément.
Enhancer-i, en une méthode appropriée pour l’étude des combinaisons d’exhausteurs de pour une poignée de gènes, est encore un peu limité en débit si l’utilisateur souhaite étudier les enrichisseurs de présumés pour des centaines de gènes. Futures applications de cette technique intégrera les technologies basées sur l’imagerie afin de quantifier plusieurs gènes dans des échantillons multiples simultanément. Ce qui est important, ces technologies sont compatibles avec la détection directe de molécules d’ARN de lysat, éliminant le besoin d’ARN chronophage. Ces adaptations facilitera l’interrogatoire de plus grands ensembles d’exhausteurs.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par le NIH/NHGRI R00 HG006922 et NIH/NHGRI R01 HG008974 à J.G. et l’Institut de Cancer de Huntsman. J.B.C. était soutenue par le programme de formation du NIH en génétique T32GM007464.
matériels
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
Références
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon