Method Article
Base d’ARN reprogrammation des fibroblastes humains de primaires sur les cellules souches pluripotentes induites
Dans cet article
Résumé
Ici, nous décrivons une méthode cliniquement pertinente, haute efficacité, exempte d’engraissement pour reprogrammer les fibroblastes humains primaires sur les cellules souches pluripotentes induites à l’aide de mis à jour l’ARNm codant pour des facteurs de reprogrammation et mature microARN-367/302 imite. Est également inclus les méthodes pour évaluer l’efficacité de reprogrammation, développez colonies clonales iPSC et confirmer l’expression du marqueur de pluripotence TRA-1-60.
Résumé
Les cellules souches pluripotentes induites (CISP) sont révélés pour être un outil précieux pour étudier les maladies et le développement humain. Encore faire progresser le CISP comme un régénérateur thérapeutique nécessite un coffre fort, robuste et un protocole de reprogrammation opportun. Nous présentons ici un protocole clinique pertinent, étape par étape pour la reprogrammation de très haute efficacité des fibroblastes dermiques humains en CISP en utilisant une approche sans intégration. Le noyau du protocole est constitué d’exprimer les facteurs de pluripotence (SOX2, KLF4, cMYC, LIN28A, NANOG, OCT4-MyoD fusion) de in vitro ARN messagers transcrits synthétisé avec modification nucléotides (mis à jour le mRNA). Les ARNm modifiées reprogrammation sont transfectés dans des fibroblastes primaires toutes les 48 h avec mature imite de microARN-367/302 axée sur les cellules souches embryonnaires pour deux semaines. Les colonies iPSC qui en résulte peuvent alors être isolés et directement étendus dans des conditions sans chargeur. Afin de maximiser l’efficacité et la cohérence de notre protocole de reprogrammation dans les trois échantillons de fibroblastes, nous avons optimisé les divers paramètres, y compris le régime de transfection RNA, calendrier de transfections, conditions de culture et densités de semis. Important, notre méthode génère CISP de haute qualité de la plupart des sources de fibroblastes, y compris les échantillons de malades, âgés ou sénescentes difficile-à-reprogrammer.
Introduction
La reprogrammation de cellules somatiques dans les cellules souches pluripotentes induites (CISP) requiert une expression étendue d’un ensemble de facteurs de transcription qui sont important dans le maintien de pluripotence1,2. Lorsque vous générez le CISP pour des applications cliniques, il est essentiel que la charge mutationnelle dans les cellules d’entrée est réduit au minimum pendant le traitement et l’efficacité de la production de l’iPSC est maintenue à un niveau relativement élevé dans les trois échantillons de patients. Cependant, la majorité de la reprogrammation de méthodes, y compris les protocoles libres intégration, souffre de la très faible efficacité de reprogrammation, quelle utilité clinique limite de ces approches3. La faible efficacité de reprogrammation peut également promouvoir sélective de reprogrammation de cellules porteuses de mutations préexistantes, augmentant la charge mutationnelle dans CISP qui en résulte. En outre, toutes basées sur l’ADN reprogrammation méthodes, telles que des lentivirus et épisomiques-approches axées sur, souffrent de la préoccupation de sécurité que l’ADN peut au hasard intégrer dans le génome et créer la possibilité pour la mutagénèse insertionnelle nuisible et indésirables ( potentiellement oncogène) expression de gènes de pluripotence dans des tissus en aval dérivés4.
Une approche prometteuse pour réaliser induction efficace de la pluripotence des cellules somatiques et réduire la charge mutationnelle dans CISP qui en résulte est d’utiliser ARN de messager plafonnés synthétique contenant des nucléobases (mis à jour le mRNA) pour la reprogrammation5modifiés. L’efficacité des approches reprogrammation mis à jour le mRNA peut être encore améliorée en ajoutant des cellules souches embryonnaires (CSE)-spécifique microARN (miARN)-367/302 s3, qui auraient dû être divulgués pour reprogrammer les cellules somatiques avec a augmenté l’efficacité6 ,,7. Toutefois, même avec l’ajout des miARN-367/302 s, l’approche modifiée reprogrammation axée sur l’ARNm échoue souvent lors de l’application à des cellules fraîchement isolées de patients3. Incohérences d’adresse de cette approche axée sur les ARNm modifiée, nous avons récemment rapporté une stratégie optimisée, exempt d’intégration qui induit la pluripotence dans les fibroblastes humains primaires avec un taux de réussite élevé et utilise les deux mis à jour l’ARNm codant reprogrammation des facteurs et mature miRNA-367/302 imite le8. Dans notre méthode, l’ARNm modifiée reprogrammation cocktail comprend une version modifiée de OCT4 fusionné avec le MyoD transactivation domaine (appelé M3O)9 et cinq autres facteurs de reprogrammation (SOX2, KLF4, cMYC, LIN28A et NANOG). Combinant mis à jour l’ARNm codant des facteurs de pluripotence avec le miRNA imite semblait avoir un effet synergique sur la reprogrammation des gains d’efficience dans le présent protocole. Des optimisations supplémentaires le régime de transfection RNA, cellulaire ensemencement, et des conditions de culture étaient également nécessaires pour accroître l’efficacité de l’approche à un très haut niveau8reprogrammation.
Contrairement à nombreux autres protocoles, notre approche reprogrammation nécessite généralement seulement quelques milliers de fibroblastes d’entrée. En outre, beaucoup sans intégration des stratégies communes à l’aide de plasmides épisomiques, virus Sendai, ou ARN autoreproducteur impliquent une vaste passage pour diluer le vecteur reprogrammation au CISP généré. À l’inverse, mis à jour le mRNA et imite les miARN matures ont une demi-vie courte et est rapidement éliminés des cellules. Pris ensemble, le laps de temps de culture cellulaire cumulative entre la collecte des échantillons de patients et la génération de CISP utilisable est minime dans cette approche, limitant efficacement l’accumulation de mutations dans CISP qui en résulte et améliorer la rentabilité économique.
Nous présentons ici le protocole détaillé étape par étape pour atteindre la haute efficacité reprogrammation des fibroblastes humains adultes en CISP à l’aide de notre approche combinatoire d’ARNm/miRNA-base mis à jour le8. Ce protocole de reprogrammation à base d’ARN fournit une méthode simple, rentable et solide pour générer le CISP exempte d’intégration pour la recherche et le potentiel des applications cliniques. En outre, elle s’applique à la reprogrammation d’une variété de lignées de fibroblastes dont difficile-à-reprogrammer, associés à la maladie, les fibroblastes sénescents et âgés. Un schéma du protocole pour la reprogrammation des fibroblastes humains est illustré à la Figure 1. Le protocole décrit plus précisément une méthode pour la reprogrammation des trois puits de fibroblastes humains de primaires adultes dans une plaque de format 6 puits. Deux puits produisent généralement un nombre suffisant de qualité iPSC colonies. Dans bien des cas, seul est bien nécessaire, et le troisième bien peut être utilisé pour l’analyse de l’efficacité de la reprogrammation. Si nécessaire, le nombre de puits peut être transposé.
Protocole
Travailler dans des conditions sans RNase et utiliser des techniques aseptiques lorsque cela est possible. Effectuer toutes les manipulations liées à la culture de cellule sous une hotte à l’aide de techniques aseptiques de sécurité biologique. Suivre les normes de biosécurité institutionnelle pour le travail avec des cellules humaines.
1. les réactifs et équipements pour la préparation de reprogrammation Initiation
- Préparer les facteurs de reprogrammation encodage cocktails modifié-ARNm.
- Suivre le protocole déjà publiés10 pour effectuer la transcription in vitro , bouchage et déphosphorylation des procédures pour chaque mis à jour l’ARNm codant le facteur individuel de reprogrammation. Après l’étape de purification finale, éluer les ARNm modifiés avec de l’eau exempte de nucléase, compléter la solution purifiée de mRNA modifiés avec 1 inhibiteur de RNase U/µL, doser les ARNm modifiés à l’aide d’un spectrophotomètre et stocker à-80 ° C pendant 6 mois. Préparer plusieurs parties aliquotes de chaque ARNm modifiés pour réduire le nombre de cycles de gel-dégel.
Remarque : Les protocoles similaires pour mis à jour le mRNA production ont été signalés ailleurs5,8,11. Modèles pour la transcription in vitro peuvent être générés par l’amplification par PCR de modèles correspondants de plasmide utilisant les amorces énumérés dans la Table des matières selon le protocole déjà publiés10. Modèles de plasmide pour chaque facteur de reprogrammation (M3O9, SOX2, KLF4, cMYC, NANOG, LIN28A) et mWasabi (contrôle pour la transfection) sont disponibles auprès de Addgene, un référentiel de plasmide à but non lucratif. - Mélanger ensemble tous les ARNm modifiés dans un rapport molaire de 3:1:1:1:1:1 (M3O : SOX2 : KLF4 : cMYC : NANOG : LIN28A) et comprennent 10 % mWasabi modifiée ARNm comme un contrôle pour l’efficacité de transfection. Ajuster la concentration d’ARNm complet mis à jour le reprogrammation mix à une concentration finale de 100 ng/µL par adjonction d’eau exempte de nucléase, additionné de 1 inhibiteur de RNase U/µL. Préparer sept 33 µL d’extraits de la complète modifiée cocktail de l’ARNm. Stocker les aliquotes de reprogrammation mixtes à-80 ° C.
Remarque : pour chaque transfection, 1 000 ng du cocktail mis à jour le mRNA est ajouté par puits (c.-à-d., un total de 3 000 ng pour 3 puits). Chaque µL 33 aliquote est dimensionnée pour transfecter 3 puits d’une plaque de format 6 puits et comprend 3 µL d’excès de volume pour tenir compte des erreurs de pipetage. Préparer sept 33 µL d’extraits est suffisante pour effectuer une reprogrammation complète fibroblaste de 3 puits dans une plaque de format 6 puits.
- Suivre le protocole déjà publiés10 pour effectuer la transcription in vitro , bouchage et déphosphorylation des procédures pour chaque mis à jour l’ARNm codant le facteur individuel de reprogrammation. Après l’étape de purification finale, éluer les ARNm modifiés avec de l’eau exempte de nucléase, compléter la solution purifiée de mRNA modifiés avec 1 inhibiteur de RNase U/µL, doser les ARNm modifiés à l’aide d’un spectrophotomètre et stocker à-80 ° C pendant 6 mois. Préparer plusieurs parties aliquotes de chaque ARNm modifiés pour réduire le nombre de cycles de gel-dégel.
- Préparer le cocktail de miRNA imite la reprogrammation.
- Imitations de miRNA dissoudre lyophilisé (Syn-a-miR - 302 a - 3P, Syn-a-miR - 302 b - 3P, Syn-a-miR - 302c - 3P, Syn-a-miR-302d-3 p, Syn-a-miR-367-3 p) à une concentration finale de 5 pmol/µL (5 µM) dans de l’eau exempte de nucléase additionné de 1 U/µL d’inhibiteur de RNase. Préparer plusieurs parties aliquotes de chaque imitateur de miRNA et stocker à-80 ° C pour un stockage prolongé.
- Mélanger tous les imitateurs de miRNA dans un rapport molaire de 1:1:1:1:1 à une concentration finale de 5 pmol/µL (5 µM). Préparer sept 14 µL d’extraits de la miRNA imite mix. Stocker les aliquotes mixtes à-80 ° C.
Remarque : Pour chaque transfection, 20 pmol du miRNA imite mix est ajoutée par puits (c.-à-d., un total de 60 pmol pour 3 puits). Chaque µL 14 aliquote est dimensionnée pour transfecter 3 puits d’une plaque de format 6 puits et comprend 2 µL de l’excès de volume pour tenir compte des erreurs de pipetage. Préparer sept 14 µL d’extraits est suffisante pour effectuer une reprogrammation complète fibroblaste de 3 puits dans une plaque de format 6 puits.
- Préparer le tampon de transfection.
- Réchauffez une bouteille de 500 mL et une bouteille de 100 mL de milieu réduit-sérum frais à température ambiante (RT) pendant environ 2 h. Ne pas utiliser un bain d’eau.
- Transférer un pH-mètre dans une armoire de sécurité biologique. Laver l’électrode de verre du compteur d’eau exempte de nucléase. Calibrer le pH mètre selon les instructions du fabricant. Laver l’électrode à nouveau avec de l’eau exempte de nucléase.
- Transférer les deux flacons de sérum réduit moyen dans la biosécurité du cabinet. La bouteille de 500 mL RT permet d’ajuster le pH.
- Mesurer le pH de base du milieu réduit-sérum en insérant l’électrode de verre du compteur pH dans la mémoire tampon. Attendre jusqu'à 1 min avant la lecture du pH sur le compteur.
- Ajouter 3 à 4 mL de 1 NaOH M dans 500 mL de milieu de sérum réduit, fermez la bouteille et bien mélanger. Attendre jusqu'à 5 min avant d’ouvrir la bouteille pour la mesure de pH. Insérer l’électrode de pH dans la mémoire tampon et attendez que la lecture du compteur pH se stabilise.
- Continuez à ajouter de petites quantités de 1 NaOH M jusqu'à ce que le pH du milieu réduit-sérum atteigne 8.15 – 8.17. Calibrer le pH mètre plusieurs fois au cours du processus. Si à tout moment, le pH devient supérieur à 8.18, le réduire en ajoutant un milieu frais réduit et le sérum de la bouteille préchauffée 100 mL (étape 1.3.1).
Remarque : Le pH du tampon réduit-sérum axée sur le milieu de transfection est essentiel. La reprogrammation sera réussie si le pH de la mémoire tampon de transfection est environ 8,2 ± 0,05 mais peut échouer si le pH est supérieur à 8,25. Par conséquent, il est conseillé d’arrêter d’ajouter NaOH, une fois que pH du tampon atteigne 8.15 – 8.17. Cela garantira que le pH final de la mémoire tampon de transfection est environ 8,2 – 8.22 après stérilisation. - Filtre de stériliser le tampon de transfection utilisant un système de filtration sous vide 0,22 µm. Aliquoter le tampon stérilisé dans des tubes de 5 ou 15 mL avec un minimum d’espace aérien.
- Stocker les aliquotes de la mémoire tampon de transfection jusqu'à 3 mois à 4 ° C. Mesurer le pH des aliquotes périodiquement. Jeter les aliquotes si le pH est supérieur à 8,25. Limiter l’utilisation aliquote à 2 transfections seulement depuis l’exposition de la mémoire tampon de transfection d’air atmosphérique augmente le pH de la mémoire tampon.
- Préparer le milieu d’expansion de fibroblastes (MEF) : milieu essentiel minimum additionné de 10 % sérum bovin fœtal (FBS), 1 x acides aminés non-essentiels, Supplément de glutamine x 1, 55 µM 2-mercaptoéthanol, 1 x stylo/strep/fungizone. Stocker la MEF à 4 ° C.
- Préparer le milieu reprogrammation : DMEM/F12 (aucun HEPES) additionné de 20 % knockout sérum remplacement (KOSR), 0,5 x acides aminés non-essentiels, Supplément de glutamine x 0,5, 55 µM 2-mercaptoéthanol, 50 µg/mL d’acide ascorbique, 1 x stylo/strep/fungizone, bFGF 100 ng/mL et 200 ng/mL B18R. Préparer le milieu lors de l’ouverture de reprogrammation sans bFGF et B18R et conserver à 4 ° C. Ne pas l’utiliser au-delà de 1 mois après préparation. Ajouter bFGF et B18R immédiatement avant chaque utilisation d’une partie aliquote de taille pour utilisation du jour.
- Préparer le milieu de placage en ajoutant 5 % inactivés par la chaleur (HI) FBS dans le milieu de reprogrammation contenant 20 % KOSR sans bFGF et B18R de l’étape 1.5. Préparer les frais moyen le jour de l’utilisation prévue. Ajouter bFGF et B18R immédiatement avant l’emploi au jour J0 de reprogrammation.
- Calibrer les incubateurs de culture de tissu avant d’initier la reprogrammation conformément aux instructions du fabricant à l’aide d’un analyseur portable de2 CO numérique. Utiliser un incubateur bas-O2 pour les étapes de reprogrammation pour assurer la génération réussie iPSC.
2. culture de fibroblastes de reprogrammation
- Préparer les fibroblastes avant le lancement de reprogrammation (journée -2).
- Enduire une nouvelle plaque de culture de tissu de 10 cm avec 5 mL de 0,1 % de gélatine. Touchez ou agiter la plaque pour faire en sorte que la totalité de la surface est enduite. Incuber pendant 15 min à 37 ° C. Aspirer la gélatine et ajouter 10 mL d’ensemble FEM. côté. Ne pas laisser la surface de la plaque à sécher avant d’ajouter le FEM.
- Soigneusement aspirer le milieu usé de fibroblastes. Rincer les cellules une fois avec 5 mL de SPD pour enlever le sérum résiduel. Ajouter 3 mL d’EDTA-trypsine. Balançant doucement la plaque afin d’assurer une couverture complète sur les cellules.
- Aspirer l’excès de liquide, laissant ~ 500 µL de la trypsine. Ne pas trop aspirer, puisque ceci peut sécher et tuer les fibroblastes. Incuber les fibroblastes avec la trypsine-EDTA pendant 3 min à 37 ° C.
- Retirez la plaque de l’incubateur et doucement mais fermement, appuyez sur le côté de la plaque pour déloger les cellules. Vérifier les cellules au microscope. Si les cellules sont jumelés et flottants, aller de l’avant. Si les cellules sont toujours attachés, incuber pendant 3 min.
- Rapidement de rinçage/collecter les cellules individuelles à l’aide de 5 mL de MEF pour neutraliser la trypsine-EDTA. Déplacer la suspension de fibroblastes dans un tube conique de 15 mL. Veiller à ce que les cellules soient bien mélangés.
- Mélanger délicatement la suspension cellulaire pour casser les grosses touffes de cellules, puis compter sur un hémocytomètre. Transfert de 2,5 x 105 cellules dans le plat de 10 cm enduit de gélatine préparée à l’étape 2.1.1. Incuber les cellules pendant la nuit dans les 37 ° C, 5 % CO2 humidifié régulièrement vitroplants incubateur.
Remarque : La densité cellulaire est essentielle pour atteindre l’efficacité élevée de reprogrammation, tel qu’il est important que les fibroblastes sont sains et division rapide. Placage de 2,5 x 105 cellules donne une confluence de 40-60 % 2 jours plus tard pour la plupart des échantillons de fibroblastes. Ajuster la quantité en conséquence pour les cellules malades ou sénescentes atteindre la confluence désirée 40-60 % 48 h plus tard. - Remplacer le support avec 10 mL de MEF frais le lendemain (jour -1).
- Plaque les fibroblastes pour initier la reprogrammation (jour 0).
- Vérifiez que les fibroblastes d’être reprogrammée à la confluence de 40 à 60 % (Figure 2, jour 0). Si les cellules sont sur - ou sous anastomosé, les fibroblastes de passage tel que décrit à l’étape 2.1 et ajuster la densité de placage en conséquence. Culture pendant encore 2 jours.
- Transférer 4 mL de SPD dans un tube conique de 15 mL. Ajouter 100 µL de recombinantes humaines (rh) laminin-521. Pipetter en haut et en bas pour bien mélanger.
- Ajouter 1 ml par puits de rhLaminin-521 dilué dans 3 puits d’une plaque 6 puits. Incuber la plaque enduite à 37 ° C pendant 2 h.
- Chauffer 6 mL de MEF et 4 mL de placage moyenne à 37 ° C. Supplément au milieu placage bFGF à une concentration finale de 100 ng/mL et B18R à une concentration finale de 200 ng/mL.
- Soigneusement aspirer le milieu usé de fibroblastes (étape 2.2.1). Rincer les cellules avec 5 mL de SPD et ajouter 3 mL d’EDTA-trypsine. En faisant doucement tourner la plaque pour couvrir les cellules avec la trypsine-EDTA.
- Aspirer l’excès de liquide, laissant ~ 500 µL de trypsine, comme fait à l’étape 2.1.3. Incuber les fibroblastes avec la trypsine-EDTA pendant 3 min à 37 ° C. Retirez la plaque de l’incubateur et doucement mais fermement, appuyez sur le côté de la plaque pour déloger les cellules. Vérifier les cellules au microscope. Si les cellules sont jumelés et flottants, aller de l’avant. Si les cellules sont toujours attachés, incuber pendant 3 min.
- Rinçage/collecter les cellules individuelles à l’aide de 5 mL de MEF pour neutraliser la trypsine-EDTA. Déplacer la suspension de fibroblastes dans un tube conique de 15 mL. Veiller à ce que les cellules sont bien mélangés et comptent sur un hémocytomètre. Pipetter 12 000 cellules dans 4 mL de milieu de placage préchauffée à l’étape 2.2.4.
Remarque : Ne pas centrifuger les cellules à tout moment. Le nombre de cellules plaqués peut être réglé vers le haut ou bas, tel que requis pour les lignes ou rapide-croissance lente, respectivement. - Retirez la plaque enduite de l’incubateur et aspirer dilué rhLaminin-521 de la microplaque. Ne laissez pas la surface des puits à sec. Doucement remettre en suspension les cellules dans le milieu d’ensemencement. Pipetter 1 mL de suspension cellulaire dans chaque enduit puits (c.-à-d., 3 000 cellules / puits).
- Placez les cellules plaqués dans un incubateur de culture tissulaire tri-gaz avec O de2 à 5 % (basse-O2). Une fois que la plaque est fixée, doucement mais complètement disperser les cellules par une alternance entre un mouvement haut/bas puis gauche/droite. Répétez les mouvements 2 fois plus. Incuber les cellules pendant la nuit. Pas agiter la plaque pour mélanger.
- Pipetter 4 mL de milieu dans un tube conique de 15 mL de reprogrammation et placez-le dans l’incubateur de bas-O2 avec un bouchon desserré s’équilibrer pendant la nuit. Ne pas ajouter de bFGF et B18R jusqu’au lendemain.
3. ouverture de reprogrammation (jour 1)
Remarque : Une fois que la reprogrammation est initiée, entretien quotidien est nécessaire pendant environ 1 mois. N’oubliez pas de planifier en conséquence. Tous les incubations de cellule suivantes doivent être effectuées dans des conditions de faible-O2 37 ° C, 5 % de CO2, incubateur de culture de tissu humidifié tri-gaz 5 % O2 .
- Remplacer le support de placage au moins 1 h avant la transfection.
- Sortir le milieu équilibré de la reprogrammation de l’incubateur de bas-O2 . Ajouter bFGF à une concentration finale de 100 ng/mL et B18R à une concentration finale de 200 ng/mL. Bien mélanger.
- Procéder avec 1 bien à la fois, utilisez une pipette 1 mL pour enlever le milieu usé et le remplacer par 1 mL de milieu reprogrammation additionné de bFGF et B18R. Répétez cette opération pour chaque puits. N’utilisez pas d’aspiration sous vide, qui sèche les cellules excessivement, cause un stress et réduit l’efficacité de reprogrammation. Remettez la plaque avec des cellules sur l’incubateur bas-O2 .
- Transfecter fibroblastes 5 µl de réactif de transfection de RNAiMax par 1 µg d’ADN messagère mis à jour le, et 1 µL de réactif de transfection par 6 pmol de miRNA imite par transfection.
Remarque : Les résultats optimaux sont obtenus lorsque transfections procéder pendant 16 à 20 h. Le réactif de transfection est dilué 10 x ; les 100 ng/µL cocktail mis à jour le mRNA est dilué x 5 ; et les imitateurs de miRNA pmol/µL 5 mix est dilué x 8.33 avec le tampon de transfection avant complexation. Voir le tableau 1 pour un résumé de la configuration de transfection.- Tout en préparant la transfection mixes, minimiser l’exposition potentielle des réactifs à RNase en suivant les pratiques de laboratoire standard.
- Equilibrer une partie aliquote de la mémoire tampon de la transfection (étape 1.3) pendant environ 1 h à température ambiante. Ne pas utiliser un bain-marie à 37 ° C ou un incubateur pour réchauffer le tampon de transfection.
- Supprimer une seule aliquote de mis à jour le mRNA (33 µL) et miRNA imite (14 µL) de-80 ° C et réchauffer à RT pendant environ 3-5 min, jusqu'à ce que décongelés. Ne pas dégeler les aliquotes à 37 ° C. Tournez en leur bas brièvement dans un microtube.
- Réchauffer le réactif de transfection de RT pour environ 3-5 min. Ne pas utiliser un bain-marie à 37 ° C ou l’incubateur. Inverser le tube fermé 2 à 3 fois pour mélanger le réactif. Faire tourner vers le bas brièvement dans un microtube.
- Transférer 279 µL du tampon RT transfection dans un tube de microcentrifuge RNase-libre. Ajouter 31 µL de réactif de transfection pour atteindre un volume final de 310 µL. mélanger soigneusement par pipetage. Ne pas vortexer. Incuber le tube à la droite pendant 1 min.
Remarque : Ce volume de réactif dilué transfection est suffisant au complexe l’ARNm modifiés et miRNA imite aliquotes de l’étape 3.2.3. - Ajouter 132 µL de la mémoire tampon de transfection RT à la quantité de 33 µL d’ARNm mis à jour le. Pipetez doucement pour mélanger : volume final est 165 µL.
- Ajouter 102.6 µL du tampon transfection RT à la quantité de 14 µL de miRNA imite. Pipetez doucement pour mélanger : volume final est 116,6 µL.
- Ajouter 165 µL de réactif dilué transfection étape 3.2.5 à la dilué modifiée ARNm de l’étape 3.2.6. Pipette pour mélanger : volume final est 330 µL.
- Ajouter 116,6 µL de réactif dilué de transfection de l’étape 3.2.5 au mélange de l’étape 3.2.7 imite miRNA dilué. Bien mélanger (volume final est 233,2 µL). Incuber pendant 15 min à ta pour laisser le tampon de transfection de complexes avec les mis à jour le mRNA et miRNA imite.
- Retirez la plaque avec les cellules de l’incubateur de bas-O2 . Ajouter 100 µL (1 µg) de la complexé modifiée mix transfection ARNm dans chaque puits, goutte à goutte dans le puits. Disperser les complexes de transfection par doucement mais bien agiter la plaque avec un mouvement vers le haut/bas puis gauche/droite. Pas agiter la plaque pour mélanger.
- Ajouter 66,7 µL (20 pmol) du miRNA complexé imite la transfection mix dans chaque puits, goutte à goutte dans le puits. Disperser des complexes de transfection par doucement mais bien agiter la plaque avec un mouvement vers le haut/bas puis gauche/droite. Pas agiter la plaque pour mélanger.
- Placez les cellules transfectées dans l’incubateur de tri-gaz avec O de2 à 5 % (basse-O2). Une fois que la plaque est fixée, disperse les complexes de transfection nouveau doucement mais bien agiter la plaque avec un mouvement vers le haut/bas puis gauche/droite.
- Pipetter 4 mL de reprogrammation moyen dans un tube conique de 15 mL et le placer dans un incubateur bas-O2 avec bouchon desserré s’équilibrer pendant la nuit. Ne pas ajouter de bFGF et B18R jusqu’au lendemain.
| Tube 1 - dilution RNAiMax (1er mix) | ||
| Réactif | Concentration de | Volume |
| Tampon de transfection | 279 ΜL | |
| RNAiMax (Ajouter 2e) | x 10 | 31 ΜL |
| Total : 310 µL | ||
| (Incuber 1 min à température ambiante) | ||
| Tube 2 - mis à jour le mRNA (2ème mélanger) | ||
| Réactif | Concentration de | Volume |
| mis à jour le mRNA mix | 100 ng/µL (x 5) | 33 ΜL |
| Tampon de transfection | ΜL 132 | |
| Total : 165 µL | ||
| (ajouter un volume égal de RNAiMax dilué de 1 Tube) | ||
| Tube 3 - miRNA imite (3e mélange) | ||
| Réactif | Concentration de | Volume |
| miRNA imite mix | 5 pmol/µL (8,33 x) | 14 ΜL |
| Tampon de transfection | 102.6 ΜL | |
| Total : 116,6 µL | ||
| (ajouter un volume égal de RNAiMax dilué de 1 Tube) | ||
Tableau 1 : Préparation du mélange de transfection.
4. remplacement de reprogrammation moyen entre Transfections (jours 2, 4, 6, 8, 10 et 12)
- Remplacez la transfection après milieu 16 – 20 h comme décrit dans 3.1.1–3.1.2. Ajouter bFGF à une concentration finale de 100 ng/mL et B18R à une concentration finale de 200 ng/mL en reprogrammation aliquotes moyens.
- Surveiller les mWasabi expression quotidiennement pour vérifier la qualité de transfection à l’aide d’un microscope configuré pour visualiser EGFP.
Remarque : mWasabi expression devrait être minimalement apparent au jour 2 et augmentation de luminosité avec chaque transfection supplémentaire.
5. autres-quotidien Transfections (jours 3, 5, 7, 9, 11 et 13)
- Effectuez la transfection tel que décrit à l’étape 3.2. Ne changez pas le milieu aux jours de la transfection.
- Préparer une portion de 4 mL de milieu de reprogrammation et placez-le dans un incubateur bas-O2 s’équilibrer pendant le changement médiatique le jour suivant. Ne pas ajouter de bFGF et B18R jusqu’au lendemain.
6. les procédures à effectuer après la Transfection Final
- Effectuer des variations moyennes quotidiennes du 14e grâce à environ 17 jours. Chauffer 7 mL de reprogrammation moyenne à 37 ° C. Ajouter bFGF à une concentration finale de 100 ng/mL.
Remarque : B18R n’est plus nécessaire après la transfection finale. Au-delà de 14 jours, il n’est plus nécessaire d’équilibrer le milieu du jour au lendemain dans un incubateur bas-O2 . - Retirez le support de tous les puits à l’aide d’une pipette sérologique. Continuer à ne pas utiliser d’aspiration. Ajouter 2 mL de reprogrammation additionné de bFGF / puits.
- Jours 17 et 18, analyser les puits reprogrammés sous un microscope inversé ou dissection. Si les colonies sont forment à proximité les uns aux autres en raison d’un rendement élevé de reprogrammation et ne peut pas être isolés manuellement, séparer les colonies en effectuant un passage facultatif des puits reprogrammés décrits à l’étape 7 ci-dessous. Si les colonies sont rares et bien séparées, choisir manuellement les clones directement à partir de la reprogrammé bien tel que décrit à l’étape 8 et CISP sous-culture conformément aux protocoles standards.
7. la procédure facultative : Passage de cellules provenant de puits reprogrammé sur EDTA
- Préparer 0,5 mM EDTA (EDTA) au SPD en diluant la solution EDTA 0,5 M. Filtre stériliser EDTA en utilisant un système de filtration sous vide de 0,22 µm.
- Préparer le milieu de cellules souches (CFP) de pluripotentes exempte d’engraissement (p. ex., mTeSR1) selon les instructions du fabricant. Préchauffer 32 mL de milieu de la CFP à 37 ° C.
- Enduire tous les puits de deux plaques 6 puits format avec CSEh qualifié de la matrice extracellulaire (ECM) en suivant les instructions du fabricant. Sceller les plaques avec le film de paraffine et les incuber pendant 1 heure à température ambiante.
- Aspirer la solution ECM des plaques pré chauffées et remplacez-le par 2 mL de milieu de CFP / puits. Ne laissez pas la surface des puits à sec. Mettez-les de côté.
- Aspirez le milieu reprogrammation de 2 puits reprogrammés sur un jour où les colonies sont grandes et bien formé (habituellement jour 18). Rincez une fois avec 1 mL d’EDTA et aspirer.
- Ajouter 1 mL d’EDTA / puits. Incuber pendant 4 min à 37 ° C.
- Délicatement enlever la plaque de l’incubateur et placez-le dans la prévention des risques biotechnologiques du cabinet.
Remarque : À ce stade, les cellules peuvent être très faiblement adhérentes et délogent facilement. - Soigneusement aspirer EDTA de deux puits. Ajouter 3 mL de milieu de CFP préchauffé à chacune bien traités avec de l’EDTA. Utilisez un grattoir de cellules pour doucement mais complètement déloger les cellules de deux puits.
- Utiliser une pipette sérologique pour doucement la suspension cellulaire, déposer et briser les grosses touffes. Ne pas pipeter jusqu'à ce que tous les bouquets de cellule ont disparu. Préserver les grappes de l’iPSC.
NOTE : Bordé de grosses touffes n’affectera pas l’excroissance des colonie iPSC. S’il y a des touffes de trop grandes cellules, tout simplement éviter de les transférer dans le plat suivant. - À l’aide d’une pipette sérologique, répartir uniformément les cellules de chaque puits traitées à l’EDTA par pipetage 0,5 mL dans chaque puits de la plaque de 6 puits ECM-enduit.
Remarque : Ne pas combiner les cellules provenant des puits reprogrammés (c.-à-d., reprogrammé bien 1 doit être répartie uniformément dans la première plaque 6 puits, et reprogrammé 2 puits doit être répartie uniformément dans la deuxième plaque 6 puits). - Remettez les cellules plaqués sur l’incubateur bas-O2 . Pour répartir les cellules, agiter chaque plaque et-avant-arrière et gauche-droite. Tourbillonner pas. Remplacer le milieu de la CFP par jour.
8. cueillette iPSC Colonies
- Préchauffer 15 mL de milieu de la CFP à 37 ° C. Enduire tous les puits d’une seule plaque 6 puits d’ECM CSEh qualifié suivant les instructions du fabricant. Sceller les plaques avec du film de paraffine et les incuber pendant 1 heure à température ambiante.
- Aspirez le milieu de culture des puits servant à recueillir le CISP. Rincez une fois avec 1 mL d’EDTA et aspirer. Ajouter 1 mL d’EDTA / puits. Incuber pendant 4 min à 37° C. Alors que les cellules sont en incubation, aspirer la solution ECM des plaques pré chauffées et remplacez par 2 mL de milieu de CFP / puits. Mettre de côté les plaques.
- Retirez délicatement la plaque avec le CISP à prélever de l’incubateur et placez-le dans la prévention des risques biotechnologiques du cabinet.
Remarque : À ce stade, les cellules peuvent être très faiblement adhérentes et délogent facilement. - Soigneusement aspirer EDTA. Très doucement, ajouter 3 mL de préchauffé moyen de CFP, prenant soin de ne pas pour déloger les colonies de l’iPSC.
- Déplacer la plaque à un champ inversé ou dissection afin de mieux visualiser les colonies. Préparer une pipette 1 mL avec un embout stérile. Enfoncer le piston à fond, puis utilisez la pipette pour gratter doucement une colonie tout en tirant lentement liquide dans la pointe pour recueillir la colonie. Dessiner comme moyen peu que possible tout en choisissant la colonie iPSC.
- Pipetter verticalement pour transférer la colonie, 3 à 4 fois dans un seul puits de la plaque de revêtement ECM de l’étape 8.2. Répétez jusqu'à ce que 6 colonies ont été cueillies et transférés dans des puits individuels. Prélever des pas plus de 2 colonies de tout puits unique si un passage en option décrit à l’étape 7 a été effectuée.
- Utiliser un protocole standard les cellules souches humaines de congeler dans tous les puits restants. Décongeler et re-plaque stocks congelés Si plus de colonies doivent être cueillies plus tard.
9. caractérisation du CISP
- Effectuer des TRA-1-60 de coloration pour l’analyse de la reprogrammation de l’efficacité (18 jours).
NOTE : 2 / 3 reprogrammé puits sont repiquées pour le prélèvement de la future colonie. Eh bien le restant peut être utilisé pour la coloration des TRA-1-60. Cette intervention peut être réalisée sur la paillasse de laboratoire.- Laver le reprogrammé bien avec 1 mL de PBS. Fixer les cellules avec du méthanol glacée à-20 ° C pendant 5 min.
- Aspirer le méthanol. Sec le puits pendant environ 2 min. Veillez ne pas trop sec. Les cellules ont séché assez quand ils deviennent une couleur translucide/mat.
- Lavez les bien 3 fois avec 1 mL de PBS pendant 5 min avec agitant doucement. Au cours de la lave, préparer 3 mL de peroxyde d’hydrogène 3 % dans du PBS.
- Aspirer le PBS. Ajouter 2 mL de solution diluée de peroxyde. Incuber pendant 15 min à ta avec agitant doucement. Préparer 4 mL de la solution de saturation : 10 % l’albumine sérique bovine (BSA) dans du PBS.
- Aspirer la solution peroxyde. Lavez les bien 2 fois avec 1 mL de PBS pendant 5 min.
- Aspirer les PBS et ajouter 2 mL de solution de blocage dans le puits. Incuber pendant 1 heure à température ambiante.
- Lavez les bien 3 fois avec 1 mL de PBS pendant 5 min avec agitant doucement.
- Diluer l’anticorps primaire anti-TRA-1-60 à 1/100 dans la solution de blocage additionné de 0.05 % d’azide de sodium. Préparer 1 mL de dilution d’anticorps pour 1 bien d’un plat de format 6 puits. Ajouter la dilution d’anticorps dans le puits et enrouler la plaque avec du parafilm pour éviter l’évaporation. Incuber une nuit à 4 ° C sous agitation douce.
Remarque : Si nécessaire, l’incubation avec l’anticorps primaire peut être faite pendant 1 h à RT avec agitant doucement. La dilution de l’anticorps primaire peut être réutilisée jusqu'à 5 fois. - Après incubation avec l’anticorps primaire, lavez les bien 3 fois avec 1 mL de PBS pendant 5 min avec agitant doucement.
- Diluer l’anticorps conjugué HRP anti-souris secondaire au 1 : 200 dans la solution de blocage. Incuber le puits avec la dilution de l’anticorps secondaire pendant 2 h à RT avec agitant doucement.
- Aspirer la dilution de l’anticorps secondaire et lavez les bien 3 fois avec 1 mL de PBS pendant 5 min avec agitant doucement.
- Lors du troisième lavage, préparer la solution de substrat suivant les instructions du fabricant. Après le lavage final, aspirer PBS. Ajouter 1 mL de la solution de substrat et incuber avant que la couleur souhaitée (environ 10 min).
- Aspirer la solution de substrat. Rincez le puits avec de l’eau pendant 5 min en secouant doucement.
- Compter les colonies, si vous le souhaitez. Reprogrammation d’efficacité = (nombre de colonies) / (nombre de fibroblastes plaqués) x 100 %.
- Pour le stockage à long terme, aspirer l’eau de la plaque teintée et sécher toute la nuit à RT. Seal la plaque sèche avec du parafilm et conserver à 4 ° C jusqu'à 2 ans évaluer l’efficacité de reprogrammation plus tard.
Résultats
Cela prend généralement environ 5 à 6 semaines de l’ouverture de fibroblastes reprogrammation au gel du premier flacon de CISP (Figure 1). Le protocole de reprogrammation peut être classé généralement en deux phases. La phase 1 comprend la culture de fibroblastes et sept transfections, avec l’ARN reprogrammation cocktail effectuées chaque 48 h. Phase 2 comprend l’isolation, expansion et caractérisation des colonies iPSC.
Avant de lancer le protocole, il convient d’assurer que les fibroblastes pour être reprogrammés sont de bonne qualité. Fibroblastes sains devraient apparaître fusiformes, bipolaire et réfringents avec un temps de doublement d’environ 24 heures. Par jour 0, 250 000 cellules plaqués dans un plat de 10 cm le jour -2 devraient croître à la confluence de 40 à 60 % (Figure 1, jour 0) et produisent environ 6 – 10 x 105 cellules. Les cellules prolifèrent à un rythme plus lent peuvent être compensées par électrodéposition à une densité plus élevée le jour -2 et le jour 0 de reprogrammation.
Fibroblastes devraient apparaître très clairsemée placage suivant dans une cupule d’une boîte de format 6 puits de reprogrammation (Figure 2, jour 1). Vingt-quatre heures après la transfection première, fibroblastes seront perdent leur forme de broche et adoptent une morphologie plus arrondie (Figure 2, jour 2), qui est maintenue dans le reste de la reprogrammation. Fluorescence verte de mWasabi ARNm devrait être minimalement observables au jour 2 et constante augmentation de luminosité pour être clairement visible par jour 4. La capacité de détecter la fluorescence mWasabi peut dépendre de la configuration et sensibilité étendue. La densité cellulaire progressivement et systématiquement augmentera dans les trois premiers transfections (jours 1-6), avec un éclat apparent dans la prolifération qui se produisent entre les jours 7 et 8. Les cellules devraient apparaître largement anastomosés au jour 10 (Figure 2, jour 10). Les premiers colons de l’iPSC peuvent apparaître dès le jour 11 (Figure 2, jour 11) ; Cependant, les colonies peuvent être non observable jusqu’au 18e jour. En règle générale, par jours 15 à 18, il y aura grande et évidente iPSC colonies qui sont clairement distincts des voisines, des fibroblastes incomplètement reprogrammés (Figure 2, jour 15 et la Figure 3, 17 jours). Immunomarquage pour le marqueur de pluripotence TRA-1-60 peut être effectuée afin d’évaluer l’efficacité de reprogrammation (Figure 3, 17, TRA-1 à 60 jours). Dans notre expérience, la plupart des lignes de fibroblastes produisent des centaines de colonies par reprogrammé bien (Figure 3, insertion de type B).
Électrodéposition sous-optimale densité est la raison la plus courante pour une efficacité réduite de reprogrammation dans notre protocole et est fréquemment associée aux fibroblastes qui sont malades, sénescentes ou haute-passage. Si la densité de placage est trop faible, il y aura de grandes plaques dénudées acellulaires à la fin de reprogrammation (Figure 4), et iPSC colonies ne peuvent pas former (Figure 4). Reprogrammé cellules devraient être très anastomosés par jour 14 (comparer la Figure 4 a et 4 b de la Figure 2, jour 14). De même, si les cellules sont plaqués trop dense ou prolifèrent trop rapidement, reprogrammation efficacité est réduite considérablement.
Pour maintenir l’homogénéité du CISP dérivé de patient, il est important de développer des lignées de cellules provenant d’une seule colonie. Puisque l’efficacité de la reprogrammation est très élevée dans notre protocole, voisins iPSC colonies pourraient se former à proximité et se développer dans chaque autre (Figure 3, jours 15-17). Cela rend parfois difficile de séparer mécaniquement une seule colonie d’expansion clonale. Nous avons trouvé qu’il est utile de premier passage bien un reprogrammé et le diluer dans un plus grand secteur de la culture. Un ratio de bon passage consiste en fractionnement uniformément une plaque 6 puits-format reprogrammée bien à travers une plaque entière de 6 puits.
Après le passage de la dilution, CISP poussent comme des colonies et est faciles à distinguer des fibroblastes (Figure 5, jour 18). Initialement, iPSC colonies peuvent être plus ou moins emballés, et les cellules individuelles ont une assez grande surface cytoplasmique. Au cours de 4 à 7 jours, le CISP prolifèrent et forme une colonie caractéristique-serrés avec bords définis. Les cellules individuelles au sein de la colonie ont une forte proportion de nucléaire avec des nucléoles proéminents (Figure 5, jour 22). Il devrait y avoir beaucoup de colonies qui se forment dans chaque puits, et seuls ceux dont la morphologie de l’iPSC classique doivent être prélevés pour l’expansion.
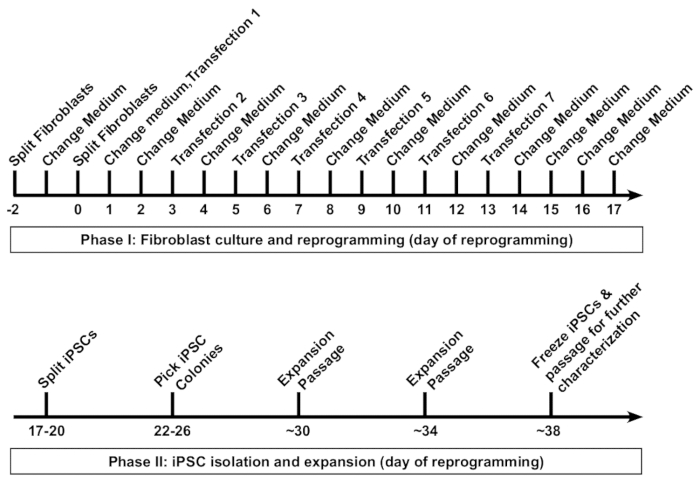
Figure 1 : Reprogrammation des fibroblastes humains en induite par les cellules souches pluripotentes (CISP). Un protocole schématique pour la reprogrammation des fibroblastes humains est présenté. Les fibroblastes sont repiquées tout d’abord à une densité faible dans une cupule d’une boîte de 6 puits format, suivi par sept transfections effectuées à des intervalles de 48 h. Le milieu est remplacé 16 – 20 h après chaque transfection. Reprogrammé CISP est repiquées tout d’abord à environ jour 18, clonale des colonies et sont recueillis par jour 26. En général, dérivé de fibroblastes iPSC lignes peuvent être congelés pour le stockage à long terme par jour 38. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
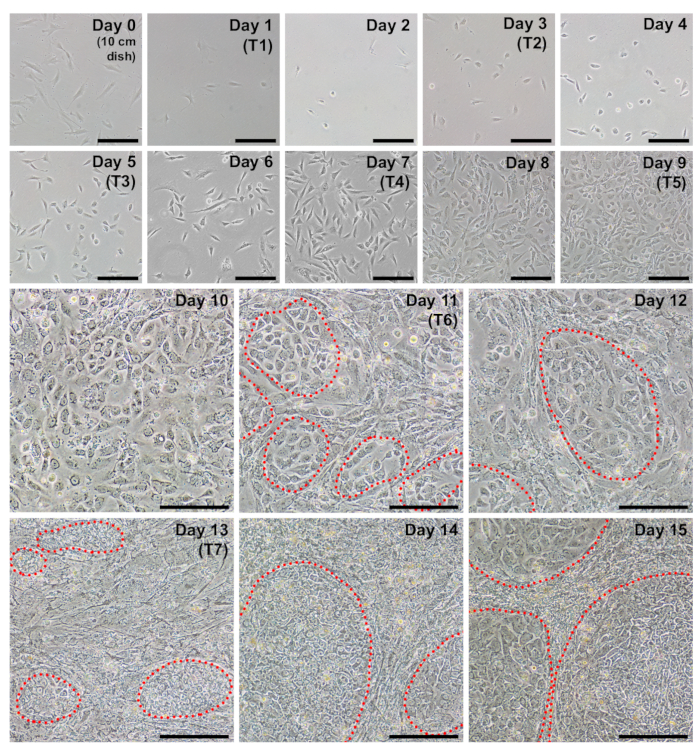
Figure 2 : Images représentatives de tous les jours au cours de chaque journée de reprogrammation. Fibroblastes devraient être environ de 40 à 60 % anastomosé au moment du passage d’ouvrir la reprogrammation (jour 0, les cellules sont plaqués dans un plat de 10 cm). La transfection de première (T1) se produit au jour 1, et les cellules devraient apparaître très clairsemées à ce stade. Le lendemain (jour 2), une morphologie plus arrondie devienne apparente. Cellules continueront d’augmenter la densité tout au long du protocole avec les clusters iPSC commence à apparaître dès le jour 11 (entouré en rouge). Jour 15, iPSC colonies sera grandes bornages discret. Echelle = 200 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
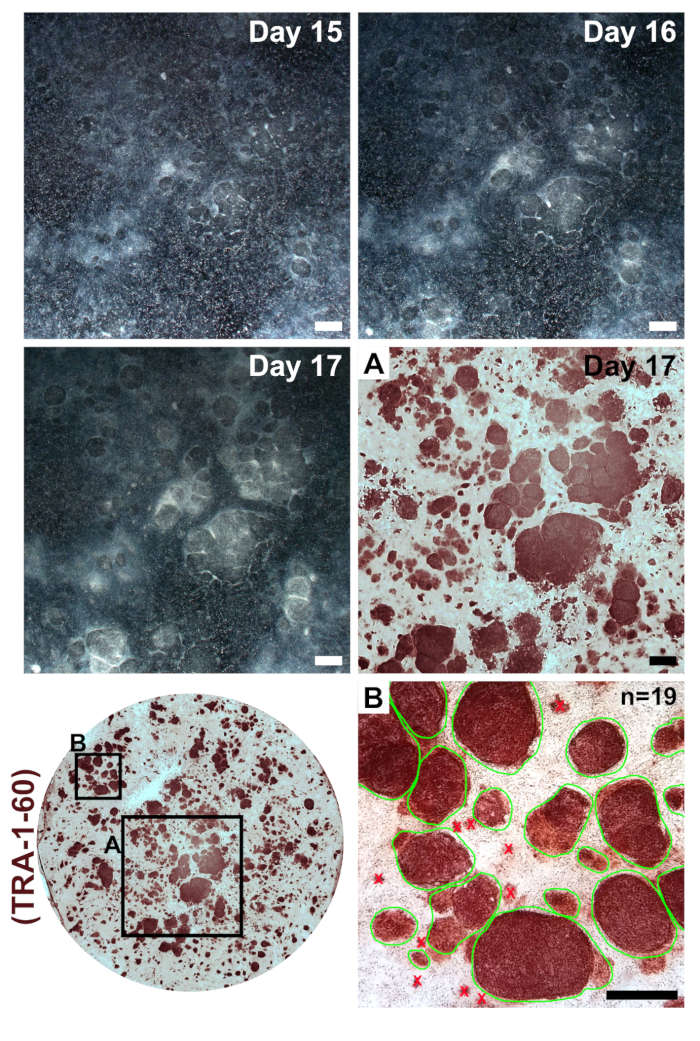
Figure 3 : Formation de colonies suite transfections avec reprogrammation modifié mRNAs et miRNA imite. Faible grossissement des images ont été prises d’un représentant de reprogrammation sur jours 15-17. Après la transfection finale, CISP reprogrammé formeront des colonies clairs avec des frontières définies que développer dans la taille et se condensent pour devenir nettement distincte de fibroblastes entourant incomplètement reprogrammés. Immunomarquage pour le marqueur de pluripotence TRA-1-60 indique la présence de CISP (schéma A) et peut être utilisé pour le calcul de l’efficacité de la reprogrammation en comptant toutes les colonies dans un puits unique (médaillon B, exemples de colonies dénombrables entouré en vert). Echelle = 1 mm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
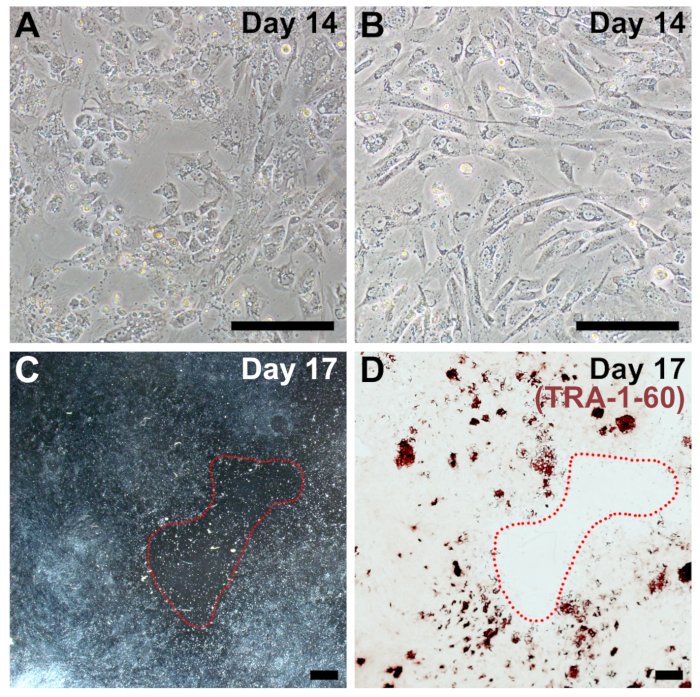
Figure 4 : Des images représentatives de densité placage sous-optimale pour reprogrammation. (A, B) Exemples de fibroblastes qui sont trop fragmentées par jour 14 de reprogrammation (comparer à la Figure 2, jour 14). (C) image de faible grossissement sur 17 jours de reprogrammation avec une grande tache aride entouré en rouge. (D) le même puits a été fixé et colorés pour TRA-1-60 confirmer les pauvres ensemble l’efficacité en raison de la faible densité de reprogrammation. Barreaux de l’échelle = 200 µm (A, B) et 1 mm (C, D). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
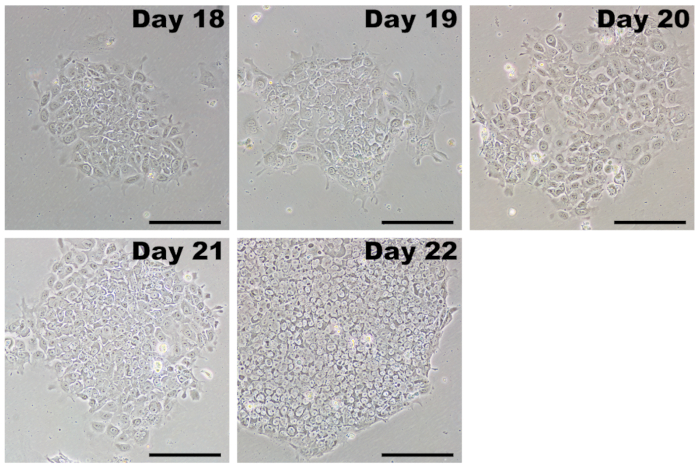
Figure 5 : Des images représentatives de CISP après passage initial. Après avoir terminé de transfections avec reprogrammation mis à jour le mRNA et des imitations de miRNA, des cellules reprogrammés sont repiquées par jours 17 à 20. CISP jouissent d’un avantage de croissance dans le milieu de la CFP et dépasser rapidement les fibroblastes qui ont été reprogrammés incomplètement. Au départ, CISP formeront des colonies qui peuvent apparaître lâches aux frontières mal définies. Quelques jours après, les cellules rapidement prolifèrent et prennent sur la morphologie caractéristique des cellules serrées avec un noyau-à-cytoplasme élevé, étroitement clustering en colonies radiovasculaires distinctement aréolées. Echelle = 200 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Ce protocole décrit une méthode ayant une pertinence clinique, sans intégration, base d’ARN qui permet la reprogrammation des lignes de fibroblastes humains normaux et associés à la maladie en CISP à une très haute efficacité. A ce jour, chaque ligne de fibroblaste humain, que nous avons tenté de reprogrammer avec le protocole décrit a donné un nombre satisfaisant de CISP de haute qualité pour des applications en aval. CISP résultant immédiatement peut être transférés et élargi dans les conditions de culture sans chargeur.
Qualité des fibroblastes de reprogrammation :
Reprogrammation de succès est fortement tributaire de la qualité d’à partir de fibroblastes. Idéalement, la reprogrammation doit être instaurée avec les fibroblastes de passage le plus bas disponibles pour réaliser le rendement le plus élevé. Reprogrammation d’efficacité est meilleure avec des fibroblastes de passage 2 – 4. Reprogrammation peut encore fonctionner avec haut-passage (passage 5 – 8), les fibroblastes sénescents même, mais avec un rendement réduit. Parfois les fibroblastes basse-passage ne sont pas disponibles ou échantillons de patients ont une mutation génétique qui empêche une croissance saine. Dans ce cas, optimisation de la densité des ensemencements initiaux peut-être être requise. La reprogrammation des lignes de fibroblastes compromis est généralement associée à la mort cellulaire accrue au cours de transfections RNA. Ainsi, les cellules de la reprogrammation bien semble être fragmentées par jours 10-14 de reprogrammation. Grandes surfaces acellulaires sera également visibles dans le puits. Si c’est le cas, le protocole de reprogrammation devront être réinitialisée avec un nombre plus élevé de départ initial des fibroblastes. 3 000 cellules d’entrée par la cupule d’une boîte de format 6 puits de placage travaille constamment pour lignes de fibroblastes plus adultes. Toutefois, le nombre de placage à 5 000 – 10 000 (50 000 pour les lignes sénescentes) peut contribuer à améliorer la reprogrammation des échantillons associés à la maladie, comme cela a été signalé dans notre précédente publication8. À l’inverse, les cellules atteignant confluence trop tôt peuvent aussi être résistants à la reprogrammation. Si les cellules subissant transfections avec reprogrammation RNAs prolifèrent trop rapidement (comme c’est parfois le cas avec des fibroblastes néonatales primaires), initier la reprogrammation avec 500 fibroblastes par puits d’un plat de 6 puits format8.
Manipulation de la mémoire tampon de transfection :
Le pH de la mémoire tampon de transfection (sérum réduit moyenne ajustée au pH de 8.2) est primordial pour atteindre l’efficacité de transfection optimal requise pour ce protocole de reprogrammation. Pour cette raison, plusieurs Précautions concernant la manipulation de la mémoire tampon de transfection sont recommandées. Nous avons trouvé que l’exposition même brève de la mémoire tampon de transfection d’air atmosphérique affecte le pH de la mémoire tampon. Par conséquent, la mémoire tampon de transfection doit être aliquotée dans un récipient de bouchon à vis avec espace d’air minimal (nous utilisons 5 ou 15 mL bouchon à vis tubes coniques). Pour minimiser encore plus l’exposition à l’air, utilisez chaque tampon de transfection aliquote pour un maximum de deux transfections. Enfin, depuis le pH d’effets de température, il est essentiel que le tampon de transfection est équilibré au RT pour l’assemblage des complexes de transfection. Il est conseillé de ne réchauffe ne pas le tampon de transfection à 37 ° C.
Passage du CISP :
Alors que bon nombre inédit de protocoles recommander cueillette iPSC individuels des colonies à la fin de la reprogrammation, cela peut être difficile à réaliser lorsque l’efficacité de la reprogrammation est très élevée ou si les colonies cluster ensemble, comme c’est souvent le cas dans notre Protocole. Donc, si les colonies de l’iPSC sont à proximité les uns aux autres, nous vous recommandons passant tout d’abord les cellules d’étaler le CISP reprogrammé avant la cueillette manuelle des colonies. Il y a plusieurs avantages à cette étape de passage au début. Les colonies s’étalant leur donne plus d’espace pour se développer, produisant des colonies beaucoup plus grandes pour le prélèvement que pourrait autrement être atteint dans le puits original. Cela améliore considérablement le taux de réussite en établissant une ligne d’iPSC d’un clone sélectionné. Nous constatons également que le temps additionnel de la culture avec des fibroblastes, bien qu’à un ratio dilué, semble améliorer la qualité moyenne des colonies de l’iPSC cueillies. Les fibroblastes incomplètement reprogrammés peuvent fournir des justificatifs facteurs paracrines, qui continuent à aider à établir le CISP et faciliter la transition directe dans les conditions de culture cellulaire exempt d’engraissement. Les fibroblastes ont fortuitement un désavantage de croissance sélective par rapport au CISP lorsqu’il est cultivé dans mTeSR1. Donc, la population de cellules de fibroblaste contaminantes est rapidement diluée à des quantités négligeables au sein de 3 à 4 passages.
Inhibiteurs de ROCK comme Y-27632 sont fréquemment utilisés pour la culture courante de CISP humaine. Nous avons constaté que la culture fréquente et/ou prolongée de certaines lignes iPSC avec Y-27632 peut avoir des effets délétères sur la qualité globale. Lorsque vous utilisez un bouquet passage de méthode, comme avec l’EDTA, Y-27632 n’est pas nécessaire pour maintenir la viabilité de l’iPSC après le fractionnement. Nous avons éliminé complètement les médias complétant avec Y-27632 pour tous les iPSC isolation, agrandissement ou culture courante.
Limitations du protocole :
Une des limitations à l’approche de reprogrammation base d’ARN décrit est le coût initial et la complexité liés à la préparation des réactifs reprogrammation. Bien que les procédures préparatoires pour générer des ARNm réactifs sont tous routinières et ont déjà été décrits (PCR, in vitro de transcription, traitement de DNase, bouchage, déphosphorylation, purification), cumulativement la fabrication de réactifs de l’ARNm est un processus relativement long et non négligeable. L’autre défi majeur de ce protocole est qu’il fallait transfecter toutes les 48 h, augmentant l’intensité de travail du protocole de reprogrammation des cellules. Ces considérations peuvent être prohibitifs si reprogrammation de seulement quelques échantillons de patients est souhaitée. Toutefois, si la considération primordiale est la génération de CISP cliniquement pertinente ou parvenir à très haute efficacité de reprogrammation, l’approche de reprogrammation base d’ARN décrit est idéal.
En résumé, la haute efficacité base d’ARN reprogrammation méthode décrite charnières sur l’efficacité de transfection optimisée du type cellules somatiques à être reprogrammé comme décrit dans notre précédente publication8. Le protocole de transfection de RNA présenté dans cette étude est très à l’écoute pour les fibroblastes humains primaires mais peut potentiellement être adapté à d’autres types de cellules pour améliorer l’efficacité de reprogrammation de diverses cellules somatiques.
Déclarations de divulgation
I.K. et G.B. sont co-inventeurs sur une demande de brevet intitulé « Méthodes et Compositions pour la reprogrammation des cellules », No de la demande PCT PCT/US2016/063258.
Remerciements
Nous sommes reconnaissants pour le soutien financier de la National Institutes of Health (T32AR007411-33) et l’Université du Colorado peau Diseases Research Core Center (P30AR057212). Nous remercions également le partenariat de recherche de l’épidermolyse bulleuse (EB), EB Medical Research Foundation, Cure EB charité, dystrophiques Epidermolyse bulleuse Research Association (DEBRA) International, de la Fondation Roi Baudouin Vlinderkindje fonds et portes Fonds de frontières.
matériels
| Name | Company | Catalog Number | Comments |
| Plasmid templates for PCR | |||
| pcDNA3.3_KLF4 | Addgene | 26815 | |
| pcDNA3.3_SOX2 | Addgene | 26817 | |
| pcDNA3.3_c-MYC | Addgene | 26818 | |
| pcDNA3.3_LIN28A | Addgene | 26819 | |
| pCR-Blunt_hM3O | Addgene | 112638 | |
| pCR-Blunt_hNANOG | Addgene | 112639 | |
| pCR-Blunt_mWasabi | Addgene | 112640 | |
| Modified mRNA in vitro transcription and miRNA mimics | |||
| Forward Primer | Integrated DNA Technologies | TTGGACCCTCGTACAGAAGC TAATACG | |
| Reverse Primer (Ordered as ultramer, 4nmol scale) | Integrated DNA Technologies | TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT CTTCCTACTCAGGCTTTATTCA AAGACCA | |
| (ARCA Cap) 3´-0-Me-m7G(5')ppp(5')G | New England Biolabs | S1411S | |
| Pfu Ultra II Hotstart 2x Master Mix | Agilent | 600850-51 | |
| 5-Methylcytidine-5'-Triphosphate | Trilink Biotechnologies | N-1014 | |
| Antarctic Phosphatase | New England Biolabs | M0289L | |
| DNase I | NEB | M0303S | |
| MEGAscript T7 Transcription Kit | ThermoFisher Scientific | AM1334 | |
| Pseudouridine-5'-Triphosphate | Trilink Biotechnologies | N-1019 | |
| Riboguard RNase Inhibitor | Lucigen | RG90910K | |
| RNA Clean & Concentrator | ZymoResearch | R1019 | |
| Syn-hsa-miR-302a-3p miScript miRNA Mimic | Qiagen | MSY0000684 | |
| Syn-hsa-miR-302b-3p miScript miRNA Mimic | Qiagen | MSY0000715 | |
| Syn-hsa-miR-302c-3p miScript miRNA Mimic | Qiagen | MSY0000717 | |
| Syn-hsa-miR-302d-3p miScript miRNA Mimic | Qiagen | MSY0000718 | |
| Syn-hsa-miR-367-3p miScript miRNA Mimic | Qiagen | MSY0000719 | |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9937 | Use to dilute modified mRNAs and miRNA mimics |
| Fibroblast culture and reprogramming | |||
| 0.1% Gelatin in H2O | StemCell technologies | #07903 | |
| Stericup-GV Sterile Vacuum Filtration System | EMD Millipore | SCGVU05RE | Use to sterilize the transfection buffer and 0.5 mM EDTA in DPBS |
| 2-Mercaptoethanol | ThermoFisher Scientific | 21985023 | |
| 6-well plates (tissue culture treated) | Corning | 3516 | |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | |
| Fetal Bovine Serum | Fisher | 26-140-079 | |
| FGF Basic | ThermoFisher Scientific | phg0263 | |
| GlutaMax Supplement | ThermoFisher Scientific | 35050061 | Glutamine supplement used for the prepration of media |
| Heat Inactivated FBS | Gibco Technologies | 10438026 | |
| KnockOut Serum Replacement | ThermoFisher Scientific | 10828010 | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher Scientific | 13778500 | The protocol is optimized for the Lipofectamine RNAiMax transfection reagent |
| MEM | ThermoFisher Scientific | 11095080 | |
| MEM Non-essential amino acids | ThermoFisher Scientific | 11140050 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985070 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 500 mL |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985062 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 100 mL |
| 10 M NaOH | Sigma-Aldrich | 72068 | Make a 1 M solution by diluting in nuclease free water and use for pH adjustment |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9932 | Use to dilute NaOH and wash a pH meter |
| Pen/Strep/Fungizone | GE Healthcare | SV30079.01 | |
| rhLaminin-521 | ThermoFisher Scientific | A29248 | Supplied at a concentration of 100 µg/mL, use as a matrix for the reprogramming procedure |
| Dulbecco's phosphate-buffered saline (DPBS) | Life Technologies | 14190144 | |
| Trypsin-EDTA 0.25% Phenol Red | Life Technologies | 2520056 | |
| Vaccinia Virus B18R (CF) | ThermoFisher Scientific | 34-8185-86 | |
| iPSC culture | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| EDTA, 0.5 M stock solution | K&D Medical | RGF-3130 | Dilute to 0.5 mM in DPBS, filter sterilize and use for iPSC passaging |
| mTeSR1 | StemCell Technologies | 85850 | Pluripotent stem cell (PSC) medium, provides growth advantage to iPSCs over fibroblasts |
| Antibodies and Detection | |||
| Rabbit anti Mouse (HRP conjugated) | Abcam | ab97046 | |
| Tra-1-60 (mouse anti human) | Stemgent | 09-0010 | |
| Hydrogen Peroxide (30%) | LabChem | LC154301 | Dilute to 3% with PBS |
| Bovine Serum Albumin (BSA) | Fisher | BP9703100 | |
| Phosphate-buffered salin (PBS) | Hyclone | SH30258.02 | Supplied as 10x, dilute to 1x |
| VECTOR NovaRED Peroxidase Substrate Kit | Vector Laboratories | SK-4800 | |
| Special Equipment | |||
| Description | Notes | ||
| Biological safety cabinet | |||
| Regular humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 37 °C. | ||
| Tri-gas humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 5% O2, 37 °C. Use for the reprogramming procedure. | ||
| pH Meter | Must have resolution to two decimal places. Designate to RNA work if possible. | ||
| Inverted microscope | Microscope configured to visualize EGFP for monitoring transfection efficiency. |
Références
- Takahashi, K., Yamanaka, S. Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors. Cell. 126 (4), 663-676 (2006).
- Takahashi, K., et al. Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors. Cell. 131 (5), 861-872 (2007).
- Schlaeger, T. M., et al. A comparison of non-integrating reprogramming methods. Nature Biotechnology. 33 (1), 58-63 (2015).
- Hacein-Bey-Abina, S., et al. LMO2-Associated Clonal T Cell Proliferation in Two Patients after Gene Therapy for SCID-X1. Science. 302 (5644), 415-419 (2003).
- Warren, L., et al. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7 (5), 618-630 (2010).
- Judson, R. L., Babiarz, J. E., Venere, M., Blelloch, R. Embryonic stem cell-specific microRNAs promote induced pluripotency. Nature Biotechnology. 27 (5), 459-461 (2009).
- Anokye-Danso, F., et al. Highly efficient miRNA-mediated reprogramming of mouse and human somatic cells to pluripotency. Cell Stem Cell. 8 (4), 376-388 (2011).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nature Communications. 9 (1), (2018).
- Hirai, H., et al. Radical acceleration of nuclear reprogramming by chromatin remodeling with the transactivation domain of MyoD. Stem Cells. 29 (9), 1349-1361 (2011).
- Mandal, P. K., Rossi, D. J. Reprogramming human fibroblasts to pluripotency using modified mRNA. Nature Protocols. 8 (3), 568-582 (2013).
- Avci-Adali, M., et al. In vitro synthesis of modified mRNA for induction of protein expression in human cells. Journal of Visualized Experiments. , e51943 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon