Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
À l’aide au transfert d’énergie Fluorescence in Vitro pour étudier la dynamique des protéines Complexes à une échelle de temps de milliseconde
Dans cet article
Résumé
Interactions protéines-protéines sont essentielles pour les systèmes biologiques, et des études de la cinétique de liaison donnent un aperçu de la dynamique et la fonction de complexes protéiques. Les auteurs décrivent une méthode qui quantifie les paramètres cinétiques d’une protéine complexe à l’aide de transfert d’énergie de fluorescence par résonance et la technique de flux stoppé.
Résumé
Les protéines sont les principaux opérateurs des systèmes biologiques, et généralement, ils interagissent avec d’autres macro - ou petites molécules pour remplir leurs fonctions biologiques. Ces interactions peuvent être très dynamiques, ce qui signifie que les sous-unités qui interagissent constamment associées et dissociées à certains taux. Tout en mesurant l’affinité de liaison à l’aide de techniques telles que quantitative déroulant révèle la force de l’interaction, étudier la cinétique de la liaison rapide permet de mieux comprendre comment l’interaction se produit et combien de temps chaque complexe peut exister. En outre, mesurer la cinétique d’une interaction en présence d’un facteur supplémentaire, comme un facteur d’échange de protéine ou d’une drogue, permet de révéler le mécanisme par lequel l’interaction est réglementée par l’autre facteur, fournissant des connaissances importantes pour la promotion de la recherche biologique et médicale. Nous décrivons ici un protocole de mesure de la cinétique de la liaison d’une protéine complexe qui a un taux élevé association intrinsèque et peut être dissocié rapidement par une autre protéine. La méthode utilise le transfert d’énergie de fluorescence resonance pour signaler la formation du complexe protéine in vitro, et il permet la surveillance de la liaison rapide et dissociation du complexe en temps réel sur un fluorimètre flux stoppé. À l’aide de ce test, les constantes de vitesse association et dissociation de la protéine complexe sont quantifiées.
Introduction
Activités biologiques sont effectuées en fin de compte par des protéines, plus de qui interagissent avec les autres pour des fonctions biologiques appropriées. En utilisant une approche computationnelle, le montant total des interactions protéine-protéine chez l’homme est estimé à ~ 650 0001et perturbation de ces interactions souvent conduit à des maladies2. En raison de leur rôle essentiel dans le contrôle des processus cellulaires et organismiques, plusieurs méthodes ont été développées pour l’étude des interactions protéine-protéine, comme deux-hybride de levure, complémentation de fluorescence bimoléculaire, split-luciférase complémentation et co-immunoprécipitation test3. Si ces méthodes sont bons à découvrir et confirmant les interactions protéine-protéine, ils sont généralement non quantitative et ainsi fournissent des informations limitées sur les affinités entre les partenaires de protéines qui interagissent. Pull-down quantitative peut être utilisé pour mesurer l’affinité de liaison (par exemple, la constante de dissociation Kd), mais elle ne mesure pas la cinétique de la liaison, ni peut il être appliqué lorsque le Kd est très faible en raison d’une insuffisante rapport signal-sur-bruit4. Plasmonique de surface (SPR) spectroscopie quantifie la cinétique de la liaison, mais il nécessite une surface spécifique et l’immobilisation d’un réactif à la surface, ce qui peut potentiellement modifier la propriété de liaison du réactif5. En outre, il est difficile pour la SPR mesurer rapidement association et dissociation taux5, et il n’est pas approprié d’utiliser la SPR pour caractériser l’événement de l’échange de sous-unités protéiques dans un complexe de protéines. Nous décrivons ici une méthode qui permet de mesurer le taux de protéine complexe montage et démontage à une échelle de temps de milliseconde. Cette méthode a été essentielle pour déterminer le rôle d’ullin -unssociated -Nedd8 -dissociated protéine C 1 (Cand1) que le F-box protéine échange facteur6,7.
Cand1 régule la dynamique des ligases de E3 des protéines (SCF) Skp1•Cul1•F-box, qui appartiennent à la grande famille d’ubiquitine ligases de Cullin-RING. Les CCS comprennent le cullin Cul1, qui se lie à la protéine de domaine RING Rbx1, et une protéine F-box interchangeable, qui recrute des substrats et lie Cul1 par l’intermédiaire de la protéine adaptatrice Skp18. Comme une ligase E3, SCF catalyse la conjugaison de l’ubiquitine sur son support, et il est activé lorsque le substrat est recruté par la protéine F-box et lorsque Cul1 est modifié par l’ubiquitine-comme la protéine Nedd89. Cand1 lie Cul1 non modifié, et lors de la liaison, il perturbe tant l’association de la protéine Skp1•F-box avec Cul1 que la conjugaison de Nedd8 Cul110,11,12,13. Ainsi, Cand1 semble être un inhibiteur de l’activité in vitro SCF, mais une carence en Cand1 dans les organismes causé défauts qui suggère un rôle positif de Cand1 dans la régulation des activités SCF dans vivo14,15,16 , 17. ce paradoxe a enfin expliqué par une étude quantitative qui a révélé les interactions dynamiques entre les protéines Cul1, Cand1 et Skp1•F-box. Utilisant la fluorescence resonance energy transfert (FRET) de détecter la formation de l’EFC et Cul1•Cand1 complexes, l’association et dissociation constantes de vitesse (ksur et koff, respectivement) ont été mesurée individuellement. Les mesures ont révélé que fois Cand1 et Skp1•F-boîte forme extrêmement serré complexe protéique avec Cul1, mais le khors du SCF est considérablement augmenté par Cand1 et augmente considérablement la khors de Cul1•Cand1 par Skp1•F-case protéine6,7. Ces résultats fournissent la prise en charge initiale et critique pour définir le rôle de Cand1 comme un facteur d’échange de protéine, qui catalyse la formation de nouveaux complexes SCF grâce au recyclage Cul1 depuis les vieux complexes SCF.
Ici, nous présentons la procédure de développement et l’utilisation de l’essai de frette pour étudier la dynamique du complexe Cul1•Cand17, et le même principe peut être appliqué afin d’étudier la dynamique des diverses biomolécules. FRETTE se produit lorsqu’un donneur est excité par la longueur d’onde appropriée et un accepteur avec spectre d’excitation qui se chevauchent le spectre d’émission de donneur est présent dans un rayon de 10-100 Å. L’état excité est transféré à l’accepteur, ce qui diminue l’intensité des donateurs et augmentant l’accepteur intensité18. L’efficacité du FRET (E) dépend de la distance entre les fluorophores donneur et accepteur (r) et le rayon de Förster (R,0) et est définie par : E = R06/ (R0 6 + r6). Le rayon de Förster (R0) dépend de quelques facteurs, y compris l’orientation angulaire du dipôle, le chevauchement spectral de la paire de donneur-accepteur et la solution utilisé19. Pour appliquer le test FRET sur un fluorimètre flux stoppé, qui surveille le changement de l’émission du donneur en temps réel et permet des mesures rapides ksur et koff, il est nécessaire d’établir la frette efficace qui entraîne une réduction significative des émissions de donateurs. Par conséquent, conception efficace frette en choisissant la paire appropriée de colorants fluorescents et sites sur les protéines cibles pour fixer les teintures est importante et est discutée dans le présent protocole.
Protocole
1. conception de l’essai de la frette.
- Télécharger le fichier de structure de la Cul1•Cand1 complexe de la Protein Data Bank (fichier 1U6G).
- Afficher la structure de la Cul1•Cand1 complexe à PyMOL.
- Utilisez la fonction de mesure sous le menu Assistant de PyMOL pour estimer la distance entre le premier acide aminé de Cand1 et le dernier acide aminé de Cul1 (Figure 1).
- Charger le spectateur de spectres en ligne (voir Table des matières) et l’affichage simultané de l’excitation et les spectres d’émission de 7-amino-4-méthylcoumarine (AMC) et FlAsH (Figure 2). Notez que AMC est le donateur de la frette et FlAsH est l’accepteur de la frette.
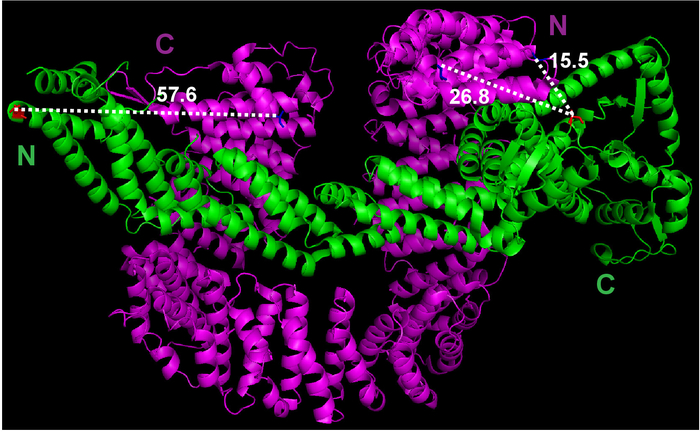
Figure 1 : la structure cristalline du Cul1•Cand1 et la mesure de la distance entre le potentiel étiquetage sites. Le fichier de structure cristalline a été téléchargé de Protein Data Bank (fichier 1U6G) et lus dans PyMOL. Mesures entre atomes sélectionnés ont été réalisés par PyMOL. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
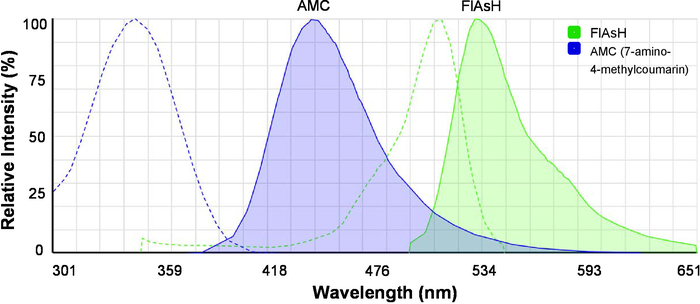
Figure 2 : l’excitation et les spectres d’émission des colorants fluorescents pour frette. Les spectres de l’AMC (7-amino-4-méthylcoumarine) et FlAsH sont indiqués. Les lignes pointillées indiquent les spectres d’excitation et les lignes pleines indiquent les spectres d’émission. L’image a été généré par la Fluorescence SpectraViewer et a été modifiée pour une meilleure clarté. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
2. préparation de Cul1AMC•Rbx1, la protéine de donateurs frette
- Construire des plasmides pour exprimer l’humaine Cul1sortase•Rbx1 en cellules d’Escherichia coli . Notez que les deux plasmides exprimant co Cul1•Rbx1 humaines en cellules d’Escherichia coli sont décrites en détail dans un précédent rapport20.
- Ajouter une séquence d’ADN codant « LPETGGHHHHHH » (sortase-His6 tag) à l’extrémité 3' de la séquence codante Cul1 par PCR standard et clonage méthodes21,22.
- Séquencer le nouveau plasmide pour confirmer l’insertion du gène est exacte.
- Express co Cul1sortase•Rbx1 en cellules d’Escherichia coli . La méthode est dérivée d’un précédent rapport20.
- Mélanger 100 ng de chacun des deux plasmides avec BL21 (DE3) cellules chimiquement compétents pour co transformation utilisant la chaleur aux chocs méthode23. La croissance de cellules sur gélose LB contenant 100 ampicilline µg/mL et au chloramphénicol de 34 µg/mL à 37 ° C pendant la nuit.
- Ensemencer à 50 mL de culture LB avec colonies fraîchement transformées et poussent en une nuit à 37 ° C sous agitation de 250 tr/min. Cela donne une culture starter.
- Ensemencer 6 flacons, chacune avec 1 L de milieu LB, avec culture starter 5 mL chacun et se développer à 37 ° C avec 250 tr/min, secouant jusqu'à ce que l' OD600 est ~ 1.0. Refroidir la culture à 16 ° C et ajouter isopropyl-β-D-thiogalactoside (IPTG) à 0,4 mM. Maintenir la culture à 16 ° C durant la nuit avec agitation de 250 tr/min.
- Récolter les cellules d’Escherichia coli par centrifugation à 5 000 x g pendant 15 min et collecter des granulés de cellule en tubes conique de 50 mL.
Remarque : Les granules cellulaires peuvent être traitées pour la purification de la protéine ou être congelés à-80 ° C avant d’effectuer les étapes de purification de protéines.
- Purification de le •Rbx1 desortasede Cul1 complexes. Cette méthode est dérivée d’un précédent rapport20.
- Ajouter 50 mL de tampon de lyse (30 mM Tris-HCl, 200 mM NaCl, 5 mM DTT, 10 % de glycérol, 1 comprimé d’inhibiteur de la protéase cocktail, pH 7,6) au culot de cellules d’Escherichia coli exprimant Cul1sortase•Rbx1.
- Lyse des cellules sur la glace avec la sonication à amplitude de 50 %. Alternez entre 1 seconde ON et OFF de 1 seconde et courir pendant 3 min.
- Répétez l’étape 2.3.2 x 2-3.
- Transférer le lysat cellulaire dans un tube de centrifugation de 50 mL et éliminer les débris cellulaires par centrifugation à 25 000 x g pendant 45 min.
- Incuber les cellules claires lysat avec 5 mL de glutathion perles à 4 ° C pendant 2 h.
- Centrifuger le mélange perles-lysat à 1 500 x g pendant 2 min à 4 ° C. Retirez le surnageant.
- Laver les billes avec 5 mL de tampon de lyse (avec sans inhibiteurs de la protéase) et enlever le surnageant après centrifugation à 1 500 g pendant 2 min à 4 ° C.
- Répétez l’étape 2.3.7 x 2.
- Ajouter 3 mL de tampon de lyse aux talons lavées et transférer le lisier de perle dans une colonne vide.
- Ajouter 5 mL de tampon d’élution (50 mM Tris-HCl, 200 mM NaCl, 10 mM réduit glutathion, pH 8,0) dans la colonne. Incuber pendant 10 minutes et recueillir l’éluat.
- Répétez l’étape 2.3.10 3-4 x.
- Ajouter 200 µL de thrombine 5 mg/mL (voir Table des matières) à l’éluat des perles de glutathion et incuber une nuit à 4 ° C.
Remarque : Le protocole peut être suspendu ici. - Diluer l’échantillon de protéine avec tampons A (25 mM HEPES, 1 millimètre DTT, 5 % de glycérol, pH 6,5) trois fois.
- Equilibrer une colonne de chromatographie échangeuse de cations (voir Table des matières) sur un système FPLC avec tampon A.
- Charger l’échantillon de protéines à la colonne de chromatographie échangeuse de cations équilibré à un débit de 0,5 mL/min.
- Éluer la protéine avec un gradient de NaCl dans 40 mL de mélange tampons A et 0 à 50 % tampon B (25 millimètres HEPES, 1 M NaCl, 1 millimètre DTT, 5 % de glycérol, pH 6,5) à un débit de 1 mL/min.
- Vérifier la protéine éluée dans différentes fractions à l’aide de SDS-PAGE24.
Remarque : Le protocole peut être suspendu ici. - Mettre en commun les fractions de l’éluat contenant Cul1sortase•Rbx1.
- Concentrer l’échantillon Cul1sortase•Rbx1 à 2,5 mL en passant le tampon à travers une membrane d’ultrafiltration (coupure de 30 kDa).
- Ajouter AMC à l’extrémité C-terminale de Cul1 via transpeptidation induite par la sortase21,22.
- Modifier la mémoire tampon dans l’échantillon de •Rbx1 Cul1sortasesortase dans la mémoire tampon (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7.5) en utilisant une colonne de dessalement (voir Table des matières).
- Equilibrer une colonne dessalement avec 25 mL de tampon de sortase.
- Charger 2,5 mL de Cul1sortase•Rbx1 échantillon dans la colonne. Jeter le cheminement.
- Éluer l’échantillon avec 3,5 mL de tampon de sortase. Recueillir le cheminement.
- Dans 900 µL de la solution de •Rbx1 desortasede Cul1 dans le tampon de la sortase, ajouter 100 µL de solution de A 600 µM purifiée sortase et 10 µL de peptide GirotAMC de 25 mM. Incuber le mélange réactionnel à 30 ° C dans la nuit sombre. Notez que cette étape va générer Cul1AMC•Rbx1.
Attention : Les colorants fluorescents sont sensibles à la lumière, afin d’éviter les exposant à la lumière ambiante lors de la préparation de protéine et l’échantillon autant que possible.
Remarque : Le protocole peut être suspendu ici. - Ajouter 50 µL de perles de Ni-NTA agarose dans le mélange réactionnel et incuber à température ambiante pendant 30 min.
- Les perles d’agarose Ni-NTA de granule par centrifugation à 5 000 x g pendant 2 min et recueillir le surnageant.
- Equilibrer une colonne de chromatographie d’exclusion taille (voir Table des matières) avec un tampon (30 mM Tris-HCl, 100 mM NaCl, 1 millimètre DTT, 10 % de glycérol) sur le système des FPLC.
- Charger tous les Cul1AMC•Rbx1 échantillon sur la colonne de chromatographie d’exclusion taille. Éluer avec 1,5 volume de colonne x du tampon.
- Vérifier les fractions de l’éluat par SDS-PAGE24.
- Mettre en commun les fractions de l’éluat contenant Cul1AMC•Rbx1.
- Mesurer la concentration de la protéine à l’aide de son absorbance à 280 nm avec un spectrophotomètre.
- Partie aliquote de la solution protéique et conserver à-80 ° C.
Remarque : Le protocole peut être suspendu ici.
- Modifier la mémoire tampon dans l’échantillon de •Rbx1 Cul1sortasesortase dans la mémoire tampon (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7.5) en utilisant une colonne de dessalement (voir Table des matières).
3. préparation du FlAsHCand1, la protéine d’accepteur de frette
NOTE : La plupart des étapes décrites dans la présente partie est les mêmes que l’étape 2. Conditions différentes sont décrites en détail ci-dessous.
- Construire le plasmide pour exprimer l’humaine TetraCysCand1 en cellules d’Escherichia coli .
- Ajouter la séquence d’ADN codant « CCPGCCGSG » (tétracystéine/TetraCys tag) avant le 15ème des acides aminés de Cand1 par régulière PCR25 (séquences d’amorces : TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG ; CTAACTAGTGTCCATTGATTCCAAG).
- Insérer le produit de la PCR dans un vecteur pGEX-4 t-2. Le plasmide pour confirmer l’insertion du gène est précis et dans le cadre de la séquence.
- Exprimer TetraCysCand1in e. coli cellules de la même manière que l’étape 2.2, sauf que le plasmide se transforme en cellules capables de Rosetta.
- Purification de TetraCysCand1 provenant des cellules d’Escherichia coli .
- Lyse des granulés de cellule d’e. coli et extrait TetraCysCand1using perles de glutathion. Ces étapes sont les mêmes que les étapes 2.3.1–2.3.12.
- Diluer l’éluat de protéine des perles du glutathion avec tampon C (50 mM Tris-HCl, 1 millimètre DTT, 5 % de glycérol, pH 7,5) par trois plis. Equilibrer une colonne chromatographie échangeuse d’anion (voir Table des matières) sur le système FPLC avec tampon C et charge l’échantillon dilué de protéine à un débit de 0,5 mL/min.
- Éluer la protéine avec un gradient de NaCl dans 40 mL de mélange tampon C et 0 à 50 % D tampon (50 mM Tris-HCl, 1 M NaCl, 1 millimètre DTT, 5 % de glycérol, pH 7,5) à un débit de 1 mL/min. Vérifiez la protéine éluée dans différentes fractions à l’aide de SDS-PAGE24et mettre en commun les fractions contenant TetraCysCand1. Notez que TetraCysCand1 a un plus grand volume de rétention que GST gratuit.
- Concentrer l’échantillon de la Cand1 de le TetraCysen faisant passer le tampon à travers une membrane d’ultrafiltration (coupure de 30 kDa).
- Equilibrer la colonne de chromatographie d’exclusion taille avec tampon de marquage (20 mM Tris-HCl, 100 mM NaCl, 2 mM TCEP, 1 mM EDTA, 5 % de glycérol) sur le système des FPLC. Charge TetraCysCand1 échantillon (500 µL chaque fois) et vérifier les fractions de l’éluat par SDS-PAGE24.
- Mettre en commun toutes les fractions contenant TetraCysCand1 et concentrer la protéine à ~ 40 µM en passant le tampon à travers une membrane d’ultrafiltration (coupure de 30 kDa). Estimation de la concentration de la protéine à l’aide de son absorbance à 280 nm. Stocker les protéines sous forme de 50 µL d’extraits à-80 ° C.
Remarque : Le protocole peut être suspendu ici.
- Préparation de Cand1 FlAsH.
- Ajouter 1 µL de solution FlAsH (voir Table des matières) à 50 µL de solution de Cand1 TetraCys.
- Bien mélanger et laisser incuber le mélange à température ambiante dans l’obscurité pendant 1-2 h obtenir FlAsHCand1.
Remarque : Le protocole peut être suspendu ici.
4. préparation de la Cand1, la protéine de chase frette
Remarque : Le protocole de préparation de protéines est similaire à l’étape 3, avec les modifications suivantes.
- Insérez la séquence codante de Cand1 pleine longueur dans le vecteur pGEX-4 t-2.
- Modifier la mémoire tampon utilisée à l’étape 3.3.5 vers un mémoire tampon contenant 30 mM Tris-HCl, 100 mM NaCl, 1 millimètre DTT, 10 % de glycérol.
- Éliminer les étapes 3.3.7 et 3.3.8.
5. tester et confirmer le dosage de la frette
- Préparer la frette tampon contenant 30 mM Tris-HCl, 100 mM NaCl, 0,5 mM DTT, ovalbumine de 1 mg/mL, pH 7,6 et utiliser à température ambiante.
- Tester la frette entre Cul1AMC•Rbx1 et Cand1 de FlAsHsur un fluorimètre.
- Dans 300 µL de tampon de la frette, ajoutez Cul1AMC•Rbx1 (frette donneur) à une concentration finale de 70 nM. Transvaser la solution dans une cuvette.
- Placez la cuve dans le porte-échantillon d’un fluorimètre. Exciter l’échantillon à 350 nm excitation et analyser les signaux d’émission de 400 nm à 600 nm par incréments de 1 nm.
- Répétez l’étape 5.2.1 mais changement Cul1AMC•Rbx1 à Cand1 FlAsH(accepteur de frette). Analyse de l’échantillon Cand1 FlAsHen utilisant la même méthode comme au point 5.2.2.
- (Facultatif) Exciter le Cand1 FlAsHà 510 nm excitation et numériser le signal d’émission de 500 nm et 650 nm.
- Dans 300 µL de tampon de frette, ajoutez les deux Cul1AMC•Rbx1 et Cand1 de FlAsHà une concentration finale de 70 nM. Analyser l’échantillon de la même manière comme au point 5.2.2.
- Confirmer la frette entre Cul1AMC•Rbx1 et Cand1 FlAsHen ajoutant la protéine chase (sans étiquette Cand1) (Figure 3).
- Dans 300 µL de tampon de la frette, ajoutez 70 nM Cul1AMC•Rbx1 et 700 nM Cand1. Analyser l’émission échantillon comme au point 5.2.2.
- Dans 300 µL de tampon de la frette, ajoutez 70 nM Cand1 FlAsHet 700 nM Cand1. Analyser l’émission échantillon comme au point 5.2.2.
- Dans 300 µL de tampon de la frette, ajoutez 70 nM Cul1AMC•Rbx1 et 70 nM Cand1 FlAsHet incuber l’échantillon à la température ambiante pendant 5 min. Puis ajouter 700 nM Cand1 et immédiatement après l’ajout, analyse l’émission échantillon comme au point 5.2.2). Notez que cette étape est semblable à l’étape 5.2.5.
- Dans 300 µL de tampon de frette, dans l’ordre ajouter 70 nM Cul1AMC•Rbx1, 700 nM Cand1 et 70 nM Cand1 FlAsH. Incuber les échantillons à la température ambiante pendant 5 min et scannez l’émission échantillon comme au point 5.2.2. Notez que c’est l’échantillon de chase (ligne verte dans la Figure 3).
6. mesurer la constante de vitesse d’association (ksur) de Cul1•Cand1
NOTE : Détails d’un fluorimètre flux stoppé de fonctionnement a été décrit dans un précédent rapport26.
- Préparer le fluorimètre arrêt d’écoulement pour les mesures.
- Allumez le fluorimètre arrêt d’écoulement selon les instructions du fabricant.
- La valeur de l’excitation lumineuse à 350 nm et utiliser un filtre passe-bande qui permet 450 nm émission pour passer la lumière et bloque l’émission lumineuse à 500-650 nm.
- Garder les valves d’échantillon à la position de la remplir et raccorder une seringue de 3 mL remplie d’eau. Lavez les deux seringues d’échantillons (A et B) avec de l’eau en déplaçant le disque échantillon de seringue et descendre plusieurs fois. Jetez toute l’eau utilisée dans cette étape.
- Garder les valves d’échantillon à la position de la remplir et raccorder une seringue de 3 mL remplie avec le tampon de la frette. Lavez les deux seringues d’échantillon avec le tampon de la frette en déplaçant le disque échantillon de seringue et descendre plusieurs fois. Jeter tout le tampon de frette utilisé dans cette étape.
- Prendre une mesure de contrôle (Figure 4).
- Connecter une seringue de 3 mL et charger seringue A avec 100 nM Cul1AMC•Rbx1 dans le tampon de la frette. Tournez la vanne de prélèvement sur le lecteur .
- Connecter une seringue de 3 mL et charger la seringue B avec le tampon de la frette. Tournez la vanne de prélèvement sur le lecteur .
- Utiliser le Panneau de configuration sous acquérir dans le logiciel pour tirer cinq coups de feu (mélanger un volume égal d’échantillons de seringue A et B de la seringue) sur le fluorimètre flux stoppé sans enregistrement des résultats.
- Ouvrez le Panneau de configuration sous acquérir dans le logiciel et programme pour enregistrer l’émission de Cul1AMC sur 60 s. Puis prendre un seul coup.
- Répétez l’étape 6.2.4 x 2.
- Tournez la vanne de prélèvement sur le remplir . Vider la seringue B et laver avec le tampon de la frette.
- Mesure a observé les constantes de vitesse d’association (kobs) de Cul1•Cand1 (Figure 4 b).
- Conserver l’échantillon dans la seringue A les mêmes comme au point 6.2.1.
- Connecter une seringue de 3 mL et charger seringue B avec 100 nM Cand1 FlAsHdans le tampon de la frette. Tournez la vanne de prélèvement sur le lecteur .
- Utiliser le Panneau de configuration sous acquérir dans le logiciel pour tirer cinq coups de feu sans enregistrement des résultats.
- Ouvrez le Panneau de configuration sous acquérir dans le logiciel et programme pour enregistrer l’émission de Cul1AMC sur 60 s. Puis prendre un seul coup.
- Répétez l’étape 6.3.4 x 2.
- Vider la seringue B et laver avec le tampon de la frette.
- Répétez les étapes 6.3.1–6.3.6 plusieurs fois avec des concentrations croissantes de FlAsHCand1 dans le tampon de la frette.
- S’adapter à la variation (diminution) en signaux fluorescents mesurées au fil du temps de chaque tir pour une seule courbe exponentielle. Cela donnera kobsdans chaque mesure, et l’unité s-1. Notez que la base de ce calcul a été bien discutée dans un précédent rapport27.
- Calculer la moyenne et l’écart type kobspour chaque concentration de Cand1 FlAsHutilisée. Tracer moyenne kobscontre Cand1 concentration (Figure 4), et la pente de la droite représente le ksur des Cul1•Cand1, avec une unité de M-1 s-1.
7. mesurer la constante de vitesse de dissociation (koff) de Cul1•Cand1 en présence de la protéine Skp1•F-box.
Remarque : Cette étape est similaire à l’étape 6, avec les modifications suivantes.
- Dans la seringue A, en vertu de la position de combler , chargez une solution de 100 nM Cul1AMC•Rbx1 et 100 nM Cand1 FlAsHdans le tampon de la frette. Mettre la vanne de prélèvement en position « DRIVE ».
- Seringue b, sous le poste à combler , chargez une solution de Skp1•Skp2 (préparé suite à un précédent rapport20). Mettre la vanne de prélèvement en position « DRIVE ».
- Ouvrez le Panneau de configuration sous acquérir dans le logiciel et programme pour enregistrer l’émission de Cul1AMC plus de 30 s. Puis prendre un seul coup. Les signaux fluorescents augmentent au fil du temps après le mélange des solutions de seringue A et B (Figure 5) de la seringue.
Résultats
Pour tester la frette entre Cul1AMC et FlAsHCand1, nous avons d’abord déterminé l’intensité des émissions de 70 nM Cul1AMC (le donateur) et 70 nM Cand1 FlAsH(accepteur), respectivement (Figure 3 a-C, bleu lignes). Dans chaque analyse, seulement un pic d’émission était présent et l’émission de FlAsHCand1 (accepteur) était faible. Lorsque 70 nM chaque de Cul1AMC ...
Discussion
FRETTE est un phénomène physique qui est d’un grand intérêt pour l’étude et la compréhension des systèmes biologiques,19. Nous présentons ici un protocole pour les essais et à l’aide de frette pour étudier la cinétique de la liaison de deux protéines qui interagissent. Lorsque vous concevez la frette, nous avons examiné trois facteurs principaux : le recouvrement spectral entre donneur d’émission et accepteur d’excitation, la distance entre les deux fluorophores et l’ori...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Shan Shu-UO (California Institute of Technology) pour discussion perspicace sur le développement de l’essai de la frette. M.G. et Y.Z. X.L. ont été financés par des fonds de démarrage de l’Université Purdue à Y.Z. et X.L.This a été financée en partie par une subvention de démarrage de Purdue University Center dans biologie végétale.
matériels
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
Références
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon