Method Article
Assemblage de Nanotiges or dans Metamolecules plasmoniques chiraux, à l’aide de modèles d’Origami ADN
Dans cet article
Résumé
Les auteurs décrivent le protocole détaillé pour l’assemblage d’axée sur l’origami en ADN des nanotiges en or en chiraux plasmoniques metamolecules avec forte chiroptiques réponses. Le protocole n’est pas limité aux configurations chirale et peut être facilement adapté pour la fabrication de diverses architectures plasmoniques.
Résumé
L’adressabilité inhérente de structures origami ADN rend les modèles idéales pour l’organisation de nanoparticules métalliques dans des nanostructures plasmoniques complexes. La haute précision spatiale d’un assembly basé sur un modèle origami ADN permet de contrôler le couplage entre les résonances plasmoniques des particules individuelles et permet d’adapter les propriétés optiques des nanostructures construites. Récemment, les systèmes plasmoniques chirales a attiré beaucoup d’attention en raison de la forte corrélation entre la configuration spatiale des assemblys plasmoniques et leurs réponses optiques (p. ex., le dichroïsme circulaire [CD]). Dans ce protocole, nous décrivons le workflow entier pour la production d’ADN axée sur l’origami chirale assemblées des nanotiges en or (AuNRs). Le protocole inclut une description détaillée des principes de conception et les procédures expérimentales pour la fabrication des gabarits d’origami ADN, la synthèse de AuNRs et l’assemblage des structures de l’origami-AuNR. En outre, la caractérisation des structures à l’aide de la microscopie électronique à transmission (TEM) et spectroscopie de CD est incluse. Le protocole décrit n’est pas limité aux configurations chirale et peut être adapté pour la construction de diverses architectures plasmoniques.
Introduction
ADN nanostructures, origami ADN en particulier, ont été largement utilisées pour organiser des molécules et des autres composants nanométriques (par exemple, les protéines et les nanoparticules [IP]), avec précision nanométrique dans des géométries presque arbitraire1,2 , 3 , 4 , 5. la capacité à organiser de NPs métalliques sur les modèles d’origami ADN avec un rendement élevé et la précision permet la fabrication de structures plasmoniques avec nouvelles propriétés optiques6,7,8, 9 , 10. technique de l’origami ADN s’avère particulièrement utile pour la génération de structures plasmoniques chiraux, nécessitant des architectures véritablement tridimensionnelle11,12,13, 14 , 15 , 16 , 17 , 18 , 19 , 20.
Ce protocole décrit en détail l’ensemble du processus de la fabrication d’assemblages chiraux modèles origami ADN AuNRs. Le logiciel utilisé pour le design21 et structure prediction22,23 de l’origami ADN est intuitive et disponible gratuitement. La fabrication de l’origami et la synthèse de AuNR utilisent commune matériel de laboratoire de biochimie (p. ex., thermocycleurs, électrophorèse, plaques chauffantes, centrifugeuses, etc.). Les structures sont caractérisés par spectroscopie de TEM et CD standard.
La fabrication de nanostructures plasmoniques similaires avec des méthodes de haut en bas (par exemple, lithographie à faisceau d’électrons) nécessiterait l’équipement assez compliqué et coûteux. En outre, ADN origami modèles fournissent la possibilité d’incorporer la reconfigurabilité structurelle dans les assemblys plasmoniques24,25,26,27,28,29 ,30,31,32,33, qui est extrêmement difficile pour les structures fabriquées avec des techniques de lithographie. Par rapport aux autres approches moléculaires34,35,36,37, fabrication de base origami ADN fournit un niveau élevé de précision spatiale et programmabilité.
Protocole
1. conception de l’origami ADN
- Identifier la disposition spatiale relative souhaitée de AuNRs et la forme appropriée de la matrice d’origami ADN (Figure 1A). Estimer les paramètres structuraux de la AuNRs et les modèles d’origami. Localiser la position approximative d’agrafes nécessitant de nouveaux modification (Figure 1B).
- Téléchargez et installez caDNAno18 pour concevoir un modèle d’origami ADN. Dans caDNAno, faites passer les brins échafaudage et agrafe selon la forme désirée du modèle et générer la séquence de brins discontinues en cliquant sur Outil Seq. Cliquez sur Outil de peinture et marquer les brins discontinues qui nécessitent davantage de modification (Figure 1C).
- Cliquez sur l' Outil d’exportation pour exporter les séquences discontinues (Figure 1C) vers un fichier .csv.
- Concevoir des écluses doubles brins pour fixer l' angle Θ entre les deux forfaits origami. Selon l’orientation relative des deux faisceaux, la construction de l’origami peut adapter left ou right handed (LH/RH) chiral configuration spatiale (Figure 1B).
- Importer fichier .csv des agrafes dans un tableur. Ajouter une séquence de10 polyA à la fin des agrafes utilisées pour l’assemblage AuNR (poignées). Modifier les brins discontinues sur les sites de verrouillage conçus avec des séquences de verrouillage.
Remarque : Les assemblys dans les résultats représentatifs contiennent 36 poignées en saillie à l’extrémité 3' des brins discontinues, 18 sur chaque forfait origami ADN, répartie sur deux hélices en parallèle chaque 21 nt. La distance entre la première et la dernière position de la poignée est 168 nt, environ 57 nm (voir le fichier ci-joint caDNAno).
2. montage des modèles origami ADN
- Préparation d’un stock de travail des brins discontinues (SM), y compris les brins avec poignées et serrures, en mélangeant des quantités égales d’oligonucléotides discontinues concentration normalisée (par exemple, 100 µM).
Remarque : Les structures Origami contiennent généralement plusieurs centaines de brins discontinues. Agrafes sont généralement achetés auprès de fournisseurs spécialisés dans la synthèse chimique d’oligonucléotides d’ADN en multiwell (p. ex., 96 puits) plaques. - Pour 500 µL de 10 origami nM, mélanger 50 µL de Tris-EDTA (TE, 10 x), de 100 µL de MgCl2 (100 mM), de 25 µL de NaCl (100 mM), 175 µL d’H2O, 100 µL de SM (0,5 µM), 5 µL de brins de verrouillage (5 µM) et un échafaudage 50 µL (100 nM).
- Recuire le mélange dans un thermocycleur de 80 ° C à 20 ° C comme indiqué dans le tableau 1.
3. purification d’origami ADN
Remarque : Cette section décrit le protocole pour la purification de gel d’agarose. Modèles d’origami ADN peuvent également être purifiés à l’aide de moyens38,,39.
- Pour 1 % gel, dissoudre 1 g d’agarose dans 100 mL de Tris-borate-EDTA (TBE, 0,5 x) en chauffant le mélange dans un four à micro-ondes. Ajouter 10 µL de 10 000 x ADN tache conformément à la spécification de la tache. Pour minimiser l’exposition aux rayons ultraviolets à l’étape d’extraction (étape 3.6), utiliser une tache d’ADN qui peut être visualisée sous excitation bleue.
- Refroidir la solution à environ 40 ° C et ajouter lentement 1 mL de MgCl2 (1,3 M) et en agitant. Monter le gel et incuber 30 min à température ambiante.
- La valeur de l’appareil d’électrophorèse et verser froid (4 ° C) tampon en cours d’exécution (0,5 x TBE avec 11 mM MgCl2) dans la boîte de gel. Placez la zone de gel dans un bain d’eau glacée.
- Tampon de charge s’ajoute les échantillons d’origami (6 x chargement mémoire tampon contient 15 % polysucrose 400 et 0,25 % de bleu de bromophénol dans l’eau). Charger les échantillons dans les puits avec un bon volume selon le peigne utilisé (par ex., 50 µL pour un peigne de 8 puits de 1,5 mm d’épaisseur).
- Exécutez l’électrophorèse pendant 2 h à 80 V.
Remarque : Pour caractériser l’origami et séparer la structure ouverte et fermée, utiliser un gel 2 % au lieu de 1 % et prolonger la durée de 4 h. - Image le gel avec l’imageur de gel (Figure 2). Un transilluminateur lumière bleu permet de visualiser les bandes, couper la bande origami, casser le gel sur une parafilm et extraire le liquide. Le rendement de récupération est d’environ 40 %.
- Pipeter le liquide dans une unité de filtration centrifuge et essorage à 3 000 x g pendant 5 min. mesurer l’absorption de la solution d’origami à 260 nm avec un spectromètre UV-visible (UV-VIS). Estimation de la concentration de l’origami en utilisant un coefficient d’extinction de 1,3 x 108 M-1·cm-1.
Remarque : La concentration typique d’origami solution après purification de gel agarose est : 1-2. - Stocker les modèles d’origami purifiée à 4 ° C pour une utilisation ultérieure.
4. synthèse des nanotiges en or
Remarque : Le protocole pour la synthèse de AuNR est adapté du précédent littérature40 avec des modifications mineures.
- Laver toute la verrerie avec eau régale pendant 5 min, rincer à l’eau, laisser agir il avec de l’eau ultrapure et sécher avant utilisation.
- Préparer le bromure d’hexadécyltriméthylammonium 0,2 M (CTAB), 1 mM HAuCl4, 4 mM AgNO3, 64 mM L (+)-acide ascorbique et 6 mM NaBH4. Utilisez l’eau froide (4 ° C) pour dissoudre NaBH4 et conservez-le au réfrigérateur à 4 ° C. Solution d’acide ascorbique doit être fraîchement préparé.
Attention : CTAB est dangereux en cas de contact cutané (irritant), contact avec les yeux (irritant), ingestion et inhalation. Porter des vêtements protecteurs appropriés. En cas de ventilation insuffisante, porter un appareil respiratoire approprié. NaBH4 est extrêmement dangereux en cas de contact cutané (irritant), contact avec les yeux (irritant), ingestion et inhalation. Portez anti-éclaboussures, une blouse de laboratoire, des gants et un appareil respiratoire de vapeur et la poussière. N’oubliez pas d’utiliser un respirateur approuvé/accrédité ou l’équivalent. - Préparer les graines de l’UA.
- Ajouter 500 µL de CTAB (0,2 M), 250 µL d’eau ultrapure et 250 µL de HAuCl4 (1 mM) dans un flacon en verre. Remuer à 450 tr/min à température ambiante pendant 5 min.
- Augmenter la vitesse en remuant à 1 200 tr/min. Ajouter 100 µL de solution de4 NaBH froide (6 mM, 4 ° C). Après 2 min, arrêter l’agitation et incuber la solution dans un bain d’eau à 30 ° C pendant 30 min avant utilisation.
- Préparer AuNRs.
- Dissoudre 0,55 g de CTAB et 0,037 g d’acide de 2, 6-dihydroxybenzoïque dans 15 mL d’eau chaude (60-65 ° C) dans un ballon à fond rond. Refroidir la solution à 30 ° C, ajouter 600 µL de AgNO3 (4 mM) et remuer à 450 tr/min pendant 2 min. Ensuite, laisser la solution reposer pendant 15 min à 30 ° C.
- Ajouter 15 mL d’HAuCl4 (1 mM) à la solution et remuer à 450 tr/min pendant 15 min. ajouter 120 µL de L (+)-acide ascorbique (64 mM), puis, immédiatement, mélanger à 1 200 tr/min pendant 30 s. ajouter 12 µL de graines Au et garder en remuant à 1 200 tr/min pendant 30 s.
- Incuber la solution dans un bain d’eau à 30 ° C pendant 18 heures. Ne pas déranger la solution et utiliser un bouchon pour refermer le flacon.
- Transférer la solution qui en résulte dans les tubes à centrifuger et centrifuger à 9 500 x g pendant 12 min à 20 ° C. Jeter le surnageant, disperse le culot dans 20 mL d’eau ultrapure et effectuer une étape de centrifugation plus.
- Disperse le granule final dans 3 mL d’eau distillée. Estimation de la concentration d’un mesurage de l’absorption UV-visible à l’aide du coefficient d’extinction pour la résonance de plasmon longitudinale AuNRs. Le coefficient d’extinction peut être prédite à l’aide de paramètres AuNR forme41. Stocker le AuNRs à 4 ° C pour une utilisation ultérieure.
5. la fonctionnalisation des nanotiges en or avec l’ADN monocaténaire
Remarque : Cette section décrit le protocole pour la fonctionnalisation AuNR avec l’ADN simple brin (ssDNA), suivant la route dite faible pH adaptée du précédent littérature42. Les AuNRs recouverts d’ADN sont purifiés par centrifugation ; Alternativement, la purification peut être effectuée à l’aide de gel d’agarose.
- Incuber 20 µL de brins d’ADN-thiol fonctionnalisées Lamb (1 mM) avec 20 µL de fraîchement préparés tris(2-carboxyethyl) chlorhydrate de phosphine (TCEP, 14 mM) pendant 1 h afin de réduire les ponts disulfures.
NOTE : La forme de groupes thiol liaisons avec AuNRs, et la séquence de Lamb s’hybride avec la poignée de10 polyA sur l’origami, dans lequel trop ou trop peu de paires de base peut conduire à un dysfonctionnement ou un assembly instable.
Attention : PTCE peut causer des brûlures graves de la peau et des lésions oculaires. Porter des gants de protection/protection vêtements/protection/visage lunettes. - Mix 150 µL de AuNRs (10 nM) et 40 µL de thiol-ADN PTCE-traitée (0,5 mM). Ajouter 1 % dodécylsulfate de sodium (SDS) à la solution AuNR pour atteindre une concentration finale de SDS de 0,05 %. Ajuster le pH à 2,5-3 ~ 1 µl de HCl (1 M).
- Incuber pendant 2 h et en agitant à 70 tr/min.
NOTE : Le rapport AuNR-à-ADN devrait être l’ordre de 1:5, 000-15 000, selon la taille des tiges. Pour les AuNRs (70 nm x 30 nm) préparé suivant le protocole décrit à l’article 4, un 13 000 excès de thiol-ADN est recommandé. - Ajouter NaCl pour parvenir à une concertation finale de NaCl de 0,5 M et incuber pendant 4 heures à température ambiante en agitant à 70 tr/min.
Remarque : Un changement de couleur à cette étape peut indiquer une fonctionnalisation d’ADN ayant échouée. - Ajuster le pH à ~8.5 avec tampon TBE (10 x) et incuber pendant la nuit.
- Laver l’ADN-AuNRs 4 x en mélangeant les échantillons avec 1 mL de tampon de lavage (0,5 x TBE avec SDS 0,1 %) et centrifuger à 7 000 x g pendant 30 min. Retirez le surnageant et remettre en suspension l’ADN-AuNRs dans le liquide restant (~ 40 µL). Estimation de la concentration d’ADN-AuNRs d’un mesurage de l’absorption UV-VIS comme indiqué au point 4.4.5.
Remarque : La solution peut devenir légèrement « trouble » à pas 5.3 et 5.4 en raison du remplacement de CTAB de la surface de la AuNRs de thiol-ADN. La solution devienne claire sur le réchauffement de la planète jusqu'à environ 35 ° C pendant 5 min.
6. montage des nanotiges en or sur les modèles d’origami ADN
- Ajouter MgCl2 à la solution d’ADN purifié-AuNRs, à une concentration finale de 10 mM. Mélanger l’ADN-AuNRs purifiée et les origami d’un ratio de 10:1.
NOTE : Un rapport plus faible peut diminuer le rendement de produit43. - Recuire le mélange dans un mixeur avec un contrôle de la température de 40 ° C à 20 ° C en agitant à 400 tr/min, la procédure décrite dans le tableau 2.
Remarque : Pour la caractérisation de la CD, l’échantillon peut être mesuré après cette étape sans plus de purification. - Électrophorèse sur gel d’agarose de 0,7 % (3,5 h à 80 V) permet de purifier les structures de l’origami-AuNR final.
- Utilisez un transilluminateur blanc léger pour l’imagerie. Couper la bande produit (origami-AuNR dimère) (Figure 3), briser le gel sur une parafilm et extraire le liquide. Pipette de liquide dans une unité de filtration centrifuge et un essorage à 3 000 x g pendant 5 min. remettre en suspension l’origami-AuNRs dans la solution. Le rendement de récupération du gel est d’environ 50 %.
- Estimation de la concentration des structures origami-AuNR d’un mesurage de l’absorption UV-VIS comme indiqué au point 4.4.5.
7. imagerie de microscopie électronique de transmission
Remarque : Cette formiate d’uranyle (UFo) souillant le protocole est adapté du précédent littérature44.
- Mix 200 µL de solution d’OVNI (0,75 %) et 1 µL de NaOH (5 M) et vortex immédiatement pour 2-3 min. centrifuger la solution tache pendant 3-4 min à 14 000 x g. Protéger la tache de l’exposition à la lumière (par exemple, en l’enveloppant dans du papier d’aluminium).
Attention : UFo est toxique si inhalé ou avalé et peut causer une irritation des yeux. Dans le cas d’une exposition brève ou peu polluants, utiliser un dispositif de filtration respiratoire. En cas d’exposition intensive ou plue, utiliser un appareil de protection respiratoire autonome. Porter des gants. Le matériau des gants doit être imperméable et résistant aux OVNI et ses solutions. Porter des lunettes très hermétiques. - Décharge luminescente carbone/formvar-enduit des grilles TEM pour 6 s juste avant utilisation pour augmenter hydrophilicité et promouvoir l’égorgement des structures. Pipetter 5 µL échantillon gouttes sur la grille TEM, incuber pendant 5-8 min et enlever la goutte en appuyant doucement sur un papier filtre avec le bord de la grille.
- Pipetter un grand (~ 20 µL) et une petite (~ 10 µL) goutte de la solution de tache sur un parafilm. Mettre la grille sur la goutte de solution de petite tache et séchez immédiatement en touchant le papier filtre avec le bord de la grille. Ensuite, mettez-le sur la goutte de solution de grosse tache pendant 30 s.
- Retirer le liquide sur la grille en touchant le papier filtre avec le bord de la grille. Placer la grille dans le porte-grille. Attendez que la grille à sécher pendant au moins 10 min.
- Caractériser les échantillons d’origami-AuNRs (Figure 6) par TEM origami (Figure 4) et AuNRs (Figure 5).
8. mesure de dichroïsme circulaire
- Purger le spectromètre CD avec N2 pendant 20 min.
Remarque : La plupart des spectromètres CD requièrent purge avec N2 avant l’allumage de la lampe. Vérifiez le manuel du spectromètre CD. - Définir la bande passante, plage de balayage et étape de l’acquisition.
NOTE : La plage de balayage dépend des propriétés optiques de AuNRs, qui dépendent de la taille de la AuNRs. - Mesure le CD vierge avec un tampon.
- Mesurer les spectres CD d’origami-AuNR échantillons (Figure 7).
Remarque : Utilisez pintes ou cuvettes de verre pour la mesure de la CD. Cuvettes en plastique ne conviennent pas pour la spectroscopie de CD. Aussi, la plupart des spectromètres de CD permettent l’acquisition simultanée de l’absorption et CD de données.
Résultats
Images TEM de modèles d’origami ADN, AuNRs et final origami-AuNR assemblées sont indiquées dans la Figure 4, Figure 5et Figure 6A, respectivement. En raison de leur préférence de liaison aux grilles TEM, origami-AuNR assemblées sont généralement vus comme faisceaux parallèles origami et tiges (Figure 6A). Recuit thermique est nécessaire pour l’alignement correct des AuNRs sur les modèles d’origami (Figure 6A, B). Le protocole permet des rendements élevés de l’assemblage de AuNRs dans metamolecules chiraux avec de fortes réponses de CD plasmoniques (Figure 7).
| Température (° C) | Temps |
| 80 | 15 min |
| 79 - 71 | 1 ° C/1 min |
| 70 - 66 | 1 ° C/5 min |
| 65 - 60 | 1 ° C/30 min |
| 59 - 37 | 1 ° C/60 min |
| 36 - 30 | 1 ° C/15 min |
| 29 - 20 | 1 ° C/5 min |
| 20 | Maintenez |
Tableau 1 : Les températures et les tarifs pour le recuit des gabarits d’origami ADN.
| Température (° C) | Temps (min) |
| 40 | 130 |
| 36 | 180 |
| 32 | 180 |
| 22 | Maintenez |
Tableau 2 : températures et durées de détention pour le recuit des modèles d’origami ADN et AuNRs. Le taux de refroidissement entre les échelons est fixé à 0,1 ° C/min. Les échantillons d’ADN origami-AuNR sont recuits en agitant à 400 tr/min.

Figure 1 : Conception d’ADN basé sur un modèle origami chiral metamolecules. (A) identifier la disposition spatiale relative souhaitée des nanotiges en or (AuNRs) et une forme convenable de la matrice d’ADN origami. (B) estimer les paramètres structuraux de la AuNRs (DAuNR, LAuNR) et le modèle d’origami (origamide la W, Lorigami, Θ). Localiser la position approximative des agrafes qui ont davantage besoin de modification. (C) conception des gabarits d’origami ADN à l’aide de caDNAno. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : L’électrophorèse de gel d’agarose de l’origami. Purification (A) avec 1 % gel d’agarose pendant 2 h à 80 c. caractérisation (B) avec 2 % gel d’agarose de 4 h à 80 c. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
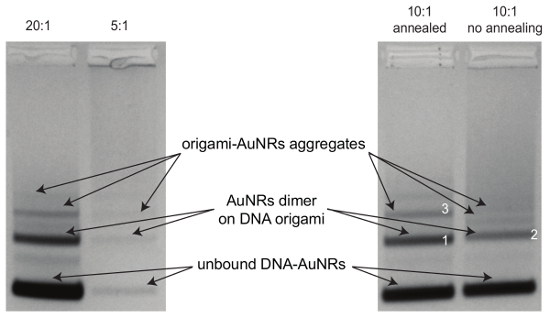
Figure 3 : La purification de l’électrophorèse sur gel d’agarose d’origami-AuNRs. Gel (0,7 %) a été exécuté pendant 3,5 h à 80 V pour les échantillons préparés conformément à la procédure de l’Assemblée avec les différents rapports d’ADN-AuNR-à-origami (20:1, 5:1) et (rapport d’ADN-AuNRs-à-origami de 10:1) avec ou sans procédure de recuit. Pour les images TEM des échantillons dans les bandes 1, 2 et 3, voir la Figure 6. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : Image de TEM représentant des modèles origami ADN. La structure de l’origami se compose de deux faisceaux 14-hélice (80 nm x 16 nm x 8 nm) reliés entre eux par le brin d’échafaudage. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 5 : Image représentant TEM de la AuNRs. Les dimensions moyennes de AuNRs synthétisés sont 70 x 30 nm. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : Images TEM des assemblées de l’origami-AuNR. (A) AuNR dimères sur origami après recuit (bande 1 sur la Figure 3). (B) AuNR dimères sur origami sans recuit (bande 2 dans la Figure 3). (C) Origami-AuNR agrégats (bande 3 à la Figure 3). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
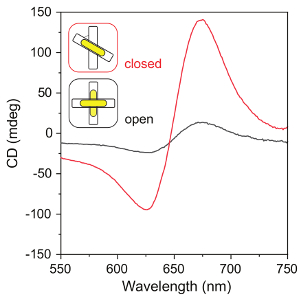
Figure 7 : Spectres CD des assemblées origami-AuNR. Les spectres de DC des structures fermées (les modèles d’origami fixés par blocage brins dans une configuration droite, avec 50° entre deux faisceaux d’origami) et la structure ouverte (les modèles d’origami sans serrure brins). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Le protocole introduit le workflow entier de conception, assemblage, purification et caractérisation d’ADN axée sur l’origami chirale assemblées de AuNRs. Les modèles d’origami ADN utilisés dans le protocole sont particulièrement adaptés pour la fabrication d’assemblages sensibles aux stimuli. Différents types de réponses et functionalizes peuvent être incorporés dans les brins de verrouillage qui définissent l’état chiraux de l’origami gabarit (Figure 1B)24,25,26,31. Pour les assemblys statiques, des modèles plus simples en forme de bloc sont souvent suffisamment14,45,46,47.
L’approche fondée sur les origami ADN la fabrication de nanostructures plasmoniques hérite des limitations de l’ADN origami technique48. La taille des modèles origami est généralement limitée par la taille de la mèche de l’échafaudage. La stabilité des structures de l’ADN est réduite dans les conditions de droit-sel. Le coût de l’aliment de base synthétique fils reste assez élevé. Toutefois, des développements récents dans le domaine de la nanotechnologie d’ADN structurelle devraient surmonter ces limitations49,50,51,52,,du5354 , 55.
Par rapport aux autres approches moléculaires pour générer des assemblys chiraux de AuNRs34,35,36,37, origami ADN fournit un niveau élevé de précision spatiale et programmabilité.
Pour parvenir à des réponses optiques fiables et reproductibles des assemblys chiraux, nous recommandons fortement d’adapter les protocoles pour AuNR synthèse40, puisque la qualité et les propriétés optiques des produits commerciaux peuvent varier entre les lots. Recuit (étape supplémentaire 6.2) est souvent cruciale pour assurer la fixation correcte de AuNRs aux modèles d’origami ADN (Figure 6).
Enfin, le protocole décrit ici n’est pas limité aux assemblys chiraux. Origami ADN fournit une plate-forme très flexible pour la fabrication de nanostructures plasmoniques complexes9,10.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs remercient Voutilainen S. pour son aide avec spectromètre de CD. Les auteurs reconnaissent la fourniture d’installations et de support technique de l’Université Aalto à OtaNano - Nanomicroscopy Center (NMC-Aalto). Ce travail a été soutenu par l’Académie de Finlande (308992 de concession) et programme de recherche et d’innovation Horizon 2020 de l’Union européenne sous le Marie Skłodowska-Curie convention no 71364 de subvention.
matériels
| Name | Company | Catalog Number | Comments |
| 2,6-Dihydroxybenzoic acid | Sigma-Aldrich | D109606-25 | 98+% |
| AgNO3 | Alfa Aesar | AA1141414 | 99.90% |
| Blue light transilluminator | Nippon Genetics | FG-06 | FastGene LED Transilluminator |
| Bromophenol Blue | Acros Organics | 403160050 | For agarorose gel loading buffer |
| Centrifugal filter units | Merck Millipore | 42600 | DNA extraction from agarose |
| Chirascan CD spectrometer | Applied Photophysics | ||
| Cuvette | Hellma | 105-202-85-40 | Quartz SUPRASIL |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Eppendorf Biospectrometer | Eppendorf | 6135000904 | |
| Eppendorf ThermoMixer C | Eppendorf | 5382000015 | |
| Ficoll 400 | Thermo Fisher Scientific | BP525-10 | Polysucrose 400 (For agarorose gel loading buffer) |
| Gel electrophoresis sets | Thermo Fisher Scientific | ||
| Gel imager | Bio-Rad | Gel Doc XR+ System | |
| HAuCl4•3H2O | Alfa Aesar | AA3640006 | 99.99% |
| HCl | Scharlau | AC07441000 | 1M |
| Hexadecyltrimethylammonium bromide (CTAB) | Sigma-Aldrich | H9151-100 | BioXtra, 98+% |
| L(+)-ascorbic acid | Acros Organics | 401471000 | 99+% |
| M13p7560 scaffold strand | Tilibit nanosystems | ||
| MgCl2•6H2O | Sigma-Aldrich | M2670-500 | BioXtra, 99+% |
| NaBH4 | Acros Organics | 200050250 | 99% |
| NaCl | Sigma-Aldrich | S7653-500 | BioXtra, 99.5+% |
| NaOH | Sigma-Aldrich | S8045-500 | BioXtra, 98+% |
| Parafilm | Sigma-Aldrich | P7668-1EA | PARAFILM M |
| PBS buffer (10X) | Thermo Fisher Scientific | BP3991 | Molecular Biology |
| ProFlex PCR System | Thermo Fisher Scientific | 4484073 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | 74255-250 | 99+% |
| Staple strands | Thermo Fisher Scientific | ||
| Sybr Safe | Invitrogen | S33102 | For DNA stain |
| TBE buffer (10X) | Invitrogen | 15581-044 | Molecular Biology |
| TE buffer (10X) | Thermo Fisher Scientific | BP24771 | Molecular Biology |
| TEM | FEI | FEI Tecnai F12 | |
| Thiol-functionalized ssDNA | Biomers.net | ||
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP-HCl) | Thermo Fisher Scientific | PI20491 | |
| UltraPure Agarose | Invitrogen | 16500-100 | |
| Ultrapure water (Type 1) | Milli-Q Direct 8 system | ||
| Uranyl Formate | Tebu-bio | 24762-1 | |
| White light transilluminator | UVP | TW-26 |
Références
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Wang, P., Meyer, T. A., Pan, V., Dutta, P. K., Ke, Y. The Beauty and Utility of DNA Origami. Chem. 2, 359-382 (2017).
- Hong, F., Zhang, F., Liu, Y., Yan, H. DNA Origami: Scaffolds for Creating Higher Order Structures. Chemical Reviews. 117, 12584-12640 (2017).
- Roller, E. -. M., Argyropoulos, C., Högele, A., Liedl, T., Pilo-Pais, M. Plasmon-Exciton Coupling Using DNA Templates. Nano Letters. 16, 5962-5966 (2016).
- Acuna, G. P., et al. Fluorescence Enhancement at Docking Sites of DNA-Directed Self-Assembled Nanoantennas. Science. 338, 506-510 (2012).
- Roller, E. -. M., et al. DNA-Assembled Nanoparticle Rings Exhibit Electric and Magnetic Resonances at Visible Frequencies. Nano Letters. 15, 1368-1373 (2015).
- Liu, Q., Song, C., Wang, Z. -. G., Li, N., Ding, B. Precise organization of metal nanoparticles on DNA origami template. Methods. 67, 205-214 (2014).
- Roller, E. -. M., et al. Hotspot-mediated non-dissipative and ultrafast plasmon passage. Nature Physics. 13, 761-765 (2017).
- Liu, N., Liedl, T. DNA-Assembled Advanced Plasmonic Architectures. Chemical Reviews. 118, 3032-3053 (2018).
- Kuzyk, A., Jungmann, R., Acuna, G. P., Liu, N. DNA Origami Route for Nanophotonics. ACS Photonics. 5, 1151-1163 (2018).
- Kuzyk, A., et al. DNA-based self-assembly of chiral plasmonic nanostructures with tailored optical response. Nature. 483, 311-314 (2012).
- Shen, X., et al. Three-Dimensional Plasmonic Chiral Tetramers Assembled by DNA Origami. Nano Letters. 13, 2128-2133 (2013).
- Liu, H., Shen, X., Wang, Z. -. G., Kuzyk, A., Ding, B. Helical nanostructures based on DNA self-assembly. Nanoscale. 6, 9331-9338 (2014).
- Shen, X., et al. 3D plasmonic chiral colloids. Nanoscale. 6, 2077-2081 (2014).
- Urban, M. J., et al. Plasmonic Toroidal Metamolecules Assembled by DNA Origami. Journal of the American Chemical Society. 138, 5495-5498 (2016).
- Hentschel, M., Schäferling, M., Duan, X., Giessen, H., Liu, N. Chiral plasmonics. Science Advances. 3, 1602735 (2017).
- Cecconello, A., Besteiro, L. V., Govorov, A. O., Willner, I. Chiroplasmonic DNA-based nanostructures. Nature Reviews Materials. 2, 17039 (2017).
- Lan, X., Wang, Q. Self-Assembly of Chiral Plasmonic Nanostructures. Advanced Materials. 28, 10499-10507 (2016).
- Shen, C., et al. Spiral Patterning of Au Nanoparticles on Au Nanorod Surface to Form Chiral AuNR@AuNP Helical Superstructures Templated by DNA Origami. Advanced Materials. 29, 1606533 (2017).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature Communications. 6, 8102 (2015).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37, 5001-5006 (2009).
- Kim, D. -. N., Kilchherr, F., Dietz, H., Bathe, M. Quantitative prediction of 3D solution shape and flexibility of nucleic acid nanostructures. Nucleic Acids Research. 40, 2862-2868 (2012).
- Maffeo, C., Yoo, J., Aksimentiev, A. De novo reconstruction of DNA origami structures through atomistic molecular dynamics simulation. Nucleic Acids Research. 44, 3013-3019 (2016).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13, 862-866 (2014).
- Kuzyk, A., et al. A light-driven three-dimensional plasmonic nanosystem that translates molecular motion into reversible chiroptical function. Nature Communications. 7, 10591 (2016).
- Kuzyk, A., Urban, M. J., Idili, A., Ricci, F., Liu, N. Selective control of reconfigurable chiral plasmonic metamolecules. Science Advances. 3, 1602803 (2017).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347, 1446-1452 (2015).
- Zhou, C., Duan, X., Liu, N. DNA-Nanotechnology-Enabled Chiral Plasmonics: From Static to Dynamic. Accounts of Chemical Research. 50, 2906-2914 (2017).
- Ijäs, H., et al. Dynamic DNA Origami Devices: from Strand-Displacement Reactions to External-Stimuli Responsive Systems. International Journal of Molecular Sciences. 19, 2114 (2018).
- Lan, X., et al. DNA-Guided Plasmonic Helix with Switchable Chirality. Journal of the American Chemical Society. 140, 11763-11770 (2018).
- Funck, T., Nicoli, F., Kuzyk, A., Liedl, T. Sensing Picomolar Concentrations of RNA Using Switchable Plasmonic Chirality. Angewandte Chemie International Edition. 57, 13495-13498 (2018).
- Zhou, C., Xin, L., Duan, X., Urban, M. J., Liu, N. Dynamic Plasmonic System That Responds to Thermal and Aptamer-Target Regulations. Nano Letters. 18, 7395-7399 (2018).
- Zhan, P., et al. Reconfigurable Three-Dimensional Gold Nanorod Plasmonic Nanostructures Organized on DNA Origami Tripod. ACS Nano. 11, 1172-1179 (2017).
- Ma, W., et al. Attomolar DNA detection with chiral nanorod assemblies. Nature Communications. 4, 2689 (2013).
- Ma, W., et al. Chiral plasmonics of self-assembled nanorod dimers. Scientific Reports. 3, 1934 (2013).
- Guerrero-Martínez, A., et al. Intense Optical Activity from Three-Dimensional Chiral Ordering of Plasmonic Nanoantennas. Angewandte Chemie International Edition. 50, 5499-5503 (2011).
- Kumar, J., et al. Detection of amyloid fibrils in Parkinson's disease using plasmonic chirality. Proceedings of the National Academy of Sciences. 115, 3225-3230 (2018).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53, 12735-12740 (2014).
- Shaw, A., Benson, E., Högberg, B. Purification of Functionalized DNA Origami Nanostructures. ACS Nano. 9, 4968-4975 (2015).
- Ye, X., et al. Improved Size-Tunable Synthesis of Monodisperse Gold Nanorods through the Use of Aromatic Additives. ACS Nano. 6, 2804-2817 (2012).
- Near, R. D., Hayden, S. C., Hunter, R. E., Thackston, D., El-Sayed, M. A. Rapid and Efficient Prediction of Optical Extinction Coefficients for Gold Nanospheres and Gold Nanorods. The Journal of Physical Chemistry C. 117, 23950-23955 (2013).
- Shi, D., Song, C., Jiang, Q., Wang, Z. -. G., Ding, B. A facile and efficient method to modify gold nanorods with thiolated DNA at a low pH value. Chemical Communications. 49, 2533-2535 (2013).
- Gür, F. N., Schwarz, F. W., Ye, J., Diez, S., Schmidt, T. L. Toward Self-Assembled Plasmonic Devices: High-Yield Arrangement of Gold Nanoparticles on DNA Origami Templates. ACS Nano. 10, 5374-5382 (2016).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature Methods. 8, 221-229 (2011).
- Lan, X., et al. Bifacial DNA Origami-Directed Discrete, Three-Dimensional, Anisotropic Plasmonic Nanoarchitectures with Tailored Optical Chirality. Journal of the American Chemical Society. 135, 11441-11444 (2013).
- Lan, X., et al. Au Nanorod Helical Superstructures with Designed Chirality. Journal of the American Chemical Society. 137, 457-462 (2015).
- Zhu, C., Wang, M., Dong, J., Zhou, C., Wang, Q. Modular Assembly of Plasmonic Nanoparticles Assisted by DNA Origami. Langmuir. , (2018).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature Nanotechnology. 6, 763-772 (2011).
- Ducani, C., Kaul, C., Moche, M., Shih, W. M., Högberg, B. Enzymatic production of 'monoclonal stoichiometric' single-stranded DNA oligonucleotides. Nature Methods. 10, 647-652 (2013).
- Praetorius, F., et al. Biotechnological mass production of DNA origami. Nature. 552, 84-87 (2017).
- Wagenbauer, K. F., Sigl, C., Dietz, H. Gigadalton-scale shape-programmable DNA assemblies. Nature. 552, 78-83 (2017).
- Zhang, T., et al. 3D DNA Origami Crystals. Advanced Materials. 30, 1800273 (2018).
- Ong, L. L., et al. Programmable self-assembly of three-dimensional nanostructures from 10,000 unique components. Nature. 552, 72-77 (2017).
- Ponnuswamy, N., et al. Oligolysine-based coating protects DNA nanostructures from low-salt denaturation and nuclease degradation. Nature Communications. 8, 15654 (2017).
- Agarwal, N. P., Matthies, M., Gür, F. N., Osada, K., Schmidt, T. L. Block Copolymer Micellization as a Protection Strategy for DNA Origami. Angewandte Chemie International Edition. 56, 5460-5464 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon