Method Article
Études ciblées utilisant le visage de bloc en série et la microscopie électronique focalisée de faisceau d'ion
Dans cet article
Résumé
Ici, nous présentons un protocole pour combiner efficacement la face de bloc de série et la microscopie électronique focalisée de faisceau d'ions pour cibler une zone d'intérêt. Cela permet une recherche efficace, en trois dimensions, et la localisation d'événements rares dans un grand champ de vision.
Résumé
Ce protocole permet l'imagerie efficace et efficace d'échantillons de cellules ou de tissus en trois dimensions au niveau de la microscopie électronique. Pendant de nombreuses années, la microscopie électronique (EM) est restée une technique intrinsèquement bidimensionnelle. Avec l'avènement des techniques d'imagerie par microscope électronique à balayage en série (volume EM), utilisant soit un microtome intégré, soit un faisceau d'ions focalisés pour trancher puis visualiser les tissus intégrés, la troisième dimension devient facilement accessible. Microscopie électronique à balayage de blocs en série (SBF-SEM) utilise un ultramicrotome enfermé dans la chambre SEM. Il a la capacité de manipuler de grands spécimens (1 000 m x 1 000 m) et d'imager de grands champs de vision à la petite taille de Pixel X, Y, mais est limité dans la dimension Z par le couteau de diamant. Le faisceau d'ions focalisé SEM (FIB-SEM) n'est pas limité en résolution 3D, (les voxels isotropes de 5 nm sont réalisables), mais le champ de vision est beaucoup plus limité. Ce protocole démontre un flux de travail pour combiner les deux techniques pour permettre de trouver des régions d'intérêt individuelles (ROI) dans un grand champ, puis d'imagerie du volume ciblé subséquent à haute résolution de voxel isotropique. La préparation de cellules ou de tissus fixes est plus exigeante pour les techniques em de volume en raison du contraste supplémentaire nécessaire pour la génération efficace de signal dans l'imagerie SEM. De tels protocoles prennent du temps et demandent beaucoup de main-d'œuvre. Ce protocole intègre également le traitement assisté par micro-ondes des tissus facilitant la pénétration des réactifs, ce qui réduit le temps nécessaire pour le protocole de traitement de jours à heures.
Introduction
Ce protocole décrit un flux de travail pour le ciblage efficace de la microscopie électronique tridimensionnelle à haute résolution (EM) vers une région d'intérêt spécifique (ROI). Depuis ses débuts dans les années 1930, EM a été une technique essentiellement bidimensionnelle. Les premières images publiées étaient des tissus entiers ou des cellules, mais qui ont rapidement cédé la place à des sections qui ont été coupées à la main à l'aide d'un ultramicrotome et photographiés à l'aide d'un microscope électronique de transmission (TEM). TEM produit des micrographies à très haute résolution où même les plus petites structures cellulaires sont clairement discernables. Cependant, la minceur de la section nécessaire pour que le tissu soit représenté par le faisceau d'électrons a rendu l'information dans la dimension Z minimale. Étant donné que les cellules sont des structures tridimensionnelles, les interactions entre les structures cellulaires et les surfaces cellulaires ont dû être déduites à partir de données limitées. Cela a soulevé le risque de mauvaise interprétation, en particulier dans les structures qui étaient complexes. Certains microscopistes ont réussi à obtenir des structures 3D plus précises par des cellules et des tissus sectionnants en série, puis en les reconstituant minutieusement à partir d'images TEM individuelles1. Il s'agissait d'un processus très laborieux et avant l'avènement de l'imagerie numérique et le rendu informatique, les résultats étaient également difficiles à visualiser. Ces dernières années, deux techniques ont été introduites qui sont devenues collectivement connues sous le nom de microscopie électronique de volume (volume EM)2 qui ont rendu l'EM en trois dimensions accessible à plus de laboratoires.
L'idée d'obtenir une pile d'images à partir d'un bloc intégré à l'intérieur d'un microscope électronique remonte à 1981, lorsque Steve Leighton et Alan Kuzirian ont construit un microtome miniature et l'ont placé dans la chambre d'un microscope électronique à balayage3 (SBF-SEM) . Ce prototype a finalement été copié et amélioré 23 ans plus tard par Denk et Horstmann4 et commercialisé par la suite. À peu près au même moment, les scientifiques biologiques ont pris connaissance d'une autre technologie utilisée principalement dans la science des matériaux, le faisceau d'ions ciblé. Cette technique utilise un faisceau d'ions d'une sorte (Gallium, plasma) pour enlever une très petite quantité de matériau de surface d'un échantillon (FIB-SEM)5. Les deux techniques utilisent la section suivie de l'imagerie fournissant une série d'images qui peuvent être combinées dans un X, Y, Z, pile. Les deux techniques fournissent des informations 3D mais à des échelles de résolution différentes. SBF-SEM est limité par les propriétés physiques du couteau diamant à des tranches pas plus minces que 50 nm pour les longues séries d'imagerie en série; cependant, la taille du bloc d'échantillon qui peut être sectionné est grande, jusqu'à 1 mm x 1 mm x 1 mm. En raison du grand format d'acquisition numérique du détecteur d'électrons dispersés arrière (32k x 32k pixels) qui reçoit un signal de la face de bloc , les tailles de pixels d'image peuvent être aussi petites que 1 nm. Il en résulte des voxels non isotropes où la dimension X,Y est souvent plus petite que la Z. En raison de la précision du faisceau d'ions, FIB-SEM a la capacité de collecter des images avec des voxels isotropes de 5 nm. Cependant, la superficie totale qui peut être imaginée est assez petite. Un tableau récapitulatif de divers échantillons et volumes photographiés avec les deux techniques a été publié précédemment3.
La préparation des tissus pour le volume EM est plus difficile que pour le TEM ou le SEM standard parce que les échantillons doivent être tachés pour fournir une génération de signal adéquate dans le SEM. Fréquemment, les colorations doivent être optimisées non seulement pour le type de tissu particulier, mais aussi pour l'ajout certaines structures cellulaires pour faciliter l'identification et la reconstruction. Le protocole utilisé ici est basé sur la norme NCMIR6. Une coloration supplémentaire signifie généralement des étapes de protocole supplémentaires. Ainsi, pour le volume EM, les protocoles standard doivent être étendus afin d'assurer suffisamment de temps pour que les réactifs pénètrent dans l'échantillon. Le traitement assisté par micro-ondes peut réduire le temps nécessaire pour la coloration de quelques heures à quelques minutes et rendre la préparation de l'échantillon EM de volume plus efficace7. Cette méthode s'applique à tous les types de cellules et de tissus8 et aux questions de recherche où l'inhomogénéité du tissu rend l'échantillonnage d'une zone spécifique essentielle9.
Une fois qu'une pile de données est obtenue, elle peut être alignée et les structures d'intérêt segmentées du reste des données et modélisées en 3D. Bien que l'automatisation de l'imagerie de nombreuses tranches de tissu a rendu l'acquisition d'image relativement simple, le processus de reconstruction numérique et de visualisation des données est une tâche longue. Les logiciels à cette fin ne sont pas encore intégrés ni entièrement automatisés. Étant donné qu'une grande partie des premiers travaux utilisant le volume EM était orientée vers les neurosciences, les techniques de coloration et de segmentation numérique des structures telles que les axones sont assez avancées par rapport à d'autres cellules et organites. Tandis que la littérature sur d'autres tissus non-neuronaux se développe rapidement, les structures non linéaires ou irrégulières exigent plus d'entrée manuelle.
L'utilisation de SBF-SEM et de FIB-SEM est une approche utile pour le ciblage et l'imagerie de structures tissulaires spécifiques et non homogènes à haute résolution en 3D. La combinaison de cela avec le traitement des tissus assistés par micro-ondes qui diminue considérablement le temps nécessaire à la préparation de l'échantillon. Ensemble, ce flux de travail fera de la génération de données d'image voxel isotropiques à haute résolution de structures fines un processus efficace et plus rapide.
Protocole
1. Fixation et traitement d'échantillon pour la microscopie électronique
- Fixer les semis d'Arabidopsis thaliana cultivés sur des plaques d'agar dans 0,5 % de paraformaldéhyde, 2,5 % de glutaraldéhyde dans un tampon de phosphate (PB) de 0,1 M (PB) pH 6,8 pour 2 h à température ambiante (RT).
CAUTION : Les aldéhydes sont irritants et corrosifs et ont un potentiel cancérogène, mutagène et tératogène. Toutes les solutions doivent être manipulées avec un équipement de protection approprié et dans une hotte de fumée. - Couper les extrémités des racines de la plante cultivée à l'étape 1.1 et mettre 2-3 pointes dans des tubes de 0,5 ml contenant le même fixatif pendant la nuit à 4 oC.
REMARQUE : Le volume de cette solution et de toutes les solutions dans les étapes restantes est déterminé par le volume de l'échantillon; un rapport minimum d'échantillon/solution est de 10:1. Les échantillons de plus de 1 mm dans n'importe quelle dimension seront difficiles à tacher, de sorte qu'il est plus difficile de travailler avec de plus gros blocs de tissus. Tous les tissus n'ont pas les mêmes caractéristiques; par exemple, les feuilles et les tiges des plantes peuvent être difficiles à tacher. Si des échantillons plus importants ou des types de tissus difficiles sont souhaités, l'optimisation du traitement de l'échantillon doit être effectuée avant de passer à la collecte de données. - Préparer la solution de thiocarbohydrazide (TCH), nécessaire frais et disponible avant l'étape 1.7. Ajouter 0,1 g de thiocarbohydrazide à 10 ml d'eau double distillée (ddH2O) et dissoudre en chauffant à 60 oC au four pendant 1 h. Avant l'utilisation, filtrez la solution TCH à l'aide d'un filtre à seringues de 0,22 m.
- Retirer le fixatif des tubes et le remplacer par un pH 6,8 de 0,1 M PB. Placez les tubes sur une table orbitale secouant à 100 tr/min et lavez-les pendant 10 min. Répétez le lavage à l'aide de PBS frais 5 fois.
- Post fixer les pointes de racine en remplaçant PB avec 2% de tétroxyde d'osmium (OsO4) et 0,2% rouge ruthénium dans 0,1 M PB pH 6,8. Mettre les tubes dans le four à micro-ondes avec les couvercles ouverts et commencer le programme 9 (tableau 1).
CAUTION: L'osmium est extrêmement dangereux en cas d'ingestion, très dangereux en cas d'inhalation, et dangereux en cas de contact cutané. Toujours manipuler en utilisant un équipement de protection approprié et dans une hotte de fumée.
REMARQUE : Tout au long du protocole, les couvercles des tubes sont toujours ouverts pendant les étapes du four à micro-ondes. - Laver les bouts de racine deux fois avec ddH2O pendant 5 min chacun sur le banc. Pour les troisième et quatrième ddH2O lavage programme d'utilisation 15 sur le micro-ondes (tableau 1). Après le premier lavage ddH2O de 40 secondes, prélever des échantillons du four à micro-ondes et remplacer le tampon par du ddH2O. Remettre les échantillons au micro-ondes et poursuivre le programme.
REMARQUE : Le micro-ondes sonnera une alarme lorsque le tampon doit être actualisé. Assurez-vous que le couvercle de la chambre à vide est remplacé correctement à chaque fois. - Incuber des échantillons dans la solution TCH préalablement préparée à RT pendant 2 min sur le banc et pour l'incubation plus loin utiliser le programme micro-ondes 8 (tableau 1). Ne changez pas de solution entre le banc et le four à micro-ondes.
- Laver les échantillons tels que décrits à l'étape 1.6.
- Placer les échantillons dans 1 % d'OsO4 dans ddH2O pour le programme micro-ondes 9 (tableau 1).
- Laver les échantillons tels que décrits à l'étape 1.6.
- Incuber des échantillons dans 1 % d'acétate uranyl en ddH2O à l'aide du programme micro-ondes 16 (tableau 1).
CAUTION: L'acétate d'uranyl est toxique, un irritant et a un potentiel cancérogène, mutagène et tératogène. Toujours manipuler en utilisant l'équipement de protection approprié. - Laver les échantillons tels que décrits à l'étape 1.6.
- Préparer la solution principale de Walton pour une utilisation à l'étape 1.14. Faites d'abord une solution de stock d'acide L-aspartique en ajoutant 0,998 g d'acide L-aspartique à 250 ml de ddH2O et en ajustant le pH à 3,8 avec 1 M KOH. Ensuite, dissoudre 0,066 g de nitrate de plomb dans 10 ml de solution de stock d'acide L-aspartique et ajuster le pH à 5,5. Laisser la solution au four à 60 oC pendant 30 min.
REMARQUE : Aucun précipité ne devrait se former. - Incuber des échantillons dans la solution de plomb de Walton pendant 30 min au four à 60 oC.
- Laver les échantillons tels que décrits à l'étape 1.6.
- Déshydrater les échantillons dans EtOH en étapes graduées de 50%, 70%, 90% en ddH2O, puis 2x dans 100% EtOH. Utilisez le programme 10 micro-ondes (tableau1) et le four à micro-ondes incitera les utilisateurs tous les 40 s à remplacer la solution par la prochaine étape EtOH. C'est la dernière étape faite dans le micro-ondes.
- Déshydrater davantage en oxyde de propylène à 100% 2x pendant 10 min chacun à RT sur le banc, remplaçant la solution entre les étapes.
REMARQUE : L'oxyde de propylène peut dissoudre certains plastiques comme les polystyrènes; soit utiliser des flacons de verre pour cette étape ou pré-test des plastiques pour la résistance.
AVERTISSEMENT : L'oxyde de propylène est très inflammable. Toujours manipuler en utilisant un équipement de protection approprié et dans une hotte de fumée. - Commencez l'infiltration des extrémités des racines en couvant dans 50% de résine de Spurr dans l'oxyde de propylène (min 2 h).
CAUTION : Les composants de résine de Spurr sont irritants. Toujours manipuler en utilisant un équipement de protection approprié et dans une hotte de fumée. - Remplacez la solution par 100% Spurr's et partez la nuit chez RT.
- Changement à la résine de Spurr fraîche 100% 2 fois (min 2 h incubations).
- Placer les échantillons dans un moule d'encastrement, contenant à nouveau la résine de 100% de Spurr fraîche et polymériser dans un four à 65 oC pendant 36 à 48 h.
REMARQUE : Le moule d'intégration utilisé dépend du type de tissu et de l'approche utilisée pour l'imagerie. Ici, un moule plat en silicone en castinerie a été utilisé (Figure 1A).
2. Préparer des échantillons intégrés pour l'imagerie
- Retirer les échantillons du four, retirer la résine du moule d'encastrement (Figure 1B).
- À l'aide d'une lame de rasoir, tailler grossièrement l'échantillon à un bloc de 0,5 mm x 0,5 mm x 0,5 mm (figure1C).
REMARQUE : Pour éviter de charger dans SBF-SEM, il est important de couper le plus de résine nue possible et de rendre l'échantillon aussi plat/mince que possible. Idéalement, tous les côtés du bloc contiennent déjà du tissu, mais surtout le côté qui sera attaché à la goupille métallique (voir l'étape 2.3) doit contenir des tissus exposés de sorte que le tissu est en contact direct avec le métal conducteur. - Retirez l'échantillon de la résine extra-terrestre et attachez-le à une broche métallique (Figure 1D) avec de la résine époxy conductrice, en s'assurant qu'une partie du tissu touche la goupille métallique. Laisser l'époxy guérir toute la nuit au four à 65 oC.
REMARQUE: Assurez-vous que l'échantillon est placé au centre de la broche, parce que le mouvement de la scène dans le SB-SEM est limité. Il peut être difficile de retirer le très petit échantillon en résine de la résine supplémentaire, car le petit échantillon aura tendance à s'envoler lorsqu'il est détaché. Une solution simple et efficace à cela est de couvrir l'échantillon avec une feuille de film de paraffine comme indiqué dans le film supplémentaire de référence1. - Attachez le support dans le support pour l'ultramicrotome. Utilisez une lame de rasoir pour enlever tout excès d'époxy et utilisez l'ultramicrotome et un couteau en diamant pour lisser le visage et les côtés du bloc, formant une pyramide. Assurez-vous qu'au moins une partie du tissu est déjà exposée sur le bloc-face.
REMARQUE: L'étape supplémentaire de l'utilisation d'un couteau en diamant est facultative, mais il rend le bloc résultant plus facile à approcher dans le SBF-SEM parce que l'ombre du couteau sur la face du bloc est plus claire rendant ainsi l'estimation de la distance entre le couteau et le visage de bloc plus facile à déterminer. - Placez le bloc d'échantillon taillé dans le pelage et enrober l'échantillon d'une fine couche (2 à 5 nm) de platine (Pt).
REMARQUE: Le platine sur le block-face sera coupé lors de l'approche dans le SBF-SEM (voir ci-dessous), mais le platine sur les côtés de la pyramide fournira une conductivité supplémentaire. Dans cet exemple, l'échantillon a été recouvert de platine, mais l'or, ou l'or / palladium est également efficace; cependant, le revêtement avec de l'or a eu comme conséquence des débris accrus sur le bloc-face pendant une course d'imagerie.
3. Imagerie dans le SBF-SEM
- Insérez le porteur dans le microscope SBF-SEM et amenez le couteau près de la surface de l'échantillon. À l'aide de la garniture de couteau de diamant outre de la partie supérieure de l'échantillon de sorte que la couche de Pt ait déjà été enlevée et au moins une partie du tissu soit exposée.
REMARQUE : Étant donné que ce processus est différent pour chaque microscope SBF-SEM, toutes les étapes ne sont pas spécifiées ici. Tant que la surface de l'échantillon est exempte de Pt et prête pour l'imagerie, les prochaines étapes seront possibles. - Commencez l'imagerie à basse résolution et à de courtes heures d'attente pour obtenir un aperçu de l'échantillon et localiser une région d'intérêt (figure 1E).
REMARQUE : Ici, 512 x 512 pixels et 1 temps de séjour ont été utilisés pour la numérisation et le positionnement rapides de la scène et 2 000 x 2 000 pixels avec 1 temps de séjour ont été utilisés pour optimiser la fenêtre d'imagerie et ajuster la mise au point. - À l'aide d'une tension d'accélération de 1,5 à 2,0 kV à un courant de 80 à 100 pA, capturez une image du tissu.
REMARQUE : L'exemple montré ici a été illustré sur un système à vide élevé où le courant du faisceau doit être ajusté pour minimiser la charge, qui dépend beaucoup de l'échantillon. Typiquement, le faisceau d'électrons est réglé à 1.5'2.0 kV mais cela sera dépendant de l'échantillon. À des grossissements plus élevés (généralement 10 000x) la résine est trop affectée par le faisceau pour garantir une section en douceur, de sorte que généralement pixel-taille est fixé à 8 -20 nm à une taille d'image de 8.000 à 10.000 x 8.000 -10.000 pixels avec une grossissement correspondante de 430 ' 1 400x et des tailles de champ de 64 X 64 mm et 200 x 200 mm respectivement. - Déterminer une région d'intérêt et décider combien de sections sont nécessaires pour couvrir le volume d'intérêt et commencer la course d'imagerie, en utilisant le détecteur d'électrons dispersés à l'arrière.
REMARQUE : Dans l'exemple présenté ici, 500 sections de 80 nm ont été représentées à 10 nm pixels et 10 000 x 10 000 pixels (temps d'arrivée 1 's). Le microscope a été réglé à 1,6 kV et 100 pA. En général, le nombre de sections dépend de l'échantillon et de la taille du retour sur investissement et peut varier de 100 à 1 000 s de sections consécutives. L'ensemble de données qui en résulte se compose d'images uniques de chaque section. Ces images doivent être converties en pile 3D.
4. Traitement des données SBF-SEM
- À l'aide de Fidji, sélectionnez la séquence d'importation de fichiers et localisez la pile d'images pour charger les images. Selon la taille de l'ensemble de données, cochez la case « Utiliser la pile virtuelle ».
REMARQUE: Si le jeu de données est vraiment grand, d'abord le convertir en 8 bits (si recueilli à 16 bits) et si nécessaire bin les données jusqu'à ce qu'il ait une taille réalisable. - L'utilisation du bouton de lecture au bas du jeu d'images passe à travers le jeu de données pour voir si l'exécution de l'imagerie a été réussie. Vérifiez les objets d'imagerie typiques pour le SBF-SEM, tels que les sections tombant du couteau sur le bloc-face, chargeant dans les zones de résine nue, coupant des artefacts du couteau (lignes horizontales sur l'image).
- À l'aide de la commande, les propriétés d'image ajustent la taille du pixel et la profondeur du voxel (c.-à-d. l'épaisseur de la section) utilisées pendant la course. Si les données sont déjà binned, en tenir compte.
- Utilisation de l'alignement de la pile linéaire d'enregistrement-enregistrement de commande avec SIFT pour enregistrer les données.
REMARQUE : L'enregistrement des données SBF-SEM est nécessaire parce qu'il peut y avoir un léger mouvement d'échantillon pendant l'imagerie en raison de la charge ou de la dérive de l'échantillon. Comme il ne s'agit que d'un mouvement minimal dans XY, seule la traduction est nécessaire. - Vérifiez l'alignement en faisant défiler le jeu de données et si OK utilisez la commande d'enregistrement sous le menu de fichier pour enregistrer le jeu de données aligné comme fichier 3D-tif.
- Analyser soigneusement l'ensemble de données pour voir si le retour sur investissement est inclus et contient les informations nécessaires pour la question biologique. Sur la dernière image de la pile (le bloc-face actuel), sélectionnez un nouveau retour sur investissement pour l'imagerie FIB-SEM.
REMARQUE : S'il n'y a pas de bonne région pour l'imagerie FIB-SEM présente sur le bloc-face actuel, plus de sections peuvent être coupées du bloc (qui est toujours dans le SBF-SEM) jusqu'à ce qu'un retour sur investissement apparaît. Il y a une limite au volume qui peut être photographié avec la FIB. Le retour sur l'image SBF-SEM peut être maximum en X, Y de 30-40 m x 15-20 'm.
5. Imagerie dans le FIB-SEM
- Prenez des images d'aperçu de l'échantillon dans le SBF-SEM, idéalement y compris un ou plusieurs bords de l'échantillon qui sont ensuite reconnaissables dans le FIB-SEM (Figure 2A,C).
REMARQUE : Dans cet exemple, les images d'aperçu SBF-SEM ont été prises à une taille de 10 nm de pixels, 8 000 x 8 000 pixels à 1's d'habit. Le microscope était toujours réglé à 1,6 kV et 100 pA. - Retirer l'échantillon du SBF-SEM et le placer dans le pelage. Enrober l'échantillon de 20 nm de platine pour l'imagerie FIB-SEM.
- Charger l'échantillon dans FIB-SEM et à l'aide d'un détecteur d'électrons secondaire à 15 kV, 1 nA localiser le retour sur investissement identifié dans le SBF-SEM sur le bloc-face (Figure 2B, D).
REMARQUE: L'imagerie à 15 kV est nécessaire pour voir à travers le revêtement de platine. - Apportez le retour sur investissement sur l'échantillon dans le point de coïncidence des faisceaux FIB et SEM, en déplaçant et en inclinant la scène (Figure 3A).
REMARQUE : La colonne FIB est généralement montée sous un angle (Figure 3A). Cela signifie que tout échantillon doit être incliné de manière à ce que la surface à imager et sectionnée soit positionnée parallèlement au faisceau FIB. La surface à être imaginée est maintenant inclinée par rapport au faisceau SEM et une tranchée de tissu doit être enlevée avant que le SEM soit en mesure d'imager le ROI (Figure 3E) - À l'aide du faisceau FIB et du système d'injection de gaz, déposez une couche protectrice de 1 mm de platine sur la surface au-dessus du ROI (Figure 3C). Ensuite, à l'aide d'un faible courant de fraisage (50-100 pA), les ridules de moulin dans le dépôt de platine pour le mise au point automatique et le suivi 3D pendant la course d'imagerie (Figure 3D). À l'aide de la colonne FIB et de l'injecteur de gaz carbonique, recouvrez ces lignes de dépôt de carbone.
REMARQUE : La taille du retour sur investissement correspond ici à la taille du retour sur investissement sur l'image SBF-SEM et la taille maximale peut donc être de 30-40 m x 15-20 m. Dans cet exemple, un retour sur investissement de 17 m x 8 m a été représenté. Le dépôt de carbone est nécessaire pour la protection des lignes et pour créer un contraste noir-blanc entre le carbone et le platine qui est idéal pour l'auto-mise au point. Les courants de fraisage utilisés pour chaque étape se trouvent dans le tableau 2. - À l'aide d'un courant de fraisage élevé, creusez une tranchée de 30 m devant le retour sur investissement, créant ainsi la surface d'imagerie du faisceau SEM (Figure 3E).
REMARQUE : Le faisceau FIB est intrinsèquement destructeur, encore plus à des courants élevés. Assurez-vous de maintenir l'imagerie à des courants élevés à un minimum et l'image à faible grossissement et des vitesses d'analyse rapide pour éviter de faire fondre la résine au retour sur investissement. - Lisser la surface d'imagerie avec un courant de fraisage plus proche du courant utilisé pendant la période d'imagerie. Arrêtez de polir lorsque toutes les marques de suivi autofocus et 3D sont clairement visibles sur la surface d'imagerie (figure 3F). La progression de la FIB peut être suivie par l'imagerie de la surface avec l'EM (à l'aide d'un détecteur d'électrons épars) tout en polissant.
- Déterminez la zone à imager sur la surface nouvellement créée et définiz les paramètres d'imagerie. Assurez-vous que le faisceau d'électrons est concentré sur la surface, fixe la luminosité et le contraste et fixez la taille du pixel et l'épaisseur de la section. Gardez le temps d'imagerie en dessous de 1 minute en ajustant la moyenne du temps et de la ligne.
REMARQUE : Il est important d'utiliser une basse tension pour l'imagerie avec le faisceau d'électrons pour s'assurer que seule la surface du bloc est image (c.-à-d., qu'aucun électron provenant de plus profond dans l'échantillon ne soit représenté). Ceci est fait en maintenant la tension en dessous de 2 kV et en utilisant un détecteur d'électrons dispersés vers l'arrière avec une tension de grille, permettant seulement des électrons à haute énergie pour être imaged. Dans cet exemple, le faisceau d'électrons a été réglé à 1,5 kV et 1 nA avec une tension de grille de 1,2 kV sur le détecteur d'électrons dispersés à l'arrière. Ici aussi, un pixel-taille de 5 nm a été utilisé avec des sections 5nm pour aboutir à un jeu de données avec voxels isotropic. Une superficie de 17 m x 10 m a été imageà 6,5 degrés de temps d'assise et une moyenne de ligne de 1,0. - Définir les fenêtres pour l'auto-tuning et le suivi 3D, en utilisant la même taille de pixel, le temps et la moyenne de ligne comme utilisé pour l'imagerie.
REMARQUE: Ce processus sera différent selon les systèmes, donc seule l'étape est mentionnée sans spécifier les différentes actions. - Démarrez la course d'imagerie et surveillez la stabilité du processus pendant les 50 à 100 premières sections. Une fois que le système fonctionne bien, quittez la pièce et assurez-vous qu'il y a le moins de perturbation possible dans la pièce.
REMARQUE : La durée de la course et le nombre de sections dépendront de la taille du retour sur investissement et de l'épaisseur de la section. Dans FIB-SEM, l'axe Z est en fait la hauteur de la boîte de retour sur l'image SBF-SEM (max 15-20 m; Figure 3E). - Enregistrez les données FIB-SEM de la même manière que décrite ci-dessus pour les données SBF-SEM.
Résultats
Les images du SBF-SEM donnent un aperçu du tissu, donnant un aperçu de l'orientation spatiale des cellules et des connexions intercellulaires (Figure 4A). L'imagerie FIB-SEM subséquente sur une nouvelle région, qui est habituellement une région d'intérêt déterminée après l'inspection de la course SBF-SEM, ajoute des détails à haute résolution de cellules et/ou structures spécifiques (figure 4B).
Figure 4C ,D montrent la différence dans le rendu des voxels non-isotropes des données SBF-SEM (Figure 4C) et les données isotropiques voxel FIB-SEM (Figure 4D). L'épaisseur z utilisée dans sBF-SEM signifie que le rendu montre clairement les sections, résultant en un effet d'escalier sur la surface. Dans les données FIB-SEM, les sections de 5 nm garantissent que le rendu semble beaucoup plus lisse et que les sections individuelles se fondent complètement dans la surface.
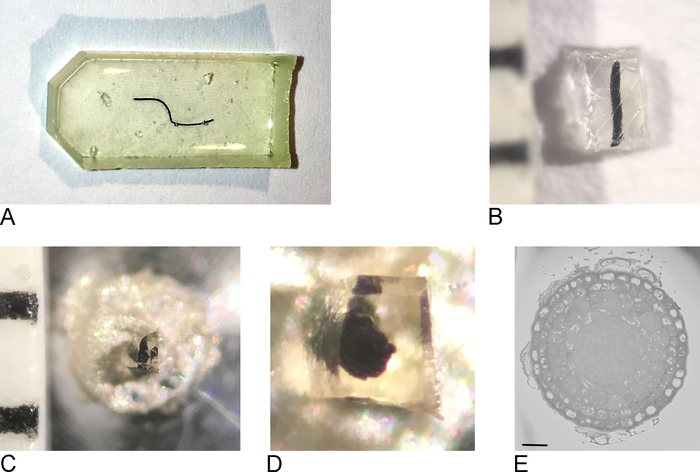
Figure 1 : Création de la face du bloc à partir d'un échantillon incorporé de résine. (A) Une pointe racine enchâssée dans la résine. (B) À l'aide d'une lame de rasoir, l'excès de résine est coupé jusqu'à ce qu'il reste un bloc de 0,5 mm2. (C,D) Le bloc taillé est collé sur une goupille métallique et après une nuit dans le four, les côtés du bloc sont taillés et la surface lissée avec un couteau en diamant à l'aide d'un ultramicrotome. (E) À l'intérieur du SBF-SEM, l'échantillon est orienté de sorte que le blockface et le retour sur investissement puissent être reconnus, barre d'échelle de 20 m. Veuillez cliquer ici pour voir une version plus large de ce chiffre.
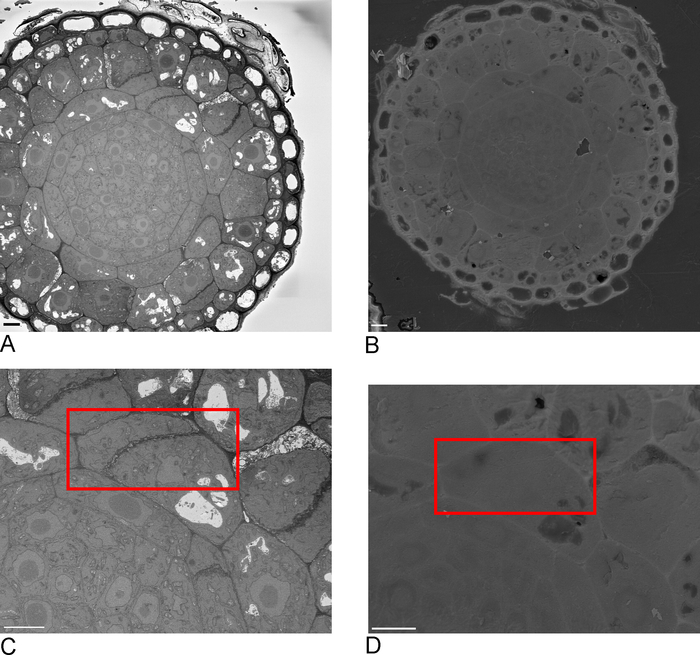
Figure 2 : Corrélation entre le SBF-SEM et le FIB-SEM. Aperçu des images du block-face à l'aide du SBF-SEM (A) et de la FIB-SEM (B), barre d'échelle de 5 m. (C,D) Zoom sur le retour sur investissement. La boîte rouge délimite la région à être représentée avec FIB-SEM, barre d'échelle de 5 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
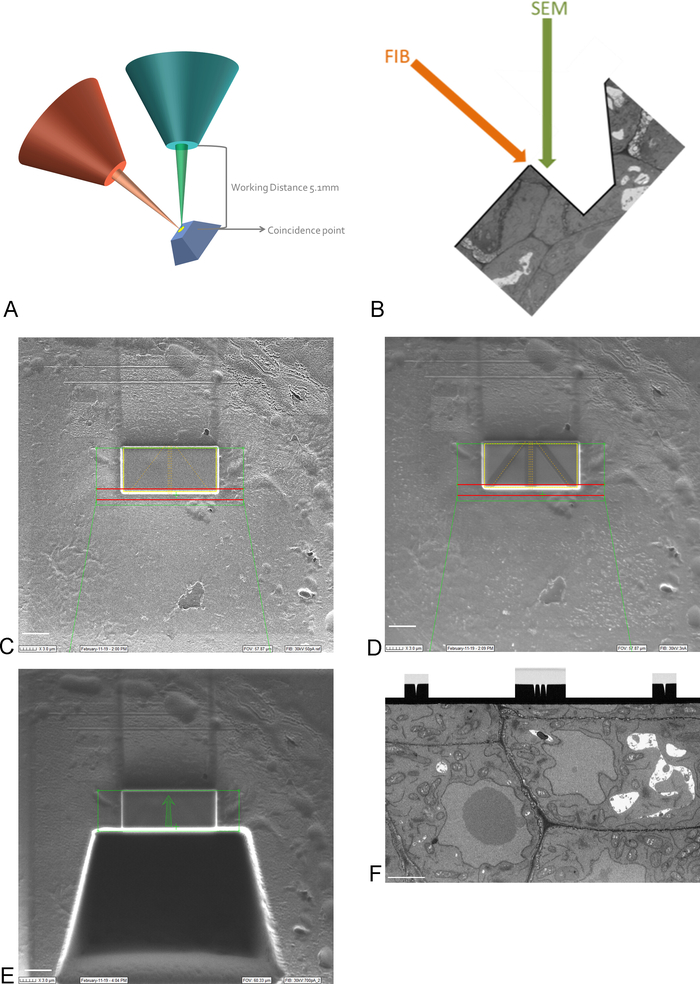
Figure 3 : Système FIB-SEM et étapes de préparation. (A) Schéma indiquant l'orientation du faisceau FIB, du faisceau SEM et de l'échantillon. L'échantillon doit être placé au point de coïncidence des faisceaux FIB et SEM pour pouvoir moudre et imager sur la même région. (B) Dessin schématique de la tranchée nécessaire à l'imagerie SEM des sections enlevées par la FIB. (C) Image prise avec le faisceau FIB montrant le dépôt de platine sur le roi-échange, barre d'échelle 5 m. (D) Image prise avec le faisceau FIB montrant les lignes utilisées pour l'auto-focus et le suivi 3D. Les lignes au milieu sont utilisées pour l'auto-focus et les lignes extérieures fournissent un suivi 3D. Dépôt de carbone sur le dessus des lignes fournit le contraste nécessaire (platine vs carbone) pour effectuer ces tâches, échelle bar 5 m. (E) Image prise avec le faisceau FIB après le fraisage de la tranchée, barre d'échelle 5 m. (F) Image prise avec le faisceau SEM montrant le région d'intérêt représentée pendant la course FIB-SEM, barre d'échelle de 2 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
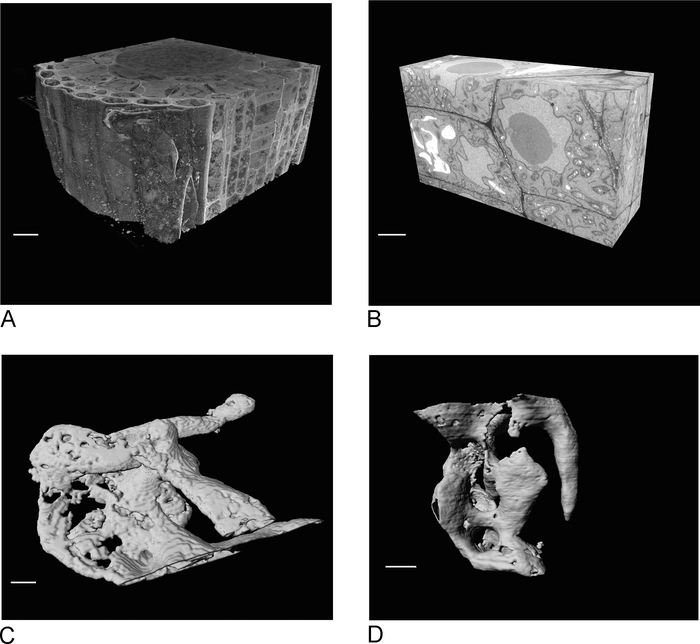
Figure 4 : Résultats SBF-SEM et FIB-SEM avant et après la segmentation. (A) vue 3D du jeu de données SBF-SEM (100 x 100 x 40 m, barre d'échelle de 10 m), (B) vue 3D du jeu de données FIB-SEM (17 x 10 x 5,4 m, barre d'échelle de 2 m),( C) Les vacuoles rendus segmentés à partir des données SBF-SEM par seuil, barre d'échelle à 2 m D. mented à partir des données FIB-SEM par seuil, barre d'échelle de 2 m. S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| Protocole pour le traitement par micro-ondes | |||||||||
| émission # | description | Prompt de l'utilisateur (on/off) | Temps (hr:min:sec) | Puissance (Watts) | Température (C) | Refroidisseur de charge (off/auto/on) | Vacuum/Bubbler Pump (off/bubb/vac cycle/vac on/vap) | Temp stable | |
| Pompe (on/off) | Température (C) | ||||||||
| 8 | Tch | de | 0:01:00 | 150 | 50 | de | CYCLE DE VIDE | sur | 30 |
| de | 0:01:00 | 0 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:01:00 | 150 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| 9 | Osmium | de | 0:02:00 | 100 | 50 | de | CYCLE DE VIDE | sur | 30 |
| de | 0:02:00 | 0 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:02:00 | 100 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:02:00 | 0 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:02:00 | 100 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| 10 | 50% EtOH | sur | 0:00:40 | 150 | 50 | de | de | sur | 30 |
| 70% EtOH | sur | 0:00:40 | 150 | 50 | de | de | sur | 30 | |
| 90% EtOH | sur | 0:00:40 | 150 | 50 | de | de | sur | 30 | |
| 100% EtOH | sur | 0:00:40 | 150 | 50 | de | de | sur | 30 | |
| 100% EtOH | sur | 0:00:40 | 150 | 50 | de | de | sur | 30 | |
| 15 | 0,1 M CACODYLATE | sur | 0:00:40 | 250 | 50 | de | CYCLE DE VIDE | sur | 30 |
| 0,1 M CACODYLATE | sur | 0:00:40 | 250 | 50 | de | CYCLE DE VIDE | sur | 30 | |
| 15 | ddH2O | sur | 0:00:40 | 250 | 50 | de | CYCLE DE VIDE | sur | 30 |
| ddH2O | sur | 0:00:40 | 250 | 50 | de | CYCLE DE VIDE | sur | 30 | |
| 16 | Acétate uranyl | de | 0:01:00 | 150 | 50 | de | CYCLE DE VIDE | sur | 30 |
| de | 0:01:00 | 0 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:01:00 | 150 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:01:00 | 0 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:01:00 | 150 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:01:00 | 0 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
| de | 0:01:00 | 150 | 50 | de | CYCLE DE VIDE | sur | 30 | ||
Tableau 1. Protocole détaillé pour le traitement par micro-ondes.
| pas | courant | Temps estimé |
| Déposition Platine | 3n A | 10-15 minutes |
| Milling Autotune et marques de suivi | 50-100 pA | 4-6 minutes |
| Déposition de carbone | 3 nA | 5-10 minutes |
| Trench grossier de fraisage | 15-30 nA | 30-50 minutes |
| Surface de polissage | 1.5-3 nA | 15-20 minutes |
| Course d'imagerie | 700 pA-1.5 nA | Heures-jours |
Tableau 2. Courants de fraisage FIB utilisés pour la préparation de l'échantillon et l'imagerie
Discussion
La microscopie électronique de volume est plus difficile et prend plus de temps que seM ou TEM conventionnel. En raison de la nécessité de tacher les tissus ou les cellules en bloc, les étapes de traitement doivent être assez longues pour assurer la pénétration des réactifs dans tout l'échantillon. L'utilisation de l'énergie micro-ondes pour faciliter la pénétration permet un traitement plus court et plus efficace et améliore la coloration. Parce que la préparation pour EM est beaucoup plus stricte que pour la microscopie légère toutes les solutions et réactifs doivent être contrôlés de qualité strictement. Les changements dans le pH, la tonicité, l'utilisation de réactifs impurs et l'introduction de contaminants dus à une mauvaise technique peuvent tous avoir des effets délétères sur l'image finale.
Volume EM nécessite également des protocoles personnalisés pour chaque type d'échantillon différent. Tissus mammifères de différents types: les plantes, les cellules simples telles que la levure, trypanosomes, C. elegans, etc, tous ont besoin de leurs propres variations pour obtenir des résultats optimaux. La fixation et la coloration doivent être conçues de manière à préserver l'intégrité structurelle et à garder l'échantillon le plus près possible de sa morphologie in vivo. La fixation des tissus à la température physiologique, au pH et à la tonicité est essentielle pour rendre l'échantillon aussi réaliste qu'il peut l'être. La congélation à haute pression (HPF) des échantillons peut aider à préserver une situation plus réaliste, (ou peut-être simplement produire des artefacts différents), mais pour d'autres que les cellules et les tissus très minces HPF échouera que la glace vitrée ne peut être générée en petits volumes. Par conséquent, pour de nombreuses questions, la fixation chimique est la seule option. Peu importe si la fixation est HPF ou chimique, dans toute expérience EM les résultats structurels doivent être soigneusement comparés aux résultats similaires de la cellule vivante ou de l'imagerie tissulaire pour voir si elles sont cohérentes. La coloration doit également être optimisée tout en tenant compte de la question spécifique à laquelle il faut répondre et du protocole qui sera utilisé pour la visualisation des images numériques.
Avoir à la fois un système SBF-SEM et FIB à proximité est un grand avantage dans de nombreuses expériences. Le grand champ de vision et la résolution élevée de X,Y de SBF-SEM facilitent la recherche de structures/cellules/événements individuels et fournissent une orientation spatiale globale des cellules dans les tissus. En outre, sa capacité à permettre l'imagerie à travers un échantillon en Z est très puissante; cependant, les reconstructions qui nécessitent des détails géométriques fins peuvent échouer ou produire des artefacts en utilisant cette technique en raison des voxels non-isotropes qu'elle génère. La FIB est limitée par la physique du processus à un champ d'imagerie plus petit, mais sa résolution 3D est suffisante pour des reconstructions très précises. La combinaison des deux techniques est simple car les échantillons peuvent passer du SBF-SEM à la FIB sans traitement ni préparation. Nous reconnaissons que l'utilisation du SBF-SEM pour la recherche à travers un échantillon pour trouver une zone particulière est une utilisation très coûteuse d'un outil beaucoup plus capable. Cependant, la possibilité de voir immédiatement le nouveau blockface et de déterminer si le retour sur investissement a été atteint est un grand avantage. En outre, les alternatives d'utiliser des sections LM semi-minces en série (0,5 m) peuvent enlever les petites structures avant qu'elles ne soient détectées, et inspecter un bloc à l'aide de sections TEM simples qui doivent être coupées, mises sur une grille et ensuite vues dans un TEM tout aussi coûteux n'est pas efficace comme la méthode présentée.
Étant donné que de nombreux programmes existent pour segmenter et rendre les données, et que les besoins d'une structure donnée peuvent ne pas être mieux servis par une seule application, aucun flux de travail standard ne peut être proposé. Certaines structures simples peuvent être segmentées à l''intérieur d'un algorithme de seuil s'ils se situent dans des valeurs à échelle grise très étroites. Les structures neuronales peuvent être segmentées semi-automatiquement à l'aide d'un programme tel qu'Ilastik11, mais il sera moins utile sur des organites de forme plus aléatoire ou complexe comme les urgences. Microscopy Image Browser est un programme très flexible qui peut aligner, segmenter et rendre des données EM volume, mais nécessite une interaction utilisateur significative12. En règle générale, le temps nécessaire pour visualiser numériquement les résultats dépassera largement le temps de préparation de l'échantillon et de l'imagerie.
Les techniques de volume EM ont ouvert la troisième dimension à l'analyse ultrastructurale. D'autres méthodes d'obtention de l'EM 3D ont des limites dans leur volume (tomographie TEM), ou leur efficacité (section série TEM). Bien que, pour la plupart, les techniques EM de volume soient trop complexes et coûteuses pour être mises en œuvre dans des laboratoires individuels, le nombre d'installations de base partagées qui les offrent a augmenté et le nombre de types d'échantillons photographiés avec succès a augmenté rapidement. Pour ceux qui ont une question spécifique et un tissu particulier, il est probable que quelqu'un sera en mesure d'offrir des conseils et des instructions sur sa préparation et l'imagerie. L'équipement EM de volume peut être amélioré pour inclure la capacité de traiter de plus grands échantillons dans le SBF-SEM et la capacité de moudre de plus grands ROIs avec le FIB. Les logiciels qui sont en mesure de segmenter les structures d'intérêt d'une manière plus automatisée simplifiera considérablement le processus d'analyse des données et l'amélioration de la vitesse de calcul permettra de réduire le temps nécessaire pour le faire. Malgré ses limites actuelles, le volume EM reste un outil utile et la combinaison du SBF-SEM et du FIB-SEM fournit un flux de travail efficace pour identifier les événements rares et les imagerie à haute résolution.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
L'équipement pour le volume EM a été fourni par une généreuse subvention du gouvernement flamand.
matériels
| Name | Company | Catalog Number | Comments |
| 3View 2XP | Gatan | NA | In chamber ultramicrotome for SBFI |
| Cacodylate buffer 0.2M solution | EM Sciences | 11652 | |
| Conductive epoxy resin (circuit works) | RS components | 496-265 | |
| Diatome Histo 4.0mm diamond knife | EM Sciences | 40-HIS | |
| Digitizing tablet | Wacom | DTV-1200W | No longer available |
| Eppendorf tubes | Eppendorf | 0030 120.094 | |
| Flat Embedding Mold | EM Sciences | 70900 | |
| Gluteraldehyde 25% solution | EM Sciences | 16220 | |
| High MW Weight Tannic Acid | EM Sciences | 21700 | |
| Lead Citrate | Sigma-Aldrich | 22861 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Osmium Tetroxide 4% solution | EM Sciences | 19170 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Pelco Biowave Pro + | Ted Pella | 36700 | |
| Potassium Ferrocyanide | Sigma-Aldrich | P3289 | |
| Quorum Q150T ES sputter coater | Quorum Technologies | 27645 | |
| Reichert-Jung Ultracut ultramicrotome | NA | NA | No longer available |
| Sodium Cacodylate 0.2M | EM Sciences | 11653 | |
| Spurrs Resin kit | EM Sciences | 14300 | |
| Uranyl Acetate | EM Sciences | 22400 |
Références
- Linberg, K. A., Fisher, S. K. An ultrastructural study of interplexiform cell synapses in the human retina. Journal of Comparative Neurology. 243, 561-576 (1986).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Leighton, S. SEM images of block faces, cut by a miniature microtome within the SEM – A technical note. Scanning Electron Microscopy. (pt 2), 71-76 (1981).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three dimensional tissue nanostructure. PLoS Biology. 11, 329 (2004).
- Heymann, J. A., Hayles, M., Gestmann, I., Giannuzzi, L. A., Lich, B., Subramaniam, S. Site specific 3D imaging of cells and tissues with a dual beam microscope. Journal of Structural Biology. 155 (1), 63-73 (2006).
- . NCMIR Methods for 3D EM: A new protocol for preparation of biological specimens for serial block face scanning electron microscopy. V7_01_10 Available from: https://ncmir.ucsd.edu/sbem-protocol (2019)
- Giberson, R. T., Austin, R. L., Charlesworth, J., Adamson, G., Herrera, G. A. Microwave and digital imaging technology reduce turnaround times for diagnostic electron microscopy. Ultrastructural Pathology. 27 (3), 187-196 (2003).
- Kremer, A., et al. Developing 3D EM in a broad biological context. Journal of Microscopy. 259 (2), 80-96 (2015).
- Vanslembrouck, B., Kremer, A., Pavie, B., van Roy, F., Lippens, S., van Hengel, J. Three-dimensional reconstruction of the intercalated disc including the intercellular junctions by applying volume scanning electron microscopy. Journal of Histochemistry and Cell Biology. 149, 479-490 (2018).
- Russel, M. R., et al. 3D correlative light and electron microscopy of cultured cells using serial blockface scanning electron microscopy. Journal of Cell Science. 130 (1), 278-291 (2017).
- Sommer, C., Strähle, C., Köthe, U., Hamprecht, F. A. ilastik: Interactive Learning and Segmentation Toolkit. Eighth IEEE International Symposium on Biomedical Imaging (ISBI). Proceedings. , 230-233 (2011).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy Image Browser: A Platform for Segmentation and Analysis of Multidimensional Datasets. PLoS Biology. 14 (1), e1002340 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon