Method Article
CRISPR/Cas9 Ribonucleoprotein-mediated Precise Gene Editing by Tube Electroporation
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Présenté ici est un protocole pour l'édition efficace de gène de ribonucleoprotein-mediated de CRISPR/Cas9 dans les cellules mammifères utilisant l'électroporation de tube.
Résumé
Les nucléases d'édition génique, représentées par la protéine 9 associée au CRISPR (Cas9), deviennent des outils courants dans la recherche biomédicale. La livraison réussie d'éléments CRISPR/Cas9 dans les cellules cibles par transfection est une condition préalable à une édition efficace des gènes. Ce protocole démontre que l'électroporation par tube (TE) de la livraison par machine de CRISPR/Cas9 ribonucleoprotein (RNP), ainsi que des modèles de donneurs d'oligodeoxynucleotide (ssODN) à un seul brin, mènent à des modèles de donneurs robustes événements précis d'édition de gènes. Tout d'abord, TE a été appliqué pour délivrer CRISPR/Cas9 RNP et ssODNs pour induire des mutations pathogènes dans le gène gamma de sous-unité de récepteur d'interleukine 2 (IL2RG) et le gène de réductase de sépiapterin (SPR) dans les cellules de fibroblaste de lapin. Des taux précis de mutation de 3.57%-20% ont été réalisés comme déterminé par le séquençage bactérien de clonage de TA. La même stratégie a ensuite été utilisée dans les iPSC humains sur plusieurs gènes cliniquement pertinents, y compris le récepteur du facteur de croissance épidermique (EGFR), la protéine de liaison de la myosine C, cardiaque (Mybpc3), et l'hémoglobine subunit bêta (HBB). Des taux de mutation constants et très précis ont été atteints (11,65 % à 37,92 %) déterminé séquençage profond (DeepSeq). Les travaux actuels démontrent que l'électroporation de tube de CRISPR/Cas9 RNP représente un protocole efficace de transfection pour l'édition de gène dans les cellules mammifères.
Introduction
CRISPR/Cas9 est la nucléase programmable la plus couramment utilisée pour l'édition de gènes. Il fonctionne à travers la reconnaissance à l'ARN (sgRNA) à guide unique des deux séquences cibles et d'une séquence adjacente de motif adjacent de motif de protospacer (PAM) dans le génome. La nucléase Cas9 génère une rupture d'ADN à double brin (DSB) située trois nucléotides en amont de la séquence PAM1. Les ORD sont réparés soit par des voies de jointure non homologue (NHEJ) sujettes aux erreurs, soit par des voies de réparation dirigées par homologie (HDR). Pour obtenir une modification précise des gènes par la voie HDR, les modèles de donneurs sont souvent fournis sous forme d'ADN plasmide (pDNA) ou d'oligodeoxynucléotide à brin unique (ssODN).
CRISPR/Cas9 et le sgRNA peuvent être livrés aux cellules en trois formats : le complexe de ribonucléoprotéines (RNP) de protéines Cas9 et de gRNA2,3; Cas9 ARNm et sgRNA4,5; ou plasmide ADN (pDNA) qui contient les promoteurs nécessaires, conduit sgRNA, et Cas9 région de codage6,7,8. De nombreux groupes ont démontré que lorsque CRISPR/Cas9 est livré en tant que RNP, l'efficacité d'édition de gènes surpasse souvent ceux obtenus dans les formats pDNA ou ARNm, attribuable s'il est beaucoup plus petit que les acides nucléiques9. En outre, il a été précédemment démontré qu'une nouvelle machine d'électroporationde tube (TE) est particulièrement efficace dans les applications d'édition de gène dans plusieurs types de cellules 9.
Présenté dans le présent travail est un protocole étape par étape dans l'utilisation de TE pour la livraison de CRISPR/Cas9 RNP aux cellules mammifères de différentes espèces à plusieurs loci cliniquement pertinents. Cette nouvelle technique de transfection TE et phénomène de taux élevé de HDR peuvent trouver de larges applications dans la recherche biomédicale.
Protocole
Toutes les procédures d'entretien, de soins et d'utilisation des animaux ont été examinées et approuvées par le Comité institutionnel de soins et d'utilisation des animaux (IACUC) de l'Université du Michigan.
1. Préparation des cellules
-
Acquérir des iPSC humains (ACS-1030) de l'American Type Culture Collection (ATCC). Culture iPScs sur matrice extracellulaire artificielle avec milieu de culture cellulaire sans chargeur (voir Tableau des matériaux) dans un incubateur de culture cellulaire (5% CO2 à 37 oC) suivant les instructions du fournisseur.
- 2 h avant la transfection, traiter les iPSC avec 10 M Rho-associés, bobine enroulée contenant des protéines kinase (ROCK) inhibiteur Y27632 (utilisation de ce qui réduit l'apoptose des hiPSC humains dissociés et augmente la survie et l'efficacité de clonage des hiPSC sans hiPSCs sans hiPSCs affectant leur pluripotence).
- Lors de la transfectilité, dissocier les iPSC avec la solution de détachement cellulaire (voir Tableau des matériaux) aux cellules simples à 37 oC pendant 5 min. Comptez le numéro de cellule.
-
Établir une culture de cellules de fibroblaste de lapin utilisant une culture primaire des biopsies de tissu de peau d'oreille de lapin, comme précédemment décrit10.
- Une biopsie de la peau de l'oreille de 0,5 cm x 0,5 cm est obtenue à partir de la pointe de l'oreille de lapin. Raser les cheveux du tissu auditif.
- Rincer 2x avec la saline tamponnée en phosphate (DPBS) de Dulbecco avec 5% de pénicilline-streptomycine. Transférer le tissu auditif dans un nouveau plat de culture tissulaire de 6 cm, puis couper le tissu en petits morceaux (1,0 mm x 1,0 mm). Ajouter quelques gouttes de sérum bovin fœtal pour empêcher le tissu de se dessécher.
- Étendre le tissu déchiqueté dans un plat de culture tissulaire de 10 cm, puis ajouter 10 ml de milieu de culture. Les cellules de fibroblaste de lapin sont cultivées dans le milieu modifié d'aigle de Dulbecco (DMEM) avec 10% de sérum bovin foetal. Mettre le plat de culture tissulaire dans l'incubateur de culture cellulaire (5 % de CO2 à 37 oC).
- Trois à cinq jours après le placage, utilisez la trypsine-EDTA pour digérer les cellules à 37 oC pendant 2 min. Comptez le numéro de cellule.
2. Conception et synthèse des gARN et des Oligos donateurs
- Pour chaque gène, la conception guide l'ARN en fonction de la séquence du locus ciblé à l'aide d'un outil en ligne (par exemple, http://crispor.tefor.net/-gt;).
- Pâte dans la séquence d'ADN d'intérêt.
- Sélectionnez un génome et un motif adjacent au protospacer (PAM). Les séquences de guidage possibles dans les séquences d'ADN d'entrée seront affichées sur la page de sortie. Il est recommandé de sélectionner l'ARNc avec une efficacité prévue plus élevée et des potentiels hors cible plus faibles.
- Synthétiser l'ADN par un fournisseur commercial pour la transcription des gARN. Effectuer la transcription in vitro de gRNA à l'aide d'un kit de synthèse gRNA selon les instructions du fabricant.
- Purifie le gRNA à l'aide d'une micro colonne de purification de l'ARN incluse dans le kit de synthèse de gRNA. Mesurer la concentration, puis stocker les gARN à -80 oC.
- Concevoir un modèle de donneur ssODN pour chaque site de mutation. Les ssODN peuvent être synthétisés par des fournisseurs commerciaux tels que IDT. En général, chaque ssODN est 120-160 nucléotides (nt) de longueur, composé de 60-80 nt dans le bras d'homologie gauche et 60-80 nt dans le bras homologie droit. Pour éviter la recoupe de l'ADN édité, une mutation silencieuse au PAM devrait être introduite dans le ssODN chaque fois que possible. Le site de coupe CRISPR doit être situé le plus près possible du changement génomique prévu.
3. Tube Electroporation de Cas9 RNP et ssODNs
- Préparer les cellules telles que décrites dans la section 1.
- Resuspendre 2-3 x 105 cellules dans 20 ll de tampon d'électroporation. Pipette de haut en bas soigneusement pour produire une suspension à une cellule.
- Pour la transfection cas9 RNP, prémélangez 2 g de protéine Cas9-NLS avec 0,67 g de gRNA à température ambiante (RT) pendant 10-15 min. Ensuite, mélangez délicatement le complexe RNP formé avec 2 g de ssODN avec des cellules.
- Transférer le mélange cellulaire dans un tube d'électroporation de 20 l à l'aide de pointes de pipette universelles fournies par le kit d'électroporation du tube. Pour obtenir une meilleure électroporation, essayez d'éviter la formation de bulles d'air pendant le transfert.
- Placez le tube d'électroporation dans la fente de l'électroporateur et appuyez sur "Go" pour finir. Suivez les paramètres suggérés par le fabricant pour chaque type de cellule. Par exemple, pour les iPSC humains et les cellules de fibroblaste de lapin, l'ensemble de tension est 420 V et le temps d'impulsion est de 30 ms. Un cycle d'électroporation réussi est indiqué par le rapport d'impulsion sur l'écran d'affichage de l'électroporateur.
- Après l'électroporation, transférez les cellules iPS humaines à 1 ml de milieu culturel préréchauffé De Y-27632-contenant décrit dans la partie de culture cellulaire. Pour les cellules de fibroblaste de lapin, transférez-les au DMEM avec 10% de sérum bovin foetal.
- Plaquer les cellules resuspension nées à un puits d'une plaque de culture de cellules de puits.
- Changer le milieu de la culture tous les jours. Y-27632 est retiré du milieu de culture iPSC humain 24 h après l'électroporation.
4. Analyse des événements d'édition de gènes
- Récolte des cellules 72 h après l'électroporation. Digiérez les cellules de la plaque de culture à l'aide de trypsine-EDTA pour les cellules de fibroblaste de lapin ou la solution de détachement cellulaire pour les iPSC humains. Après centrifugeuse, resuspendre les cellules avec 350 ml de tampon de lyse (1 M Tris HCl, 5 M NaCl, 0,5 M EDTA; pH 8,0, 10% SDS, ajouter 20 'L de 20 mg/mL de stock protéase K par 1 ml de tampon de lyse), puis incuber à 55 oC pendant la nuit.
- Extraire l'ADN génomique avec du phénol-chloroforme en utilisant des procédures standard.
- Amplifier 100-200 bp fragments d'ADN contenant la région ciblée à l'aide de polymérase d'ADN haute fidélité, puis purifier les fragments d'ADN à partir de gels à l'aide d'un kit d'extraction de gel ou directement à partir de produits PCR à l'aide d'un mini kit PCR SV.
- Pour déterminer l'efficacité de l'édition génétique par séquençage des colonies bactériennes, ligales les produits PCR purifiés dans un vecteur pCR4-TOPO à l'aide d'un kit de clonage TOPO TA. Prenez au hasard des clones bactériens, puis séquencez les inserts à l'aide d'une amorce de séquençage universelle fournie par le kit de clonage TOPO TA.
- Pour déterminer l'efficacité de l'édition génétique par séquençage profond, envoyez les produits PCR purifiés (100-200 pb) à partir de l'étape 4.3 pour le séquençage de l'amplicon CRISPR dans un noyau de séquençage de l'ADN.
Résultats
TE de Cas9 RNP et ssODNs aux cellules de fibroblaste de lapin
Le processus global de livraison par voie TE de Cas9 RNP aux cellules de mammifères est illustré à la figure 1. Tout d'abord, des mutations C231Y et Q235X ont été produites dans le gène IL2RG, et la mutation R150G a été produite dans le gène SPR dans les cellules de fibroblaste de lapin. Les mutations de perte de fonction dans les gènes IL2RG et SPR sont connues pour causer l'immunodéficience primaire11 et les déficits moteurs et cognitifs12,respectivement.
Les dessins spécifiques de sgRNA sont illustrés dans la figure 2A. Les amorces utilisées pour amplifier les régions ciblées sont énumérées dans le tableau 3. Les séquences des ssODN sont affichées dans le tableau 1. Les taux d'édition génétique ont été déterminés par le clonage bactérien TA (figure 2B). Au locus IL2RG C231, sur les 28 clones qui ont été séquencés, un (3,57%) a porté la mutation Précise c231Y, quatre (14,28%) mutations d'insertion ou de suppression (indel) et les 23 autres (82 %) étaient de type sauvage. Au locus IL2RG Q235, sur les 27 clones qui ont été séquencés, deux (7,41 %) a porté la mutation Q235X précise, trois mutations d'indel portées (11,11%) et les autres étaient de type sauvage. Au sPG R150 locus, sur les 20 clones séquencés, cinq (25%) a porté la mutation R150G précise, 10 (50%) mutations d'indel, et les autres étaient de type sauvage.
TE de Cas9 RNP et ssODNs aux iPSC humains
TE a ensuite été utilisé pour délivrer Cas9 RNP et ssODNs aux iPSC humains et cibler les loci cliniquement pertinents dans les gènes EGFR, Mybpc3 et HBB. Les mutations ponctuelles dans la région proximale de t790 d'EGFR confèrent la résistance aux inhibiteurs de kinase de tyrosine d'EGFR dans les patients du cancer du poumon de non-petite cellule (NSCLC) hébergeant des mutations activantes de l'EGFR13. Une mutation de frameshift dans l'exon 16 dans Mybpc3 est impliquée dans la cardiomyopathie hypertrophique14. La mutation de point d'E6V dans le gène hBB mène à la maladie de drépanocytose15.
Les dessins spécifiques de sgRNA sont illustrés dans la figure 3A. Les amorces utilisées pour amplifier les régions ciblées sont énumérées dans le tableau 3. Les séquences des ssODN sont affichées dans le tableau 1. Les taux d'édition génétique ont été déterminés par DeepSeq (figure 3B). Au locus de l'EGFR, 15,68 % des allèles portaient les mutations ponctuelles précises (6 315 lectures), 22,75 % portaient des mutations d'indel (9 162 lectures), et les 61,57 % restants étaient de type sauvage (24 797 lectures). Au locus Mybpc3, 37,92 % ont porté la suppression précise de TGAA de 4 pb (11 654 lectures), 2,24 % ont porté des mutations d'indel (410 lectures) et les 59,84 % restants étaient de type sauvage (18 692 lectures). Au locus HBB, 11,65 % portaient la mutation E6V précise (6 565 lectures), 23,35 % portaient des mutations d'indèle (13 163 lectures) et les 65 % restants étaient de type sauvage (36 644 lectures).

Figure 1: Graphique de flux de l'électroporation de tube de Cas9 RNP.

Figure 2 : Modification génétique des cellules de fibroblaste de lapin. (A) Illustration des séquences cibles. Les boîtes indiquent des loci ciblés. Les lettres soulignées correspondent à des séquences de gRNA. Les lettres de couleur rouge indiquent des séquences PAM. (B) TA clonage des résultats des événements d'édition de gènes. Les boîtes indiquent précisément les loci mutés. La séquence D'Indel montrée n'est représentative que d'un type d'allele. D'autres séquences d'indel ne sont pas affichées. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
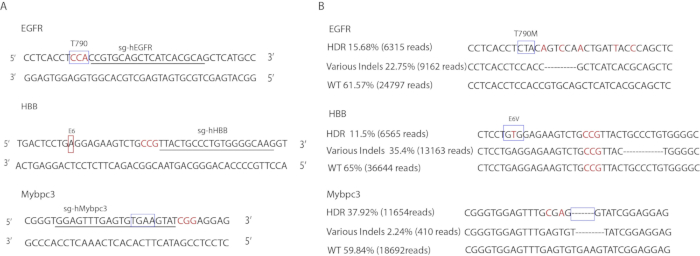
Figure 3 : Édition génétique des iPSC humains. (A) Illustration des séquences cibles. Les boîtes indiquent des loci ciblés. Les lettres soulignées correspondent à la séquence de gRNA. Les lettres de couleur rouge indiquent des séquences PAM. (B) Résultats Deepseq des événements d'édition de gènes. Les boîtes indiquent précisément les loci mutés. Les lettres de couleur rouge indiquent des mutations silencieuses qui ont été introduites dans les modèles de donneur. La séquence D'Indel montrée n'est représentative que d'un type d'allele. D'autres séquences d'indel ne sont pas affichées. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Locus |
Séquence Oligo | |||||||
| (mutation ciblée) | ||||||||
| Lapin IL2RG (C231Y) | AGCGTGGATGGGCAGAAACTATACACGTTCCGAGTCCGGAGCCGTTTTAACCCTTTTTTGTATGGGAGTGCTCAGCATTGGAGT GAATGGAGCACCCACCCCACACTACTGGGGGAGCAAAACTTCAAAGGGTAAAATGGGCCT | |||||||
| Lapin IL2RG (Q235X) | AGCGTGGATGGGCAGAAACTATACACGTTCCGAGTCCGGAGCCGTTTTAACCCTTTGTGTGGGAGTGCTTAGCATTGGAGT GAATGGAGCACCCACCCCACACTACTGGGGGAGCAAAACTTCAAAGGGTAAAATGGGCCT | |||||||
| Lapin SPR | gacctccatgctctgcctgacctcctgccctgaaggcgttcctgccagtcctggtggCctcagcgggactgtggtgacatctcgtcgtcgtgtgtgccctgcagcccttcaagggctggggggggggtgtcgtcttcattcaagggctgggggggt cgctgtac (cgctgtac) | |||||||
| (R150G) | ||||||||
| EGFR humain | ACGTGATGGCCAGCGTGGACACCCACGTGTGCCGCCGCTGCTGCTGCTGCTCACCCTACTCACCCCTACAGTCCAACTACTACCC AGCTCATGCCCTTCGGCTGCCTCCTGGACTATGTCCGAACACAAGACAATATTGGCTCCCAGTAC | |||||||
| (Les mutations ponctuelles se sitorquent dans le T790) | ||||||||
| Mybpc3 humain | GCCCCCTGTGCTCATCACCACCGCCCCCTGGGGGGGGGCTGATGGGGGGGGCGCGGGGAGTTGCGAGTTGCGAGTCGG GGAGGGGGCGCAGTCAAATGGTGAGTTCCAGAAGCACGGGGCATGTGTTGGGGGCAT | |||||||
| (suppression de 4 pb) | ||||||||
| HBB humain | TCTGACACAACTGTGTTCACCACACACCACTCAAACAGACACCATGGTGCATCTGACTACTTGTGTTGCTACTACTACT CTGTGGGGCAAGGTGAACGTGGATGAAGTTGGTTGAGGCCCTGGGCAG | |||||||
| (E6V) | ||||||||
Tableau 1 : Séquences de ssODN.
| pas | problème | Raisons possibles | Solutions |
| 2.1 (en) | Faible taux d'indel | Mauvaise conception d'ARN de guide, stocks d'ARN de guide -gt;6 mois, faible concentration d'ARN de guide | Refonte de l'ARN guide, produire / commander l'ARN nouveau guide. |
| 2,3 | Faible efficacité PGE | mauvaise conception de l'ADN des donneurs, ARN guide peu efficace, quantité incorrecte d'ADN de donneur ou ADN de mauvaise qualité | Augmenter la longueur des bras homologiques, introduire la mutation PAM, introduire des mutations silencieuses à l'ADN du donneur, utiliser un ARN guide plus efficace, Optimiser le rapport de la protéine Cas9 sur l'ARN guide. |
| 3,4 | Transfection ratée | Bulles d'air formées pendant le transfert du mélange cellules-tampon au tube d'électroporation, réglage incorrect de tension/durée | Essayez d'éviter la formation de bulles d'air, ajustez le réglage tension/durée. |
| 3,6 Annonces | faible viabilité cellulaire après électroporation | Faible survie de l'ipsc humain simple | Ajouter l'inhibiteur ROCK après l'électroporation, augmenter le nombre de cellules. |
| 4.1 (en) | Échec du PCR | Contenu élevé de GC, ou séquence répétitive | Optimisez l'état PCR, ajoutez Du DMSO au système PCR. |
Tableau 2 : Guides de dépannage pour les problèmes fréquents.
| Nom d'amorce | ordre | note |
| Rb-IL2RG-F | CATGACAGTGACAGGGTCCC | Pour amplifier le fragment d'ADN du lapin IL2RG |
| Rb-IL2RG-R | TGCCAGAGACACAAGCGAAC | |
| Rb-SPR-F | GTACTTTGGAGGGACAGAGG GTACTTTTGGAGGGACAGAGG GTACTTTTGGAGGGACAGAGG | Pour amplifier le fragment d'ADN SPR de lapin |
| RB-SPR-R | CTCAGCACCCTGACACTGGG | |
| H-EGFR-F | TGATGGCCAGCGTGGACAAC | Pour amplifier le fragment d'ADN humain EGFR |
| H-EGFR-R | ACCAGTTGAGCAGGTACTGGG | |
| H-Mybpc3-F | ATGCCCCGTGCTTTTGGAAC | Pour amplifier le fragment d'ADN humain Mybpc3 |
| H-Mybpc3-R | TCAGGGGAGCCAACCCTCAT | |
| H-HBB-F | TAACCTTGATACCAACCTGC | Pour amplifier le fragment d'ADN humain HBB |
| H-HBB-R | CATTTGCTTTTGACACAACT |
Tableau 3 : Amorces utilisées à l'étape 4.3.
Discussion
La méthode d'électroporation de tube a été efficace en fournissant LE RNP de CRISPR/Cas9 et les ssODNs au lapin et aux cellules humaines, menant à l'édition précise précise de gène (PGE). La principale différence entre TE et d'autres dispositifs d'électroporation conventionnels est l'utilisation d'un tube, dans lequel deux électrodes sont sur le dessus et le bas du tube et l'échantillon est chargé en entier puis scellé lors de l'électroporation (Figure 1). En revanche, dans une cuvette conventionnelle, les électrodes sont sur les côtés et l'échantillon n'est pas entièrement scellé lors de l'électroporation. Cette nouvelle conception réduit la génération de bulles d'air et comprime la taille des bulles d'air, ce qui améliore par conséquent même la distribution de la tension électrique, et conduit ainsi à la mort cellulaire réduite et l'efficacité de transfection élevée9. Dans le présent travail, des taux élevés de PGE (15%-37%) ont été atteints en ciblant les gènes EGFR, Mybpc3 et HBB dans les iPSC humains. Ces résultats sont compatibles avec un rapport antérieur dans lequel des taux élevés de PGE ont été atteints dans les cellules souches humaines9.
Des mutations pathogènes ont été ciblées dans les gènes IL2RG et SPR dans les cellules de lapin. Récemment, les lapins IL2RG-knockout ont été produits comme modèles pour l'immunodéficience combinée grave X-liée humaine (SCID-X1)16,17. Les travaux actuels montrent que les mutations DU patient IL2RG (p. ex. C231Y et Q235X) peuvent être générées efficacement dans les cellules de lapin, démontrant ainsi la faisabilité de créer des modèles de lapins SCID-X1 porteurs de mutations patientes. Il a également été démontré que les mutations SPR R150G peuvent être efficacement créées dans les cellules de lapin. Cette mutation provoque des déficits moteurs et cognitifs chez les enfants12. Ces modèles de lapin mutation IL2RG et SPR, une fois générés, peuvent servir de modèles précliniques précieux pour les études translationnelles. Ils peuvent également être utilisés pour établir des thérapies basées sur l'édition génétique pour ces maladies monogéniques.
L'une des préoccupations concernant les applications d'édition de gènes à médiation CRISPR/Cas9 est les événements d'édition hors cible. Les taux d'indel ont été analysés dans les sites hors cible prévus pour les sgARN utilisés dans cette étude (tableau S1), en utilisant des méthodes précédemment décrites9. Au total, sept loci potentiels hors cible ont été analysés pour sg-rb-IL2RG-01, cinq pour sg-rb-SPR, sept pour sg-hEGFR, cinq pour sg-hMybpc3, et sept pour sg-hHB), en utilisant les amorces énumérées dans le tableau S2. Aucun indel hors cible n'a été révélé par les essais T7E1 (Figure S1), indiquant des risques minimes hors cible pour l'édition génétique à médiation CRISPR/Cas9 à l'aide de ces sgRNAs. Il indique également que la méthode d'électroporation du tube elle-même ne provoque pas ou n'augmente pas les modifications hors cible. Néanmoins, des efforts devraient être consacrés à la réduction ou à l'élimination des modifications indésirables hors cible. Le séquençage du génome entier peut être nécessaire pour exclure de tels événements pour les cellules qui sont destinées à être utilisées dans des applications cliniques.
Au niveau technique, les facteurs suivants sont considérés comme des facteurs clés pour atteindre l'édition efficace et précise du génome par l'électroporation du tube CRISPR/Cas9 RNP. Tout d'abord, il est conseillé de sélectionner un sgRNA efficace avec un faible potentiel hors cible prévu. Il est important de valider l'efficacité indel de l'ARSS sélectionné avant de l'utiliser pour les applications PEG. Il n'est pas rare qu'un logiciel prédit bon sgRNA échoue à l'étape de validation.
Deuxièmement, pour atteindre le PGE élevé, il est recommandé d'induire une mutation de PAM au donateur de ssODN chaque fois que possible. La raison en est que, ce faisant, CRISPR/Cas9 recoupe après l'intégration des modèles de donneurs est empêchée. Dans certains cas, le PGE lui-même introduit des mutations PAM. Dans d'autres cas, il est possible d'introduire des mutations silencieuses dans la séquence PAM. Dans le cas où une mutation PAM n'est pas possible, il est conseillé d'essayer d'inclure plusieurs mutations silencieuses dans le donneur qui correspond à la séquence sgRNA.
Troisièmement, particulièrement pertinent pour TE, il est important d'éviter la formation de bulles d'air lors du transfert des cellules et du mélange RNP au tube d'électroporation. Tandis que la conception d'un tube de TE minimise déjà la formation de bulle d'air, la manipulation soigneuse réduira davantage et peut même accomplir éviter la formation de bulle d'air. Un guide de prise de vue de difficulté pour des problèmes fréquents qui peuvent être rencontrés dans l'application de l'électroporation de tube pour CRISPR/Cas9 ribonucleoprotein négocié l'édition précise de gène est fourni dans le tableau 2.
En conclusion, il est démontré ici que l'électroporation de tube est un moyen efficace pour la livraison de CRISPR/Cas9 RNP et de ssODNs aux cellules mammifères pour atteindre des taux élevés de PGE. Cette nouvelle technique de transfection TE et son taux d'édition génétique précis et robuste peuvent faciliter le développement d'applications d'édition de gènes.
Déclarations de divulgation
J. C. travaille chez Celetrix LLC, fabricant de l'électroporateur de tube. L. M., L. J., J. S., D. Y., J. Z., Y. E. C., et J. X. ne déclarent aucun intérêt concurrent.
Remerciements
Ce travail a été soutenu par les National Institutes of Health (R21OD023194 à JX). Ce travail a utilisé les services de base soutenus par center for Advanced Models for Translational Sciences and Therapeutics (CAMTraST) au Centre médical de l'Université du Michigan.
matériels
| Name | Company | Catalog Number | Comments |
| Accutase | STEMCELL Technologies | 792 | Cell detachment solution for human iPSCs, first used in Step 1.1.2. |
| Cas9 Nuclease 3NLS | IDT | 1074182 | Cas9 protein, first used in Step 3.3. |
| DMEM | Thermo Fisher | 11965092 | For cell culture, first used in Step 1.2.3. |
| DPBS | Thermo Fisher | 1708075 | For preparing cell culture, first used in Step 1.2.2. |
| EDTA | Lonza | 51201 | For making lysis buffer, first used in Step 4.1. |
| Electroporation buffer | Celetrix | 13–0104 | The electroporation buffer, first used in Step 3.2. |
| Electroporation tubes | Celetrix | 20 μL: 12–0107; 120 μL: 12–0104 | The electroporation tube, first used in Step 3.4. |
| Electroporator | Celetrix | CTX-1500A LE | The tube electroporation machine, first used in Step 3.5 |
| Fetal bovine serum | Sigma Aldrich | 12003C | For cell culture, first used in Step 1.2.2. |
| Forma CO2 Incubators | Thermo Fisher | Model 370 | For cell culture, first used in Step 1.1. |
| Gel Extraction Kit | Qiagen | 28115 | For gel purification, first used in Step 4.3. |
| Human induced pluripotent stem cells | American Type Culture Collection | ACS-1030 | Human iPSCs, first used in Step 1.1. |
| Matrigel | Corning | 354277 | Artificial extracellular matrix; for precoating cell culture plate, first used in Step 1.1. |
| mTeSR 1 medium | STEMCELL Technologies | 85850 | Feeder-free cell culture medium for human iPSCs, first used in Step 1.1. |
| PCR SV mini | GeneAll | 103-102 | For PCR product purification, first used in Step 4.3. |
| Penicillin-Streptomycin | Thermo Fisher | 15140163 | For preparing cell culture, first used in Step 1.2.2. |
| Phenol-chloroform | Thermo Fisher | 15593031 | For DNA extraction, first used in Step 4.2. |
| Precision gRNA Synthesis Kit | Invitrogen | A29377 | For the generation of full length gRNA (guide RNA), first used in Step 2.4. |
| Proteinase K Solution | Thermo Fisher | AM2548 | For DNA extraction, first used in Step 4.1. |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification, first used in Step 4.3. |
| Sodium dodecyl sulfate | Sigma Aldrich | L3771 | For making lysis buffer, first used in Step 4.1. |
| TA Cloning Kit | Thermo Fisher | K457502 | For TA clone sequencing, first used in Step 4.4. |
| Tissue Culture Dish (10 cm) | FALCON | 353003 | For cell culture, first used in Step 1.2.3. |
| Tissue Culture Dish (12 well) | FALCON | 353043 | For cell culture, first used in Step 3.7. |
| Tissue Culture Dish (6 cm) | FALCON | 353004 | For cell culture, first used in Step 1.2.2. |
| Tris HCl | Thermo Fisher | BP1757-500 | For making lysis buffer, first used in Step 4.1. |
| Trypsin-EDTA | Thermo Fisher | 25200056 | For cell digestion, first used in Step 1.2. 4. |
| Universal Fit Pipette Tips | Celetrix | 14-0101 | For electroporation, first used in Step 3.4. |
| Y27632 | LC Labs | Y-5301 | The apoptosis inhibotor, first used in Step 1.1.1. |
Références
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mout, R., et al. Direct Cytosolic Delivery of CRISPR/Cas9-Ribonucleoprotein for Efficient Gene Editing. ACS Nano. 11 (3), 2452-2458 (2017).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo. Nature Biotechnology. 33 (1), 73-80 (2015).
- Miller, J. B., et al. Non-Viral CRISPR/Cas Gene Editing In Vitro and In Vivo Enabled by Synthetic Nanoparticle Co-Delivery of Cas9 mRNA and sgRNA. Angew Chem Int Ed Engl. 56 (4), 1059-1063 (2017).
- Finn, J. D., et al. A Single Administration of CRISPR/Cas9 Lipid Nanoparticles Achieves Robust and Persistent In Vivo Genome Editing. Cell Reports. 22 (9), 2227-2235 (2018).
- Liang, C., et al. Tumor cell-targeted delivery of CRISPR/Cas9 by aptamer-functionalized lipopolymer for therapeutic genome editing of VEGFA in osteosarcoma. Biomaterials. 147, 68-85 (2017).
- Luo, Y. L., et al. Macrophage-Specific in Vivo Gene Editing Using Cationic Lipid-Assisted Polymeric Nanoparticles. ACS Nano. 12 (2), 994-1005 (2018).
- Wang, H. X., et al. Nonviral gene editing via CRISPR/Cas9 delivery by membrane-disruptive and endosomolytic helical polypeptide. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4903-4908 (2018).
- Xu, X., et al. Efficient homology-directed gene editing by CRISPR/Cas9 in human stem and primary cells using tube electroporation. Scientific Reports. 8 (1), 11649 (2018).
- Du, F., et al. Beneficial effect of young oocytes for rabbit somatic cell nuclear transfer. Cloning Stem Cells. 11 (1), 131-140 (2009).
- Allenspach, E., Rawlings, D. J., Scharenberg, A. M., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Friedman, J., et al., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Hidaka, N., et al. Most T790M mutations are present on the same EGFR allele as activating mutations in patients with non-small cell lung cancer. Lung Cancer. 108, 75-82 (2017).
- Ma, H., et al. Correction of a pathogenic gene mutation in human embryos. Nature. 548 (7668), 413-419 (2017).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Song, J., et al. Bacterial and Pneumocystis Infections in the Lungs of Gene-Knockout Rabbits with Severe Combined Immunodeficiency. Frontiers in Immunology. 9, 429 (2018).
- Song, J., et al. Production of immunodeficient rabbits by multiplex embryo transfer and multiplex gene targeting. Scientific Reports. 7 (1), 12202 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon