Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Optimisation des processus à l’aide de micro-bioréacteurs automatisés à haut débit dans la culture chinoise de cellules d’ovaires de Hamster
Dans cet article
Résumé
Ici, nous présentons une procédure détaillée pour exécuter une conception de l’expérience dans un micro-bioréacteur automatisé suivi par la récolte cellulaire et la quantification des protéines à l’aide d’une colonne de protéines A.
Résumé
L’optimisation des bioprocessus pour augmenter le rendement des produits désirés est importante dans l’industrie biopharmaceutique. Ceci peut être réalisé par la sélection des souches et par le développement de paramètres de bioprocessus. Des flacons de secousse ont été utilisés à cette fin. Ils n’ont toutefois pas la capacité de contrôler les paramètres du processus tels que le pH et l’oxygène dissous (DO). Cette limitation peut être surmontée à l’aide d’un micro-bioréacteur automatisé. Ces bioréacteurs imitent la culture à plus grande échelle. L’un des principaux avantages de ce système est l’intégration de la conception de l’expérience (DOE) dans le logiciel. Cette intégration permet d’établir une conception où plusieurs paramètres de processus peuvent être variés simultanément. Les paramètres critiques du processus et les conditions optimales de bioprocessus peuvent être analysés dans le logiciel. L’objectif du travail présenté ici est d’introduire l’utilisateur aux étapes impliquées dans la conception de processus dans le logiciel et l’incorporation du DOE dans la culture.
Introduction
Le marché biopharmaceutique mondial valait plus de 250 milliards de dollars AMÉRICAINS en 2018 et n’a cessé d’augmenterde 1. Les sociétés pharmaceutiques s’éloignent de la production de petits médicaments moléculaires à des thérapies biotechno logiquement produites telles que des protéines recombinantes. À eux seuls, ils représentent un chiffre d’affaires de plus de 150 milliards de dollars1. Les cellules mammifères sont maintenant largement utilisées pour la production de ces protéines recombinantes pharmaceutiques. Dans la période actuelle, parmi les 68 produits approuvés produits par des cellules de mammifères, 57 sont produits par les cellules ovaires hamster chinois (CHO)2. Les cellules CHO sont spécifiquement utilisées pour la production de protéines recombinantes qui nécessitent des modifications post-traductionnelles. Ces cellules sont préférées car elles se développent dans une suspension et permettent ainsi des résultats reproductibles dans un milieu chimiquement défini de sérum3,4. L’autre avantage de l’utilisation des cellules DE CHO est que la structure de glycan du produit ressemble à celle de l’anticorps monoclonal humain (mAb) et a comme conséquence le rendement plus élevé de protéine recombinante et la productivité spécifique due à l’amplification de gène5.
Le rendement de la culture cellulaire recombinante CHO (rCHO) a été multiplié par cent au cours des deux dernières décennies. Cette amélioration est attribuée à l’optimisation des paramètres du processus, à la stratégie d’alimentation et au développement du milieu6défini chimiquement par le sérum. Avec l’augmentation des besoins des produits pharmaceutiques, la pression augmente sur l’efficacité des coûts et du temps pour le développement du processus de production7. Pour réduire la pression tout en assurant la qualité du produit a redirigé l’accent de l’industrie pharmaceutique sur la qualité par la conception (QbD). QbD est utilisé pour comprendre la production du produit ainsi que le processus. Un outil essentiel utilisé dans l’ObD est la conception de l’expérience (DOE). Il contribue à mieux comprendre le processus en révélant la relation entre les différentes variables d’entrée et les données de sortie qui en résultent. L’application de l’approche DOE pour optimiser le bioprocessus est bénéfique au cours des premières étapes du projet dans l’assimiler les conditions du processus et d’augmenter la quantité et la qualité de titer. Cette approche est bénéfique par rapport à la stratégie à l’ancienne : un facteur à la fois (OFAT). Les approches statistiques du DOE à l’aide de Classical, Shainin ou Taguchi sont de loin supérieures à l’OFAT8.
Le processus et l’optimisation des médias peuvent être effectués dans des flacons de secousse. Les flacons sont relativement peu coûteux. Cependant, il n’est pas possible de contrôler des paramètres tels que la température, le pH et l’oxygène dissous (DO). Pour surmonter ces inconvénients, des bioréacteurs multi-utilisés de banc-top allant du volume de travail de 0,5 L à 5 L peuvent être utilisés. Les réacteurs assurent une surveillance et un contrôle complets des procédés en ligne. Cependant, l’utilisation du bioréacteur multi-utilisation est exigeante en temps et en main-d’œuvre. Afin de surmonter ces inconvénients, un nouveau bioréacteur à usage unique qui combine le processus complet de surveillance du bioréacteur de banc-dessus et la manipulation facile du flacon de secousse est utilisé. Le système de criblage à haut débit et la technologie à usage unique ont contribué à améliorer l’efficacité de la performance et du développement des processus9.
Dans cet article, les lignes directrices pour charger la recette dans le logiciel automatisé de micro-bioréacteur (AMBR) sont énumérées. L’influence de différentes vitesses d’agitateur et de pH sur la concentration de cellules viables (VCC) et le titer est étudiée au cours de cette expérience. Le résultat expérimental et l’analyse sont effectués avec la conception du logiciel d’expérience MODDE 12. L’analyse du produit est effectuée dans un système de chromatographie liquide à haute pression (HPLC) avec une colonne Protéique A. Il est basé sur le principe que la région de Fc du mAb se lie à la protéine A avec une affinité élevée10,11. Avec cette méthode, il est possible d’identifier et de quantifier le mAb. La quantification est effectuée sur les zones mesurées de pointe d’elution à 280 nm.
Protocole
1. Procédure de préculture
REMARQUE: Recombinant CHO DG44 cellules avec une concentration cellulaire viable de 1 x 107 cellules/mL sont utilisés pour ce protocole.
- Décongeler la fiole contenant 1,2 ml de cellules à température ambiante et transférer immédiatement la suspension cellulaire dans un tube de centrifugeuse conique de 15 ml contenant 10 ml de milieu de graines froides.
- Centrifuge le tube de centrifugeuse conique pendant 5 minutes à 190 x g et température ambiante et jeter le supernatant.
- Préchauffer 150 ml du milieu de graine dans un flacon de secouement de 500 ml à 36,8 oC.
- Réutilisez délicatement la pastille cellulaire dans 10 ml de milieu de semence préchauffé et transférez les cellules dans le flacon de secousse.
- Utilisez 1 ml d’échantillon du flacon pour mesurer le VCC initial et la viabilité à l’aide d’un compteur cellulaire.
REMARQUE: La viabilité devrait être supérieure à 70 % après le dégel pour une culture réussie. - Incuber le flacon de secousse dans un shaker orbital (diamètre de shaker de 19 mm) à 36,8 oC et 7,5% de CO2 avec un taux de secousse de 120 tr/min.
REMARQUE: Ces conditions varient en fonction de la souche cellulaire et moyenne. - Trois jours après avoir passé les cellules, retirer le flacon de shake du shaker et le placer sous l’armoire à écoulement lamineuse. Prenez 1 ml d’échantillon pour mesurer la concentration cellulaire finale. Calculer le volume à transférer au milieu frais des graines pré-réchauffées de telle sorte que la concentration cellulaire initiale dans le nouveau passage est de 2 x 105 cellules/mL.
- Passer les cellules 5 fois au total avant de passer au bioréacteur pour la culture principale.
2. Culture principale
- Mesurer la concentration cellulaire finale de la préculture. Calculer le volume à transférer au bioréacteur de telle sorte que la concentration cellulaire initiale dans le réacteur est de 3 x 105 cellules/mL.
- Remplissez le réacteur avec le milieu de production un jour avant l’inoculation pour équilibrer le réacteur et définir les paramètres de processus tels que la température, le pH et le DO.
REMARQUE: Les conditions de culture sont de 36,8 oC et de 60 % de concentration dissoute en oxygène (DO). Nous avons testé des vitesses d’agitateur de 1050 tr/min et 1300 tr/min avec des pH de 6,9, 7,1 et 7,3. La durée totale de la culture est de 12 jours jusqu’à ce que les cellules soient récoltées. Le processus de lot s’exécute pendant 72 heures après quoi le milieu d’alimentation est ajouté tous les 24 h. Le protocole à utiliser pour la culture est répertorié en détail dans le segment suivant.
3. Rédaction de la recette dans le logiciel micro-bioréacteur automatisé
REMARQUE: Il existe deux façons d’écrire une recette dans le logiciel de culture cellulaire AMBR: il est créé soit en utilisant un assistant ou en ajoutant chaque étape manuellement. Aux fins de ce protocole, les étapes utilisant l’assistant sont affichées.
- Création d’une nouvelle expérience
- Ouvrez le logiciel de culture cellulaire AMBR et dans l’onglet Introduction cliquez sur Créer une nouvelle expérience.
- Chargement de la recette
- Dans l’onglet Nouvelle Expérience, entrez le nom de l’expérience avec la date à laquelle elle doit être menée.
- Activez le point de contrôle de la station de culture et des navires à utiliser pendant la culture. Les étiquettes Auto Add DOE seront également activées pour une transition facile pendant la programmation de l’expérience DOE. Cliquez sur Next pour passer à l’onglet suivant.
- Définissez des informations sur l’ajout de médias dans le vaisseau avec l’antifoam, l’inoculum, l’alimentation et le glucose.
- Activez le point de contrôle Add Media Plate. Définissez le type de plaque, le nom et l’emplacement de la plaque contenant le support.
CAUTION: Selon le type de plaque et si la plaque contient un couvercle, activez la vérification sur Est Lidded pour assurer le bon fonctionnement du gestionnaire liquide - Cliquez sur Ajouter des médias aux navires. Entrez le volume des médias à ajouter dans les navires. Définir la cartographie du transfert des supports de la plaque vers les navires. Cliquez sur Next pour passer à l’onglet suivant.
- Activez le point de contrôle Add Media Plate. Définissez le type de plaque, le nom et l’emplacement de la plaque contenant le support.
- Définissez les conditions de culture dans le réacteur.
- Après que les informations des médias ont été introduites dans le logiciel, assignez les conditions de culture. Cliquez sur Condition Media et remplissez la température, ciblez DO, limite de pH supérieure et remuez RPM (Up stirring ou Down stirring).
- Définissez l’ajout d’inoculums dans les navires.
- Activez Ajouter la plaque de cellules. Définissez le type de plaque, le nom et l’emplacement de la plaque contenant le support.
- Cliquez sur Ajouter des cellules aux navires. Entrez l’heure de l’inoculation et le volume des médias à ajouter aux navires.
- Décrivez le chemin parcouru par le gestionnaire liquide jusqu’au transfert de la cellule de la plaque vers les navires. Cliquez sur Next pour passer à l’onglet suivant.
REMARQUE: Assurez-vous que les conseils de pipette de réutilisation sont désactivés pour éviter la contamination croisée et une mauvaise concentration initiale viable des cellules.
- Définir l’ajout d’aliments pour animaux, de glucose et d’antifoam.
REMARQUE: La procédure d’ajout d’aliments, de glucose et d’antifoam est similaire les unes aux autres. Pour le bien de ce protocole, la procédure est répertoriée pour "Feed". Cela peut être reproduit pour le glucose et l’antifoam.- Activez la plaque d’alimentation Add et définissez le type, le nom et l’emplacement de la plaque. Cliquez sur Ajouter de l’alimentation aux navires et entrez le volume de l’alimentation à ajouter aux navires. Définir la cartographie du transfert de l’alimentation de la plaque vers les navires.
- Selon la culture, ajouter le nombre d’ajouts d’aliments pour animaux. Pour cette culture, le réacteur est alimenté après 72 heures toutes les 24 heures.
- Ajouter manuellement le délai entre l’alimentation en entrant les données dans Delay from cells added. Le premier jour d’alimentation est après 72 heures d’inoculation et le prochain est après 96 heures et ainsi de suite.
REMARQUE: L’ajout d’antifoam est programmé pour être ajouté tous les jours pour éviter l’écume pendant la culture.
- Définir l’échantillonnage pendant la culture.
- Activez la plaque d’échantillon d’ajouter et définissez le type de plaque, le nom et l’emplacement.
- Vérifiez l’échantillon de prélèvement des navires et entrez le volume de l’échantillon à retirer des navires. Définir la cartographie du transfert de l’échantillon des navires vers la plaque. Assurez-vous que le volume ne diminue pas en dessous de 10 ml pendant toute la culture.
- Ajouter le nombre d’échantillons à prélever pendant la culture. Semblable à l’alimentation, ajoutez le temps de l’échantillon retiré du navire pour chaque point d’échantillonnage d’entrée.
- Enregistrer le processus. Il est maintenant prêt pour l’exécution.
REMARQUE: Pour assurer le bon fonctionnement du protocole, passez à l’onglet Étapes de processus dans le logiciel de culture cellulaire AMBR et sélectionnez la vue Process Step pour visualiser le flux de la recette.
- Dans l’onglet Nouvelle Expérience, entrez le nom de l’expérience avec la date à laquelle elle doit être menée.
- Conception de l’expérience dans le microréacteur automatisé
- Afin d’exécuter le logiciel DOE du bioréacteur, assurez-vous que la recette dans le logiciel principal est enregistrée et prête à l’emploi.
- Ouvrez le logiciel AMBR 15 DOE et cliquez sur Investigation et sélectionnez Nouveau.
- Entrez le nom de la nouvelle enquête du DOE dans la boîte de dialogue Create Investigation.
- Afin d’attribuer une expérience à l’enquête du DOE, ouvrez la recette créée pour étudier les différents paramètres. Cliquez sur Parcourir et sélectionnez l’expérience respective.
- Définissez le facteur DOE.
- Les étiquettes du navire sont déjà enrôlées dans la colonne. Pour définir le facteur DOE souhaité, sélectionnez le paramètre et cliquez sur le facteur DOEétiqueté colonne . Sélectionnez Nouveau et ajoutez les unités, l’abréviation, la limite inférieure et supérieure des facteurs (p. ex., température, DO, pH).
- Définissez le facteur de réponse.
- Une fois que les facteurs du DOE ont été définis, définissez la réponse en fonction de laquelle l’analyse expérimentale serait structurée.
- Dans l’onglet Réponses, définissez les valeurs à prendre en considération pour l’analyse des données.
- Cliquez sur Modifier les réponses doE et définir le nom de la réponse, l’abréviation, les unités, les limites minimales et maximales (p. ex., titer, concentration cellulaire viable).
- Une fois les réponses définies, sélectionnez la variable AMBR pour chaque réponse et définissez la variable. Une réponse peut être automatiquement associée à une variable micro-bioréacteur, Choisissez la variable requise de la liste déroulante.
- Modifier l’équation pour chacune des réponses en fonction de l’exigence. Le choix se situe entre les données minimales, maximales, d’abord, dernières et moyennes.
- Créez un design.
- Utilisez le Start Design Wizard afin de sélectionner le type de conception expérimentale, pour ajouter ou supprimer le nombre de répliques et de points de centre.
- Sélectionnez l’objectif, qui détermine le choix des conceptions et des modèles :
Dépistage : Utilise des modèles linéaires et d’interaction pour trouver les facteurs importants
Optimisation (RSM), utilise des modèles quadratiques et cubiques pour la modélisation et l’optimisation détaillées
Objectif fractionné : Les modèles de formulation et de facteurs de processus peuvent être choisis séparément - Une fois l’objectif décidé, sélectionnez le modèle et la conception ainsi que le nombre de points de centre et de répliques.
- Cliquez sur Finition et passez à l’onglet suivant.
- Définissez l’expérience.
REMARQUE: Les facteurs DOE sont répertoriés dans la colonne droite du logiciel. En sélectionnant les facteurs souhaités, les navires qui exécutent cette expérience avec le paramètre souhaité seraient mis en évidence. Les navires à l’intérieur de la station de culture peuvent être déplacés en cliquant à droite sur le navire et en le déplaçant à l’endroit désiré.- Créez des paquets de travail qui peuvent être importés dans le logiciel de culture cellulaire AMBR. Selon le nombre d’expériences, les différents paquets de travail sont créés et stockés pour une mise en œuvre ultérieure
- Exécution de l’expérience dans les paquets de travail créés sur l’ordinateur portable de contrôle AMBR
- Dans l’onglet Expérience, cliquez sur Create DOE Experiment et naviguez pour le paquet de travail créé à l’aide du logiciel DOE.
- Initialiser le processus en cliquant sur Démarrer.
- Analyse des résultats expérimentaux
- Une fois l’expérience exécutée, exportez les données à l’aide des résultats du DOE à l’exportation. La fenêtre des résultats du DOE à l’exportation s’ouvre et les rangées indiquant le navire et la station de culture sont répertoriées dans le tableau.
- Sélectionnez les lignes souhaitées et cliquez sur les lignes sélectionnées exportées ou les données expérimentales d’exportation pour stocker tous les résultats et enregistrer le fichier pour une analyse plus approfondie.
- Importer les données dans le module AMBR DOE en passant à l’onglet Résultats et en sélectionnant les résultats d’importation.
- Parcourez le fichier de données souhaité et cliquez sur les résultats d’analyse.
- Analyser les résultats plus loin dans MODDE.
4. Exécution de la culture dans le micro-bioréacteur automatisé
REMARQUE: Les étapes suivantes sont exécutées par l’utilisateur à l’aide du protocole écrit dans le logiciel susmentionné. Les étapes sont effectuées par l’utilisateur sauf indication contraire.
- Chargement des navires
- Ouvrez les récipients de culture gamma stérilisés sous un armoire à débit lamineur et orientez-vous dans la station de culture telle qu’elle est représentée à la figure 2.
- Nettoyer la plaque de pince avec 70% d’éthanol et d’eau distillée double. Ensuite, autoclavez la plaque et placez-le au-dessus du navire.
- Monter la plaque de pince avec une plaque d’agitateur, en veillant à ce que chaque broche soit fixée fermement.
- Resserrer à la fois la plaque d’agitation et serrer la plaque sur l’assemblage en remuant.
- Exécution du logiciel de micro-bioréacteur
- Utilisez le programme écrit à la section 3 pour exécuter la culture.
- Visualiser les étapes du processus prévues ou complétées dans l’onglet Processus. Modifier les étapes de culture pendant le processus exécuté au besoin en arrêtant d’abord le gestionnaire liquide, puis en modifiant la recette du processus.
- Ajouter l’antifoam aux vaisseaux avant que l’agitateur ne commence à s’assurer qu’il n’y a pas d’écume excessive pendant la culture. L’antifoam sera ajouté régulièrement, et la mousse détectée visuellement.
- Ajout de médias dans le navire
- Remplissez manuellement la plaque de puits 24 fournie avec les micro-bioréacteurs avec des supports stériles et placez-les dans le pont désigné du système. Assurez-vous que la plaque est placée dans le tablier désigné par le programme écrit (article 3). Le remplissage du navire aura lieu tel que prévu à la section 3.2.2.
REMARQUE: La température et l’agitateur commencent immédiatement après l’ajout des médias et de l’antifoam. Le lecteur du capteur est activé 1 heure après la remplissage du vaisseau (étape Démarrer le moniteur). Le gazage à chaque navire commence une fois que le lecteur a été activé. Les médias sont laissés à l’équilibre pendant au moins 6 heures avant le recalibrage et l’inoculation du pH. Les paramètres du processus peuvent être modifiés dans le logiciel comme mentionné dans la section 3.2.3.
- Remplissez manuellement la plaque de puits 24 fournie avec les micro-bioréacteurs avec des supports stériles et placez-les dans le pont désigné du système. Assurez-vous que la plaque est placée dans le tablier désigné par le programme écrit (article 3). Le remplissage du navire aura lieu tel que prévu à la section 3.2.2.
- Inoculation
- Mesurer la concentration cellulaire viable après le 5e passage. Calculer le nombre de cellules à transférer aux vaisseaux pour s’assurer que la concentration cellulaire initiale dans tous les vaisseaux est de 3 x 105 cellules/mL.
- Transférer les cellules à une plaque de puits de 24 profondeurs de telle sorte que le volume de la suspension est au moins 1,6 fois le volume requis. Pour un volume requis de 2 ml de l’inoculum, transférer 3,2 ml de suspension cellulaire dans chaque puits dans la plaque.
- Placez la plaque de puits 24 dans le pont désigné. Les navires seront inoculés comme à la section 3.2.4.
- Échantillonnage et analyse quotidiens
- Retirez chaque jour un échantillon de 460 L des navires à l’aide du gestionnaire liquide. Diluer 200 L de l’échantillon avec 800 l de tampon 1x PBS filtré (5x dilution), puis placer dans le compteur cellulaire.
- Centrifuge l’échantillon restant pendant 5 min à 190 x g et température ambiante et stockez le supernatant pour une analyse plus approfondie (glucose, lactate, glutamine et glutamate).
- Congeler 100 l’un des supernatants à -20 oC jusqu’à la fin de la culture pour la quantification des protéines.
- Fin de la culture
- Lorsque le contrôle des paramètres du processus (c.-à-d. la température, l’agitation, le pH et le DO) a pris fin, arrêtez la surveillance du processus.
- Dévisser la plaque d’agitateur et la plaque de pince.
- Enlevez les vaisseaux culturels et nettoyez les stations de culture. Placez les plaques de séchage sur les stations de culture et visser.
- Pendant ce temps, nettoyer les plaques de pince à fond avec 70% d’éthanol et d’eau distillée double.
- Cliquez sur Stop dans le logiciel bioréacteur une fois le cycle de séchage terminé.
- Récolte de culture cellulaire
- Récolter les cellules le jour 12 de la culture en enlevant manuellement le contenu des vaisseaux en tubes de centrifugeuse de 50 ml. Centrifuge le bouillon de cellules à 190 x g pendant 30 min.
- Jetez le granule cellulaire et entreposez le supernatant à -20 oC.
5. Mesurer la concentration de mAb
- Utilisez une colonne de protéines A de 1,7 ml pour la quantification de la protéine pendant la course de culture.
-
Préparer l’équilibre et le tampon d’elution avant de décongeler les échantillons.
- Utilisez une solution de 0,5 M Na2HPO4 contenant 0,5 M NaCl avec un pH de 7,9 comme tampon d’équilibre et une solution de glycine de 100 mM contenant 0,5 M NaCl avec un pH de 2 comme tampon d’élétisation.
- Filtrer les deux tampons à l’intermédiaire d’une membrane de 0,2 m et de dégazer avant d’être placé pour l’analyse.
- Purger le système de chromatographie liquide haute performance (HPLC) avec tampon d’équilibre fraîchement préparé.
- Chargez la colonne de protéine A sur le système HPLC.
- Effectuer la chromatographie avec un débit de 1 mL/min. Réglez la température du four à colonne à 30 oC et l’autosampler température à 10 oC
- Décongeler les échantillons congelés à température ambiante et filtrer 225 L de chaque échantillon à travers une membrane de 0,22 m PVDF. Diluer les échantillons avec une concentration plus élevée de la protéine désirée sont dans un rapport de 1:20 avec tampon d’équilibre et filtrer à travers la membrane avant de placer dans l’autosampler.
- Placez les échantillons dans l’autosampler. Chargez la méthode et la séquence dans le logiciel et démarrez la séquence.
REMARQUE: La méthode est composée de trois phases (voir la figure 1) : injection de l’échantillon dans la colonne pendant les deux premières minutes; suivi d’un tampon d’elution pendant 8 min et d’une régénération de colonne avec tampon d’équilibre pendant 10 min.
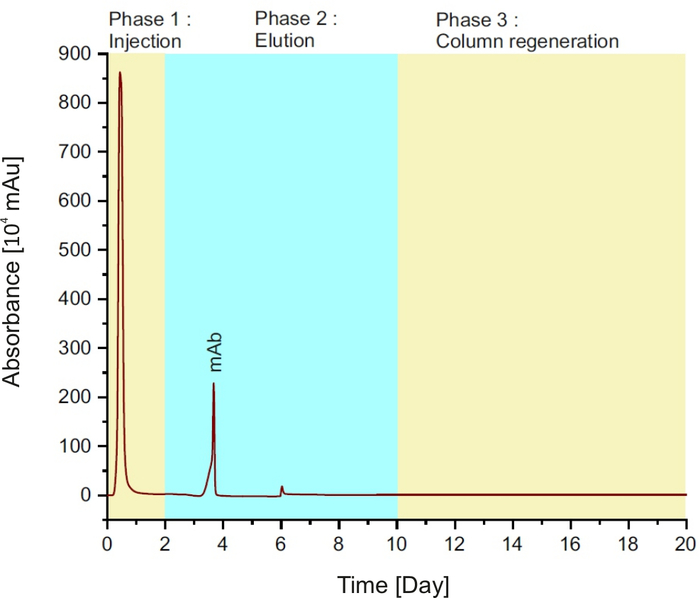
Figure 1 : chromatogramme de protéine A, représentant les différentes phases au cours d’une seule course. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Résultats
Un aperçu de la culture pratiquée dans cette étude est présenté à la figure 2.
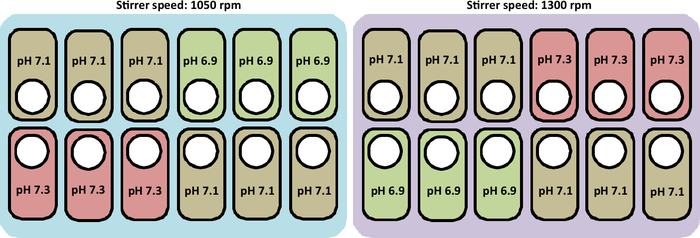
Figure 2 : Représentation schématique des conditions expérimentales pour tester les profils de pH et de vitesse d’agitateur dans les stations de culture. Le chiffre représente également la disposition correcte pour placer les navires.
Discussion
L’optimisation du processus d’augmentation du rendement est d’une importance cruciale dans l’industrie biopharmaceutique. Des flacons de secousse pourraient être utilisés pour le criblage de la souche; cependant, la surveillance des paramètres du processus tels que le pH et le DO ne sont pas disponibles dans les flacons. Les micro-bioréacteurs ont un avantage car ils permettent une surveillance et un contrôle continus du processus. Ces boucles de contrôle dans le micro-bioréacteur fournissent également un...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs souhaitent remercier le Bundesministerium f’r Bildung und Forschung (BMBF), le Ministère fédéral de l’éducation et de la recherche, en Allemagne, et l’équipe bioprocessue de Sartorius Stedim Biotech GmbH, en Allemagne, pour leur soutien.
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL disposable pipette tips, sterilized | Sartorius Stedim Biotech GmbH | A-0040 | |
| 200 mM L-glutamine | Corning, Merck | 25-005-CV | |
| 24 Well deep well plates | Sartorius Stedim Biotech GmbH | A-0038 | |
| 5 mL disposable pipette tips, sterilized | Sartorius Stedim Biotech GmbH | A-0039 | |
| ambr 15 automated microbioreactor system | Sartorius Stedim Biotech GmbH | 001-2804 | |
| ambr 15 Cell Culture 24 Disposable Bioreactors - Sparged | Sartorius Stedim Biotech GmbH | 001-1B86 | |
| Antifoam C Emulsion | Sigma-Aldrich, Merck | A8011 | |
| Bottle Top Sterile filter | Corning, Merck | CLS431474 | 0.1 μm pore size |
| CEDEX Detergent (3% Mucosol) | Roche Innovatis AG | 05-650-658-001 | |
| Cell counter | Roche Innovatis AG | 05-650-216-001 | CEDEX HiRes |
| CHO DG44 cell line | Cellca, Sartorius Stedim Biotech GmbH | ||
| CHOKO Feed Media A (FMA) | Sigma-Aldrich, Merck | CR80025 | |
| CHOKO Feed Media B (FMB) | Sigma-Aldrich, Merck | CR80026 | |
| CHOKO Production Medium | Sigma-Aldrich, Merck | CR80027 | |
| CHOKO Stock Culture Meium | Sigma-Aldrich, Merck | CR80028 | |
| Chromaster high pressure liquid chromatography system | VWR International | ||
| Conical Centrifuge tube | Corning, Merck | SIAL0790 | |
| Ethanol | Merck | 1070179026 | |
| Glycine | Carl Roth | 56-40-6 | |
| HPLC Vials | VWR International | SUPLSU860181 | |
| PBS | Sigma-Aldrich,Merck | P4417 | |
| Protein A Column | Thermo Fisher Scientific | 1502226 | POROS™ A 1.7 mL |
| Sodium chloride | Sigma-Aldrich,Merck | 7647-14-5 | |
| Sodium phosphate dibasic anhydrous | Sigma-Aldrich,Merck | 7558-79-4 | |
| Trypan Blue | VWR International | VWRVK940 | |
| YSI | YSI Inc | 2900D | YSI 2900 Select |
Références
- Walsh, G. Biopharmaceutical benchmarks 2018. Nature Biotechnology. 36, 1136 (2018).
- Kim, J. Y., Kim, Y., Lee, G. M. CHO cells in biotechnology for production of recombinant proteins: current state and further potential. Applied Microbiology and Biotechnology. 93 (3), 917-930 (2012).
- Lai, T., Yang, Y., Ng, S. K. Advances in Mammalian cell line development technologies for recombinant protein production. Pharmaceuticals (Basel). 6 (5), 579-603 (2013).
- Carlage, T., et al. Analysis of dynamic changes in the proteome of a Bcl-XL overexpressing Chinese hamster ovary cell culture during exponential and stationary phases. Biotechnology Progress. 28 (3), 814-823 (2012).
- Hacker, D. L., de Jesus, M., Wurm, F. M. 25 years of recombinant proteins from reactor-grown cells - where do we go from here. Biotechnology Advances. 27 (6), 1023-1027 (2009).
- Shukla, A. A., Gottschalk, U. Single-use disposable technologies for biopharmaceutical manufacturing. Trends in Biotechnology. 31 (3), 147-154 (2013).
- Ao, S., Gelman, L. . Advances in electrical engineering and computational science. Lecture notes in electrical engineering. 39, (2009).
- Bareither, R., et al. Automated disposable small scale reactor for high throughput bioprocess development: a proof of concept study. Biotechnology and Bioengineering. 110 (12), 3126-3138 (2013).
- Kang, Y., Ludwig, D. L., Balderes, P. What can cell culture flocculation offer for antibody purification processes. Pharmaceutical Bioprocessing. 2 (6), 483-485 (2014).
- Choe, W., Durgannavar, T. A., Chung, S. J. Fc-Binding Ligands of Immunoglobulin G: An Overview of High Affinity Proteins and Peptides. Materials (Basel). 9 (12), (2016).
- Schäpper, D., et al. Application of microbioreactors in fermentation process development: a review. Analytical and Bioanalytical Chemistry. 395 (3), 679-695 (2009).
- Zhang, Z., et al. Microbioreactors for Bioprocess Development. Journal of the Association for Laboratory Automation. 12 (3), 143-151 (2007).
- Claßen, J., et al. Spectroscopic sensors for in-line bioprocess monitoring in research and pharmaceutical industrial application. Analytical and Bioanalytical Chemistry. 409 (3), 651-666 (2017).
- Janoschek, S., et al. A protocol to transfer a fed-batch platform process into semi-perfusion mode: The benefit of automated small-scale bioreactors compared to shake flasks as scale-down model. Biotechnology Progress. 35 (2), 2757 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon