Method Article
Protocole d’électroporation universel et efficace pour le génie génétique des organoïdes gastro-intestinaux
Dans cet article
Résumé
Ce protocole décrit une méthode efficace d’électroporation pour la transfection de quatre entités organoïdes gastro-intestinales différentes avec de plus grands plasmides (à la mesure de 10 kB). Il peut être exécuté dans un jour et n’a pas besoin de préparation étendue ou de tampons d’électroporation spéciaux et coûteux.
Résumé
L’électroporation est une méthode courante pour la transfection avec différents types de molécules par perméabilisation électrique de la membrane plasmatique. Avec l’utilisation croissante des organoïdes comme méthode de culture pour le matériel patient primaire dans les dernières années, des méthodes efficaces de transfert des composants pour le génie génétique dans ce système de culture 3D sont dans le besoin. En particulier pour les organoïdes, l’efficacité des manipulations génétiques dépend d’une transfection réussie. Ainsi, ce protocole a été développé pour faciliter l’électroporation des organoïdes et pour prouver sa fonctionnalité universelle dans différentes entités. Les organoïdes de cancer côlorectal, pancréatique, hépatique et gastrique humain ont été électroporated avec avec avec de petits et grands plasmides en comparaison. Sur la base des vecteurs de codage GFP, l’efficacité de la transfection a été déterminée par LE FACS. Aucune préparation complète des cellules ou des tampons d’électroporation spéciaux et coûteux n’est nécessaire, et le protocole peut être exécuté en un jour.
Introduction
Ces dernières années, un nouveau système de culture cellulaire 3D, appelé organoïdes, a été développé pour divers tissus normaux et cancéreux. Les organoïdes sont fonctionnellement et morphologiquement très proches de leur tissu d’origine. Ils peuvent être générés à partir de différentes espèces, sont facilement extensibles, génomiquement stables et génétiquement modifiables, ce qui en fait un système modèle idéal pour les recherches génétiques1,2,3. Les techniques de génie génétique comme le système CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas9 permettent diverses manipulations. La sélection des clones peut être réalisée par des conditions médiatiques définies, par exemple, par wNT ligand retrait pour APC (Adenomatosis Polyposis Coli) clones knock-out4,5. Alternativement, les marqueurs de sélection doivent être introduits par la réparation dirigée homologue d’un vecteur de ciblage6,7. En raison du fait que souvent plus d’un plasmide doit être introduit, une transfection efficace devient un paramètre crucial. En outre, pour réduire les effets hors cible non spécifiques, une expression transitoire de l’endodoucane Cas9 est souhaitable8.
L’électroporation est une méthode relativement simple pour transfect cellules avec de l’ADN, de l’ARN, des protéines ou d’autres macromolécules. Au moyen d’impulsions électriques, la membrane cellulaire devient plus perméable et provoque une augmentation de l’utilisation9. Dans un protocole d’électroporation précédemment édité des organoïdes du côlon une efficacité de 30 % avec un GFP de piggy-bac (protéine fluorescente verte) exprimant le vecteur (7.4 kB) a été atteint dans une procédure de quatre jours10. Le protocole suivant a été développé pour faciliter une transfection efficace des organoïdes cancéreux ou sains avec de grands plasmides codant pour l’ARN de guide simple (sgRNA) et la séquence d’endonucb de Cas9 (par exemple px458 comme vecteur avec 9.3 kb). L’ensemble du processus d’électroporation peut être effectué en un jour, sans tampons d’électroporation spéciaux, et avec au moins des efficacités comparables entre les différents organoïdes gastro-intestinaux, à savoir l’adénocarcinome canalaire pancréatique (PDAC), le cancer colorectal (CRC), le cholangioccinome (CCC) et les organoïdes du cancer gastrique (GC).
Protocole
L’approbation éthique a été obtenue du comité d’éthique de la TU Dresde (#EK451122014).
1. Culture organoïde et préparations avant l’électroporation
- Établir les organoïdes par digestion des tissus tel que décrit précédemment et les étendre avec leur entité correspondante médium de culture spécifique dans une matrice de sous-sol (aperçu voir tableau 1 et tableau des matériaux)11,12,13,14,15,16,17.
REMARQUE : Pour les échantillons de tissus humains, un consentement éclairé et l’approbation de l’étude par un comité d’éthique sont nécessaires. - Préémindre les plaques de 48 puits à 37 oC pour l’ensemencement post-électroporation.
- Préparer les antibiotiques basiques w/o moyens ainsi que les antibiotiques organiques de culture organoïde spécifiques à l’entité (voir le tableau 1) y compris 10 M Y-27632 et 3 M CHIR99021.
REMARQUE : Le retrait des antibiotiques est important pour réduire les effets toxiques. Y-27632 et CHIR99021 améliorent la récupération cellulaire10. -
Préparation des organoïdes (voir Figure 1)
- Cultiver 5 puits d’organoïdes dans une plaque de 48 puits par échantillon d’électroporation dans le milieu de culture.
REMARQUE : Les organoïdes proliférants doivent être utilisés (environ 2-3 jours après la dernière fractionnement). - Préparer 230 L de réactif ssocion (voir Tableau des matériaux)dont 10 M Y-27632 par puits.
- Retirer le milieu de culture de chaque puits et dissocier mécaniquement les organoïdes en 230 l du mélange de dissociation préparé. Piscine 5 puits par échantillon d’électroporation dans un tube de 15 ml.
- Mélanger par vortex et incuber de 5 à 15 min à 37 oC jusqu’à ce que des grappes de 10 à 15 cellules se produisent. Par conséquent, vérifiez la dissociation au microscope. Arrêter la digestion en ajoutant des antibiotiques basaux moyens w /o jusqu’à 10 ml.
REMARQUE: Cette étape est très critique! L’efficacité de l’électroporation sera réduite, lorsque l’incubation est trop courte, mais une longue digestion réduira la capacité de survie. - Centrifugeuse à 450 x g pendant 5 min à température ambiante, jetez le supernatant et lavez deux fois avec 4 ml de tampon d’électroporation (voir Tableau des matériaux).
- Cultiver 5 puits d’organoïdes dans une plaque de 48 puits par échantillon d’électroporation dans le milieu de culture.
2. Électroporation
REMARQUE : Le protocole suivant est développé pour les électroporateurs capables d’ondes carrées et de séquences d’impulsions séparées (voir figure 2). En option, les valeurs d’impédance ainsi que les tensions, les courants et les énergies transférés dans l’échantillon peuvent être mesurés comme contrôle pour les expériences reproductibles.
- Resuspendre la pastille organoïde dans 100 l de tampon d’électroporation (voir Tableau des matériaux) contenant 30 g d’ADN plasmide.
REMARQUE : La concentration de l’ADN plasmide utilisé devrait dépasser 5 g/L pour une concentration optimale de sel pendant le processus d’électroporation. Par conséquent, endofree plasmide maxi kits (voir Tableau des matériaux) pour la préparation des vecteurs sont recommandés. Une quantité totale de 45 g d’ADN peut être utilisée sans effets cytotoxiques. - Distribuez le mélange complet ADN-organoïde dans une cuvette d’électroporation de 2 mm de largeur sans produire de bulles d’air.
- Définiz les paramètres d’électroporation en fonction de Fujii et coll.10 (voir tableau 2, Figure 2).
- Mélanger légèrement les cellules sans mousser en tapant sur la cuvette avec un doigt. Placer la cuvette dans la chambre de cuvette.
- Appuyez sur le bouton '' de l’électroporateur et notez la valeur de l’impédance.
REMARQUE: Une impédance entre 30-40 a montré les meilleurs résultats. En général, il devrait être dans la gamme entre 30-55 . Si ce n’est pas le cas, s’il vous plaît contrôler les aspects suivants: largeur d’écart de la cuvette utilisée, connexions par câble de l’électroporateur, bulles d’air possibles, volume correct et la concentration de sel du mélange d’électroporation. - Appuyez sur le bouton Démarrer pour exécuter le programme d’électroporation et contrôler les valeurs des courants, des tensions et des énergies affichées.
REMARQUE : Les valeurs des tensions, des courants et des énergies mesurés doivent correspondre aux paramètres d’électroporation fixés. Pour la comparaison des expériences répétées, il peut être utile de noter ces données. - Après l’électroporation, ajoutez immédiatement 500 'L d’antibiotiques w/o moyens de culture (avec CHIR99021 et Y-27632 ; voir l’étape 1.3). Mélanger en pipetting de haut en bas pour dissocier la mousse blanche.
REMARQUE : La mousse blanche apparaît après le processus d’électroporation et un nombre significatif de cellules y est attachée. Donc, la dissociation de celui-ci est très important pour ne pas perdre des cellules. - Transférer complètement l’échantillon de la cuvette dans un nouveau tube de 15 ml à l’aide de la pipette appartenant aux cuvettes d’électroporation (voir Tableau des matériaux). Il est recommandé de rincer à nouveau la cuvette avec le milieu basal pour obtenir les cellules restantes.
- Pour la régénération des cellules, incuber pendant 40 min à température ambiante.
3. Ensemencement des cellules
- Centrifuger les cellules à 450 x g pendant 5 min à température ambiante et jeter le supernatant.
- Resuspendre le granule dans 100 l de matrice de sous-sol et de graines 20 gouttes de L dans une plaque préchauffée de 48 puits (voir l’étape 1.2). Incuber pendant 10 min à 37 oC pour la polymérisation et ajouter 250 l de milieu de culture, qui est complété par Y-27632 et CHIR99021 jusqu’à la prochaine division des organoïdes cultivés (environ 5-7 jours).
4. Détermination de l’efficacité de la transfection
REMARQUE : En général, il est recommandé d’électroporater un vecteur porteur d’un marqueur de fluorescence comme contrôle de transfection supplémentaire. Selon le marqueur choisi et sa maturation chromophore la fluorescence sera visible dans environ 24-48 h après la transfection18.
- Vérifier la fluorescence au microscope après 24-48 h dans le contrôle de la transfection (voir La figure 4B).
-
Numérisation des cellules activées fluorescentes (FACS)
- Récoltez les cellules de façon analogue à l’étape 1.4.2-1.4.4 et digérez autour de 10-20 min jusqu’à ce qu’il y ait des cellules simples. Ajouter jusqu’à 10 ml de salin tamponné par le phosphate (PBS).
- Centrifugeuse à 450 x g pendant 5 min à température ambiante et jetez le supernatant.
- Optionnel pour discrimination des cellules vivantes : suspendez le granule dans 1 ml de saline tamponnée en phosphate (PBS) et ajoutez un anticorps approprié (voir Tableau des matériaux)ou de l’iodure de propidium (PI). Mélanger très soigneusement seulement en tapant et incuber pendant 30 min à température ambiante dans l’obscurité. Laver avec 10 ml de PBS, centrifuger et jeter le supernatant.
- Resuspendre la pastille cellulaire dans 200 L de PBS et filtrer la suspension par une passoire cellulaire de 100 m dans un tube FACS.
- Analyser les cellules à l’aide d’une machine FACS à l’aide d’une stratégie de gating appropriée (voir la figure 3; Figure 4A) et déterminer l’efficacité de la transfection.
Résultats
Les organoïdes de quatre entités cancéreuses différentes (CRC, CCC, PDAC, GC) ont été électroporated au moins 3 fois utilisant 30 g d’un petit plasmide (pCMV-EGFP, 4.2 kb) ou un grand plasmide (px458, 9.3 kb). Les deux vecteurs portent une cassette GFP permettant la détermination de l’efficacité de la transfection 48 h après l’électroporation par cytométrie de flux. Pour analyser seulement les cellules vivantes, coloration avec un anticorps de vie-mort avant balayage a été exécuté. La stratégie de gating est présentée à la figure 3.
Dans les quatre entités organoïdes, le plasmide de 4,2 kB a été transfecté avec une plus grande efficacité que le plasmide de la taille 4 ( La transfection la plus efficace du petit plasmide a été atteinte chez les organoïdes PDAC avec 92,1 - 5,2 % de cellules positives GFP, tandis que le grand plasmide a été transfecté avec une efficacité de 46,7 à 3,7 % (écart standard moyen, n - 3). Comparé aux organoïdes du cancer du pancréas, le plasmide plus grand a été transfecté plus efficacement en organoïdes crC avec une efficacité moyenne de 53,4 à 11,7 %, tandis que le petit plasmide a été transfecté avec une efficacité moyenne de 84,3 à 5,8 %. L’entité la plus difficile à transfect étaient les organoïdes gastriques de cancer : pour le grand et le petit plasmide, l’efficacité la plus basse de transfection a été atteinte dans cette entité (32.3 - 12.7 % et 74.1 - 5.5%, respectivement). Les organoïdes de CCC ont montré une efficacité moyenne de transfection de 83,0 - 13,1 % pour le petit plasmide et pour le grand plasmide 39,5 - 10,4 % ont été obtenus.
Comme preuve de concept, les organoïdes normaux humains d’estomac ont été électroporated avec un px458_Conc2 codage de plasmide pour Cas9, GFP et deux sgRNAs ciblant TP53. Les ruptures de double brin induites par Cas9 sur l’exon 8 ont été réparées par l’assemblage final non-homologue (NHEJ), ce qui a entraîné des changements de cadre et, par conséquent, un ko du gène (voir la figure supplémentaire 1).
Tableau 1 : Composition des médias basaux, mélanges de digestion et de culture. S’il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).

Tableau 2 : Paramètres d’électroporation selon Fujii et coll.10.
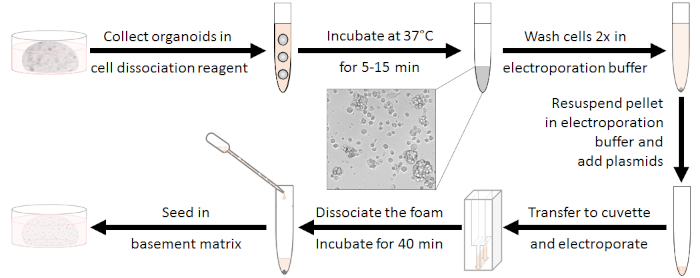
Figure 1 : Flux de travail de préparation à l’électroporation. Tout d’abord, les organoïdes doivent être dissociés à des grappes de 10-15 cellules et les antibiotiques devraient être lavés. Après l’électroporation, la mousse blanche doit être dissociée. Les cellules peuvent être ensemencées après avoir régénération pendant 40 min à température ambiante. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
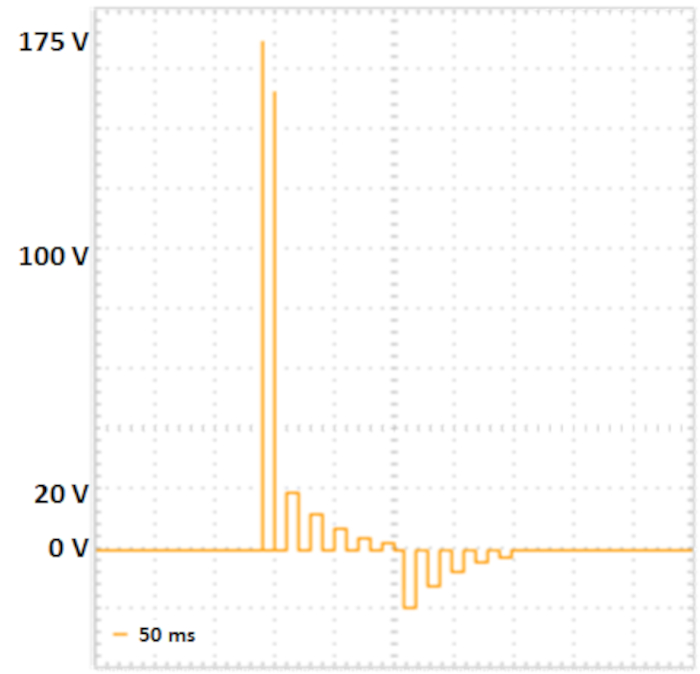
Figure 2 : Électroporation en deux étapes. Deux impulsions de poring avec une plus grande tension une courte durée (175 V et 157.5 V, chacun e pendant 5 ms, pause pendant 50 ms, carie de tension 10%) conduire à la formation de pores dans les membranes cellulaires. Les impulsions de transfert suivantes transmettent l’ADN dans les cellules : cinq impulsions de transfert positives (avec 20 V, 12 V, 7,2 V, 4,32 V et 2 592 V, chacun pour 50 ms, pause de 50 ms, carie de tension 40%), suivie de cinq impulsions de transfert échangées polarity (avec 20 V, 12 V, 7,2 V, 4,32 V et 2 592 V, chacun e pendant 50 ms, pause de 50 ms, tension de 40%). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
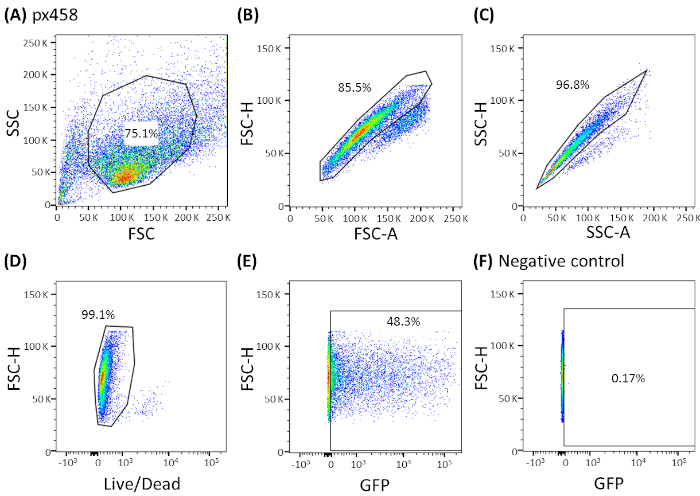
Figure 3 : Stratégie de gating représentative montrée par les organoïdes du CCC. Tous les organoïdes électroporated ont été analysés par cytométrie de flux 48 h après électroporation. Les cellules électroporated sans ADN plasmide ont été employées comme contrôles négatifs. Les portes ont été fixées comme suit: (A) gating pour la forme cellulaire, (B,C) gating pour les cellules simples (doublet discrimination), (D) gating pour les cellules vivantes (taché d’un anticorps pour les cellules apoptotiques) et (E,F) finalement gating pour les cellules exprimant eGFP (canal FITC). FSC - diffusion vers l’avant; SSC et la dispersion latérale. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
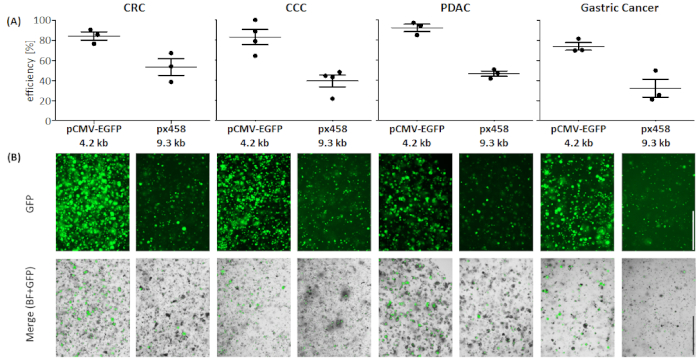
Figure 4 : Efficacité d’électroporation de quatre entités organoïdes. (A) Analyse FACS (n - 34, écart standard moyen et chaque valeur unique sont montrées) et (B) comparaison visuelle par microscope à fluorescence. Barre d’échelle de 1 000 m BF et champ lumineux; CCC - cholangioccarcinome; CRC - Cancer colorectal; GC - cancer gastrique; PDAC - adénocarcinome canalaire pancréatique. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
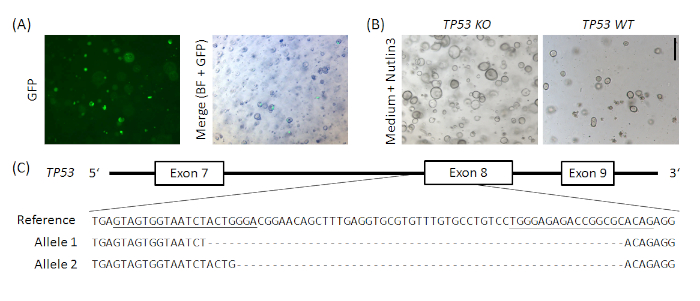
Figure supplémentaire 1 : KO exemplaire de CRISPR/Cas9-basé de TP53 dans les organoïdes humains normaux d’estomac. Le px458_Conc2 vecteur (voir Tableau des Matériaux)a été cloné en combinant le vecteur concatemer 2 gRNA, un don généreux de Bon-Kyoung Koo19, avec px45820, résultant en un codage plasmide pour 2 sgRNAs, Cas9 et GFP. Deux sgRNAs ciblant TP53 ont été introduits dans px458_Conc2 vecteur par clonage de porte d’or (analoguement à Andersson-Rolf et al.,19). 10 g d’ADN plasmide ont été électroporated dans les organoïdes gastriques normaux humains (A). Clones ont été sélectionnés par l’administration Nutlin3 (B) et le KNOCK TP53 a été confirmé par toPO TA clonage et le séquençage des allèles, ici exemplaire montré pour un clone (C). Les sgRNAs sont soulignés dans la référence. Barre d’échelle de 200 m. BF - champ lumineux. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
Ce protocole donne des instructions détaillées pour une électroporation efficace, rapide et facile à effectuer de différentes entités organoïdes. En plus des organoïdes tumoraux présentés par PDAC, CRC, CCC et GC, il fonctionne avec succès pour les organoïdes dérivés de tissus sains ainsi. Le protocole peut être exécuté dans un délai d’un jour. Dans les protocoles de transfection organoïde publiés, l’ensemble de la procédure a duré quatre jours, y compris deux jours de préparations avec différents types de médias de culture10,21. Dans notre protocole, aucun prétraitement spécial n’est requis. En se lavant avec le tampon d’électroporation avant l’électroporation les composants antibiotiques des médias ont été lavés et un ajustement des concentrations saline pour des valeurs optimales d’impédance a été réalisé. Néanmoins, certains aspects critiques devraient être considérés pour une électroporation réussie :
Cellules
Dans le protocole d’électroporation de Fujii et coll.10, il est recommandé de dissocier les organoïdes à des cellules individuelles et de les filtrer à travers une passoire cellulaire de 20 m. Dans nos mains, la digestion des cellules individuelles diminue fortement la survie des cellules. Comme suggéré dans Merenda et autres21,nous avons également dissocié des organoïdes aux groupes de 10-15 cellules et ne pouvions pas déterminer une efficacité diminuée comparée à la dissociation de cellules simples. Après l’électroporation, il est très important de dissocier la mousse blanche, de sorte qu’aucune cellule attachée ne se perd.
Pour la culture cellulaire 2D, il a été démontré qu’un temps de régénération après l’électroporation de plus de 10 min jusqu’à 40 min augmente la capacité de survie et l’efficacité de transfection en particulier des grands plasmides22. Dans les expériences d’essai, la même chose pourrait être documentée pour les organoïdes, menant à une étape d’incubation de 40 min après électroporation dans ce protocole. Afin d’augmenter la récupération de l’électroporation, nous les avons cultivés avec Rho-associé protéine kinase (ROCK) inhibiteur Y-27632 pendant cinq à sept jours23. De même, la supplémentation supplémentaire de glycogène synthase kinase 3 (GSK3) inhibiteur CHIR99021 est destiné à aider les cellules simples à récupérer10.
Paramètres
L’un des avantages de l’électroporateur utilisé est que l’impédance peut être mesurée avant l’électroporation pour des conditions optimales. Selon le fabricant, les valeurs d’impédance devraient être de 30-55 euros. Dans nos mains, les valeurs d’impédance de 30-40 ont montré des efficacités optimales. Dans une expérience préliminaire, différentes tensions et valeurs de longueur d’impulsion de l’impulsion de poring ont été variées pour trouver la proportion optimale de l’efficacité à la survie. En résumé, nous avons pu confirmer les valeurs décrites de Fujii et coll.10 dans les différentes entités décrites ici.
Adn
L’effet de différentes quantités d’ADN a été testé dans des expériences préliminaires jusqu’à 45 g d’ADN par échantillon. Aucun effet cytotoxique n’a pu être détecté. L’efficacité de la transfection a été augmentée d’une manière dépendante de la dose avec la saturation 'gt; 30 'g. Nous avons donc utilisé 30 g par échantillon dans le protocole final, mais bien sûr il peut être augmenté (par exemple, pour l’électroporation de plus de plasmides en parallèle). En outre, la pureté et la concentration de l’ADN semble être très important. Une concentration supérieure à 5 g/L a montré des efficacités de transfection optimales.
Comme prévu, le plasmide de 9,3 kB pourrait être transfecté avec une efficacité inférieure à celle du plasmide plus petit de 4,2 kB (voir la figure 4). L’utilisation de plasmides encore plus gros que 10 kB devrait diminuer encore l’efficacité. Pour les applications futures, il pourrait être intéressant de tester l’ADN minicercle comme vecteur, puisque ces porteurs de gènes n’ont pas l’épine dorsale bactérienne d’un plasmide qui les rend plus petits24. Cela devrait se traduire par une efficacité de transfection améliorée. En outre, pour les manipulations à base de CRISPR d’organoïdes une électroporation directe de sgRNAs liés à Cas9 comme un complexe de ribonucléoprotéine (RNP) pourrait être une alternative ou l’ajout25.
Déclarations de divulgation
Les auteurs n’ont rien à révéler.
Remerciements
Nous remercions Juliane Fohgrub, Ann-Christin Meinecke et Max Heiduk pour leur excellente assistance technique. Le financement a été fourni par Deutsche Krebshilfe (No 111350 et 70112925), Sander Stiftung (No 2014.104.1), Hector Stiftung (No M65.2) et l’Union européenne (ERC no 639050).
matériels
| Name | Company | Catalog Number | Comments |
| For establishment and culture medium | |||
| [Leu15] Gastrin | Sigma-Aldrich | G9145 | |
| A83-01 | Tocris Bioscience | 2939 | |
| Advanced DMEM/F-12 | Invitrogen | 12634010 | |
| B27 | Invitrogen | 17504044 | |
| B27 Supplement, minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| CHIR99021 | Stemgent | 04-0004 | |
| Collagenase II | Life Technologies | 17101-015 | |
| Collagenase XI | Sigma-Aldrich | C9407-100MG | |
| Collagenase D | Roche | 11088866001 | |
| Dispase II | Roche | 4942078001 | |
| Dnase I | Sigma-Aldrich | D5319 | |
| D-sorbitol | Roth | 6213.1 | |
| Dithiothreitol | Thermo Scientific | 1859330 | |
| EDTA | Roth | 8040 | |
| Forskolin | Tocris Bioscience | 1099 | |
| Glutamax | Life Technologies | 35050061 | |
| Hepes | Thermo Fisher Scientific | 15630106 | |
| hFGF-10 | Preprotech | 100-26 | |
| KCl | Sigma-Aldrich | P9541 | |
| KH2PO4 | Roth | 3904.2 | |
| Matrigel | Corning | 356231 | basement matrix |
| mEGF | Invitrogen | PMG8043 | |
| N2 | Invitrogen | 17502048 | |
| NaCl | Roth | 3957.1 | |
| Na2HPO4 | Roth | K300.2 | |
| N-Acetyl-L-Cystein | Sigma-Aldrich | A9165 | |
| Nicotinamid | Sigma-Aldrich | N0636 | |
| Noggin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (Hek293-mNoggin-Fc) |
| PBS | Gibco | 14190169 | |
| Penicillin Streptomycin | Life Technologies | 15140122 | |
| Primocin | InvivoGen | ant-pm-1 | |
| Prostaglandin E2 | Tocris Bioscience | 2296 | |
| Recombinant Human HGF | Preprotech | 100-39H | |
| Rspondin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (HA-Rspo1-Fc-293T) |
| SB202190 | Sigma-Aldrich | S7067 | |
| Sucrose | VWR | 27,480,294 | |
| TrypLE Express | Gibco | 12604021 | Dissociation reagent |
| Wnt3A | n.a. | n.a. | Conditioned medium produced from L-Wnt3a cells (from Sylvia Boj) |
| Y-27632 | Sigma-Aldrich | Y0503 | |
| Consumables | |||
| Cell Strainer 100 µm | Falcon | 352360 | |
| 48-well plate | Corning | 3548 | |
| Nepa Electroporation Cuvettes 2mm gap w/pipettes | Nepa Gene Co., Ltd. | EC-002S | |
| Tubes 15 ml | Greiner | 188271 | |
| Tubes 15 ml low binding | Eppendorf | 30122208 | Tubes for FACS preparing |
| Equipment | |||
| Electroporator Nepa21 | Nepa Gene Co., Ltd. | n.a. | |
| EVOS FL Auto | Invitrogen | AMAFD1000 | Fluorescence microscope |
| LSRFortessa | BD Bioscience | 647800E6 | FACS |
| Reagents and plasmids | |||
| Live/dead fixable blue dead cell stain kit | Invitrogen | L34962 | Includes antibody for live/dead staining for FACS analysis |
| EndoFree Plasmid Maxi Kit | Qiagen | 12362 | |
| Nutlin-3 | Selleckchem | S1061/07 | |
| Opti-MEM | Gibco | 31985047 | Electroporation buffer |
| 2 gRNA concatemer vector | AddGene | 84879 | |
| pCMV-EGFP | Nepa Gene Co., Ltd. | n.a. | |
| px458 plasmid | AddGene | 48138 | coding for sgRNA and Cas9 |
| px458_Conc2 plasmid | AddGene | 134449 | px458 plasmid containing 2x U6 promotors for two different sgRNAs |
| sgRNA_hTP53_1a | Eurofins Genomics | n.a. | 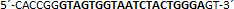 |
| sgRNA_hTP53_1b | Eurofins Genomics | n.a. | 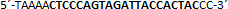 |
| sgRNA_hTP53_2a | Eurofins Genomics | n.a. | 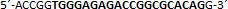 |
| sgRNA_hTP53_2b | Eurofins Genomics | n.a. | 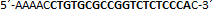 |
Références
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
- Werner, K., Weitz, J., Stange, D. E. Organoids as Model Systems for Gastrointestinal Diseases: Tissue Engineering Meets Genetic Engineering. Current Pathobiology Reports. 4 (1), 1-9 (2016).
- Olayanju, A., Jones, L., Greco, K., Goldring, C. E., Ansari, T. Application of porcine gastrointestinal organoid units as a potential in vitro tool for drug discovery and development. Journal of applied toxicology. 39 (1), 4-15 (2019).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Matano, M., et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nature Medicine. 21 (3), 256-262 (2015).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Drost, J., et al. Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer. Science. 358 (6360), 234-238 (2017).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy Nucleic Acids. 4, e264 (2015).
- Tsong, T. Y. Electroporation of cell membranes. Biophysical journal. 60 (2), 297-306 (1991).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Bartfeld, S., et al. In vitro expansion of human gastric epithelial stem cells and their responses to bacterial infection. Gastroenterology. 148 (1), 126-136 (2015).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Broutier, L., et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nature Medicine. 23 (12), 1424-1435 (2017).
- Hennig, A., et al. CFTR Expression Analysis for Subtyping of Human Pancreatic Cancer Organoids. Stem Cells Int. 2019, 1024614 (2019).
- Seidlitz, T., et al. Human gastric cancer modelling using organoids. Gut. 68 (2), 207-217 (2019).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Current protein & peptide science. 9 (4), 338-369 (2008).
- Andersson-Rolf, A., et al. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 271-277 (2016).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8, 2281 (2013).
- Merenda, A., et al. A Protocol for Multiple Gene Knockout in Mouse Small Intestinal Organoids Using a CRISPR-concatemer. Journal of Visualized Experiments. (125), (2017).
- Lesueur, L. L., Mir, L. M., Andre, F. M. Overcoming the Specific Toxicity of Large Plasmids Electrotransfer in Primary Cells In Vitro. Molecular Therapy Nucleic Acids. 5, e291 (2016).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Darquet, A. M., Cameron, B., Wils, P., Scherman, D., Crouzet, J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Therapy. 4 (12), 1341-1349 (1997).
- Jacobi, A. M., et al. Simplified CRISPR tools for efficient genome editing and streamlined protocols for their delivery into mammalian cells and mouse zygotes. Methods. 121-122, 16-28 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon