Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une plate-forme de spectrométrie de masse d’échange hydrogène-deutérium (HDX-MS) pour étudier les enzymes biosynthétiques peptidiques
Dans cet article
Résumé
Les synthetases de lanthipeptide catalysent les réactions en plusieurs étapes pendant la biosynthèse des produits naturels peptidiques. Ici, nous décrivons un flux de travail continu, ascendant, de spectrométrie de masse d’échange hydrogène-deutérium (HDX-MS) qui peut être employé pour étudier la dynamique conformationnelle des synthetases de lanthipeptide, aussi bien que d’autres enzymes semblables impliquées dans la biosynthèse de produit naturel de peptide.
Résumé
La spectrométrie de masse d’échange hydrogène-deutérium (HDX-MS) est une méthode puissante pour la caractérisation biophysique des changements conformationnels enzymatiques et des interactions enzyme-substrat. Parmi ses nombreux avantages, HDX-MS ne consomme que de petites quantités de matière, peut être effectuée dans des conditions quasi natives sans avoir besoin d’étiquetage enzymatique/substrat, et peut fournir des informations spatialement résolues sur la dynamique conformationnelle des enzymes, même pour les grandes enzymes et les complexes multiprotéines. La méthode est initiée par la dilution de l’enzyme d’intérêt dans tampon préparé en D2O. Cela déclenche l’échange de protium dans les amides de liaison peptidique (N-H) avec le deutérium (N-D). Aux points de temps d’échange désirés, les aliquots de réaction sont trempés, l’enzyme est protéolysée en peptides, les peptides sont séparés par chromatographie liquide ultra-performante (UPLC), et le changement de masse de chaque peptide (dû à l’échange d’hydrogène contre du deutérium) est enregistré par la SEp. La quantité d’absorption de deutérium par chaque peptide dépend fortement de l’environnement local de liaison hydrogène de ce peptide. Les peptides présents dans les régions très dynamiques de l’échange d’enzymes deutérium très rapidement, tandis que les peptides dérivés de régions bien ordonnées subissent des échanges beaucoup plus lentement. De cette façon, le taux HDX rend compte de la dynamique conformationnelle des enzymes locales. Les perturbations des niveaux d’absorption du deutérium en présence de différents ligands peuvent ensuite être utilisées pour cartographier les sites de liaison ligand, identifier les réseaux allotériques et comprendre le rôle de la dynamique conformationnelle dans la fonction enzymatique. Ici, nous montrons comment nous avons utilisé HDX-MS pour mieux comprendre la biosynthèse d’un type de peptide produits naturels appelés lanthipeptides. Les lanthipeptides sont des peptides génétiquement codés qui sont modifiés post-translationnellement par de grandes enzymes multifonctionnelles et conformationnelles dynamiques qui sont difficiles à étudier avec les approches traditionnelles de biologie structurale. HDX-MS fournit une plate-forme idéale et adaptable pour étudier les propriétés mécanistes de ces types d’enzymes.
Introduction
Les protéines sont des molécules structurellement dynamiques qui échantillonnent différentes conformations sur des échelles de temps allant des vibrations des liaisons femtosecondes aux réarrangements de domaines protéiques entiers qui peuvent se produire sur plusieurs secondes1. Ces fluctuations conformationnelles sont souvent des aspects critiques de la fonction enzymatique/protéique. Par exemple, les changements conformationnels induits par la liaison ligand sont souvent d’une importance cruciale pour moduler la fonction enzymatique, soit en organisant les résidus actifs du site nécessaires à la catalyse, en définissant les sites de liaison du substrat dans les mécanismes cinétiques séquentiels, en protégeant les intermédiaires réactifs de l’environnement, soit en modulant la fonction enzymatique par l’intermédiaire de réseaux allotériques. Des études récentes ont également montré que la dynamique conformationnelle peut être conservée tout au long de l’évolution et que les perturbations des mouvements moléculaires conservés peuvent être corrélées avec des changements dans la spécificité du substrat et l’émergence de nouvelles fonctions enzymatiques2,3.
Ces dernières années, la spectrométrie de masse d’échange hydrogène-deutérium (HDX-MS) est rapidement apparue comme une technique puissante pour sonder la façon dont les paysages conformationnels protéiques réagissent aux perturbations telles que la liaison ligand ou la mutagenèse4,5,6,7. Dans une expérience typique de HDX-MS (figure 1), une protéine d’intérêt est placée dans le tampon préparé dans D2O, ce qui déclenche le remplacement des protons échangeables de solvants par deutérium. Le taux de change de l’amide moiety des liaisons peptidiques dépend fortement du pH, de la séquence locale d’acides aminés, et de l’environnement structurel local de l’amide8. Les amidons engagés dans des interactions de liaison hydrogène (comme celles présentes dans les α-hélices et les feuilles de β) s’échangent plus lentement que les amides dans les régions non structurées de la protéine qui sont exposées au solvant en vrac. Ainsi, l’étendue de l’absorption de deutérium est un reflet de la structure de l’enzyme. On s’attendrait à ce que les enzymes qui sont conformationally dynamiques, ou qui subissent des transitions structurelles sur la liaison ligand, produisent une réponse mesurable de HDX.
La base mécaniste du taux de change lent d’un amide structuré est indiquée dans la figure 25,8,9. Pour subir le HDX, la région structurée doit d’abord échantillonner transitoirement une conformation dépliée, de sorte que les molécules solvantes qui catalysent l’échange HDX via un mécanisme chimique acide/de base spécifique, aient accès à l’amide échangeable. En fin de compte, les magnitudes relatives du taux de change chimique (kchem) et les taux de pliage et de refolding (kouvert et kfermer) déterminer le taux HDX mesuré dans l’expérience5,8. De ce modèle cinétique simple, il est clair que l’étendue de l’absorption de deutérium reflétera la dynamique conformationnelle sous-jacente (telle que définie par kouvert et kfermer). La plupart des expériences HDX-MS sont réalisées dans un flux de travail ascendant où, suite à la réaction d’échange, la protéine d’intérêt est digérée en peptides et l’absorption de deutérium par chaque peptide est mesurée comme une augmentation de la masse7. De cette façon, HDX-MS permet de cartographier les perturbations de la dynamique conformationnelle des enzymes à l’échelle spatiale locale des peptides, ce qui permet au chercheur d’évaluer comment la perturbation modifie la dynamique dans différentes régions de l’enzyme d’intérêt.
Les avantages de l’approche HDX-MS pour élucider la dynamique structurelle des protéines sont nombreux. Tout d’abord, la méthode peut être effectuée avec de petites quantités de protéines indigènes ou sur des complexes protéiques dans des systèmes à structure quaternaire10. Il n’est même pas nécessaire que la préparation enzymatique utilisée dans l’analyse soit fortement purifiée11,12, tant que le flux de travail HDX-MS ascendant fournit un nombre suffisant de peptides identifiés avec confiance qui couvrent la séquence protéique d’intérêt. En outre, HDX-MS peut fournir des informations sur la dynamique conformationnelle dans des conditions quasi natives sans avoir besoin d’étiquetage protéique spécifique au site comme cela serait utilisé dans les études de fluorescence à moléculeunique 13, et il n’y a pas de limite de taille au complexe protéique ou protéique qui peut être étudié (ce qui rend difficiles des approches telles que la spectroscopie par résonance magnétique nucléaire [NMR])7,14. Enfin, des méthodes HDX-MS résolues dans le temps peuvent être utilisées pour étudier les protéines intrinsèquement désordonnées, qui sont difficiles à étudier avec la cristallographie aux rayons X15,16,17,18. La principale limitation de HDX-MS est que les données sont de faible résolution structurelle. Les données HDX-MS sont utiles pour indiquer où la dynamique conformationnelle évolue et pour révéler des changements conformationnels couplés, mais elles ne fournissent pas souvent beaucoup d’informations sur le mécanisme moléculaire précis qui conduit au changement observé. Les progrès récents dans la combinaison des méthodes de dissociation de capture d’électrons avec les données de protéine HDX-MS se sont révélés prometteurs pour cartographier les sites d’échange aux résidus d’acides aminéssimples 19, mais le suivi des études biochimiques et structurelles sont encore souvent nécessaires pour fournir la clarté aux modèles structurels transmis par les données HDX-MS.
Ci-dessous, un protocole détaillé pour le développement d’un essai HDX-MS est présenté20. Les protocoles de préparation de l’échantillon présentés ci-dessous devraient être généralement applicables à toute protéine qui présente une bonne solubilité dans les tampons aqueux. Des méthodes de préparation d’échantillons plus spécialisées et des workflows HDX-MS sont disponibles pour les protéines que nécessaire en présence de détergent ou de phospholipides21,22,23,24. Les paramètres instrumentaux de collecte de données HDX-MS sont décrits pour un spectromètre de masse quadrupole haute résolution couplé au système de chromatographie liquide. Des données d’une complexité et d’une résolution similaires pourraient être recueillies sur l’un ou l’autre d’un certain nombre de systèmes de spectrométrie de masse chromatographie liquide (LC-MS) disponibles dans le commerce. Des aspects clés du traitement des données à l’aide d’un logiciel disponible dans le commerce sont également fournis. Nous présentons également des lignes directrices pour la collecte et l’analyse des données qui sont conformes aux recommandations formulées par l’ensemble de la communauté HDX-MS12. Le protocole décrit est utilisé pour étudier les propriétés structurelles dynamiques de HalM2, une synthétase lanthipeptide qui catalyse la maturation en plusieurs étapes d’un produit naturel peptidique antimicrobien20. Nous montrons comment HDX-MS peut être utilisé pour révéler les sites de liaison du substrat et les propriétés allostériques qui ont échappé à la caractérisation précédente. Plusieurs autres protocoles sur la protéine HDX-MS ont été publiés ces dernières années25,26. Avec le présent travail, ces contributions antérieures devraient fournir au lecteur une certaine flexibilité dans la conception expérimentale.
Protocole
1. Préparation de réaccérateurs déréflés et de solutions de stock enzymatique
- Préparez les réachagents nécessaires aux réactions HDX (y compris les tampons, sels, substrats, ligands, etc.) sous forme de solutions de stock concentré 100−200x en D2O (99,9 % de fraction atome D). Préparez au moins 50 mL de solution de stock tampon.
NOTE: Pour la caractérisation de HalM2, les solutions suivantes ont été préparées: 500 mM MgCl2, 100 mM tris (2 carboxyethyl)phosphine (TCEP), 750 mM ATP (en tampon HEPES), 800 mM HEPES 7,1, 500 μM HalA2, et 500 mM AMPPNP. - Congeler et lyophiliser les solutions de stock à la sécheresse.
- Re-dissoudre dans D2O, et répéter le cycle de lyophilisation au moins une fois de plus pour remplacer autant de protons échangeables par des deutéons que possible.
- Ajustez le du stock tampon HEPES déréflé à la valeur désirée avec naod/DCl concentré, en gardant à l’esprit la relation suivante27:
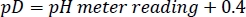
REMARQUE : Le taux d’amide HDX dépend fortement du pL de la solution (pL = pH ou pD)5. Différents lots de solutions de stock tampon doivent être préparés, stockés et utilisés de la même manière pour éviter une légère dérive pL entre les expériences. - Calculez la quantité de chaque reagent nécessaire pour un essai HDX de 300 μL et stockez comme aliquots à usage unique à -80 °C.
- Préparez une solution concentrée de stock enzymatique (~100−200 μM) dans un tampon de stockage d’enzymes protiated à l’aide d’un filtre centrifuge(Tableau des matériaux)ou d’un dispositif équivalent.
REMARQUE : Le tampon exact et le seuil de poids moléculaire du filtre centrifuge dépendront de la protéine/enzyme d’intérêt. HalM2 est stocké dans 50 mM HEPES, pH 7,5, 100 mM KCl, et 10% glycérol. 10 filtres kDa ont été utilisés pour préparer l’enzyme concentrée. - Aliquot l’enzyme en portions à usage unique et conserver à -80 °C.
REMARQUE : Il peut s’agir d’un point d’arrêt. Toutes les solutions de stock décrites dans la section 1 peuvent être préparées avant les réactions HDX. Si elles sont stockées à -80 °C, la plupart des enzymes/solutions de stock stérilisés seront stables pendant de nombreux mois.
2. Étalonnage du volume d’étanchéation HDX
- Préparer une réaction HDX de 300 μL dans D2O à l’aide des réaccérateurs stérilisés et des stocks concentrés d’enzymes préparés dans la section 1.
- Utilisez une concentration finale en enzymes de 1−5 μM.
- Utilisez une concentration tampon HEPES stérilisé finale d’au moins 50−100 mM.
- Assurez-vous que les concentrations d’autres composants sont suffisantes pour maintenir l’activité/fonction enzymatique désirée.
- Préparer 1 L de solution d’étanchéité HDX (phosphate de 100 mM, 0,8 M guanidine-HCl, pH 1,9). Congeler et conserver en portions de 50 mL (pour le stock à long terme) et de 1 mL (pour les aliquots à usage unique).
REMARQUE : La composition exacte du tampon d’étanchéation dépendra de l’enzyme utilisée dans l’étape de protéolyse du flux de travail HDX-MS ascendant (étape 3.3.3). Le tampon d’étanchéation donné ici est compatible avec la pepsine, la protéase la plus couramment utilisée pour HDX-MS. Si une protéase différente est utilisée, vérifiez auprès du fournisseur de protéase pour assurer la compatibilité tampon. - Calibrez le volume de tampon d’étanchéation nécessaire pour ajuster le pL final du mélange de réaction HDX trempé à une valeur de lecture de mètre de pH de 2,3.
REMARQUE : Le taux de change solvant H/D de la liaison peptidique amide N-H est un processus dépendant du pH qui est soumis à la catalyse acide et de base. Le taux de change minimum se produit à une valeur de pH 2,5 (lecture du mètre de pH = 2,3 pour un mélange 50:50 H2O:D2O). Ainsi, une valeur finale de pL près de 2,5 permettra de minimiser l’échange de dos d’hydrogène qui se produit au cours de l’analyse lC-MS ascendante, préservant ainsi l’étiquette de deutérium dans les peptides.- Mélanger 50 μL du mélange de réaction HDX de l’étape 2.1 avec 50 μL de tampon étanche et mesurer le pL du mélange trempé avec une électrode micro-pointe.
- Augmentez le volume de solution d’étanchéation au besoin pour ajuster la lecture finale du compteur de pH à une valeur de 2,3.
- Une fois que le volume d’étanchéisation approprié a été déterminé, répétez le processus d’assaisage plusieurs fois à l’aide d’aliquots frais de 50 μL de la réaction HDX (étape 2.1) pour s’assurer qu’un pL final cohérent est atteint à l’ajout d’une quantité fixe de tampon d’assaisons.
3. Préparation d’échantillons de référence et optimisation du flux de travail LC-MS ascendant
- Préparer des échantillons de référence non stérilisés pour la protéine d’intérêt dans le triplicate dans des tubes de 0,5 mL. Assurez-vous que les conditions du mélange de réaction finale sont identiques à celles utilisées dans les réactions HDX authentiques (étape 2.1), sauf que les réactions sont préparées en H2O à l’aide de solutions de stock de réaccente également préparées en H2O.
- Étanchez les échantillons comme à l’étape 2.3 en ajoutant le volume approprié de tampon d’étanchéation pour ajuster le pH final à 2,5. Flash congeler les échantillons dans de l’azote liquide et stocker à -80 °C jusqu’à ce qu’ils soient prêts pour l’analyse.
- Analyser les échantillons de référence enzymatiques protiated à l’aide d’un flux de travail LC-MS ascendant.
REMARQUE : Avant d’exécuter ces étapes, le système LC-MS qui sera utilisé pour l’acquisition de données doit être correctement calibré et prêt à l’emploi. Le moment et la température de toutes les étapes du flux de travail LC-MS ascendant doivent être rigoureusement contrôlés afin de minimiser les différences dans l’échange de dos entre les échantillons. Avec l’instrumentation MS utilisée dans ce protocole (Tableau des matériaux et Informations à l’appui), la plupart des étapes peuvent être contrôlées par le logiciel de l’instrument. Pour assurer la collecte de répliques précises, il est recommandé d’automatiser autant d’étapes dans le flux de travail que possible.- Retirer un échantillon individuel de référence enzymatique (préparé comme à l’étape 3.2) du congélateur et décongeler à 37 °C pendant 1 min dans un bain d’eau.
- À exactement 2 minutes après avoir retiré l’échantillon du congélateur et de la décongélation, injectez une partie de 40 μL de l’échantillon de référence trempé dans une colonne de chromatographie liquide ultra-performante (UPLC) (2,1 x 30 mm, 300 Å, 5 μM) contenant une phase stationnaire fonctionnalisée avec de la pepsine (une protéase acide stable).
- Digérer l’échantillon à un débit de 100 μL/min pendant 3 min à 15 °C en utilisant 0,1 % d’acide formique dans H2O (pH = 2,5) comme solvant.
- Recueillir les peptides peptiques comme ils s’élit de la colonne de pepsine sur une colonne de piège C18 tenue à 0,4 °C pour minimiser l’échange de dos.
- Passez les peptides peptiques desaltés de la colonne de piégeage à une colonne analytique C18 (1 mm x 100 mm, 1,7 μM, 130 Å) maintenue et actionnée à 0,4 °C pour la séparation des peptides peptiques.
REMARQUE : Les étapes 3.3.3−3.3.5 peuvent être automatisées par certains systèmes LC-MS utilisés pour l’acquisition de données HDX-MS. Alternativement, ces étapes peuvent être effectuées indépendamment, en gardant à l’esprit que le moment et la température de chaque étape doit être soigneusement contrôlée pour atteindre un échange constamment bas du dos. - Elute la colonne C18 avec un système de solvant d’acide formic d’acetonitrile/eau/0.1%. Optimisez le gradient LC pour la protéine d’intérêt afin de maximiser la séparation et de préserver l’étiquette de deutérium dans les peptides peptiques.
REMARQUE : Les détails de l’élitution du gradient sont fournis dans l’information à l’appui. - Soumettons le digeste peptique à la spectrométrie de masse de l’ionisation électrospray (ESI).
REMARQUE : Les conditions de source fournies dans l’information à l’appui fourniront une ionisation suffisante pour la plupart des peptides peptiques.- Une fois ionisé dans l’instrument ms, effectuer une séparation de la mobilité ion phase de gaz en utilisant l’azote comme gaz tampon pour améliorer la capacité maximale de la méthode.
- Après la séparation de la mobilité iriontique, soumettons les ions précurseurs peptiques à un fluxde travail MS E impliquant des cycles alternatifs de faible énergie de collision (4 V) et d’énergie de collision élevée (21−40 V).
REMARQUE : Les régimes alternés d’énergie de collision faible et élevée permettent la collecte de données sur la SP (faible énergie de collision) simultanément avec les données MSMS (énergie de collision élevée). Ceci, à son tour, permet la corrélation de temps des ions précurseurs avec leurs ions de fragment respectifs. Cette corrélation est essentielle pour l’identification confiante de peptide décrite dans la section 4. - Détecter le précurseur du peptide et les ions fragmentaires à l’aide d’un analyseur de masse d’une puissance de résolution d’au moins 20 000.
- Parallèlement à l’acquisition de données, acquérir des données MS pour une norme externe [Glu-1]-fibrinopeptide B (GluFib).
REMARQUE : Le flux de travail sur la SP décrit à l’étape 3.3.7 est appelé protocole MSE. Des paramètres instrumentaux complets pourun protocole MS E qui convient à HDX-MS ascendant sont fournis dans l’information à l’appui.
- Évaluer la qualité des données LC-MS.
REMARQUE : À l’aide du protocole décrit ci-dessus et des paramètres instrumentaux fournis dans l’information àl’appui, les échantillons de référence devraient produire un chromatogramme iion total d’une intensité maximale du signal d’environ 1 x 108. Il devrait y avoir beaucoup de peptides peptiques qui s’échappent entre 3−9 min (Figure 3A−C). - Injecter 40 μL d’échantillons vierges (0,1% d’acide formique dans l’eau) pour nettoyer la pepsine et les colonnes analytiques C18.
REMARQUE : En général, 2−3 blancs devraient suffire. - Répétez les étapes 3.3.1−3.3.9 pour chacun des échantillons de protéines de référence triplicate.
4. Traitement des données de référence et définition d’une liste peptidique
- Analyser les données msE brutes (étape 3.3.7) à l’aide d’un logiciel protéomique (Table of Materials). À l’aide du logiciel protéomique, accédez aux bibliothèques | Databanks séquence protéique pour définir la base de données protéique en important la séquence d’acides aminés de la protéine d’intérêt.
REMARQUE : L’objectif de cette étape est de rechercher les données de référence sur la SP pour les peptides peptiques dérivés de la protéine d’intérêt et d’utiliser les données msms (acquises simultanément avec les données sur la SP) pour valider toute identification putative au peptide. - Donnez un nom à la séquence protéique d’intérêt. Importer la séquence protéique (en format FASTA). Le logiciel effectuera une digestion in silico de la protéine de base de données pour générer une liste de peptides qui seront utilisés pour rechercher les données LC-MS.
- Définissez les paramètres de traitement (situés sous le menu bibliothèque). Sélectionnez Electrospray MSE comme type d’acquisition de données. Dans le champ Lock Mass for Charge 2, entrez 785.8426 pour le m/z pour le 2+ ion de [Glu-1]-fibrinopeptide B (GluFib) et cliquez sur finition.
- Définissez les paramètres workflow (situés sous le menu Bibliothèque).
- Sélectionnez Electrospray MSE pour le type de recherche. Sous le flux de travail | Titre de requête de recherche de base de données, sélectionnez la protéine de base de données créée à l’étape 4.2 dans le champ Databank.
- Changez le reagent de digest primaire à non spécifique, et effacez le champ de reagent de modificateur fixe en tenant le bouton de Ctrl tout en cliquant sur Carbamidomethyl C.
- Spécifiez l’annuaire de sortie en naviguant vers les options | configuration d’automatisation | Identité E. Cochez les cases pour apex 3D et Peptide sortie 3D et sortie comptable ion et spécifiez l’annuaire désiré.
- Traiter les données de l’échantillon de référence.
- Sur la barre d’outils gauche de l’espace de travail de la plate-forme protéomique, créez une nouvelle plaque en cliquant à droite sur Microtiter Plate. Mettre en évidence trois puits dans la plaque de microtiter (un pour chaque échantillon de référence prélevé à la section 3). Cliquez à gauche dans un puits, maintenez et faites glisser vers trois puits.
- Cliquez à droite et sélectionnez ajouter des données brutes. Dans la fenêtre qui apparaît, accédez à l’annuaire contenant les trois fichiers de référence de la section 3 et sélectionnez-les en même temps.
- Cliquez ensuite et choisissez les paramètres de traitement définis à l’étape 4.3. Cliquez ensuite et sélectionnez les paramètres de workflow définis à l’étape 4.4. Puis cliquez sur la finition.
- Une fois que les données brutes, les paramètres de traitement et les paramètres de flux de travail ont été attribués à chaque puits de la plaque, les puits apparaîtront bleus. Sélectionnez les puits, cliquez à droite et sélectionnez les dernières données brutes du processus. Cliquez sur le coin inférieur droit de la fenêtre pour suivre le traitement des données. Une fois que le message Aucun travail à exécuter n’apparaît, le traitement est entièrement terminé.
- Une fois le traitement des données terminé, les puits de la plaque passeront au vert. Cliquez à droite sur les puits et sélectionnez afficher les résultats du flux de travail. Une fenêtre séparée s’ouvrira pour chaque fichier de données de référence.
- Inspecter les données pour s’assurer que la majorité des signaux de SP dans les données de l’échantillon de référence ont été cartographiés avec succès aux peptides prévus à partir de la digestion in-silico de la protéine d’intérêt. Les peptides appariés seront de couleur bleue dans le spectre de sortie( Figure 4). Double-cliquez sur le filtre OK et vérifiez que la couverture en pourcentage est supérieure à 99%.
REMARQUE : Lors du traitement, la sortie des données sera automatiquement enregistrée avec l’extension defichier (raw_data_file_name_IA_final_peptide) dans l’annuaire spécifié à l’étape 4.5. - Importer la sortie logicielle protéomique dans le logiciel de traitement HDX (Table of Materials) pour un seuil supplémentaire.
- Cliquez sur Données dans le coin gauche de la fenêtre logicielle de traitement HDX. Cliquez sur les résultats PLGS d’importation et cliquez sur l’icône ajouter. Choisissez les fichiers de données traités à partir de l’étape 4.9 en naviguant vers l’annuaire approprié.
- Cliquez sur Suivant et spécifiez les paramètres suivants : minimum d’ions consécutifs ≥ 2, erreur de masse = 5 ppm, et seuil de fichier = 3. Cliquez sur la finition.
- Une fois satisfait des paramètres de seuil, enregistrez le projet HDX. Toutes les données HDX seront importées dans ce projet pour analyse et affichage.
REMARQUE : Les échantillons échangés de deutérium décrits dans la section suivante devront être traités avec un flux de travail identique LC-MS. Par conséquent, avant de procéder aux analyses HDX (section 5), assurez-vous que la préparation de l’échantillon (section 2), le flux de travail bottom-up LC-MS (section 3) et les workflows de traitement des données (section 4) fournissent la reproductibilité souhaitée et la couverture séquence de la protéine cible. Si l’un ou l’autre de ces processus doit être modifié pour améliorer la couverture, il est conseillé de revenir à l’étape 2.1, de préparer de nouveaux échantillons de référence en triplicate et de répéter les sections 2−4 (tout en faisant les ajustements nécessaires au protocole) pour s’assurer que chaque peptide peut être généré et détecté reproductiblement.
5. Effectuer des réactions HDX
- Préparez un espace de travail pour les réactions HDX.
- Pré-aliquot étanchez le tampon dans des tubes correctement étiquetés de 0,5 mL. Préparez un tube différent pour chaque point de temps, chaque réplique, et chaque état biochimique à analyser. Utilisez le volume approprié de tampon d’étanchéation à partir de l’étape 2.2 nécessaire pour ajuster la lecture finale du compteur de pH d’une partie de 50 μL de la réaction HDX à une valeur de 2,3.
- Centrifugez brièvement les cuves de 0,5 mL pour transférer tout le tampon d’étanchéation au fond du tube. Placez les tubes sur la glace.
- Remplissez un petit Dewar d’azote liquide et gardez-le à côté de l’espace de travail.
- Préparez les réactions HDX. Assurez-vous qu’il y a suffisamment de volume de réaction pour recueillir le nombre désiré de points de temps d’échange (un aliquot de 50 μL pour chaque point de temps désiré). Recueillir au moins 4−5 points de temps sur 3−4 ordres de grandeur dans l’échelle de temps (par exemple, des temps d’étanchéité de 15 s, 60 s, 300 s [5 min], 1 800 s [30 min], et 14 400 s [4 h] fournissent une couverture adéquate de la dynamique d’échange pour la plupart des enzymes).
- Pré-mélanger tous les composants déréflés (moins l’enzyme) de l’étape 1.1 en D2O.
- Pour chaque état biochimique à examiner (enzyme libre, enzyme + ligand, enzyme + inhibiteur, etc.), préparez les réactions HDX au moins en triplicate.
- Incuber les mélanges de réaction dans un bain d’eau à température contrôlée à 25 °C pendant 10 minutes avant l’ajout de l’enzyme.
REMARQUE : L’enzyme doit être préparée comme solution de stock concentré (~100−200 μM, étape 1.6) afin de minimiser l’ajout de protium dans l’analyse HDX. - Lorsque vous ajoutez l’enzyme à une concentration finale de 1−5 μM, démarrez la émietté. Mélanger soigneusement et rapidement la solution à l’aide d’une pipette de 200 μL pour s’assurer que l’enzyme est répartie uniformément dans l’échantillon.
- Aux points de temps d’échange désirés, retirez 50 aliquots de μL de la réaction HDX et mélangez rapidement et uniformément avec le tampon d’assaisons pré-aliquoted et glacé dans un tube de 0,5 mL.
REMARQUE : Les volumes de mélange et la procédure de mélange doivent être aussi précis et reproductibles que possible pour s’assurer que le pL d’étanchéité final désiré de 2,3 est atteint rapidement dans tous les échantillons. Garder le froid de glace tampon étanche aidera à minimiser l’échange de dos lors de la dénaturation de l’enzyme. - Immédiatement après avoir étanché l’échantillon HDX, plafonnez le tube et congèlez le flash dans de l’azote liquide.
- Continuer à recueillir des points de temps jusqu’à ce que tous les analyses soient terminées, puis transférer les échantillons au congélateur de -80 °C pour l’entreposage.
REMARQUE : Il peut s’agir d’un point d’arrêt. Après avoir recueilli tous les points de temps HDX, les échantillons peuvent être stockés à -80 °C jusqu’à ce qu’ils soient prêts pour l’analyse LC-MS. Idéalement, les réactions triplicate HDX devraient être effectuées pour tous les états biochimiques d’intérêt le même jour. Au minimum, toutes les réactions HDX répliquées pour un état biochimique donné doivent être exécutés en parallèle le même jour.
- Après avoir recueilli tous les points de temps HDX trempés, soumettez les échantillons au flux de travail LC-MS ascendant optimisé développé tel que décrit à l’étape 3.3. Injectez des échantillons HDX dans un ordre randomisé avec un nombre approprié de blancs entre les échantillons pour s’assurer que tout report de peptide est minime.
REMARQUE : Les données HDX n’ont pas besoin d’être collectées en mode MSE. Ainsi, le segment de l’énergie de collision élevée (étape 3.3.7.2) devrait être retiré du cycle de service de la SP. Il devrait s’agir de la seule modification apportée au flux de travail décrit à l’étape 3.3. - Évaluer la qualité des données HDX au fur et à mesure qu’elles sont collectées.
- S’assurer que les pics chromatographiques présents dans le chromatogramme iion total des échantillons de référence non stérilisés apparaissent en même temps de rétention dans les échantillons stérilisés (comme dans la figure 3D−F).
- S’assurer que les spectres de masse résumés sur des intervalles de temps spécifiques à partir d’échantillons de référence et d’échantillons stérilisés présentent des signes de stérilisation (c.-à-d. un déplacement de l’enveloppe isotopique des peptides individuels vers des valeurs m/z plus élevées dans les échantillons stérilisés [figure 6]).
6. Traitement des données HDX
- Importer les données HDX dans le projet HDX créé à l’étape 4.11 en cliquant sur Données | Fichiers MS dans la barre d’outils supérieure.
- Cliquez sur Nouvel État et nouvelle exposition au besoin pour définir les états biochimiques (p. ex., enzyme libre, enzyme + ligand, etc.) et les temps d’exposition au deutérium, respectivement, qui sont pertinents à l’analyse.
- Cliquez sur New Raw pour sélectionner les fichiers de données HDX à analyser. Attribuez les temps d’échange appropriés et l’état biochimique à chaque fichier de données brutes importé.
REMARQUE : Les données peuvent être importées et traitées par lots, ou toutes en même temps. L’ajout de données au projet n’annulera aucune analyse qui a déjà été effectuée dans le cadre de ce projet.
- Une fois que les fichiers de données ont été ajoutés, cliquez sur la finition pour commencer le traitement des données. Après un court délai, le logiciel demandera si l’utilisateur veut enregistrer les données avant de continuer. Cliquez sur oui.
REMARQUE : Le traitement initial peut prendre jusqu’à plusieurs heures selon le nombre d’échantillons analysés, le nombre de peptides dans la liste finale du peptide (étape 4.10), la taille de la fenêtre chromatographique et la fréquence de l’acquisition spectrale. - Si vous le souhaitez, modifiez les paramètres de traitement dans le menu Configuration pour modifier les paramètres de recherche iion. Assurez-vous d’utiliser les mêmes paramètres de recherche iion pour toutes les données d’un projet donné.
7. Analyse et visualisation des données HDX
REMARQUE : Une fois le traitement initial des données brutes terminé (étape 6.2), le logiciel de traitement HDX aura localisé les peptides de la liste des peptides (générés à l’étape 4.10) dans chacun des fichiers de données brutes analysés. Une fois que la distribution d’isotopes pour un peptide dans la liste est située dans un fichier de données brutes, le logiciel de traitement HDX représente chaque isotope avec un « bâton » (comme dans la figure 6C−E). Les intensités relatives des bâtonnets pour un peptide donné sont ensuite utilisées pour calculer l’absorption du deutérium par rapport aux spectres de référence. Tandis que le logiciel hdx-traitement fait un travail admirable d’attribuer correctement des « bâtons » à la plupart des peptides, la curation manuelle significative des valeurs d’absorption de deutérium sera toujours exigée.
- Analyse des valeurs d’absorption du peptide deutérium
- Sélectionnez le premier peptide dans la liste peptidique et ouvrez l’intrigue spectrale empilée dans le menu Vues. Faites défiler de haut en bas dans la fenêtre empilée de parcelle de spectre pour voir les spectres de masse pour le peptide sélectionné en fonction du temps d’échange de deutérium (Figure 7D,E).
- Assigner et affecter des bâtons au besoin à l’aide de clics de souris pour s’assurer que la distribution isotopique appropriée a été localisée dans les données et que chaque pic isotopique a été attribué (les bâtons assignés apparaîtront bleus). En cliquant sur l’un des spectres de l’intrigue spectrale empilée (Figure 7D,E ) permettra àl’utilisateurd’attribuer / bâtons non assignés dans la fenêtre de visionneuse de données active (Figure 7C).
- Vérifiez les affectations de bâton pour chaque état de charge en retraciant l’état de charge en haut de la fenêtre spectrale empilée de parcelle.
- Répétez les étapes 7.1.1−7.1.3 pour chaque état biochimique d’intérêt. L’état biochimique peut également être basculé en haut de la fenêtre spectrale empilée. Pour les mesures de différence d’absorption de deutérium les plus précises, assurez-vous que les bâtons sont attribués pour le même ensemble d’états de charge pour chaque état biochimique.
- Répétez les étapes 7.1.1−7.1.4 pour chaque peptide dans la liste peptidique.
- Vérifiez l’écart type des valeurs d’absorption du peptide deutérium à l’aide de la carte de couverture.
- Accédez à la carte de couverture du menu Vues, qui affiche chaque peptide dans la liste peptidique cartographiée le long de la séquence d’acides aminés de la protéine d’intérêt (Figure 8C). Colorer les peptides en fonction de l’écart type relatif (unités de Da).
- Recherchez visuellement sur la carte des peptides aberrants avec un écart type relatif élevé. Cliquez sur les peptides aberrants de la carte de couverture pour remplir les parcelles spectrales empilées (figure 8B) et la fenêtre de visionneuse de données (figure 8A) avec le peptide cible.
- À l’aide de la parcelle spectrale empilée, vérifiez soigneusement que tous les états de charge et tous les points de temps du peptide aberrant ont correctement attribué des bâtons.
REMARQUE : Le plus souvent, les peptides avec de grands écarts types (>0.3 Da) ont des pics isotopiques qui n’ont pas été convenablement assignés bâtons par le logiciel (comme indiqué dans la figure 8B). L’attribution des bâtons manquants permettra généralement de réduire l’écart type relatif d’un peptide à 0,3 Da. - Cachez le peptide de la liste si l’écart type relatif ne peut pas être réduit à moins de 0,3 Da.
- Exporter des données de différence HDX entre deux états biochimiques pour cartographier sur un modèle structurel de la protéine d’intérêt.
- Affichez la différence d’intérêt dans la carte de couverture. Cliquez à droite sur la carte de couverture pour exporter les données de différence vers .csv fichier. Exporter les données de l’État (.csv format) en naviguant vers les données | Export State Data dans la barre d’outils principale.
REMARQUE : Le formatage approprié des données de différence et des fichiers de données d’état est fourni dans l’information à l’appui. - Importez les données de différence, les données d’état, et le fichier de pdb de la protéine d’intérêt dans Deuteros28. Choisissez l’intervalle de confiance de 99 %, sélectionnez activer lasomme et traiter les données.
REMARQUE : MATLAB doit être installé sur le PC pour faire fonctionner Deuteros. Deuteros utilisera les mesures de reproduction dans l’ensemble de données pour calculer l’écart type des données d’absorption pour chaque peptide. Cet écart type sera utilisé pour définir l’intervalle de confiance pour les échanges significatifs, qui seront affichés sur les parcelles. - Dans le cadre de PyMOL Options, sélectionnez l’absorption à l'| exporter pour générer un script Pymol pour cartographier les régions de différence d’échange significative sur la structure pdb de la protéine d’intérêt à l’aide du logiciel PyMOL.
REMARQUE : En utilisant le flux de travail décrit dans ce protocole, l’intervalle de confiance de 99 % pour la différence significative d’absorption de deutérium pour un peptide donné à un seul moment est généralement de 0,3−0,5 Da. L’intervalle de confiance de 99 % pour l’écart résumé sur tous les points de temps d’échange est généralement de 0,7−1,0 Da.
- Affichez la différence d’intérêt dans la carte de couverture. Cliquez à droite sur la carte de couverture pour exporter les données de différence vers .csv fichier. Exporter les données de l’État (.csv format) en naviguant vers les données | Export State Data dans la barre d’outils principale.
Résultats
Il est nécessaire d’évaluer la qualité de la digestion protéolytique et la reproductibilité du flux de travail pour chaque ensemble d’injections d’échantillons. Ainsi, avant d’effectuer des essais HDX-MS, il est essentiel d’établir des conditions efficaces pour la protéolyse de la protéine d’intérêt, pour la séparation des peptides à l’aide de la chromatographie liquide de phase inverse et de la mobilité des ions de phase gazeuse, et pour la détection des peptides à l’aide de SP. À cette ...
Discussion
Le flux de travail HDX-MS présenté dans ce protocole fournit une plate-forme remarquablement robuste pour cartographier la distribution spatiale des éléments structurellement dynamiques dans les protéines et pour étudier comment ces dynamiques changent en réponse à la perturbation (liaison ligand, mutagenèse enzymatique, etc.). HDX-MS présente plusieurs avantages distincts par rapport à d’autres approches de biologie structurelle qui sont couramment utilisées pour étudier la dynamique conformationnelle. Pl...
Déclarations de divulgation
Nous n’avons rien à divulguer.
Remerciements
Ces travaux ont été appuyés par le Conseil de recherches en sciences naturelles et en génie du Canada, le Fonds de recherche du Québec nature et technologie, la Fondation canadienne pour l’innovation et les fonds de démarrage de l’Université McGill.
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| [glu-1]-fibrinopeptide B (Glu-Fib) | BioBasic | NA | |
| 0.5 mL Amicon Ultracel 10k centrifugal filtration device (Millipore) | Milipore Sigma | UFC501096 | |
| acetonitrile | Fisher | A955-1 | |
| AMP-PNP | SIGMA | A2647-25MG | |
| ATP | SIGMA | a2383-5G | |
| D2O | ALDRICH | 435767-100G | |
| formic acid | Thermo Fisher | 28905 | |
| guanidine-HCl | VWR | 97063-764 | |
| HEPES | Fisher | BP310-1 | |
| Magnesium chloride | SiGMA-Aldrich | 63068-250G | |
| Potassium chloride | BioBasic | PB0440 | |
| potassium phosphate | BioBasic | PB0445 | |
| TCEP Hydrochloride | TRC Canada | T012500 | peptide was synthesized upon request |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| software | |||
| Deuteros | Andy M C Lau, et al | version 1.08 | |
| DynamX | Waters | version 3.0 | |
| MassLynx | Waters | version 4.1 | |
| Protein Lynx Global Server (PLGS) | Waters | version 3.0.3 | |
| PyMOL | Schrödinger | version 2.2.2 | |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| Instrument and equipment | |||
| ACQUITY UPLC BEH C18 analytical Column | Waters | 186002346 | |
| ACQUITY UPLC BEH C8 VanGuard Pre-column | Waters | 186003978 | |
| ACQUITY UPLC M-Class HDX System | Waters | ||
| HDX Manager | Waters | ||
| microtip pH electrode | Thermo Fisher | 13-620-291 | |
| Waters Enzymate BEH column or Pepsin solumn | Waters | 186007233 | |
| Waters Synapt G2-Si | Waters |
Références
- Karplus, M., McCammon, J. A. Molecular dynamics simulations of biomolecules. Nature Structural & Molecular Biology. 9 (9), 646-652 (2002).
- Campbell, E., et al. The role of protein dynamics in the evolution of new enzyme function. Nature Chemical Biology. 12 (11), 944-950 (2016).
- Tokuriki, N., Tawfik, D. S. Protein dynamism and evolvability. Science. 324 (5924), 203-207 (2009).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chemical Society Reviews. 40 (3), 1224 (2011).
- Katta, V., Chait, B. T. Conformational changes in proteins probed by hydrogen-exchange electrospray-ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 5 (4), 214 (1991).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Science. 2 (4), 522 (1993).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Xiao, K., et al. Revealing the architecture of protein complexes by an orthogonal approach combining HDXMS, CXMS, and disulfide trapping. Nature Protocols. 13 (6), 1403-1428 (2018).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Masson, G. R., et al. Recommendations for performing, interpreting and reporting hydrogen deuterium exchange mass spectrometry (HDX-MS) experiments. Nature Methods. 16 (7), 595-602 (2019).
- Liu, J., et al. An Efficient Site-Specific Method for Irreversible Covalent Labeling of Proteins with a Fluorophore. Scientific Reports. 5, 16883 (2015).
- Marion, D., et al. Overcoming the overlap problem in the assignment of proton NMR spectra of larger proteins by use of three-dimensional heteronuclear proton-nitrogen-15 Hartmann-Hahn-multiple quantum coherence and nuclear Overhauser-multiple quantum coherence spectroscopy: application to interleukin 1.beta. Biochemistry. 28 (15), 6150-6156 (1989).
- Balasubramaniam, D., Komives, E. A. Hydrogen-exchange mass spectrometry for the study of intrinsic disorder in proteins. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1834 (6), 1202-1209 (2013).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of the American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Keppel, T. R., Howard, B. A., Weis, D. D. Mapping Unstructured Regions and Synergistic Folding in Intrinsically Disordered Proteins with Amide H/D Exchange Mass Spectrometry. Biochemistry. 50 (40), 8722-8732 (2011).
- Mitchell, J. L., et al. Functional Characterization and Conformational Analysis of the Herpesvirus saimiri Tip-C484 Protein. Journal of Molecular Biology. 366 (4), 1282-1293 (2007).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Habibi, Y., Uggowitzer, K. A., Issak, H., Thibodeaux, C. J. Insights into the Dynamic Structural Properties of a Lanthipeptide Synthetase using Hydrogen-Deuterium Exchange Mass Spectrometry. Journal of the American Chemical Society. 141 (37), 14661-14672 (2019).
- Hebling, C. M., et al. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (13), 5415-5419 (2010).
- Duc, N. M., et al. Effective application of bicelles for conformational analysis of G protein-coupled receptors by hydrogen/deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 26 (5), 808-817 (2015).
- Canul-Tec, J. C., et al. Structure and allosteric inhibition of excitatory amino acid transporter 1. Nature. 544 (7651), 446-451 (2017).
- Reading, E., et al. Interrogating Membrane Protein Conformational Dynamics within Native Lipid Compositions. Angewandte Chemie International Edition. 56 (49), 15654-15657 (2017).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. Journal of Visualized Experiments. (81), e50839 (2013).
- Lento, C., et al. Time-resolved ElectroSpray Ionization Hydrogen-deuterium Exchange Mass Spectrometry for Studying Protein Structure and Dynamics. Journal of Visualized Experiments. (122), e55464 (2017).
- Schowen, K. B., Schowen, R. L. Solvent isotope effects on enzyme systems. Methods in Enzymology. 87, 551-606 (1982).
- Lau, A. M. C., Ahdash, Z., Martens, C., Politis, A. Deuteros: software for rapid analysis and visualization of data from differential hydrogen deuterium exchange-mass spectrometry. Bioinformatics. 35 (7), 3171-3173 (2019).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. Journal of Pharmaceutical Sciences. 100 (6), 2071 (2011).
- Burkitt, W., O'Connor, G. Assessment of the repeatability and reproducibility of hydrogen/deuterium exchange mass spectrometry measurements. Rapid Communications in Mass Spectrometry. 22 (23), 3893-3901 (2008).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13, 2528-2532 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon