Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation de l’oxydation cellulaire à l’aide d’une protéine fluorescente verte rougeox-sensible spécifique à compartiment subcellulaire
Dans cet article
Résumé
Ce protocole décrit l’évaluation de l’état redox subcellulaire spécifique au compartiment dans la cellule. Une sonde fluorescente sensible au redox permet une analyse ratiométrique pratique dans les cellules intactes.
Résumé
La mesure de l’équilibre oxydation/réduction intracellulaire donne une vue d’ensemble du statut physiologique et/ou pathophysiologique redox d’un organisme. Les thiols sont particulièrement importants pour éclairer le statut redox des cellules par l’intermédiaire de leurs rapports réduits de dithiol et de disulfure oxydé. Les protéines fluorescentes contenant de la cystéine d’ingénierie ouvrent une nouvelle ère pour les biocapteurs sensibles au redox. L’une d’entre elles, la protéine fluorescente verte sensible au redox (roGFP), peut facilement être introduite dans les cellules avec transduction adénovirale, permettant d’évaluer le statut redox des compartiments subcellulaires sans perturber les processus cellulaires. Les cysteines réduites et les cystines oxydées de roGFP ont des maxima d’excitation à 488 nm et 405 nm, respectivement, avec l’émission à 525 nm. L’évaluation des ratios de ces formes réduites et oxydées permet le calcul pratique de l’équilibre redox dans la cellule. Dans cet article de méthode, les cellules humaines immortalisées de cancer du sein triple-négative (MDA-MB-231) ont été employées pour évaluer le statut de redox dans la cellule vivante. Les étapes de protocole incluent la transduction de ligne cellulaire MDA-MB-231 avec l’adénovirus pour exprimer le roGFP cytosolique, le traitement avec H2O2,et l’évaluation du rapport de cystéine et de cystine avec la cytométrie de flux et la microscopie de fluorescence.
Introduction
Le stress oxydatif a été défini en 1985 par Helmut Sies comme « une perturbation de l’équilibre prooxydant-antioxydant en faveur de l’ancien »1, et une pléthore de recherches ont été menées pour obtenir le statut de redox spécifique à la maladie, à la nutrition et au vieillissement des organismes1,2,3. Depuis lors, la compréhension du stress oxydatif est devenue plus large. Tester les hypothèses d’utilisation d’antioxydants contre les maladies et / ou le vieillissement a montré que le stress oxydatif non seulement provoque des dommages, mais a également d’autres rôles dans les cellules. En outre, les scientifiques ont montré que les radicaux libres jouent un rôle important pour la transduction du signal2. Toutes ces études renforcent l’importance de déterminer les changements dans le rapport réduction-oxydation (redox) des macromolécules. L’activité enzymatique, les antioxydants et/ou les oxydants, et les produits d’oxydation peuvent être évalués avec diverses méthodes. Parmi ceux-ci, les méthodes qui déterminent l’oxydation du thiol sont sans doute les plus utilisées parce qu’elles rendent compte de l’équilibre entre les antioxydants et les prooxidants dans les cellules, ainsi que les organismes4. Plus précisément, les rapports entre le glutathion (GRATHione disulfure (GSSG) et/ou la cystéine (CyS)/cystine (CySS) sont utilisés comme biomarqueurs pour surveiller l’état redox des organismes2.
Les méthodes utilisées pour évaluer l’équilibre entre les prooxydants et les antioxydants reposent principalement sur les niveaux de protéines réduites/oxydées ou de petites molécules dans les cellules. Les taches occidentales et la spectrométrie de masse sont utilisées pour évaluer de façon générale les ratios de macromolécules réduites/oxydées (protéines, lipides, etc.), et les ratios GSH/GSSG peuvent être évalués avec spectrophotométrie5. Une caractéristique commune de ces méthodes est la perturbation physique du système par la lyse cellulaire et/ou l’homogénéisation des tissus. Ces analyses deviennent également difficiles lorsqu’il est nécessaire de mesurer l’état d’oxydation des différents compartiments cellulaires. Toutes ces perturbations causent des artefacts dans l’environnement d’essai.
Les protéines fluorescentes sensibles à Redox ont ouvert une ère avantageuse pour évaluer l’équilibre redox sans causer de perturbation dans les cellules6. Ils peuvent cibler différents compartiments intracellulaires, permettant la quantification d’activités spécifiques au compartiment (p. ex., l’analyse de l’état redox des mitochondries et du cytosol) pour étudier le croisement entre les organites cellulaires. Les protéines fluorescentes jaunes (YFP), les protéines fluorescentes vertes (GFP) et les protéines HyPeR sont examinées par Meyer et ses collègues6. Parmi ces protéines, les résidus de GFP (roGFP) sensibles au redox (roGFP) sont uniques en raison des différentes lectures fluorescentes de ses résidus de CyS (ex. 488 nm/em. 525 nm) et de CySS (ex. 405 nm/525 nm), ce qui permet l’analyse ratiométrique, contrairement à d’autres protéines sensibles au redox comme YFP7,8. La sortie ratiométrique est précieuse car elle contrebalance les différences entre les niveaux d’expression, les sensibilités de détection et le photobleaching8. Les compartiments subcellulaires des cellules (cytosol, mitochondries, noyau) ou de différents organismes (bactéries ainsi que cellules de mammifères) peuvent être ciblés en modifiant le roGFP7,9,10.
Les tests roGFP sont effectués à l’aide de techniques d’imagerie fluorescente, en particulier pour des expériences de visualisation en temps réel. Des analyses cytométriques de flux de roGFP sont également possibles pour des expériences avec des points de temps prédéterminés. L’article actuel décrit à la fois l’utilisation de la microscopie fluorescente et de la cytométrie de flux pour effectuer une évaluation ratiométrique du statut de redox dans les cellules de mammifères surexprimant le roGFP (ciblé au cytosol) par transduction adénovirale.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE : Ce protocole a été optimisé pour les cellules MDA-MB-231 confluentes à 70 %à 80 %. Pour les autres lignées cellulaires, le nombre de cellules et la multiplicité de l’infection (MOI) doivent être réoptisés.
1. Préparation des cellules (jour 1)
- Maintenir la ligne cellulaire MDA-MB-231 dans des flacons de 75 cm2 avec 10 ml de milieu eagle modifié (DMEM) de Dulbecco complété par 10 % de sérum bovin fœtal (FBS) à 37 °C dans une atmosphère humidifiée de 5 % de CO2.
REMARQUE : DMEM complété par 10 % de FBS, 37 °C et une atmosphère humidifiée de CO2 de 5 % sont utilisés pour toutes les incubations d’attachement et de traitement tout au long du protocole. - Préparez les cellules MDA-MB-231 pour l’expérience.
- Apirate le milieu dans la fiole, détacher les cellules avec 2 ml de 0,25% trypsine-EDTA solution pendant 2 min, et inactiver l’activité trypsine avec 6 ml de milieu complet (DMEM avec 10% FBS). Centrifugez les cellules à 150 x g pendant 5 min. Apirate le supernatant et suspendre les cellules dans 5 mL de milieu complet.
- Mélangez une suspension de cellule à volume égal et 0,4 % de bleu trypan. Prenez 10 μL de ce mélange et comptez les cellules avec le compteur cellulaire automatisé.
REMARQUE : Un compteur Coulter ou un hémocytomètre peut également être utilisé pour le comptage cellulaire. - Ensemencez les cellules dans une plaque de 6 puits pour les analyses de cytométrie de débit et ensemencez 150 000 cellules dans 1 mL de milieu par puits. Attendez 16 h pour l’attachement cellulaire.
- Ensemencez les cellules dans une lame de chambre de 4 puits pour l’imagerie fluorescente et ensemencez 25 000 cellules dans 0,5 mL de milieu par puits. Attendez 16 h pour l’attachement cellulaire.
REMARQUE : Puits de contrôle des semences en plus des puits de traitement. Utilisez l’un des puits de commande pour déterminer le numéro de cellule (facultatif : si la période de fixation des cellules est plus courte que le temps de doublement, le nombre de cellules peut être supposé être le même que la densité d’ensemencement) et l’autre pour un contrôle non infecté (0 MOI).
2. Transduction adénovirale de roGFP (jour 2 et 3)
ATTENTION : Les adénovirus peuvent causer des maladies. Lors de la transducation des cellules, utilisez des pointes filtrées et décontaminez les pointes, les pipettes Pasteur et les tubes de microcentrifuge avec 10 % d’eau de Javel.
REMARQUE : Ce protocole a été démontré avec le roGFP cytosol-spécifique, mais d’autres compartiments cellulaires (par exemple, mitochondrial ou espace intermembrane mitochondrial) peuvent être visés avec ce même protocole.
- Générer une courbe dose-réponse pour que le moi obtienne l’efficacité de transduction la plus élevée en calculant le volume d’adénovirus (mL) requis pour chaque valeur d’OI pour la lignée cellulaire MDA-MB-231 (Tableau 1) :
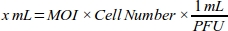
REMARQUE : Le titer fonctionnel de chaque lot de stock adénoviral, qui est exprimé sous forme d’unité de formation de plaque (PFU) par mL, est fourni par la société. Le MOI optimal pour la transduction diffère d’un type de cellule à l’autre. Pour la plupart des cellules de mammifères, la plage optimale d’OI se situe entre 10 et 300. Selon la réponse cellulaire, les valeurs d’OI devraient être recalculées (p. ex., la plage d’OI devrait être réduite si les cellules ont une réponse cytotoxique, ou si la gamme devrait être augmentée si les cellules ont une faible efficacité de transduction). - Faire 1:100 dilution de 6 x 1010 PFU/mL adénoviral roGFP solution avec milieu de culture cellulaire (DMEM avec 10% FBS) pour pipetage fiable.
- Pipette et ajouter 0,0125 mL (12,5 μL), 0,025 mL (25 μL), 0,05 mL (50 μL) de dilution adénovirale de roGFP dans chaque puits de la plaque de puits 6 afin de transduire les 150 000 cellules avec 50, 100 et 200 moi respectivement pour l’analyse de cytométrie de flux (tableau 1).
- Pipette et ajouter 0,0042 mL (4,2 μL) de dilution adénovirale de roGFP dans les puits de diapositives à 4 chambres pour transduire 25 000 cellules avec 100 MOI pour l’imagerie par fluorescence (tableau 1).
REMARQUE : Une quantité minimale de milieu doit être utilisée dans les puits pour assurer l’interaction la plus élevée entre la construction adénovirale de roGFP et les cellules. La teneur en sérum du milieu de culture peut devoir être diminuée pour différentes lignées cellulaires parce que des niveaux élevés de sérum peuvent affecter négativement l’efficacité de transduction dans certains types de cellules. - Incuber les cellules pendant 16 à 24 h dans les conditions d’entretien cellulaire. Le lendemain (jour 3), changer le milieu de culture moyenne à cellule (DMEM avec 10% FBS) pour permettre la récupération cellulaire pour 24 h supplémentaires. Visualiser les cellules au microscope pour évaluer leur morphologie; les cellules peuvent exprimer le roGFP même si elles ont des changements morphologiques.
REMARQUE : Le jour 3, les cellules doivent commencer à exprimer le roGFP; par conséquent, l’efficacité de la transduction peut être surveillée à l’aide de la microscopie par fluorescence (filtres avec l’ex. 488/em. 525). Pour obtenir des résultats d’analyse cohérents, soyez conscient et documentez les changements morphologiques sous le microscope de contraste de phase et observez la morphologie tout en évaluant l’efficacité de transduction. - Construire une courbe de réponse à la dose à l’aide des échantillons de 50, 100 et 200 moi préparés à l’étape 2.3 et de leur efficacité de transduction résultant de l’analyse de cytométrie du débit (étapes 3.1 et 4.1). Évaluer l’efficacité optimale de la transduction grâce à la documentation des changements morphologiques (étape 2.5) et à la courbe dose-réponse de l’OI.
REMARQUE : Bien que plus de 98 % de la population cellulaire à 100 MOI et 200 moi express roGFP (voir résultats représentatifs), 200 groupes de moi ont montré des changements substantiels dans la morphologie cellulaire des cellules MDA-MB-231. Par conséquent, le MOI le plus efficace pour les cellules MDA-MB-231 a été déterminé à 100 MOI. - Après que le MOI optimal (ici, 100 MOI) a été choisi pour la ligne cellulaire MDA-MB-231, effectuer une expérience avec des matériaux d’essai (10 μM H2O2 et son véhicule 0,1% d’eau déionisée).
- Préparer et semer les cellules selon la section 1. En utilisant le volume de transduction adénoviral pour 100 MOI calculé à l’étape 2.1, répétez les étapes 2.2−2.4 pour 100 transduction adénovirale de cellules de MOI. Incuber ensuite la plaque et les lames de chambre selon l’étape 2.5.
3. Acquisition du solde CyS/CySS
- Cytométrie de flux (jour 4)
- Le jour 4, incuber les cellules de l’étape 2.7.1 avec 10 μM H2O2 pendant 1 h.
REMARQUE : 10 μM H2O2 ont été utilisés comme substance d’essai et 0,1 % d’eau déionisée a été utilisée comme traitement du véhicule dans ce protocole. D’autres agents oxydants peuvent être utilisés comme contrôles positifs ici. - Remplacer les supports de la plaque de 6 puits, remplacer par une solution de 750 μL de 0,25 % trypsine-EDTA et attendre 2 min pour que les cellules se détachent. Inactiver la trypsine avec 2 mL de milieu complet (DMEM avec 10% FBS) et recueillir le volume en tubes coniques de 15 mL.
- Centrifugez les tubes à 150 x g pendant 5 min à 4ºC. Jeter le supernatant et suspendre les cellules dans 500 μL de solution saline tamponnée par phosphate (PBS).
- Répétez l’étape 3.1.3
- Filtrer les suspensions cellulaires dans des tubes compatibles avec la cytométrie à l’aide d’un maillage de 40 μm. Gardez les tubes sur la glace et loin de la lumière et suivez l’étape 4.1 pour l’analyse des données.
- Le jour 4, incuber les cellules de l’étape 2.7.1 avec 10 μM H2O2 pendant 1 h.
- Imagerie microscopique (jour 4)
- Le jour 4, traiter les cellules avec 10 μM H2O2, acquérir des images immédiatement (point de temps 0) et 1 h après le traitement et suivre l’étape 4.2 pour l’analyse des données.
4. Analyse des données
- Quantification de cytométrie de flux
- Méthode de cytométrie du débit défini pour 3 analyses différentes via un logiciel d’acquisition d’échantillons(voir tableau des matériaux) : diffusion avant (FCS) sur l’axe x et la dispersion latérale (SSC) sur l’axe y afin d’évaluer la taille et la complexité des cellules (SSC peut être utilisé pour l’identification approximative des cellules mortes et vivantes); ex. 488 nm/em. filtre de pass de bande de 525 nm (fluorescéine isothiocyanate [FITC]) sur l’axe x et SSC sur l’axe y pour évaluer CyS-roGFP; ex. 405 nm/em. Filtre de passe-groupe de 525 nm (Brilliant Violet 510 [BV510]) sur l’axe x et SSC sur l’axe y pour évaluer CySS-roGFP.
- Acquérir 0 protocole d’assurance-vie et visualiser des cellules avec un logiciel d’acquisition d’échantillons. Répétez cette étape pour les échantillons restants (50, 100, 200 groupes d’OI et plus tard sur 10 μM H2O2 cellules traitées et cellules traitées par véhicule). Enregistrez les fichiers pour l’analyse des données.
- Logiciel d’analyse de données ouvertes (voir Tableau des matériaux)et ouvrir 0 fichier d’exemples d’information. Évaluer la population cellulaire d’intérêt (Porte 1). Gluez les gatings suivants pour minimiser la fluorescence de fond pour ex. 488 nm/em. 525 nm (Porte 2) et ex. 405 nm/em. Filtres de bande de 525 nm (Gate 3) avec les cellules témoins non infectées (0 MOI).
- Ouvrez 50, 100 et 200 fichiers d’échantillons d’OI dans un logiciel d’analyse de données pour évaluer la courbe dose-réponse. Analyser les intensités moyennes de fluorescence avec les portes 2 et 3 pour chaque échantillon. Répétez cette étape pour les échantillons d’essai (10 μM H2O2 cellules traitées et cellules traitées par véhicule).
- Calculez le rapport d’intensité fluorescent moyen entre les formes oxydées et les formes réduites de roGFP avec l’équation suivante.

- Évaluation de l’image
- Utilisez un microscope contenant des filtres de fluorescence pour les filtres CyS-roGFP et CySS-roGFP (ex. 488 nm/em. 525 nm et ex. 405 nm/em. 525 nm, respectivement).
- Dans chaque puits de la diapositive de chambre, choisissez 4 zones aléatoires pour acquérir des images, en utilisant l’objectif 4x pour visualiser de plus grandes zones.
REMARQUE : L’objectif 20x peut également être utilisé pour les affichages d’images. - Ouvrez l’image avec le logiciel ImageJ11. Appliquer l’analyse | Mesurer les commandes de chaque image et utiliser l’équation à l’étape 4.1.5 pour quantifier les données.
REMARQUE : La quantification des images est ratiométrique; par conséquent, le protocole n’inclut pas la soustraction de l’arrière-plan. Toutefois, pour pouvoir comparer les images, la luminosité, le contraste et la saturation doivent être les mêmes pour chaque image. L’importance statistique a été évaluée à l’issue de l’analyse à sens unique de la variance (ANOVA) et du test post-hoc de Tukey.
Access restricted. Please log in or start a trial to view this content.
Résultats
L’état redox de CyS/CySS est facilement analysé avec des ropFPP transduct. La sonde fluorescente quantifie le rapport entre les formes réduites et oxydées (longueurs d’onde d’excitation 488 nm et 405 nm, respectivement). Les données de fluorescence peuvent être obtenues par cytométrie de flux et microscopie.
Un grand nombre de cellules peuvent être acquises de façon cohérente et pratique à l’aide de la cytométrie du flux. L’analyse se compose de 3 étapes principales : 1)...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’équilibre thiol/disulfide dans un organisme reflète le statut redox des cellules. Les organismes vivants ont le glutathion, la cystéine, les thiols protéiques et les thiols à faible poids moléculaire, qui sont tous affectés par le niveau d’oxydation et l’écho du statut redox des cellules4. Les ropFPs conçus permettent la quantification non perturbatrice de l’équilibre thiol/disulfure via leurs résidus de CyS7. La propriété ratiométrique du roGFP four...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
La construction et l’adénovirus recombinant pour l’expression du roGFP spécifique au cytosol dans les cellules ont été générés dans le laboratoire de Paul T. Schumacker, PhD, Freiberg School of Medicine, Northwestern University, et ViraQuest Inc., respectivement. Cette étude a été soutenue par le Center for Studies of Host Response to Cancer Therapy grant P20GM109005 par l’intermédiaire du NIH National Institute of General Medical Sciences Centers of Biomedical Research Excellence (COBRE NIGMS), National Institute of General Medical Sciences Systems Pharmacology and Toxicology Training Program grant T32 GM106999, UAMS Foundation/Medical Research Endowment Award AWD00053956, UAMS Year-End Chancellor’s Awards AWD00053484. L’installation de base de cytométrie de flux a été soutenue en partie par le Center for Microin microbicetal Pathogenesis et host Inflammant Responses grant P20GM103625 par l’intermédiaire du COBRE NIGMS. Le contenu est uniquement de la responsabilité des auteurs et ne représente pas nécessairement les vues officielles des NIH. L’ATA a reçu le soutien du Conseil de recherche scientifique et technologique de Turquie (TUBITAK) 2214-A.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco by Life Sciences | 25200-056 | Cell culture |
| 4-well chamber slide | Thermo Scientific | 154526 | Cell seeding material for fluorescent imaging |
| 5 ml tubes with cell strainer cap | Falcon | 352235 | Single cell suspension tube for flow cytometry analysis |
| 6-well plate | Corning | 353046 | Cell seeding material for flow cytometry analysis |
| 15 ml conical tubes | MidSci | C15B | Cell culture |
| 75 cm2 ventilated cap tissue culture flasks | Corning | 4306414 | Cell culture |
| Adenoviral cytosol specific roGFP | ViraQuest | VQAd roGFP | roGFP construct kindly provided by Dr. Schumaker |
| Class II, Type A2 Safety Hood Cabinet | Thermo Scientific | 1300 Series A2 | Cell culture |
| Countess automated cell counter | Invitrogen | C10227 | Cell counting |
| Countess cell counter chamber slides | Invitrogen | C10283 | Cell counting |
| DMEM | Gibco by Life Sciences | 11995-065 | Cell culture |
| FBS | Atlanta Biologicals | S11150 | Cell culture |
| Filtered pipette tips, sterile, 20 µl | Fisherbrand | 02-717-161 | Cell culture |
| Filtered pipette tips, sterile, 1000 µl | Fisherbrand | 02-717-166 | Cell culture |
| Flow Cytometer | BD Biosciences | LSRFortessa | Instrument equipped with FITC and BV510 bandpass filters for flow cytometry analyses |
| Fluorescent Microscope | Advanced Microscopy Group (AMG) | Evos FL | Fluorescent imaging |
| Hydrogen Peroxide 30% | Fisher Scientific | H325-100 | Positive control |
| Light Cube, Custom | Life Sciences | CUB0037 | Fluorescent imaging of roGFP expressing cells (ex 405 nm) |
| Light Cube, GFP | Thermo Scientific | AMEP4651 | Fluorescent imaging of roGFP expressing cells (ex 488 nm) |
| MDA-MB-231 | American Tissue Culture Collection | HTB-26 | Human epithelial breast cancer cell line |
| Microcentrifuge tubes, 2 ml | Grenier Bio-One | 623201 | Cell culture |
| PBS | Gibco by Life Sciences | 10010-023 | Cell culture |
| Pipet controller | Drummond | Hood Mate Model 360 | Cell culture |
| Serologycal pipet, 1 ml | Fisherbrand | 13-678-11B | Cell culture |
| Serologycal pipet, 5 ml | Fisherbrand | 13-678-11D | Cell culture |
| Serologycal pipet, 10 ml | Fisherbrand | 13-678-11E | Cell culture |
| Tissue Culture Incubator | Thermo Scientific | HERACell 150i | CO2 incubator for cell culture |
| Trypan blue stain 0.4% | Invitrogen | T10282 | Cell counting |
Références
- Sies, H. Oxidative stress: A concept in redox biology and medicine. Redox Biology. 4, 180-183 (2015).
- Jones, D. P. Redefining Oxidative Stress. Antioxidants & Redox Signalling. 8 (9-10), (2006).
- Pizzino, G., et al. Oxidative Stress: Harms and Benefits for Human Health. Oxidative Medicine and Cellular Longevity. 20117, 8416763(2017).
- Go, Y. M., Jones, D. P. Thiol/disulfide redox states in signaling and sensing. Critical Reviews in Biochemistry and Molecular Biology. 48 (2), 173-191 (2013).
- Hansen, J. M., Go, Y., Jones, D. P. Nuclear and Mitochondrial Compartmentation of Oxidative Stress and Redox Signaling. Annual Review of Pharmacology and Toxicology. 46 (1), 215-234 (2006).
- Meyer, A. J., Dick, T. P. Fluorescent protein-based redox probes. Antioxidants and Redox Signaling. 13 (5), 621-650 (2010).
- Dooley, C. T., et al. Imaging dynamic redox changes in mammalian cells with green fluorescent protein indicators. Journal of Biological Chemistry. 279 (21), 22284-22293 (2004).
- Björnberg, O., Østergaard, H., Winther, J. R. Measuring intracellular redox conditions using GFP-based sensors. Antioxidants and Redox Signaling. 8 (3-4), 354-361 (2006).
- Bhaskar, A., et al. Reengineering Redox Sensitive GFP to Measure Mycothiol Redox Potential of Mycobacterium tuberculosis during Infection. PLoS Pathogens. 10 (1), 1003902(2014).
- Loor, G., et al. Mitochondrial oxidant stress triggers cell death in simulated ischemia-reperfusion. Biochimica et Biophysica Acta - Molecular Cell Research. 1813 (7), 1382-1394 (2011).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Loor, G., et al. Menadione triggers cell death through ROS-dependent mechanisms involving PARP activation without requiring apoptosis. Free Radical Biology and Medicine. 49 (12), 1925-1936 (2010).
- Esposito, S., et al. Redox-sensitive GFP to monitor oxidative stress in neurodegenerative diseases. Reviews in the Neurosciences. 28 (2), 133-144 (2017).
- Meyer, A. J., et al. Redox-sensitive GFP in Arabidopsis thaliana is a quantitative biosensor for the redox potential of the cellular glutathione redox buffer. Plant Journal. 52 (5), 973-986 (2007).
- Galvan, D. L., et al. Real-time in vivo mitochondrial redox assessment confirms enhanced mitochondrial reactive oxygen species in diabetic nephropathy. Kidney International. 92 (5), 1282-1287 (2017).
- Swain, L., Nanadikar, M. S., Borowik, S., Zieseniss, A., Katschinski, D. M. Transgenic organisms meet redox bioimaging: One step closer to physiology. Antioxidants and Redox Signaling. 29 (6), 603-612 (2018).
- Gutscher, M., et al. Proximity-based protein thiol oxidation by H2O2-scavenging peroxidases. Journal of Biological Chemistry. 284 (46), 31532-31540 (2009).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring EGSH and H2O2 with roGFP2-based redox probes. Free Radical Biology and Medicine. 51 (11), 1943-1951 (2011).
- Dey, S., Sidor, A., O'Rourke, B. Compartment-specific control of reactive oxygen species scavenging by antioxidant pathway enzymes. Journal of Biological Chemistry. 291 (21), 11185-11197 (2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon