Method Article
Profilage métabolique à haut débit pour les raffinements de modèles de microalgues
Dans cet article
Résumé
Ce protocole démontre l’utilisation d’une plate-forme technologique de microréseau phénotype (PM) pour définir les besoins métaboliques de Chlamydomonas reinhardtii, une microalgue verte, et affiner un modèle de réseau métabolique existant.
Résumé
Les modèles métaboliques sont reconstruits sur la base de l’annotation du génome disponible d’un organisme et fournissent des outils prédictifs pour étudier les processus métaboliques au niveau des systèmes. Les modèles métaboliques à l’échelle du génome peuvent inclure des lacunes ainsi que des réactions qui ne sont pas vérifiées expérimentalement. Les modèles reconstruits d’espèces de microalgues nouvellement isolées entraîneront des faiblesses en raison de ces lacunes, car il existe généralement peu de preuves biochimiques disponibles pour le métabolisme de ces isolats. La technologie des microréseaux phénotypiques (PM) est une méthode efficace à haut débit qui détermine fonctionnellement les activités métaboliques cellulaires en réponse à un large éventail de métabolites d’entrée. La combinaison des essais phénotypiques à haut débit avec la modélisation métabolique peut permettre de reconstruire ou d’optimiser rapidement les modèles de réseau métabolique existants en fournissant des preuves biochimiques pour soutenir et élargir les preuves génomiques. Ce travail montrera l’utilisation des tests PM pour l’étude des microalgues en utilisant l’espèce modèle de microalgue verte Chlamydomonas reinhardtii comme exemple. Des preuves expérimentales de plus de 254 réactions obtenues par PM ont été utilisées dans cette étude pour étendre et affiner d’environ 25% un modèle de réseau métabolique de C. reinhardtii à l’échelledu génome, i RC1080. Le protocole créé ici peut être utilisé comme base pour profiler fonctionnellement le métabolisme d’autres microalgues, y compris les mutants de microalgues connus et les nouveaux isolats.
Introduction
L’optimisation du métabolisme des algues pour une production améliorée et stable de métabolites ciblés nécessite le développement de stratégies complexes d’ingénierie métabolique grâce à des analyses des réseaux métaboliques au niveau des systèmes. Les modèles de réseaux métaboliques peuvent guider les conceptions rationnelles pour le développement rapide de stratégies d’optimisation1,2,3,4. Bien qu’environ 160 espèces de microalgues aient été séquencées5, il n’existe, à notre connaissance, que 44 modèles métaboliques d’algues disponibles4,6,7. En raison de la difficulté d’obtenir des données phénotypiques métaboliques à haut débit pour la validation expérimentale de l’information génomique, la reconstruction de modèles de réseau de haute qualité est à la traîne par rapport au développement rapide du séquençage du génome des algues.
C. reinhardtii est un système modèle attrayant pour les études à base d’algues. Cette espèce peut croître photoautotrophe ou hétérotrophe et a été largement utilisée comme organisme modèle dans la recherche fondamentale et appliquée. Sa séquence génomique a été publiée en 20078, avec des modèles métaboliques à l’échelle du génome reconstruits par la suite pour les espèces9,10,11. Le modèle à l’échelle du génome de C. reinhardtii (iRC1080) a été reconstruit par Chang et al. 10 basé sur des preuves génomiques et documentaires (impliquant environ 250 sources). Il a 1 706 métabolites avec 2 190 réactions10; toutefois, l’exhaustivité du modèle n’a pas pu être vérifiée au-delà des preuves expérimentales publiées disponibles à l’époque.
La technologie des microréseaux phénotypiques (PM) est une plate-forme à haut débit qui peut fournir des informations de profilage métabolique pour les micro-organismes hétérotrophes ainsi que pour les cellules de culture tissulaire. En particulier, il peut être utilisé pour combler le déficit de connaissances entre le phénotype et le génotype dans les microalgues, comme cela a été signalé pour La première fois pour Chlamydomonas reinhardtii12 et par la suite pour une espèce de Chloroidium13 et Chlorella14. En étudiant les réponses cellulaires à des milliers de métabolites, de molécules de signalisation, d’osmolytes et de molécules effectrices, les tests PM peuvent fournir un profilage métabolique fonctionnel et offrir des informations sur la fonction, le métabolisme et la sensibilitéenvironnementale 15,16,17. Plus précisément, les tests PM détectent l’utilisation des métabolites des cellules dans des microplaques de 96 puits contenant différents nutriments, métabolites ou osmolytes contenus dans chaque puits. De plus, il est également possible de tester des molécules bioactives, telles que des antibiotiques et des hormones. Comme déterminé par l’intensité de la production de couleur par la réduction du NADH d’un colorant redox à base de tétrazolium, l’utilisation métabolique des substrats est évaluée en termes de respiration cellulaire15,16,17. Les expériences sur des microplaques de 96 puits peuvent être surveillées et déterminées automatiquement au fil du temps avec la plate-forme d’instrument de microréseau phénotypique (PMI). Vingt microplaques de 96 puits sont conçues pour représenter les métabolites communs pour étudier les phénotypes cellulaires afin d’utiliser des sources de carbone, d’azote, de soufre et de phosphore, ainsi que différents effets osmotiques / ioniques et pH. La technologie PM a été utilisée avec succès pour mettre à jour et mettre à niveau un certain nombre de modèles métaboliques existants à l’échelle du génome pourlesmicro-organismes15, 16,17,18.
Le protocole et les données présentées ici sont basés sur des travaux précédemment publiés par Chaiboonchoe et al. 12 Les travaux présentés détaillent l’utilisation de la méthode d’essai des particules pour caractériser les phénotypes métaboliques des microalgues et pour élargir un modèle métabolique algal existant de C. reinhardtii ainsi que pour guider la reconstruction de nouveaux modèles métaboliques.
Protocole
1. Expériences sur les microréseaux phénotypiques
- Obtenez la souche CC-503 de C. reinhardtii auprès du Chlamydomonas Resource Center de l’Université du Minnesota, États-Unis (https://www.chlamycollection.org).
- Cultiver les cellules dans des milieux frais de tris-acétate-phosphate (TAP)19 avec des concentrations finales de 400 μg/mL de timentine, de 50 μg/mL d’ampicilline et de 100 μg/mL de kanamycine (pour inhiber la croissance bactérienne) sous 400 photons micromol/m2 s, à 25 °C, pendant deux jours à mi-phase logarithmique.
- Faire tourner la culture à 2 000 x g pendant 10 min à 22 °C et jeter le surnageant sans déranger la pastille.
- Préparer un milieu TAP frais contenant 0,1% de colorant violet de tétrazolium « D ».
REMARQUE : Modifier les milieux TAP à cette étape pour exclure certains éléments nutritifs en fonction de la catégorie de métabolites testés dans chaque plaque (p. ex., exclure le chlorure d’ammonium pour les plaques sources d’azote). - Resuspendez la pastille dans un milieu TAP frais préparé (à partir de l’étape 1.2) jusqu’à une concentration finale de 1 x 106 cellules/mL.
- Utilisez des plaques d’essai de réseau de composés chimiques (sources de carbone, sources d’azote, plaques de sources de phosphore et de soufre et sources d’azote peptidique).
- Inoculer une aliquote de 100 μL de milieux contenant des cellules dans chaque puits des plaques d’essai.
REMARQUE: Assurez-vous de dupliquer les tests. - Striez les cellules sur les plaques d’extrait de levure / peptone et effectuez une coloration au gramme, comme dans Smith et al. 20 avant et après le test pour surveiller la contamination bactérienne.
- Insérez les plaques d’essai du réseau de composés chimiques dans le système de lecteur de microplaques.
- Incuber toutes les plaques à 30 °C jusqu’à 7 jours et programmer le système de lecteur de microplaques pour lire le changement de couleur du colorant toutes les 15 minutes.
REMARQUE: Comme la plupart des lecteurs de microplaques ne fournissent pas une source de lumière continue pendant l’incubation, les algues devraient pouvoir effectuer une respiration hétérotrophe.
2. Analyse des données
- Exportez les données cinétiques brutes du lecteur de microplaques sous forme de fichiers CSV, qui seront ensuite utilisés comme entrée dans le package Omnilog Phenotype Microarray (OPM) dans R. Ajoutez les informations biologiques sous forme de métadonnées (par exemple, désignation de la souche, milieu de croissance, température, etc.).
- Utilisation du logiciel de conversion de données PM Kinetic; chargez les fichiers de données D5E et convertissez-les en fichiers OKA à l’aide des lignes de commande suivantes dans le logiciel d’analyse cinétique PM :
| de chargement Importer (localiser le dossier des fichiers OKA) | Remplir les filtres | Importer | Ajouter toutes les plaques | Fermer.
| d’exportation choisissez les données de lecture (cinétique), choisissez le format (CSV) (en-tête de tabulation)et choisissez les plaques (chaque plaque (fichiers individuels)) | Exporter des données | Sauvegarder. - Pour effectuer l’analyse des données du phénotype Microarray (PM), utilisez le progiciel OPM21,22 qui s’exécute dans l’environnement logiciel R. Le package, le didacticiel et la documentation de référence sont disponibles à l’adresse suivante : http://www.goeker.org/opm/. Dans RStudio, une interface utilisateur graphique pour R, installez le package opm et ses dépendances à l’aide des commandes suivantes :
source (http://www.goeker.org/opm/install_opm.R)
bibliothèque (opm) - Accédez au répertoire qui contient les fichiers CSV des données cinétiques et importez les données à l’aide de la fonctionopm read_ :
x <- read_opm(« . », convert="grp », include=list (« csv: »)) - Agrégez et discrétisez les données cinétiques à l’aide de l’estimation des paramètres de courbe.
Pour (i en 1 :longueur(x)) {
x[[i]]
pour (I en 1 :length(x)) {x[[i]] <- include_metadata(x[[i]], md = métadonnées, remplacer = TRUE)} - Utilisez la fonction xy_plot pour cartographier les mesures de respiration (ou de croissance) (axe des y) en fonction du temps (axe des x) pour les plaques de 96 puits d’essai.
print (xy_plot(x[[ 1 ]], include ="Strain », theor.max = FALSE)) - Visualisez les données sous forme de carte thermique à l’aide de la fonction level_plot pour permettre un aperçu comparatif rapide des données cinétiques.
level_plot(x, main = list(), colors = opm_opt(« color.borders »), panel.headers = metadata$Strain, cex = NULL, strip.fmt = list(), striptext.fmt = list(), legend.sep = » « , space ="Lab », bias = 0.7 , num.colors = 200 L) - Extraire des informations biologiques importantes, les paramètres de la courbe, à partir des courbes cinétiques brutes et inclure la phase de décalage (λ), le taux de croissance (μ), la respiration cellulaire maximale (A) et l’aire sous la courbe (ASC)21. Pour identifier les métabolites positifs, utilisez les valeurs A du témoin négatif, qui représente la réactivité abiotique du colorant avec le milieu, en plus de l’ébauche de chaque plaque de micropuit comme valeurs de soustraction de fond. La fonction d’extraction est utilisée pour obtenir le paramètre A.
opm_opt(« curve.param »)
param <- extrait (x, as.labels = list(« Strain »)))
- Utilisation du logiciel de conversion de données PM Kinetic; chargez les fichiers de données D5E et convertissez-les en fichiers OKA à l’aide des lignes de commande suivantes dans le logiciel d’analyse cinétique PM :
3. Identification des réactions et des gènes associés aux nouveaux métabolites
- Recherchez KEGG (Kyoto Encyclopedia of Genes and Genomes) (http://www.genome.jp/kegg/) et MetaCyc (http://metacyc.org/) pour identifier les numéros de commission des enzymes (CE) pour les réactions utilisant des métabolites trouvés dans des réseaux de composés chimiques23,2423,24.
- Utilisez les numéros CE identifiés comme base de recherche dans plusieurs ressources d’annotation d’algues disponibles telles que joint Genome Institute (JGI), Phytozome (http://www.phytozome.net) et les publications évaluées par des pairs23,25,26,27.
- Si une requête ne renvoie aucune preuve génétique pour un numéro CE donné, identifier les protéines associées pertinentes dans d’autres organismes, en commençant par les espèces les plus proches du C. reinhardtii, puis effectuer une recherche basée sur un profil à l’aide du serveur NCBI PSI-BLAST avec les paramètres par défaut et utiliser des protéines non redondantes (nr) dans C. reinhardtii (taxid:3055) pour identifier les gènes candidats associés à la réaction12.
- Organisez manuellement les résultats PSI-BLAST avec des valeurs E de < 0,05 pour la pertinence par rapport au numéro EC recherché en interrogeant ces résultats BLAST via les serveurs de prédiction de domaine protéique EMBL-EBI Pfam (http://pfam.xfam.org/search) ou InterPro (http://www.ebi.ac.uk/interpro/). Notez que les deux derniers scans sont des étapes critiques pour assurer l’identification de l’activité enzymatique correcte pour la protéine.
4. Perfectionnement et évaluation du modèle
- Utiliser la dernière version de COBRA Toolbox v.3.028dans MATLAB29,30pour effectuer les étapes suivantes pour affiner le modèle. La boîte à outils COBRA peut être installée en suivant les étapes décrites dans : https://opencobra.github.io/cobratoolbox/stable/installation.html . Vous pouvez également noter que la boîte à outils COBRA est également implémentée dans d’autres langages de programmation open source, tels que Python (COBRApy31) et est disponible à l’adresse suivante: https://opencobra.github.io/cobrapy/ .
- Après avoir installé COBRA Toolbox v.3.0, ouvrez MATLAB et exécutez la commande suivante pour initialiser la boîte à outils :
initCobraToolbox; - Ajoutez les réactions identifiées avec leurs gènes associés au modèle métabolique, tels que iRC1080, à l’aide des fonctions de la boîte à outils COBRA addReaction et changeGeneAssociation. Accédez au répertoire qui contient le modèle iRC1080, téléchargé à partir de http://bigg.ucsd.edu/models/iRC1080 et exécutez les commandes suivantes pour charger le modèle, le renommer et ajouter une nouvelle réaction et son gène associé.
Chargement('iRC 1080 .mat')
modelNew = iRC 1080;
modelNew = addReaction(modelNew, 'D-ALA 2' , ...
{'d-ala[c]' , 'atp[c]' , ...
'D-aladata[c]' , 'adp[c]' , 'pi[c]' , ...
'h[c]' },[- 2 - 1 1 1 1 1 ],false);
modelNEW = changeGeneAssociation(modelNew, ...
« D-ALA 2 », « au.g 14655 _t 1 »); - Dans certains cas, lorsque le métabolite n’est pas produit intracellulairement mais est repris du milieu, ajoutez des réactions de transport pour les nouveaux métabolites au modèle. Ces réactions de transport représentent la diffusion passive d’un métabolite du milieu extracellulaire vers le cytosol. En outre, ajoutez une réaction d’échange artificielle correspondante à l’aide de la fonction addExchangeRxn pour entrer ou sortir le métabolite dans le milieu extracellulaire.
modelNew = addReaction(modelNew, 'CYCPt' , ...
{'cycp[e]','cycp[c]' },[- 1 1 ],true)'
modelNew = addExchangeRxn(modelNew, 'cycp[e]' ,- 1000 ,1000 ); - Testez le comportement du nouveau modèle résultant, par exemple iBD1106, en effectuant une analyse du balance de flux (FBA) à l’aide de la fonction optimizeCbModel dans des conditions claires et sombres pour la maximisation de la biomasse en tant que fonction objective. Pour la croissance de la lumière, réglez les limites inférieure et supérieure des réactions lumineuses de la litho' solaire PRISM à 646,07 (taux maximal). Pour la croissance sombre, définissez les limites de toutes les réactions lumineuses PRISM à zéro. Utilisez la fonction Biomasse définie précédemment10 pour la croissance dans des conditions sombres et claires. La solution Expédié par Amazon produira deux vecteurs correspondant aux flux de réaction (solution.v) et au coût réduit (solution.w), ainsi qu’un vecteur correspondant aux prix fictifs des métabolites (solution.y).
%Simuler la croissance à l’état de lumière :
modelNew = changeRxnBounds(modelNew,{ ...
% « PRISM_solar_litho », ...
« PRISM_solar_exo », ...
'PRISM_incandescent_ 60 W' , ...
'PRISM_fluorescent_cool_ 215 W' , ...
« PRISM_metal_halide », ...
« PRISM_high_pressure_sodium », ...
« PRISM_growth_room », ...
« PRISM_white_LED », ...
« PRISM_red_LED_array_ 653 nm », ...
« PRISM_red_LED_ 674 nm », ...
'PRISM_fluorescent_warm_ 18 W' , ...
« PRISM_design_growth », ...
}, 0, 'b' );
modelNew = changeObjective(modelNew, 'BIOMASS_Chlamy_mixo');
FBAsolutionNew = optimizeCbModel(modelNew, 'max'); - Répétez l’étape 4.1.4 pour iRC1080 afin de comparer les solutions Expédié par Amazon obtenuespour i BD1106 avec celles obtenuespour i RC1080.
- Il existe une gamme de méthodes COBRA disponibles qui peuvent être utilisées pour comparer des modèles (par exemple, analyse de la variabilité du flux, études de délétion de gènes, analyses de robustesse, prédictions de fractionnement de flux, FBA, échantillonnage, etc.). Des tutoriels détaillés peuvent être trouvés sur https://opencobra.github.io/cobratoolbox/stable/tutorials/index.html. Ici, un exemple est fourni où le modèle iRC1080 est comparé à sa version raffinée, iBD1106, en obtenant les prix fictifs (sensibilité de la fonction objectif biomasse aux changements de variable système) des métabolites pris en compte dans chaque modèle.
Obtenez les prix de l’ombre pour les métabolites:
shadowPrices = table(modelNew.mets, ...
modelNew.metNames, FBAsolutionNew.y);
- Après avoir installé COBRA Toolbox v.3.0, ouvrez MATLAB et exécutez la commande suivante pour initialiser la boîte à outils :
Résultats
Phénotype Microréseau de criblage de l’algue modèle Chlamydomonas reinhardtii
Les tests PM testent la capacité de l’algue à utiliser diverses sources de carbone, d’azote, de soufre et de phosphore dans un milieu minimal. Dans cette description des méthodes, nous avons démontré comment les tests de particules ont été utilisés pour identifier le métabolisme du carbone et de l’azote. La cinétique d’utilisation du carbone et de l’azote a été mesurée à l’aide d’un lecteur de microplaques. Les données ont été analysées à l’aide du logiciel PMI. La cinétique sommaire des plaques d’essai des particules sélectionnées (PM01 et PM03) est illustrée à la figure 1. Les « diagrammes xy » affichent les mesures de respiration au fil du temps tracées pour les essais des plaques à 96 puits, où l’axe y et l’axe x représentent les valeurs des mesures brutes et du temps, respectivement. Les données ont été converties en un modèle de carte thermique pour analyser comparativement l’assemblage des données cinétiques.
Le pipeline de raffinage du réseau métabolique à l’échelle du génome à l’aide de données sur les particules(figure 2)illustre l’intégration des essais de particules à haut débit avec les preuves expérimentales fournies par les recherches génomiques qui peuvent élargir un modèle de réseau métabolique.
Pour déterminer la reproductibilité des données PM obtenues à partir de plaques PM01 -04 et PM10, une régression linéaire a été analysée pour tracer les données de deux expériences répliquées indépendantes l’une par rapport à l’autre (Figure 3). La figure 3 montre que la majorité des données étaient presque similaires car elles tombent sur la ligne des 45°, avec seulement quelques valeurs aberrantes présentes, et leur coefficient de détermination R2 était de 0,9. La cohérence et la reproductibilité des expériences pour l’algue sont vérifiées par ce graphique.
Identification de nouveaux métabolites
Le test PM a identifié 662 métabolites dans sept plaques; PM01-PM04 et PM06-PM08, tandis que la chromatographie en phase gazeuse à temps de vol (GC-TOF) avait identifié 77 métabolites32 (Figure 4). En comparant ces deux ensembles avec les 1068 métabolites comptabilisés dans le iRC1080, seuls six métabolites se chevauchaient entre les trois ensembles, et 149 se chevauchaient entre le iRC1080 et le PM. Ce résultat démontre que la plateforme de profilage métabolique peut être une source importante de nouvelles informations métaboliques.
L’acide acétique était la seule source de carbone détectée dans la plaque PM01 en tant que carbone de soutien après soustraction du signal de fond. Ce résultat est conforme à la littérature33 et montre la spécificité des tests pm. Les tests PM ont révélé de nouvelles sources de soufre, de phosphore et d’azote que C. reinhardtii peut utiliser pour la croissance. Les métabolites soufrés étaient le sulfate, le thiosulfate, le tétrathionate et le DL-lipoamide. Les sources de phosphore étaient le thiophosphate, le dithiophosphate, l’acide D-3-phospho-glycérique et la cystéamine-S-phosphate. Les métabolites sources d’azote étaient les acides L-aminés et D-aminés, y compris les acides aminés moins courants; L-homosérine, L-pyroglutamique, méthylamine, éthylamine, éthanolamine et D,L-α-amino-butyrique, et 108 Di-peptides et cinq tri-peptides (Tableau 1). Les 128 métabolites nouvellement identifiés ont été recherchés dans KEGG et MetaCyc pour leurs réactions associées, leur nombre d’EC et leurs voies.
Les 128 nouveaux métabolites ont été associés à 49 numéros CE uniques. Parmi ceux-ci, 15 CE ont été liés à leurs preuves génomiques à l’aide de cinq sources, notamment: Phytozome Version 10.0.234 JGI Version 435, AUGUSTUS 5.0 et 5.210 annotations de Manichaikul et al. 36 et KEGG13. Les métabolites sans preuve génomique ont été saisis sur le site Web Universal Protein Resource (UniProt, http://www.uniprot.org/)37,38 où leurs séquences connexes ont été trouvées dans d’autres organismes. Les séquences homologues chez C. reinhardtii ont été identifiées en exécutant le BLAST itéré spécifique à la position (PSI-BLAST, https://blast.ncbi.nlm.nih.gov/Blast.cgi) à partir du site Web du NCBI en ne considérant que les séquences qui produisaient des alignements significatifs (valeur E <0,005).
Raffinement du modèle
Les réactions associées aux nouveaux métabolites 128, ainsi qu’à leurs gènes codés, ont été ajoutées au modèle iRC1080, élargissant ainsi le réseau. Le modèle résultant iBD1106, représente 2 444 réactions, 1 959 métabolites et 1 106 gènes (Tableau 2). Les 254 nouvelles réactions ajoutées étaient 20 réactions d’oxydation d’acides aminés, 108 réactions d’hydrolyse di-peptidique, cinq réactions d’hydrolyse de tripeptides et 120 réactions de transport, codées par quatre gènes (Cre02.g096350.t1.3, au.g14655_t1, e_gwW.1.243.1, Cre12.g486350.t1.3).
Au total, 113 nouvelles réactions ajoutées expliquent l’hydrolyse des di-peptides et des tri-peptides. L’hydrolyse des di-peptides et des tri-peptides est associée à deux gènes, l’un pour les di-peptides (Cre02.g078650.t1.3) et l’autre pour les tri-peptides (Cre16.g675350.t1.3).
En ce qui concerne les sources de phosphore, une réaction d’hydrolyse de la cystéamine-S-phosphate en cystéamine et en phosphate a été ajoutée associée au gène JLM_162926.
L’outil WoLF PSORT39 (http://www.genscript.com/psort/wolf_psort.html) et les résultats rapportés par Ghamsari et al. 35 ont été appliqués pour obtenir la spécification des compartiments cellulaires où les nouvelles réactions ont lieu. En analysant les séquences protéiques associées aux nouvelles réactions, WoLF PSORT a prédit le cytosol comme compartiment cellulaire des réactions.
Un modèle métabolique généré peut contenir des lacunes lorsque l’information biochimique est incomplète. Dans de tels cas, gapFind, une commande COBRA, est utilisée. Il répertorie les lacunes profondes et permet d’identifier les nouvelles lacunes dans le nouveau modèle. Les métabolites qui ne peuvent pas être produits dans un modèle métabolique sont appelés lacunes radiculaires40,41. L’analyse de l’écart radiculaire a indiqué que les modèles iRC1080 et iBD1106 contiennent les mêmes écarts 91. Cela montre que l’ajout des nouveaux métabolites et de leurs réactions associées n’a pas introduit de lacunes radiculaires supplémentaires. Il convient de noter que la méthode de phénotypage utilisée dans ce protocole ne comble pas les lacunes racinaires, car les métabolites de l’écart racinaire d’origine manquent de mécanismes de transport ou de production, qui n’ont pas été abordés dans les essais de phénotypage. L’analyse du bilan de flux a été réalisée pour tester le comportement métabolique de l’iBD1106 dans des conditions claires et sombres; (pas d’acétate) et (avec de l’acétate), respectivement. L’algorithme maximise les réactions précurseurs de la biomasse pour une fonction objective (croissance de la biomasse). Pour évaluer l’implication de chaque métabolite dans la fonction objective définie, des « prix fictifs » pour tous les métabolites ont été calculés. Le changement de la fonction objective concernant les changements de flux du métabolite définit le prix de l’ombre d’un métabolite30,42. L’indication de la question de savoir si un métabolite est en « excès » ou « limite » la fonction objective peut être déterminée par une analyse des prix fictifs, par exemple la production de biomasse. Les valeurs de prix de l’ombre négatives ou positives révèlent des métabolites qui, en cas d’addition, diminueront ou augmenteront la fonction objective. Les valeurs nulles des prix fictifs révèlent des métabolites qui n’affecteront pas la fonction objective. La comparaison des prix fictifs entre iBD1106 et iRC1080 à la figure 5 montre que, pour la plupart des métabolites, aucun changement significatif n’est observé; cependant, des différences sont trouvées dans 105 et 70 cas dans des conditions de croissance claires et sombres, respectivement. Le tableau 4 comprend des exemples de ces métabolites.
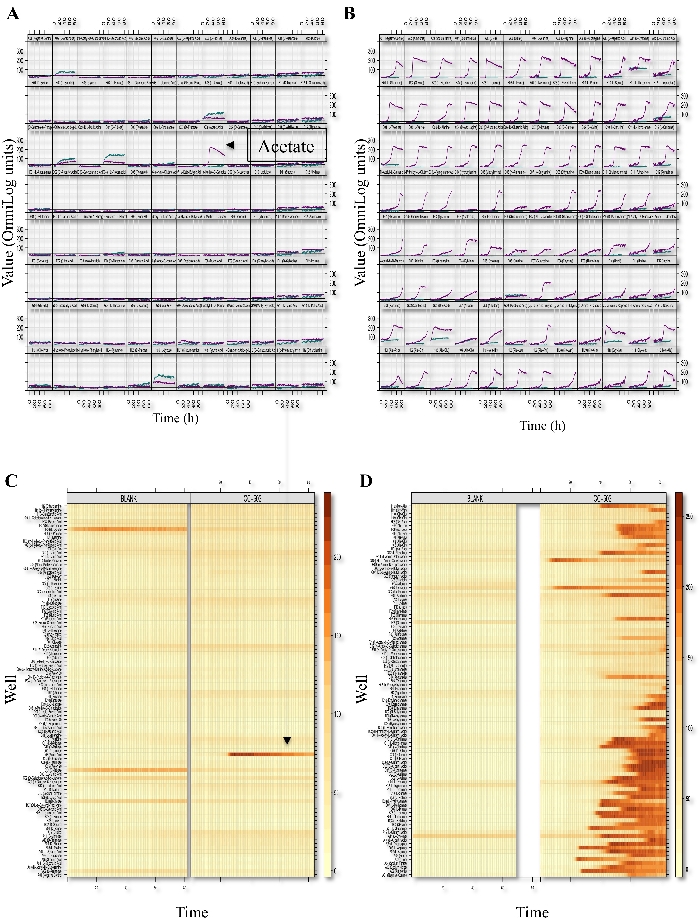
Figure 1: Profilage phénotypique des microréseaux de C. reinhardtii. Diagrammes XY de respiration et diagrammes de niveau des PM01 (sources de carbone; A, C) et PM03 (sources d’azote; B, D) les plaques d’essai sont montrées. La figure est un réseau 8x12 où chaque cellule représente une plaque de puits et, par conséquent, un métabolite ou un environnement de croissance donné. Dans chaque représentation de cellule ou de puits, les courbes représentent la conversion de colorant par réduction (axe des y) en fonction du temps (axe des x). Les courbes de respiration des particules du CC-503 et des puits vierges sont indiquées dans chaque cellule et sont indiquées par couleur (la couleur sarcelle représente les puits vierges et la couleur pourpre représente CC-503). Le diagramme de niveau représente chaque courbe de respiration sous la forme d’une fine ligne horizontale changeant de couleur (ou restant inchangée) au fil du temps. Les changements de couleur de la carte thermique vont du jaune clair (peu ou pas de respiration a eu lieu) à l’orange foncé ou au brun (une respiration importante a eu lieu). Les métabolites utilisés par C. reinhardtii (CC-503) et les plaques vierges sont représentés. Cette figure est tirée d’un travail précédemment publié par Chaiboonchoe et al. 12Veuillez cliquer ici pour voir une version agrandie de cette figure.
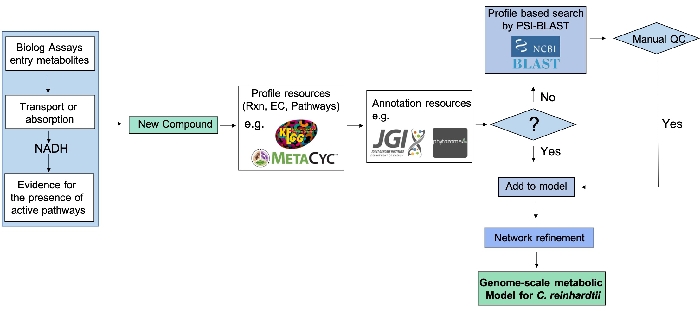
Figure 2: Pipelined’affinement du réseau métabolique à l’échelle du génome à l’aide de données sur les particules. Une fois qu’un nouveau composé est positif dans un essai de particules, son numéro de commission des enzymes (EC), sa réaction et sa voie sont identifiés à partir des bases de données disponibles, par exemple KEGG et MetaCyc. Les preuves génomiques sont ensuite extraites des ressources génomiques et d’annotation lorsqu’elles sont disponibles et constituent un lien entre le génotype et le phénotype. Lorsque les preuves génomiques directes ne sont pas disponibles, la séquence protéique est identifiée à partir des numéros EC, et les preuves génétiques sont identifiées via PSI-Blast. Le réseau métabolique reconstruit est ensuite affiné sur la base de composés nouvellement identifiés, mais seulement après une étape de contrôle de la qualité qui consiste à interroger les domaines protéiques à l’aide de bases de données pertinentes. Ce chiffre a été modifié à partir de travaux précédemment publiés par Chaiboonchoe et al. 12Veuillez cliquer ici pour voir une version agrandie de cette figure.
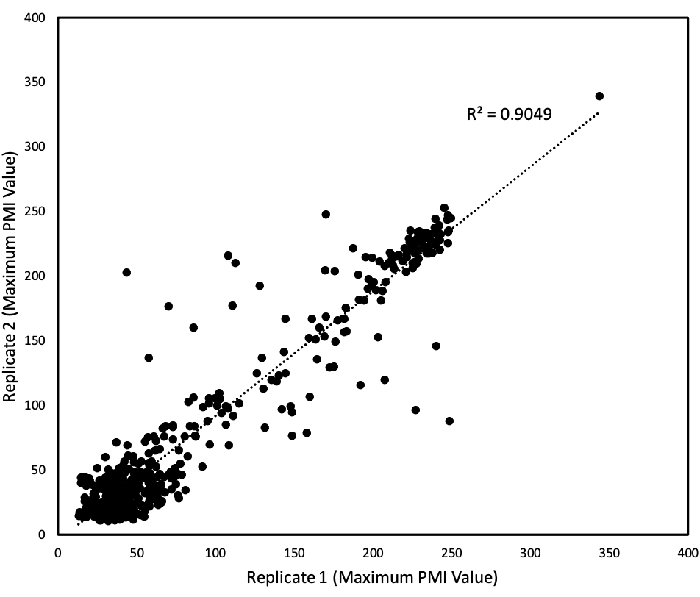
Figure 3: Reproductibilité des essais pm. Les valeurs PMI ont été collectées sur une période de 168 heures, et les valeurs PMI maximales ont été tracées pour deux études répliquées. Chaque axe représente les valeurs PMI maximales pour chaque étude (l’axe des x étant une étude répliquée et l’axe des y une autre). Les valeurs reproduites sont équidistantes de chaque axe. Chaque point représente une valeur maximale unique. La régression linéaire a été effectuée par un tableur, et le coefficient de détermination résultant (R2)est affiché. Ce chiffre a été modifié à partir de travaux précédemment publiés par Chaiboonchoe et al. 12Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4: Diagramme de Venn des métabolites. Le diagramme de Venn énumère les métabolites identifiés par les plaques PM, le modèle métabolique iRC1080 et les expériences de temps de vol par chromatographie en phase gazeuse (GC-TOF). Chaque cercle indique le nombre total de métabolites qui existent dans chaque méthode d’étude respective. Dans le même temps, les régions qui se chevauchent représentent le nombre de métabolites partagés entre ces méthodes. Le modèle métabolique iRC1080 contient un total de 1 068 métabolites uniques. Le GC-TOF a identifié un total de 77 métabolites32, tandis qu’il y a un total de 662 métabolites identifiés à l’aide des plaques PM. Ce chiffre est tiré de travaux précédemment publiés par Chaiboonchoe et al. 12Veuillez cliquer ici pour voir une version agrandie de cette figure.
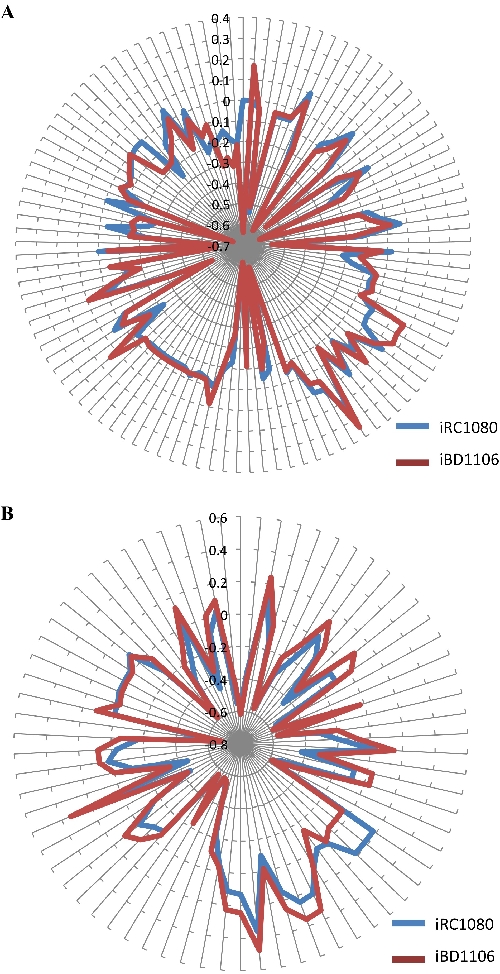
Figure 5: Prix fictifs des métabolites dans iRC1080 et iBD1106 dans des conditions différentes pour la maximisation de la biomasse. Chaque cercle sur les « tracés radar » correspond à une valeur de prix d’ombre, tandis que chaque ligne s’étendant du centre d’un tracé indique un métabolite. (A) Prix de l’ombre et comportements métaboliques de iRC1080 et iBD1106 dans une condition de croissance légère; (B), différents comportements métaboliques de iRC1080 et iBD1106 dans un état de croissance sombre. Ce chiffre est tiré de travaux précédemment publiés par Chaiboonchoe et al. 12Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Biologie Chimique | CE* | Annotation de gènes | PSI-BLAST |
| Cystéamine-S-Phosphate | 3.1.3.1 | JLM_1629261,2,3,4 | |
| Tétrathionate | 1.8.2.2 | valeur E insignifiante | |
| 1.8.5.2 | valeur E insignifiante | ||
| D-Alanine | 1.4.1.1 | XP_001700222.1 | |
| 1.5.1.22 | échec du contrôle qualité manuel | ||
| 2.1.2.7 | valeur E insignifiante | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 2.3.2.10 | valeur E insignifiante | ||
| 2.3.2.14 | valeur E insignifiante | ||
| 2.3.2.16 | valeur E insignifiante | ||
| 2.3.2.17 | valeur E insignifiante | ||
| 2.3.2.18 | valeur E insignifiante | ||
| 2.6.1.21 | échec du contrôle qualité manuel | ||
| 3.4.13.22 | XP_001698572.1, XP_001693532.1, XP_001701890.1, XP_001700930.1 | ||
| 3.4.16.4 | Chlre2_kg.scaffold_ 140000391,2,3 | ||
| 3.4.17.8 | échec du contrôle qualité manuel | ||
| 3.4.17.13 | valeur E insignifiante | ||
| 3.4.17.14 | valeur E insignifiante | ||
| 4.5.1.2 | valeur E insignifiante | ||
| 6.1.1.13 | échec du contrôle qualité manuel | ||
| 6.1.2.1 | échec du contrôle qualité manuel | ||
| 6.3.2.4 | au.g14655_t11,2,3 | ||
| 6.3.2.10 | échec du contrôle qualité manuel | ||
| 6.3.2.16 | valeur E insignifiante | ||
| 6.3.2.35 | valeur E insignifiante | ||
| D-Asparagine | 1.4.5.1 | valeur E insignifiante | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 3.1.1.96 | valeur E insignifiante | ||
| 2.3.1.36 | valeur E insignifiante | ||
| 1.4.99.1 | XP_001692123.1 | ||
| 3.5.1.77 | e_gwW.1.243.11,2 | ||
| 3.5.1.81 | valeur E insignifiante | ||
| 5.1.1.10 | échec du contrôle qualité manuel | ||
| Acide D-aspartique | 6.3.1.12 | valeur E insignifiante | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Acide D-glutamique | 1.4.3.7 | valeur E insignifiante | |
| 1.4.3.3 | valeur E insignifiante | ||
| D-Lysine | 5.4.3.4 | valeur E insignifiante | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 6.3.2.37 | échec du contrôle qualité manuel | ||
| D-Sérine | 2.7.11.8 | valeur E insignifiante | |
| 2.7.11.17 | Cre12.g486350.t1.31,2,3,4 | ||
| 3.4.21.78 | échec du contrôle qualité manuel | ||
| 3.4.21.104 | échec du contrôle qualité manuel | ||
| 4.3.1.18 | g6244.t14 | échec du contrôle qualité manuel | |
| 6.3.2.35 | valeur E insignifiante | ||
| 6.3.3.5 | valeur E insignifiante | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-Valine | 1.21.3.1 | échec du contrôle qualité manuel | |
| 6.3.2.26 | échec du contrôle qualité manuel | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Acide L-pyroglutamique | |||
| Thiophosphate | |||
| Dithiophosphate | |||
| Éthylamine | 6.3.1.6 | ||
| Acide D,L-a-amino-butyrique | 2.1.1.49 | valeur E insignifiante | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Di-peptide | 3.4.13.18 | Cre02.g078650.t1.31 | |
| Tri-peptide | 3.4.11.4 | Cre16.g675350.t1.31 |
Tableau 1: Liste des métabolites d’utilisation du substrat positif identifiés (C, P, S, N) non présents dans le modèle métabolique iRC1080. *La réaction n’a pas été incluse si aucun gène n’a été identifié. 1 Phytozome version 10.0.2 (http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Creinhardtii). deux JGI version 4 35. 3 Auguste version 510. 4 KEGG (http://www.genome.jp/kegg/kegg1.html). 5 JGI version 3.136. Ce tableau est tiré de travaux précédemment publiés par Chaiboonchoe et al. 12
| Modèle | Réactions | Métabolites | Gènes |
| iRC1080 | 2,191 | 1,706 | 1,086 |
| iBD1106 | 2,445 | 1,959 | 1,106 |
Tableau 2: Contenu des iRC1080 et iBD1106. Ce tableau est tiré de travaux précédemment publiés par Chaiboonchoe et al. 12
| Catégorie ou classe de réactions | Nombre de réactions |
| Acides aminés | 20 |
| Dipeptides | 108 |
| Tripeptides | 5 |
| Réaction de transport | 120 |
Tableau 3: Résumé des nouvelles réactions dans le BD1106. Ce tableau est tiré de travaux précédemment publiés par Chaiboonchoe et al. 12
| Conditions de croissance | Métabolite | Nom | iRC1080 | iBD1106 | |
| 4r5au | 4-(1-D-Ribitylamino)-5-aminouracile | 0 | 0.168 | ||
| 5aprbu | 5-Amino-6-(5'-phosphoribitylamino)uracile | -0.009 | 0.158 | ||
| Lumière | pa1819Z18111Z | 1-(9Z)-octadécénoyl,2-(11Z)-octadénoyl-sn-glycérol3-phosphate | -0.009 | -0.65 | |
| Sombre | 4amais | 4-aminobutanoate | 0.18 | -0.05 | |
Tableau 4: Exemple de prix fictifs significatifs pour iRC1080 et iBD1106. Ce tableau est tiré de travaux précédemment publiés par Chaiboonchoe et al. 12
Discussion
Le phénotypage métabolique de la microalgue verte, C. reinhardtii,a été décrit ici à l’aide de plaques de dosage des particules à haut débit et d’un PMI non modifié. Les essais ont été utilisés pour un total de 190 sources de carbone (PM01 et PM02), 95 sources d’azote (PM03), 59 sources de phosphore et 35 sources de soufre (PM04), ainsi que des sources d’azote peptidique (PM06-08). Une respiration positive a été observée pour 148 nutriments (un test positif pour l’utilisation de la source C, quatre tests positifs pour chacune des utilisations de la source S et de la source P, et 139 tests positifs pour l’utilisation de la source N). Les substrats ou les éléments nutritifs (carbone, azote, phosphore ou soufre) du milieu ne doivent pas être ajoutés au milieu défini lorsqu’ils sont appliqués sur les microplaques de particules pertinentes qui testent pour chacune de ces sources.
La méthode présentée ici est efficace pour caractériser les phénotypes de microalgues métaboliques qui peuvent être utilisés pour étendre les modèles de réseau métabolique existants ou diriger la reconstruction de nouveaux modèles. De plus, comme les besoins nutritionnels de la plupart des microalgues ne sont pas connus, cette plate-forme peut être utilisée pour les définir rapidement. Nelson et coll. 43 avait appliqué avec succès ces méthodes pour identifier de nouveaux composés qui soutiennent la croissance des microalgues Chloroidium sp. UTEX 3007 et a utilisé les informations obtenues pour définir les métabolites d’entrée de l’espèce, qui, contrairement à Chlamydomonas, comprennent 40 sources de carbone différentes.
L’une des principales limites du PM pour le profilage des microalgues est que le PMI n’a pas d’éclairage dans la chambre d’incubation et que les microalgues doivent pouvoir effectuer un métabolisme hétérotrophe. L’absence de lumière pourrait affecter l’interprétation des modèles qui incorporent la lumière pour calculer les flux métaboliques. Les paires de gènes avec les fonctions de coordination ont co-évolué pour constituer des centres de réseau métabolique, et la distinction entre les hubs de réseau photosynthétique et non photosynthétique peut être faite44. En général, les concentrateurs de réseau photosynthétiques (c.-à-d. les nœuds hautement connectés dans le modèle) seraient exclus des modèles hétérotrophes. À des fins pratiques, la modélisation de l’hétérotrophisme chez les espèces mixotrophes devrait omettre les réactions connues pour être entraînées par la lumière et tenir compte des différences de bilan énergétique entre les conditions. Ainsi, la modélisation du métabolisme dépendant de la lumière et indépendant de la lumière est une pratique courante dans la modélisation métabolique de Chlamydomonas6,45.
Certaines microalgues vertes, comme les trébouxiophytes, sont connues pour assimiler une variété de molécules de carbone pour la croissance, et on pense que cela est issu de leur longue histoire évolutive en tant que membres des lichens46. Alors que les chlorophytes comme Chlamydomonas peuvent utiliser l’acétate pour la croissance, la microalgue marine brune Tisochrysis lutea, connue pour son potentiel à produire commercialement des acides gras polyinsaturés à très longue chaîne (AGPI-VLC), ne peut pas utiliser l’acétate mais peut utiliser le glycérol pour la croissance47. Une concentration de biomasse supérieure à 100 g l−1 de poids de cellule sèche a été atteinte avec Chlorella avec ajout optimisé de sources de carbone organique en mode batch nourri48. De plus, l’ajout de sucre à chlorellavulgaris peut élever sa séquestration de CO2, offrant ainsi un avantage additif lors de la croissance photosynthétique49. La plupart des microalgues hétérotrophes peuvent également se développer de manière mixotrophique, mais il a été démontré que le chlorophyte Chromochloris zofingiensis arrête la photosynthèse lors de l’ajout de sucre50.
Les diatomées, appartenant à la division Bacillariophyta, sont un groupe majeur de phytoplancton. Bien que la plupart des diatomées ne puissent se développer que photoautotrophement, certaines d’entre elles peuvent être cultivées de manière mixotrophe ou hétérotrophe51. Par exemple, le glycérol s’est avéré soutenir la croissance de la lumière en l’absence de CO2 chez certaines diatomées, y compris l’espèce modèle Phaeodactylum tricornutum52. En outre, certaines diatomées benthiques comme Nitzschia linearis peuvent se développer sur les glucides dans l’obscurité53. Il est probable qu’il étende les tests de particules aux diatomées et à d’autres groupes d’algues en complétant les sources de carbone organique appropriées pour permettre aux cellules de se développer de manière hétérotrophe, et une stratégie de mixotrophie peut également être utilisée pour les microalgues autotrophes obligatoires fournissant un apport de lumière minimalement requis.
Pour évaluer la reproductibilité des données, il est fortement recommandé d’effectuer des tests en double pour toutes les plaques. Un essai ne peut être considéré comme positif que si, après soustraction du témoin négatif et des puits à blanc respectifs, l’absorbance (valeur PMI) est positive. Cette description, en présence du composé testé, est le reflet de la réaction abiotique du colorant avec le milieu.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Un soutien majeur pour ce travail a été fourni par le NYUAD Center for Genomics and Systems Biology (CGSB), financé par Tamkeen dans le cadre d’une subvention de l’Institut de recherche de l’Université de New York Abu Dhabi (73 71210 CGSB9) et des Fonds de recherche de la faculté de NYU Abu Dhabi (AD060). W.F. a également été soutenu par le programme des cent talents de l’Université du Zhejiang. Nous remercions Ashish Jaiswal pour son aide dans l’enregistrement de la vidéo. Nous remercions Hong Cai d’avoir généré les données sur le phénotype métabolique.
matériels
| Name | Company | Catalog Number | Comments |
| Ampicillin | VWR | 97062-796 | |
| Biolog assay plates [ PM01-08] | Biolog, Hayward, CA, USA | ||
| Biolog Omnilog Instrument | Biolog, Hayward, CA, USA | ||
| Chlamydomonas reinhardtii strain CC-503 | Chlamydomonas Resource Center at the University of Minnesota, USA. | Regents of the University of Minnesota | |
| Kanamycin | VWR | 0408-EU-10G | |
| Tetrazolium Violet Dye “D” | Biolog, Hayward, CA, USA | ||
| Timentin | GlaxoSmithKline Australia Pty Ltd | 42010012-2 |
Références
- Oberhardt, M. A., Palsson, B. &. #. 2. 1. 6. ;., Papin, J. A. J. M. Applications of genome-scale metabolic reconstructions. Molecular Systems Biology. 5 (1), 320 (2009).
- Schmidt, B. J., Lin-Schmidt, X., Chamberlin, A., Salehi-Ashtiani, K., Papin, J. A. Metabolic systems analysis to advance algal biotechnology. Biotechnology Journal. 5 (7), 660-670 (2010).
- Koskimaki, J. E., Blazier, A. S., Clarens, A. F., Papin, J. A. J. I. B. Computational models of algae metabolism for industrial applications. Industrial Biotechnology. 9 (4), 185-195 (2013).
- Koussa, J., Chaiboonchoe, A., Salehi-Ashtiani, K. J. B. Computational approaches for microalgal biofuel optimization: a review. BioMed Research. 2014, 649453 (2014).
- Nelson, D. R., et al. Large-scale genome sequencing reveals the driving forces of viruses in microalgal evolution. Cell Host & Microbe. 29 (2), 250-266 (2021).
- Shene, C., Asenjo, J. A., Chisti, Y. Metabolic modelling and simulation of the light and dark metabolism of Chlamydomonas reinhardtii. The Plant Journal. 96 (5), 1076-1088 (2018).
- Tibocha-Bonilla, J. D., Zuñiga, C., Godoy-Silva, R. D., Zengler, K. Advances in metabolic modeling of oleaginous microalgae. Biotechnology for Biofuels. 11 (1), 241 (2018).
- Merchant, S. S., et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions. Science. 318 (5848), 245-250 (2007).
- May, P., Christian, J. -. O., Kempa, S., Walther, D. J. B. G. ChlamyCyc: an integrative systems biology database and web-portal for Chlamydomonas reinhardtii. BMC Genomics. 10 (1), 209 (2009).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), (2011).
- de Oliveira Dal'Molin, C. G., Quek, L. -. E., Palfreyman, R. W., Nielsen, L. K. AlgaGEM - a genome-scale metabolic reconstruction of algae based on the Chlamydomonas reinhardtii genome. BMC Genomics. 12 (5), (2011).
- Chaiboonchoe, A., et al. Microalgal metabolic network model refinement through high-throughput functional metabolic profiling. Frontiers in Bioengineering and Biotechnology. 2, 68 (2014).
- Kanehisa, M., et al. Data, information, knowledge and principle: back to metabolism in KEGG. Nucleic Acids Research. 42 (1), 199-205 (2014).
- Zuñiga, C., et al. Genome-scale metabolic model for the green alga Chlorella vulgaris UTEX 395 accurately predicts phenotypes under autotrophic, heterotrophic, and mixotrophic growth conditions. Plant Physiology. 172 (1), 589-602 (2016).
- Bochner, B. R. New technologies to assess genotype-phenotype relationships. Nature Reviews Genetics. 4 (4), 309-314 (2003).
- Bochner, B. R. Global phenotypic characterization of bacteria. FEMS Microbiology Reviews. 33 (1), 191-205 (2009).
- Bochner, B. R., Gadzinski, P., Panomitros, E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Research. 11 (7), 1246-1255 (2001).
- Bartell, J. A., Yen, P., Varga, J. J., Goldberg, J. B., Papin, J. A. Comparative metabolic systems analysis of pathogenic Burkholderia. Journal of Bacteriology. 196 (2), 210-226 (2014).
- Gorman, D. S., Levine, R. J. P. o. t. N. A. o. S. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. PNAS. 54 (6), 1665-1669 (1965).
- Smith, A. C., Hussey, M. A. Gram stain protocols. American Society for Microbiology. , 1-9 (2005).
- Vaas, L. A. I., et al. opm: an R package for analysing OmniLog phenotype microarray data. Bioinformatics. 29 (14), 1823-1824 (2013).
- Vaas, L. A. I., Sikorski, J., Michael, V., Göker, M., Klenk, H. -. P. Visualization and Curve-Parameter Estimation Strategies for Efficient Exploration of Phenotype Microarray Kinetics. PLoS ONE. 7 (4), 34846 (2012).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes-a 2019 update. Nucleic Acids Research. 48 (1), 445-453 (2020).
- Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M., Tanabe, M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Research. , (2020).
- Lopez, D., Casero, D., Cokus, S. J., Merchant, S. S., Pellegrini, M. Algal Functional Annotation Tool: a web-based analysis suite to functionally interpret large gene lists using integrated annotation and expression data. BMC Bioinformatics. 12 (1), 282 (2011).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Research. 46, 633-639 (2018).
- Sahoo, S., et al. dEMBF v2. 0: An Updated Database of Enzymes for Microalgal Biofuel Feedstock. Plant and Cell Physiology. 61 (5), 1019-1024 (2020).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 14 (3), 639-702 (2019).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 1, (2019).
- Orth, J. D., Thiele, I., Palsson, B. &. #. 2. 1. 6. ;. What is flux balance analysis. Nature Biotechnology. 28 (3), 245 (2010).
- Ebrahim, A., Lerman, J. A., Palsson, B. O., Hyduke, D. R. COBRApy: constraints-based reconstruction and analysis for python. BMC Systems Biology. 7 (1), 74 (2013).
- Bölling, C., Fiehn, O. Metabolite profiling of Chlamydomonas reinhardtii under nutrient deprivation. Plant Physiology. 139 (4), 1995-2005 (2005).
- Harris, E. H. . The Chlamydomonas sourcebook: introduction to Chlamydomonas and its laboratory use. 1, (2009).
- Goodstein, D. M., et al. Phytozome: a comparative platform for green plant genomics. Nucleic Acids Research. 40, 1178-1186 (2012).
- Ghamsari, L., et al. Genome-wide functional annotation and structural verification of metabolic ORFeome of Chlamydomonas reinhardtii. BMC Genomics. 12 (1), 4 (2011).
- Manichaikul, A., et al. Metabolic network analysis integrated with transcript verification for sequenced genomes. Nature Methods. 6 (8), 589-592 (2009).
- Apweiler, R., et al. UniProt: the Universal Protein knowledgebase. Nucleic Acids Research. 32, 115-119 (2004).
- Consortium, T. U. Activities at the universal protein resource (UniProt). Nucleic Acids Research. 42 (11), 7486-7486 (2014).
- Horton, P., et al. PSORT: protein localization predictor. Nucleic Acids Research. 35, 585-587 (2007).
- Becker, S. A., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox. Nature Protocols. 2 (3), 727-738 (2007).
- Schellenberger, J., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature Protocols. 6 (9), 1290 (2011).
- Varma, A., Boesch, B. W., Palsson, B. O. Stoichiometric interpretation of Escherichia coli glucose catabolism under various oxygenation rates. Applied and Environmental Microbiology. 59 (8), 2465-2473 (1993).
- Nelson, D. R., et al. The genome and phenome of the green alga Chloroidium sp. UTEX 3007 reveal adaptive traits for desert acclimatization. eLife. , 25783 (2017).
- Chaiboonchoe, A., et al. Systems level analysis of the Chlamydomonas reinhardtii metabolic network reveals variability in evolutionary co-conservation. Molecular BioSystems. 12 (8), 2394-2407 (2016).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), 518 (2011).
- Rajendran, A., Hu, B. Mycoalgae biofilm: development of a novel platform technology using algae and fungal cultures. Biotechnology for Biofuels. 9 (1), 112 (2016).
- Hu, H., et al. Effect of cultivation mode on the production of docosahexaenoic acid by Tisochrysis lutea. AMB Express. 8 (1), 50 (2018).
- Bumbak, F., Cook, S., Zachleder, V., Hauser, S., Kovar, K. Best practices in heterotrophic high-cell-density microalgal processes: achievements, potential and possible limitations. Applied Microbiology and Biotechnology. 91 (1), 31 (2011).
- Fu, W., et al. Sugar-stimulated CO2 sequestration by the green microalga Chlorella vulgaris. Science of the Total Environment. 654, 275-283 (2019).
- Roth, M. S., et al. Regulation of oxygenic photosynthesis during trophic transitions in the green alga Chromochloris zofingiensis. The Plant Cell. , (2019).
- Villanova, V., et al. Investigating mixotrophic metabolism in the model diatom Phaeodactylum tricornutum. Philosophical Transactions of the Royal Society B: Biological Sciences. 372 (1728), 20160404 (2017).
- Cerón-García, M., et al. Mixotrophic growth of Phaeodactylum tricornutum on fructose and glycerol in fed-batch and semi-continuous modes. Bioresource Technology. 147, 569-576 (2013).
- Tuchman, N. C., Schollett, M. A., Rier, S. T., Geddes, P. . Advances in Algal Biology: A Commemoration of the Work of Rex Lowe. , 167-177 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon