Method Article
Cryo-microscopie électronique à particule unique : de l’échantillon à la structure
Dans cet article
Résumé
La détermination de la structure des complexes macromoléculaires à l’aide de cryoEM est devenue une routine pour certaines classes de protéines et de complexes. Ici, ce pipeline est résumé (préparation des échantillons, criblage, acquisition et traitement des données) et les lecteurs sont dirigés vers des ressources et des variables plus détaillées qui peuvent être modifiées dans le cas d’échantillons plus difficiles.
Résumé
La cryo-microscopie électronique (cryoEM) est une technique puissante pour la détermination de la structure des complexes macromoléculaires, via l’analyse de particules uniques (SPA). Le processus global consiste à i) vitrifier l’échantillon dans un film mince soutenu sur une grille cryoEM; ii) le criblage de l’échantillon pour évaluer la distribution des particules et la qualité de la glace; iii) si la grille est appropriée, la collecte d’un seul ensemble de données de particules pour analyse; et iv) le traitement d’images pour obtenir une carte de densité EM. Dans ce protocole, une vue d’ensemble de chacune de ces étapes est fournie, en mettant l’accent sur les variables qu’un utilisateur peut modifier au cours du flux de travail et le dépannage des problèmes courants. Le fonctionnement du microscope à distance devenant la norme dans de nombreuses installations, des variantes des protocoles d’imagerie pour aider les utilisateurs à fonctionner efficacement et à imager lorsque l’accès physique au microscope est limité seront décrites.
Introduction
CryoEM à particule unique
Pour étudier la vie au niveau moléculaire, nous devons comprendre la structure. De nombreuses techniques pour sonder la structure des protéines sont disponibles, telles que la RMN, la cristallographie aux rayons X, la spectrométrie de masse et la microscopie électronique (EM). À ce jour, la majorité des structures déposées dans la banque de données sur les protéines (PDB) ont été résolues à l’aide de la cristallographie aux rayons X. Cependant, à partir de ~2012, la cryo-microscopie électronique (cryoEM) est devenue une technique courante pour la détermination de la structure des protéines et son utilisation a considérablement augmenté. Le nombre total de cartes EM déposées dans la banque de données de microscopie électronique (EMDB) (en décembre 2020) était de 13 421 contre 1 566 en 2012 (Figure 1, www.ebi.co.uk). En 2012, le nombre de coordonnées atomiques modélisées dans les cartes de densité cryoEM, déposées dans l’APB, n’était que de 67, mais en décembre 2020, 2 309 structures avaient été déposées jusqu’à présent, soit une multiplication par 35. Cette croissance sous-jacente de la qualité et de la quantité des cartes de densité cryoEM produites, parfois appelée « révolution de résolution »1, a été causée par une coalescence d’avancées dans de multiples domaines : le développement de nouvelles caméras pour l’imagerie connues sous le nom de détecteurs d’électrons directs ; nouveaux logiciels; et des microscopes plus stables2,3,4.
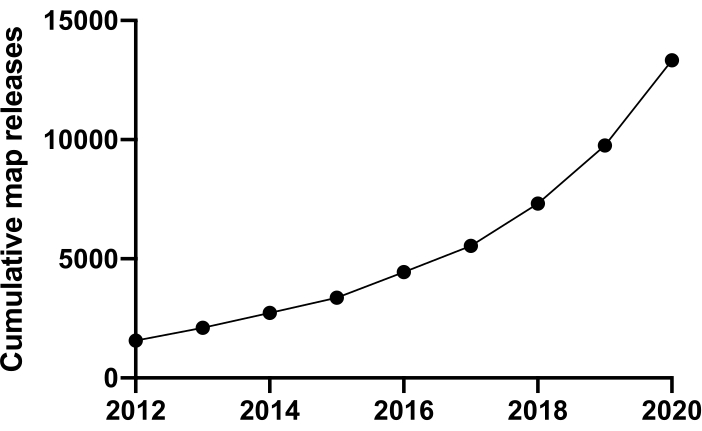
Figure 1 : Soumissions cumulatives à la BDIM de 2012 à décembre 2020. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
L’analyse de particules uniques (SPA) est un outil puissant pour générer des informations biologiques dans une grande variété de types d’échantillons en élucidant les structures à haute résolution de complexes isolés5,6, y compris les virus7,8, les protéines membranaires9,10, les assemblages hélicoïdaux11 et d’autres complexes macromoléculaires dynamiques et hétérogènes12,13, dont les tailles varient selon les ordres de grandeur (à partir de 39 kDa 14,15 à des dizaines de mégadaltons). Ici, un protocole pour un pipeline standard pour cryoEM SPA de l’échantillon à la structure est décrit.
Avant de se lancer dans ce pipeline, un échantillon purifié doit être soumis à une analyse biochimique pour évaluer ses chances de succès en aval. La préparation d’un échantillon approprié est sans doute la plus grande barrière à la SPA, en particulier pour les complexes transitoires et hétérogènes (compositionnels et conformationnels). La préparation du complexe macromoléculaire doit contenir le moins de contaminants possible, à une concentration suffisante pour produire de nombreuses particules dans chaque micrographie cryoEM, et dans une composition tampon bien adaptée à l’analyse cryoEM. Certains constituants tampons, y compris le saccharose, le glycérol et des concentrations élevées (~> 350 mM de sels, selon la taille de l’échantillon, les propriétés et d’autres constituants tampons) peuvent interférer avec le processus de vitrification ou réduire le rapport signal/bruit dans les images, entravant la détermination de la structure16.
En règle générale, au minimum, la chromatographie d’exclusion de taille (SEC) et l’analyse de gel SDS-PAGE doivent être utilisées pour évaluer la pureté de l’échantillon17,18, mais le dichroïsme circulaire, les essais fonctionnels, le SEC couplé à la diffusion de la lumière multi-angle et les tests de stabilité thermique sont tous des outils utiles pour l’analyse qualitative des préparations complexes macromoléculaires avant l’analyse cryoEM. Cependant, les résultats de ces analyses biochimiques peuvent donner peu d’informations sur l’hétérogénéité structurelle de l’échantillon et son comportement sur une grille cryoEM. Pour cette raison, la coloration négative EM est couramment utilisée comme un outil rapide, bon marché et puissant pour évaluer l’hétérogénéité de la composition et de la conformation, et donc un bon moyen de déterminer quelle fraction d’élution d’une purification est la plus prometteuse, ou de filtrer différentes compositions tampons19,20. Une fois qu’un échantillon prometteur a été identifié, nous pouvons passer au pipeline SPA cryoEM. La coloration négative ne correspond pas toujours aux résultats ultérieurs observés dans cryoEM; parfois, un échantillon semble pauvre par coloration négative, mais s’améliore lorsqu’il est vu dans la glace vitrée dans cryoEM. En revanche, les échantillons ont parfois l’air excellent pendant les étapes de coloration négative, mais nécessitent une optimisation supplémentaire significative lors de la progression vers cryoEM. Cependant, dans la majorité des cas, la coloration négative constitue une étape utile du contrôle de la qualité.
Vitrification
L’environnement hostile dans le système de vide du microscope électronique provoque à la fois une déshydratation et des dommages causés par les radiations aux échantillons biologiques non fixés21. Par conséquent, pour imager l’échantillon dans un état semblable à celui d’un natif, le spécimen biologique doit être conservé avant l’imagerie. Pour les préparations purifiées de complexes macromoléculaires, la vitrification est la méthode de choix pour permettre sa visualisation par cryoEM tout en préservant les détails atomiques du complexe. La découverte de la vitrification comme méthode de préparation des échantillons a été une avancée fondamentale en microscopie électronique de spécimens biologiques, pour laquelle Dubochet a été reconnu dans le prix Nobel de chimie 2017. La vitrification de l’échantillon consiste à créer une fine couche de solution contenant l’échantillon d’intérêt, généralement des dizaines de nm d’épaisseur, suspendue sur un support de grille cryoEM. Le film mince est ensuite congelé extrêmement rapidement dans un cryogène tel que l’éthane liquide à ~-175 °C. Le taux de congélation est d’environ 106 °C/s, assez rapide pour que de la glace amorphe ou vitreuse se forme, suspendant l’échantillon dans un film mince et solide22.
La variable initiale à considérer est le support de réseau cryoEM choisi23. Une grille EM se compose généralement d’un film de carbone amorphe avec des perforations (régulières ou irrégulières), sur une structure de support. La structure de support est généralement une grille métallique circulaire de 3,05 mm de diamètre, généralement en cuivre, mais d’autres métaux tels que l’or ou le molybdène (qui a des propriétés de dilatation thermique préférées24) peuvent être utilisés. Parfois, un support mince et continu supplémentaire est appliqué sur la grille, tel que du graphène, de l’oxyde de graphène ou une fine couche de carbone amorphe (~ 1-2 nm). Alors que les grilles cryoEM standard (le plus souvent du cuivre à mailles 400-200 avec un support en carbone perforé (trous ronds de 1,2 μm séparés par 1,3 μm (r1,2 / 1,3), ou 2 μm séparés par 2 μm de carbone (r2/2)) - bien que de nombreux modèles différents soient disponibles) ont été utilisées dans la grande majorité des structures signalées à ce jour, de nouvelles technologies de grille avec une conductivité améliorée et un mouvement réduit des échantillons ont été signalées25 . Les grilles sélectionnées sont soumises à un traitement de nettoyage par décharge incandescente/plasma pour les rendre hydrophiles et adaptées à l’application d’échantillons26.
Après la décharge luminescente, l’étape suivante est la formation de couches minces. Ce film mince est le plus souvent formé à l’aide de papier filtre pour éliminer l’excès de liquide de la grille. Bien que cela puisse être effectué manuellement, un certain nombre de dispositifs de congélation à piston sont disponibles dans le commerce, notamment le Vitrobot Mk IV (Thermo Fisher Scientific), l’EM GP II (Leica) et le CP3 (Gatan). Avec ces dispositifs, environ 3 à 5 μL d’échantillon en solution sont appliqués sur la grille EM, puis effacent l’excès de solution à l’aide de papier filtre. La grille, avec une fine pellicule en suspension à travers elle, est ensuite plongée dans de l’éthane liquide refroidi par de l’azote liquide (LN2) à ~-175 °C. Une fois gelée, la grille est maintenue à une température inférieure au point de dévitrification (-137 °C) avant et pendant l’imagerie.
Sélection d’échantillons et collecte de données
Après la vitrification d’une grille cryoEM, l’étape suivante consiste à examiner la grille pour évaluer sa qualité et déterminer si la grille est adaptée à la collecte de données à haute résolution. Une grille cryoEM idéale a de la glace vitrée (par opposition à la glace cristalline) avec l’épaisseur de glace juste suffisante pour accueillir la dimension la plus longue de l’échantillon, garantissant que la glace environnante contribue le moins possible à l’image résultante. Les particules à l’intérieur de la glace devraient avoir une taille et (si elles sont connues) compatibles avec la biochimie, et idéalement être monodispersées avec une distribution aléatoire des orientations des particules. Enfin, la grille devrait avoir suffisamment de zones de qualité suffisante pour satisfaire à la longueur de collecte de données souhaitée. Selon l’échantillon, cela peut prendre de nombreuses itérations de vitrification et de criblage jusqu’à ce que des grilles optimales soient produites. Heureusement et malheureusement, il existe une vaste gamme de variables qui peuvent être testées empiriquement pour modifier la distribution des particules sur les grilles cryoEM (examinées en 16,27). Dans ce manuscrit, des résultats représentatifs pour un projet de protéine membranaire10 sont présentés.
Une fois qu’une grille appropriée a été identifiée, la collecte de données peut se poursuivre. Plusieurs modèles de microscopes électroniques à cryotransmission pour échantillons biologiques sont optimisés pour collecter des données à haute résolution de manière automatisée. En règle générale, les données sont collectées sur des systèmes de 300 kV ou 200 kV. La collecte automatisée de données peut être réalisée à l’aide de logiciels tels que EPU (Thermo Fisher Scientific)28, Leginon29, JADAS30 et SerialEM31,32. Une collecte de données automatisée avec des détecteurs modernes donne généralement des téraoctets (To) de données brutes sur une période de 24 heures (la taille moyenne des ensembles de données est d’environ 4 To).
En raison des restrictions COVID-19 en place dans une grande partie du monde (au moment de la rédaction de cet article, décembre 2020), de nombreux établissements de microscopie sont passés à l’offre d’un accès à distance. Une fois que les grilles ont été chargées dans le chargeur automatique d’un microscope, l’acquisition de données peut être effectuée à distance.
Traitement d’images et construction de modèles
Lorsqu’une session de collecte de données peut durer généralement de 0,5 à 4 jours, le traitement ultérieur de l’image peut prendre plusieurs semaines et mois, en fonction de la disponibilité des ressources informatiques. Il est standard pour les étapes initiales de traitement d’image, à savoir la correction de mouvement et l’estimation de la fonction de transfert de contraste (CTF) qui doivent avoir lieu « à la volée » 33,34. Pour le traitement en aval, il existe une pléthore de suites logicielles disponibles. Les particules sont « cueillies » et extraites de micrographies35,36. Une fois les particules extraites, un protocole standard consisterait à traiter les particules à travers plusieurs cycles de classification (en deux dimensions (2D) et en trois dimensions (3D) et/ou en se concentrant sur des régions d’intérêt spécifiques) pour atteindre un sous-ensemble homogène de particules. Ce sous-ensemble homogène de particules est ensuite moyenné ensemble pour produire une reconstruction 3D. À ce stade, les données sont souvent corrigées davantage pour produire la carte de la plus haute qualité possible, par exemple par le raffinement CTF, les corrections de distorsion37 et le polissage bayésien38 . Le résultat de ce traitement d’image est une carte cryoEM 3D du spécimen biologique d’intérêt. La plage de résolution atteinte dans une expérience automatisée « standard » à partir d’une grille de qualité suffisante, avec des données collectées sur un système de microscope de 300 kV est généralement comprise entre 10 Å et 2 Å en fonction de la taille et de la flexibilité du complexe protéique. Avec un spécimen idéal, des résolutions d’environ 1,2 Å ont maintenant été atteintes à l’aide des workflows SPA5. Bien que ce protocole détaille les étapes vers l’obtention d’une carte de densité EM, une fois que celle-ci est en main, elle peut être interprétée plus avant en ajustant et en affinant un modèle de protéine (si la résolution est < 3,5 Å) ou en construisant de novo39. Les données associées aux expériences de détermination de la structure peuvent être déposées dans des dépôts publics en ligne, y compris des cartes de densité EM (banque de données de microscopie électronique)40, des coordonnées atomiques résultantes (banque de données sur les protéines)41 et des ensembles de données bruts (archives d’images publiques de microscopie électronique)42.
Dans ce protocole, le complexe protéique de la membrane externe RagAB (~340 kDa) de Porphyromonas gingivalis est utilisé comme exemple de complexe macromoléculaire10 (EMPIAR-10543). Pour ceux qui découvrent cryoEM, la prise en charge des échantillons via ce pipeline, de l’échantillon à la structure, est disponible, sous réserve d’un examen par les pairs, via des systèmes d’accès financés tels que iNEXT Discovery et Instruct.
Protocole
1. Vitrification du réseau
REMARQUE: Pour toutes les étapes des étapes 1 et 2, assurez-vous que tous les outils sont propres, secs et à température ambiante avant de les refroidir à la température LN2 , en utilisant du LN2 fraîchement décanté pour réduire la contamination par la glace. Dans la mesure du possible, travaillez dans un environnement à humidité contrôlée avec < humidité relative de 20 %. S’assurer que l’équipement de protection individuelle approprié et la documentation H&S sont en place avant de commencer les travaux.
- Assurez-vous que le spécimen d’intérêt est prêt pour la préparation de l’échantillon.
- Choisissez les grilles cryoEM appropriées et assurez-vous qu’elles ont été rendues hydrophiles à l’aide d’une décharge luminescente ou d’un traitement au plasma. Une grande variété de systèmes et de variantes du protocole sont disponibles, mais tous impliquent de placer les grilles dans un système de nettoyage par décharge incandescente / plasma et d’exécuter un programme qui pompera la chambre au niveau de vide souhaité, avant d’introduire un mélange de gaz / vapeur chimique ou air spécifique dans le système. Un courant électrique traverse le système, ionisant les particules de gaz et induisant la surface des grilles à rendre plus hydrophile.
- Démarrez le dispositif de congélation à piston pour la vitrification du réseau en allumant le système à l’aide de l’interrupteur d’alimentation à l’arrière et attendez que l’écran tactile se charge.
- À l’aide du stylet ou des doigts fournis, dans la console, réglez la température de fonctionnement souhaitée de la chambre (plage disponible de 4 à 60 ° C, recommandée pour la plupart des macromolécules de 4 à 6 ° C).
- Remplissez l’humidificateur avec 50 mL d’eau de laboratoire de type II à l’aide d’une seringue, via le tube en caoutchouc au fond de l’humidificateur. Assurez-vous d’enlever tout air emprisonné dans la seringue avant le remplissage. Veillez à ne pas trop remplir l’humidificateur ou de l’eau s’infiltrera dans la chambre. Une fois l’humidificateur rempli, retirez le piston de la seringue de 5 à 10 mL pour créer un joint sous vide.
- Dans console, réglez l’humidité relative souhaitée pour la chambre (la plage disponible est de 0 à 100%, une humidité de 95 à 100% est généralement utilisée). Laissez l’humidité réglée sur « off » jusqu’à ce qu’immédiatement avant la fabrication de la grille afin que la chambre ne soit pas trop humide.
- Procurez-vous la pince à épiler du dispositif de congélation et le papier filtre coupé à la bonne taille pour l’adapter aux tampons, achetés ou à l’aide d’un tampon pour couper une ouverture de la taille appropriée.
- Préparez le cryogène pour la congélation à piston.
- Placez le porte-boîte en cryo-grille métallique, la tasse en cryogène et les pattes d’araignée en métal dans le récipient de liquide de refroidissement.
- Refroidissez le récipient en remplissant la chambre extérieure de LN2. Gardez la chambre extérieure remplie pour couvrir le haut du support de boîte cryo-grille. Ajouter ~ 1 cm de LN2 supplémentaire à la coupelle de cryogène pour faciliter l’équilibrage du système à la température LN2 .
REMARQUE: L’anneau anti-contamination peut être utilisé pour limiter la condensation de l’air humide autour de la tasse de cryogène et entraîner une contamination par le cryo-liquide de refroidissement / éthane. Cela n’est généralement pas nécessaire dans un environnement à humidité contrôlée. Si vous utilisez l’anneau anti-contamination, veillez à ne pas trop remplir le récipient avec du LN2 , sinon il pourrait se renverser lorsque l’anneau est pressé dans le récipient plus tard dans le processus. - Attendez 3-5 min pour observer l’ébullition des pattes d’araignée, puis attendez encore 3 min pour vous assurer que la tasse de cryogène est suffisamment froide pour condenser le milieu de vitrification.
- Liquéfiez le cryogène (éthane liquide) dans la tasse de cryogène.
- Prenez le tuyau du cylindre d’éthane avec un tube mince et une buse pour distribuer le gaz. Une pointe de pipette P200 avec l’ouverture ouverte en coupant la pointe même à l’aide d’une lame de rasoir est idéale ici. Une ouverture plus large est nécessaire pour empêcher l’éthane de se solidifier à la pointe et de bloquer l’écoulement du gaz.
- Assurez-vous que la tasse de cryogène ne contient pas de LN2 restant, prenez la buse de gaz éthane et placez-la dans la tasse de cryogène. À l’aide du régulateur de bouteille de gaz, démarrez un faible débit et distribuez du gaz cryogénique dans la tasse de cryogène pour condenser le gaz. Gardez la pointe à partir de laquelle le gaz s’écoule directement pressé contre la paroi de la tasse de cryogène, mais déplacez-la doucement d’avant en arrière dans un mouvement de tapotement contre la surface. Réguler le débit du gaz pour permettre à un débit faible et régulier de commencer à se condenser / liquéfier de manière contrôlée dans la tasse de cryogène.
- Remplissez la tasse juste en dessous du bord de l’araignée argentée et arrêtez le flux de gaz, puis retirez soigneusement la conduite de gaz pour éviter de contaminer le LN2 environnant avec de l’éthane.
- Remplissez le récipient de liquide de refroidissement avec du LN2, en faisant très attention à ne pas en renverser dans l’éthane liquide.
- Laissez les pattes des araignées en position pendant ~ 3-5 min pour s’assurer que l’éthane liquide est équilibré à une température suffisamment froide. Le cryogène commencera à paraître trouble / légèrement opaque. Cela indique qu’il est proche de son point de congélation. À ce stade, utilisez une pince à épiler pour enlever l’araignée. Tant que le LN2 est conservé dans le récipient entourant la tasse de cryogène, l’éthane restera liquéfié et adapté à la vitrification pendant 1-2 h. Cependant, essayez de terminer la procédure le plus rapidement possible, en particulier dans les pièces non contrôlées par l’humidité, afin de réduire la contamination par la glace.
REMARQUE: Si l’araignée semble être « coincée », utilisez un objet métallique tel qu’un écrou et tenez-la contre les pattes de l’araignée pour les réchauffer légèrement, puis retirez les pattes.
- Préparez le dispositif de congélation à piston et les accessoires pour la vitrification des échantillons.
- Ajoutez des boîtes de stockage de grille au support de boîte de cryo-grille métallique et tout au long de la procédure, assurez-vous que le LN2 est maintenu rempli juste au-dessus du niveau des boîtes de grille (généralement toutes les ~ 5 min).
- Sur l’écran du dispositif de congélation à piston, dans la zone Paramètres du processus , entrez les paramètres choisis, notamment: le temps de transfert (le temps pendant lequel les coussinets du dispositif de congélation du piston se réuniront), la force (la distance entre les coussinets de transfert et la grille, ce qui modifie le gradient de formation de glace) et le total (nombre de fois que les coussinets de buvard entreront pour se rencontrer). Choisissez ces paramètres en fonction du dispositif de congélation à piston individuel et du comportement de la macromolécule. Les valeurs typiques sont une force de transfert comprise entre 0 et 5, un temps de transfert de 1 à 6 s et un total de transfert de 1. Le temps d’attente typique (temps entre le début du blot et le début du blot) et le temps de vidange (temps après le blotting avant de plonger) est de 0-2 s.
REMARQUE: Selon les préférences de l’utilisateur, des options supplémentaires dans la section Options, Divers peuvent être sélectionnées, y compris Utiliser la pédale pour passer à l’étape suivante à chaque pression, Sauter le transfert de grille (saute la dernière étape où le bras de la pince est légèrement relevé), Désactiver l’humidité pendant le processus (pendant que l’échantillon est appliqué, arrête l’humidification active de la chambre, ce qui peut rendre plus difficile la vue de la grille) et Autoraise Ethanelift (combine l’étape de la pince à épiler soulevée dans la chambre et soulevant le récipient de liquide de refroidissement - saute l’étape du récipient d’éthane Raise ). Ici, toutes ces options sont activées. - Placez le récipient de liquide de refroidissement solidement sur le bras de la plate-forme mobile sous la chambre
- Insérez du papier buvard frais sur chaque bras de buvard en vous assurant que les clips annulaires en plastique sont fixés. Chaque papier filtre permettra 16 blots (les bras font pivoter le papier buvard). Appuyez sur le bouton Réinitialiser le papier de transfert dans la section Contrôles .
- Exécutez 1 cycle complet du processus de vitrification du dispositif de congélation à piston pour vous assurer que chaque pièce mobile se comporte comme prévu.
- Appuyez sur (ou utilisez la pédale) pour placer une nouvelle grille, puis Démarrer le processus, puis Processus , puis Continuer. À ce stade, surveillez pour vous assurer que les bras de buvard sont en contact les uns avec les autres comme prévu.
- Allumez l’humidificateur. De la vapeur d’eau sera produite (tant que l’humidité réglée est plus élevée que celle actuellement dans la chambre).
- Le spécimen d’intérêt peut maintenant être vitrifié. Utilisez la pédale ou Placez une nouvelle grille et la tige plongeante descendra hors de la chambre permettant aux pinces d’être fixées dans le support.
- À l’aide de la pince à épiler le dispositif de congélation à piston, ramassez la grille cryoEM à décharge lumineuse / nettoyée au plasma souhaitée, en prenant soin de noter quel côté est le bon côté à utiliser pour l’application de l’échantillon selon le fabricant de la grille. Ramassez la grille par la jante, en prenant soin d’éviter tout contact excessif / inutile avec la pince à épiler car cela endommagerait le support. Fixez la grille dans la pince à épiler en déplaçant le clip noir vers la partie striée de la pince à épiler. La grille doit être solidement maintenue, mais le clip ne doit pas être trop bas car il entrera en contact avec les coussinets de buvard, ce qui entraînera un buvard irreproductible et, plus tard, la pince à épiler devra être maintenue en dessous de ce point lors de la libération du clip.
- Placez la pince à épiler du dispositif de congélation par piston qui maintient la grille cryoEM sur le bras pneumatique avec le bon côté face à votre main dominante. La conception de la pince à épiler et de la chambre du dispositif de congélation à piston est telle que l’échantillon peut être appliqué à travers le côté droit ou gauche de la chambre, en fonction de la main de l’utilisateur.
REMARQUE: L’application de l’échantillon sur différents côtés avec les mêmes paramètres de buvard donne rarement des résultats comparables, de sorte que les chercheurs gauchers peuvent avoir besoin d’ajuster leurs paramètres de buvard indépendamment de leurs collègues droitiers. - Appuyez sur Démarrer le processus et la grille maintenue dans la pince à épiler sera prise dans la chambre et le récipient de liquide de refroidissement sera soulevé.
- Appuyez sur Processus et la pince à épiler déplacera la grille à l’endroit où une pipette peut être utilisée pour appliquer l’échantillon à la grille. Ouvrez l’orifice latéral face au bon côté de la grille et appliquez l’échantillon par pipetage, en vous assurant que la pointe de la pipette ne touche pas la grille car cela pourrait endommager le support de la grille / plier la grille, mais distribuez le liquide suffisamment près pour que la gouttelette se distribue sur la grille. En règle générale, 3 à 5 μL sont appliqués.
- Appuyez sur Continuer et les paramètres prédéfinis par l’utilisateur épongeront la grille, puis plongeront la pince à épiler avec la grille montée dans la tasse de liquide de refroidissement pour la vitrification de l’échantillon. La pince à épiler descendra en conjonction avec le bras tenant le récipient de liquide de refroidissement et le liquide de refroidissement, gardant la grille immergée dans le cryogène.
- Transférez la grille de la tasse de cryogène à la boîte de stockage de la grille immergée dans LN2.
- Détachez la pince à épiler du bras de la pince à épiler, en prenant grand soin de ne pas entrer en contact avec la grille vitrifiée avec les côtés de la coupelle de cryogène. Ajustez la poignée pour que les pinces à épiler soient maintenues confortablement. Aussi rapidement et soigneusement que possible, déplacez la grille du cryogène vers le LN2. D’une main, tenez la pince à épiler fermée à l’aide de vos doigts et de l’autre main, faites glisser le clip noir vers le haut, en maintenant la pince à épiler fermée. Réajustez la poignée et manipulez la grille dans la boîte de stockage de la grille.
- Répétez les étapes 1.10-1.19 jusqu’à ce que toutes les grilles soient faites (une session typique impliquera de faire 4-12 grilles). Stockez toutes les boîtes de stockage de grille contenant des grilles dans LN2 dewar jusqu’aux étapes suivantes.
2. Grilles d’écrêtage pour le chargement dans un microscope à chargement automatique
- Clipsez les grilles dans un assemblage de grille automatique selon le protocole décrit précédemment 28.
3. Connexion à distance sécurisée aux microscopes
REMARQUE: Avec les contrôles COVID-19 au moment de la rédaction du présent rapport, mais aussi avec les préoccupations environnementales associées aux voyages internationaux, de plus en plus d’installations de microscopie offrent des services où l’utilisateur opère à distance. La méthode de mise en œuvre pour cela variera en fonction de la configuration informatique locale de chaque installation et des besoins de sa communauté d’utilisateurs internes et externes. Ici, le processus d’accès à distance aux cryoEM à eBIC et de contrôle du microscope via le logiciel EPU est décrit.
- Connectez-vous à distance à cryoEM. L’ouverture de session à distance est assurée par le logiciel NoMachine pour accéder au PC de support du microscope et est configurée pour autoriser uniquement l’accès aux utilisateurs enregistrés lors d’une visite via les informations d’identification d’ouverture de session FedID des utilisateurs. L’accès reste actif uniquement pendant la durée de la session.
- Ouvrez NoMachine et démarrez une nouvelle connexion NX pour nx-cloud.diamond.ac.uk avec l’authentification par mot de passe.
- Ouvrez la connexion et connectez-vous avec le nom d’utilisateur fedid@fed.cclrc.ac.uk et le mot de passe FedID. Double-cliquez sur l’icône correspondant au microscope concerné parmi les options disponibles pour ouvrir une connexion au PC de support concerné.
- Entrez le nom d’utilisateur clrc\FedID et le mot de passe sur l’écran d’ouverture de session Windows.
- Ouvrez le logiciel TeamViewer à partir de l’icône du bureau et connectez-vous à PartnerID: TEM avec le mot de passe fourni. Cela établit la connexion entre le PC de support et le PC TEM. Le bouton Moniteur suivant du ruban TeamViewer permet de basculer entre l’interface utilisateur du microscope et la fenêtre EPU.
- Les fonctions du microscope peuvent ensuite être contrôlées par les utilisateurs directement via l’interface EPU.
4. Chargement des échantillons dans un microscope à chargement automatique et dépistage de la glace et de la qualité des échantillons
REMARQUE: Dans cette section, un microscope avec un chargeur automatique et un logiciel EPU est utilisé pour le criblage des échantillons, mais cela peut être réalisé à l’aide d’autres logiciels et d’un système d’entrée latérale et de cryoEM d’autres fabricants.
- Chargez les grilles découpées dans le chargeur automatique du microscope comme décrit précédemment28.
- Dans l’onglet Chargeur automatique de l’interface utilisateur du microscope, appuyez sur la touche Options à l’aide de la flèche et appuyez sur le bouton Inventaire . Cela vérifiera séquentiellement chaque position dans la cassette pour déterminer si une cartouche est présente. Les emplacements occupés seront étiquetés en bleu. Si tous les emplacements occupés ont été mappés, appuyez à nouveau sur le bouton Inventaire pour vous arrêter après la position actuelle, sinon laissez-les en cours d’exécution jusqu’à ce que tous les emplacements occupés aient été mappés. Étiquetez tous les emplacements occupés avec les détails de l’échantillon dans les cases fournies.
- Mettez en surbrillance la grille à transférer dans la colonne du microscope et cliquez sur Charger. L’étiquette de la fente passera du bleu au jaune une fois que la grille aura été chargée avec succès sur la scène. Passez à l’écran des grilles.
- Ouvrez le logiciel EPU. Sur la page Préparation , sélectionnez Optique et paramètres d’acquisition , puis sélectionnez le paramètre prédéfini Atlas dans le menu déroulant. Choisissez les préréglages de réglage de faisceau appropriés (par exemple, mag nominal 64x, taille de spot 5, Microsonde, avec une zone éclairée dans la plage parallèle pour le détecteur Falcon - pour plus d’informations, choisissez des préréglages de réglage de faisceau voir28). Appuyez sur Définir pour transmettre les paramètres au microscope.
- Appuyez sur Ouvrir les vannes à colonne et insérez le FluScreen. Vérifiez qu’un faisceau est visible et suffisamment étalé et centré pour couvrir le détecteur. Si nécessaire, accédez à une région plus mince de la grille à l’aide du joystick ou du menu de scène pour contrôler les mouvements de la scène en X et Y.
- Soulevez le FluScreen et prenez une image à l’aide du bouton Aperçu dans EPU. Sur la base de l’image acquise, la dose peut être augmentée en passant à une taille de point de nombre inférieure, et vice versa.
- Dans EPU, accédez à la page Atlas et appuyez sur Nouvelle session. Sélectionnez le format d’image MRC et entrez un nom de dossier et un emplacement appropriés pour enregistrer la session de filtrage, puis cliquez sur Appliquer.
- Sélectionnez Filtrage dans le menu de gauche. Cochez les cases à côté de chaque grille pour obtenir un montage d’atlas. Commencez la session de dépistage dans EPU. Un atlas sera acquis pour chaque grille vérifiée, avec un certain nombre de carrés de grille disponibles répertoriés à la fin. Chaque atlas peut être consulté en le mettant en évidence sur la page de criblage, avec un balisage montrant des carrés de grille avec une épaisseur de glace prévue similaire regroupée par couleur.
- Une fois terminés, examinez les atlas recueillis et identifiez les grilles appropriées pour évaluer la qualité des échantillons à des grossissements plus élevés (c.-à-d. celles avec un nombre approprié de carrés de grille qui ne sont ni secs ni obscurcis par une glace épaisse). Mettez en surbrillance la grille choisie dans le menu de filtrage EPU et cliquez sur Charger l’exemple.
- Utilisez les préréglages de réglage de faisceau (voir 28 pour l’explication des préréglages de réglage de faisceau souhaités pour chaque étape) et la fonction de prévisualisation pour examiner plus en détail les carrés de grille souhaités.
- Dans le menu de sélection de l’Atlas, sélectionnez la grille actuellement chargée et déplacez la scène vers un carré de grille contenant des trous remplis en cliquant avec le bouton droit de la souris sur l’emplacement souhaité sur l’image de la grille et en sélectionnant Déplacer vers le carré de la grille.
- Revenez à la page EPU, Préparation et sélectionnez le préréglage GridSquare .
- Ouvrez la page EPU, Fonctions automatiques et exécutez Auto-eucentric by stage tilt avec le préréglage GridSquare pour déplacer l’échantillon à la hauteur eucentrique.
REMARQUE: L’inclinaison auto-eucentrique par faisceau est également disponible, ce qui est plus rapide mais généralement moins précis que l’inclinaison auto-eucentrique par étage. - Dans EPU, Préparation, prenez une nouvelle image GridSquare Preview . Notez les différentes valeurs de gris entre les différents trous indiquant différentes épaisseurs de glace. Déplacez la scène sur un trou en cliquant avec le bouton droit de la souris > déplacer la scène ici. Sélectionnez le préréglage Trou/Hauteur eucentrique et Aperçu.
REMARQUE: Selon le poids moléculaire et la forme de la particule d’intérêt, il peut être possible de l’identifier au grossissement trou/hauteur eucentrique. - Sélectionnez le préréglage Acquisition de données et définissez un grossissement qui permet d’identifier facilement les particules (correspondant à un échantillonnage d’objet généralement <2 Å/pixel). Réglez le décalage de mise au point sur ~-3 à -5 μm avec une dose d’électrons d’exposition de ~40-80 e-/ Å2.
- Itérez à travers les étapes de la section 4.4 pour évaluer une gamme d’épaisseurs de glace pour la distribution, l’orientation et la contamination des particules à travers le réseau. La distribution des particules peut varier près des bords par rapport au centre du trou et il est donc important de surveiller différents endroits avec le trou.
- Filtrez toutes les grilles qui sont prometteuses à partir d’atlas comme ayant suffisamment de carrés de grille. Conservez-les dans le microscope et procédez à l’acquisition de données à l’aide de l’EPU, ou déchargez les échantillons du microscope et stockez-les sous LN2 jusqu’à ce que la collecte des données soit planifiée.
5. Collecte de données cryoEM à particule unique (en mettant l’accent sur le fonctionnement à distance)
REMARQUE: Un protocole détaillé pour l’acquisition de données avec EPU est décrit dans le manuel du fabricant et ailleurs28. Ici, les modifications de ce protocole pour le fonctionnement à distance (à savoir la réduction de l’utilisation des panneaux manuels pour effectuer des tâches et l’utilisation d’alternatives logicielles) sont mises en évidence.
- À moins qu’un n’ait déjà été collecté au cours de la session, collectez un Atlas pour la grille.
- Définissez chacun des préréglages de réglage du faisceau en fonction des besoins expérimentaux du projet.
- Effectuez des étalonnages de décalage d’image28.
- Configurez la session EPU.
- Dans EPU, sélectionnez la page EPU puis Configuration de la session, sélectionnez Nouvelle session puis Nouveau dans les préférences.
- Sélectionnez Nouvelle session , une fenêtre contextuelle apparaîtra fournissant une option pour utiliser les paramètres précédents. Yes chargera automatiquement les paramètres de l’EPU précédent (c’est-à-dire le support d’échantillon, la plage de mise au point, les paramètres de mise au point automatique, le type de grille) dans la session EPU en cours. La sélection de Nouveau dans les préférences permet à l’utilisateur de choisir un fichier avec des préférences enregistrées (par exemple, plage de mise au point, paramètres de mise au point automatique, type de grille) et ces informations seront préchargées dans EPU.
- Remplissez le nom de la session avec quelque chose d’informatif. L’installation locale peut suggérer une convention de dénomination.
- Dans Type, sélectionnez Manuel.
- Pour le mode Acquisition, sélectionnez un centrage précis des trous ou une acquisition plus rapide.
- Dans Format d’image, sélectionnez le format souhaité.
- Sélectionnez un dossier de stockage approprié et EPU créera un répertoire avec le nom de la session.
- Sélectionnez le porte-spécimen approprié en fonction du type de grille et de l’espacement des trous utilisés (par exemple, Quantifoil 1.2/1.3) et appuyez sur Appliquer. Ce protocole décrit le processus de génération d’un modèle pour un tableau régulier de trous
- Sélectionnez un carré de grille initial et définissez un modèle d’acquisition.
- Aller à la sélection carré, si tous les carrés sont verts, cliquez sur désélectionner tout en haut à gauche.
- Ouvrir les vignettes (clic droit > ouvrir la vignette). Sélectionnez un carré (cliquez avec le bouton droit > ajouter, cliquez avec le bouton droit de la souris > Déplacer la scène vers le carré de la grille).
- Accédez à Sélection des trous et appuyez sur Auto Eucentric. Attendez que cela soit terminé et qu’une image Grid Square ait été prise. Si la fonction automatique échoue, c’est peut-être parce que la hauteur est considérablement réduite; si c’est le cas, il peut être ajusté manuellement à l’aide du fluScreen au grossissement Grid Square.
- Mesurez la taille des trous. Déplacez et ajustez les cercles jaunes afin qu’ils soient au-dessus des trous avec une taille et un espacement corrects.
- Appuyez sur Find holes (Trouver des trous). Vérifiez que les trous ont été trouvés correctement. Si ce n’est pas le cas, changez la taille du trou et retrouvez les trous. Répétez cette opération jusqu’à ce qu’il trouve le trou correctement. S’il échoue systématiquement, envisagez de passer à un nombre inférieur (plus lumineux) de taille de point au grossissement carré de la grille.
- Utilisez l’histogramme filtrer la qualité de la glace sur la droite pour ajuster la sélection des trous. Cela peut être utile pour exclure les zones avec de la glace épaisse et de la glace mince. On s’en souviendra pour les futurs carrés de grille sélectionnés au cours de cette session.
- Optimisez la sélection des trous avec les outils du menu Sélectionner en haut. Par exemple, cliquez sur Supprimer les trous près de la barre de grille.
- Accédez à Définition du modèle et appuyez sur Acquérir.
- Cliquez sur Rechercher et centrer le trou. Il y aura maintenant une image d’un trou avec un cercle jaune autour du trou.
REMARQUE: S’il a du mal à trouver le trou, insérez l’ouverture de l’objectif. S’il ne trouve toujours pas le trou, essayez d’augmenter le temps d’exposition pour le trou / hauteur eucentrique prédéfinie ou d’augmenter la mise au point pour ce préréglage ou bin l’image. Un changement de mise au point important peut modifier l’alignement du décalage de l’image. - Modifiez les temps de décalage de délai après le décalage d’étape et de délai après le décalage d’image à 1-5 s.
- Vérifiez que la valeur de décalage maximal de l’image (si l’option est disponible) est comme vous le souhaitez. Si une collection de décalage d’image sans aberration est utilisée, cette valeur est définie dans le fichier de configuration EPU, sinon 5 μm est une valeur standard.
- Cliquez sur Ajouter une zone d’acquisition, puis cliquez n’importe où sur l’image. Déplacez la zone d’acquisition à l’endroit souhaité (c.-à-d. au bord d’un trou) afin que les zones d’acquisition ne soient pas doublement exposées avec le faisceau (le carré dans le cercle vert représente la zone du détecteur, le cercle vert est le diamètre du faisceau).
- En haut à droite, ajoutez la plage de défocalisation. Ajoutez ensuite d’autres zones d’acquisition. Une liste de défocalisation typique pour un projet de protéine membranaire est de -0,8 à -3 μm de défocalisation.
- Cliquez sur Ajouter une zone de mise au point automatique et cliquez n’importe où sur l’image. Déplacez la zone de mise au point automatique vers le carbone entourant un trou. La pratique standard consiste à effectuer la mise au point automatique après centrage lors de l’utilisation d’AFIS, ou tous les 5 à 15 μm, en fonction de la variation de hauteur z sur le carré.
- Cliquez sur Ajouter une zone de mesure de dérive, la mesure de dérive effectuée une fois par carré de grille, avec un seuil défini de 0,05 nm/s est un paramètre standard. La zone de mesure de dérive peut (et c’est une bonne idée de) se chevaucher directement avec la zone de mise au point automatique. Assurez-vous qu’aucune zone de dérive ou de mise au point automatique ne chevauche une zone d’acquisition.
REMARQUE : Le modèle peut être vérifié à l’aide de la fonction d’exécution du modèle. C’est une bonne idée pour voir si les zones d’acquisition ont besoin d’être déplacées (par exemple, trop / pas assez de carbone dans les images), mais ce n’est pas nécessaire. - Revenez à la sélection Carré et, sur la grille, sélectionnez les carrés à acquérir. Utilisez le nombre de zones d’acquisition et le taux d’acquisition de données attendu (à partir de l’installation en fonction des détecteurs et de la configuration expérimentale) pour prédire le nombre de zones d’acquisition nécessaires.
- Lorsque tous les carrés souhaités sont sélectionnés, appuyez sur Préparer tous les carrés.
- Une fois chaque carré collecté, naviguez entre les carrés de la grille et affinez les trous à l’aide du pinceau de sélection.
- Déplacez-vous vers un emplacement de scène sur l’échantillon et utilisez les fonctions automatiques pour définir la hauteur eucentrique. Effectuez des alignements de microscope comme décrit précédemment28, mais au lieu d’effectuer manuellement un alignement sans coma et de corriger l’astigmatisme objectif, utilisez des outils d’alignement dans le logiciel. En bref, réglez les conditions du faisceau d’acquisition, assurez-vous que l’ouverture de l’objectif (OA) est retirée et que l’étage est positionné sur une zone stable du faisceau de l’échantillon à hauteur eucentrique. Effectuez un alignement sans coma dans les fonctions automatiques avant de réinsérer et de centrer l’OA et de corriger l’astigmatisme de l’objectif avec l’EPU. Assurez-vous que les deux alignements convergent vers des valeurs appropriées (<150 nm de coma et un astigmatisme proche de zéro.
- Avant de commencer l’acquisition automatisée, assurez-vous que la turbopompe du chargeur automatique est éteinte et que l’ouverture de l’objectif est insérée.
- Dans Acquisition automatisée, appuyez sur Démarrer pour lancer l’acquisition automatisée de données.
6. Traitement d’image pour obtenir une carte de densité EM
REMARQUE: La majorité des installations cryoEM offrent un prétraitement des films de micrographie « à la volée ». Il existe une grande variété de progiciels et d’approches disponibles pour cela, y compris les pipelines RELION28,33, cryoSPARC43, Scipion34 et WarpEM44. Un pipeline basé sur RELION est décrit ici et il est supposé que l’utilisateur a déplacé les films de micrographie vers un emplacement de stockage approprié avec accès aux ressources informatiques. Un aperçu du processus et des résultats représentatifs d’un projet de protéine membranaire sont fournis, une description détaillée et un tutoriel étape par étape peuvent être trouvés sur la page d’accueil de RELION: https://www3.mrc-lmb.cam.ac.uk/relion.
- Effectuer une analyse « à la volée » de la correction du mouvement de la micrographie et de l’estimation CTF. Démarrez RELION dans le répertoire du projet. Planifiez les tâches d’importation, de correction de mouvement et d’estimation CTF en boucle de manière à ce qu’elles soient simultanées avec la collecte et le transfert de données. Un script d’analyse micrographique28 fournit un retour visuel en temps réel sur l’astigmatisme et les valeurs de défocalisage estimées (voir les résultats représentatifs).
- Choisissez des particules à partir de micrographies prétraitées. Il existe un certain nombre de logiciels automatisés de prélèvement de particules parmi lesquels choisir. Des options de sélection sans référence et basées sur des modèles sont disponibles dans l’onglet Sélection automatique de RELION37. D’autres programmes peuvent être utilisés pour différentes étapes, par exemple en utilisant crYOLO pour la sélection de particules35.
- Extraire les particules des micrographies corrigées par le CTF.
REMARQUE: Pour réduire le temps de calcul requis pour les étapes précoces de « nettoyage », de traitement, de mise à l’échelle / bac des particules lors de l’extraction. Vous trouverez des détails sur la façon d’exécuter le travail d’extraction dans le didacticiel RELION 3.1. Pour ce projet, les particules ont d’abord été regroupées par un facteur 2. - Effectuez une moyenne de classe 2D. La classification sur 100 à 200 classes fonctionne bien pour la plupart des ensembles de données contenant ≥ 100 000 particules. Il n’est pas recommandé d’utiliser beaucoup plus de 200 classes ou moins de 50 classes, même lorsque les ensembles de données sont petits, sauf si l’échantillon a une symétrie élevée (c’est-à-dire un virus icosaédrique), auquel cas moins de 50 classes peuvent encore donner un bon résultat. Réglez le diamètre du masque assez grand pour accueillir la dimension la plus longue de la particule, mais suffisamment serré pour exclure toute particule voisine (cela peut nécessiter des essais et des erreurs).
- Sélectionnez les bonnes classes (c’est-à-dire celles avec des détails structurels) à l’aide de la tâche de sélection de sous-ensembles. Des exemples de bonnes et de mauvaises moyennes de classe 2D peuvent être trouvés dans la section des résultats représentatifs.
- Générez un modèle initial de novo à partir des données à l’aide de la tâche de modèle initial 3D dans RELION.
REMARQUE: Les piles de particules moins propres peuvent bénéficier d’un raffinement SGD ab initio (descente de gradient stochastique) multi-références, car cela offre une occasion supplémentaire de trier les particules indésirables / sous-optimales. Sélectionnez un diamètre de masque qui peut accueillir la particule d’intérêt et laissez les valeurs par défaut des champs dans l’onglet 'SGD' car ceux-ci fonctionnent généralement bien. Assurez-vous que le modèle initial semble raisonnable dans Chimera (ou un autre programme de visualisation approprié) (voir les résultats représentatifs). - Effectuez une classification 3D pour traiter l’hétérogénéité des données en utilisant la sortie de l’étape 6.6 comme modèle de référence. Évaluez les cartes résultantes dans Chimera. Traiter indépendamment les empilements de particules correspondant à des états conformationnels uniques. Utilisez la tâche de sélection de sous-ensembles pour sélectionner une ou plusieurs classes d’intérêt et générer des fichiers particles.star pour les piles de particules associées.
- Exécutez l’auto-raffinement 3D. Utilisez les moyennes de classe 3D obtenues à l’étape précédente comme références pour affiner leurs piles de particules correspondantes. Si la résolution du raffinement se rapproche de la limite de Nyquist des données, réextraitez les particules sans réduction d’échelle. Après la réextraction, répétez le travail d’affinage automatique 3D avec la pile de particules non liées. Dans ce cas, les modèles de référence 3D doivent être redimensionnés de manière à ce que les tailles de pixels et de boîtes soient cohérentes avec celles des images de particules réextraitées. Utilisez l’outil de ligne de commande relion_image_handler pour effectuer cette opération.
- Utilisez la symétrie dans le raffinement si nécessaire. Si une carte reconstruite possède une symétrie, alignez-la sur l’axe de symétrie approprié à l’aide de l’outil de ligne de commande relion_align_symmetry. Utilisez la carte alignée résultante comme référence dans une nouvelle tâche d’affinement automatique 3D avec l’opérateur de symétrie approprié spécifié dans l’onglet référence.
- Affinez les cartes à partir de l’auto-raffinement 3D. Cela se fait à l’aide de la tâche de post-traitement dans RELION, mais un masque approprié doit d’abord être créé à partir de la carte affinée. Les étapes de création et de post-traitement du masque sont détaillées dans le tutoriel RELION (voir également les résultats représentatifs).
REMARQUE: La résolution de nombreuses reconstructions peut être encore améliorée en utilisant les fonctionnalités de polissage bayésien et de raffinement CTF dans RELION. Utilisez le type de travail de raffinement CTF pour estimer et corriger les aberrations d’ordre supérieur (inclinaison du faisceau, aberrations du trèfle et aberrations du 4e ordre) et, en tant que tâches distinctes, le grossissement anisotrope et le défocalisage par particule. Ensuite, utilisez le travail de polissage bayésien (entraîné ou avec des valeurs par défaut) pour traiter le mouvement induit par faisceau sur une base par particule. Comme abordé dans le tutoriel RELION 3.1, ces travaux bénéficieront probablement d’une approche itérative (raffinement CTF → polissage bayésien → auto-raffinement 3D → post-traitement →... loop) puisque les deux bénéficient de modèles à plus haute résolution. - Corrigez la maniabilité des cartes de densité EM si nécessaire. Examinez les cartes pour déterminer si la maniabilité est correcte, soit en essayant de s’adapter à un modèle atomique existant, soit en évaluant la maniabilité des régions hélicoïdales alpha. Si nécessaire, retournez la carte le long de l’axe z dans UCSF Chimera45 à l’aide de la commande 'vop zflip'.
Résultats
Lors du criblage, les grilles peuvent être jetées au stade de l’atlas, où les caractéristiques résolues à faible grossissement marquent la grille comme ne convenant pas à l’acquisition de données. Par exemple, si une grille a subi des dommages mécaniques importants avec la majorité des carrés de grille brisés (figure 2A), ou lorsque la grille semble être « sèche », sans glace vitreuse (figure 2B). Ces grilles sont généralement identifiables car les bords des carrés de la grille semblent nets et distincts. Sur la majorité des grilles réalisées à l’aide du dispositif de congélation à plongeon, un gradient de glace est observé (Figure 2C,D). La distribution des particules, selon le spécimen d’intérêt, peut varier considérablement avec l’épaisseur de la glace et il est donc recommandé de filtrer une gamme de carrés de grille pour évaluer la distribution des particules. Des outils ont été mis en œuvre dans le logiciel EPU au cours de l’étape de criblage de l’atlas pour aider l’utilisateur à identifier les carrés de grille d’épaisseur de glace similaire ou différente, ce qui peut être particulièrement utile pour les utilisateurs qui débutent dans l’examen des grilles cryoEM (Figure 2E, F).

Figure 2 : Exemple de montages « atlas » à faible grossissement provenant de séances de projection. A) Une grille qui a subi des dommages importants avec la majorité des carrés de grille cassés - impropre à la collecte. B) Une grille sèche sans glace vitreuse - impropre à la collecte. C) Une grille démontrant un gradient de glace avec ~ 50% de la grille utilisable. D) Un gradient de glace avec ~ 33% de la grille utilisable. C et D conviennent tous deux à la collecte de données si les carrés de grille utilisables ont une épaisseur de glace appropriée pour la collecte et s’il y a suffisamment de zones d’acquisition pour satisfaire à la durée minimale d’une collecte (par exemple, 24 h) E) Un exemple d’atlas avec une gamme d’épaisseurs de glace. F) Le même atlas présenté en E mais avec des carrés de grille catégorisés et colorés par logiciel EPU en fonction de l’épaisseur de la glace. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Lors du dépistage de la distribution des particules, assurez-vous que les paramètres d’imagerie, tels que le grossissement et la dose totale d’électrons, sont similaires à ceux qui devraient être utilisés lors de l’acquisition de données afin de fournir une image précise des résultats attendus. Au cours du criblage, une distribution idéale des particules est monodispersée avec une gamme d’orientations de particules visibles (selon le spécimen et les connaissances existantes sur la morphologie de la particule, cela peut être difficile à déterminer) (Figure 3A). La glace doit être aussi mince que possible tout en accueillant les particules de plus grande dimension, si la glace est trop mince, elle peut fondre lorsqu’elle est éclairée par le faisceau d’électrons. Cela provoque un mouvement excessif dans la micrographie, et les zones qui présentent cette caractéristique doivent être évitées (Figure 3B). D’après l’expérience collective, cet effet est le plus souvent observé lorsqu’il y a du détergent dans le tampon. Cela peut entraîner une glace très mince au centre du trou et donc des particules peuvent être physiquement exclues et forcées vers le bord. Cet effet est observé à la figure 3C, mais dans ce cas, il ne s’agit pas d’un exemple extrême et ces images contribueraient tout de même utilement à un ensemble de données. Enfin, la glace doit être vitreuse; exclure toutes les zones de la grille (ou des grilles) où la majorité ou la totalité des images prises montrent de la glace cristalline (figure 3D) de l’acquisition de données. Souvent, de la glace non vitreuse est observée au bord des carrés de grille. Les lecteurs sont invités à consulter des examens détaillés des variables qui peuvent être modifiées au cours de la vitrification de la grille16 et des descriptions du comportement des particules dans l’environnement à couche mince46,47 pour plus d’informations.

Figure 3 : Micrographies représentatives montrant différentes distributions de particules. A) Une distribution « idéale » de particules monodispersées adoptant une gamme d’orientations. B) Glace trop mince au milieu du trou qu’elle déforme lors de l’exposition au faisceau d’électrons provoquant un mouvement excessif dans la micrographie. Cet effet est le plus souvent observé lorsque du détergent est présent dans le tampon C) Lorsque la glace est plus mince au centre du trou, cela exclut physiquement les particules du centre, provoquant un encombrement des particules vers le bord du trou. Dans ce cas, il n’est pas assez extrême pour empêcher ces images d’être utiles, mais cela suggère qu’il vaut la peine de filtrer des zones légèrement plus épaisses. D) La glace n’est pas vitrée, les données ne doivent pas être collectées sur les zones qui ressemblent à cet exemple de micrographie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Le traitement d’image à la volée peut aider à détecter les erreurs et les problèmes d’acquisition de données et est donc toujours recommandé dans la mesure du possible. Par exemple, un mouvement excessif dans les micrographies peut indiquer que la turbopompe du chargeur automatique est active ou que des données sont collectées sur un carré de grille fissuré où la glace se déplace de manière significative dans le faisceau d’électrons, ce qui indique que le carré de la grille doit être ignoré. À la volée, l’estimation CTF peut révéler des circonstances où un point focal positif (plutôt que de se déconcentrer) est appliqué (où des programmes d’estimation CTF et des paramètres pour trouver ces points sont utilisés), et déterminer le déphasage où une plaque de phase Volta48 est utilisée. Les pipelines de traitement d’images à la volée incluent souvent un résumé graphique des données (Figure 4A) pour permettre aux utilisateurs d’évaluer rapidement la qualité des micrographes et de décider si des modifications de collecte de données sont nécessaires.
La sélection des particules à partir de micrographies, tout en évitant les « faux positifs » tels que la contamination ou le film de support de la grille, peut nécessiter une optimisation. Cependant, les sélecteurs de particules tels que crYOLO fonctionnent souvent suffisamment bien en utilisant les paramètres par défaut pour un « premier passage » des données (Figure 4B), permettant une progression vers la moyenne des classes 2D où il peut être plus facile d’évaluer la qualité des données et la probabilité de succès en aval. Pour la plupart des projets, la classification 2D d’environ > 10k particules devrait commencer à révéler des classes qui ont des détails de structure secondaire. Pour passer à la 3D, l’étape de classification 2D doit généralement révéler des classes représentant une gamme d’orientations de particules. Si une orientation privilégiée est révélée, d’autres itérations de préparation de l’échantillon16 ou une acquisition de données supplémentaire avec l’échantillon incliné peuvent être nécessaires49. Toutes les classes qui montrent des détails de structure secondaire doivent être choisies pour passer à l’analyse 3D, tandis que les particules « indésirables » sont éliminées (Figure 4C).
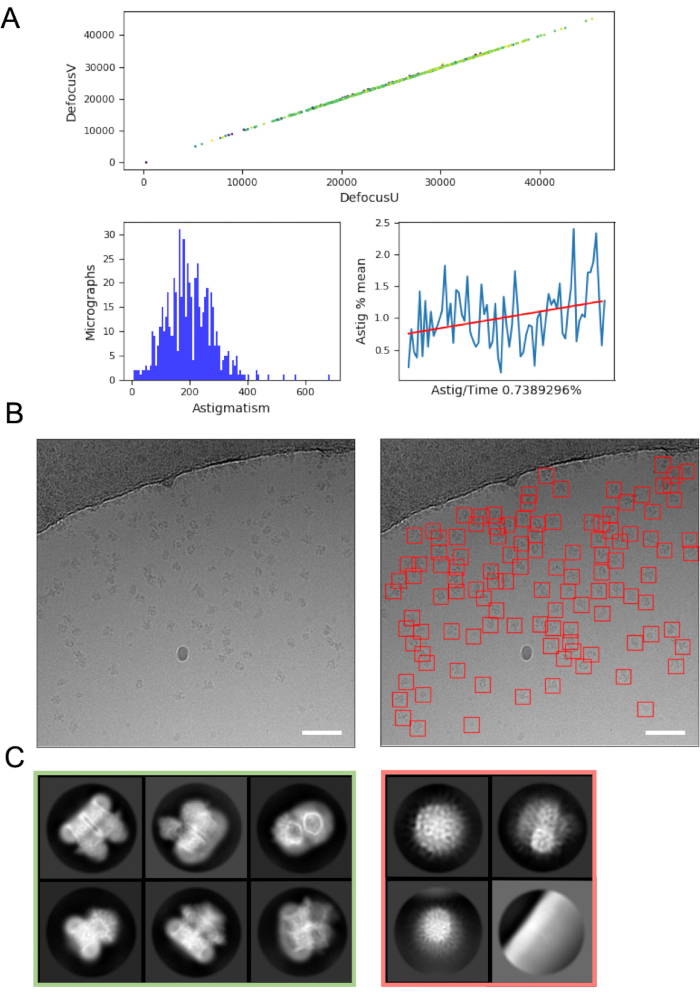
Figure 4 : Étapes initiales de traitement de l’image. A) Sortie à partir d’un script de traitement d’image « à la volée ». B) Exemple de micrographie (à gauche) avec des particules correctement choisies automatiquement identifiées à l’aide du modèle général crYOLO (à droite, avec des particules délimitées par des carrés rouges) Les barres d’échelle (blanches) sont de 50 nm. C) Résultats de la classification 2D montrant les classes qui ont été rejetées dans le carré rouge, et les classes à partir desquelles les particules ont été sélectionnées pour un traitement ultérieur en vert. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Un petit sous-ensemble de particules peut être utilisé pour générer un modèle initial (Figure 5A). Ce modèle initial peut ensuite être utilisé comme modèle de départ dans la classification et le raffinement 3D. Dans le cas de RagAB, l’ensemble de données contenait trois conformateurs distincts qui peuvent être séparés lors de la classification 3D (Figure 5B). Les particules contribuant à chacune de ces classes peuvent ensuite être traitées indépendamment et utilisées pour affiner une carte de densité EM qui peut ensuite faire l’objet d’une interprétation plus poussée et de la construction de modèles.
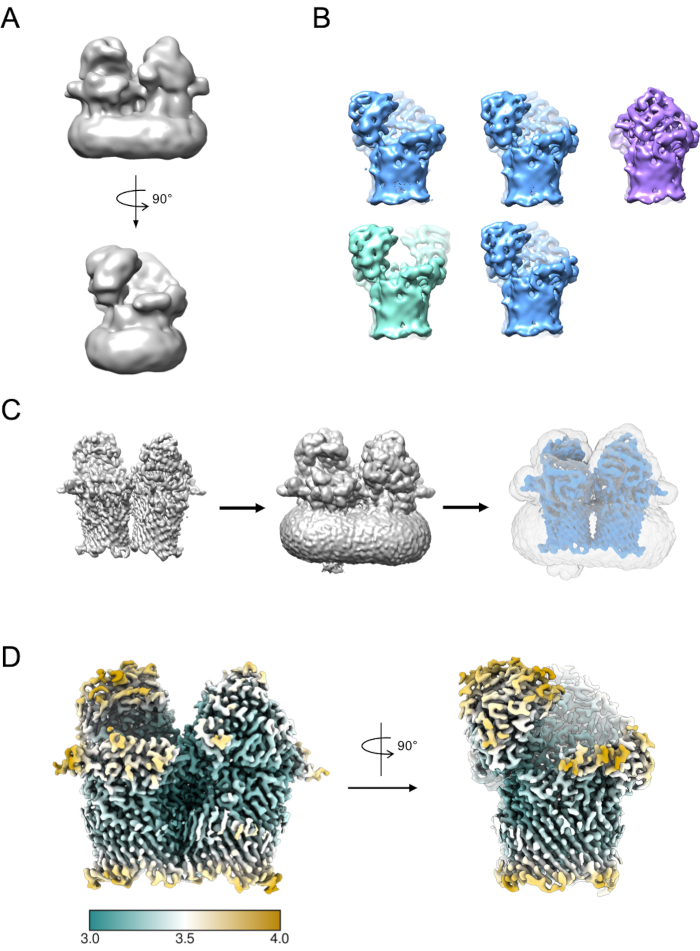
Figure 5 : Génération d’une carte de densité EM 3D. A) Modèle initial typique généré à l’aide de RELION. B) Classification 3D sur 5 classes montrant la séparation des particules en trois états conformationnels distincts : ouvert-ouvert (vert), ouvert-fermé (bleu), fermé-fermé (violet). C) Processus de création de masques. La carte du raffinement 3D (à gauche) doit être visualisée en chimère. La visionneuse de volume peut ensuite être utilisée pour identifier le seuil le plus bas à partir duquel la carte est exempte de densité disjointe et bruyante (milieu). Cette valeur de seuil est entrée en tant que seuil de binarisation initial dans la tâche de création de masque RELION. Un exemple de sortie de masque est affiché en gris (à droite). D) Carte de densité EM haute résolution de l’état ouvert-fermé de RagAB (EMD-10245), filtrée et colorée par résolution locale (Å). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Dans ce protocole, nous avons décrit un pipeline de base applicable aux spécimens pouvant faire l’objet d’une SPA de routine. Bien que cette méthode de buvard de papier filtre de formation de couches minces et de vitrification soit sans aucun doute réussie compte tenu de son utilisation dans la grande majorité des projets SPA à ce jour, elle présente un certain nombre d’inconvénients. Il s’agit notamment du gaspillage de l’échantillon, des délais lents (secondes) nécessaires pour former le film mince et congeler l’échantillon, de l’irréproductibilité signalée27 et des effets négatifs signalés de l’utilisation de papier filtre pour éliminer l’excès de liquide50. Récemment, de nouvelles technologies ont été développées pour améliorer la reproductibilité de la production de couches minces51,52. D’autres technologies ont été développées qui réduisent le temps entre l’application de l’échantillon et la vitrification53,54,55. Alors que les méthodes à base de papier filtre pour la formation de couches minces restent la méthode la plus répandue de préparation d’échantillons SPA cryoEM au moment de la rédaction, ces nouvelles technologies peuvent apporter une gamme d’avantages en termes d’efficacité et de reproductibilité de la vitrification de grille, ainsi que créer de nouvelles opportunités pour apporter des dimensions expérimentales supplémentaires, telles que la résolution temporelle et le mélange rapide avant la vitrification.
Le processus de criblage de grille pour la plupart des utilisateurs est actuellement un processus qualitatif qui implique l’acquisition d’atlas à faible grossissement, suivie de la prise d’images à fort grossissement à travers la grille pour évaluer la distribution des particules. Bien qu’il s’agisse d’une approche suffisamment robuste pour certains types d’échantillons, il peut être difficile d’évaluer à l’œil nu si le spécimen est effectivement ce que le chercheur espère imager ou a une orientation préférée, par exemple avec de petits échantillons (<200 kDa) ou lorsque la morphologie à basse résolution rend difficile l’identification à l’œil nu si une gamme de distributions de particules est présente. Pour certains projets, il est impossible de déterminer si l’échantillon est comme souhaité, par exemple lorsqu’un ligand est lié ou lorsque l’échantillon est examiné pour évaluer si une petite sous-unité (par exemple, 10 kDa) est toujours présente en association avec un complexe. Pour ces projets, des pipelines entièrement automatisés pour l’analyse des données combinés à des collections « courtes » de 0,5 à 1 h, qui peuvent passer par des étapes de traitement d’image à la classification 2D ou même à la classification et au raffinement 3D, aideraient à déterminer efficacement si une collecte plus longue est justifiée. Ces pipelines sont encore en cours de développement et ne sont pas largement mis en œuvre à l’heure actuelle, mais ils ont le potentiel d’améliorer l’efficacité du criblage de grille cryoEM, en particulier pour les échantillons difficiles.
Les améliorations apportées aux détecteurs d’électrons directs, ainsi que les modifications apportées à la microscopie, combinées aux progrès du traitement des images, telles que la collecte de données par décalage d’image, ont augmenté le débit et la qualité des images produites lors de l’acquisition de données. Cette augmentation du taux de collecte de données souligne la nécessité d’un criblage approfondi des réseaux cryoEM avant l’acquisition de nombreux To de données.
CryoEM SPA est devenu une technique de biologie structurale vraiment courante et, dans de nombreux cas, l’approche « aller à » pour certaines classes de spécimens, tels que les complexes macromoléculaires hétérogènes et labiles. Bien que le protocole décrit ici un aperçu de base du pipeline SPA, chaque section couverte ici (vitrification et criblage en grille, cryoEM et traitement d’image) est un sujet à part entière et mérite d’être exploré lors du développement d’un projet SPA. À mesure que les technologies de préparation des échantillons et de microscopie progressent, et que de nouveaux algorithmes et approches de traitement d’images sont mis en ligne, SPA continuera de se développer en tant que pipeline, aidant les chercheurs à mieux comprendre les systèmes biologiques complexes.
Déclarations de divulgation
Aucun conflit d’intérêts n’est signalé.
Remerciements
Ce travail a été soutenu par l’iNEXT-Discovery (Grant 871037) financé par le programme Horizon 2020 de la Commission européenne. J B. R. White est financé par le Wellcome Trust (215064/Z/18/Z). Les microscopes FEI Titan Krios ont été financés par l’Université de Leeds (prix UoL ABSL) et Wellcome Trust (108466/Z/15/Z). Nous remercions M Iadanza pour l’utilisation de son script d’analyse micrographique. Nous remercions Diamond Light Source pour l’accès et le soutien des installations cryo-EM du Centre national de bio-imagerie électronique (eBIC) du Royaume-Uni financé par le Wellcome Trust, le MRC et le BBRSC.
matériels
| Name | Company | Catalog Number | Comments |
| Blunt tweezers | Agar Scientific | AGT5022 | |
| Cryo EM round storage box | Agar Scientific | AGG3736 | |
| CryoEM autogrid boxes | ThermoFisher Scientific | 1084591 | |
| CryoEM grids | Quantifoil | N1-C14nCu30-01 | |
| Ethane gas | Boc | 270595-F | |
| LN2 foam dewar | Agar Scientific | AG81760-500 | |
| LN2 storage dewar | Worthington industries | HC 34 | |
| Pipette | Gilson | 10082012 | |
| Pipette tips | Star labs | s1111-1706 | |
| Syringe | BD | BD 300869 | |
| Type II lab water | Suez | select fusion | |
| Vitrobot | ThermoFisher Scientific | 1086439 | |
| Vitrobot filter paper | Whatman | 1001-055 | |
| Vitrobot styrophome container assembly | ThermoFisher Scientific | 1086439 | |
| Vitrobot tweesers | ThermoFisher Scientific | 72882-D | |
| Software | |||
| EPU | ThermoFisher Scientific | 2.8.1.10REL | |
| TEM server | ThermoFisher Scientific | 6.15.3.22415REL | |
| Tia | ThermoFisher Scientific | 5.0.0.2896REL | |
| Titan krios microscope | ThermoFisher Scientific | Titan Krios G2 |
Références
- Kuehlbrandt, W. The Resolution Revolution. Science. 343 (6178), 1443-1444 (2014).
- McMullan, G., Faruqi, A. R., Henderson, R. Direct Electron Detectors. Methods in Enzymology. , (2016).
- Elmlund, D., Le, S. N., Elmlund, H. High-resolution cryo-EM: the nuts and bolts. Current Opinion in Structural Biology. , (2017).
- Lyumkis, D. Challenges and opportunities in cryo-EM single-particle analysis. Journal of Biological Chemistry. , (2019).
- Nakane, T., et al. Single-particle cryo-EM at atomic resolution. Nature. , (2020).
- Yip, K. M., Fischer, N., Paknia, E., Chari, A., Stark, H. Atomic-resolution protein structure determination by cryo-EM. Nature. , (2020).
- Conley, M. J., et al. Calicivirus VP2 forms a portal-like assembly following receptor engagement. Nature. 565 (7739), 377-381 (2019).
- Hesketh, E. L., et al. The 3.3 Å structure of a plant geminivirus using cryo-EM. Nature communications. 9 (1), 2369 (2018).
- Malone, L. A., et al. Cryo-EM structure of the spinach cytochrome b6 f complex at 3.6 A resolution. Nature. 575 (7783), 535-539 (2019).
- Madej, M., et al. Structural and functional insights into oligopeptide acquisition by the RagAB transporter from Porphyromonas gingivalis. Nature Microbiology. , (2020).
- Gallardo, R., et al. Fibril structures of diabetes-related amylin variants reveal a basis for surface-templated assembly. Nature Structural and Molecular Biology. , (2020).
- Scarff, C., et al. Structure of the shutdown state of myosin-2. Nature. , (2020).
- Scarff, C. A., et al. Structure of the protective nematode protease complex H-gal-GP and its conservation across roundworm parasites. PLoS Pathogens. 16 (4), 1008465 (2020).
- Wu, M., Lander, G. C. How low can we go? Structure determination of small biological complexes using single-particle cryo-EM. Current Opinion in Structural Biology. , (2020).
- Khoshouei, M., Radjainia, M., Baumeister, W., Danev, R. Cryo-EM structure of haemoglobin at 3.2 Å determined with the Volta phase plate. Nature Communications. , (2017).
- Drulyte, I., et al. Approaches to altering particle distributions in cryo-electron microscopy sample preparation. Acta crystallographica. Section D, Structural biology. 74, 560-571 (2018).
- Thompson, R. F., Walker, M., Siebert, C. A., Muench, S. P., Ranson, N. A. An introduction to sample preparation and imaging by cryo-electron microscopy for structural biology. Methods. 100, 3-15 (2016).
- Cheng, Y., Grigorieff, N., Penczek, P. A., Walz, T. A primer to single-particle cryo-electron microscopy. Cell. 161 (3), 438-449 (2015).
- Scarff, C. A., Fuller, M. J. G., Thompson, R. F., Iadanza, M. G. Variations on negative stain electron microscopy methods: tools for tackling challenging systems. Journal of Visualized Experiments. (132), e57199 (2018).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative Staining and Image Classification - Powerful Tools in Modern Electron Microscopy. Biological procedures online. 6, 23-34 (2004).
- Baker, L. A., Rubinstein, J. L. Radiation Damage in Electron Cryomicroscopy. Methods in enzymology. 481, 371-388 (2010).
- Dubochet, J., et al. Cryo-electron microscopy of vitrified specimens. Quarterly Reviews of Biophysics. 21 (02), 129-228 (1988).
- Passmore, L. A., Russo, C. J. Specimen Preparation for High-Resolution Cryo-EM. Methods in enzymology. 579, 51-86 (2016).
- Carragher, B., et al. Current outcomes when optimizing ‘standard’ sample preparation for single-particle cryo-EM. Journal of Microscopy. , (2019).
- Naydenova, K., Jia, P., Russo, C. J. Cryo-EM with sub-1 Å specimen movement. Science. , (2020).
- Passmore, L. A., Russo, C. J. Specimen Preparation for High-Resolution Cryo-EM. Methods in Enzymology. , (2016).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature protocols. 14 (1), 100-118 (2019).
- Suloway, C., et al. Automated molecular microscopy: the new Leginon system. Journal of Structural Biology. 151 (1), 41-60 (2005).
- Zhang, J., et al. JADAS: A customizable automated data acquisition system and its application to ice-embedded single particles. Journal of Structural Biology. , (2009).
- Mastronarde, D. N. SerialEM: A program for automated tilt series acquisition on Tecnai microscopes using prediction of specimen position. Microscopy and Microanalysis. , (2003).
- Schorb, M., Haberbosch, I., Hagen, W. J. H., Schwab, Y., Mastronarde, D. N. Software tools for automated transmission electron microscopy. Nature Methods. , (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta crystallographica. Section D, Structural biology. 73, 496-502 (2017).
- Gómez-Blanco, J., et al. Using Scipion for stream image processing at Cryo-EM facilities. Journal of Structural Biology. , (2018).
- Wagner, T., et al. SPHIRE-crYOLO is a fast and accurate fully automated particle picker for cryo-EM. Communications biology. 2 (1), 213-218 (2019).
- Bepler, T., et al. TOPAZ: A Positive-Unlabeled Convolutional Neural Network CryoEM Particle Picker that can Pick Any Size and Shape Particle. Microscopy and Microanalysis. , (2019).
- Zivanov, J., et al. New tools for automated high-resolution cryo-EM structure determination in RELION-3. eLife. 7, 163 (2018).
- Zivanov, J., Nakane, T., Scheres, S. H. W. A Bayesian approach to beam-induced motion correction in cryo-EM single-particle analysis. IUCrJ. , (2019).
- Cianfrocco, M. A., Kellogg, E. H. What Could Go Wrong? A Practical Guide to Single-Particle Cryo-EM: From Biochemistry to Atomic Models. Journal of Chemical Information and Modeling. , (2020).
- Tagari, M., Newman, R., Chagoyen, M., Carazo, J. M., Henrick, K. New electron microscopy database and deposition system. Trends in Biochemical Sciences. , (2002).
- Berman, H. M., et al. The Protein Data Bank. Nucleic Acids Research. , (2000).
- Iudin, A., Korir, P. K., Salavert-Torres, J., Kleywegt, G. J., Patwardhan, A. EMPIAR: A public archive for raw electron microscopy image data. Nature Methods. , (2016).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. CryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. , (2017).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nature Methods. , (2019).
- Goddard, T. D., et al. UCSF ChimeraX: Meeting modern challenges in visualization and analysis. Protein Science. 27 (1), 14-25 (2018).
- Klebl, D. P., et al. Need for Speed: Examining Protein Behavior during CryoEM Grid Preparation at Different Timescales. Structure. , (2020).
- Noble, A. J., et al. Routine single particle CryoEM sample and grid characterization by tomography. eLife. 7, 32 (2018).
- Danev, R., Buijsse, B., Khoshouei, M., Plitzko, J. M., Baumeister, W. Volta potential phase plate for in-focus phase contrast transmission electron microscopy. Proceedings of the National Academy of Sciences. , (2014).
- Zi Tan, Y., et al. Addressing preferred specimen orientation in single-particle cryo-EMthrough tilting. Nature Methods. , (2017).
- Armstrong, M., Han, B. -. G., Gomez, S., Turner, J., Fletcher, D. A., Glaeser, R. M. Microscale Fluid Behavior during Cryo-EM Sample Blotting. Biophysical Journal. 118 (3), 708-719 (2020).
- Arnold, S. A., et al. Blotting-free and lossless cryo-electron microscopy grid preparation from nanoliter-sized protein samples and single-cell extracts. Journal of Structural Biology. , (2017).
- Dandey, V. P., et al. Spotiton: New Features and Applications. Journal of Structural Biology. , (2018).
- Rubinstein, J. L., et al. Shake-it-off: a simple ultrasonic cryo-EM specimen-preparation device. Acta crystallographica. Section D, Structural biology. 75, 1063-1070 (2019).
- Tan, Y. Z., Rubinstein, J. L. Through-grid wicking enables high-speed cryoEM specimen preparation. bioRxiv. , (2020).
- Klebl, D. P., et al. Sample deposition onto cryo-EM grids: From sprays to jets and back. Acta Crystallographica Section D: Structural Biology. , (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon