Method Article
Optogénétique bioluminescente 2.0 : Exploiter la bioluminescence pour activer les protéines photosensorielles in vitro et in vivo
Dans cet article
Résumé
La bioluminescence-lumière émise par une enzyme luciférase oxydant un substrat de petite molécule, une luciférine-peut être exploitée pour activer des protéines photosensorielles, ajoutant ainsi une autre dimension à la stimulation lumineuse et permettant la manipulation d’une multitude de fonctions médiées par la lumière dans les cellules à travers les échelles temporelles et spatiales.
Résumé
La bioluminescence - lumière émise par une enzyme luciférase oxydant un substrat de petite molécule, une luciférine - a été utilisée in vitro et in vivo pour activer les canaux ioniques et les pompes à lumière dans les neurones. Bien que cette approche optogénétique bioluminescente (BL-OG) confère une composante chimiogénétique aux outils optogénétiques, elle ne se limite pas à une utilisation en neurosciences. Au contraire, la bioluminescence peut être exploitée pour activer n’importe quelle protéine photosensorielle, permettant ainsi la manipulation d’une multitude de fonctions médiées par la lumière dans les cellules. Une variété de paires luciférase-luciférine peuvent être associées à des protéines photosensorielles nécessitant différentes longueurs d’onde d’intensité lumineuse et lumineuse.
Selon l’application spécifique, l’apport de lumière efficace peut être obtenu en utilisant des protéines de fusion luciférase-photorécepteur ou par simple co-transfection. Les protéines photosensorielles basées sur la dimérisation dépendante de la lumière ou les changements conformationnels peuvent être entraînées par la bioluminescence pour affecter les processus cellulaires de la localisation des protéines, la régulation des voies de signalisation intracellulaire à la transcription. Le protocole ci-dessous détaille l’exécution expérimentale de l’activation de la bioluminescence dans les cellules et les organismes et décrit les résultats en utilisant des recombinases et des facteurs de transcription basés sur la bioluminescence. Le protocole fournit aux chercheurs les procédures de base pour réaliser l’optogénétique bioluminescente in vitro et in vivo. Les approches décrites peuvent être étendues et individualisées à une multitude de paradigmes expérimentaux différents.
Introduction
Les protéines photosensorielles peuvent être activées par la lumière provenant soit d’une source de lumière physique, soit d’une enzyme luciférase en présence de son substrat, la luciférine, pour générer une bioluminescence. Pour les applications qui nécessitent des échelles de temps milli- ou même femtosecondes et/ou une résolution spatiale unicellulaire, les sources lumineuses physiques (lasers et diodes électroluminescentes (LED)) sont les seules à pouvoir être réglées à ces échelles. Des exemples sont la restriction spatiale de la lumière utilisée pour stimuler les pôles opposés dans le développement des larves de drosophiles avec un contrôle temporel de la milliseconde1 ou la stimulation précise de structures subcellulaires uniques telles que les tubules mitochondriaux2. Cependant, de nombreuses autres applications pour les commutateurs optiques ont des priorités différentes, y compris le contrôle spatial étendu et l’application répétée de manière non invasive et sans dommages causés par la lumière, mais avec un contrôle temporel défini dans des échelles de temps minuscules et des intensités accordables. Ici, l’utilisation de luciférases comme source de lumière alternative pour activer les domaines de détection de la lumière présente plusieurs avantages. Contrairement à l’activation de la lumière par fibre optique, la bioluminescence atteint tous les domaines de détection de la lumière exprimés dans la population cellulaire cible lorsque la source lumineuse est génétiquement codée. L’utilisation de la bioluminescence atténue les inquiétudes concernant les dommages aux tissus et aux cellules par la fibre optique et l’exposition physique prolongée à la lumière. La lumière est allumée avec l’application du substrat de luciférase. L’apparition est immédiate in vitro et in vivo en fonction de la voie d’administration et dure environ 15-30 min; une présence prolongée ou une stimulation phasique de la lumière peut être obtenue avec différentes luciférines et avec des applications supplémentaires ou répétées de substrat3. Enfin, l’émission de bioluminescence peut être réglée en faisant varier la concentration de luciférine.
L’utilisation de la bioluminescence pour activer les photorécepteurs en mouvement ionique, c’est-à-dire les éléments optogénétiques, tels que les channelrhodopsines ou les pompes, a été largement démontrée4,5,6,7,8. Cette approche d’optogénétique bioluminescente (BL-OG) a été utilisée dans des expériences in vivo chez la souris et le rat5,6,7,9,10,11,12. L’activation des opsines par BL-OG nécessitait une quantité de bioluminescence d’au moins ~33 μW/mm2, l’efficacité de l’activation augmentant avec une émission lumineuse plus élevée6,9. Les photorécepteurs sensoriels qui se déplacent d’ions sont un sous-groupe du grand contingent de photorécepteurs sensoriels trouvés dans la nature qui ne se déplacent pas d’ions13,14. L’extension de la bioluminescence à l’activation de photorécepteurs mobiles non ioniques, tels que les domaines de photosension de plantes ou de bactéries, est encouragée par des rapports15,16 selon lesquels les photocapteurs mobiles non ioniques sont significativement plus sensibles à la lumière que les channelrhodopsines, assurant un entraînement encore meilleur des capteurs de lumière avec bioluminescence que ceux déjà obtenus avec des éléments optogénétiques déplaçant des ions. Récemment, plusieurs publications ont signalé l’utilisation de la bioluminescence comme source lumineuse pour l’activation d’une variété de photorécepteurs, y compris les domaines lumière-oxygène-tension-détection (LOV), les domaines BLUF (blue-light-using-flavin) et les cryptochromes (CRYs)3,17,18,19,20,21,22 (Tableau 1 ). Les applications de l’activation par bioluminescence de commutateurs optiques ciblaient des processus intracellulaires allant de la mort cellulaire induite par les espèces réactives de l’oxygène, à la synthèse de l’AMPc, au recrutement et à la dissociation des protéines, en passant par la recombinaison génomique et l’induction de la transcription.
Ce protocole décrit la conception générale des outils optogénétiques basés sur la bioluminescence et détaille les procédures d’exécution expérimentale de l’activation de la bioluminescence dans les cellules et les organismes. Il comprend des descriptions sur la façon de mettre en place une pièce, une hotte de culture tissulaire et un incubateur, et un microscope pour travailler avec la bioluminescence, ainsi que les étapes de la préparation de la luciférine à son application. Ce protocole fournit aux chercheurs les procédures de base pour réaliser l’optogénétique bioluminescente (BL-OG) in vitro et in vivo. Les approches décrites peuvent être étendues et individualisées à différents paradigmes expérimentaux. Nous prévoyons que ce protocole facilitera l’adoption de l’utilisation de la bioluminescence dans les études biologiques optogénétiques.
Protocole
Toutes les procédures de la présente étude ont été effectuées à l’aide de protocoles approuvés par l’Institutional Animal Care and Use Committee (IACUC) pour la manipulation des animaux à la Central Michigan University, MI.
1. Activation par bioluminescence des protéines photosensorielles in vitro
- Construit
- Sélectionnez une séquence de luciférase ou une séquence de fusion de protéines fluorescentes de luciférase qui entraînera l’expression d’un émetteur de lumière produisant de la lumière d’une longueur d’onde correspondant au photorécepteur à activer.
REMARQUE: Par exemple, les luciférases émettrices de lumière bleue, telles que les variantes de Gaussia luciferase ou NanoLuc, peuvent être associées à des photorécepteurs de détection de lumière bleue tels que CRY / Ca2 + - et la protéine de liaison à l’intégrine (CIB), LOV ou Vivid (VVD). - Si ce n’est pas déjà le cas auprès d’autres chercheurs ou de gisements de plasmides, utilisez des techniques de biologie moléculaire standard pour cloner l’ADN dans un plasmide d’expression de mammifère.
NOTE: Le choix des promoteurs est dicté par la nécessité de fournir une expression forte et constitutive du module électroluminescent, tel que celui fourni par les promoteurs CAG et CMV. - Pour les études initiales, utilisez des plasmides distincts pour la co-transfection de l’émetteur de lumière et du capteur de lumière. Générer des protéines de fusion des deux fractions au besoin et pour des études ultérieures.
- Obtenez des ADN plasmidiques de haute qualité à l’aide de kits mini-, midi ou maxiprep selon les protocoles du fabricant.
- Sélectionnez une séquence de luciférase ou une séquence de fusion de protéines fluorescentes de luciférase qui entraînera l’expression d’un émetteur de lumière produisant de la lumière d’une longueur d’onde correspondant au photorécepteur à activer.
- Culture cellulaire et transfection
REMARQUE: Les cellules HeLa et les cellules HEK293 sont utilisées comme exemples dans ce protocole.- Cellules de plaque dans des formats et des nombres en fonction de l’utilisation finale souhaitée.
NOTE: Des exemples spécifiques sont donnés dans le tableau 2. La densité cellulaire au moment du placage déterminera à quel moment les cellules peuvent être transfectées.- Pour évaluer la transcription activée par bioluminescence par microscopie à fluorescence, des cellules HEK293 sur des couvercles de 12 mm revêtus de poly-D-lysine (PDL) placés dans des boîtes à 24 puits.
- Pour évaluer la transcription activée par la bioluminescence en mesurant l’émission lumineuse d’une luciférase rapporteure orthogonale dans un luminomètre, plaquez les cellules HeLa initialement dans des boîtes à 6 ou 12 puits pour la transfection, mais les recollez après la transfection (voir étape 4).
- Si une stimulation répétée par bioluminescence est effectuée dans des chambres d’imagerie à cellules vivantes, sélectionnez des couvercles de la taille appropriée et placez-les dans des plaques multipuits de la taille appropriée (plaques de 24 puits pour les couvercles de 12 mm; plaques de 12 puits pour les couvercles de 15 mm et de 18 mm). Ensemencez les cellules au-dessus des fiches de couverture à l’aide des numéros de cellule spécifiés dans le tableau 2. Si le type de cellule sélectionné n’adhère pas bien à la surface de culture, plaquez les cellules sur des plats revêtus de PDL.
- Effectuer la transfection par lipofection selon la recommandation du fabricant ou utiliser toute méthode de transfection appropriée pour le type de cellule sélectionné.
NOTE : Le tableau 3 détaille les expériences de transfection pour deux photorécepteurs différents, EL222 et CRY2/CIB, et leurs plasmides rapporteurs respectifs, en plus de différentes protéines électroluminescentes. Les rapports des différents plasmides fonctionnent bien pour les exemples sélectionnés mais devront être optimisés pour chaque paire émetteur de lumière/capteur de lumière. - Après la transfection, placez les cellules dans un incubateur complètement étanche à la lumière (Figure 1).
- Selon l’utilisation finale souhaitée, utilisez les cellules pour la stimulation par bioluminescence le lendemain dans leurs puits / plats d’origine, ou replaquez-les 3-4 h après la lipofection. Pour lire la transcription d’un gène rapporteur de luciole luciférase dans un luminomètre, replaquez les cellules dans des plaques blanches de 96 puits.
REMARQUE: Effectuez toutes les manipulations dans une pièce étanche à la lumière dans une hotte à flux laminaire éclairée par une lumière rouge (Figure 2).- Lavez les cellules transfectées une fois avec le milieu Eagle modifié (DMEM) de Dulbecco ou une solution saline tamponnée au phosphate (PBS).
- Ajouter le volume minimal d’un réactif trypsinisant aux puits (24 puits : 100 μL ; 12 puits : 150 μL ; 6 puits : 300 μL) et incuber les cellules pendant 3 min à 37 °C.
- Ajouter un milieu de culture pour obtenir une concentration cellulaire qui donnera la densité cellulaire appropriée pour l’étape de placage suivante (par exemple, remettre en suspension des cellules dans un puits 24 dans un volume final de 1,2 mL pour le placage dans 10 puits d’une plaque de 96 puits; remettre en suspension des cellules dans un puits 12 dans un volume final de 2,4 mL pour le placage dans 20 puits d’une plaque de 96 puits). Mettre en commun les cellules transfectées de plusieurs puits en fonction du nombre de puits nécessaires à la fin.
- Plaquez les cellules transfectées dans leur format final et retournez les plaques dans l’incubateur protégé de la lumière.
- Cellules de plaque dans des formats et des nombres en fonction de l’utilisation finale souhaitée.
- Activation de la bioluminescence in vitro
- Préparer le substrat de luciférase (luciférine).
- Préparer des stocks de 50 mM en dissolvant 5 mg de coelenterazine lyophilisée (CTZ) dans 250 μL de son solvant spécifique. Assurez-vous que tout le CTZ le long des parois du flacon est dissous par pipetage ou vortex. Protéger le flacon de la lumière directe.
- Préparer des aliquotes de 50 μL dans des tubes de microcentrifugation noirs de 0,5 mL et les conserver à -80 °C pour une utilisation future.
REMARQUE: Le CTZ dissous dans le solvant ne gèle pas à -80 °C. Les aliquotes peuvent être retirées et retournées au congélateur plusieurs fois pour créer des solutions de travail tant que l’exposition à la lumière et à la température ambiante est réduite au minimum.
- Stimulation lumineuse à bioluminescence unique
REMARQUE: Toutes les manipulations sont effectuées dans une pièce étanche à la lumière dans une hotte à flux laminaire éclairée par une lumière rouge (Figure 2).- Préparer une solution de travail de luciférine dans un milieu de culture cellulaire (DMEM ou NeuroBasal). Ajuster la concentration de la luciférine de manière à ce que la concentration finale soit de 100 μM. Préparer toutes les dilutions de CTZ dans un milieu peu de temps avant de les ajouter aux cellules, car le CTZ s’oxyde avec le temps.
REMARQUE: Si le volume entier du milieu est remplacé, la solution de travail sera de 100 μM. Si un milieu contenant de la luciférine est ajouté aux cellules, la concentration sera plus élevée par le facteur de dilution (par exemple, l’ajout de 50 μL de milieu contenant 300 μM de luciférine à 100 μL de milieu dans le puits entraînera une dilution de 1:3 et donc une concentration finale de 100 μM de luciférine). - Ajouter un milieu contenant de la luciférine aux cellules et incuber pendant la durée souhaitée de la stimulation lumineuse.
REMARQUE: Cela peut être aussi court que 1 min ou aussi long que 15 min et peut être encore plus court ou plus long. La durée de la sortie du milieu contenant de la luciférine sur les cellules dépend de la demi-vie et de la cinétique de la combinaison luciférase-luciférine sélectionnée. - Surveiller l’émission de lumière à une concentration finale de luciférine de 100 μM à l’œil nu après avoir éteint la lumière rouge; attendez quelques secondes jusqu’à ce que les yeux se soient ajustés à l’obscurité complète. Documentez l’émission de lumière en prenant une photo (même avec un téléphone portable).
- Terminez la stimulation lumineuse en retirant le milieu contenant de la luciférine et en le remplaçant par un milieu de culture. Selon la sensibilité des expériences, laver les cellules avec un milieu de culture une ou deux fois après avoir retiré le milieu contenant de la luciférine pour éliminer toute la luciférine. Si les cellules n’adhèrent pas bien à la surface de culture, plaquez-les sur de la vaisselle enduite de PDL pour éviter de perdre les cellules pendant les lavages.
- Renvoyer les cellules dans l’incubateur protégé par la lumière pendant 16 à 24 h.
- Préparer une solution de travail de luciférine dans un milieu de culture cellulaire (DMEM ou NeuroBasal). Ajuster la concentration de la luciférine de manière à ce que la concentration finale soit de 100 μM. Préparer toutes les dilutions de CTZ dans un milieu peu de temps avant de les ajouter aux cellules, car le CTZ s’oxyde avec le temps.
- Stimulation lumineuse par bioluminescence répétée
REMARQUE: Toutes les manipulations sont effectuées dans une pièce qui peut être rendue étanche à la lumière et être éclairée par une lumière rouge.- Configurez la chambre d’imagerie des cellules vivantes. Créez un compartiment étanche à la lumière autour du microscope d’imagerie à cellules vivantes à l’aide d’une boîte et de feuilles de plastique noires ou de rideaux noirs (Figure 3). Couvrez toutes les sources lumineuses présentes à l’intérieur du compartiment étanche à la lumière et de la pièce (par exemple, les indicateurs LED sur le microscope ou les instruments).
- Installez le système de perfusion avec la solution souhaitée pour l’admission et l’abri de la chambre menant à un conteneur à déchets.
REMARQUE: Par exemple, la solution d’imagerie peut être la solution de Tyrode (chlorure de sodium (124 mM), chlorure de potassium (3 mM), HEPES (10 mM), chlorure de calcium dihydraté (2 mM), chlorure de magnésium hexahydraté (1 mM), D-glucose (20 mM)). - Préparez une solution fonctionnelle de luciférine dans la solution d’imagerie. Aliquote dans autant de tubes de microcentrifugation que le nombre de stimulations répétées. Ajuster la concentration de la luciférine de telle sorte que la concentration finale dans la chambre d’imagerie soit de 100 μM.
- Placez un couvercle avec des cellules transfectées dans la chambre.
- Tout en maintenant la pompe en marche, retirez le tube d’entrée de la pompe du bécher d’admission et immergez-le rapidement dans la solution de luciférine, en gardant le temps de transition aussi court que possible pour éviter tout vide d’air dans le tube.
- Dès que la solution de luciférine a été absorbée, replacez le tube d’entrée dans le bécher d’admission. Répétez ce processus autant de fois que nécessaire et à des intervalles de plusieurs minutes à quelques heures, en fonction du schéma physiologique auquel les cellules sont censées être exposées.
- Renvoyer les cellules dans l’incubateur protégé par la lumière pendant 16 à 24 heures pour la transcription ou pendant la durée pendant laquelle l’effet de la stimulation lumineuse doit être évalué.
- Préparer le substrat de luciférase (luciférine).
2. Activation par bioluminescence des protéines photosensorielles in vivo
- Construit
- Sélectionnez une séquence de luciférase ou une séquence de fusion de protéines fluorescentes de luciférase qui entraînera l’expression d’un émetteur de lumière produisant de la lumière d’une longueur d’onde correspondant au photorécepteur à activer.
- Utilisez des techniques de biologie moléculaire standard pour cloner l’ADN dans un plasmide pAAV, s’il n’est pas déjà disponible auprès d’autres chercheurs ou de dépôts de plasmides.
- Choisissez des promoteurs puissants pour l’expression des modules électroluminescents, tels que CAG ou CMV.
- Utiliser des approches standard pour préparer des stocks viraux à titre élevé6 ou faire préparer commercialement des vecteurs viraux.
- Pour les études initiales, utilisez des vecteurs viraux distincts pour la co-transduction de l’émetteur de lumière et du capteur de lumière afin de permettre l’ajustement des rapports des différents composants si nécessaire.
- Transduction AAV
- Injecter à l’organe cible de l’animal expérimental des vecteurs viraux de l’émetteur de lumière, du capteur de lumière et du rapporteur analogues aux rapports de concentration utilisés pour les transfections in vitro (tableau 3).
- Remettez les animaux dans leurs cages d’origine pendant au moins 2 semaines pour permettre une expression maximale de tous les composants.
REMARQUE: Si l’organe cible est à l’intérieur du corps et protégé de la lumière ambiante, les animaux peuvent être logés dans des conditions de lumière normales.
- Activation de la bioluminescence in vivo
- Préparer le substrat de luciférase (luciférine).
- Sortez un flacon de CTZ soluble dans l’eau du congélateur à -80 °C et laissez-le chauffer à température ambiante. Gardez-le à l’abri de la lumière.
- Par flacon de 500 μg, ajouter 250 μL d’eau stérile, soit à l’aide d’une seringue, soit en ouvrant le flacon et en ajoutant de l’eau avec une pipette, puis en remettant le bouchon en caoutchouc sur le flacon en verre.
- Incuber le flacon en verre reconstitué dans un bain-marie à 55 °C pendant quelques minutes pour dissoudre complètement la poudre.
- Transférer la solution dans un tube de microcentrifugation noir. Rincez les parois du flacon en verre pour récupérer tout le CTZ.
- Retirez la quantité de solution nécessaire pour la journée. Conserver le reste de la solution à 4 °C pour une utilisation le lendemain. Ne pas congeler!
- Effectuer les mêmes étapes (2.3.1.1.-2.3.1.5) pour un flacon de véhicule.
- Stimulation lumineuse par bioluminescence
- Retirer le volume de luciférine/véhicule nécessaire pour la taille de l’animal et la voie d’application choisie (tableau 4).
- Injecter aux animaux de la luciférine ou du véhicule. Répétez la stimulation lumineuse par bioluminescence conformément à la conception expérimentale. Par exemple, si l’activation d’une recombinase est souhaitée au cours d’un paradigme comportemental spécifique, injectez les animaux juste avant le test comportemental. Si la transcription phasique d’une molécule est le but, injectez les animaux à plusieurs reprises pendant des jours.
- Recueillir des données auprès des animaux stimulés par la bioluminescence comme prévu.
- Préparer le substrat de luciférase (luciférine).
Résultats
Il existe de nombreux événements intracellulaires qui peuvent être manipulés avec des actionneurs répondant à la lumière, et qui se prêtent à une activation bimodale avec des sources de lumière physiques et biologiques. Vous trouverez ci-dessous des exemples utilisant un intégrateur de calcium photosensible (Ca2+), une translocation de protéines induite par la lumière, un facteur de transcription de détection de la lumière et une recombinase photosensible. Les exemples illustrent la faisabilité de l’utilisation de la bioluminescence pour activer divers types de photorécepteurs. Les expériences présentées n’ont pas été spécifiquement optimisées en ce qui concerne l’application de diodes électroluminescentes (LED), la luciférase choisie, ou en ce qui concerne les concentrations et le moment de l’application de la luciférine.
L’expression rapide régulée par la lumière et l’activité (FLARE) est un système optogénétique qui permet la transcription d’un gène rapporteur avec la coïncidence d’une augmentation du Ca2+ intracellulaire et de la lumière23 (Figure 4A). La présence de Ca2+ est nécessaire pour amener la protéase à proximité du site de clivage de la protéase accessible uniquement avec une stimulation lumineuse, ce qui entraîne la libération du facteur de transcription. Les cellules HEK293 ont été co-transfectées avec les composants FLARE d’origine, une construction de rapporteur double Firefly (FLuc)-dTomato et une variante de gaussia luciférase sbGLuc6 ancrée dans la membrane. En présence d’une augmentation du Ca2+ intracellulaire par l’exposition des cellules à 2 μM d’ionomycine et à 5 mM de chlorure de calcium (CaCl2), l’application de LED bleue a conduit à une expression robuste du rapporteur de fluorescence par rapport aux cellules laissées dans l’obscurité, ainsi qu’à l’expression de FLuc déterminée en mesurant la luminescence lors de l’ajout du substrat FLuc, D-luciférine. Des niveaux similaires d’expression de FLuc ont été atteints avec la bioluminescence émise par sbGLuc lors de l’application du substrat sbGLuc (CTZ) avec l’ionomycine et CaCl2. Notez que les luciférases utilisées pour l’activation de la lumière (sbGLuc) et pour rapporter l’effet de l’activation de la lumière (transcription de FLuc) ne produisent de la lumière qu’avec leurs luciférines respectives (CTZ vs D-luciférine) et ne réagissent pas de manière croisée.
Différents composants ont été combinés pour générer un système de transcription induit par la lumière basé sur l’hétérodimérisation des cryptochromes23,24 (Figure 4B). CRY2 a été fusionné à une protéase tandis que le CIB lié à la membrane a été fusionné au site de clivage de la protéase et au facteur de transcription. La translocation des protéines induite par la lumière a libéré le facteur de transcription, conduisant à l’expression de FLuc et de dTomato, comme le montre la figure 4A. Alors que la présence de la composante du facteur de transcription à elle seule a entraîné un signal de fond considérable, peut-être dû à une protéolyse spontanée, la lumière physique (LED) et la bioluminescence (CTZ) ont fortement augmenté l’expression de FLuc mesurée dans un système d’imagerie in vivo (IVIS).
Dans une autre série d’expériences, NanoLuc (luciférine: furimazine ou hCTZ) a été utilisé pour la régulation optogénétique de la transcription par la dimérisation de CRY / CIB et le facteur de transcription photosensible, EL22225,26,27. La figure 5A,B montre les schémas des différents composants dans les états sombre et clair et de la luciférase co-transfectée ou fusionnée au capteur de lumière. Diverses comparaisons sont présentées à la figure 5C. La bioluminescence, induite par l’ajout de hCTZ aux cellules HEK293 exprimant les constructions et en les retirant après 15 minutes, était plus efficace pour conduire la transcription du rapporteur que 20 minutes d’exposition à la lumière LED pour CRY / CIB et EL222. Pour CRY/CIB, une heure d’exposition aux LED était suffisante pour atteindre un niveau de transcription comparable à 15 min de bioluminescence. En revanche, pour EL222, même 60 minutes de LED étaient à peine deux fois moins efficaces qu’une brève exposition à la bioluminescence. Il n’y avait pas de différences significatives dans l’efficacité de la transcription entre les deux systèmes lorsqu’ils étaient co-transfectés, bien que les protéines de fusion de CRY/CIB soient plus efficaces que celles d’EL222. Pour les deux systèmes, les protéines de fusion ont conduit à des niveaux de transcription significativement plus élevés que les composants co-transfectés. CRY/CIB a montré des niveaux de fond constamment plus élevés avec l’application du véhicule par rapport à EL222, qui avait une transcription de fond négligeable. L’augmentation des concentrations de hCTZ seule n’a eu aucun effet sur la transcription du gène rapporteur.
Les recombinases photoactivables constituent un outil polyvalent pour les manipulations optogénomiques. Nous avons testé l’activation par bioluminescence d’une cre recombinase fractionnée photosensible basée sur la protéine Vivid LOV, iCreV28. La figure 6A montre un schéma des différents composants, sbGLuc, iCreV, et un rapporteur de fluorescence lox-stop-lox (tdTomato) avant et après l’application de CTZ. Les résultats de l’application CTZ par rapport aux commandes (pas de CTZ ou de LED) sont illustrés à la Figure 6B. Il y a une certaine expression de fond même dans l’obscurité (pas de CTZ); cependant, en présence de CTZ, l’expression est fortement augmentée sur l’arrière-plan et similaire à celle induite avec l’application de LED.

Figure 1 : Incubateur scellé à la lumière. Rabat de boîte en carton recouvrant la lumière du panneau de commande éclairé (flèche supérieure). Couvercle imperméable à la lumière sur la porte vitrée de l’incubateur (flèche du bas) pour protéger les cellules de l’exposition à la lumière. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Hotte à écoulement laminaire éclairée par une lumière rouge. Configuration montrant une hotte de culture tissulaire à flux laminaire standard éclairée par une lumière rouge. La flèche indique une lampe de bureau standard avec une ampoule rouge. Toutes les manipulations sous la lumière rouge sont effectuées dans une pièce autrement sombre et étanche à la lumière. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Compartiments étanches à la lumière autour des microscopes d’imagerie à cellules vivantes. Deux exemples de configurations de microscope d’imagerie à cellules vivantes montrant l’utilisation d’une boîte solide avec des rideaux en plastique uniquement sur la face avant (panneaux de gauche: haut et bas) ou de rideaux noirs tout autour de la configuration d’imagerie (panneaux de droite: haut et bas). Les faces avant des deux exemples restent ouvertes et enroulées lorsqu’elles ne sont pas utilisées (panneaux supérieurs: gauche et droite). Les rideaux noirs avant sont enroulés vers le bas pour empêcher toute lumière dans la pièce (p. ex., écrans d’ordinateur) d’entrer dans la zone d’imagerie lors de la stimulation et/ou de l’imagerie par bioluminescence des cellules vivantes (panneaux du bas : gauche et droite). Veuillez cliquer ici pour voir une version agrandie de cette figure.
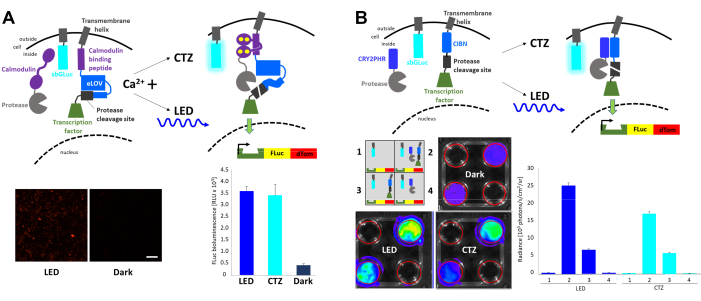
Figure 4 : Bioluminescence pour l’intégration des événements de signalisation intracellulaire. (A) Schémas des composants FLARE co-transfectés avec sbGLuc. En présence de Ca2+ et de la proximité résultante de la protéase avec le site de clivage de la protéase, la bioluminescence ou la LED entraînera le déploiement du LOV, l’exposition du site de clivage et la libération du facteur de transcription. Les cellules ont été exposées à des LED (rapport cyclique de 33 %, 2 s marche/4 s d’arrêt pendant 40 min ; puissance lumineuse de 3,5 mW, irradiance de 4,72 mW/cm2) ou à la bioluminescence (concentration finale de 100 μM de CTZ pendant 15 min) ou laissées dans l’obscurité. Images microscopiques de cellules HEK293 exprimant les composants ci-dessus après traitement pour augmenter les niveaux de Ca2+ et l’exposition aux LED (à gauche). Luminescence FLuc mesurée dans un luminomètre comparant l’exposition aux LED, à la bioluminescence (CTZ) ou à gauche dans l’obscurité (droite). (B) Schémas d’un système de transcription non dépendant du Ca2+ co-transfecté avec sbGLuc. Les cellules HEK293 dans des plaques à 4 puits ont été transfectées avec quatre arrangements différents de composants comme décrit dans le schéma. Les plaques ont été exposées soit à des LED (rapport cyclique de 33 %, 2 s marche/4 s d’arrêt pendant 40 min; puissance lumineuse de 3,5 mW, irradiance de 4,72 mW/cm2) ou à une bioluminescence (concentration finale de 100 μM de CTZ) en ajoutant du CTZ et en le laissant allumé pendant 15 min; les plaques de contrôle ont été laissées dans l’obscurité. La transcription du rapporteur FLuc a été mesurée dans un IVIS. Des images IVIS de plats représentatifs sont montrées à gauche; Les mesures de radiance de plusieurs répliques de référence aux commandes sombres sont indiquées à droite. Barre d’échelle = 100 μm. Abréviations : FLARE = Expression rapide régulée par la lumière et l’activité; LOV = détection lumière-oxygène-tension; LED = diode électroluminescente; CTZ = coelenterazine; FLuc = luciole luciférase; dTom = dTomato; CRY2 = cryptochrome 2; CRY2PHR = région d’homologie de la photolyase CRY2; CIB1 = Ca2+- et protéine de liaison à l’intégrine 1; CIBN = N-terminus de CIB1; IVIS = système d’imagerie in vivo. Veuillez cliquer ici pour voir une version agrandie de cette figure.
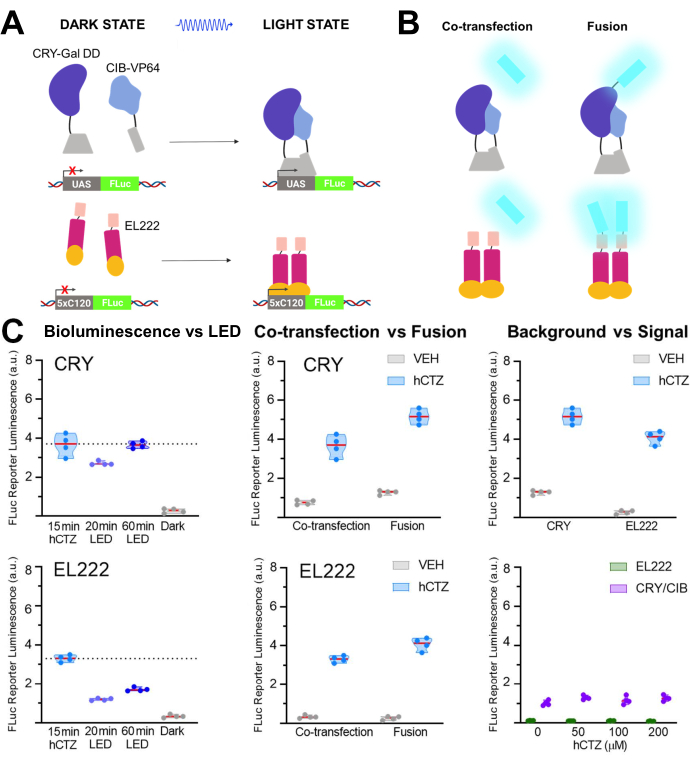
Figure 5: Bioluminescence pour la transcription de conduite. (A) Schémas de deux systèmes de transcription photoactivables dans leurs états sombre et clair. (B) NanoLuc a été co-transfecté ou fusionné aux fractions de détection de lumière comme illustré (N-NanoLuc-CRY-GalDD-C; N-NanoLuc-VP16-EL222-C). (C) Comparaisons utilisant les deux systèmes en ce qui concerne les sources lumineuses, la conception de la construction et le signal au bruit. Les cellules ont été exposées à des DEL (rapport cyclique de 33 %, 2 s marche/4 s d’arrêt pendant 40 min; puissance lumineuse de 3,5 mW, irradiance de 4,72 mW/cm2 ) ou à la bioluminescence pendant 15 min (concentration finale de 100 μM hCTZ; sauf si des concentrations différentes sont notées). Sombres, les plaques ont été laissées intactes dans l’incubateur entre la transformation initiale des plasmides et la mesure du FLuc; VEH, les plaques ont été manipulées de la même manière que celles recevant hCTZ, mais ont reçu le véhicule à la place. Différences dans les niveaux de transcription: hCTZ, CRY co-transfecté vs EL222 - non significatif; hCTZ, luciférase - fusion de photoprotéines CRY vs EL222 - p < 0,005; hCTZ, cry co-transfection vs fusion - p < 0,005; hCTZ, EL222 co-transfection vs fusion - p < 0,01; véhicule, CRY vs EL222 - p < 0,05. Abréviations : UAS = séquence d’activation en amont; LED = diode électroluminescente; CTZ = coelenterazine; FLuc = luciole luciférase; CRY = cryptochrome; CIB = Ca2+- et protéine de liaison à l’intégrine; VEH = véhicule. Veuillez cliquer ici pour voir une version agrandie de cette figure.
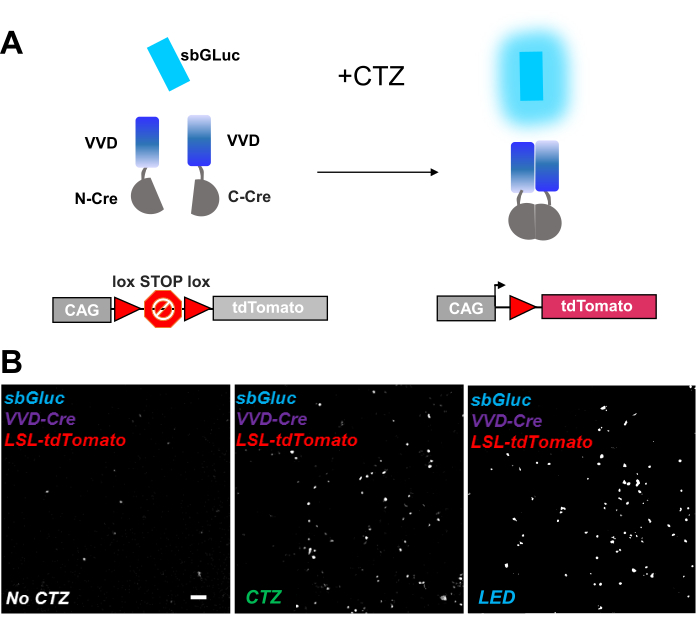
Figure 6: Bioluminescence pour la manipulation optogénomique. (A) Schémas de la manipulation optogénomique pilotée par la bioluminescence à l’aide de sbGLuc, des composants iCreV divisés et d’une cassette rapporteur LSL, avant et après l’application de la lumière. (B) Les cellules HEK293 ont été lipofectées avec des plasmides, puis maintenues dans l’obscurité. Vingt-quatre heures plus tard, les cellules ont été traitées pendant 30 min avec un milieu (pas de CTZ) ou avec du CTZ (concentration finale de 100 μM) ou avec des LED (rapport cyclique de 25 %, 5 s marche/15 s d’arrêt pendant 5 min ; puissance lumineuse de 14,81 mW, irradiance de 20 mW/cm2 ) comme témoin positif. Images microscopiques de la fluorescence de tdTomato en utilisant les conditions indiquées. Barre d’échelle = 100 μm. Abréviations: LSL = lox-stop-lox; CTZ = coelenterazine; LED = diode électroluminescente; VVD = Vif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Activation par bioluminescence des photorécepteurs. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Lignes directrices pour le placage et la transfectation des cellules dans différents formats. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Ratios de divers plasmides pour la transfection. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 4 : Voies d’injection, volumes et concentrations de luciférine pour des applications in vivo (souris de 25 g). Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Il existe une gamme de luciférases et de luciférines avec des longueurs d’onde d’émission lumineuse correspondant aux spectres d’activation des protéines photosensorielles de la lumière bleue à la lumière rouge14,29. En dehors de l’alignement des longueurs d’onde d’émission et d’excitation, il n’existe aucun moyen fiable de déterminer a priori quel couplage fonctionnera le mieux. Ainsi, la nécessité de déterminer expérimentalement comment les paires luciférine-luciférase fonctionnent dans les cellules et les organismes dans les systèmes photosensoriels moteurs.
Les protocoles décrits dans cette présentation décrivent comment préparer la luciférine et comment l’appliquer in vitro et in vivo, ainsi que des directives sur la mise en place de salles, de hottes de culture tissulaire, d’incubateurs et de microscopes pour des expériences utilisant la bioluminescence. Dans les expériences représentatives, différentes luciférases (NanoLuc, Gaussia luciferase) avec plusieurs protéines photosensorielles (CRY / CIB, EL222, VVD, LOV) ont été utilisées, démontrant les effets de la bioluminescence par rapport à la lumière physique, de la co-transfection par rapport aux protéines de fusion, des comparaisons signal-bruit et différents tests de lecture. D’autres applications des protéines photosensorielles activatrices de la bioluminescence sont décrites dans des publications de plusieurs groupes, ciblant l’induction de la mort cellulaire, la synthèse de l’AMPc et le mouvement des protéines en plus de la transcription (tableau 1).
La simple co-transfectation des composants émetteurs et capteurs de lumière est un bon début. Les variables sont les rapports molaires de l’émetteur et du capteur; les inconnues sont les niveaux de fond de l’activité du capteur dans l’obscurité, l’activité du capteur par rapport à l’intensité et à la durée de la lumière, et l’efficacité de l’activation du capteur en comparant la lumière physique et biologique. Alors que les constructions de fusion ont l’avantage de maintenir le rapport molaire de l’émetteur et du capteur à 1:1 et de rapprocher l’émetteur de lumière du domaine de détection de la lumière, d’autres considérations entrent en jeu, telles que l’endroit où attacher (N- ou C-terminus) et comment se lier (longueur et composition de l’éditeur de liaison) sans affecter les performances de l’actionneur photosensoriel.
Pour les expériences in vitro et in vivo, il existe de multiples options pour régler l’émission de lumière bioluminescente, soit en faisant varier la concentration de la luciférine, soit en faisant varier le temps pendant lequel la luciférine est mise à la disposition du capteur respectif. La quantité et le temps minimum sont déterminés par la présence ou l’absence de l’effet attendu avec l’activation de la lumière. En revanche, les maxima respectifs sont principalement déterminés par la tolérance des cellules à des concentrations élevées de luciférine sur des périodes prolongées. La concentration de CTZ choisie dans les exemples ci-dessus, 100 μM, est proche de la limite supérieure pour divers types de cellules, des cellules HEK293 aux neurones. L’objectif est d’utiliser une concentration aussi faible que possible pendant le temps le plus court pour obtenir l’activation du domaine de photosension ciblé. Cela sera réalisé plus facilement en utilisant des luciférases à forte émission de lumière et des photorécepteurs à haute sensibilité à la lumière.
La bioluminescence pour piloter les photorécepteurs a été utilisée chez les rongeurs (souris, rats) avec des protéines photosensibles exprimées dans le foie, les muscles, la moelle épinière et le cerveau, ainsi que via des cellules exprimant des photorécepteurs transplantées par voie sous-cutanée ou intrapéritonéale. En principe, il n’y a pas de limites empêchant l’approche d’être appliquée à différentes espèces, des primates non humains aux poissons ou aux mouches. Selon la perméabilité de l’organisme à la luciférine, l’application peut être aussi simple que l’application de la luciférine dans l’eau environnante (p. ex., chez les larves de poisson30). Avant d’utiliser BL-OG dans tout nouvel organisme, des expériences pilotes doivent être menées pour s’assurer que la luciférine atteint ses cibles par la voie d’application choisie.
Les aspects critiques de la conception expérimentale sont les divers contrôles qui sont importants pour l’interprétation des résultats. Les cellules exprimant un rapporteur entraîné par une luciférase agissant sur une protéine photosensorielle doivent être comparées à des cellules dépourvues de luciférase ou dépourvues de protéine photosensorielle. En outre, des comparaisons doivent être faites entre les cellules exposées à la luciférine, au véhicule ou maintenues dans l’obscurité. Il est également important de réaliser les limites des différents tests pour évaluer les effets de l’activation des photorécepteurs par bioluminescence. Par exemple, l’efficacité de la transcription activée par la bioluminescence peut être testée de différentes manières, selon que le gène rapporteur est une luciférase orthogonale (luminomètre, IVIS) ou une protéine fluorescente (tri cellulaire activé par fluorescence, analyse d’images par microscopie). Bien que les effets de base devraient être reproductibles sur toutes les plates-formes de test, les aspects quantitatifs des effets peuvent varier considérablement.
L’activation par bioluminescence des photorécepteurs a été démontrée jusqu’à présent pour un nombre limité de luciférases et de protéines photosensorielles, respectivement, in vitro et in vivo. Il peut être étendu à la grande classe de photorécepteurs pour activer beaucoup plus de processus biologiques. Une telle expansion de l’approche est encore favorisée par le développement continu de nouvelles luciférases et de paires de protéines de fluorescence de luciférase avec une émission de lumière beaucoup plus élevée que les luciférases naturelles et avec des caractéristiques cinétiques adaptables à différentes applications. Ces progrès sont mis en parallèle avec la génération de nouvelles luciférines, ce qui ajoute encore à une luminosité et à des palettes de couleurs accrues29. Cette plate-forme d’outils offre des applications pour manipuler et étudier la dynamique intracellulaire et les interactions cellulaires à l’intérieur des cellules, des tissus et des organismes vivants.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions nos collègues pour les constructions, en particulier A. Ting pour la protéase Ca-FLARE, le facteur de transcription et le rapporteur (Addgene # 92214, 92213, 92202), H. Kwon pour TM-CIBN-BLITz1-TetR-VP16 et NES-CRY2PHR-TevC (Addgene # 89878, 89877), C. Tucker pour CRY-GalΔDD (B1013) et CIB-VP64 (B1016) (Addgene # 92035, 92037), M. Walsh pour pGL2-GAL4-UAS-Luc (Addgene #33020), K. Gardner pour VP-EL222 et C120-Fluc, et A. Cetin et H. Zeng pour avoir rendu iCreV disponible avant publication. Ce travail a été soutenu par des subventions de NSF (NeuroNex 1707352), NIH (U01NS099709), la Fondation W.M. Keck et le Conseil suédois de la recherche à A.B. (2016-06760).
matériels
| Name | Company | Catalog Number | Comments |
| ABI 25W Deep Red 660 nm LED Light Bulb | Amazon | to be used with any lamp stand | |
| Black Microcentrifuge Tubes, 0.5 mL, Argos Technologies | Fisher Scientific | 03-391-166 | |
| Black Microcentrifuge Tubes, 1.5 mL, Argos Technologies | Fisher Scientific | 03-391-161 | |
| Black Nylon, Polyurethane-Coated Fabric (1.5 m x 2.7 m) x 0.12 mm (thick) | THOR LABS | BK-5 | |
| C120-Fluc | K. Gardner | ||
| CaCl2 | Sigma | C8106; CAS: 10035-04-8 | |
| Ca-FLARE protease, transcription factor and reporter | Addgene # 92214, 92213, 92202 | A. Ting | |
| CIB-VP64 (B1016) | Addgene # 92037 | C. Tucker | |
| CRY-GalΔDD (B1013) | Addgene # 92035 | C. Tucker | |
| CTZ | Prolume Inc. (NanoLight) | 303 | formulation for in vitro applications with Gaussia luciferases |
| CTZ (Water soluble native coelenterazine) | Prolume Inc. (NanoLight) | 3031 | formulation for in vivo applications with Gaussia luciferases |
| D-(+)-Glucose | Sigma | G8270; CAS: 50-99-7 | |
| D-Luciferin, Potassium Salt | Gold Biotechnology | LUCK | |
| DMEM | Thermo Fisher | 11960044 | |
| D-PBS, no calcium, no magnesium | Thermo Fisher | 14190144 | |
| hCTZ | Prolume Inc. (NanoLight) | 301 | formulation for in vitro applications with Oplophorus luciferases |
| HEK293 | ATCC | CRL-1573 | |
| HeLa | ATCC | CCL-2 | |
| HEPES | Sigma | H3375; CAS: 7365-45-9 | |
| iCreV | A. Cetin and H. Zeng | ||
| In Vivo Imaging System (IVIS) | Perkin-Elmer | Lumina LT | |
| KCl | Sigma | P5405; CAS: 7447-40-7 | |
| LED Array Driver | Amuza | LAD-1 | |
| LED Array for Multiwell Plates | Amuza | LEDA-x | |
| Lipofectamine 2000 Reagent | Invitrogen | 11668-019 | Transfection reagent |
| Luminometer | Molecular Devices | SpectraMax L | |
| MgCl2 Hexahydrate | Sigma | M2670; CAS: 7791-18-6 | |
| NaCl | Sigma | S7653; CAS: 7647-14-5 | |
| NanoFuel Solvent | Prolume Inc. (NanoLight) | 399 | for dissolving CTZ preparations for in vitro use |
| NaOH | Sigma | 221465; CAS: 1310-73-2 | |
| NES-CRY2PHR-TevC | Addgene # 89877 | H. Kwon | |
| Opti-MEM | Thermo Fisher | 11058021 | transfection medium |
| PDL coated coverslips (12 mm, 15 mm, 18 mm) | Neuvitro Corporation | GG-12-PDL, GG-15-PDL , GG-18-PDL | |
| pGL2-GAL4-UAS-Luc | Addgene #33020 | M. Walsh | |
| Prizmatix USB Pulser TTL Generator for Optogenetics | Goldstone Scientific | ||
| TM-CIBN-BLITz1-TetR-VP16 | Addgene # 89878 | H. Kwon | |
| TrypLE Express | Gibco | 12604-013 | |
| Vehicle (Water-soluble carrier without CTZ) | Prolume Inc. (NanoLight) | 3031C | control for in vivo applications with CTZ |
| VP-EL222 | K. Gardner |
Références
- Johnson, H. E., et al. The spatiotemporal limits of developmental Erk signaling. Developmental Cell. 40 (2), 185-192 (2017).
- Wang, Y., et al. Photostimulation by femtosecond laser triggers restorable fragmentation in single mitochondrion. Journal of Biophotonics. 10 (2), 286-293 (2017).
- Li, T., et al. A synthetic BRET-based optogenetic device for pulsatile transgene expression enabling glucose homeostasis in mice. Nature Communications. 12 (1), 615(2021).
- Berglund, K., Birkner, E., Augustine, G. J., Hochgeschwender, U. Light-emitting channelrhodopsins for combined optogenetic and chemical-genetic control of neurons. PLoS One. 8 (3), 59759(2013).
- Tung, J. K., Gutekunst, C. -A., Gross, R. E. Inhibitory luminopsins: genetically-encoded bioluminescent opsins for versatile, scalable, and hardware-independent optogenetic inhibition. Scientific Reports. 5, 14366(2015).
- Berglund, K., et al. Luminopsins integrate opto- and chemogenetics by using physical and biological light sources for opsin activation. Proceedings of the National Academy of Sciences of the United States of America. 113 (3), 358-367 (2016).
- Gomez-Ramirez, M., More, A. I., Friedman, N. G., Hochgeschwender, U., Moore, C. I. The BioLuminescent-OptoGenetic in vivo response to coelenterazine is proportional, sensitive and specific in neocortex. Journal of Neuroscience Research. 98 (3), 471-480 (2020).
- Moore, C. I., Berglund, K. BL-OG: BioLuminescent-OptoGenetics. Journal of Neuroscience Research. 98 (3), 469-470 (2020).
- Park, S. Y., et al. Novel luciferase-opsin combinations for improved luminopsins. Journal of Neuroscience Research. 98 (3), 410-421 (2020).
- Jaiswal, P. B., Tung, J. K., Gross, R. E., English, A. W. Motoneuron activity is required for enhancements in functional recovery after peripheral nerve injury in exercised female mice. Journal of Neuroscience Research. 98 (3), 448-457 (2020).
- Zenchak, J. R., et al. Bioluminescence-driven optogenetic activation of transplanted neural precursor cells improves motor deficits in a Parkinson's disease mouse model. Journal of Neuroscience Research. 98 (3), 458-468 (2020).
- Tung, J. K., Shiu, F. H., Ding, K., Gross, R. E. Chemically activated luminopsins allow optogenetic inhibition of distributed nodes in an epileptic network for non-invasive and multi-site suppression of seizure activity. Neurobiology of Disease. 109, Pt A 1-10 (2018).
- Hegemann, P. Algal sensory photoreceptors. Annual Review of Plant Biology. 59, 167-189 (2008).
- Losi, A., Gardner, K. H., Moglich, A. Blue-light receptors for optogenetics. Chemical Reviews. 118 (21), 10659-10709 (2018).
- Proshkina, G. M., Shramova, E. I., Shilova, O. N., Ryabova, A. V., Deyev, S. M. Phototoxicity of flavoprotein miniSOG induced by bioluminescence resonance energy transfer in genetically encoded system NanoLuc-miniSOG is comparable with its LED-excited phototoxicity. Journal of Photochemistry and Photobiology B: Biology. 188, 107-115 (2018).
- Kawano, F., Okazaki, R., Yazawa, M., Sato, M. A photoactivatable Cre-loxP recombination system for optogenetic genome engineering. Nature Chemical Biology. 12 (12), 1059-1064 (2016).
- Shramova, E. I., Proshkina, G. M., Chumakov, S. P., Khodarovich, Y. M., Deyev, S. M. Flavoprotein miniSOG cytotoxisity can be induced by bioluminescence resonance energy transfer. Acta Naturae. 8 (4), 118-123 (2016).
- Naim, N., et al. Luminescence-activated nucleotide cyclase regulates spatial and temporal cAMP synthesis. Journal of Biological Chemistry. 294 (4), 1095-1103 (2019).
- Kim, C. K., Cho, K. F., Kim, M. W., Ting, A. Y. Luciferase-LOV BRET enables versatile and specific transcriptional readout of cellular protein-protein interactions. Elife. 8, 43826(2019).
- Parag-Sharma, K., et al. Engineered BRET-based biologic light sources enable spatiotemporal control over diverse optogenetic systems. ACS Synthetic Biology. 9 (1), 1-9 (2020).
- Kim, E. H., et al. Self-luminescent photodynamic therapy using breast cancer targeted proteins. Science Advances. 6 (37), (2020).
- Kim, C. K., et al. A Molecular calcium integrator reveals a striatal cell type driving aversion. Cell. 183 (7), 2003-2019 (2020).
- Wang, W., et al. A light- and calcium-gated transcription factor for imaging and manipulating activated neurons. Nature Biotechnology. 35 (9), 864-871 (2017).
- Lee, D., Hyun, J. H., Jung, K., Hannan, P., Kwon, H. -B. A calcium- and light-gated switch to induce gene expression in activated neurons. Nature Biotechnology. 35 (9), 858-863 (2017).
- Pathak, G. P., et al. Bidirectional approaches for optogenetic regulation of gene expression in mammalian cells using Arabidopsis cryptochrome 2. Nucleic Acids Research. 45 (20), 167(2017).
- Nishio, H., Walsh, M. J. CCAAT displacement protein/cut homolog recruits G9a histone lysine methyltransferase to repress transcription. Proceedings of the National Academy of Sciences of the United States of America. 101 (31), 11257-11262 (2004).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Yao, S., et al. RecV recombinase system for in vivo targeted optogenomic modifications of single cells or cell populations. Nature Methods. 17 (4), 422-429 (2020).
- Love, A. C., Prescher, J. A. Seeing (and using) the light: Recent developments in bioluminescence technology. Cell Chemical Biology. 27 (8), 904-920 (2020).
- Naumann, E. A., Kampff, A. R., Prober, D. A., Schier, A. F., Engert, F. Monitoring neural activity with bioluminescence during natural behavior. Nature Neuroscience. 13 (4), 513-520 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon