Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Préparation de microcapsules multifonctionnelles à base de soie chargées de plasmides d’ADN codant pour des aptamères d’ARN et des riboswitches
Dans cet article
Résumé
Le protocole décrit la formation de microcapsules chargées d’ADN robustes et biocompatibles sous forme de biocapteurs in vitro multiplexés capables de suivre plusieurs ligands.
Résumé
Nous introduisons un protocole pour la préparation de microcapsules de fibroïne de soie chargées d’ADN via la méthode d’assemblage couche par couche (LbL) sur des noyaux sphériques sacrificiels. Après l’adsorption d’une couche première et de plasmides d’ADN, la formation de microcapsules robustes a été facilitée par l’induction de feuilles de β dans la structure secondaire de la soie lors de la déshydratation aiguë d’une seule couche de soie. Par conséquent, la stratification s’est produite via de multiples liaisons hydrogène et interactions hydrophobes. Lors de l’adsorption de coquilles multicouches, les structures noyau-coquille peuvent être fonctionnalisées davantage avec des nanoparticules d’or (AuNP) et / ou des anticorps (IgG) à utiliser pour la télédétection et / ou la livraison ciblée. L’ajustement de plusieurs paramètres clés lors du dépôt séquentiel de macromolécules clés sur des noyaux de silice, tels que la présence d’un apprêt polymère, la concentration d’ADN et de protéines de soie, ainsi qu’un certain nombre de couches adsorbées, a abouti à des microcapsules biocompatibles chargées d’ADN avec une perméabilité et des charges d’ADN variables. Lors de la dissolution des noyaux de silice, le protocole a démontré la formation de microcapsules creuses et robustes avec des plasmides d’ADN immobilisés sur la surface interne de la membrane de la capsule. La création d’une membrane biocompatible sélectivement perméable entre les plasmides d’ADN et l’environnement externe a préservé l’ADN pendant le stockage à long terme et a joué un rôle important dans l’amélioration de la réponse de sortie des plasmides confinés dans l’espace. L’activité des modèles d’ADN et leur accessibilité ont été testées lors de réactions de transcription et de traduction in vitro (systèmes acellulaires). Les plasmides d’ADN codant pour les aptamères et les riboswitches d’ARN ont été activés avec succès avec les analytes correspondants, comme cela a été visualisé lors de la localisation de transcrits d’ARN marqués par fluorescence ou de la protéine GFPa1 dans les membranes de la coquille.
Introduction
Le domaine de la biologie synthétique offre des opportunités uniques de développer des capacités de détection en exploitant les mécanismes naturels développés par les micro-organismes pour surveiller leur environnement et les menaces potentielles. Il est important de noter que ces mécanismes de détection sont généralement liés à une réponse qui protège ces micro-organismes contre l’exposition nocive, régulant l’expression des gènes pour atténuer les effets négatifs ou empêcher l’absorption de matières toxiques. Des efforts importants ont été déployés pour concevoir ces microorganismes afin de créer des capteurs de cellules entières tirant parti de ces réponses naturelles, mais en les redirigeant pour reconnaître de nouvelles cibles et/ou produire un signal mesurable pouvant être mesuré à des fins de quantification (généralement la fluorescence)1,2. Actuellement, les préoccupations concernant l’utilisation de micro-organismes génétiquement modifiés (OGM), en particulier lorsqu’ils sont disséminés dans l’environnement ou le corps humain, en raison de fuites de cellules entières ou d’une partie de leur matériel génétique, même encapsulées dans une matrice polymère, suggèrent que d’autres moyens d’exploiter ces approches de détection sont nécessaires3.
Une approche puissante pour exploiter les avantages de la détection basée sur les micro-organismes sans se soucier du déploiement d’OGM est l’utilisation de systèmes de transcription/traduction in vitro (IVTT). D’un point de vue pratique, les systèmes IVTT consistent en un mélange contenant la plupart des composants cellulaires à l’état actif qui a été « extrait » des cellules par différents moyens, y compris la sonication, le battement de perles ou autres4. Le produit final de ce procédé est un mélange réactionnel biochimique déjà optimisé pour effectuer la transcription et la traduction qui peut être utilisé pour tester différents capteurs dans un format « récipient ouvert », sans les contraintes liées à l’utilisation de cellules entières (diffusion membranaire, efficacité de transformation, toxicité cellulaire, etc.). Il est important de noter que différents composants de capteurs peuvent être ajoutés quantitativement et leur effet étudié par différentes techniques optiques et spectrométriques, comme nous l’avons démontré5. Il a été remarqué que les performances des systèmes IVTT peuvent être incohérentes; Cependant, des études récentes ont montré des approches pour standardiser leur préparation et leur caractérisation, ce qui est d’une grande aide lors de l’étude de leurs performances dans la conception de capteurs6. Récemment, de nombreux exemples de systèmes IVTT utilisés pour créer des tests sur papier grâce à la lyophilisation de leurs composants dans des matrices papier ont été démontrés, y compris la détection d’ions de métaux lourds, de médicaments, d’éléments de détection de quorum et autres 7,8,9. Un espace d’application passionnant pour les capteurs basés sur IVTT est leur utilisation dans des applications de détection dans différents types d’environnements, y compris le sol, l’eau et le corps humain. Afin de déployer ces systèmes IVTT dans ces environnements difficiles, une approche d’encapsulation doit être mise en œuvre pour contenir les composants IVTT et les protéger de la dégradation.
Les approches d’encapsulation les plus courantes pour les systèmes IVTT comprennent l’utilisation de capsules lipidiques, de micelles, de polymères et d’autres microconteneurs hermétiquement fermés10,11,12. L’un des inconvénients de cette approche est la nécessité d’incorporer des mécanismes passifs ou actifs pour transporter les matériaux à l’intérieur et à l’extérieur des conteneurs afin de permettre la communication avec l’environnement extérieur et de fournir des capacités de détection. Pour surmonter certains de ces problèmes, l’étude présente une méthode qui fournit une approche simple mais efficace pour encapsuler les matériaux de codage pour différentes conceptions de capteurs à exprimer dans les systèmes IVTT. Cette approche est basée sur l’utilisation du dépôt couche par couche (LbL) d’un biopolymère en présence des plasmides d’intérêt pour créer des microcapsules creuses à porosité élevée, ce qui permet au matériel génétique protégé d’interagir avec les différents composants de l’IVTT de choix. L’étude a démontré que les plasmides encapsulés pouvaient diriger la transcription et la traduction lorsqu’ils étaient activés dans cette matrice polymère, comme le montre la réponse d’un aptamère codé par un plasmide et d’un riboswitch à leurs cibles correspondantes. De plus, ce revêtement LbL protège les plasmides pendant des mois sans conditions de stockage particulières.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Construction du vecteur plasmidique.
- Construire un vecteur plasmidique (pSALv-RS-GFPa1, 3,4 kb) par amplification de la séquence codante d’un riboswitch théophylline (ThyRS) couplé à GFPa1 à partir du vecteur pJ201:23976-RS-GFPa1 (conçu et créé par DNA2.0) et insertion dans le vecteur d’expression E. coli, pSAL13. Utiliser des amorces avant (5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3') et inverse (5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') pour amplifier la séquence codante de ThyRS couplée à GFPa1 et effectuer une réaction de PCR dans un volume de 50 μL en utilisant l’ADN polymérase selon le protocoledu fabricant 14.
- Préparer un gel d’agarose à 1 % à partir de 0,5 g d’agarose, 50 mL de tampon TAE (40 mM d’acétate de tris, 1 mM d’EDTA, pH 8,0) et 3 μL de coloration d’ADN.
- Mélanger 5 μL aliquote du produit amplifié par PCR avec 5 μL d’eau exempte de RNase/DNase et 2 μL de colorant de chargement de gel 6x et analyser par électrophorèse sur gel d’agarose. Chargez une échelle d’ADN (0.1-10.0 kb) comme référence. Faites fonctionner le gel à 120 V jusqu’à ce que la ligne de colorant ait presque atteint le fond du gel.
- Visualisez les fragments d’ADN à l’aide d’un système d’imagerie par transilluminateur UV pour vérifier la taille correcte de l’ADN15.
- Purifier le produit PCR à l’aide d’un kit de purification PCR selon le protocole du fabricant16.
- Digérer le produit de PCR et le vecteur d’expression pSAL avec des enzymes de restriction KpnI et BlpI dans une réaction de 15 μL, contenant 10 μL de produit PCR ou de vecteur plasmidique (concentration 20-50 ng / μL), 1,5 μL de tampon enzymatique 10x, 1 μL de chaque enzyme et 1,5 μL d’eau exempte de RNase/DNase, à 37 °C pendant 2 h.
- Ajouter 3 μL de colorant de charge de gel 6x au mélange réactionnel et séparer les fragments digérés sur un gel d’agarose à 1% comme décrit aux étapes 1.3-1.5.
- Purifier les fragments d’ADN à l’aide d’un kit d’extraction de gel selon le protocole16 du fabricant.
- Lilifier le produit PCR digéré en un vecteur plasmidique linéarisé digéré, pSAL, en utilisant l’ADN ligase T4 et un tampon ligase complété dans une réaction de 10 μL contenant 3-20 fmol du vecteur digéré, 9-60 fmol du produit PCR digéré, 2 μL de tampon ligase, 1 μL (1 unité) de T4 DNA ligase, et de l’eau sans DNase/RNase. Incuber la réaction de ligature à 25 °C pendant 3 h.
REMARQUE : S’assurer que la teneur totale en ADN du mélange réactionnel est de 0,01 à 0,1 μg. - Transformer les cellules compétentes en E. coli DH5α avec 10 ng du mélange réactionnel de ligature selon le protocole du fabricant17.
- Faire pousser les cellules transformées à 37 °C pendant une nuit sur des plaques de LB-agar supplémentées en ampicilline (100 μg/mL).
- Prélever 3-4 colonies bactériennes dans la plaque et transférer de manière aseptique chacune d’elles dans 5 mL de milieu LB complété par de l’ampicilline (100 μg/mL). Cultiver les cultures pendant la nuit à 37 °C dans un incubateur à agitation à 225 tr/min.
- Ensemencer les cultures de nuit par centrifugation à 11 x g pendant 3 min à température ambiante.
- Utilisez un kit de purification pour purifier les plasmides selon le protocole16 du fabricant.
- Vérifier les séquences des plasmides purifiés par séquençage de l’ADN. La carte plasmidique et la séquence de la construction résultante (pSALv-RS-GFPa1) sont illustrées à la figure 1.
2. Purification de l’ADN à grande échelle.
- Transformer le vecteur plasmidique pSALv-RS-GFPa1 (3,4 kb) (codant pour le riboswitch théophylline couplé au gène rapporteur GFPa1) ou pET28c-F30-2xBrocoli (5,4 kb) (codant pour Broccoli aptamère) en cellules compétentes E. coli DH5α selon le protocoledu fabricant 17.
- Cultiver les cellules transformées à 37 °C pendant une nuit sur des plaques de LB-agar supplémentées en ampicilline (100 μg/mL) pour les cellules transformées avec pSALv-RS-GFPa1 ou kanamycine (50 μg/mL) pour les cellules transformées avec du brocoli pET28c-F30-2x.
- Prélever 3 à 4 colonies bactériennes dans la plaque et transférer de façon aseptique chaque colonie dans 5 mL de milieu LB additionné d’un antibiotique approprié (100 μg/mL d’ampicilline ou 50 μg/mL de kanamycine). Cultiver les cultures pendant la nuit à 37 °C dans un incubateur à agitation à 225 tr/min.
- Utiliser 3 mL de la culture de nuit pour inoculer 150 mL de LB complétée par un antibiotique approprié (100 μg/mL d’ampicilline ou 50 μg/mL de kanamycine) et cultiver les cultures pendant la nuit à 37 °C dans un incubateur à agitation à 225 rpm.
- Enduire les cellules par centrifugation à ≥3400 x g pendant 10 min à 4 °C.
- Utilisez un kit de purification pour purifier les plasmides selon le protocole16 du fabricant.
- Éluer l’ADN avec 0,5 mL d’eau pure sans DNase/RNase. Mesurer la concentration d’ADN et préparer 1 mL de solutions mères d’ADN (100 ng/μL). Conserver les tubes avec de l’ADN à 4 °C jusqu’à une utilisation ultérieure.
3. Extraction de la fibroïne de soie et préparation des matériaux initiaux.
- Préparer une solution aqueuse de protéine de fibroïne de soie (SF) reconstituée à partir de cocons de vers à soie Bombyx mori selon le mode opératoire décrit en détail ailleurs pour représenter 10% de la solution Silk-LiBr18.
- Déterminer la concentration finale de la solution aqueuse de SF. Pipeter 0,5 ml de solution de soie sur une boîte de Petri de 60 mm, laisser sécher à 60 °C et mesurer le poids d’un film de soie sec. Diviser le poids sec par 0,5 mL pour calculer le pourcentage de poids par volume.
- Diluer la solution de soie concentrée avec de l’eau distillée sans DNase/RNase en ajoutant lentement de l’eau à l’aide d’une pipette sérologique pour obtenir une concentration finale de 1 mg/mL. Conservez la solution à 4 °C pour une utilisation ultérieure.
- Préparez de la fibroïne de soie marquée par fluorescence à l’aide d’un kit d’étiquetage des anticorps. Utilisez 1 mL de solution de fibroïne de soie à 2 mg/mL pour coupler les groupes α-aminés N-terminaux de la protéine avec un colorant dérivé activé par l’ester NHS selon le protocole19 du fabricant.
- Préparer 50 mL de solution aqueuse de polyéthylèneimine (PEI) à une concentration de 6 mg/mL, ajuster le pH à 4 avec HCl (1 M). Filtrer la solution à travers une membrane stérile de 0,2 μm. Le stockage est possible dans des conditions ambiantes pendant des mois.
- Préparez les cœurs SiO2 . Pipeter 300 μL de particules de SiO 2 dans un tube microcentrifuge de2 mL. Laver les microparticules deux fois avec 1 mL d’eau exempte de DNase/RNase par centrifugation à 0,2 x g pendant 1 min.
4. Effectuez le dépôt couche par couche d’une couche première, de plasmides d’ADN et de couches de soie.
- Pour déposer la couche principale de PEI sur les microparticules de SiO2 , ajouter 1 mL de solution de PEI à la pastille filée de l’étape 3.6 et agiter le mélange aux conditions ambiantes sur un thermomélangeur à 800 tr/min pendant 15 min. Laver les particules quatre fois avec 1 mL d’eau désionisée exempte de DNase/RNase par centrifugation à 0,2 x g pendant 1 min.
- Pour effectuer le dépôt de la couche d’ADN, ajouter 1 mL de la solution aqueuse de plasmides d’ADN de l’étape 2.7 aux microparticules amorcées par le PEI et agiter doucement le mélange à 4 °C sur un thermomélangeur à 800 rpm pendant 15 min. Pour préparer des microcapsules avec différentes charges d’ADN, ajuster la concentration de plasmides d’ADN de 50 à 200 ng / μL en utilisant de l’eau distillée sans DNase / RNase et utiliser 1 mL de ces solutions pour déposer l’ADN. Recueillir les microparticules par centrifugation à 0,2 x g pendant 1 min.
- Marquez les tubes pour les plasmides d’ADN codant pour le riboswitch théophylline couplé avec GFPa1 comme ThyRS-GFPa1, et les plasmides d’ADN codant pour l’aptamère de brocoli comme BrocApt.
REMARQUE: Gardez les tubes microcentrifugés avec de l’ADN sur de la glace. - Retirez délicatement le surnageant et lavez les microparticules quatre fois avec 1 mL d’eau distillée sans DNase/RNase, en jetant chaque fois le surnageant après centrifugation à 0,2 x g pendant 1 min. Effectuer toutes les expériences à température ambiante (RT), sauf indication contraire.
- Pour effectuer le dépôt de la couche de fibroïne de soie, ajouter 1 mL de la solution aqueuse de SF reconstituée de l’étape 3.3 aux microparticules adsorbées par l’ADN, tourbillonner doucement et agiter le mélange à 10 °C sur le thermomélangeur à 750 rpm pendant 15 min. Recueillir les microparticules par centrifugation à 0,2 x g pendant 1 min à 4 °C, retirer le surnageant, puis les laver une fois avec 1 mL d’eau distillée sans DNase/RNase. Répétez la centrifugation et jetez le surnageant.
REMARQUE: Pendant l’expérience, gardez la solution de soie sur de la glace pour éviter la gélification induite par la température. - Traiter progressivement les particules avec du méthanol pour induire la formation de feuilles de β dans la structure des protéines de soie. Tout d’abord, ajoutez 0,5 mL d’eau distillée DNase/RNase, faites tourbillonner le tube microcentrifugeur, puis ajoutez 0,5 mL de méthanol à 100 %. Agiter doucement les particules sur le thermomélangeur à 10 °C pendant 5 min. Recueillir les particules par centrifugation à 0,2 x g pendant 1 min. Retirez le surnageant.
- Traiter les particules avec du méthanol pour favoriser la formation de feuilles de β et assurer une forte adsorption physique de la couche de soie. Ajouter 1 mL de méthanol à 100 %. Agiter doucement les particules sur le thermomélangeur à 750 tr/min pendant 10 min à 10 °C.
- Recueillir les particules par centrifugation à 0,2 x g pendant 1 min à 4 °C et les laver deux fois avec 1 mL d’eau distillée sans DNase/RNase à chaque fois, en jetant le surnageant et en tourbillonnant doucement avant la prochaine centrifugation.
- Répétez les étapes 4,5-4,8 20 fois pour obtenir des structures noyau-coque multicouches en soie. Pour la dernière étape de dépôt, utilisez de la soie marquée par fluorescence à partir de l’étape 3.4 (Silk-DyLight550, 1 mL).
- Effectuez la dernière étape de lavage et conservez les microparticules dans 1 mL d’eau distillée exempte de DNase/RNase aux conditions ambiantes.
NOTE: Pour éviter l’agrégation des particules pendant le dépôt des couches de soie, effectuez une inspection visuelle de la suspension de particules et pipetez-la de haut en bas à l’aide d’une pointe de pipette de 1 mL pour favoriser une distribution homogène des particules. - Calculer le nombre de copies de plasmides d’ADN encapsulées dans chaque microcapsule, ADN N en utilisant l’équation 1:
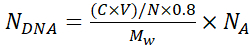 (1)
(1)
Où N = 6,769 × 1011 - le nombre de cœurs SiO2 utilisés pour l’encapsulation. Calculer à partir d’une courbe étalon pour les concentrations connues de particules de silice en utilisant des dilutions en série et l’absorption A 320 à λ =320 nm;
C- concentration initiale de l’ADN utilisé pour l’adsorption
V- le volume d’ADN utilisé pour l’adsorption
0.8- Efficacité de l’adsorption de l’ADN sur les cœurs
Mw- Poids moléculaire du plasmide d’ADN
NA- Nombre d’Avogadro (6.022 × 1023)
5. Dissolution des noyaux pour obtenir des microcapsules de soie.
- Préparer une solution d’acide fluorhydrique (HF) à 8 %, pH 5,5, en diluant la solution mère (48 %) avec de l’eau distillée. Acquérir un tube à centrifuger de 50 mL. Pipeter soigneusement 5 mL de HF et ajouter 25 mL d’eau distillée pour obtenir une solution HF à 8%.
ATTENTION : L’HF est un acide hautement corrosif et peut causer de graves brûlures aux tissus. Une extrême prudence doit être prise lors de la manipulation et de l’utilisation de HF pour les expériences. Respecter la procédure opérationnelle normalisée (SOP) pour l’utilisation et la manipulation appropriées des HF développées par l’organisation afin d’éviter les accidents indésirables de déversement. N’utilisez pas de récipients en verre pour diluer l’acide HF. Utilisez la hotte chimique pour effectuer cette étape du protocole. - Dissoudre les noyaux SiO2 en ajoutant 1,5 mL de solution HF à 8 % aux microparticules noyau-enveloppe granulées à partir de l’étape 4.10. Tourbillonner doucement et laisser les noyaux se dissoudre pendant la nuit dans des conditions ambiantes avec une légère agitation à 450 tr/min.
REMARQUE: Pour éviter le déversement de HF, utilisez du ruban de greffage pour sceller le tube de microcentrifugeuse. Utilisez une hotte chimique pour effectuer cette étape du protocole. - Préparez un bécher en verre de 2 L rempli de 2 L d’eau désionisée. Transférer la solution de microcapsules dans des appareils de dialyse (50 kDa MWCO) et les dialyser contre de l’eau désionisée avec un changement répété de l’eau toutes les 3 heures pendant les 3 prochains jours.
NOTE: Recueillir le surnageant lors des trois premiers échanges d’eau et jeter la solution conformément au protocole établi pour les déchets dangereux. - Utilisez une pipette de 1 mL pour transférer la suspension des appareils de dialyse dans de nouveaux tubes microcentrifugeuses de 2 mL pour recueillir les microcapsules.
NOTE: Conservez les solutions aqueuses de microcapsules aux conditions ambiantes pendant plusieurs années.
6. Imagerie de microcapsules de fibroïne de soie à l’aide d’un microscope confocale à balayage laser (CLSM).
- Effectuer la coloration de l’ADN à l’aide d’un colorant à ADN.
- Transférer 300 μL de microcapsules creuses de fibroïne de soie dans un tube microcentrifuge frais de 1 mL. Ajouter 500 μL d’eau distillée sans RNase/DNase.
- Ajouter 5 μL de colorant colorant à l’ADN, brièvement vortex, et incuber à TA pendant 2 h à l’abri de la lumière.
- Effectuer quatre étapes de lavage par centrifugation à 0,1 x g pendant 20 min à 4 °C à chaque fois, en retirant soigneusement 400 μL du surnageant et en le reconstituant avec 400 μL d’eau distillée sans RNase/DNase.
- Réaliser l’imagerie de capsules de soie sur des systèmes confocaux inversés équipés de trois lasers majeurs (405 nm, 488 nm, 561 nm) en utilisant un objectif d’immersion dans l’huile 100x (NA 1.49). Transférer 100 μL de l’échantillon de capsule dans un seul puits de lames de verre chambrées à 8 puits, permettre aux capsules de sédimenter pendant 20 à 30 minutes avant l’imagerie.
NOTE: Les colorants sont très sensibles au photoblanchiment. Protégez les échantillons en recouvrant les lames de papier d’aluminium.
7. Estimation de la perméabilité des microcapsules creuses à l’aide de la méthode de seuil de masse moléculaire (MWCO).
- Préparer 2 mL chacune de solutions de fluorophore de dextran marquées FITC (20 μM, diH 2 O) de différents Mw (4 kDa, 20 kDa, 40 kDa, 70 kDa, 150 kDa, 250 kDa, 500 kDa et2MDa).
- Pipeter 100 μL de la suspension de capsules dans un seul puits d’une lame de verre chambrée. Analysez chaque conception de microcapsule (concentration de PEI, nombre de plasmides d’ADN de charge, concentration de fibroïne de soie et nombre de couches) séparément.
- À chaque puits, ajouter 300 μL de la solution spécifique de fluorophore en partant du Mw le plus bas jusqu’au plus élevé, de sorte que chaque puits corresponde à la solution spécifique de fluorophore. Mélanger en pipetant de haut en bas et laisser le mélange incuber pendant 1 h à TA jusqu’à ce que la diffusion des solutions de fluorophores atteigne l’équilibre.
- Transférer la lame dans un microscope confocal à balayage laser (CLSM) et imager chaque puits à l’aide d’un objectif d’immersion dans l’huile 100x à l’excitation λ = 488 nm.
- Identifiez la zone d’intérêt en ajustant le plan focal pour vous assurer que les capsules apparaissent sous forme de cercles du plus grand diamètre. Cela se produit généralement lors de la visualisation des échantillons plus près du fond du puits lorsque les capsules sédimentent en raison de la gravité.
- Recueillir plusieurs images d’échantillons de microcapsules en déplaçant la lame dans la direction XY. Capturez des images pour représenter jusqu’à 100-150 capsules pour chaque échantillon.
- Utilisez le logiciel ImageJ pour analyser la perméabilité de la membrane de la capsule dans chaque solution de fluorophore Mw en comparant les intensités de fluorescence à l’intérieur et à l’extérieur des capsules. Pour cela, dessinez une région d’intérêt (ROI) sous la forme d’un cercle pour délimiter la circonférence de la capsule et cliquez sur Analyser / Mesurer pour mesurer l’intensité de fluorescence à l’intérieur. Tabulez les données dans une feuille de calcul. Effectuez cette opération pour chaque microcapsule pour un total de 200-300 capsules.
- Évaluez l’intensité de fluorescence extérieure de la même manière en décrivant le retour sur investissement et en mesurant l’intensité loin des capsules. Effectuez 3 à 5 mesures pour l’analyse statistique.
- Pour effectuer une analyse statistique, comparez les intensités de fluorescence à l’intérieur et à l’extérieur des capsules à l’aide du test t apparié (p < 0,05).
- Utiliser le tableau de conversion 2 pour estimer la perméabilité des microcapsules en fonction des rayons hydrodynamiques pour FITC-Dextran avec variable Mw.
8. Activation in vitro du riboswitch synthétique de théophylline dans des microcapsules de soie
- Préparer 1 mL de solution mère de théophylline (100 mM, DMSO). Préparer le système d’extrait d’E. coli S30 pour l’ADN circulaire en décongelant les composants sur la glace pendant 40 min.
- Procurez-vous un tube microcentrifuge sans DNase/RNase de 0,5 mL. Effectuer une réaction de transcription/traduction in vitro , combiner les composants acellulaires avec un échantillon de microcapsules dans l’ordre suivant (volume total de 50 μL) : Prémélange S30 sans acides aminés (20 μL) ; Extrait de S30, circulaire (15 μL); mélange complet d’acides aminés (5 μL); microcapsules creuses contenant des plasmides ThyRS-GFPa1 de l’étape 4.10 (9 μL); et théophylline, 100 mM de DMSO (1 μL).
REMARQUE: Après avoir ajouté tous les composants, vortex brièvement le tube et prélever l’échantillon pendant une brève centrifugation à 0,2 × g pendant quelques secondes. - Incuber le tube à 30 °C pendant 4 h et vérifier la fluorescence sur un lecteur de plaques en utilisant l’excitation à λ = 488 nm et l’émission pour le filtre GFP/FITC (510 nm ± 20 nm).
- Imagez les capsules sur n’importe quel système LCSM à l’aide de lasers 488 nm et 561 nm. Obtenez des images de la meilleure qualité en utilisant un objectif d’immersion dans l’huile 100x et des diapositives chambrées à 8 puits.
9. Activation in vitro de l’aptamère de brocoli dans des microcapsules de soie
- Préparer 1 mL de solution mère de colorant DFHBI-1T (30 μM,diH2O). Préparer le kit de réaction du système acellulaire PURE (synthèse des protéines à l’aide d’éléments recombinants) en décongelant les composants sur de la glace pendant 40 min.
- Procurez-vous un tube microcentrifuge sans DNase/RNase de 0,5 mL. Effectuer une réaction de transcription in vitro en combinant des composants de réaction acellulaires avec un échantillon de microcapsules dans l’ordre suivant (volume total de 50 μL) : solution A (20 μL); solution B (15 μL); microcapsules creuses contenant des plasmides BrocApt de l’étape 4.10 (14 μL); et colorant DFHBI-1T (1 μL).
REMARQUE: Après avoir ajouté tous les composants, vortex brièvement le tube et prélever l’échantillon pendant une brève centrifugation à 0,2 × g pendant quelques secondes. - Incuber le tube à 37 °C pendant 6 h et vérifier la fluorescence sur un lecteur de plaques en utilisant l’excitation à λex = 470 nm et l’émission à λem = 510 nm ± 20 nm.
- Imagez les capsules sur n’importe quel système LCSM à l’aide de lasers 488 nm et 561 nm. Obtenez des images de la meilleure qualité en utilisant un objectif d’immersion dans l’huile 100x et des lames chambrées en verre couvercle à 8 puits.
Access restricted. Please log in or start a trial to view this content.
Résultats
Ici, l’étude aborde la fonctionnalité des modèles d’ADN codant différents modèles de capteurs (deux types d’éléments de transcription / traduction régulés par l’ARN) après encapsulation dans des capsules de protéines de soie. Les microcapsules ont été préparées au moyen d’un modèle d’assemblage couche par couche (LbL) des composants clés : une couche primaire, des plasmides d’ADN codant pour des conceptions de capteurs et un biopolymère de fibroïne de soie (Figure 2
Access restricted. Please log in or start a trial to view this content.
Discussion
Des microcapsules d’hydrogel sélectivement perméables chargées de différents types de conceptions de capteurs codés par l’ADN peuvent être préparées en suivant ce protocole. L’une des caractéristiques distinctives de l’approche LbL est la capacité d’adapter la complexité des microcapsules lors de l’assemblage ascendant, qui commence généralement par l’adsorption d’espèces moléculaires sur des modèles sacrificiels. En ajustant soigneusement les concentrations des composants initiaux, les co...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les points de vue et opinions présentés ici sont ceux des auteurs et ne représentent pas nécessairement les points de vue du DoD ou de ses composantes.
Remerciements
Ce travail a été soutenu par la subvention LRIR 16RH3003J du Bureau de la recherche scientifique de l’armée de l’air, ainsi que par le programme Synthetic Biology for Military Environments Applied Research for the Advancement of S & T Priorities (ARAP) du Bureau du sous-secrétaire à la Défense pour la recherche et l’ingénierie des États-Unis.
La séquence plasmidique vectorielle de ThyRS (pSALv-RS-GFPa1, 3,4 kb) a été généreusement fournie par le Dr J. Gallivan. Les cocons de vers à soie de Bombyx mori ont été généreusement donnés par le Dr D.L. Kaplan de l’Université Tufts, MA.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
Références
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940(2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027(2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469(2014).
- Frey, B. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. Reischl, U. 13, Humana Press. Totowa, NJ. 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923(2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870(2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466(2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504(2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046(2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707(2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283(2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon