Method Article
Production efficace et évolutive de variantes de huntingtine humaine pleine longueur dans des cellules de mammifères à l’aide d’un système d’expression transitoire
Dans cet article
Résumé
Nous fournissons des protocoles évolutifs couvrant la conception de construction, la transfection transitoire, l’expression et la purification de variantes complètes de la protéine huntingtine humaine dans les cellules HEK293.
Résumé
La huntingtine pleine longueur (FL HTT) est une grande protéine (aa 1-3 144), exprimée de manière ubiquitaire, contenant de la polyglutamine (polyQ) avec une masse d’environ 350 kDa. Bien que la fonction cellulaire de FL HTT ne soit pas entièrement comprise, une expansion mutante du tractus polyQ au-dessus de ~36 répétitions est associée à la maladie de Huntington (MH), la longueur polyQ étant à peu près corrélée avec l’âge d’apparition. Pour mieux comprendre l’effet de la structure sur la fonction du HTT mutant (mHTT), de grandes quantités de la protéine sont nécessaires. La production submilligramme de FL HTT dans des cellules de mammifères a été réalisée en utilisant l’expression de lignées cellulaires stables inductibles par la doxycycline. Cependant, la production de protéines à partir de lignées cellulaires stables présente des limites qui peuvent être surmontées par des méthodes de transfection transitoire.
Cet article présente une méthode robuste pour la production en faible quantité de milligrammes de FL HTT et de ses variantes à partir de plasmides optimisés pour le codon par transfection transitoire à l’aide de polyéthylènemine (PEI). La méthode est évolutive (>10 mg) et donne constamment 1 à 2 mg/L de culture cellulaire de FL HTT hautement purifié. Conformément aux rapports précédents, l’état de la solution purifiée de FL HTT s’est avéré très dynamique; La protéine a une propension à former des dimères et des oligomères d’ordre élevé. Une clé pour ralentir la formation d’oligomères est de travailler rapidement pour isoler les fractions monomères des fractions dimériques et oligomères d’ordre élevé pendant la chromatographie d’exclusion de taille.
La chromatographie d’exclusion de taille avec diffusion de la lumière multiangle (SEC-MALS) a été utilisée pour analyser le dimère et la teneur oligomérique d’ordre supérieur du HTT purifié. Aucune corrélation n’a été observée entre la longueur du polyQ FL HTT (Q23, Q48 et Q73) et la teneur en oligomères. La construction exon1-supprimée (aa 91-3,144) a montré une propension à l’oligomérisation comparable à FL HTT (aa 1-3,144). Les méthodes de production, de purification et de caractérisation par indice de réfraction SEC/MALS (IR), électrophorèse sur gel de dodécylsulfate-polyacrylamide de sodium (SDS-PAGE), Western blot, Native PAGE et Blue Native PAGE sont décrites ici.
Introduction
La maladie de Huntington (MH) est une maladie neurodégénérative rare caractérisée principalement par des mouvements moteurs instables et involontaires, ainsi que par des altérations cognitives et psychiatriques, telles que des changements de personnalité et de l’apathie 1,2. La MH est associée à une expansion du tractus de répétition CAG situé dans l’exon 1 du gène huntingtin (HTT) à plus de 35 répétitions, avec un nombre plus élevé de répétitions CAG corrélées à une apparition plus précoce de la maladie 3,4. Le produit translationnel de HTT, la protéine huntingtine (HTT), est impliqué dans la viabilité neuronale et le développement du cerveau 5,6,7,8,9.
HTT est une protéine d’échafaudage qui participerait à un large éventail de processus cellulaires, au transport des vésicules, à la division cellulaire, à la ciliogenèse et à l’autophagie10,11. Cependant, la pathogenèse moléculaire de la MH n’est pas tout à fait claire, et l’identification des interacteurs protéiques clés médiant l’impact pathologique du mHTT expansé polyQ fait défaut. Certaines recherches suggèrent un gain de fonction toxique du mHTT entraîné par la propension à l’oligomérisation de la protéine HTT expansée, car des agrégats HTT ont été identifiés dans les neurones et la glie chez les patients MH et les modèles animaux de la maladie 12,13,14,15,16,17 . Pour alimenter l’étude de la fonction et de la structure des variantes FL HTT et mHTT et fournir aux chercheurs des normes protéiques de haute qualité pour le développement de tests, un approvisionnement robuste et évolutif de protéines recombinantes homogènes est nécessaire.
En raison de sa taille (aa 1-3 144, numérotation basée sur la longueur polyQ Q23), de l’instabilité protéolytique et de sa propension à s’agréger, FL HTT s’est avéré difficile à exprimer et à isoler en tant que protéine soluble. Auparavant, la région de l’exon 1 (aa 2-90) du HTT a été exprimée et purifiée à grande échelle à l’aide de divers marqueurs qui peuvent augmenter la solubilité de la protéine dans Escherichia coli18,19,20. FL HTT a d’abord été exprimé et purifié dans un système d’expression cellulaire d’insecte utilisant le baculovirus 21,22, et des structures de microscopie électronique (EM) 30 Å à basse résolution de FL Q23-HTT et Q78-HTT réticulées chimiquement ont été signalées23. L’étude de la structure du HTT a encore progressé lorsque la production de FL Q17, Q46 et Q128-HTT avec des modifications post-traductionnelles natives (PTM) a été réalisée dans des cellules humaines utilisant des lignées cellulaires stables ou des systèmes d’expression d’adénovirus24. Ces études suggèrent que, bien que le HTT purifié existe principalement à l’état monomère, il tend également à former des oligomères et des agrégats d’ordre élevé.
L’ultracentrifugation analytique du FL Q128-HTT, avec une région polyQ fortement expansée, a permis d’obtenir plus de fractions oligomères et agrégées que la protéine avec la région polyQ non expansée24. En utilisant une lignée cellulaire stable, une stratégie a été adaptée avec succès pour stabiliser FL HTT par co-expression avec le partenaire d’interaction HAP40. Une structure cryo-EM du complexe FL HTT et HAP40 a été résolue à une résolution moyenne de 4 Å en utilisant le complexe protéique purifié (PDB:6EZ8)25. Cette stratégie de co-expression a été adaptée avec succès à un système de baculovirus, et une série de variantes HTT de haute qualité avec différentes longueurs de polyQ ont été exprimées et purifiées à partir de cellules d’insectes26. Depuis lors, d’autres structures cryo-EM du complexe de HTT avec des longueurs polyQ variables et des structures HAP40 et de résolution supérieure ont été résolues et déposées dans la base de données sur les protéines27,28 (PDB: 7DXK, 7DXH, 6X9O).
Nous avons optimisé une méthode de transfection et d’expression dans les cellules HEK293, en utilisant la polyéthylènemine (PEI), pour une expression transitoire rapide de FL HTT. Comme preuve de principe, les variantes FL HTT contenant 23 glutamines (FL Q23-HTT) ont d’abord été purifiées et caractérisées à l’aide d’une modification d’une méthode de purification décrite précédemment24. Cette méthode de transfection transitoire est pratique, très efficace et évolutive ; il peut produire du HTT purifié avec des rendements de 1-2 mg/L, comparables à la méthode de la lignée cellulaire stable rapportée24. Parce que la protéine est produite dans une lignée cellulaire humaine, le HTT produit est plus susceptible d’avoir des PTM humains natifs lorsqu’il est soumis à une analyse protéomique par spectrométrie de masse 11,29,30,31. Des quantités de milligrammes des variantes FL Q48-HTT, FL Q73-HTT et exon1-deleted (ΔExon1-HTT) de FL HTT ont été produites, démontrant que la méthode d’expression transitoire est particulièrement utile pour produire rapidement des variantes alternatives de HTT sans dépendre de l’effort fastidieux requis pour établir des lignées cellulaires stables pour la production.
Le protocole suivant illustre la méthode standard utilisée dans le laboratoire de ces auteurs pour la culture cellulaire, la transfection, la purification des protéines et la caractérisation des protéines post-purification pour produire FL Q23-HTT à partir d’une culture cellulaire de 2 L. Le protocole peut être étendu à des cultures plus grandes ou adapté pour purifier d’autres variantes HTT. Jusqu’à 10 L de cultures cellulaires de FL HTT et diverses mutations de site ou de troncature d’homologues HTT et HTT ont été réalisées avec succès en laboratoire en utilisant le même protocole. Le FL HTT purifié contient un pourcentage élevé de monomères ainsi que des dimères et des oligomères d’ordre supérieur. Le même profil agrégé est observé parmi les variantes produites (Q23, Q48, Q73 et Exon1 supprimé). Comme l’agrégation peut se produire lorsque les précautions appropriées ne sont pas prises, une étude de la formulation et de la stabilité au gel-dégel a été menée pour identifier les meilleures conditions pour la manipulation des protéines. Des méthodes, telles que Blue Native PAGE et SEC/MALS-RI, sont également décrites pour analyser la teneur en oligomères HTT dans le cadre du processus de contrôle de la qualité. Au profit de la communauté de recherche MH, les plasmides et les protéines HTT décrits dans cette étude sont également déposés dans le référentiel communautaire MH de l’Institut Coriell (www.coriell.org/1/CHDI).
Protocole
1. Conception et production de constructions pour l’expression des mammifères HTT marqués FLAG
- Récupérez la séquence complète de la protéine HTT humaine (P42858) du National Center for Biotechnology Information (https://www.ncbi.nlm.nih.gov/).
REMARQUE: Les chercheurs doivent être familiers avec les organisations du domaine de HTT et maintenir la structure 3D du noyau HTT lors de la conception de constructions pour les mutants HTT. - Demander un service de synthèse de gènes pour effectuer l’optimisation du codon pour l’expression cellulaire humaine basée sur la séquence de P42858. Changez le nombre polyQ de Q16 à la longueur Q souhaitée (Q23 a été choisi comme première construction ici) et synthétisez le gène HTT pleine longueur.
REMARQUE : La construction Q23-HTT pleine longueur optimisée pour les codons synthétisés a été livrée sous forme d’insertion dans le plasmide pUC18 dans cette étude. - Facultatif : Ajoutez des fonctionnalités pour faciliter le clonage de différentes longueurs Q et la purification dans les constructions.
REMARQUE : Un site de clivage du virus de la gravure du tabac (TEV) et une étiquette de purification FLAG (AAAENLYFQGDYKDDDDK) ont été ajoutés à l’extrémité C-terminale des constructions. Deux sites HindIII ont été conçus dans les constructions pour englober la région polyQ (la séquence protéique traduite n’est pas modifiée par l’introduction de sites HindIII). Cela permet au chercheur de modifier la longueur Q de HTT par restriction de la digestion et de la ligature des enzymes sans resynthétiser le gène HTT complet.
2. Clonez les constructions HTT synthétisées dans pcDNA3.1.
- Condenser 5 μg de pUC18-Q23-HTT et 5 μg de pcDNA3.1 en utilisant 2 μL de NheI et de PmeI chacun à 37 °C pendant 2 h.
- Exécutez un gel d’agarose à 0,5% p/v et purifiez le fragment de Q23-HTT et le vecteur pcDNA3.1 digéré à l’aide d’un kit d’extraction de gel d’agarose. Quantifier les concentrations d’ADN purifié par OD280 à l’aide d’un spectromètre UV capable de mesurer les microlitres d’échantillons.
NOTE: OD260/280 allant de 1,8 à 2,0 est généralement observé. Le FL HTT synthétisé est fourni sous forme d’insert avec NheI et PmeI aux deux extrémités dans un plasmide pUC18. Utilisez d’autres enzymes de restriction si HTT est synthétisé différemment. - Utilisez 10 ng de vecteur pcDNA3.1 digéré dans la réaction. Litruer les ADN purifiés à un rapport molaire de 1:1 (HTT:pcDNA3.1) dans une réaction de 10 μL à température ambiante pendant 5 min en utilisant l’ADN ligase T4.
- Transformer le produit ligaturé en cellules E. coli compétentes (voir le tableau des matériaux) en utilisant le protocole spécifié par le fabricant de la ligase.
- Prélever 6 colonies simples et faire des cultures de nuit dans 4-6 mL de LB complétés par 100 μg/mL de carbénicilline à 37 °C.
- Allouer 1 mL de chaque culture de nuit. Ajouter du glycérol à 25% v/v et conserver le stock de glycérol à -80 °C. Purifiez la culture restante pendant la nuit à l’aide d’un kit de mini-préparation selon les étapes spécifiées dans le manuel de l’utilisateur.
- Séquencer tous les plasmides à l’aide d’amorces de séquençage couvrant la région de transcription du plasmide. Choisissez un stock de glycérol qui a la bonne séquence comme stock de glycérol principal et jetez le reste.
- Facultatif : Demandez un service de synthèse de gènes pour synthétiser l’ADN avec les différentes longueurs Q (Q48, Q73 et Exon1) qui couvrent les deux sites HindIII dans le plasmide pcDNA3.1-Q23-HTT. Digérer pcDNA3.1-Q23-HTT et les ADN nouvellement synthétisés à l’aide de HindIII, et les relier avec la ligase T4 comme dans les étapes 2.2-2.7 pour fabriquer FL HTT avec différentes longueurs polyQ dans le plasmide pcDNA3.1.
NOTE: Les constructions plasmidiques utilisées dans cette étude sont également disponibles directement auprès du HD Community Repository de l’Institut Coriell (www.coriell.org/1/CHDI); voir le tableau des matières.
3. GIGA prépara l’ADN plasmidique exempt d’endotoxines pour la transfection à grande échelle
- Étaler les stocks de glycérol bactérien de pcDNA3.1-Q23-HTT-TEV-FLAG sur une plaque de gélose LB avec de la carbénicilline (100 μg/mL). Incuber la plaque à 37 °C pendant 16-24 h jusqu’à l’apparition de colonies individuelles.
- Prélever une seule colonie, inoculer une culture de démarrage de 5 mL dans un milieu riche formulé pour l’amplification plasmidique avec de la carbénicilline (100 μg/mL) et cultiver à 37 °C pendant 8 h.
- Choisissez un kit de purification plasmidique GIGA sans endotoxines. Suivez les étapes décrites dans le manuel du plasmide GIGA kit pour purifier le plasmide pcDNA3.1-Q23-HTT-TEV-FLAG.
- Mesurer les niveaux d’endotoxines plasmidiques à l’aide d’un kit de quantification d’endotoxines à base de lysat amibocytaire de limulus (LAL). Suivez la procédure spécifiée dans le manuel du fabricant.
REMARQUE: Une purification plasmidique de haute qualité et à faible teneur en endotoxines est essentielle pour obtenir une bonne efficacité de transfection. En utilisant ce protocole, 20 à 40 mg de plasmide (forme superenroulée >80%) peuvent être obtenus par L de culture bactérienne à des concentrations plasmidiques > 4 mg / mL. Un plasmide correctement purifié doit avoir un taux d’endotoxines < 30 EU/mg. OD260/280 allant de 1,8 à 2,0 est généralement observé.
4. Transfection à grande échelle de 2 L de cellules HEK293 par la polyéthylèneimine (PEI)
- Ajouter 1 g de PEI 25K à 1 L d’eau exempte d’endotoxines sous agitation. Ajuster le pH à 2,0 en utilisant 100 mM HCl et remuer jusqu’à ce que tout le PEI 25K se dissolve. Ajuster le pH à 7,0 à l’aide d’une solution de NaOH de 100 mM et filtrer à travers un filtre de 0,2 μm. Aliquote et conserver à -20 °C jusqu’à un an.
REMARQUE : Les substances aliquotes de PEI peuvent être conservées à 4 °C jusqu’à deux semaines, mais ne doivent jamais être recongelées après la décongélation. - Propager des cellules HEK293 dans le milieu de croissance (voir le tableau des matières) supplémentées en pénicilline-streptomycine (concentration finale à 5 U/mL pour la pénicilline et 5 μg/mL pour la streptomycine) dans un incubateur humidifié à 37 °C, 90 tr/min, 5 % de CO2 pendant 18-24 h. Diluer les cellules à 2 L à une densité de ~1,2 × 106 cellules/ml en utilisant le milieu de croissance dans des flacons d’erlenmeyer de 5 L un jour avant la transfection.
- Continuer à cultiver les cellules à 37 °C, 90 tr/min, 5% CO2 pendant 18-24 h. Mesurez les paramètres de la cellule à l’aide d’un compteur automatique de cellules capable de mesurer la densité et la viabilité des cellules en suivant le manuel d’utilisation.
REMARQUE: La densité cellulaire devrait doubler et la viabilité devrait être de >95%. La densité cellulaire avant la transfection devrait être d’environ 2,0 × 10 6-2,4 × 106 cellules/mL. Diluer les cellules à la densité désirée avant la transfection si nécessaire. - Calculer les quantités de plasmide et d’IPE requises pour la transfection; utiliser 1 mg de plasmide et 3 mg de PEI pour la transfection de chaque litre de culture cellulaire. Allouer 2 mg de plasmide et 6 mg de PEI nécessaires pour une transfection de 2 L.
- Diluer le plasmide et le PEI individuellement dans un volume de solution saline tamponnée au phosphate égal à 1/20edu volume total de culture cellulaire (100 mL chacun pour une transfection de 2 L) et incuber à température ambiante pendant 5 min. Mélanger le plasmide dilué et le PEI en remuant doucement et incuber le mélange à température ambiante pendant 30 min.
NOTE: Le mélange apparaîtra légèrement trouble après l’incubation. - Ajouter le mélange à la culture cellulaire et agiter doucement pour les mélanger.
- Faire pousser les cellules à 37 °C, 5% CO2, 90 rpm pendant 24 h.
- Ajouter une solution de butyrate de sodium 2 M jusqu’à une concentration finale de 2 mM. Ajouter un agent antimousse 1:1000 (v/v) et un antimousse 1:1000 (v/v) à la culture.
- Déplacer le ballon dans un incubateur humidifié à 32 °C, 90 tr/min, 5 % de CO2, et continuer à pousser pendant 48 h.
- Mesurez les paramètres de la cellule, y compris la densité et la viabilité des cellules, à l’aide du compteur automatique de cellules suivant le manuel de l’utilisateur.
- Transférer 2,0 × 10 6 cellules (Vol = 2,0 × 106/densité cellulaire) dans un tube microcentrifugeux. Ensemencer les cellules à 2 000 × g pendant 1 min dans une centrifugeuse pour transfert Western dans la section 5.
- Récolter les cellules par centrifugation à 2 000 × g pendant 30 min et conserver la pastille cellulaire à -80 °C avant purification.
5. SDS-PAGE et Western blot de lysat cellulaire HEK293 pour estimer le niveau d’expression de HTT
- Prélever une partie aliquote de 2,0 × 106 cellules préalablement congelées (étape 4.11) de la transfection à grande échelle de la culture cellulaire HEK293. Ajouter 250 μL de solution saline tamponnée Tris (TBS) complétée par 50 μg/mL de digitonine, 5 mM d’EDTA et 1x cocktail d’inhibiteurs de protéase, et remettre en suspension la pastille cellulaire en l’aspirant plusieurs fois à l’aide d’une pipette.
- Faire tourner doucement les tubes pendant 30 min à 4 °C à l’aide d’un minirotateur pour lyser les cellules. Enduire la matière insoluble par centrifugation à 17 000 × g pendant 5 min.
- Ajouter 1/3 du volumedu tampon de charge 4x de dodécylsulfate de lithium (LDS) réducteur au surnageant et chauffer à 70 °C pendant 10 min.
- Charger 5-20 μL de lysat cellulaire sur un gel préfabriqué Tris-acétate PAGE à 3-8%. À l’aide du tampon de fonctionnement SDS 1x tris-acétate compatible avec le gel, faites fonctionner le gel en mode tension constante à 150 V pendant 60 min.
REMARQUE: Le tris-acétate SDS-PAGE a été utilisé pour l’analyse FL HTT car il génère une résolution plus élevée que les autres types de SDS-PAGE pour les protéines de poids moléculaire supérieur à 300 kDa. Les protéines utilisées dans cette étude sont également disponibles directement auprès du HD Community Repository de l’Institut Coriell (www.coriell.org/1/CHDI) ; voir le tableau des matières. - Pour effectuer le transfert Western blot, assemblez un sandwich de transfert à l’aide d’un papier transfert épais équilibré par tampon de transfert, d’une membrane de polyfluorure de vinylidène activé au méthanol (PVDF) et d’un gel SDS-PAGE. Transférer les protéines sur la membrane PVDF à l’aide d’un buvard occidental semi-sec conformément au manuel d’utilisation du fabricant.
NOTE: En règle générale, 20-30 min à 135 mA est suffisant pour une membrane de 10 cm x 10 cm. - Démonter le sandwich de transfert et bloquer la membrane dans du TBST (20 mM Tris pH 7,4, 150 mM NaCl et 0,1 % v/v Tween-20) complété par du lait écrémé à 5 % p/v.
- Incuber la membrane sur une bascule pendant 1 h à température ambiante avec 15 mL d’anticorps primaire (dilution de 1:2 500 pour l’anticorps monoclonal anti-FLAG et 1:2 000 pour tous les autres anticorps primaires).
REMARQUE : Les principaux anticorps utilisés dans cette étude sont les anticorps anti-FLAG M2, MAB5492, MAB5490, MAB2166, MAB3E10, MAB4E10, MAB2168, MAB8A4 (voir le tableau des matériaux). - Laver la membrane 3 x 5 min avec 30-50 mL de TBST.
- Incuber la membrane sur une bascule avec un anticorps secondaire IgG anti-souris conjugué à un colorant fluorescent de chèvre à 1:15 000, température ambiante, dans 15 mL de TBST contenant 5 % p/v de lait en poudre.
- Visualisez les bandes de transfert Western sur un imageur fluorescent en utilisant la longueur d’onde spécifique à l’anticorps secondaire. Quantifier le signal de bande à l’aide du logiciel accompagnant l’imageur conformément au manuel de l’utilisateur.
REMARQUE: Le transfert Western quantitatif peut être effectué en utilisant du HTT purifié comme norme. Une gamme standard linéaire de HTT est spécifique à l’instrument et a été établie dans ce laboratoire de 25 ng à 250 ng de HTT par voie en utilisant un anticorps anti-FLAG. Le transfert Western de HTT doit être exempt de dégradation; un niveau d’expression HTT total de 2-4 pg/cellule est généralement observé. Se reporter au protocole32 publié précédemment pour plus de détails sur la façon d’effectuer un transfert Western quantitatif.
6. Purification rapide par chromatographie liquide protéique (FPLC) de HTT à l’aide d’une colonne anti-FLAG et d’une SEC
- Purification anti-FLAG
- Estimer la quantité de résine FLAG nécessaire à la purification (généralement, 12 mL de résine d’affinité anti-FLAG M2 pour la purification de 2 à 4 L de culture cellulaire transfectée). Emballer 12 à 25 mL de résine anti-FLAG sur une colonne vide (voir le tableau des matériaux) en utilisant FPLC à un débit de 4 mL/min en utilisant le tampon A (tableau 1). Ajustez la hauteur du piston afin qu’il n’y ait pas d’espace entre l’extrémité du piston et le lit de résine.
- En utilisant un rapport de 10 mL de tampon de lyse pour 1 g de pastille de cellule, décongeler et suspendre la pastille de cellule dans un tampon de lyse froid (tableau 1).
- Passez la suspension cellulaire une fois à travers un homogénéisateur à cisaillement élevé à 10 000 psi. Clarifier le lysat par centrifugation à 20 000 × g pendant 1 h dans une centrifugeuse équipée d’un rotor compatible à angle fixe.
- Programmez le FPLC (voir le tableau des matériaux pour le logiciel utilisé dans l’étude) et exécutez les séquences suivantes.
- Chargez le lysat clarifié via la pompe à échantillon.
- Laver avec 4 volumes de colonne (CV) de tampon A (tableau 1).
- Laver avec 4 CV de tampon B (tableau 1).
- Laver avec 8 CV de tampon C (tableau 1).
- Laver avec 3 CV de tampon D (tableau 1).
- Laver avec 3 CV de tampon d’élution (tableau 1).
- Analysez 10 μL des fractions de pic à l’aide de SDS-PAGE. Recueillir et combiner les fractions de pointe avec la pureté désirée. Économisez ~50 μL des éluats combinés pour l’analyse SDS-PAGE.
REMARQUE: Normalement, un seul pic apparaîtra, et toutes les fractions éluées dans le pic contiennent ~ 90% de HTT pur. - Régénérez une colonne anti-FLAG en utilisant 5 CV de Regeneration Buffer (Tableau 1) et rééquilibrez la colonne en utilisant 5 CV de Buffer A.
REMARQUE: La résine anti-FLAG peut être réutilisée jusqu’à cinq fois ou jusqu’à ce que le rendement relatif / litre tombe à 50% de la première purification.
- Purification de la colonne d’exclusion de taille (SEC) à l’aide d’une colonne SEC
- Prééquilibrer une colonne SEC qui permet la séparation de protéines de masse moléculaire (MW) > 500 kDa (voir le tableau des matériaux pour la colonne utilisée) en utilisant 2 ×CV de tampon SEC (tableau 1).
- Charger directement l’éluat anti-FLAG (à partir de l’étape 6.1.5) via une superloop de 50 mL. Exécuter 1,2 × CV de tampon SEC par injection. Exécutez la séparation SEC pendant la nuit à 4 °C.
REMARQUE : Un maximum de 5 mL ou 15 mL d’échantillon de protéines peut être chargé sur les colonnes SEC sélectionnées dans cette étude. Programmez le FPLC afin que plusieurs injections puissent être effectuées automatiquement. Des exemples de scripts de méthode sont également inclus en tant que fichier supplémentaire 1 et fichier supplémentaire 2. - Comparez le profil d’élution avec le profil d’élution HTT standard pour distinguer les pics monomères, dimères et oligomères d’ordre supérieur. Regrouper les fractions HTT monomères en fonction du profil d’élution de la colonne SEC. Si vous le souhaitez, mettez en commun séparément les fractions HTT oligomères et dimères d’ordre supérieur.
- Concentrer la protéine HTT mélangée à l’aide d’un concentrateur centrifuge de 100 kDa à 4 °C. Calculez les concentrations de protéines en divisant leurs valeurs de DO280 par les coefficients d’extinction respectifs (les coefficients d’extinction théoriques de Q23-HTT, Q48-HTT, Q73-HTT et ΔExon1-HTT sont respectivement 0,776, 0,769, 0,762 et 0,798 (mg/mL)-1 cm-1 cm-1 pour le calcul). Maintenir la concentration de HTT ≤ 1,0 mg/mL.
REMARQUE: Il est essentiel de surveiller le processus de concentration car une surconcentration entraînera une agrégation. - Aliquote la protéine HTT purifiée dans des tubes microcentrifugeuses cryo-sécuritaires dans un volume < 100 μL. Congeler les aliquotes à l’aide d’azote liquide et les conserver à -80 °C.
7. Analytique HPLC SEC-MALS-dRI pour analyser la polydispersité HTT
- Effectuer toutes les analyses SEC-MALS à 4 °C sur un système de chromatographie liquide (CLHP) haute performance couplé à un détecteur UV, un détecteur multiangle de diffusion de la lumière et un détecteur à indice de réfraction différentiel (dRI).
- Avant de connecter la colonne UHPLC au système, purgez la pompe et les détecteurs avec de l’eau filtrée (0,1 μm) de qualité CLHP.
- Connectez la colonne UHPLC (voir le tableau des matériaux pour la colonne utilisée) au système. Équilibrer la colonne avec de l’eau filtrée (0,1 μm), puis un tampon SEC-MALS (tableau 1) jusqu’à ce que tous les signaux du détecteur atteignent la ligne de base.
- Injecter 2 μL de 6 mg/mL d’albumine sérique bovine (BSA) à un débit de 0,3 mL/min pendant 15 min par injection et inspecter la qualité des données. Effectuez la normalisation, l’alignement des pics et la correction de l’élargissement de la bande en fonction du profil BSA, et créez un modèle pour les exemples HTT suivants.
- Décongeler rapidement un flacon de l’échantillon FL Q23-HTT dans un bain-marie à température ambiante à l’aide d’un flotteur. Filtrer le HTT à travers un filtre de spin de 0,1 μm. Injecter 2 à 4 μL de l’échantillon HTT et faire fonctionner pendant 15 min à 4 °C à un débit de 0,3 mL/min.
- Analyser les données chromatographiques et de diffusion de la lumière à l’aide du logiciel d’accompagnement (voir le tableau des matériaux). Utilisez le détecteur dRI comme détecteur de concentration et utilisez 0,185 comme incrément d’indice de réfraction (dn/dc) pour HTT. Générer un diagramme de Zimm pour déterminer la masse moléculaire moyenne en poids pour chaque pic33,34.
NOTE: L’incrément de l’indice de réfraction de HTT est calculé comme 0,185 en utilisant le logiciel SEDFIT35 du programme et la séquence d’acides aminés primaires de HTT en entrée.
REMARQUE: Le monomère HTT MW est déterminé par SEC-MALS à ~370 kDa ± 30 kDa. Le HTT purifié a généralement une teneur en monomères comprise entre 60 et 75% (dans ce laboratoire). Une faible teneur en monomères peut indiquer qu’il faut prendre plus de précautions dans la manipulation pour éviter l’agrégation.
8. Blue Native PAGE pour analyser la polydispersité HTT
- Préparer 1 L de tampon anodique en mélangeant 50 mL de 20x tampon courant Blue Native PAGE (voir le tableau des matériaux) avec 950 mL deH2O. Préparer 2 L de tampon cathodique bleu foncé en mélangeant 100 mL de tampon courant Blue Native PAGE et 100 mL d’additif de cathode Blue Native PAGE (20x) avec 1 800 mL de H2O. Refroidir les tampons à 4 °C avant utilisation.
- Décongeler rapidement un flacon d’échantillon de FL Q23-HTT dans un bain-marie à température ambiante à l’aide d’un flotteur. Gardez la protéine décongelée sur la glace avant utilisation.
- Mélanger 5 μg de FL Q23-HTT (~1 mg/mL), 1 μL d’additif G250 à 0,5 %, 2,5 μL de 4x tampon d’échantillon Blue Native PAGE et de l’eau pour porter le volume final à 10 μL.
- Charger l’échantillon mélangé FL Q23-HTT sur un gel Bis-Tris préfabriqué à 3-12%. Charger 7,5 μL de l’étalon protéique non coloré dans le même gel que l’étalon.
- Remplissez l’avant du réservoir avec un tampon cathodique bleu foncé et l’arrière du réservoir avec un tampon d’anode.
REMARQUE : Remplissez les mémoires tampons après le chargement de l’échantillon pour faciliter la visualisation lors du chargement des échantillons. - Faites fonctionner le gel à 150 V pendant 120 min dans une chambre froide.
- Colorer le gel avec une solution décolorante (tableau 1) jusqu’à ce que des bandes soient observées; Transférer le gel dans l’eau. Visualisez et documentez le gel sur une station d’imagerie.
REMARQUE: Blue Native PAGE a été conçu à l’origine pour analyser les protéines membranaires. Il a été adapté dans ce laboratoire comme méthode alternative pour estimer la teneur en monomères de HTT. Il se lie aux régions hydrophobes du HTT et l’empêche de former des agrégats dans des conditions tampons dépourvues de détergent. Le PAGE natif traditionnel sans utiliser le bleu de Coomassie G250 provoque la formation d’oligomères et d’agrégats solubles par HTT, probablement en raison des nombreuses poches hydrophobes existant dans le HTT.
9. PAGE FDS suivie d’une coloration Coomassie ou argentée pour analyser la pureté HTT
- Ajouter 4x tampon d’échantillon LDS et 10x réactif réducteur au FL Q23-HTT purifié pour que la concentration finale du tampon de charge et du réactif réducteur soit de 1x.
- Chauffer l’échantillon sur un bloc chauffant sec à 70 °C pendant 10 min.
- Charger un maximum de 1 μg de protéines par puits sur un gel d’acétate de Tris à 3-8% et faire fonctionner à 150 V pendant 1 h en utilisant un tampon de fonctionnement Tris-acétate SDS.
REMARQUE : Les protéines utilisées dans cette étude sont également disponibles directement auprès du HD Community Repository de l’Institut Coriell (www.coriell.org/1/CHDI) ; voir le tableau des matières. - Tache de Coomassie
- Laver le gel avecH2Opendant 5 min.
- Colorer le gel dans la solution de coloration de Coomassie (tableau 1) en berçant le gel dans 30 mL de solution de coloration pendant 15 min.
- Décolorer en balançant le gel dans 50 mL deH2Opendant 5 min. Répéter deux fois. Visualisez et documentez le gel coloré à Coomassie sur une station d’imagerie.
- Teinture d’argent à l’aide d’un kit de teinture d’argent commercial.
- Après SDS-PAGE, fixer le gel à l’aide de la solution de fixation (Tableau 1) pendant 1 h à une nuit à température ambiante.
- Effectuez la coloration, lavez et développez selon les instructions du kit.
- Arrêtez l’étape de développement immédiatement une fois que les bandes ont atteint l’intensité souhaitée.
- Documenter le gel dans un système de documentation du gel équipé d’une source de lumière visible.
REMARQUE: HTT purifié à >95% peut être détecté par Coomassie et coloration d’argent avec ce protocole. Reportez-vous au protocole32 publié précédemment pour plus de détails sur la façon d’effectuer une analyse quantitative des protéines.
Résultats
Un vecteur d’expression transitoire (pcDNA3.1-Q23-HTT-TEV-FLAG, Figure 1A) est conçu pour une production rapide de FL Q23-HTT dans des cellules de mammifères (aa 1-3 144, basé sur la numérotation Q23). Cette construction possède les caractéristiques conçues pour générer rapidement diverses constructions de mutation HTT par clonage de cassette, faciliter la purification de la protéine HTT à une qualité et une homogénéité élevées avec un minimum d’étapes chromatographiques, et avoir la possibilité de produire FL HTT non marqué. La liste des fonctionnalités comprend 1. Les sites de digestion de restriction HindIII, entourant la répétition CAG dans l’exon 1 de HTT, peuvent être utilisés pour générer des mutants FL HTT avec un étirement polyQ de différentes longueurs par digestion et ligature d’enzymes de restriction; 2. l’extrémité C-terminale du FL HTT est marquée avec un épitope FLAG avec un site de reconnaissance de la protéase TEV pour une purification par affinité en une étape du FL HTT avec une grande pureté et la génération facultative de la protéine FL HTT sans marquage en utilisant le clivage de la protéase TEV; 3. Séquence FL HTT optimisée pour le codon pour l’utilisation de codons de cellules humaines pour une expression de haut niveau dans les cellules HEK293. Le vecteur pcDNA 3.1 (+) est utilisé comme épine dorsale de la construction pour tirer parti de la forte activité d’activation transcriptionnelle du promoteur du CMV dans les lignées cellulaires de mammifères.
En utilisant pcDNA3.1-Q23-HTT-TEV-FLAG comme modèle de départ, les constructions Q48 et Q73 FL HTT ont été produites en synthétisant des fragments d’ADN avec une longueur Q appropriée couvrant deux sites d’enzymes de restriction HindIII et en échangeant la même région dans le modèle. Le mutant ΔExon1 de FL HTT (aa 91-3 144) (Figure 1B) a été produit à l’aide d’amorces dirigées vers les résidus supprimés couvrant la région de l’exon 1 dans le gabarit. Des cellules HEK293 transfectées avec pcDNA3.1-Q23-HTT-TEV-FLAG à l’aide de PEI ont été cultivées dans des flacons agitateurs de 5 L contenant moins de 5% de CO2. Une purification typique à grande échelle utilise une pastille cellulaire de 2 à 10 L contenant 6,0 × 10 9-3,0 × 1010 cellules. Avant de procéder à la purification, le niveau d’expression de HTT de chaque transfection a été estimé par transfert Western quantitatif en utilisant du HTT recombinant purifié marqué FLAG comme anticorps standard et un anticorps anti-FLAG comme premier anticorps. Des pastilles dont le niveau d’expression de HTT est estimé à ≥2 pg HTT/cellule ont été utilisés pour la purification.
La purification du FL HTT consiste en un processus en 2 étapes, d’abord avec purification d’affinité anti-FLAG, puis avec SEC sur une colonne de filtration sur gel avec une plage de séparation appropriée pour HTT (Figure 2A; voir le tableau des matériaux pour des exemples). Après les deux étapes, le HTT a été obtenu à > 95% de pureté de l’échantillon, telle que déterminée par SDS-PAGE avec du bleu de Coomassie et une teneur en monomère de >65% sur la base de l’analyse SEC-MALS. Étant donné que le temps et la température de purification prolongés ont un impact négatif sur la teneur finale en monomère HTT, le FPLC a été utilisé dans les deux étapes de purification afin de minimiser la manipulation et d’obtenir une qualité d’échantillon constante. Le principal contaminant lors de la purification anti-FLAG était le chaperon Hsp70 déterminé par spectrométrie de masse (Figure 2B, voie 2). Ceci est cohérent avec la découverte selon laquelle Hsp70 est co-purifié avec FL HTT exprimé de manière stable dans les lignées cellulaires humaines24, ce qui suggère que Hsp70 peut être un stabilisateur commun pour FL HTT in vivo.
La contamination par le Hsp70 peut être éliminée en effectuant un lavage intensif au chlorure de magnésium et à l’ATP pendant l’étape de purification par affinité anti-FLAG (Figure 2B, voie 1). Lors de l’élimination de Hsp70, FL HTT est susceptible de former des oligomèresd’ordre supérieur 24 et doit être maintenu à une concentration ≤ 1 mg / mL. L’étape de concentration avant la SEC peut souvent entraîner une agrégation importante. Par conséquent, la meilleure pratique consiste à charger directement les fractions de crête de la purification anti-FLAG sur la colonne d’exclusion de taille sans concentration. Après SEC, l’échantillon a été concentré à ≤1 mg/mL pour une récupération maximale du FL HTT monomère. La quantité de HTT récupérée à chaque étape de purification a été estimée par transfert bleu de Coomassie ou Western quantitatif en utilisant le FL HTT purifié comme étalon de quantification (tableau 2). Le rendement typique des protéines FL HTT purifiées produites par la méthode décrite est d’environ 1 mg/L de culture cellulaire, mais peut tomber bien en deçà de ce rendement (tableau 3) en raison de la variabilité d’un lot à l’autre, ou si la résine de purification anti-FLAG est réutilisée plusieurs fois.
La surexpression de FL HTT peut entraîner une fragmentation de la protéine22. FL Q23-HTT produit par la méthode décrite ici résolu comme une bande unique avec le MW correct de 350 kDa par SDS PAGE, coloré soit par Coomassie G250, soit par coloration à l’argent (figure 2C). Par transfert western, FL Q23-HTT a réagi avec des anticorps élevés contre les épitopes au niveau du N-terminal, du C-terminal et de plusieurs domaines intermédiaires, sans qu’aucune bande supplémentaire liée aux fragments n’ait été observée, ce qui indique que la protéine a été isolée sans troncature détectable significative (Figure 3A). Les variantes de longueur polyQ FL HTT Q23, Q48 et Q73 ont réagi comme prévu dans le transfert western, montrant un signal progressivement plus fort pour l’AcM MW1 dirigé par polyQ en corrélation avec l’augmentation de la longueur Q: Q23-HTT < Q48-HTT < Q73-HTT (Figure 3B). Aucun signal n’a été observé pour ΔExon1-HTT (aa 91-3 144) lorsqu’il a été sondé avec les anticorps MW1 et MAB549, qui ciblent l’exon terminal N 1 (Figure 3B).
SEC-MALS a été utilisé pour analyser l’état d’agrégation et la masse moléculaire de la protéine HTT purifiée. Les échantillons ont été analysés par SEC analytique surveillé par des détecteurs UV, MALS et dRI. La masse molaire absolue obtenue à partir de SEC-MALS ne dépend pas de la forme des molécules33,34 ; par conséquent, SEC-MALS fournit une estimation non biaisée de MW pour les fractions monomères et oligomères lorsqu’elles sont bien séparées. Parmi les colonnes CLHP testées, la colonne SEC (voir le tableau des matériaux) a montré une résolution suffisante entre le monomère HTT et le dimère pour que les masses molaires puissent être distinguées (figure 4). La concentration en protéines a été déterminée par détection dRI. Les incréments de l’indice de réfraction (dn/dc) de FL HTT sont de 0,1853 mL/g tel que calculé par le logiciel SEDFIT35. Des profils d’élution analytiques SEC similaires ont été observés pour ΔExon1 HTT (91-3 144), FL Q23, Q48 et Q73 HTT (1-3 144), chacun consistant en un pic majeur de monomère avec des pics dimères et oligomères mineurs (tableau 4). Le MW calculé pour la forme monomère est supérieur au MW théorique. Ceci est probablement causé par le chevauchement d’espèces provenant de pics oligomères d’ordre supérieur et par des erreurs résultant de signaux dRI faibles lorsque les protéines HTT sont maintenues en faible concentration pour éviter la formation d’oligomères d’ordre supérieur. En intégrant les pics UV de plusieurs lots de variantes FL HTT purifiées, aucune corrélation claire entre la longueur du polyQ et le profil agrégé n’a été observée (tableau 4).
En plus de la SEC analytique, le PAGE natif traditionnel a été réalisé pour déterminer s’il peut être utilisé comme méthode complémentaire pour caractériser l’état oligomère FL HTT. Les oligomères d’ordre supérieur ont été résolus par des gels de tris-acétate à 3-8% en utilisant un tampon natif sans détergent. Le FL HTT purifié de la SEC a montré plusieurs bandes correspondant aux états d’oligomérisation (figure 5A). La bande la plus basse était située entre le marqueur natif 480 kDa et 720 kDa, similaire aux résultats précédents rapportés pour FL HTT purifié à partir de cellules d’insectes22. Cependant, le monomère HTT n’était pas la bande la plus abondante lors de l’utilisation du PAGE natif traditionnel, et les résultats ne sont pas corrélés avec le profil agrégé déterminé par SEC-MALS analytique. Plusieurs patchs hydrophobes présents dans FL HTT36,37,38, en particulier l’interface hydrophobe entre HAP40 et FL HTT25, sont susceptibles de contribuer à la formation d’oligomères d’ordre supérieur lors de la migration dans le gel. En effet, les régions hydrophobes sont connues pour interagir les unes avec les autres en l’absence d’interactions protéine-protéine détergente ou stabilisatrice. Conformément aux propriétés hydrophobes de HTT, FL HTT forme des quantités croissantes de fractions oligomères d’ordre supérieur en l’absence de CHAPS pendant l’étape de purification SEC.
Blue Native PAGE, qui est largement utilisé pour étudier les protéines membranaires et les grands complexes protéiques contenant des patchs hydrophobes39, a été comparé au PAGE natif traditionnel. Le HTT purifié a montré trois bandes principales sur Blue Native PAGE avec des MW estimés à 643, 927 et 1070 kDa (Figure 5B) qui représentent probablement les espèces monomères, dimériques et trimères de HTT, respectivement. La bande monomère est restée la bande la plus abondante dans le Blue Native PAGE, correspondant bien au profil analytique SEC des mêmes échantillons. La surestimation de MW du monomère HTT par Blue Native PAGE peut résulter de la structure sphérique creuse unique ou des régions hydrophobes de HTT qui provoquent une migration plus lente par rapport aux marqueurs de poids moléculaire correspondants 11,23,25. Dans l’ensemble, FL Q23-HTT, FL Q48-HTT, FL Q73-HTT et ΔExon1-HTT ont des profils PAGE natifs bleus similaires avec seulement de légères différences dans la migration de la bande protéique en raison de leurs différences de poids moléculaire.
Comme contrôle supplémentaire de la qualité des protéines purifiées, l’étiquette C-terminal FLAG peut être retirée de FL HTT par traitement avec la protéase TEV. Après le clivage protéolytique, les échantillons ont été analysés par transfert Western à l’aide de quatre anticorps pour confirmer le retrait du marqueur FLAG et détecter la dégradation du HTT. L’immunoréactivité à l’anti-FLAG M2 et à trois anticorps spécifiques de la huntingtine avec des épitopes à l’extrémité N, aux domaines intermédiaires et à l’extrémité C du HTT a montré une élimination réussie du marqueur FLAG et aucun produit de dégradation spécifique du HTT (figure supplémentaire S1).
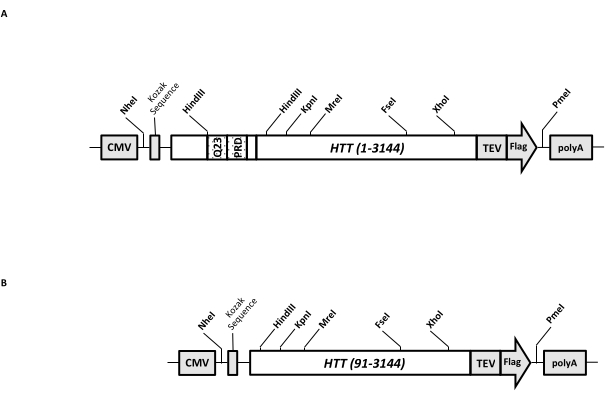
Figure 1 : Construire pour l’expression HTT pleine longueur. (A) Le Q23 HTT complet a été optimisé pour le codon et cloné en plasmide pcDNA3.1 (+). L’extrémité 3' de HTT a été marquée avec l’épitope Flag et le site de clivage de la protéase TEV pour produire une protéine HTT sans marqueur. L’étirement de la polyglutamine et le domaine riche en proline ont été conçus avec des sites d’endonucléase de restriction HindIII flanqués pour insérer des répétitions CAG supplémentaires en utilisant le clonage de cassettes, c’est-à-dire Q48 et Q73, pour produire des variantes HTT avec différentes longueurs de polyQ. (B) La construction ΔExon1 a été réalisée par mutagénèse PCR en utilisant pcDNA3.1-Q23-HTT comme modèle. Les résidus 91-3,144 de HTT sont restés dans la construction ΔExon1 pour expression. Abréviations : HTT = huntingtine; CMV = cytomégalovirus; Q23 = étirement de polyglutamine; PRD = domaine riche en proline; TEV = site de clivage du virus de la gravure du tabac. Veuillez cliquer ici pour voir une version agrandie de cette figure.
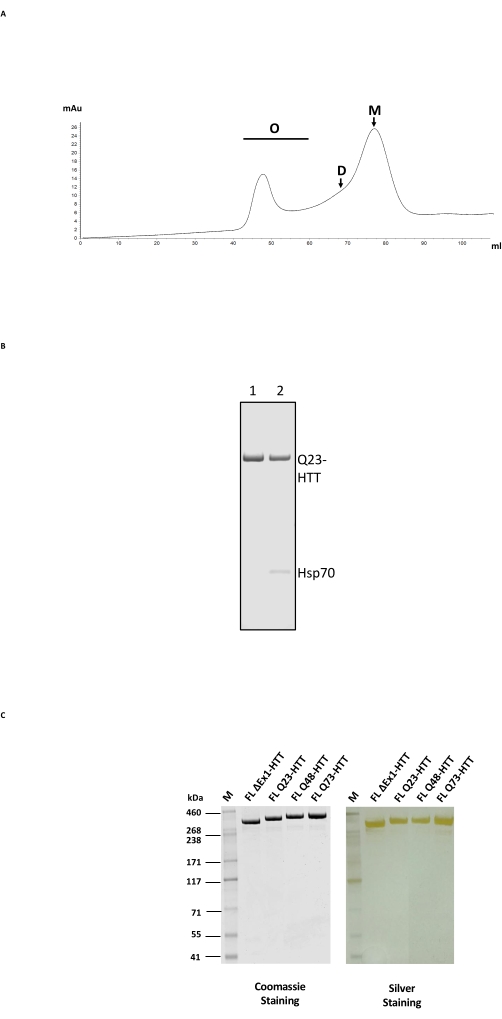
Figure 2 : Purification à grande échelle de HTT. (A) Profil SEC du Q23-HTT pleine longueur purifié anti-drapeau sur une colonne FPLC. Les pics d’oligomères, de dimères et de monomères de Q23-HTT hautement ordonnés sont étiquetés. Des fractions contenant du monomère ont été prélevées en tant qu’échantillon final de HTT. (B) La FDS-PAGE de Q23-HTT purifié avec étape de lavage ATP/magnésium (voie 1) ou sans lavage ATP/magnésium entraîne une co-élution Hsp70 (voie 2). (C) Variantes HTT purifiées sur toute la longueur sur FDS-PAGE colorées au bleu Coomassie G-250 ou à la teinture argentée. Abréviations : FL = pleine longueur; HTT = huntingtine; SEC = chromatographie d’exclusion de taille; FPLC = chromatographie liquide à protéines rapides; O = oligomère; D = dimère; M = monomère; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium polyacrylamide; Hsp70 = protéine de choc thermique 70. Veuillez cliquer ici pour voir une version agrandie de cette figure.
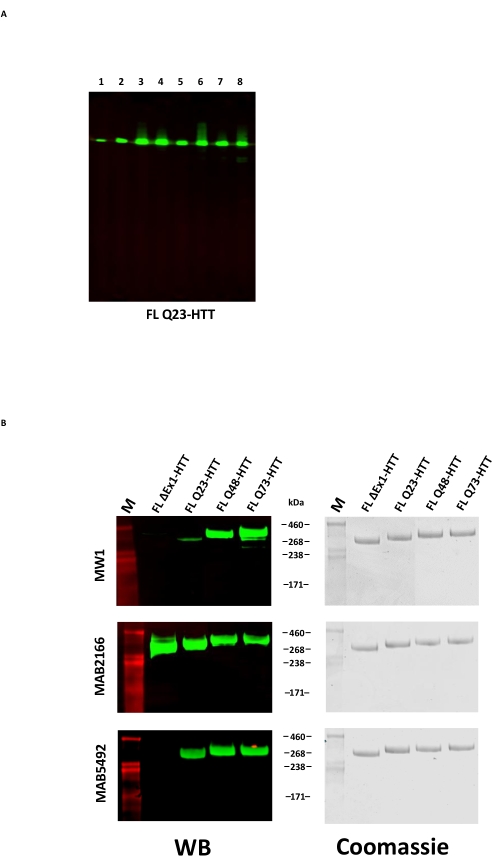
Figure 3 : Analyse par transfert Western des variantes HTT purifiées. (A) Le FL Q23-HTT purifié a été exécuté sur SDS-PAGE et transféré sur une membrane PVDF. Les anticorps primaires et les épitopes en interaction sont Lane 1, α-FLAG M2, FLAG tag; Voie 2, MAB5492, HTT aa. 1-82; Lane 3, MAB5490, HTT aa 115-129; Lane 4, MAB2166, HTT aa 181-810; Lane 5, MAB3E10, HTT aa 1 171-1 177; Lane 6, MAB4E10, HTT aa 1 844-2 131; Lane 7, MAB2168, HTT aa 2 146-2 541; Lane 8, MAB8A4, HTT aa 2 703-2 911. (B) 1 μg de variantes FL HTT purifiées ont été exécutées sur SDS-PAGE et transférées au PVDF (à gauche), et un double gel SDS a été exécuté et coloré avec du bleu de Coomassie (à droite). Les anticorps primaires et les épitopes en interaction sont les répétitions PolyQ étendues de la rangée 1, MW1; Rangée 2, MAB2166, HTT aa 181-810; Ligne 3, MAB5492, HTT aa 1-82. Abréviations : FLL Q23-HTT = protéine huntingtine pleine longueur contenant 23 résidus de glutamine; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium polyacrylamide; WB = Western blot; M = marqueur; PVDF = fluorure de polyvinylidène. Veuillez cliquer ici pour voir une version agrandie de cette figure.
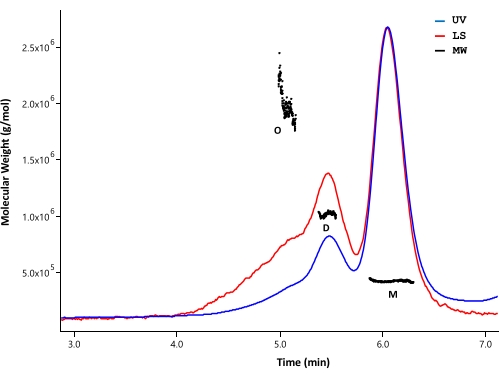
Figure 4 : Analyse SEC-MALS du HTT pleine longueur. Le Q23-HTT purifié pleine longueur a été élué sur une colonne UPLC. Les positions de pointe des monomères, dimères et oligomères prévus sont indiquées. Les masses moléculaires ont été calculées pour les pics de monomères, de dimères et de trimères et indiquées dans le tableau 5. Des profils d’élution similaires sont observés pour Q48, Q73 et ΔExon1 HTT, avec des teneurs variables en monomères, dimères et oligomères dans chaque purification. Abréviations : SEC-MALS = chromatographie d’exclusion de taille avec diffusion de la lumière multiangle; UV = Ultra Violet; LS = diffusion de la lumière; MW = poids moléculaire; Q23-HTT = protéine huntingtine contenant 23 résidus de glutamine; M = monomère; D = dimère; O = oligomère. Veuillez cliquer ici pour voir une version agrandie de cette figure.
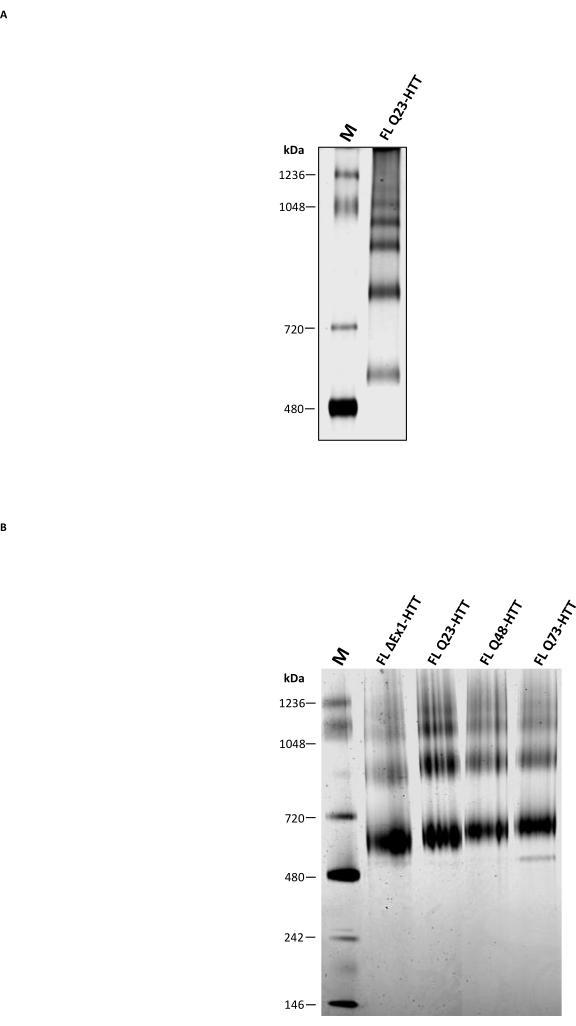
Figure 5 : Caractérisation du HTT purifié à l’aide d’un gel PAGE natif clair ou d’un gel PAGE natif bleu. Le marqueur natif et le Q23-HTT monomère apparent de la SEC ont été résolus sur des gels de tris-acétate à 3-8% dans un système PAGE non dénaturant (A) et un système PAGE natif bleu (B). Abréviations : FL = intégrale; Q23-HTT = protéine huntingtine contenant 23 résidus de glutamine; PAGE = électrophorèse sur gel de polyacrylamide; M = marqueur. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Pas | Nom | Composition | ||
| 6.1.1 | Tampon A | 50 mM Tris, 500 mM NaCl, 5% v/v glycérol, 5 mM EDTA, 0,01% v/v Tween-20, pH 8,0. | ||
| 6.1.2 | Tampon de lyse | 50 mM Tris, 500 mM NaCl, 5% v/v glycérol, 5 mM EDTA et 1x cocktail inhibiteur de protéase | ||
| 6.1.4.2 | Tampon A | 50 mM Tris, 500 mM NaCl, 5% v/v glycérol, 5 mM EDTA, 0,01% v/v Tween-20, pH 8,0. | ||
| 6.1.4.3 | Tampon B | 50 mM Tris; 500 mM KCl; 5 mM MgCl2; 5% v/v glycérol; 0,01 % v/v Tween-20, pH 8,0 | ||
| 6.1.4.4 | Tampon C | 20 mM Tris; 200 mM KCl; 5 mM MgCl2; 5 mM d’ATP; 0,01 % v/v Tween-20; 5 % v/v de glycérol, pH 8,0 | ||
| 6.1.4.5 | Tampon D | 50 mM Tris; 500 mM NaCl; 5% v/v glycérol; 5 mM d’EDTA; 0,5 % p/v CHAPS, pH 8,0 | ||
| 6.1.4.6 | Tampon d’élution | 50 mM Tris; 500 mM NaCl; 5% v/v glycérol; 0,5 % p/v CHAPS; 0,2 mg/mL de peptide DYKDDDK, pH 8,0 | ||
| 6.1.6 | Tampon de régénération | 0,1 M glycine HCl, pH 3,5; 0,01 % v/v Tween-20 | ||
| 6.2.1 | Tampon SEC | 50 mM Tris, 500 mM NaCl, 5% v/v glycérol, 0,5% p/v CHAPS, 1 mM TCEP | ||
| 7.3 | Tampon SEC-MALS | 50 mM HEPES, pH 7,2, 500 mM NaCl, 5% v/v glycérol, 0,5% p/v CHAPS | ||
| 8.7 | Solution de décoloration | 40 % v/v de méthanol et 7 % v/v d’acide acétique | ||
| 9.4.2 | Solution de coloration Coomassie | 0,01 % p/v Coomassie G250, 50 % v/v/ méthanol, 10 % v/v acide acétique | ||
| 9.5.1 | Solution de réparation | 50 % v/v de méthanol, 10 % v/v d’acide acétique, 50 μL de formaldéhyde/100 mL de solution | ||
Tableau 1 : Composition des tampons et des solutions
| Escalier | Concentration de HTT (mg/ml) | Volume total (mL) | Teneur en HTT (mg) | Rendement HTT par cellule (pg/cellule) | % Rendement |
| Surnageant | 0.1792 | 220 | 39.4 | 4.4 | 100 |
| Anti-drapeau | 1.524 | 8.6 | 13.1 | 1.47 | 33.4 |
| SECONDE | 0.91 | 3.9 | 3.54 | 0.4 | 9.1 |
Tableau 2 : Rendement en HTT d’une pastille HEK293 de 2 L transfectée avec pcDNA3.1-Q23-HTT-TEV-Flag. Abréviations : FL Q23-HTT = protéine huntingtine pleine longueur contenant 23 résidus de glutamine; TEV = site de clivage du virus de la gravure du tabac; SEC = chromatographie d’exclusion de taille.
| Exemple HTT | Rendement HTT (mg/L) | Pureté moyenne (%) | ||
| BCA | A280 | |||
| 1 | FL DEx1-HTT (N=3) | 0.67-1.30 | 0.69-1.18 | 99.3 |
| 2 | FL Q23-HTT (N=3) | 0.25-0.92 | 0.28-0.98 | 96.9 |
| 3 | FL Q48-HTT (N=3) | 0.28-1.15 | 0.38-1.16 | 97.4 |
| 4 | FL Q73-HTT (N=3) | 0.58-1.05 | 0.57-0.97 | 98.8 |
Tableau 3 : Résumé du rendement en protéines de quatre purifications de variantes FL HTT et de leur pureté finale. Abréviation : FLL HTT = protéine huntingtine pleine longueur.
| Exemple HTT | Un | D | M |
| FL Q23-HTT | 4.2-6.9% | 18.7-29.3% | 66.5-76.0% |
| FL Q48-HTT | 4.0-9.4% | 10.6-17.8% | 73.6-85.4% |
| FL Q73-HTT | 2.0-14.0% | 16.9-24.6% | 65.1-81.1% |
Tableau 4 : Résumé de la teneur représentative en agrégats, dimères et monomères des variantes FL HTT issues de la purification. Abréviations : FL HTT = protéine huntingtine pleine longueur; A = agrégat; D = dimère; M = monomère; SEC = chromatographie d’exclusion de taille.
Figure supplémentaire S1 : Analyse par transfert Western après digestion de la protéase TEV. Les FL Q23-HTT et FL Q48-HTT purifiés ont été exécutés sur SDS-PAGE, transférés sur des membranes PVDF et analysés par transfert Western après digestion TEV. Les principaux anticorps utilisés étaient anti-Flag M2 (Flag tag), MAB5492 (HTT aa 1-82), MAB3E10 (HTT aa 997-1 276) et MAB2168 (HTT aa 2 146-2 541). Voie 1, Norme de protéines; Voie 2, Q23-HTT-TEV-Flag; Voie 3, Q48-HTT-TEV-Flag; Lane 4, Q23-HTT-TEV-Flag traitée avec de la protéase TEV à 1:5, pendant la nuit à 4 °C; Voie 5, Q48-HTT-TEV-Flag traitée avec de la protéase TEV à 1:5, pendant la nuit à 4 °C. Abréviations : FL HTT = protéine huntingtine pleine longueur; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium polyacrylamide; TEV = virus de la gravure du tabac; PVDF = fluorure de polyvinylidène. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S2 : Analyse SEC-MALS des variantes FL HTT soumises à des cycles de gel-dégel. Le Q23-HTT (A) et le Q48-HTT (B) purifiés ont été congelés à -80 °C et décongelés jusqu’à 6 fois à température ambiante. Le Q23-HTT et le Q48-HTT après le premier cycle de gel-dégel et le sixième cycle de gel-dégel ont ensuite été analysés par SEC-MALS. Une légère diminution de la fraction monomère et une augmentation des fractions dimères et oligomères d’ordre élevé ont été observées par diffusion de la lumière après des cycles répétés de gel-dégel. Les positions de pointe des monomères, des dimères et des oligomères d’ordre élevé prévus sont indiquées. Abréviations : FL HTT = protéine huntingtine pleine longueur; O = oligomère; D = dimère; M = monomère; SEC-MALS = Chromatographie d’exclusion de taille avec diffusion de la lumière multi-angle. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S3 : PAGE DE LA FDS des variantes FL HTT soumises à des cycles de gel-dégel. Le Q23-HTT purifié (voies 2 à 7) et le Q48-HTT (voies 9 à 14) ont été congelés à -80 °C et décongelés à température ambiante jusqu’à 6 fois. Les aliquotes de Q23-HTT et de Q48-HTT ont été stockées après chaque cycle de gel-dégel, puis analysées par SDS PAGE. Aucune augmentation des agrégats ou des produits de dégradation n’a été observée; Les échantillons ont été considérés comme stables et purs à > 95 % par densitométrie en bande. Abréviations : FL HTT = protéine huntingtine pleine longueur; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium et de polyacrylamide. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 1 : FPLC 15 mL script anti-FLAG HTT. Abréviations = FPLC = chromatographie liquide à protéines rapides; HTT = protéine huntingtine. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2 : FPLC SEC_MALS script HTT. Abréviations : SEC-MALS = chromatographie d’exclusion de taille avec diffusion de la lumière multiangle; FPLC = chromatographie liquide à protéines rapides; HTT = protéine huntingtine. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Nous décrivons ici une méthode transitoire de transfection, d’expression et de purification pour générer de multiples constructions de protéines FL HTT avec une pureté et une homogénéité appropriées pour une utilisation comme normes pour le développement d’immunoessais et de tests MS, de contrôles pour l’analyse par transfert Western et pour les études structure-fonction. Cette méthode d’expression transitoire est évolutive et polyvalente et permet à l’utilisateur de générer des quantités de faibles milligrammes de variantes FL HTT plus efficacement que l’utilisation de lignées cellulaires stables ou de méthodes basées sur des virus décrites précédemment21,22,23,24. Régulièrement, 2 à 5 mg de FL HTT hautement purifié peuvent être générés à partir d’une production de protéines à l’échelle de 2 L en moins d’une semaine en utilisant la méthode d’expression transitoire une fois le plasmide construit, avec un rendement typique de 1 à 2,5 mg de FL HTT par litre de culture cellulaire.
La méthode d’expression transitoire décrite ici surmonte de nombreux obstacles à l’expression de lignées cellulaires stables, tels que le temps nécessaire pour établir les lignées cellulaires et les difficultés de stockage et de maintien de lignées cellulaires stables. Le PEI est également relativement peu coûteux par rapport aux autres réactifs de transfection sur le marché, ce qui rend la transfection à grande échelle économiquement viable. Le protocole comporte également des limites : l’efficacité de la transfection dépend en grande partie de la qualité des plasmides, de la croissance cellulaire optimale et de la qualité du stockage et de la préparation du PEI. Les opérateurs doivent prendre des précautions particulières et effectuer des contrôles de qualité dans ces étapes critiques pour éviter une chute drastique des rendements en protéines. La résine anti-FLAG utilisée dans le protocole est également relativement coûteuse et montre une capture réduite de FL HTT après plusieurs purifications et régénérations. Certains chercheurs peuvent trouver plus pratique de passer à une étiquette différente pour permettre une régénération plus robuste de la résine d’affinité.
Diverses lignées cellulaires et conditions d’expression ont été testées pour optimiser les niveaux d’expression de FL HTT. Les cellules HEK293 ont été choisies pour l’expression de FL HTT en raison de la forte expression des protéines et de la facilité de manipulation dans un format de culture en suspension, ce qui rend la méthode adaptée à une expression à grande échelle dans des agitateurs ou des bioréacteurs. Un niveau d’expression plus élevé de la protéine FL HTT peut être atteint à des températures de culture plus basses telles que 32 ° C plutôt qu’en utilisant la température habituelle de 37 ° C. Il est possible que la température plus basse ralentisse la synthèse des protéines et favorise le repliement correct de FL HTT40. Cependant, ce phénomène n’est pas spécifique à FL HTT ou aux lignées cellulaires testées. La température post-transfection réduite a été largement utilisée dans l’expression des protéines pharmaceutiques dans les cellules CHO. Bien que le mécanisme ne soit pas entièrement compris, on pense que les basses températures arrêtent le cycle cellulaire dans la phase G1 et détournent l’énergie cellulaire vers la production de protéines41.
Le HTT pleine longueur purifié à partir de cellules de mammifères co-élue avec le chaperon Hsp7024, et les étapes de lavage Mg-ATP peuvent éliminer la protéine Hsp70. Fait intéressant, Hsp70 co-élué n’est pas observé dans FL HTT purifié à partir d’un système d’expression cellulaire d’insecte21,22,23. Cela peut refléter une différence dans les PTM de FL HTT ou les réponses protéiques de choc thermique à la surexpression de FL HTT dans les cellules de mammifères et d’insectes. Une fois que la protéine recombinante a été dépouillée de Hsp70, des détergents non ioniques tels que CHAPS ou DDM sont nécessaires pour stabiliser la forme monomère de FL HTT.
Les états d’oligomérisation des variantes FL HTT ont été analysés à l’aide de Blue Native PAGE et SEC-MALS. Une petite fraction de HTT dimérique et oligomère d’ordre supérieur était présente lors de l’analyse par Blue Native PAGE ou SEC-MALS. Il est à noter que les oligomères d’ordre supérieur formés par FL HTT ne semblent pas être en corrélation avec la longueur polyQ, et même le mutant de délétion Exon1 affiche un rapport oligomère-dimère-monomère similaire. Les variations réelles de la teneur en oligomères de ces constructions sont probablement dues à des différences mineures dans la production et la manutention de chaque lot. Contrairement aux agrégats et aux fibrilles formés par HTT Exon140,41, les oligomères d’ordre supérieur de FL HTT sont restés solubles et ont pu être analysés par SEC et Native PAGE.
Le FL HTT monomère purifié n’est que relativement stable. Un stockage prolongé à 4 °C, de courtes incubations à température ambiante ou des concentrations > 1 mg/mL convertiront tous le FL HTT monomère en formes dimères et oligomères d’ordre supérieur, même si aucune précipitation visible n’est observée dans ces conditions. Le FL HTT monomère purifié maintenu à ≤1 mg/mL est resté relativement stable à -80 °C dans un tampon de stockage (50 mM Tris, pH 8,0, 500 mM NaCl, 5 % v/v glycérol, 0,5 % p/v CHAPS et 5 mM DTT) comme décrit précédemment24. Jusqu’à 6 cycles de gel-dégel de FL HTT préparés et stockés de cette manière n’ont pas provoqué de précipitation visible de la protéine, bien qu’un léger changement vers un état oligomérique plus élevé ait été observé par SEC-MALS (figure supplémentaire S2). Les échantillons ont également été analysés par SDS PAGE à la suite de cycles répétés de gel-dégel. Aucun précipité visible n’a été observé; aucun agrégat ou produit de dégradation supplémentaire n’a été observé par SDS-PAGE (figure supplémentaire S3). La stabilité à long terme du FL HTT purifié est toujours à l’étude. En l’absence de données concluantes à long terme, nous recommandons de conserver le FL HTT purifié à -80 °C pendant 6 mois maximum.
Les variantes de la protéine FL HTT recombinante de haute qualité et les méthodes pour les produire sont très demandées par la communauté de recherche MH. Ces protéines sont utilisées comme étalons d’immunoessais et d’analyse de la SEP, dans des études structurales et pour le développement de nouveaux tests spécifiques FL HTT. Les méthodes d’expression transitoire à grande échelle décrites ici ont toujours produit des quantités de milligrammes de variantes FL HTT avec une pureté de >95%, fournissant des outils essentiels pour les études HTT. La production de dizaines de milligrammes de variantes polyQ FL HTT hautement purifiées et d’autres mutants à l’appui de la recherche MH est devenue une routine.
Déclarations de divulgation
Les auteurs déclarent n’avoir aucun conflit d’intérêts avec le contenu de cet article.
Remerciements
Nous remercions le Département des sciences pharmaceutiques de l’Université d’État de New York à Buffalo d’avoir effectué une analyse de la SEP du HTT. Ce travail était un effort de collaboration avec la Fondation CHDI. Nous remercions tout particulièrement Elizabeth M. Doherty; Ignacio Munoz-Sanjuan; Douglas Macdonald, Fondation CHDI; et Rory Curtis, Curia, pour leur contribution inestimable lors de la préparation de ce manuscrit. Nous sommes également reconnaissants à Michele Luche, Mithra Mahmoudi et Stephanie Fox pour leur soutien à cet effort de recherche.
matériels
| Name | Company | Catalog Number | Comments |
| 100 kDa concentrator-Amicon | Millipore | UFC910096 | Protocol Section Number-6.2.4 |
| 20x blue native PAGE running buffer | Invitrogen | BN2001 | Protocol Section Number-8.1 |
| 20x TBS | Thermo Fisher | PI28358 | Protocol Section Number-5.1 |
| 4x blue native PAGE sample buffer | Invitrogen | BN2003 | Protocol Section Number-8.3 |
| 4x LDS loading buffer | Invitrogen | NP0007 | Protocol Section Number-5.3 |
| 5 L Erlenmeyer flasks | Corning | 431685 | Protocol Section Number-4.2 |
| Agarose gel extraction kit | Qiagen | 28704 | Protocol Section Number-2.2 |
| Anti-clumping agent | Thermo Fisher | 0010057AE | Protocol Section Number-4.8 |
| anti-FLAG M2 affinity gel | Sigma | A2220 | Protocol Section Number-6.1.1 |
| anti-FLAG M2 | Sigma | F3165 | Protocol Section Number-5.7 |
| Anti foam-Excell anti foam | Sigma | 59920C-1B | Protocol Section Number-4.8 |
| ATP | Sigma | A6419 | Protocol Section Number-6.1.4.4 |
| BEH 450 SEC | Waters | 186006851 | 2.5 µm x 4.6 mm x 150 mm Protocol Section Number-7.3 |
| blue native PAGE 5% G-250 sample additive | Invitrogen | BN2004 | Protocol Section Number-8.3 |
| carbenicillin | Thermo Fisher | 10177012 | Protocol Section Number-2.5 |
| centrifuge - Sorvall Lynx 6000 | Thermo Fisher | 75006590 | Protocol Section Number-6.1.3 |
| Cell Counter - ViCELL | BECKMAN COULTER | Protocol Section Number-4.3 | |
| CHAPS | Anatrace | C316S | Protocol Section Number-6.1.4.6 |
| Competent E. coli cells-TOP10 | Invitrogen | C404010 | Protocol Section Number-2.4 |
| digitonin | Sigma | D141 | Protocol Section Number-5.1 |
| differential refractive index detector | Wyatt | Protocol Section Number-7.1 | |
| DYKDDDDK peptide | Genscript | Peptide synthesis service Protocol Section Number-6.1.4.6 | |
| EDTA | Sigma | EDS | Protocol Section Number-5.1 |
| EndoFree Plasmid Giga Kit | Qiagen | 12391 | Protocol Section Number-3.3 |
| Endotoxin free water | Cytiva | SH30529.03 | Protocol Section Number-4.1 |
| endotoxin quantification kit-CRL Endosafe Nexgen-PTS detection system | Charles River | PTS150K | Protocol Section Number-3.4 |
| fixed angle rotor A23-6x100 rotor | Thermo Fisher | 75003006 | Protocol Section Number-6.1.3 |
| FPLC software- Unicorn 6.2 | Cytiva | Protocol Section Number-6.1.4 | |
| Gene synthesis | Genscript | Gene synthesis service Protocol Section Number-1.2 | |
| Glycerol | Fisher Scientific Glycerol (Certified ACS) | G33-4 | Protocol Section Number-5.6 |
| Growth Medium-Expi293 expression medium | Thermo Fisher | A1435102 | Protocol Section Number-4.2 |
| HEK293 cells | Thermo Fisher | R79007 | Protocol Section Number-4 |
| high shear homogenizer-Microfluidizer | MicroFluidics | LM10 | Protocol Section Number-6.1.3 |
| HPLC - 1260 infinity II Bio-Insert HPLC | Agilent | Protocol Section Number-7.1 | |
| Image Studio | LiCor | Image analysis software Protocol Section Number-5.1 | |
| MAB2166 | Sigma | MAB2166 | Protocol Section Number-5.7 |
| MAB2168 | EMD | MAB2168 | Protocol Section Number-5.7 |
| MAB3E10 | Santa Cruz | SC-47757 | Protocol Section Number-5.7 |
| MAB4E10 | Santa Cruz | SC-7757 | Protocol Section Number-5.7 |
| MAB5490 | Sigma | MAB5490 | Protocol Section Number-5.7 |
| MAB5492 | Sigma | MAB5492 | Protocol Section Number-5.7 |
| MAB8A4 | Santa Cruz | SC-47759 | Protocol Section Number-5.7 |
| multi-angle light scattering detector | Wyatt | Protocol Section Number-7.1 | |
| NativeMark Unstained Protein Standard | Invitrogen | LC0725 | Protocol Section Number-8.4 |
| NaCl | Sigma | S9888 | Protocol Section Number-5.6 |
| NheI | New England Biolab | R0131S | Hi-Fi version available Protocol Section Number-2.2 |
| NuPAGE 3–8% Tris acetate gels | Invitrogen | EA0375PK2 | Protocol Section Number-5.4 |
| NuPAGE Tris-Acetate SDS Running buffer | Invitrogen | LA0041 | Protocol Section Number-5.4 |
| PEI 25K | Polysciences | 23966-1 | Protocol Section Number-4.1 |
| Penicillin-Streptomycin | Thermo Fisher | 15070063 | Protocol Section Number-4.2 |
| Phosphate Buffered Saline (PBS) | Cytiva | SH30256.02 | Protocol Section Number-4.5 |
| plasmid miniprep kit | Qiagen | 27104 | Protocol Section Number-2.6 |
| PmeI | New England Biolab | R0560S | Protocol Section Number-2.2 |
| precast Bis-tris gel- 3-12% NativePAGE Novex Bis-Tris Gel | Invitrogen | BN1003BOX | Protocol Section Number-8.4 |
| protease inhibitor cocktail | GoldBio | GB-331-1 | Protocol Section Number-5.1 |
| SEC-MALS analysis software - Astra 7 | Wyatt Technology | Protocol Section Number-7.6 | |
| secondary antibody -IRdye 800 CW goat anti-mouse IgG | LiCor | 926-32210 | Protocol Section Number-5.9 |
| Superose 6 pg XK 16/70 | Cytiva | 90100042 | Protocol Section Number-6.2 |
| Tris base | Fisher | BP152 | Protocol Section Number-5.6 |
| Tween-20 | Thermo Fisher | AAJ20605AP | Protocol Section Number-6.1.1 |
| UV spectrometer - Nanodrop 8000 | Thermo Fisher | ND-8000-GL | Protocol Section Number-2.2 |
| XK26/100 | Cytiva | 28988951 | Protocol Section Number-6.1.1 |
Références
- Walker, F. O. Huntington's disease. Lancet. 369 (9557), 218-228 (2007).
- McColgan, P., Tabrizi, S. J. Huntington's disease: a clinical review. European Journal of Neurology. 25 (1), 24-34 (2018).
- Duyao, M., et al. Trinucleotide repeat length instability and age of onset in Huntington's disease. Nature Genetics. 4 (4), 387-392 (1993).
- MacDonald, M. E., et al. A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington's disease chromosomes. Cell. 72 (6), 971-983 (1993).
- Nasir, J., et al. Targeted disruption of the Huntington's disease gene results in embryonic lethality and behavioral and morphological changes in heterozygotes. Cell. 81 (5), 811-823 (1995).
- Dragatsis, I., Levine, M. S., Zeitlin, S. Inactivation of Hdh in the brain and testis results in progressive neurodegeneration and sterility in mice. Nature Genetics. 26 (3), 300-306 (2000).
- Anne, S. L., Saudou, F., Humbert, S. Phosphorylation of huntingtin by cyclin-dependent kinase 5 is induced by DNA damage and regulates wild-type and mutant huntingtin toxicity in neurons. Journal of Neuroscience. 27 (27), 7318-7328 (2007).
- Dietrich, P., Johnson, I. M., Alli, S., Dragatsis, I. Elimination of huntingtin in the adult mouse leads to progressive behavioral deficits, bilateral thalamic calcification, and altered brain iron homeostasis. PLoS Genetics. 13 (7), 1006846(2017).
- Dragatsis, I., et al. Effect of early embryonic deletion of huntingtin from pyramidal neurons on the development and long-term survival of neurons in cerebral cortex and striatum. Neurobiology of Disease. 111, 102-117 (2018).
- Benn, C. L., et al. Huntingtin modulates transcription, occupies gene promoters in vivo, and binds directly to DNA in a polyglutamine-dependent manner. Journal of Neuroscience. 28 (42), 10720-10733 (2008).
- Saudou, F., Humbert, S. The biology of huntingtin. Neuron. 89 (5), 910-926 (2016).
- Davies, S. W., et al. Formation of neuronal intranuclear inclusions underlies the neurological dysfunction in mice transgenic for the HD mutation. Cell. 90 (3), 537-548 (1997).
- DiFiglia, M., et al. Aggregation of huntingtin in neuronal intranuclear inclusions and dystrophic neurites in brain. Science. 277 (5334), 1990-1993 (1997).
- Gutekunst, C. A., et al. Nuclear and neuropil aggregates in Huntington's disease: Relationship to neuropathology. Journal of Neuroscience. 19 (7), 2522-2534 (1999).
- Hodgson, J. G., et al. A YAC mouse model for Huntington's disease with full-length mutant huntingtin, cytoplasmic toxicity, and selective striatal neurodegeneration. Neuron. 23 (1), 181-192 (1999).
- Hoffner, G., Djian, P. Polyglutamine aggregation in Huntington disease: does structure determine toxicity. Molecular Neurobiology. 52 (3), 1297-1314 (2015).
- Waldvogel, H. J., Kim, E. H., Tippett, L. J., Vonsattel, J. P. G., Faull, R. L. M. The neuropathology of Huntington's disease. Current Topics in Behavioral Neurosciences. 22, 33-80 (2014).
- Kim, M. Beta conformation of polyglutamine track revealed by a crystal structure of huntingtin N-terminal region with insertion of three histidine residues. Prion. 7 (3), 221-228 (2013).
- Hoop, C. L., et al. Huntingtin exon 1 fibrils feature an interdigitated β-hairpin-based polyglutamine core. Proceedings of the National Academy of Sciences of the United States of America. 113 (6), 1546-1551 (2016).
- Vieweg, S., Ansaloni, A., Wang, Z. M., Warner, J. B., Lashuel, H. A. An intein-based strategy for the production of tag-free huntingtin exon 1 proteins enables new insights into the polyglutamine dependence of Httex1 aggregation and fibril formation. Journal of Biological Chemistry. 291 (23), 12074-12086 (2016).
- Seong, I. S., et al. Huntingtin facilitates polycomb repressive complex 2. Human Molecular Genetics. 19 (4), 573-583 (2009).
- Li, W., Serpell, L. C., Carter, W. J., Rubinsztein, D. C., Huntington, J. A. Expression and characterization of full-length human huntingtin, an elongated HEAT repeat protein. Journal of Biological Chemistry. 281 (23), 15916-15922 (2006).
- Vijayvargia, R., et al. Huntingtin's spherical solenoid structure enables polyglutamine tract-dependent modulation of its structure and function. eLife. 5, 11184(2016).
- Huang, B., et al. Scalable production in human cells and biochemical characterization of full-length normal and mutant huntingtin. PLoS ONE. 10 (3), 0121055(2015).
- Guo, Q., et al. The cryo-electron microscopy structure of huntingtin. Nature. 555 (7694), 117-120 (2018).
- Harding, R. J., et al. Design and characterization of mutant and wildtype huntingtin proteins produced from a toolkit of scalable eukaryotic expression systems. Journal of Biological Chemistry. 294 (17), 6986-7001 (2019).
- Harding, R. J., et al. HAP40 orchestrates huntingtin structure for 1 differential interaction with polyglutamine 2 expanded exon 1. bioRxiv. , (2021).
- Huang, B., et al. Pathological polyQ expansion does not alter the conformation of the Huntingtin-HAP40 complex. Structure. 29 (8), 804-809 (2021).
- Colin, E., et al. Huntingtin phosphorylation acts as a molecular switch for anterograde/retrograde transport in neurons. EMBO Journal. 27 (15), 2124-2134 (2008).
- Thompson, L. M., et al. IKK phosphorylates Huntingtin and targets it for degradation by the proteasome and lysosome. Journal of Cell Biology. 187 (7), 1083-1099 (2009).
- Ratovitski, T., et al. Post-translational modifications (PTMs), identified on endogenous Huntingtin, cluster within proteolytic domains between HEAT repeats. Journal of Proteome Research. 16 (8), 2692-2708 (2017).
- Taylor, S. C., Berkelman, T., Yadav, G., Hammond, M. A defined methodology for reliable quantification of western blot data. Molecular Biotechnology. 55 (3), 217-226 (2013).
- Tarazona, M. P., Saiz, E. Combination of SEC/MALS experimental procedures and theoretical analysis for studying the solution properties of macromolecules. Journal of Biochemical and Biophysical Methods. 56 (1-3), 95-116 (2003).
- Folta-Stogniew, E. Oligomeric states of proteins determined by size-exclusion chromatography coupled with light scattering, absorbance, and refractive index detectors. Methods in Molecular Biology. 328, Clifton, N.J. 97-112 (2006).
- McMeekin, T. L., Wilensky, M., Groves, M. L. Refractive indices of proteins in relation to amino acid composition and specific volume. Biochemical and Biophysical Research Communications. 7 (2), 151-156 (1962).
- Atwal, R. S., et al. Huntingtin has a membrane association signal that can modulate huntingtin aggregation, nuclear entry and toxicity. Human Molecular Genetics. 16 (21), 2600-2615 (2007).
- Kegel-Gleason, K. B. Huntingtin interactions with membrane phospholipids: Strategic targets for therapeutic intervention. Journal of Huntington's Disease. 2 (3), 239-250 (2013).
- Michalek, M., Salnikov, E. S., Werten, S., Bechinger, B. Membrane interactions of the amphipathic amino terminus of huntingtin. Biochemistry. 52 (5), 847-858 (2013).
- Wittig, I., Braun, H. P., Schägger, H. Blue native PAGE. Nature Protocols. 1 (1), 418-428 (2006).
- Nissley, D. A., O'Brien, E. P. Altered co-translational processing plays a role in huntington's pathogenesis-A hypothesis. Frontiers in Molecular Neuroscience. 9, 54(2016).
- Kumar, N., Gammell, P., Clynes, M. Proliferation control strategies to improve productivity and survival during CHO based production culture: A summary of recent methods employed and the effects of proliferation control in product secreting CHO cell lines. Cytotechnology. 53 (1-3), 33-46 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon