Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Flux de travail expérimental et d’analyse de données pour la nanoindentation de matière molle
Dans cet article
Résumé
Le protocole présente un flux de travail complet pour les expériences de nanoindentation de matériaux mous, y compris les hydrogels et les cellules. Tout d’abord, les étapes expérimentales pour acquérir des données de spectroscopie de force sont détaillées; ensuite, l’analyse de ces données est détaillée par un logiciel Python open-source nouvellement développé, téléchargeable gratuitement à partir de GitHub.
Résumé
La nanoindentation fait référence à une classe de techniques expérimentales où une sonde de force micrométrique est utilisée pour quantifier les propriétés mécaniques locales des biomatériaux mous et des cellules. Cette approche a acquis un rôle central dans les domaines de la mécanobiologie, de la conception de biomatériaux et de l’ingénierie tissulaire, afin d’obtenir une caractérisation mécanique correcte des matériaux mous avec une résolution comparable à la taille des cellules individuelles (μm). La stratégie la plus populaire pour acquérir de telles données expérimentales consiste à utiliser un microscope à force atomique (AFM); Bien que cet instrument offre une résolution sans précédent en force (jusqu’au pN) et en espace (sub-nm), sa facilité d’utilisation est souvent limitée par sa complexité qui empêche les mesures de routine d’indicateurs intégraux des propriétés mécaniques, tels que le module de Young (E). Une nouvelle génération de nanopénétrateurs, tels que ceux basés sur la technologie de détection de fibre optique, a récemment gagné en popularité pour sa facilité d’intégration tout en permettant d’appliquer des forces inférieures à nN avec une résolution spatiale en μm, ce qui convient pour sonder les propriétés mécaniques locales des hydrogels et des cellules.
Dans ce protocole, un guide étape par étape détaillant la procédure expérimentale pour acquérir des données de nanoindentation sur des hydrogels et des cellules à l’aide d’un nanopénétrateur de détection de fibre optique sur virole disponible dans le commerce est présenté. Alors que certaines étapes sont spécifiques à l’instrument utilisé ici, le protocole proposé peut servir de guide pour d’autres dispositifs de nanoindentation, à condition que certaines étapes soient adaptées conformément aux directives du fabricant. En outre, un nouveau logiciel Python open-source équipé d’une interface utilisateur graphique conviviale pour l’analyse des données de nanoindentation est présenté, qui permet le criblage des courbes mal acquises, le filtrage des données, le calcul du point de contact par différentes procédures numériques, le calcul conventionnel de E, ainsi qu’une analyse plus avancée particulièrement adaptée aux données de nanoindentation unicellulaire.
Introduction
Le rôle fondamental de la mécanique en biologie est aujourd’hui établi 1,2. Des tissus entiers aux cellules individuelles, les propriétés mécaniques peuvent renseigner sur l’état physiopathologique du biomatériau étudié 3,4. Par exemple, le tissu mammaire affecté par le cancer est plus rigide que le tissu sain, un concept qui est à la base du test de palpation populaire5. Il a notamment été récemment démontré que la maladie à coronavirus 2019 (COVID-19) causée par le coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2) est soulignée par des modifications des propriétés mécaniques des cellules sanguines, notamment une diminution de la déformabilité des érythrocytes et une diminution de la rigidité des lymphocytes et des neutrophiles par rapport aux cellules sanguines d’individus naïfs du SRAS-CoV-26.
En général, la mécanique des cellules et des tissus est intrinsèquement imbriquée : chaque tissu possède des propriétés mécaniques spécifiques qui influencent et dépendent simultanément de celles des cellules constitutives et de la matrice extracellulaire (MEC)5. Pour cette raison, les stratégies d’étude de la mécanique en biologie impliquent souvent des substrats d’ingénierie avec des stimuli mécaniques physiologiquement pertinents pour élucider le comportement cellulaire en réponse à ces stimuli. Par exemple, les travaux fondateurs d’Engler et de ses collègues ont démontré que l’engagement de la lignée des cellules souches mésenchymateuses est contrôlé par l’élasticité de la matrice, comme étudié sur des hydrogels de polyacrylamide bidimensionnels (PAAm) mous et rigides7.
Il existe de nombreuses stratégies pour caractériser mécaniquement le biomatériau à l’étude, variant en échelle spatiale (c.-à-d. locale à en vrac) et en mode de déformation (p. ex., axial vs cisaillement), ce qui donne des informations différentes, qui nécessitent une interprétation prudente 3,8,9,10. La mécanique des biomatériaux mous est généralement exprimée en termes de rigidité. Cependant, la rigidité dépend à la fois des propriétés du matériau et de la géométrie, tandis que les modules élastiques sont des propriétés fondamentales d’un matériau et sont indépendants de la géométrie du matériau11. Ainsi, différents modules d’élasticité sont liés à la rigidité d’un échantillon donné, et chaque module d’élasticité englobe la résistance du matériau à un mode de déformation spécifique (p. ex., axial vs cisaillement) dans différentes conditions aux limites (p. ex., expansion libre vs confinement)11,12. Les expériences de nanoindentation permettent de quantifier les propriétés mécaniques grâce au E qui est associé à une déformation uniaxiale (indentation) lorsque le biomatériau n’est pas confiné latéralement10,11,12.
La méthode la plus populaire pour quantifier l’E des systèmes biologiques à l’échelle microscopique est AFM13,14,15,16. AFM est un outil extrêmement puissant avec une résolution de force jusqu’au niveau pN et une résolution spatiale jusqu’à l’échelle sub-nm. En outre, AFM offre une flexibilité extrême en termes de couplage avec des outils optiques et mécaniques complémentaires, étendant ainsi ses capacités à extraire une mine d’informations du biomatériau à l’étude13. Ces caractéristiques attrayantes, cependant, s’accompagnent d’une barrière à l’entrée représentée par la complexité du dispositif expérimental. L’AFM nécessite une formation approfondie avant que les utilisateurs puissent acquérir des données robustes, et son utilisation pour la caractérisation mécanique quotidienne des matériaux biologiques est souvent injustifiée, en particulier lorsque sa force unique et ses résolutions spatiales ne sont pas requises.
Pour cette raison, une nouvelle classe de nanopénétrateurs a récemment gagné en popularité en raison de leur facilité d’utilisation, tout en offrant des données comparables AFM avec une résolution de force inférieure à nN et une résolution spatiale μm, reflétant les forces exercées et perçues par les cellules sur des échelles de longueur pertinentes2. En particulier, les dispositifs de nanoindentation à virole basés sur la technologie de détection de fibre optique17,18 ont gagné en popularité parmi les chercheurs actifs dans le domaine de la mécanobiologie et au-delà; et une multitude de travaux rapportant les propriétés mécaniques des biomatériaux utilisant ces dispositifs, y compris les cellules19,20, les hydrogels8,21 et les tissus22,23 ont été publiés. Malgré la capacité de ces systèmes à sonder les propriétés mécaniques dynamiques locales (c.-à-d. le module de stockage et de perte), les expériences quasi statiques donnant E restent le choix le plus populaire 8,19,20,21. En bref, les expériences quasi-statiques de nanoindentation consistent à indenter l’échantillon avec une vitesse constante jusqu’à un point de consigne défini soit par un déplacement maximal, une force ou une profondeur d’indentation, et à enregistrer à la fois la force et la position verticale du porte-à-faux dans des courbes dites force-distance (F-z). Les courbes F-z sont ensuite converties en courbes d’indentation de force (F-δ) par l’identification du point de contact (CP), et équipées d’un modèle de mécanique de contact approprié (généralement le modèleHertz 13) pour calculer E.
Bien que le fonctionnement des nanopénétrateurs à dessus de la ferrule ressemble aux mesures AFM, il existe des spécificités à prendre en compte. Dans ce travail, un guide étape par étape pour acquérir de manière robuste des courbes F-z à partir de cellules et d’hydrogels imitant les tissus à l’aide d’un nanopénétrateur de virole disponible dans le commerce est fourni, afin d’encourager la normalisation des procédures expérimentales entre les groupes de recherche utilisant ce dispositif et d’autres dispositifs similaires. En outre, des conseils sur la meilleure façon de préparer des échantillons d’hydrogel et des cellules pour effectuer des expériences de nanoindentation sont donnés, ainsi que des conseils de dépannage tout au long de la voie expérimentale.
De plus, une grande partie de la variabilité des résultats de nanoindentation (c.-à-d. E et sa distribution) dépend de la procédure spécifique utilisée pour analyser les données, qui n’est pas triviale. Pour résoudre ce problème, des instructions pour l’utilisation d’un logiciel open source nouvellement développé programmé en Python et équipé d’une interface utilisateur graphique (GUI) conviviale pour l’analyse par lots des courbes F-z sont fournies. Le logiciel permet un filtrage rapide des données, le filtrage des données, le calcul du CP à travers différentes procédures numériques, le calcul conventionnel de E, ainsi qu’une analyse plus avancée appelée spectres d’élasticité24, permettant d’estimer le module de Young de la cellule, le module de Young du cortex d’actine et l’épaisseur du cortex d’actine. Le logiciel peut être téléchargé gratuitement à partir de GitHub et peut être facilement adapté pour analyser des données provenant d’autres systèmes en ajoutant un analyseur de données approprié. Il est souligné que ce protocole peut être utilisé pour d’autres dispositifs de nanoindentation à haut de la virole, et d’autres dispositifs de nanoindentation en général, à condition que certaines étapes soient adaptées selon les directives de l’instrument spécifique. Le protocole est schématiquement résumé à la figure 1.
Protocole
1. Préparation des substrats/cellules pour les mesures de nanoindentation
- Suivez les étapes indiquées dans le Protocole additionnel pour la préparation des hydrogels/cellules PAAm pour les expériences de nanoindentation. La procédure est résumée à la figure 2.
NOTE: Les hydrogels PAAm ont été choisis car ce sont les hydrogels les plus couramment utilisés dans le domaine de la mécanobiologie. Cependant, le protocole est également applicable à tout type d’hydrogel25 (voir Discussion, modifications de la méthode).
2. Démarrage de l’appareil, choix de la sonde et étalonnage de la sonde
- Suivez les étapes indiquées dans le protocole supplémentaire pour démarrer l’appareil. Pour plus de détails techniques sur le fonctionnement des nanopénétrateurs à fibre optique, consultez ces références17,18.
- Sélectionnez la sonde de nanoindentation comme décrit ci-dessous.
REMARQUE : Toutes les sondes disponibles dans le commerce, telles que celles utilisées dans ce protocole, sont équipées d’une pointe sphérique (Figure 3A). Par conséquent, le choix se réduit à deux variables : la rigidité du porte-à-faux et le rayon de pointe (Figure 3B).- Choisissez la rigidité d’un porte-à-faux (k dans N/m) qui correspond à la rigidité attendue de l’échantillon pour obtenir les meilleurs résultats15 (voir Figure 3C et Discussion, étapes critiques du protocole). Pour les cellules, sélectionnez une sonde avec k compris entre 0,01 et 0,09 N/m. Pour les hydrogels, sélectionnez une sonde avec k dans la plage de 0,1 à 0,9 N/m, ce qui donne des résultats optimaux pour les gels dont l’indice E attendu se situe entre quelques kPa et 100 kPa (voir Résultats représentatifs).
- Choisissez le rayon de la pointe (R en μm) en fonction de la résolution spatiale souhaitée du processus d’indentation. Pour les petites cellules telles que les cellules 293T (rein embryonnaire humain) (HEK293T) (diamètre moyen de ~10-15 μm26), sélectionnez une sphère avec R = 3 μm. Pour les hydrogels, sélectionnez une sphère avec R = 10-250 μm pour sonder les propriétés mécaniques du biomatériau sur une grande surface de contact et éviter les hétérogénéités locales.
- Consultez la figure 3C et les directives supplémentaires du fabricant pour choisir la sonde appropriée.
- Une fois la sonde sélectionnée, suivez les étapes du protocole additionnel pour la monter sur le nanopénétrateur.
3. Calibrage de la sonde
REMARQUE: Les étapes suivantes sont spécifiques aux dispositifs de nanoindentation à virole basés sur la technologie de détection de fibre optique, et elles sont détaillées pour la version logicielle 3.4.1. Pour les autres dispositifs de nanoindentation, suivez les étapes recommandées par le fabricant de l’appareil.
- Dans la fenêtre principale du logiciel, cliquez sur Initialiser. Un menu d’étalonnage apparaîtra. Entrez les détails de la sonde (se trouvent sur le côté de la boîte de la sonde; k en N/m, R en μm et le facteur d’étalonnage en air) dans les zones d’entrée.
- Préparez la boîte d’étalonnage : une boîte de Petri en verre épais à fond plat (voir Discussion, étapes critiques dans le protocole). Remplissez le plat avec le même milieu que le plat d’échantillon (cela peut aussi être de l’air). Faire correspondre la température du milieu avec celle de l’échantillon.
- Placez le plat d’étalonnage sous la sonde. Si nécessaire, faites glisser la sonde du support de bras du nanopénétrateur et tenez-la dans une main pour faire de la place pour le placement de la parabole d’étalonnage. Faites glisser la sonde en place.
- Effectuez les deux étapes suivantes pour l’étalonnage dans un liquide. Si vous mesurez dans l’air, utilisez le substrat en polytétrafluoroéthylène fourni pour l’étalonnage et passez à l’étape 5.
- Prémouiller la sonde avec une goutte d’éthanol à 70% à l’aide d’une pipette Pasteur avec l’extrémité de la pipette en contact léger avec la virole en verre, pour que la goutte glisse sur le porte-à-faux et la pointe sphérique27 (voir Discussion, étapes critiques dans le protocole).
- Faites glisser manuellement le bras du nanopénétreur vers le bas jusqu’à ce que la sonde soit complètement immergée, mais toujours loin du fond de la boîte de Pétri. Si nécessaire, ajouter plus de milieu à la boîte d’étalonnage. Attendez 5 minutes pour que les conditions d’équilibre soient atteintes dans le liquide.
- Dans le menu Initialiser du logiciel, cliquez sur Scan Wavelength. L’écran de l’interféromètre affiche une barre de progression et la fenêtre Live Signal du logiciel de l’ordinateur affiche le motif illustré à la figure S1A, à gauche. Pour vérifier si le balayage optique a réussi, accédez au panneau Balayage de longueur d’onde sur le boîtier de l’interféromètre. En cas de succès, une onde sinusoïdale devrait être visible (Figure 1A, à droite). Voir Discussion (dépannage de la méthode) si une erreur apparaît.
- Dans le menu Initialiser , cliquez sur Rechercher une surface, ce qui abaissera progressivement la sonde jusqu’à ce qu’un seuil défini dans la flexion du porte-à-faux soit atteint. La sonde cesse de bouger lorsque le contact avec la boîte de Petri en verre est établi.
- Vérifiez si la sonde est en contact avec la surface. Déplacez la sonde vers le bas de 1 μm à l’aide du bouton flèche y vers le bas de la fenêtre principale du logiciel. Observez le signal vert (déviation du porte-à-faux) dans la fenêtre Live du logiciel pour les changements dans la ligne de base à chaque étape, lorsque le porte-à-faux est en contact avec le substrat (Figure S1B). S’il n’y a pas de changement, le porte-à-faux n’est pas en contact (voir étape suivante).
- Augmentez la valeur de seuil dans le menu Options sous l’onglet Rechercher une surface par rapport à sa valeur par défaut de 0,01 d’une étape de 0,01 à la fois et répétez l’étape Rechercher une surface jusqu’au contact. Sinon, abaissez la sonde au contact par petits pas de 1 μm jusqu’à ce que la ligne de base verte commence à se déplacer à chaque étape vers le bas.
REMARQUE: Pour les porte-à-faux les plus mous (k = 0,025 Nm-1), augmentez la valeur de seuil dans le menu Options sous l’onglet Rechercher une surface a priori de l’exécution de l’étape 6. Partez d’une valeur seuil de 0,06 ou 0,07 et augmentez-la jusqu’à 0,1 si nécessaire. En effet, le bruit ambiant entraînera probablement une courbure du porte-à-faux au-dessus du seuil avant le contact. Pour les sondes souples, la diminution de la vitesse d’approche (μm/s) dans le même menu améliore également la procédure. - Dans le menu Initialiser , cliquez sur Calibrer.
- Vérifiez la fenêtre Live Signal du logiciel et assurez-vous que les signaux de déplacement du piézo et de déviation du porte-à-faux montent en même temps (Figure S1C).
- S’il y a un décalage dans le temps, la sonde n’est pas complètement en contact avec le verre. Déplacez la sonde vers le bas par pas de 1 μm jusqu’à ce que la ligne de base du signal en porte-à-faux change (voir étape 7) et répétez l’étape 9.
- Si le signal en porte-à-faux ne change pas du tout pendant l’étape d’étalonnage , la sonde est loin de la surface. Augmentez le seuil de contact de manière itérative (voir étape 8) jusqu’à ce que la surface soit trouvée correctement et répétez l’étalonnage depuis le début en commençant par le balayage de longueur d’onde.
- Lorsque l’étalonnage est terminé, vérifiez les anciens et les nouveaux facteurs d’étalonnage dans la fenêtre contextuelle affichée. Si le nouveau facteur d’étalonnage se situe dans la plage correcte, cliquez sur Utiliser un nouveau facteur. Si l’étalonnage échoue et que le nouveau facteur est NaN ou n’est pas dans la plage attendue, reportez-vous à la section Discussion (dépannage de la méthode) pour la résolution.
NOTE: Le nouveau facteur doit être ~n fois inférieur à celui fourni sur la boîte de la sonde si l’étalonnage a été effectué dans un milieu liquide avec un indice de réfraction n (n = 1,33 pour l’eau). Si l’étalonnage a été effectué dans l’air, les nouveaux et les anciens facteurs d’étalonnage doivent être approximativement égaux. - Vérifiez si le cercle de démodulation a été correctement calibré comme suit. Accédez à l’onglet Démodulation sur le bureau de l’interféromètre. Tapotez doucement sur la table optique ou sur le nanopénétrateur pour induire suffisamment de bruit. Un cercle blanc composé de points de données discrets devrait couvrir approximativement le cercle rouge (figure S1D).
- Si le cercle blanc ne chevauche pas le cercle rouge ou si un avertissement apparaît sur l’écran de l’interféromètre, le cercle de démodulation doit être recalibré. Cela peut être fait de deux manières, comme décrit ci-dessous.
- Appuyez continuellement sur le corps du nanopénétrateur pour induire un cercle complet de bruit et appuyez sur le bouton Calibrer de l’interféromètre.
- Entrez en contact avec le substrat de verre et appuyez sur Calibrer dans le menu Initialiser de la fenêtre principale du logiciel. N’enregistrez pas le facteur d’étalonnage. À ce stade, vérifiez à nouveau et assurez-vous que le cercle blanc chevauche le cercle rouge.
REMARQUE: Si le signal n’est pas seulement légèrement déplacé du cercle de démodulation, mais est devenu très petit ou n’est pas visible du tout, cela signifie que le porte-à-faux est collé à la fibre optique. Suivez les conseils de dépannage pour ce problème (voir Discussion, dépannage de la méthode) et répétez les étapes 13.1 ou 13.2. Une fois que le porte-à-faux revient à sa position horizontale (Figure 3A), le signal reviendra dans le cercle de démodulation.
- Vérifiez l’étalonnage en effectuant une indentation sur le substrat de verre directement après l’étalonnage comme décrit ci-dessous.
- Chargez ou créez un fichier d’expérience en cliquant sur Configurer l’expérience et ajoutez une étape Rechercher une surface et une étape Indentation . Pour l’étape d’indentation, utilisez les paramètres de mode de déplacement par défaut et définissez le déplacement maximal sur la distance d’étalonnage (3 000 nm) pour déplacer la sonde contre le substrat rigide.
- Cliquez sur Run Experiment et vérifiez le cercle de démodulation dans la fenêtre de l’interféromètre. Vérifiez le signal blanc et assurez-vous qu’il se trouve au-dessus du cercle rouge lors de l’indentation.
- Vérifiez les résultats dans la fenêtre principale du logiciel dans le graphique Données temporelles et assurez-vous que le déplacement du piézo (ligne bleue) est égal à la déviation du porte-à-faux (ligne verte) lorsque l’indentation commence au contact et qu’aucune déformation du matériau n’est attendue. Si les signaux ne sont pas parallèles, voir Discussion (dépannage de la méthode).
- Modifiez le chemin local dans le menu d’étalonnage en définissant le chemin d’enregistrement de l’étalonnage sur un répertoire approprié.
- Lorsque la sonde a été calibrée avec succès, déplacez le piézo vers le haut de 500 μm.
4. Mesure du module de Young des matériaux souples
- Nanoindentation d’hydrogels
- Chargez la boîte de Petri contenant le(s) échantillon(s) sur la platine du microscope et déplacez manuellement la sonde du nanopénétrateur à une position x-y souhaitée au-dessus de l’échantillon.
- Faites glisser manuellement la sonde en solution, en prenant soin de laisser 1-2 mm entre la sonde et la surface de l’échantillon à ce stade. Attendez 5 minutes que la sonde s’équilibre dans le milieu.
- Focalisez le plan z du microscope optique de manière à ce que la sonde soit clairement visible.
- Effectuez une seule indentation pour régler les paramètres expérimentaux comme décrit ci-dessous.
- Configurez une nouvelle expérience dans la fenêtre principale du logiciel. Cliquez sur Configurer l’expérience, ce qui ouvrira une nouvelle fenêtre. Ajoutez une étape Rechercher une surface . Tous les paramètres de l’étape Rechercher une surface peuvent être modifiés dans le menu Options du logiciel si nécessaire.
REMARQUE : La surface de recherche abaissera la sonde jusqu’à ce que la surface soit trouvée, puis rétractera la sonde à une distance définie par Z au-dessus de la surface (μm), au-dessus de la surface de l’échantillon. Si le porte-à-faux choisi est trop rigide pour l’échantillon ou si l’échantillon est collant, après l’étape, la sonde est susceptible d’être encore en contact avec l’échantillon, ce qui entraînera une courbe sans ligne de base (Figure 4C). Pour résoudre ce problème, augmentez le Z au-dessus de la surface (μm). - Ajoutez une étape de retrait . Sélectionnez l’onglet Profil et cliquez sur Contrôle de déplacement. Conservez le profil de retrait par défaut.
- Cliquez sur Run Experiment dans la fenêtre principale du logiciel. Cela permettra de trouver la surface et d’effectuer une seule indentation. Si l’indentation unique ne ressemble pas aux attentes, ajustez les paramètres expérimentaux comme indiqué à la figure 4 et à la section Discussion (dépannage de la méthode).
- Configurez une nouvelle expérience dans la fenêtre principale du logiciel. Cliquez sur Configurer l’expérience, ce qui ouvrira une nouvelle fenêtre. Ajoutez une étape Rechercher une surface . Tous les paramètres de l’étape Rechercher une surface peuvent être modifiés dans le menu Options du logiciel si nécessaire.
- Une fois que l’indentation semble comme vous le souhaitez, configurez l’analyse matricielle de sorte qu’une zone suffisante de l’échantillon soit mise en retrait. Cliquez sur Configurer l’expérience, ajoutez une étape de recherche de surface avec les paramètres expérimentaux précédemment déterminés et ajoutez une étape de balayage matriciel .
- Pour les hydrogels plats, configurez un balayage matriciel contenant 50 à 100 points (c.-à-d. 5 x 10 ou 10 x 10 po x et y) espacés de 10 à 100 μm (c.-à-d. dx = dy = 10-100 μm). Cliquez sur Utiliser la position de la scène pour lancer l’analyse matricielle à partir de la position actuelle de l’étape. Assurez-vous que la case Recherche automatique de surface est cochée pour trouver la surface à chaque indentation à l’aide des paramètres expérimentaux définis.
- Pour éviter le suréchantillonnage, définissez la taille de l’étape sur au moins deux fois le rayon de contact (
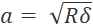 , où δ est la profondeur d’indentation).
, où δ est la profondeur d’indentation). - Configurez le profil de balayage matriciel dans le contrôle de déplacement. Assurez-vous que le profil ne viole pas les hypothèses du modèle Hertz (voir Discussion, étapes critiques dans le protocole).
- Laissez le nombre de segments à 5, qui est la valeur par défaut, et utilisez le profil de déplacement par défaut. Si nécessaire, modifiez le profil de déplacement en termes de déplacement maximal et de temps pour chaque segment incliné, ce qui affectera respectivement la profondeur d’indentation maximale et la vitesse de déformation. Ne pas dépasser les vitesses de déformation > 10 μm/s (voir Discussion, limites de la méthode).
- Entrez une valeur pour la vitesse d’approche, qui détermine la vitesse à laquelle la sonde est déplacée vers l’échantillon avant le contact. Faire correspondre la vitesse de rétraction à la vitesse d’approche (voir note ci-dessous).
REMARQUE: Pour les porte-à-faux souples et les environnements bruyants, une vitesse d’approche de 1 000 à 2 000 nm / s est recommandée. Pour les porte-à-faux plus rigides et les environnements contrôlés, cela peut être augmenté. - Enregistrez l’expérience configurée dans le chemin d’expérience souhaité et sélectionnez un répertoire dans lequel les données seront enregistrées dans le chemin d’enregistrement, dans l’onglet Général de la fenêtre Configurer l’expérience . Cliquez sur Run Experiment (Exécuter l’expérience).
- Pour éviter le suréchantillonnage, définissez la taille de l’étape sur au moins deux fois le rayon de contact (
- Une fois le balayage matriciel terminé, augmenter la sonde de 200 à 500 μm et déplacer la sonde vers une autre zone de l’échantillon suffisamment éloignée de la première zone.
- Répéter l’expérience au moins deux fois afin d’obtenir suffisamment de données sur chaque échantillon (c.-à-d. au moins deux balayages matriciels par échantillon, contenant 50 à 100 courbes chacun).
- Nanoindentation de cellules
- Chargez l’échantillon sur le microscope comme décrit ci-dessus.
- Pour l’indentation d’une seule cellule, concentrez le plan z de sorte que les cellules et la sonde soient visibles à un grossissement de 20x ou 40x, selon la taille et l’étalement de la cellule.
- Déplacez la sonde au-dessus de la cellule à mettre en retrait.
- Configurez une nouvelle expérience dans la fenêtre principale du logiciel. Cliquez sur Configurer l’expérience, ce qui ouvrira une nouvelle fenêtre. Ajoutez une étape Rechercher une surface et une indentation avec les paramètres par défaut en mode déplacement.
- Cliquez sur Run Experiment, qui trouvera la surface et effectuera une seule indentation. Vérifiez si l’indentation a réussi. Si la courbe ne ressemble pas aux attentes, ajustez les paramètres expérimentaux (voir Figure 4 et Discussion, dépannage de la méthode).
- Si l’indentation a réussi, ajoutez un balayage matriciel à l’expérience. Suivez les étapes données pour les expériences de nanoindentation des hydrogels; configurer l’analyse matricielle de sorte que la taille de l’étape permette de mettre en retrait une petite zone de la cellule ; 25 points espacés de 0,5 à 5 μm pour les cellules HEK293T.
- Selon la taille de la cellule, adaptez le balayage matriciel pour vous assurer que la pointe ne s’indente pas en dehors de la limite de la cellule, c’est-à-dire en effectuant une géométrie de carte différente ou en sondant moins de points.
- Cliquez sur Run Experiment et attendez qu’il soit terminé.
- Une fois le balayage matriciel terminé, soulevez la sonde hors de contact (50 μm dans le plan z ).
- Déplacez la sonde au-dessus d’une nouvelle cellule et répétez le processus (voir Discussion, étapes critiques dans le protocole).
- Nettoyage de la sonde et extinction de l’instrument
- Suivez les étapes indiquées dans le protocole additionnel pour nettoyer la sonde et éteindre le dispositif de nanoindentation.
5. Analyse des données
- Téléchargement et installation du logiciel
- Suivez les étapes indiquées dans le Protocole additionnel pour télécharger et installer le logiciel d’analysedes données 28,29.
- Criblage des courbes F-z et production d’un ensemble de données nettoyées au format JSON
- Lancez prepare.py à partir de la ligne de commande sur l’ordinateur de laboratoire, comme indiqué aux étapes 2 et 3.
- Si vous utilisez un ordinateur Windows, maintenez la touche Maj enfoncée tout en cliquant avec le bouton droit sur le dossier NanoPrepare et cliquez sur Ouvrir la fenêtre PowerShell ici. Tapez la commande python prepare.py et appuyez sur la touche Entrée . Une interface graphique apparaîtra sur votre écran (Figure S2).
- Si vous utilisez un ordinateur MacOS, cliquez avec le bouton droit de la souris sur le dossier NanoPrepare et cliquez sur Nouveau terminal dans le dossier. Tapez la commande python3 prepare.py et appuyez sur la touche Entrée , ce qui lancera l’interface graphique (Figure S2).
- Sélectionnez le format de données O11NEW dans la liste déroulante. Si les données ne sont pas chargées correctement, relancez l’interface graphique et sélectionnez O11OLD.
REMARQUE: Le format O11NEW fonctionne pour les données obtenues à l’aide de dispositifs de nanoindentation à fibre optique à base de virole avec la version logicielle 3.4.1. Ce format fonctionnera également pour les versions précédentes du logiciel, du moins celles appartenant aux nanopénétrateurs installés en 2019-2020. - Cliquez sur Charger le dossier. Sélectionnez un dossier contenant les données à analyser : analyse matricielle unique ou analyse matricielle multiple. Le graphique du haut (courbes brutes) sera rempli avec l’ensemble de données téléchargé. Pour visualiser une courbe spécifique, cliquez dessus. Cela le mettra en surbrillance en vert et l’affichera sur le graphique du bas (courbe actuelle).
- Nettoyez l’ensemble de données à l’aide des onglets présents dans la partie droite de l’interface graphique, comme indiqué ci-dessous.
- Utilisez le bouton Segment pour sélectionner le segment correct à analyser, qui est le segment avant des courbes F-z . Le nombre spécifique dépend du nombre de segments sélectionnés dans le logiciel de nanoindentation lors de la réalisation d’expériences.
- Utilisez le bouton Recadrer 50 nm pour rogner les courbes de 50 nm à l’extrême gauche (si L est coché), à droite (si R est coché) ou des deux côtés (si R et L sont cochés). Cliquez plusieurs fois sur ce bouton pour recadrer autant que nécessaire. Utilisez cette option pour supprimer les artefacts présents au début/à la fin des courbes F-z.
- Examinez la languette Cantilever pour la constante du ressort, la géométrie de la pointe et le rayon de la pointe. Inspectez l’onglet pour vous assurer que les métadonnées ont été lues correctement.
- Utilisez l’onglet Filtrage pour définir un seuil de force qui annulera toutes les courbes qui n’ont pas atteint la force donnée. Les courbes abandonnées seront surlignées en rouge.
- Utilisez le bouton bascule manuel pour supprimer manuellement les courbes qui n’ont pas été correctement acquises. Supprimez toutes les courbes en cliquant sur la courbe spécifique et en sélectionnant OUT, ce qui mettra la courbe en surbrillance en rouge.
- Cliquez sur Enregistrer JSON. Entrez un nom approprié pour l’ensemble de données nettoyé, qui est un fichier JSON unique. Envoyez le fichier JSON à l’ordinateur sur lequel le logiciel NanoAnalysis a été installé.
6. Analyse formelle des données
- Lancez le fichier nano.py à partir de la ligne de commande en accédant au dossier NanoAnalysis et en lançant un terminal comme expliqué précédemment. Tapez la commande python nano.py ou python3 nano.py (selon le système d’exploitation) et appuyez sur la touche Entrée . Une interface graphique apparaîtra sur votre écran (Figure S3).
- En haut à gauche de l’interface graphique, cliquez sur Load Experiment et sélectionnez le fichier JSON. Cela remplira la liste des fichiers et le graphique Courbes brutes montrant l’ensemble de données en termes de courbes F-z. Les axes F et z sont affichés par rapport aux coordonnées CP, qui sont calculées en arrière-plan lors du chargement du jeu de données (voir la remarque suivante). Dans la zone Statistiques, vérifiez les valeurs des trois paramètres : Nactivé, N a échoué et Nexclu.
REMARQUE : Nactivé représente le nombre de courbes qui seront analysées dans l’analyse ultérieure des spectres Hertz/élasticité et qui apparaissent en noir dans le graphique Courbes brutes . Nfailed représente le nombre de courbes sur lesquelles un CP fiable n’a pas pu être trouvé et apparaissent en bleu dans le graphique. Ces courbes seront automatiquement écartées dans l’analyse ultérieure. Lors de l’ouverture du logiciel, certaines courbes peuvent être automatiquement déplacées vers le jeu défaillant. En effet, le CP est calculé à l’ouverture du logiciel avec l’algorithme de seuil par défaut (voir ci-dessous). Nexclu représente les courbes sélectionnées manuellement pour être exclues de l’analyse et apparaîtra en rouge dans le graphique (voir ci-dessous). - Vérifiez si le nombre de courbes échouées, exclues et activées est raisonnable. Consultez le graphique Courbes brutes pour visualiser les courbes.
- Pour visualiser une courbe spécifique plus en détail, cliquez dessus. Cela le mettra en surbrillance en vert et l’affichera sur le graphique Courbe actuelle . Une fois qu’une seule courbe a été sélectionnée, les paramètres R et k (qui doivent être les mêmes pour toutes les courbes) seront renseignés dans la zone Statistiques de l’interface graphique.
- Modifiez l’état d’une courbe donnée à l’aide de la zone Basculer . Cliquez sur la courbe spécifique dont vous souhaitez modifier l’état, puis cliquez sur Activé, Échec ou Exclu. Le nombre dans la zone Statistiques est automatiquement mis à jour.
Remarque : Pour modifier l’affichage de l’ensemble de données dans le graphique Courbes brutes , utilisez la zone Affichage . Cliquez sur Tout pour afficher toutes les courbes (c’est-à-dire activées, échouées et exclues dans les couleurs respectives). Cliquez sur Impossible d’afficher les courbes activées et les courbes échouées et cliquez sur Activé pour afficher uniquement les courbes activées. Pour réinitialiser l’état de toutes les courbes entre activé et exclu, cliquez sur Activé ou Exclu dans la zone Réinitialiser . - Une fois que le jeu de données a été nettoyé, suivez le pipeline d’analyse des données décrit ci-dessous.
- Filtrez tout bruit présent dans les courbes à l’aide des filtres implémentés dans l’interface graphique (Filtering Box), à savoir un filtre personnalisé appelé filtre Prominency, le filtre Savitzky Golay30,31 (SAVGOL) et un filtre de lissage basé sur le calcul de la médiane des données dans une fenêtre donnée (filtre médian). Voir Discussion (étapes critiques du protocole) pour plus de détails sur les filtres.
- Examinez les courbes filtrées dans le graphique Courbe actuelle . La courbe filtrée est représentée en noir tandis que la version non filtrée de la courbe est représentée en vert.
REMARQUE: Il est conseillé de filtrer les données le moins possible pour préserver les caractéristiques du signal d’origine. Le filtrage excessif peut lisser les différences présentes dans les données. Travailler avec le filtre de proéminence activé est suffisant pour l’analyse Hertz des données de nanoindentation. Si les données sont particulièrement bruyantes, un filtre SAVGOL ou médian peut également être appliqué. - Sélectionnez un algorithme pour trouver le CP. Dans la zone Point de contact, choisissez l’une des nombreuses procédures numériques implémentées dans le logiciel, à savoir la qualité de l’ajustement (GoF)32, le rapport de variances (RoV)32, la dérivée secondaire 33 ou le seuil 33. Voir Discussion, étapes critiques du protocole pour plus de détails sur les algorithmes.
REMARQUE: Le CP est le point auquel la sonde entre en contact avec le matériau et doit être identifié afin de convertir les données F-z en données F-δ (pour les matériaux mous, δ est fini et doit être calculé). L’algorithme sélectionné sera appliqué à toutes les courbes actives de l’ensemble de données, et les courbes où l’algorithme ne parvient pas à localiser le CP de manière robuste seront déplacées vers l’ensemble défaillant. - Ajustez les paramètres de l’algorithme en fonction de votre jeu de données afin que le CP soit localisé correctement, comme indiqué dans la discussion, étapes critiques du protocole. Pour voir où le CP a été trouvé sur une seule courbe, sélectionnez la courbe en cliquant dessus et cliquez sur Inspecter. Vérifiez la fenêtre contextuelle qui apparaît pour identifier l’emplacement du CP.
- Vérifiez si la ligne rouge qui apparaît, qui est le paramètre que l’algorithme a calculé dans la région d’intérêt sélectionnée, a une valeur maximale ou minimale qui correspond à l’emplacement du CP (par exemple, pour le GdF, le paramètre est le R2). Répétez ce processus pour toutes les courbes si nécessaire.
REMARQUE: Les axes de la fenêtre contextuelle sont en coordonnées absolues afin que l’emplacement du CP puisse être affiché. Inversement, les axes des courbes brutes et du graphique de la courbe actuelle sont représentés par rapport au CP, c’est-à-dire que l’emplacement du CP est (0,0). - Cliquez sur Analyse Hertz. Cela générera trois graphiques décrits ci-dessous.
- Vérifiez les courbes F-δ individuelles dans l’ensemble de données ainsi que l’ajustement moyen en Hertz (ligne pointillée rouge). Ajustez l’indentation en nm jusqu’à laquelle le modèle Hertz est ajusté dans la zone Résultats sous Indentation maximale (nm). Réglez-le sur un maximum de ~10% de R pour que le modèle Hertz soit valide (voir Discussion, étapes critiques dans le protocole).
- Vérifiez la courbe moyenne F-δ avec la bande d’erreur montrant un écart type (ET) ainsi que l’ajustement Hertz moyen (ligne pointillée rouge). Visualisez l’ajustement Hertz moyen sur le graphique Courbes brutes pour référence et l’ajustement Hertz pour chaque courbe sur la courbe actuelle.
- Vérifiez le nuage de points de E provenant de l’ajustement du modèle Hertz à chaque courbe individuelle.
- Cliquez sur le nom du fichier, la courbe F-z , la courbe F-δ ou un point sur le nuage de points pour mettre en surbrillance la courbe de chaque tracé. Si un point de données dans le nuage de points semble se trouver en dehors de la distribution des données, cliquez dessus et inspectez la courbe à laquelle il appartient. Vérifiez que le CP a été localisé correctement en cliquant sur le bouton Inspecter . Exclure la courbe de l’analyse si nécessaire.
- Examinez la boîte Résultats pour la moyenne calculée E et son écart-type (Eγ ± σ) et assurez-vous qu’ils sont raisonnables pour l’expérience donnée.
- Dans la zone Enregistrer , cliquez sur Hertz. Dans la fenêtre contextuelle, entrez le nom du fichier et le répertoire. Une fois terminé, cliquez sur Enregistrer. Un fichier .tsv sera créé. Ouvrez le fichier .tsv dans n’importe quel logiciel supplémentaire de votre préférence et utilisez les valeurs pour l’analyse statistique et le traçage ultérieur.
Remarque : Le fichier contient le E obtenu à partir de chaque courbe et la moyenne E et son écart-type. En outre, le fichier contient les métadonnées associées à l’analyse, y compris le nombre de courbes analysées, R, k et l’indentation maximale utilisée pour le modèle Hertz. - Cette étape est facultative. Cliquez sur Average F-Ind pour exporter la force moyenne et l’indentation moyenne, ainsi qu’un SD dans la force.
- Pour les données de nanoindentation cellulaire, cliquez sur Analyse des spectres d’élasticité (voir Résultats représentatifs et discussion). Inspecter les deux tracés produits, à savoir, E en fonction de la profondeur d’indentation (E(δ)) pour chaque courbe, et la moyenne E(δ) avec bande d’erreur montrant un SD (ligne rouge continue et zone ombrée) ajustée par un modèle (ligne pointillée noire), ce qui permet d’estimer le module de Young du cortex d’actine de la cellule, le module de Young de la cellule, et l’épaisseur du cortex d’actine. De plus, vérifiez la moyenne E(δ) dans le graphique du haut en rouge.
- Assurez-vous que la case Interpoler est cochée, ce qui garantit que la dérivée nécessaire pour effectuer l’analyse des spectres d’élasticité est calculée sur le signal interpolé (voir Résultats représentatifs).
- Inspectez la boîte Résultats, en indiquant le module de Young du cortex (E 0 ± σ), le module de Young de la cellule (Eb ± σ) et l’épaisseur du cortex (d 0 ± σ).
REMARQUE: Les spectres d’élasticité moyens peuvent sembler bruyants au début, avec des oscillations sinusoïdales proéminentes. Par conséquent, l’équation (3) peut ne pas être ajustée correctement. Si tel est le cas, l’augmentation itérative de la longueur de la fenêtre du filtre de lissage SAVGOL34 résout ce problème. - Une fois l’analyse terminée, cliquez sur ES dans la case Enregistrer . Cela exportera un fichier .tsv dans le répertoire spécifié, contenant l’élasticité moyenne en fonction de la profondeur d’indentation moyenne et du rayon de contact, les métadonnées associées à l’expérience (voir ci-dessus) et les paramètres estimés du modèle expliqués ci-dessus. Enfin, l’élasticité moyenne sans tenir compte de la dépendance à l’égard de δ et de son écart-type est également rapportée.
- Fermez le logiciel et entrez les résultats enregistrés dans tout autre logiciel de votre préférence pour tracer davantage les données et effectuer une analyse statistique.
- Cette étape est facultative : Exportez des graphiques à partir de l’interface graphique en cliquant avec le bouton droit sur le graphique et en sélectionnant Exporter. Exportez le graphique dans .svg, de sorte que les paramètres tels que la police, la taille de la police, le style de ligne, etc. peut être édité dans un autre logiciel de votre choix.
REMARQUE:Des algorithmes et des filtres CP personnalisés peuvent être programmés et ajoutés à ceux déjà existants. Voir la note complémentaire 1 pour plus de détails.
Résultats
En suivant le protocole, un ensemble de courbes F-z est obtenu. L’ensemble de données contiendra très probablement de bonnes courbes et des courbes à écarter avant de poursuivre l’analyse. En général, les courbes doivent être écartées si leur forme est différente de celle de la figure 4A. La figure 5AI montre un ensemble de données de ~100 courbes obtenues sur un hydrogel PAAm mou de E 0,8 KPa35 attendu t?...
Discussion
Ce protocole montre comment acquérir de manière robuste des données de nanoindentation par spectroscopie de force à l’aide d’un nanopénétrateur de virole disponible dans le commerce sur des hydrogels et des cellules individuelles. En outre, des instructions pour l’utilisation d’un logiciel open-source programmé en Python comprenant un flux de travail précis pour l’analyse des données de nanoindentation sont fournies.
Étapes critiques du protocole
Les ét...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le CG et le MAGO reconnaissent tous les membres du CeMi. MSS reconnaît son soutien par le biais d’une subvention de programme EPSRC (EP/P001114/1).
GC : logiciel (contribution au développement de logiciels et d’algorithmes), analyse formelle (analyse des données de nanoindentation), validation, investigation (expériences de nanoindentation sur gels de polyacrylamide), curation de données, rédaction (ébauche originale, révision et édition), visualisation (figures et graphiques). MAGO: investigation (préparation d’échantillons de gels et de cellules, expériences de nanoindentation sur cellules), rédaction (ébauche originale, révision et édition), visualisation (figures et graphiques). NA : validation, rédaction (révision et édition). IL: logiciel (contribution au développement de logiciels et d’algorithmes), validation, rédaction (révision et édition); MV : conceptualisation, logiciel (conception et développement de logiciels et d’algorithmes originaux), validation, ressources, rédaction (ébauche originale, révision et révision), supervision, administration de projet, acquisition de financement MSS: ressources, rédaction (révision et révision), supervision, administration de projet, acquisition de financement. Tous les auteurs ont lu et approuvé le manuscrit final.
matériels
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
Références
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.