Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Spectroscopie de force d’ensemble par forces de cisaillement
Dans cet article
Résumé
La spectroscopie de force d’ensemble (EFS) est une technique robuste pour le déploiement mécanique et la détection en temps réel d’un ensemble de structures biomoléculaires dans les domaines biophysiques et biosensoriels.
Résumé
Les techniques monomoléculaires basées sur la fluorescence et les principes mécanochimiques offrent une sensibilité supérieure dans la détection biologique. Cependant, en raison du manque de capacités de haut débit, l’application de ces techniques est limitée en biophysique. La spectroscopie de force d’ensemble (EFS) a démontré un débit élevé dans l’étude d’un ensemble massif de structures moléculaires en convertissant les études mécanochimiques de molécules individuelles en celles d’ensembles moléculaires. Dans ce protocole, les structures secondaires de l’ADN (i-motifs) ont été déployées dans le flux de cisaillement entre le rotor et le stator d’une pointe d’homogénéisateur à des vitesses de cisaillement allant jusqu’à 77796/s. Les effets des débits et des tailles moléculaires sur les forces de cisaillement subies par le motif i ont été démontrés. La technique EFS a également révélé l’affinité de liaison entre les motifs de l’ADN i et les ligands. De plus, nous avons démontré une réaction de chimie de clic qui peut être actionnée par la force de cisaillement (c’est-à-dire la chimie mécano-clic). Ces résultats établissent l’efficacité de l’utilisation de la force de cisaillement pour contrôler la conformation des structures moléculaires.
Introduction
En spectroscopie de forcemonomoléculaire 1 (SMFS), les propriétés mécaniques de structures moléculaires individuelles ont été étudiées par des instruments sophistiqués tels que le microscope à force atomique, les pinces optiques et les pinces magnétiques 2,3,4. Limité par la même exigence de directionnalité des molécules dans les configurations de génération de force / détection ou le petit champ de vision dans les pinces magnétiques et le microscope de force de centrifugeuse miniature (MCF)5,6,7,8, seul un nombre limité de molécules peut être étudié simultanément en utilisant SMFS. Le faible débit de SMFS empêche sa large application dans le domaine de la reconnaissance moléculaire, qui nécessite l’implication d’un grand ensemble de molécules.
L’écoulement de cisaillement fournit une solution potentielle pour appliquer des forces à un ensemble massif de molécules9. Dans un écoulement de liquide à l’intérieur d’un canal, plus le débit est proche de la surface du canal, plus le débit10 est lent. Un tel gradient de vitesse d’écoulement provoque une contrainte de cisaillement parallèle à la surface limite. Lorsqu’une molécule est placée dans cet écoulement de cisaillement, la molécule se réoriente de sorte que son axe long s’aligne avec la direction de l’écoulement, car la force de cisaillement est appliquée à l’axe long11. Du fait de cette réorientation, toutes les molécules du même type (taille et longueur des poignées) devraient s’aligner dans la même direction tout en subissant la même force de cisaillement.
Ce travail décrit un protocole permettant d’utiliser un tel flux de cisaillement pour exercer une force de cisaillement sur un ensemble massif de structures moléculaires, comme en témoigne le motif i de l’ADN. Dans ce protocole, un flux de cisaillement est généré entre le rotor et le stator dans une pointe d’homogénéisateur. La présente étude a révélé que la structure du motif i de l’ADN plié pouvait être dépliée par des taux de cisaillement de 9724-97245 s−1. En outre, une constante de dissociation de 36 μM a été trouvée entre le ligand L2H2-4OTD et le i-motif. Cette valeur est cohérente avec celle de 31 μM mesurée par le test de décalage de gel12. De plus, la technique actuelle est utilisée pour déplier le i-motif, qui peut exposer le cuivre chélaté (I) pour catalyser une réaction de clic. Ce protocole permet ainsi de déplier un grand ensemble de structures i-motif avec des instruments à faible coût dans un temps raisonnable (moins de 30 min). Étant donné que la technique de la force de cisaillement augmente considérablement le débit de la spectroscopie de force, nous appelons cette technique spectroscopie de force d’ensemble (EFS). Ce protocole vise à fournir des lignes directrices expérimentales pour faciliter l’application de cet EFS basé sur la force de cisaillement.
Protocole
REMARQUE : Tous les tampons et les réactifs chimiques utilisés dans ce protocole sont énumérés dans le tableau des matériaux.
1. Préparation du microscope à force de cisaillement
REMARQUE: Le microscope à force de cisaillement contient deux parties, une unité de réaction (homogénéisateur) et une unité de détection (microscope à fluorescence). Le grossissement de l’oculaire est de 10x et le grossissement de la lentille de l’objectif (air) est de 4x.
- Assemblez l’homogénéisateur et le microscope sur une table de montage. Portez des lunettes et allumez le microscope à fluorescence, puis ajustez l’homogénéisateur pour vous assurer que le faisceau lumineux d’excitation d’une longueur d’onde appropriée (ici, 488 nm a été utilisé) passe par le centre de l’extrémité dispersante de l’homogénéisateur.
- Préparer une chambre de réaction à fond plat de 5 cm de hauteur et de 1,5 cm de section transversale de 1,5 cm2 x 1,5cm2 . Pour réduire le bruit de fond, assurez-vous que le matériau de chambre sélectionné ne devient pas fluorescent, comme les verres (certains plastiques ont une fluorescence).
- Choisissez une pointe de dispersion homogénéiseur appropriée qui peut fournir la force de cisaillement souhaitée.
NOTE: La vitesse de cisaillement dépend de la distance entre le rotor et le stator à une vitesse de rotation fixe11 selon l’équation suivante: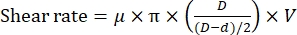
où μ est la viscosité dynamique de l’eau à 20 °C; D est le diamètre intérieur du stator; d est le diamètre extérieur du rotor et V est la vitesse de cisaillement (tr/min/s). - Serrez la chambre de réaction sur l’étage d’échantillonnage du microscope à fluorescence, puis ajustez la chambre pour maintenir l’extrémité dispersante de l’homogénéisateur (Figure 1). Assurez-vous que l’embout de dispersion est légèrement au-dessus (~1 mm) de la surface inférieure de la chambre de réaction.
- Ajustez la position verticale de l’homogénéisateur et de la chambre ensemble pour vous assurer que le microscope est focalisé sur la surface de la pointe de dispersion. Ajustez ensuite la position horizontale de l’embout dispersant pour vous assurer que la zone de détection (champ de vision) est définie entre le rotor et le stator (Figure 1).
- Allumez les canaux de fluorescence en fonction du colorant fluorescent utilisé dans l’expérience.
- Avant l’expérience de cisaillement à grande vitesse, utilisez de l’eau désionisée (DI) pour tester le cisaillement à faible vitesse de cisaillement (p. ex. 2 000 tr/min) afin de vous assurer que l’extrémité de dispersion peut fonctionner correctement sans toucher la chambre de réaction.
2. Déplier des i-motifs avec et sans ligands
- Préparer un ADN télomérique humain i-motif (Table of Materials) marqué avec un colorant et un quencher à ses deux extrémités, respectivement, dans de l’eau DI, comme décrit dans Hu et al.11.
NOTE: i-motif contenant la séquence: 5'-TAA CCC TAA CCC TAA CCC TAA CCC TAA. - Diluer l’ADN à 5 μM dans le tampon MES de 30 mM à pH 5,5 ou pH 7,4. À la solution d’ADN, ajouter le ligand L2H2-4OTD, qui a été synthétisé selon Abraham Punnoose et al.13, dans une plage de concentration de 0-60 μM. Mélanger doucement la solution pendant 10 min pour plier les structures i-motif sans lumière.
- Vérifiez et minimisez l’intensité de fluorescence de fond de la chambre de réaction remplie d’eau DI désionisée à l’aide du microscope fluorescent sans cisaillement. Un moyen facile de minimiser la fluorescence de fond est de laver la chambre de réaction avec de l’eau DI. La valeur de fluorescence de fond doit être soustraite dans les analyses de données ultérieures.
REMARQUE: La lumière parasite doit être évitée à partir de cette étape. - Réglez les paramètres de la caméra CCD à l’aide du logiciel. Les paramètres recommandés sont les suivants : temps d’exposition = 0,5 s, sensibilité CCD = 1600 et temps d’enregistrement = 20 min.
- À l’aide d’une longue pipette, ajouter la solution d’ADN dans la chambre de réaction vide et propre. Couvrir la chambre de réaction avec une boîte noire. Ensuite, démarrez l’homogénéisateur pour effectuer un cisaillement à une fréquence de cisaillement sélectionnée allant de 9 724 s−1 à 97 245 s−1 (sélectionnée à l’aide du logiciel associé à l’homogénéisateur) pendant 20 minutes avec la caméra CCD allumée pour enregistrer les données.
- Après l’expérience, retirez la chambre et lavez-la avec de l’eau DI.
3. Réaction de clic actionnée par la force de cisaillement
- Préparer l’ADN i-motif dans de l’eau DI. Incuber 10 μM d’ADN i-motif dans 300 μL de tampon Tris 30 mM (pH 7,4) complété par 150 μM CuCl et 300 μM d’acide ascorbique pendant 10 min pour plier les structures i-motif (toutes les concentrations sont des concentrations finales dans la solution).
REMARQUE: CuCl est le catalyseur de la réaction de clic. L’acide ascorbique empêchera l’oxydation du cuivre (I). - Ultrafiltrez la solution avec un dispositif d’ultrafiltration à une force centrifuge de 14 300 x g. Reconstituer la solution à ~500 μL avec un tampon Tris 30 mM (pH 7,4) complété par de l’acide ascorbique 300 μM après chaque filtration.
- Répétez la filtration 3x.
- Recueillir la solution résiduelle et obtenir un volume final de 300 μL en ajoutant 30 mM de Tris (pH 7,4) complété par 300 μM d’acide ascorbique ainsi que 20 μM d’azoture de Calfluor 488, 20 μM de HPG et 10 μM de TBTA. Une fois les réactifs ajoutés, déplacez la solution dans la chambre noire.
REMARQUE: La lumière doit être évitée après cette étape. - Vérifiez et minimisez l’intensité de fluorescence de fond de la chambre de réaction remplie d’eau DI à l’aide du microscope avant les expériences de cisaillement. Un moyen facile de minimiser la fluorescence de fond est de laver la chambre de réaction avec de l’eau DI.
- Ajouter la solution d’ADN dans la chambre de réaction vide avec une longue pipette, puis démarrer le cisaillement de l’homogénéisateur à une vitesse de cisaillement de 63 209 s−1 pendant 20 min avec la caméra CCD allumée.
- Après l’expérience, retirez la chambre et lavez-la avec de l’eau DI.
Résultats
La figure 1 décrit le déploiement mécanique et la détection en temps réel des molécules d’ensemble dans EFS. Dans la figure 1B, on a observé que l’intensité de fluorescence de l’ADN i-motif augmentait avec le taux de cisaillement allant de 9 724 s−1 à 97 245 s−1 dans un tampon de pH 5,5 MES. En tant que témoin, l’intensité de fluorescence n’a pas augmenté lorsque le même ADN i-motif a été cisai...
Discussion
Le protocole décrit dans ce manuscrit permet d’étudier en temps réel le déroulement d’un ensemble de structures biomoléculaires par force de cisaillement. Les résultats présentés ici soulignent que les structures à motif d’ADN peuvent être dépliées par la force de cisaillement. Le déploiement du motif i lié au ligand et les réactions de clic actionnées par la force de cisaillement étaient des applications de preuve de concept pour cette méthode de spectroscopie de force d’ensemble.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts.
Remerciements
Ce travail de recherche a été soutenu par la National Science Foundation [CBET-1904921] et les National Institutes of Health [NIH R01CA236350] à H. M.
matériels
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Références
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon