Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Bioréacteur de perfusion multi-flux intégré au fractionnement de sortie pour la culture cellulaire dynamique
Dans cet article
Résumé
Cet article présente une méthode pour construire et exploiter un système de culture cellulaire de perfusion multicanal à faible coût pour mesurer la dynamique des taux de sécrétion et d’absorption des solutés dans les processus cellulaires. Le système peut également exposer les cellules à des profils de stimulus dynamiques.
Résumé
Certaines fonctions cellulaires et tissulaires fonctionnent dans un délai dynamique de quelques minutes à quelques heures qui sont mal résolues par les systèmes de culture conventionnels. Ce travail a permis de développer un système de bioréacteur de perfusion à faible coût qui permet de perfuser continuellement le milieu de culture dans un module de culture cellulaire et de fractionner dans un module en aval pour mesurer la dynamique à cette échelle. Le système est construit presque entièrement à partir de pièces disponibles dans le commerce et peut être parallélisé pour mener simultanément des expériences indépendantes dans des plaques de culture cellulaire conventionnelles multi-puits. Cet article vidéo montre comment assembler la configuration de base, qui ne nécessite qu’une seule pompe à seringue multicanal et un collecteur de fraction modifié pour perfuser jusqu’à six cultures en parallèle. Des variantes utiles de la conception modulaire sont également présentées qui permettent une dynamique de stimulation contrôlée, telle que des impulsions de soluté ou des profils de type pharmacocinétique. Il est important de noter que lorsque les signaux de soluté traversent le système, ils sont déformés en raison de la dispersion des solutés. En outre, une méthode de mesure des distributions de temps de séjour (RTD) des composants de la configuration de perfusion avec un traceur utilisant MATLAB est décrite. Les RTD sont utiles pour calculer comment les signaux de soluté sont déformés par le flux dans le système à compartiments multiples. Ce système est très robuste et reproductible, de sorte que les chercheurs de base peuvent facilement l’adopter sans avoir besoin d’installations de fabrication spécialisées.
Introduction
De nombreux processus biologiques importants se produisent dans les cultures cellulaires et tissulaires sur une échelle de minutes àquelques heures 1,2,3. Bien que certains de ces phénomènes puissent être observés et enregistrés de manière automatisée à l’aide de la microscopie time-lapse4, de la bioluminescence1 ou d’autres méthodes, les expériences impliquant la collecte d’échantillons de surnageants de culture pour analyse chimique sont souvent effectuées manuellement dans des cultures cellulaires statiques. L’échantillonnage manuel limite la faisabilité de certaines études en raison des inconvénients des points de temps d’échantillonnage fréquents ou après les heures d’ouverture. D’autres lacunes des méthodes de culture statique comprennent des expériences impliquant des expositions contrôlées et transitoires à des stimuli chimiques. Dans les cultures statiques, les stimuli doivent être ajoutés et supprimés manuellement, et les profils de stimulus sont limités aux changements d’étape au fil du temps, tandis que les changements de milieu ajoutent et suppriment également d’autres composants du milieu, ce qui peut affecter les cellules de manière incontrôlée5. Les systèmes fluidiques peuvent surmonter ces défis, mais les dispositifs existants posent d’autres défis. Les dispositifs microfluidiques s’accompagnent des coûts prohibitifs de l’équipement spécialisé et de la formation à la production et à l’utilisation, nécessitent des méthodes microanalytiques pour traiter les échantillons et les cellules sont difficiles à récupérer des dispositifs après perfusion6. Peu de systèmes macrofluidiques ont été créés pour les types d’expériences décrits ici 7,8,9,10, et ils sont construits à partir de plusieurs pièces personnalisées fabriquées en interne et nécessitent plusieurs pompes ou collecteurs de fractions. En outre, les auteurs n’ont connaissance d’aucun système de culture cellulaire de perfusion macrofluidique disponible dans le commerce autre que les bioréacteurs à réservoir agité pour la culture en suspension, qui sont utiles pour la biofabrication, bien qu’ils ne soient pas conçus pour la modélisation et l’étude de la physiologie.
Les auteurs ont déjà fait état de la conception d’un système de bioréacteur de perfusion à faible coût composé presque entièrement de pièces11 disponibles dans le commerce. La version de base du système permet de conserver plusieurs cultures dans une plaque de puits dans un incubateur à CO2 et de les perfuser en continu avec du milieu provenant d’une pompe à seringue, tandis que les flux de milieu d’effluent des cultures sont automatiquement fractionnés en échantillons au fil du temps à l’aide d’un collecteur de fractions avec une modification personnalisée. Ainsi, ce système permet l’échantillonnage automatisé du milieu de culture surnageant et l’entrée continue de soluté dans les cultures au fil du temps. Le système est macrofluidique et modulaire et peut être facilement modifié pour répondre aux besoins de nouvelles conceptions d’expériences.
L’objectif global de la méthode présentée ici est de construire, de caractériser et d’utiliser un système de culture cellulaire par perfusion qui permet des expériences dans lesquelles les taux de sécrétion ou d’absorption de substances par les cellules au fil du temps sont mesurés et / ou les cellules sont exposées à des signaux de soluté précis et transitoires. Cet article vidéo explique comment assembler la configuration de base, qui est capable de perfuser jusqu’à six cultures cellulaires simultanément à l’aide d’une seule pompe à seringue et d’un collecteur de fraction modifié. Deux variantes utiles du système de base qui utilisent des pompes et des pièces supplémentaires pour permettre des expériences qui exposent les cellules à des signaux de concentration transitoires de soluté, y compris de brèves impulsions et des profils pharmacocinétiques12, sont également présentées, illustrées à la figure 1.
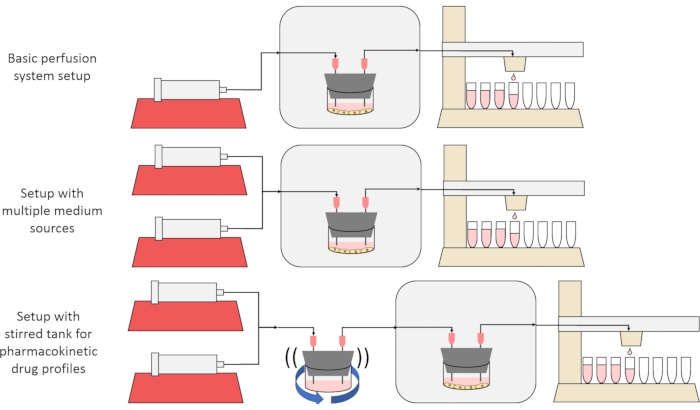
Figure 1: Trois variantes de la conception du système de perfusion. (En haut) Le système de perfusion de base. (Au milieu) Le système de perfusion avec un robinet d’arrêt pour plusieurs sources de milieu. (En bas) Le système de perfusion avec un réservoir agité pour imiter un volume de distribution bien mélangé. Veuillez cliquer ici pour voir une version agrandie de cette figure.
En raison de la dispersion et de la diffusion dans le flux, les signaux de soluté se déforment ou « s’enduire » lorsqu’ils se déplacent dans le système d’écoulement. Cette distorsion peut être quantifiée par l’utilisation de distributions de temps de séjour (RDT)13. Cet article explique comment effectuer des expériences de traçage sur des composants du système de perfusion (Figure 2) et fournit des scripts MATLAB pour générer des RTD à partir de données mesurées. Une explication détaillée de cette analyse peut être trouvée dans l’article précédent des auteurs11. Des scripts MATLAB supplémentaires adaptent les fonctions appropriées aux RTD et extraient les paramètres physiques, et effectuent la convolution du signal à l’aide de RTD pour prédire comment l’entrée du signal de soluté par l’utilisateur se propagera et se déformera à travers le système de perfusion14.
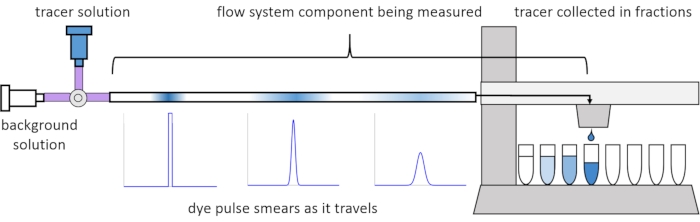
Figure 2 : Distributions du temps de résidence. Les RTD des composants du système d’écoulement, tels que cette longueur de tuyau, sont mesurés en entrant une impulsion de traceur dans le système et en mesurant comment il « frottis » au moment où il sort dans les fractions collectées. Ce chiffre a été modifié à partir d’Erickson et al.11. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparer les pièces pour la perfusion de la plaque de puits
- Préparer les tubes
- Couper deux longueurs de tube en silicone (1,6 mm de diamètre intérieur) pour chaque culture cellulaire à perfuser. Assurez-vous que la pièce utilisée comme tube en amont est suffisamment longue pour atteindre la pompe à seringue à la culture cellulaire à l’intérieur de l’incubateur, et que la pièce en aval peut atteindre de la culture cellulaire à la position la plus étendue du collecteur de fractions.
- Donnez à chaque morceau de tube une étiquette unique à ses deux extrémités avec du ruban adhésif étiqueté.
- Préparer les bouchons pour la plaque de puits
- Obtenez un bouchon en silicone pour chaque puits de la plaque de puits à perfuser, avec un diamètre approprié pour s’adapter confortablement dans les puits avec un joint étanche à l’air.
- Coupez l’excès de matière du fond du bouchon afin qu’il s’insère dans les puits tout en laissant de l’espace à l’intérieur pour l’air au-dessus du niveau de liquide prévu.
- Poussez deux aiguilles émoussées de 18 G à travers chaque bouchon, dans le haut et hors du bas pour servir d’entrée et de sortie pour le flux à travers le puits, diamétralement opposées l’une à l’autre pour maximiser la distance entre leurs extrémités à l’intérieur du puits.
- Ajustez la hauteur des aiguilles dans le puits bouché, car la hauteur de l’aiguille de sortie déterminera la hauteur stable du niveau de liquide dans le puits pendant la perfusion.
REMARQUE: Si la perfusion est commencée avec l’aiguille de sortie au-dessus du niveau de liquide, le liquide s’accumulera dans le puits jusqu’à ce que le niveau atteigne l’aiguille. Si la perfusion est démarrée avec l’aiguille de sortie en dessous du niveau de liquide, le niveau de liquide restera stable à moins que des bulles d’air ne s’écoulent dans le puits, ce qui entraînera une baisse de la hauteur du liquide jusqu’à ce qu’elle soit à la même hauteur que l’aiguille de sortie.
- Rassembler des pièces supplémentaires
- Obtenez une seringue stérile pour chaque culture cellulaire à perfuser qui est suffisamment grande pour contenir suffisamment de milieu pour toute la perfusion, plus une quantité supplémentaire de milieu pour remplir initialement le tube.
- Pour chaque culture à perfuser, obtenez un connecteur Luer femelle-barbe et deux connecteurs Luer mâle-barbe, ainsi que deux bonnets Luer femelles et deux mâles.
- Nettoyer et stériliser les pièces
- Si des pièces ont déjà été utilisées, nettoyez-les en les parfumant avec 0,1 N NaOH, suivi d’un rinçage à l’eau désionisée.
- Par autoclavage ou autre, assurez-vous de la stérilité de toutes les pièces énumérées ci-dessus.
2. Découpez au laser le distributeur multi-têtes et fixez-le à un collecteur de fractions
- Recréez le modèle de distributeur multi-têtes de la Figure 3 dans un programme de conception assistée par ordinateur ou téléchargez le fichier DXF du modèle fourni (fichier supplémentaire 1).
- Utilisez une découpeuse laser pour découper le dessin à partir d’une feuille acrylique 1/8.
- Retirez les trois vis qui fixent la tête de distribution à la base mobile du collecteur de fractions.
- Alignez les trois plus petits trous du distributeur à plusieurs têtes avec les trous de vis dans la base mobile et revissez les vis à travers les trous à fixer.
- Ajustez l’étanchéité des trois vis pour incliner le distributeur multi-têtes de haut en bas jusqu’à ce que la rangée de trous de distribution soit alignée avec les tubes de collecte en dessous.
- Placez soigneusement les embouts de pipette de 300 μL à travers les trous souhaités pour servir d’embouts de distribution.
REMARQUE: Le collecteur de fraction peut être utilisé sans le distributeur à plusieurs têtes pour perfuser une seule culture cellulaire.
3. Mesurer les RTD des composants et effectuer la convolution du signal
- Configurez des pompes et des seringues pour l’impulsion de traçage comme illustré à la figure 2.
- Procurez-vous deux pompes à seringues monocanaux ou multicanaux.
- Choisissez une solution d’arrière-plan pour représenter le milieu qui sera utilisé dans le système d’écoulement pendant les expériences de culture cellulaire. Assurez-vous que la solution d’arrière-plan possède des propriétés de transfert de masse similaires à celles du support. Dans de nombreux cas, l’eau désionisée est un choix approprié.
- Choisissez une substance traceuse pour représenter le soluté qui sera d’intérêt lors des expériences de culture cellulaire. S’assurer que le traceur a des propriétés de transfert de masse similaires à celles du soluté d’intérêt et que sa concentration doit pouvoir être mesurée. Dans de nombreux cas, le colorant alimentaire est un choix approprié.
- Dissoudre la substance traceuse dans la solution de fond pour obtenir la solution traceuse.
- Remplissez une seringue avec une solution de fond et chargez-la dans une pompe à seringue. Remplissez une autre seringue avec une solution traceuse et chargez-la dans la deuxième pompe à seringue.
- Connectez les deux seringues à deux des trois ports d’un robinet d’arrêt à quatre voies à l’aide de connecteurs Luer.
- Fermez le robinet d’arrêt à la solution d’arrière-plan et pompez la solution de traceur dans le robinet d’arrêt jusqu’à ce qu’il commence à s’égoutter sur le port ouvert. Arrêtez la pompe et n’ajustez pas la seringue davantage.
REMARQUE: Il est important que la barre mobile de la pompe à seringue soit poussée contre les pistons de la seringue avant le début des parties chronométrées de l’expérience. Cela permettra au débit de commencer immédiatement au démarrage de la pompe. Sinon, la pompe peut être démarrée, mais le débit ne commencera pas réellement tant que la barre mobile n’aura pas rattrapé la position du piston. - Fermez le robinet d’arrêt à la solution de traceur et pompez la solution d’arrière-plan dans le robinet d’arrêt jusqu’à ce que toute la solution résiduelle de traceur ait été évacuée du port ouvert. Arrêtez la pompe et n’ajustez pas la seringue davantage.
- Configurer le composant d’intérêt du système d’écoulement et le collecteur de fractions
- Configurez le composant du système de flux souhaité pour l’analyse RTD. Assurez-vous que le composant à mesurer se termine par un morceau de tuyau en aval de longueur et de flexibilité appropriées pour atteindre le collecteur de fraction pendant le fonctionnement.
- Insérez l’extrémité du tuyau en aval dans un distributeur de pointe de pipette dans le distributeur à plusieurs têtes de manière à ce qu’il soit bien connecté.
- Fixez l’orifice ouvert du robinet d’arrêt à quatre voies à l’entrée du composant à mesurer. Pomper la solution de fond à travers le composant jusqu’à ce qu’il soit entièrement rempli comme il le serait lors d’une expérience de culture cellulaire, et il commence à s’égoutter hors de la pointe de distribution du collecteur de fractions. Arrêtez la pompe.
- Injecter l’impulsion du traceur, collecter les fractions et mesurer le traceur
- Réglez la pompe de la solution de traçage sur le débit souhaité. Fermez le robinet d’arrêt sur la solution d’arrière-plan et démarrez le flux de la solution de traçage. Dans le même temps, démarrez le collecteur de fractions.
- Continuez l’écoulement de la solution traceuse pendant une courte période de temps pour approximer une entrée d’impulsion du traceur. Une durée d’impulsion de 10 minutes s’est avérée efficace pour les RTD à un débit de 1 mL/h.
REMARQUE: Si l’impulsion du traceur est trop brève, le traceur n’entrera pas suffisamment dans le flux pour être mesurable. Si l’impulsion est trop longue, elle ne se rapprochera plus d’une impulsion et changera la forme du RTD. - À la fin de la période d’impulsion de la solution traceuse, arrêtez la pompe de solution traceuse. Fermez rapidement le robinet d’arrêt à la solution de traçage et démarrez le flux de la solution d’arrière-plan au même débit.
- Laissez la solution d’arrière-plan s’écouler et les fractions être collectées jusqu’à ce que tout le traceur ait traversé le système et dans les fractions collectées.
- Arrêtez le système et mesurez la concentration du traceur dans les fractions. N’incluez que les fractions qui ont été complètement distribuées. Si la collection est arrêtée à mi-chemin de la collection d’une fraction, n’incluez pas cette fraction.
- Calculer la distribution du temps de séjour (RTD) à partir des données mesurées dans MATLAB
REMARQUE: Une explication écrite de l’analyse effectuée par ce script MATLAB peut être trouvée dans la publication précédente des auteurs11, et les discussions sur la théorie sont largement disponibles dans la littérature13.- Produire un fichier .xlsx contenant les données de concentration dans le format de la feuille de calcul example_tracer_data.xlsx fournie dans le fichier supplémentaire 2. Entrez les valeurs de concentration du traceur dans les fractions (toutes les unités) dans l’ordre chronologique de gauche à droite à la ligne 2. Entrez le temps écoulé entre le début de l’impulsion et la fin de la dernière fraction dans la cellule A5 et entrez la longueur de l’impulsion traceur en minutes dans la cellule A8.
- Enregistrez le fichier .xlsx dans le répertoire MATLAB.
- Ouvrez le script RTD_From_Data.m, à partir du fichier supplémentaire 3, dans l’éditeur MATLAB.
- Remplacez le nom du fichier .xlsx entre parenthèses dans la première ligne de la section Charger les données du script par le nom du nouveau fichier de données .xlsx, en suivant les instructions écrites dans le fichier de script. Exécutez le script.
- Assurez-vous que le script effectue avec succès l’analyse RTD13, en produisant un tracé de la RTD et en renvoyant la valeur de l’intégrale numérique sur la RTD égale à 1. Recherchez le vecteur temps (t) et le vecteur de valeurs RTD (Et) associés enregistrés par le script dans le répertoire MATLAB.
- Adapter une fonction de modèle au RTD dans MATLAB
- Ouvrez le script Fit_RTD_Function.m dans l’éditeur MATLAB à partir du fichier supplémentaire 4.
- Choisissez l’une des trois fonctions du modèle commenté pour s’adapter au RTD : le modèle de dispersion axiale13, qui s’adapte aux RTD pour l’écoulement laminaire dans les tubes cylindriques ; le modèle CSTR13, qui s’adapte aux réservoirs bien agités; et le modèle n-CSTR15, qui s’adapte approximativement à des plaques de puits plus grandes. Pour adapter un autre modèle non inclus ici, ajoutez-le au script dans le même format.
- Supprimez les commentaires dans la section du script contenant le modèle choisi pour le raccord.
- Remplacez les valeurs des estimations initiales des paramètres par celles appropriées pour le RDT.
- Exécutez le script pour produire un tracé de la fonction d’ajustement superposée aux données RTD et pour imprimer les valeurs des paramètres d’ajustement de la fonction. Si l’ajustement est très mauvais ou si des erreurs se produisent, modifiez les suppositions initiales du paramètre et exécutez à nouveau le script.
- Effectuer la convolution du signal dans MATLAB
- Choisissez un signal et un RTD ou deux RTD à convolve.
- Ouvrez le script Signal_Convolution.m, à partir du fichier supplémentaire 5, dans l’éditeur MATLAB.
- Pour chacun des deux signaux à convoluer (c’est-à-dire un signal et un RTD, ou deux RTD), définissez un vecteur de points temporels uniformément espacés dans les unités souhaitées et un vecteur correspondant de valeurs de signal à ces moments.
REMARQUE: Les vecteurs des deux signaux doivent avoir le même nombre d’éléments et les mêmes pas de temps de taille. C’est pourquoi il est utile d’avoir le RTD comme une fonction continue qui peut être échantillonnée pour un nombre arbitraire de points à n’importe quel intervalle de temps. - Entrez les deux signaux dans MATLAB et exécutez le script pour obtenir les vecteurs de temps et de signal du signal de sortie.
4. Mettre en place le système de perfusion de base avec des cellules dans une plaque de puits
- Préparer la plaque de puits
- Assurez-vous que la culture sur plaque de puits a la profondeur moyenne appropriée pour l’expérience de perfusion. Effectuez tout changement de milieu final, stimulation ou autre étape souhaitée avant de commencer la perfusion. Si des cellules de suspension sont perfusées, centrifugez la plaque pour vous assurer qu’elles sont au fond.
- Dans des conditions stériles, insérez les bouchons avec des aiguilles dans les cultures de plaques de puits avec les aiguilles tirées vers le haut. Une fois le bouchon en place, abaissez les aiguilles à la hauteur souhaitée pour la perfusion, car la hauteur de l’aiguille de sortie détermine le niveau de liquide stable.
- Boucher les aiguilles avec des bouchons Luer mâles et garder toute la plaque de puits dans un incubateur jusqu’à utilisation.
- Préparer les seringues et les tubes en amont
- Dans des conditions stériles, remplissez une seringue pour chaque culture à perfuser avec suffisamment de milieu pour la durée souhaitée de la perfusion, plus suffisamment de milieu supplémentaire pour remplir le tube en amont.
- Fixez le tube en amont à la seringue à l’aide d’un connecteur Luer femelle-barbe. À l’autre extrémité du tube, insérez un connecteur Luer mâle-barbe.
- Distribuer le milieu de la seringue jusqu’à ce que le tube en amont soit entièrement rempli de milieu.
- Coiffez l’extrémité ouverte du tube avec un capuchon Luer femelle.
REMARQUE: Toutes les seringues répliquées doivent avoir exactement le même volume à ce stade. Si leurs volumes ne sont pas égaux, leurs pistons seront dans des positions différentes et ils ne s’intégreront pas tous bien dans une seule pompe à seringue multicanal.
- Dans des conditions stériles, insérez un connecteur Luer mâle-barbe dans une extrémité du tube en aval et recouvrez-le d’un capuchon Luer femelle.
- Apportez soigneusement tous les tubes préparés, les seringues et la plaque de puits à l’incubateur qui sera utilisé pour la perfusion.
- Placez la pompe à seringue et le collecteur de fractions aux endroits souhaités près de l’incubateur. Placez la pompe à seringue sur ou près de l’incubateur et placez le collecteur de fraction à côté de l’incubateur, près du port.
- Regroupez les extrémités coiffées de tous les tubes en amont et en aval et poussez-les de l’extérieur de l’incubateur vers l’intérieur à travers le port.
- Chargez les seringues dans la pompe à seringue et insérez les extrémités ouvertes des tubes en aval dans les embouts de la pipette de distribution du distributeur à plusieurs têtes du collecteur de fractions.
- À l’intérieur de l’incubateur, tirez autant de mou que possible des tubes en amont dans l’incubateur pour maximiser la longueur des tubes à travers lesquels le fluide qui circule peut recevoir de la chaleur et du CO2 de l’air de l’incubateur. Tout en les maintenant en place, retirez les tubes en aval de l’incubateur, juste assez pour qu’ils puissent atteindre le point le plus étendu du collecteur de fractions tout en gardant les extrémités coiffées à l’intérieur de l’incubateur.
- Pour chaque puits bouché, décapsulez rapidement les aiguilles et les tubes en amont et en aval de ce puits et fixez-les ensemble avec leurs connecteurs Luer.
- Une fois que toutes les pièces sont connectées, faites fonctionner brièvement la pompe à seringue à une vitesse relativement élevée pour vous assurer que tous les flux s’écoulent correctement.
- À ce stade, si vous souhaitez commencer l’expérience avec les tubes en aval remplis du milieu, continuez à faire fonctionner la pompe jusqu’à ce que tous soient remplis. Sinon, arrêtez la pompe.
- Réglez le débit de la pompe à seringue et la fréquence de collecte des fractions et démarrez les deux machines simultanément pour commencer l’expérience. Collectez des fractions pour la durée d’expérience souhaitée.
5. Configurez le système de perfusion avec un robinet d’arrêt pour plusieurs sources de milieu
- Effectuez toutes les sous-étapes de l’étape 4.1 ci-dessus.
- Préparez les deux milieux à utiliser dans la perfusion, en étiquetant le milieu qui sera distribué en premier comme 1 et l’autre comme milieu 2.
- Pour chaque culture à perfuser, remplissez une seringue avec suffisamment de milieu 1 pour la durée de sa dispensation, plus suffisamment de volume pour remplir initialement le système de perfusion. Remplissez une deuxième seringue avec suffisamment de milieu 2 pour la durée de sa dispensation.
- Connectez les deux seringues à deux des trois orifices d’un robinet d’arrêt à quatre voies.
REMARQUE: Une longueur de tuyau pour connecter les seringues aux robinets d’arrêt peut être nécessaire. - Préparez le robinet d’arrêt et les seringues de la même manière que les étapes 3.1.7 à 3.1.8 ci-dessus en fermant le robinet d’arrêt au milieu 1 et en distribuant le milieu 2 dans le robinet d’arrêt jusqu’à ce qu’il commence à s’égoutter sur le port ouvert.
- Fermez le robinet d’arrêt au milieu 2 et distribuez le milieu 1 dans le robinet d’arrêt jusqu’à ce que tout le milieu résiduel 2 ait été évacué du port ouvert.
- Fixez le tube en amont à l’orifice ouvert du robinet d’arrêt à l’aide d’un connecteur Luer femelle-barbe. À l’autre extrémité du tube, insérez un connecteur Luer mâle-barbe.
- Distribuer le milieu 1 de la seringue jusqu’à ce que le tube en amont soit entièrement rempli de milieu.
- Passez aux étapes 4.3 à 4.11 ci-dessus, en chargeant les deux seringues dans des pompes à seringues séparées et en ne distribuant que le milieu 1.
- Réglez le débit de la pompe à seringue pour le milieu 1 et la fréquence de collecte des fractions, puis démarrez les deux machines simultanément pour commencer l’expérience.
- Lorsque la source de milieu doit être changée, arrêtez rapidement la pompe à seringue pour le milieu 1, fermez le robinet d’arrêt au milieu 1 et démarrez la pompe à seringue pour le milieu 2. Si vous le souhaitez plus tard, remettez la source au milieu 1 de la même manière.
- Collectez des fractions pour la durée d’expérience souhaitée.
6. Configurez le système de perfusion avec un réservoir agité pour imiter la pharmacocinétique
- Obtenir des données pharmacocinétiques pour le médicament d’intérêt et s’assurer qu’il se compose d’un pic de concentration suivi d’une désintégration exponentielle.
- Après avoir réglé le pic sur l’heure 0 et supprimé les points de données avant le pic, utilisez le script Stirred_Tank_Fit.m (fichier supplémentaire 6) pour adapter l’équation RTD du réservoir agité aux données. L’entrée v (le débit de perfusion souhaité) et les données à ajuster sous forme de paire de vecteurs directement dans le script, ainsi que t et C pour les valeurs de temps et de concentration, respectivement. Exécutez le script pour imprimer le paramètre V, qui est le volume de réservoir agité requis.
- Planifiez une disposition du système de perfusion comprenant deux pompes à seringues et un agitateur à plaques en amont de l’incubateur.
- Mesurer les RTD des composants du système de perfusion au-delà du robinet d’arrêt et effectuer la convolution du signal des RTD avec différentes durées et concentrations d’impulsions médicamenteuses pour trouver un profil pharmacocinétique approprié. Utilisez l’équation RTD du réservoir agité d’ajustement dans ce calcul.
- Passez aux étapes 5.1 à 5.8 ci-dessus.
- Utilisez une plaque de puits supplémentaire de taille appropriée comme réservoir agité pour l’installation. Chaque puits peut servir de réservoir agité pour une culture perfusée. Remplissez le puits avec le volume requis de milieu, V, et branchez le puits avec les aiguilles initialement tirées vers le haut, puis poussées au fond du puits, et boucher les aiguilles.
- Chargez les seringues dans les pompes à seringues et connectez rapidement les tubes des seringues aux aiguilles d’entrée bouchées des réservoirs agités. Ensuite, connectez les entrées du tube de culture cellulaire en amont aux aiguilles de sortie des réservoirs agités.
- Passez aux étapes 5.9 à 5.10.
- Au moment souhaité, passez au milieu de distribution 2 contenant le médicament, comme décrit à l’étape 5.11. Revenez au milieu 1 lorsque la perfusion du médicament est terminée.
- Passez à l’étape 5.12.
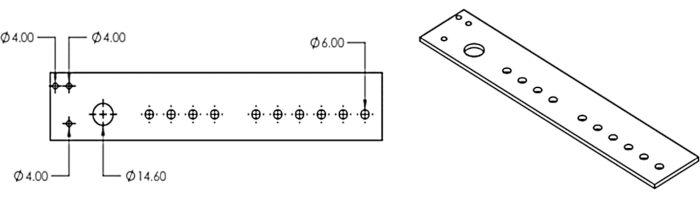
Figure 3 : Le distributeur multi-têtes. Conception pour le distributeur multi-têtes découpé au laser. Ce chiffre a été modifié à partir d’Erickson et al.11. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Le système de perfusion avec plusieurs sources de milieu de la section 5 du protocole a été utilisé pour mesurer la dynamique d’expression d’un gène rapporteur piloté par le facteur nucléaire kappa-light-chain-enhancer of activated B cells (NF-κB) transcription factor in human embryonic kidney 293 (HEK293) cells en réponse à une impulsion de 1,5 h de facteur de nécrose tumorale alpha (TNF-α). Les cellules HEK293 ont été transduites de manière stable à l’aide de vecteurs lentiviraux avec une construc...
Access restricted. Please log in or start a trial to view this content.
Discussion
Ce travail décrit l’assemblage et le fonctionnement d’un système de culture cellulaire par perfusion avec de multiples sources de milieu démontré avec un exemple spécifique dans lequel la dynamique de l’expression génique induite par NF-κB en réponse à une impulsion transitoire de TNF-α a été mesurée. Les RTD des composants du système de perfusion ont été mesurées et modélisées, et la convolution du signal a été utilisée pour prédire à la fois l’exposition des cellules à l’impulsion α ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Cette recherche a été menée avec le soutien de Grant Nos. R01EB012521, R01EB028782 et T32 GM008339 des National Institutes of Health.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 18 Gauge 1 1/2- in Disposable Probe Needle For Use With Syringes and Dispensing Machines | Grainger | 5FVK2 | |
| 293T Cells | ATCC | CRL-3216 | HEK 293T cells used in the Representative Results experiment. |
| 96-Well Clear Bottom Plates, Corning | VWR | 89091-010 | Plates for measuring dye concentrations in RTD experiments and GLuc in representative results experiment. |
| BD Disposable Syringes with Luer-Lok Tips, 5 mL | Fisher Scientific | 14-829-45 | |
| BioFrac Fraction Collector | Bio-Rad | 7410002 | Fraction collector that can be used for a single stream, or modified using our method to enable collection from multiple streams. |
| Clear High-Strength UV-Resistant Acrylic 12" x 12" x 1/8" | McMaster-Carr | 4615T93 | This sheet is cut using a laser cutter according to the DXF file in the supplemental materials to produce the multi-head dispenser that can be attached to the BioFrac fraction collector. |
| Coelenterazine native | NanoLight Technology | 303 | Substrate used in Gaussia luciferase bioluminescence assay in representative results. |
| Corning Costar TC-Treated Multiple Well Plates, size 48 wells, polystyrene plate, flat bottom wells | Millipore Sigma | CLS3548 | Used to grow and perfuse 293T cells in representative results. |
| Corning Costar Flat Bottom Cell Culture Plates, size 12 wells | Fisher Scientific | 720081 | Can be plugged and used as a stirred tank to produce pharmacokinetic profiles in perfusion. Can also contain cells for perfusion. |
| DMEM, high glucose | ThermoFisher Scientific | 11965126 | |
| Epilog Zing 24 Laser | Cutting Edge Systems | Epilog Zing 24 | Laser cutter used to produce multi-head dispenser from acrylic sheet. Other laser cutters may be used. |
| Fisherbrand Sterile Syringes for Single Use, Luer-Lock, 20 mL | Fisher Scientific | 14-955-460 | |
| Fisherbrand Sterile Syringes for Single Use, Luer-Lock, 60 mL | Fisher Scientific | 14-955-461 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-129 | Microcentrifuge tubes for collecting fractions. |
| Fisherbrand Round Bottom Disposable Borosilicate Glass Tubes with Plain End | Fisher Scientific | 14-961-26 | Glass tubes for collecting fractions. |
| Fisherbrand SureOne Micropoint Pipette Tips, Universal Fit, Non-Filtered | Fisher Scientific | 2707410 | 300 ul pipette tips that best fit the multi-head dispenser and tubing to act as dispensing tips. |
| Gibco DPBS, powder, no calcium, no magnesium | Fisher Scientific | 21600010 | Phosphate buffered saline. |
| Labline 4625 Titer Shaker | Marshall Scientific | Labline 4625 Titer Shaker | Orbital shaker used to keep stirred tanks mixed. |
| Masterflex Fitting, Polycarbonate, Four-Way Stopcock, Male Luer Lock, Non-Sterile; 10/PK | Cole-Parmer | EW-30600-04 | Used to join multiple inlet streams for RTD experiments and cell culture experiments. |
| Masterflex Fitting, Polycarbonate, Straight, Female Luer x Cap; 25/PK | Masterflex | UX-45501-28 | |
| Masterflex Fitting, Polypropylene, Straight, Female Luer to Hosebarb Adapters, 1/16" | Cole-Parmer | EW-45508-00 | |
| Masterflex Fitting, Polypropylene, Straight, Male Luer Lock to Hosebarb Adapter, 1/16" ID | Cole-Parmer | EW-45518-00 | |
| Masterflex Fitting, Polypropylene, Straight, Male Luer Lock to Plug Adapter; 25/PK | Masterflex | EW-30800-30 | |
| Masterflex L/S Precision Pump Tubing, Platinum-Cured Silicone, L/S 14; 25 ft | Masterflex | EW-96410-14 | |
| MATLAB | MathWorks | R2019b | Version R2019b. Newer versions may also be used. Some older versions may work. |
| NE-1600 Six Channel Programmable Syringe Pump | New Era Pump Systems | NE-1600 | |
| Rack Set F1 | Bio-Rad | 7410010 | Racks to hold collecting tubes in the fraction collector. |
| Recombinant Human TNF-alpha (HEK293-expressed) Protein, CF | Bio-Techne | 10291-TA-020 | Cytokine used to stimulate 293T cells in representative results. |
| Saint Gobain Solid Stoppers, Versilic Silicone, Size: 00, Bottom 10.5mm | Saint Gobain | DX263015-50 | Fits 48-well plates. |
| Saint Gobain Solid Stoppers, Versilic Silicone, Size: 4 Bottom 21mm | Saint Gobain | DX263027-10 | Fits 12-well plates. |
| Sodium Hydroxide, 10.0 N Aqueous Solution APHA; 1 L | Spectrum Chemicals | S-395-1LT | |
| SolidWorks | Dassault Systems | SolidWorks | CAD software used to create the multi-head dispenser DXF file. |
| Varioskan LUX multimode microplate reader | ThermoFisher Scientific | VL0000D0 | Plate reader. |
| Wilton Color Right Performance Color System Base Refill, Blue | Michaels | 10404779 | Blue food dye containing Brilliant Blue FCF, used as a tracer in RTD experiments. Absorbance spectrum peaks at 628 nm. |
Références
- Welsh, D. K., Yoo, S. H., Liu, A. C., Takahashi, J. S., Kay, S. A. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Current Biology. 14 (24), 2289-2295 (2004).
- Talaei, K., et al. A mathematical model of the dynamics of cytokine expression and human immune cell activation in response to the pathogen Staphylococcus aureus. Frontiers in Cellular and Infection Microbiology. 11, 711153(2021).
- Kemas, A. M., Youhanna, S., Zandi Shafagh, R., Lauschke, V. M. Insulin-dependent glucose consumption dynamics in 3D primary human liver cultures measured by a sensitive and specific glucose sensor with nanoliter input volume. FASEB Journal. 35 (3), 21305(2021).
- Muzzey, D., van Oudenaarden, A. Quantitative time-lapse fluorescence microscopy in single cells. Annual Review of Cell and Developmental Biology. 25, 301-327 (2009).
- Calligaro, H., Kinane, C., Bennis, M., Coutanson, C., Dkhissi-Benyahya, O. A standardized method to assess the endogenous activity and the light-response of the retinal clock in mammals. Molecular Vision. 26, 106-116 (2020).
- Battat, S., Weitz, D. A., Whitesides, G. M. An outlook on microfluidics: the promise and the challenge. Lab on a Chip. 22 (3), 530-536 (2022).
- Petrenko, V., Saini, C., Perrin, L., Dibner, C. Parallel measurement of circadian clock gene expression and hormone secretion in human primary cell cultures. Journal of Visualized Experiments. (117), e54673(2016).
- Yamagishi, K., Enomoto, T., Ohmiya, Y. Perfusion-culture-based secreted bioluminescence reporter assay in living cells. Analytical Biochemistry. 354 (1), 15-21 (2006).
- Watanabe, T., et al. Multichannel perfusion culture bioluminescence reporter system for long-term detection in living cells. Analytical Biochemistry. 402 (1), 107-109 (2010).
- Murakami, N., Nakamura, H., Nishi, R., Marumoto, N., Nasu, T. Comparison of circadian oscillation of melatonin release in pineal cells of house sparrow, pigeon and Japanese quail, using cell perfusion systems. Brain Research. 651 (1-2), 209-214 (1994).
- Erickson, P., Houwayek, T., Burr, A., Teryek, M., Parekkadan, B. A continuous flow cell culture system for precision cell stimulation and time-resolved profiling of cell secretion. Analytical Biochemistry. 625, 114213(2021).
- Saltzman, W. M. Drug Delivery: Engineering Principles for Drug Therapy. , Oxford University Press. (2001).
- Fogler, H. S. Elements of Chemical Reaction Engineering. 4th edn. , Prentice Hall PTR. Boston. (2006).
- Conesa, J. A. Chemical Reactor Design: Mathematical Modeling and Applications. , Wiley. (2019).
- Toson, P., Doshi, P., Jajcevic, D. Explicit residence time distribution of a generalised cascade of continuous stirred tank reactors for a description of short recirculation time (bypassing). Processes. 7 (9), 615(2019).
- Tamayo, A. G., Shukor, S., Burr, A., Erickson, P., Parekkadan, B. Tracking leukemic T-cell transcriptional dynamics in vivo with a blood-based reporter assay. FEBS Open Biology. 10 (9), 1868-1879 (2020).
- Newell, B., Bailey, J., Islam, A., Hopkins, L., Lant, P. Characterising bioreactor mixing with residence time distribution (RTD) tests. Water Science and Technology. 37 (12), 43-47 (1998).
- Dubois, J., Tremblay, L., Lepage, M., Vermette, P. Flow dynamics within a bioreactor for tissue engineering by residence time distribution analysis combined with fluorescence and magnetic resonance imaging to investigate forced permeability and apparent diffusion coefficient in a perfusion cell culture chamber. Biotechnology and Bioengineering. 108 (10), 2488-2498 (2011).
- Gaida, L. B., et al. Liquid and gas residence time distribution in a two-stage bioreactor with cell recycle. HAL Open Science. , (2008).
- Rodrigues, M. E., Costa, A. R., Henriques, M., Azeredo, J., Oliveira, R. Wave characterization for mammalian cell culture: residence time distribution. New Biotechnology. 29 (3), 402-408 (2012).
- Olivet, D., Valls, J., Gordillo, M. A., Freixó, A., Sánchez, A. Application of residence time distribution technique to the study of the hydrodynamic behaviour of a full-scale wastewater treatment plant plug-flow bioreactor. Journal of Chemical Technology and Biotechnology. 80 (4), 425-432 (2005).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon