Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un pipeline pour étudier les structures et les voies de signalisation des récepteurs de la sphingosine 1-phosphate
Dans cet article
Résumé
S1P exerce ses divers effets physiologiques à travers la sous-famille des récepteurs S1P (S1PR). Ici, un pipeline est décrit pour expliquer les structures et la fonction des S1PR.
Résumé
Les lysophospholipides (LPL) sont des lipides bioactifs qui comprennent la sphingosine 1-phosphate (S1P), l’acide lysophosphatidique, etc. S1P, un produit métabolique des sphingolipides dans la membrane cellulaire, est l’un des LPL les mieux caractérisés qui régule une variété de réponses physiologiques cellulaires via des voies de signalisation médiées par les récepteurs de la sphingosine 1-phosphate (S1PR). Cela implique que le système de signalisation S1P-S1PRs est une cible thérapeutique potentielle remarquable pour des troubles, notamment la sclérose en plaques (SEP), les maladies auto-immunes, le cancer, l’inflammation et même la COVID-19. Les S1PR, un petit sous-ensemble de la famille des récepteurs couplés aux protéines G (RCPG) de classe A, sont composés de cinq sous-types: S1PR1, S1PR2, S1PR3, S1PR4 et S1PR5. Le manque d’informations structurelles détaillées, cependant, entrave la découverte de médicaments ciblant les RP S1. Ici, nous avons appliqué la méthode de cryo-microscopie électronique pour résoudre la structure du complexe S1P-S1PRs et élucidé le mécanisme d’activation, la reconnaissance sélective des médicaments et le couplage des protéines G en utilisant des tests fonctionnels cellulaires. D’autres récepteurs des lysophospholipides (LPLR) et RCPG peuvent également être étudiés en utilisant cette stratégie.
Introduction
La sphingosine-1-phosphate (S1P), un produit métabolique des sphingolipides dans la membrane cellulaire, est une molécule de signalisation lysophosphatique omniprésente qui implique diverses activités biologiques, y compris le trafic de lymphocytes, le développement vasculaire, l’intégrité endothéliale et la fréquence cardiaque 1,2,3. Le S1P exerce ses divers effets physiologiques par l’intermédiaire de cinq sous-types de récepteurs S1P (S1PRs 1-5); Les S1PR se trouvent dans une variété de tissus et présentent des préférences uniques pour les protéines G en aval 4,5. S1PR1 est principalement couplé à la protéine Gi, qui inhibe par la suite la production d’AMPc; S1PR2 et S1PR3 sont couplés avec Gi, Gq et G12/13, et S1PR4 et S1PR5 transduisent le signal via Gi et G12/136.
La signalisation S1P-S1PR est une cible thérapeutique essentielle pour de multiples maladies, notamment les maladies auto-immunes7, l’inflammation8, le cancer9 et même la COVID-1910. En 2010, le fingolimod (FTY720) a été homologué en tant que médicament de première classe ciblant les RP S1 pour traiter la sclérose en plaques (SEP)11. Cependant, il est capable de se lier à tous les S1PR sauf S1PR2, tandis que la liaison non spécifique à S1PR3 entraîne un œdème du cortex cérébral, une constriction vasculaire et bronchique et une fuite épithélialepulmonaire 12. Comme stratégie alternative pour augmenter la sélectivité thérapeutique, des ligands spécifiques au sous-type pour le récepteur ont été produits. Le siponimod (BAF312) a été approuvé en 2019 pour le traitement de la SEP en rechute13; il cible efficacement S1PR1 et S1PR5, alors qu’il n’a aucune affinité pour S1PR3, présentant moins d’effets secondaires dans la pratique clinique14. En 2020, la Food and Drug Administration des États-Unis a autorisé l’ozanimod pour le traitement de la SEP15. Il a été rapporté que l’ozanimod détient une sélectivité 25 fois plus grande pour S1PR1 que pour S1PR516. Notamment, dans le contexte de la pandémie actuelle de COVID-19, il a été découvert que des médicaments agonistes ciblant les RP S1 peuvent être utilisés pour traiter la COVID-19 en utilisant des techniques de thérapie immunomodulatrice17. En comparaison avec le fingolimod, l’ozanimod a montré sa supériorité dans la réduction des symptômes chez les patients atteints de COVID-19 et fait actuellement l’objet d’essais cliniques10. Comprendre la base structurelle et la fonction des RP S1 jette des bases importantes pour le développement d’un médicament qui cible sélectivement les PRS11 18.
De nombreuses techniques sont utilisées pour étudier l’information structurelle des biomacromolécules, y compris la cristallographie aux rayons X, la résonance magnétique nucléaire (RMN) et la microscopie électronique (EM). En mars 2022, plus de 180 000 structures ont été déposées sur la banque de données sur les protéines (PDB), et la plupart d’entre elles ont été résolues par cristallographie aux rayons X. Cependant, avec la première structure de résolution quasi atomique de TPRV1 (résolution de 3,4 Å) rapportée par Yifan Cheng et David Julius en 2013 19, la cryo-microscopie électronique (cryo-EM) est devenue une technique courante pour les structures protéiques, et le nombre total de structures EM PDB était supérieur à10 000. Les domaines de percée critiques sont le développement de nouvelles caméras pour l’imagerie connues sous le nom de caméras de détection directe d’électrons et de nouveaux algorithmes de traitement d’image. Cryo-EM a révolutionné la biologie des structures et la découverte de médicaments basés sur la structure au cours de la dernière décennie20. Comme comprendre comment les complexes macromoléculaires remplissent leurs rôles complexes dans la cellule vivante est un thème central en sciences biologiques, la cryo-EM a le potentiel de révéler des conformations de complexes moléculaires dynamiques, en particulier pour les protéines transmembranaires21. Les récepteurs couplés aux protéines G (RCPG) constituent la plus grande superfamille de protéines transmembranaires et les cibles de plus de 30 % des produits pharmaceutiques actuellement commercialisés22. Le développement de la cryo-EM a contribué à une explosion de structures à haute résolution de complexes protéiques GPCR-G, permettant la détermination structurelle de cibles « réfractaires » qui ne sont toujours pas accessibles à l’analyse cristallographique aux rayons X dans la conception de médicaments23. Par conséquent, l’application cryo-EM offre une chance de déterminer la structure tridimensionnelle des RCPG dans des conditions quasi natives à une résolution atomique proche de24. Les progrès de la cryo-EM permettent de visualiser les fondements mécanistes de la stimulation ou de l’inhibition des RCPG, et d’autres avantages dans la découverte de nouveaux sites de liaison pour la création de médicaments ciblant les RCPG25.
En nous appuyant sur les énormes progrès de la technologie cryo-EM, nous avons identifié des structures de complexes de signalisation agonisés S1PR1-, S1PR3- et S1PR5-Gi récemment26,27. Chez l’homme, les S1PR se trouvent dans divers tissus et présentent des préférences uniques pour les protéines G en aval 4,5. S1PR1 est principalement couplé à la protéine Gi, qui inhibe par la suite la production d’adénosine monophosphate 3′,5′-cyclique (AMPc). S1PR3 et S1PR5 sont également capables de se coupler avec Gi 6,28. Étant donné que l’activation des récepteurs gicouplés diminue la production d’AMPc29, un test d’inhibition de l’AMPc de Gi a été introduit pour mesurer les effets d’inhibition de l’AMPc pour capturer les altérations fonctionnelles26,27. Utilisant une version mutante de Photinus pyralis luciferase dans laquelle une fraction protéique de liaison à l’AMPc a été insérée, ce test d’AMPc offre une méthode simple et fiable pour surveiller l’activité des RCPG par des changements dans la concentration intracellulaire d’AMPc30. Il s’agit d’un essai fonctionnel sensible et non radioactif qui peut être appliqué pour surveiller la signalisation en aval en temps réel d’un large éventail de RCPG à des fins de découverte de médicaments31.
Ici, un résumé est fourni des méthodes critiques pour résoudre les modes d’activation et de reconnaissance des médicaments des S1PR, y compris principalement des manipulations cryo-EM et un test d’AMPc d’inhibition de Gi. Cet article vise à fournir des conseils expérimentaux complets pour d’autres explorations des structures et des fonctions des RCPG.
Protocole
1. Purification du complexe protéique S1PRs-G
- Pour purifier le complexe protéique humain S1PRs-G, cloner les ADNc de S1PR1 dépourvus de résidus C-terminaux 338-382, le type sauvage S1PR3, S1PR5 tronqué avec 345-398 à l’extrémité C, et le Gi1 de type sauvage dans le vecteur pFastBac1 et les ADNc des Gβ1 et Gγ2 de type sauvage dans le vecteur pFastBacdual (Table of Materials).
REMARQUE: Toutes les constructions pour les S1PR contiennent également la séquence de signal d’hémagglutinine (HA) suivie d’une étiquette épitope Flag à l’extrémité N et d’une étiquette 10x sa balise à l’extrémité C. En outre, une séquence d’ADN synthétique (Table of Materials) pour traduire le lysozyme T4 (T4L) a été insérée dans l’extrémité N des S1PR afin de faciliter l’expression et la purification des récepteurs. - Préparation du baculovirus codant pour les S1PR, Gi1 et Gβ1γ2
- Ajouter les vecteurs recombinants à 50 μL d’Escherichia coli (E. coli) compétent en DH5α stocké dans un tube de 1,5 mL à -80 °C et incuber sur de la glace pendant 30 min.
- Choquer les cellules à 42 °C pendant 90 s, les transférer immédiatement sur la glace et les refroidir pendant 2 min.
- Agiter le tube à 37 °C pendant 3-5 h après avoir complété avec 300 μL de bouillon lysogénique (LB) milieu. Plaquer 100 μL de cellules sur la gélose LB et incuber à 37 °C en maintenant à l’obscurité pendant 48 h.
- Inoculer la colonie blanche dans 5 mL de milieu LB contenant 50 μg/mL de kanamycine, 10 μg/mL de tétracycline et 7 μg/mL de gentamicine, et cultiver à 37 °C pendant 16 h.
- Isoler le bacmide recombinant avec la trousse de miniprep plasmidique (Table of Materials) en suivant les instructions du fabricant, pour produire le baculovirus P0.
REMARQUE: Avant utilisation, le bacmide purifié a été analysé par PCR avec des amorces pUC / M13 avant et arrière. Pour la PCR, nombre de cycles = 30 cycles, température de fusion = 58 °C et temps d’extension = 1 min par 1 Kb. - Préparer le baculovirus P0 tel que décrit dans un protocole antérieur32.
- Cultivez les cellules sf9 (milieu ESF921) dans des plaques à six puits et vérifiez que les cellules sont en phase logarithmique (1,0-1,5 x 106 cellules/mL).
- Diluer 8 μL de réactif de transfection du baculovirus dans 100 μL du milieu de Grace, et incuber pendant 30 min à température ambiante. Diluer 10 μg de bacmide recombinant dans 100 μL du milieu de Grace, et mélanger délicatement. Combiner le bacmide dilué avec le réactif de transfection du baculovirus dilué, mélanger doucement et incuber pendant 30 minutes à température ambiante.
- Ajouter le mélange (bacmide et réactif) sur les cellules (étape 1.2.6.1) et les plaquer à 27 °C pendant 3 h.
- Retirez le milieu de la Grace et remplacez-le par 2 ml de milieu de culture cellulaire ESF921. Plaquer les plaques à six puits à 27 °C et recueillir le milieu de culture cellulaire ESF921 après 5 jours après la transfection.
- Centrifuger à 500 x g à 4 °C pendant 10 min pour enlever les cellules et les débris. Transférer le surnageant dans des tubes de 2 mL. Il s’agit du stock de baculovirus P0.
- Isoler le stock de virus P1
- Transférer 30 mL de cellules sf9 dans un flacon conique, les cultiver à 27 °C en agitant à 270 tr/min et vérifier que les cellules atteignent une phase logarithmique (1,0-1,5 x 106 cellules/mL).
- Ajouter 2 mL de bouillon du virus P0 dans le flacon et agiter à 270 rpm pendant 4 jours à 27 °C.
- Transférer les cellules dans un tube de 50 mL, centrifuger à 1 800 x g pendant 10 min à 4 °C pour éliminer les cellules et les débris, et transférer le surnageant dans des tubes de 50 mL. Il s’agit du stock de baculovirus P1.
- Amplifier le stock de baculoviraux
- Répétez l’étape 1.2.7 en utilisant 50 mL de cellules sf9 en phase logarithmique (1,0-1,5 x 106 cellules/mL) et 1 mL de P1 stock.
- Conservez le stock de baculovirus P2 obtenu à 4 °C, en le protégeant de la lumière.
NOTE: Ne pas amplifier le baculovirus indéfiniment, car les mutants délétères ont été produits à chaque passage.
- Expression du complexe protéique S1PRs-G
- Culture de cellules d’insectes sf9 pour atteindre une densité de 2,5 x 106 cellules/mL, co-infection avec le baculovirus P2 codant pour S1PRs, Gi1 et Gβ1γ2 à un rapport volumique de 1:2:1, et culture à nouveau à 27 °C pendant 48 h.
- Recueillir les cellules par centrifugation à 700 x g à 4 °C pendant 15 min, les congeler dans de l’azote liquide et les stocker à -80 °C pour utilisation.
- Purification des protéines
- Décongeler la pastille cellulaire obtenue à l’étape 1.3 à température ambiante, puis la remettre en suspension dans un tampon de lyse (20 mM HEPES pH 7,5, 50 mM NaCl, 5 mM MgCl2, 5 mM CaCl2) complété par 100 μg/mL de benzamidine, 100 μg/mL de leupeptine, 100 μg/mL d’aprotinine, 25 mU/mL d’apyrase et 10 μM d’agoniste. Remuer la suspension cellulaire à température ambiante pendant 2 h pour induire la formation du complexe protéique S1PRs-G.
NOTE: L’apyrase est une ATP diphosphohydrolase. Il catalyse l’élimination du phosphate gamma de l’ATP et du phosphate bêta de l’ADP. - Transférer la solution dans les tubes, centrifuger à 70 000 x g pendant 10 min et retirer le surnageant soigneusement. Remettre la pastille en suspension dans un tampon solubilisant (20 mM HEPES pH 7,5, 100 mM NaCl, 5 mM MgCl2, 5 mM CaCl2, 0,5 % (p/v) LMNG, 0,1 % (p/v) CHS, 1 % (p/v) cholate desodium, 10 % (v/v) glycérol).
- Transférer la suspension dans un verre Rebondir et homogénéiser complètement la pastille. Ajouter 10 μM d’agoniste, 4 mg de scFv16, 100 μg/mL de benzamidine, 100 μg/mL de leupeptine, 100 μg/mL d’aprotinine et 25 mU/mL d’apyrase à la suspension, et agiter à 4 °C pendant 2 h.
REMARQUE: Les étapes d’homogénéisation des granulés sont cruciales pour la production du complexe protéique GPCR-G. - Transférer la solution dans les tubes et centrifuger à 100 000 x g pendant 30 min à 4 °C.
- Pré-équilibrer la résine drapeau avec le tampon de lavage (20 mM HEPES pH 7,5, 100 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2, 10 μM agoniste, 0,0375% (p/v) LMNG, 0,0125% (p/v) GDN, 0,01% (p/v) CHS).
- Transférer le surnageant dans les tubes et incuber avec la résine drapeau à 4 °C pendant 2 h.
- Charger le mélange ci-dessus sur la colonne de verre et laver la colonne avec 50 mL de tampon de lavage additionné de 100 μg/mL de benzamidine, 100 μg/mL de leupeptine et 100 μg/mL d’aprotinine.
- Éluer la colonne avec 10 mL du tampon d’élution contenant 20 mM HEPES pH 7,5, 100 mM NaCl, 5 mM EDTA, 200 μg/mL Flag peptide, 10 μM agoniste, 0,0375 % (p/v) LMNG, 0,0125 % (p/v) GDN, 0,01 % (p/v) CHS, 100 μg/mL benzamidine, 100 μg/mL de leupeptine et 100 μg/mL d’aprotinine.
- Prélever le complexe protéique S1PRs-G et le concentrer à 1 mL à l’aide d’un concentrateur de coupure de 100 kDa à 1 300 x g à 4 °C. Filtrer à travers un filtre de 0,22 μM et centrifuger à 13 000 x g pendant 10 min à 4 °C pour éliminer les agrégats.
- Charger le complexe protéique S1PRs-G sur une colonne de filtration sur gel de chromatographie d’exclusion de taille (SEC) prééquilibrée avec le tampon SEC contenant 20 mM HEPES pH 7,5, 100 mM NaCl, 10 μM agonist, 100 μm TCEP, 0,00375% (p/v) LMNG, 0,00125% (p/v) GDN et 0,001% (p/v) CHS à un débit de 0,5 mL/min à 4 °C.
- Recueillir les fractions de crête et concentrer à l’aide d’un concentrateur de coupure de 100 kDa à 1 300 x g à 4 °C pour la cryo-EM.
- Décongeler la pastille cellulaire obtenue à l’étape 1.3 à température ambiante, puis la remettre en suspension dans un tampon de lyse (20 mM HEPES pH 7,5, 50 mM NaCl, 5 mM MgCl2, 5 mM CaCl2) complété par 100 μg/mL de benzamidine, 100 μg/mL de leupeptine, 100 μg/mL d’aprotinine, 25 mU/mL d’apyrase et 10 μM d’agoniste. Remuer la suspension cellulaire à température ambiante pendant 2 h pour induire la formation du complexe protéique S1PRs-G.
2. Microscopie électronique pour résoudre la structure S1PRs
- Collecte de données
- Pour préparer les grilles cryo-EM, maintenez les grilles R1.2/1.3 de 300 mailles Au pendant 10 s et déchargez par lueur pendant 60 s à 15 mA à l’aide d’un système de nettoyage par décharge luminescente.
- Effectuer la vitrification de l’échantillon comme décrit précédemment33,34. Dans la console de congélation, réglez la température à 4 °C et l’humidité relative à 100% pour l’environnement de travail de la chambre. Utilisez la force de transfert pendant 0, le temps d’attente de 0 s, le temps de transfert de 2 ou 3 s et le temps de vidange de 0 s. Il ne nécessite généralement que 3 μL de l’échantillon à des concentrations de 5 à 10 mg / mL pour une vitrification unique.
- Clipser et charger les grilles dans l’assemblage de la grille automatique, charger l’assemblage de la grille automatique dans Nanocab et charger Nanocab dans le microscope par chargeur automatique comme décrit précédemment35. Qualité des échantillons d’écran avec le logiciel EPU234. Habituellement, les données recueillies dans les zones d’épaisseur de glace appropriée étaient meilleures.
- Recueillir des données cryo-EM comme décrit en détail précédemment35. Pour le récepteur S1P, régler le décalage de défocalisation entre -1,0 μm et -1,8 μm avec la dose électronique d’exposition de 50-65 e-/Å2. Pour les complexes S1PR1-Gi, collectez automatiquement la pile de films à l’aide du logiciel EPU2 en mode comptage avec le détecteur K2 à un temps d’exposition total de 2 s, une fréquence d’enregistrement de cinq images brutes par seconde et une dose totale de 56 e-/Å2 pour produire 35 images par pile.
REMARQUE: Habituellement, plus de 5 000 films sont nécessaires pour reconstruire la structure du récepteur.
- Traiter les données à l’aide d’une combinaison de RELION36 et cryoSPARC37 pour obtenir une carte de densité cryo-EM idéale. Utiliser RELION-3.1_gpu_ompi4 pour traiter les données initialement, ce qui implique des opérations similaires à celles décrites précédemment34.
- Dans le terminal système Linux, entrez le répertoire parent du répertoire de stockage des données.
- Entrez la commande relion dans le terminal pour ouvrir l’interface utilisateur graphique (GUI) RELION.
REMARQUE: Si c’est la première fois qu’une interface graphique RELION a été ouverte dans ce répertoire, une fenêtre d’invite apparaîtra; cliquez sur Oui. - Cliquez sur Importer dans la barre de fonctions sur le côté droit de l’interface graphique RELION pour importer les données brutes dans le RELION.
- Dans l’option Films/micros, sélectionnez Oui pour Importer des films/micrographies bruts ?, entrez le chemin d’accès aux données dans le champ Fichiers d’entrée bruts (des caractères génériques sont recommandés), puis sélectionnez Oui pour Ces films sont-ils multi-images ?. Entrez la taille en pixels des films dans le champ Taille de pixel (angström), la tension de fonctionnement du microscope (en kV) dans le champ Tension (kV) et l’aberration sphérique du microscope dans le champ Aberration sphérique (mm). Ce sont les paramètres qui ont été enregistrés au moment de la collecte des données.
- Dans l’option En cours d’exécution , modifiez le nom de la file d’attente en fonction du serveur sur lequel le programme s’exécute (d’autres fonctions doivent également modifier ce paramètre). Laissez les autres paramètres aux valeurs par défaut définies par RELION. Enfin, lorsque tous les paramètres sont détectés correctement, cliquez sur RUN! pour exécuter le programme.
- Utilisez la fonction de correction de mouvement pour l’alignement de toutes les images38.
- Dans l’option E / S, cliquez sur Parcourir et choisissez la sortie de la fonction d’importation nommée movies.star comme entrée du fichier STAR des films d’entrée. Entrez la dose par base de sondage dans le champ Dose par base de sondage (e/A2) qui est égale à la dose totale divisée par le nombre de bases de sondage. Sélectionnez Non pour Enregistrer la somme des spectres de puissance ?.
- Dans l’option Motion, entrez 250 pour le facteur B, 5,5 pour le nombre de patchs X, Y et 2 pour les images de groupe (assurez-vous de la dose du groupe >3). Si les données n’ont pas été référencées par gain pendant la période de collecte, une image de référence de gain est nécessaire et peut être obtenue en acquérant une zone de grille vide. Sélectionnez Non pour utiliser la propre implémentation de RELION? et entrez le répertoire contenant le fichier exécutable de MOTIONCOR2 39dans le champ exécutable MOTIONCOR2.
- Dans l’option En cours d’exécution , choisissez l’IPM et le numéro de thread appropriés en fonction de la puissance de calcul du serveur ; ici, MPI = 8 et threads = 3 ont été utilisés.
- Utiliser la fonction d’estimation CTF pour moduler l’image cryo-EM de l’échantillon vitrifié40. Dans l’option E / S, cliquez sur Parcourir et choisissez la sortie de Motion cor nommé corrected micrographs.star comme entrée du fichier STAR des films d’entrée. Dans l’option Gctf, sélectionnez Oui pour UseGctf à la place ?.
- Utilisez la fonction de sélection de sous-ensembles pour supprimer les micrographies avec une valeur de rlnCtfMaxResolutin >4.
- Dans l’option E /S , cliquez sur Parcourir situé à droite de OU sélectionnez parmi micrographs.star et choisissez la sortie de CtfFind nommée micrographs_ctf.star comme entrée. Dans l’option Sous-ensemble , sélectionnez Oui pour Sélectionner en fonction des valeurs de métadonnées ? et entrez 4 pour la valeur Nombre maximal de métadonnées afin de supprimer les données incorrectes.
- Sélection manuelle: choisissez certaines images manuellement pour la sélection préliminaire et la classification.
- Dans l’option E /S , cliquez sur Parcourir et choisissez micrographs_selected.star dans le répertoire de sélection précédent (étape 2.2.6) comme entrée.
- Cliquez sur RUN! (une fenêtre apparaît). Cliquez sur Fichier dans le coin supérieur gauche de la nouvelle fenêtre et cliquez sur Inverser la sélection pour annuler la sélection de toutes les images. Cochez la case de sélection la plus à gauche dans la ligne correspondante de chaque entrée et cliquez sur choisir pour vérifier les images et sélectionner ~ 500 bonnes images. Cliquez sur Fichier > Enregistrer la sélection pour enregistrer les images sélectionnées et fermer la fenêtre.
- Auto-picking: les progiciels de prélèvement automatisé de particules sont utiles et puissants41.
- Dans l’option E /S , cliquez sur Browse to the right of Input micrographs for autopick (Micrographies d’entrée) pour autopick et choisissez micrographs_selected.star dans le répertoire ManualPick précédent (Étape 2.2.7) comme entrée. L’algorithme laplacien-de-gaussien est utilisé au début, donc sélectionnez Oui pour OU: utilisez Laplacien-de-Gaussien.
- Dans l’option Laplacien, définissez Min.diameter pour le filtre LoG (A) sur 80 et Max.diameter pour le filtre LoG (A) sur 130. Dans l’option Autopicking, définissez Distance interparticulaire minimale (A) sur 65 et sélectionnez Oui pour Utiliser l’accélération GPU si le GPU est accessible.
- Extraire les particules pour les prochaines étapes.
- Dans l’option E /S , cliquez sur Parcourir à droite du fichier STAR de la micrographie et choisissez le fichier micrographs_selected.star de l’étape 2.2.6. Cliquez sur Parcourir à droite de Coordonnées d’entrée et choisissez le cords_suffix_autopick.star de l’étape 2.2.8.
- Dans l’option Extraire, sélectionnez Oui pour Redimensionner les particules et définissez la taille redimensionnée (pixels) sur 128 pour accélérer les étapes ultérieures.
- Classification 2D pour la classification préliminaire des particules
- Dans l’option E /S , cliquez sur Browse to the right of Input images STAR file (Parcourir à droite de Input images STAR file) et choisissez particles.star à l’étape 2.2.9. Dans l’option Optimisation , définissez Nombre de classes sur 100 et Diamètre du masque (A) sur 140.
- Dans l’option Calculer , définissez Nombre de particules regroupées sur 10, entrez le répertoire qui se trouve sur un lecteur local rapide (par exemple, un lecteur SSD) dans le champ Copier la particule dans le répertoire de travail , puis sélectionnez Oui pour Utiliser l’accélération GPU ? pour une vitesse de traitement plus rapide.
- Sélection de sous-ensembles pour sélectionner de bons résultats 2D comme modèles pour choisir les particules
- Dans l’option E /S , cliquez sur Browse (Parcourir à droite de Select classes from model.star ) et choisissez la sortie de l’étape 2.2.10 nommée run_it025_model.star comme entrée. Cliquez sur RUN!. Dans la fenêtre pop-up, cochez Trier les images sur et Trier inversé? et cliquez sur Afficher!.
- Choisissez de bons résultats 2D représentatifs comme référence pour Référence de la fonction de prélèvement automatique .
REMARQUE : Les résultats sélectionnés sont encadrés en rouge. Les bons et les mauvais résultats de classification 2D sont affichés plus tard. - Cliquez avec le bouton droit de la souris et sélectionnez Enregistrer les classes sélectionnées.
- Utilisez le modèle pour le deuxième tour de sélection automatique. Dans l’option E /S , cliquez sur Browse to the right of Input micrographs for autopick (Parcourir à droite de Input micrographs) pour autopick et choisissez micrographs_selected.star à l’étape 2.2.6. Cliquez sur Parcourir à droite des références 2D, choisissez class_averages.star à l’étape 2.2.11 et sélectionnez Non pour OU: utilisez Laplacian-of-Gaussian.
- Effectuez à nouveau l’extraction des particules à l’aide de coord_suffix_autopick.star de l’étape 2.2.12 et micrographs_selected.star de l’étape 2.2.6.
- Effectuez à nouveau la classification 2D à l’aide de particles.star de l’étape 2.2.13.
- Effectuez à nouveau la sélection des sous-ensembles à l’aide de run_it025_optimiser.star de l’étape 2.2.14.
REMARQUE: Toutes les images 2D avec des contours clairs et des formes correctes doivent être choisies. - Effectuez l’extraction des particules comme suit. Dans l’option E /S , cliquez sur Parcourir à droite du fichier STAR de la micrographie, choisissez le micrographs_selected.star de l’étape 2.2.6 et sélectionnez Oui pour réextraire les particules raffinées?. Cliquez sur Browse (Parcourir à droite du fichier STAR des particules raffinées ) et choisissez particles.star à l’étape 2.2.15.
- Modèle initial 3D et génération d’une carte de référence : Dans l’option E /S , cliquez sur Browse to the right of Input images STAR file ( Parcourir à droite de Input images STAR file) et choisissez particles.star à l’étape 2.2.16. Définissez Nombre de classes sur 1 et Diamètre du masque (A) sur 140 dans l’option Optimisation .
- Classification 3D et génération d’une carte 3D préliminaire : Dans l’option E /S , cliquez sur Browse to the right of Input images STAR file (Images d’entrée ) et choisissez particles.star à l’étape 2.2.16. Cliquez sur Parcourir à droite de Carte de référence et choisissez le fichier initial_model.mrc de l’étape 2.2.17. Définissez Nombre de classes sur 4-6 et Diamètre du masque (A) sur 140 dans l’option Optimisation .
- Génération de masque : sélectionnez la ou les cartes 3D correctes de l’étape 2.2.17 comme entrée dans l’option E/S . Définissez le seuil de binarisation initial sur 0,05 (ajustez en fonction de la sortie), étendez le mappage binaire de ce nombre de pixels à 3 et ajoutez le bord souple de ce nombre de pixels à 8 dans l’option Masque .
- Utilisez cryoSPARC pour le prochain traitement.
- Créez un nouvel espace de travail et cliquez sur Job Builder pour la première tâche.
- Pour importer la pile de particules, entrez le chemin de particule (étape 2.2.16) dans le champ Chemin méta particule et le tracé du film (Étape 2.2.16) dans le champ Chemin de données de particule.
REMARQUE: Les paramètres Accélération de la tension (kV), Aberration sphérique (mm) et Taille des pixels (Angstrom) sont les mêmes que précédemment. La valeur du contraste d’amplitude (fraction) est de 0,1. - Importez des volumes 3D en saisissant le chemin du meilleur volume 3D à l’étape 2.2.18 dans le chemin des données du volume et en sélectionnant Mapper pour Type de volume importé.
- Importez le masque en entrant le chemin du masque (étape 2.2.19) dans le chemin des données du volume et en sélectionnant Masque pour Type de volume importé.
- Raffinement non uniforme : Prenez les sorties des étapes 2.2.22, 2.2.23 et 2.2.24 comme entrée.
REMARQUE: Cette fonction est très utile pour les protéines membranaires.- Faites glisser la sortie de l’étape 2.2.22 (imported_particles) en tant qu’entrée des particules (particule) de Non-uniform Refinement, la sortie de l’étape 2.2.23 (imported_volume_1) en entrée du volume ( volume) de Non-uniform Refinement et la sortie de l’étape 2.2.24 (imported_mask_1) en entrée du masque ( masque) de Non-uniform Refinement.
REMARQUE: Parfois, de meilleurs résultats peuvent être obtenus sans masque. - Cliquez sur File d’attente pour lancer le traitement.
- Faites glisser la sortie de l’étape 2.2.22 (imported_particles) en tant qu’entrée des particules (particule) de Non-uniform Refinement, la sortie de l’étape 2.2.23 (imported_volume_1) en entrée du volume ( volume) de Non-uniform Refinement et la sortie de l’étape 2.2.24 (imported_mask_1) en entrée du masque ( masque) de Non-uniform Refinement.
- Exécutez les étapes 2.2.18-2.2.25 pour de meilleurs résultats. Grâce à la série de traitement ci-dessus, une carte 3D S1PR-Gi de bonne résolution peut être obtenue.
3. Essai d’inhibition de l’AMPc médiée par S1PRs-Gi
REMARQUE: L’expérience d’inhibition de l’AMPc médiée par S1PRs-Gi a été divisée en plusieurs parties, et les procédures expérimentales suivantes sont détaillées. Le principe expérimental et le processus expérimental général sont représentés sous la forme d’un organigramme à la figure 1.
- Construction de plasmides
- Sous-cloner les ADNc de type sauvage S1PR1, S1PR3 et S1PR5 dans le vecteur pcDNA3.1+ avec une séquence de signaux HA suivie d’une étiquette Flag à l’extrémité N (Table of Materials).
REMARQUE : Les mutations pour tous les récepteurs ont été générées à l’aide du kit de mutagénèse (Tableau des matériaux).
- Sous-cloner les ADNc de type sauvage S1PR1, S1PR3 et S1PR5 dans le vecteur pcDNA3.1+ avec une séquence de signaux HA suivie d’une étiquette Flag à l’extrémité N (Table of Materials).
- Préparation du plasmide
- Ajouter les vecteurs pcDNA3.1+ recombinants ou le plasmide capteur (Table des matériaux) à 50 μL d’Escherichia coli ( E. coli) compétent en DH5α stocké dans un tube de 1,5 mL à -80 °C séparément, et incuber sur de la glace pendant 30 min. Choquer les cellules à 42 °C pendant 90 s, les transférer immédiatement sur la glace et les refroidir pendant 2 min.
REMARQUE: Le plasmide capteur fourni par le kit de dosage de l’AMPc d’inhibition de l’UGc (Table of Materials) exprime un gène de luciférase modifié fusionné avec un domaine de liaison à l’AMPc et augmente l’activité de luminescence lorsque l’AMPc est liée. - Agiter le tube à 37 °C pendant 1 h après avoir complété avec 300 μL de bouillon lysogène (LB). Plaquer 100 μL de cellules sur la gélose LB et incuber à 37 °C en maintenant dans l’obscurité pendant 48 h. Inoculer la colonie blanche dans 5 mL de milieu LB contenant 100 μg/mL d’ampicilline, et la cultiver à 37 °C pendant 16 h.
- Isoler l’ADN à l’aide de la trousse de minipréparation plasmidique (tableau des matériaux) en suivant les instructions du fabricant; le plasmide était à une concentration de plus de 350 ng/μL avec la valeur A260/A280 entre 1,7 et 1,9.
- Ajouter les vecteurs pcDNA3.1+ recombinants ou le plasmide capteur (Table des matériaux) à 50 μL d’Escherichia coli ( E. coli) compétent en DH5α stocké dans un tube de 1,5 mL à -80 °C séparément, et incuber sur de la glace pendant 30 min. Choquer les cellules à 42 °C pendant 90 s, les transférer immédiatement sur la glace et les refroidir pendant 2 min.
- Culture cellulaire
- Placez les cellules CHO-K1 dans une boîte de Petri de 10 cm, cultivez-les dans un incubateur à 37 °C contenant 5% de CO2 et récoltez-les lorsque la monocouche est à 80%-90% de confluence.
- Aspirer le milieu de croissance des cellules CHO-K1, ajouter 4 mL de trypsine-EDTA à 0,05 % préchauffé à 37 °C sur la boîte de Petri doucement et incuber pendant 15 s. Ensuite, ajoutez 4 mL de milieu de croissance composé de milieu F12 + 10% FBS.
- Délogez les cellules de la surface de la boîte de Petri en balançant doucement et en tapotant le côté de la boîte de Pedia. Remplissez un tube conique avec une suspension cellulaire. Remuer et pipeter lentement pour enlever doucement les amas cellulaires.
- Centrifuger les cellules à 250 x g pendant 5 minutes à température ambiante, aspirer le surnageant et réanimer avec 3 mL de PBS. Répétez cette étape.
- Déterminer le nombre de cellules à l’aide de l’hémocytomètre et centrifuger les cellules à 250 x g pendant 5 min à température ambiante.
- Aspirer le tampon PBS et remettre en suspension les cellules CHO-K1 avec 3 mL de milieu de croissance composé de milieu F12 et de FBS à 10%.
- Ajouter 2 mL du milieu de croissance composé de milieu F12 et de FBS à 10 % dans chaque puits de la plaque à six puits, et ensemencer 150 μL de suspension cellulaire dans chaque puits pour maintenir les cellules à la densité de 1,5 x 105 cellules/mL. Incuber la plaque à six puits dans un incubateur de culture tissulaire à 37 °C avec 5% de CO2 pendant environ 24 h.
- Transfection transitoire
- Diluer 2 μg d’ADN (étape 3.2.3) dans 200 μL de tampon de réactif de transfection (Tableau des matériaux). Mélanger en tourbillonnant pendant 10 s et en tournant brièvement avant utilisation.
REMARQUE: Ici, les 2 μg d’ADN contiennent 0,5 μg de vecteur récepteur (S1PR1, S1PR3 ou S1PR5) et 1,5 μg du plasmide capteur. - Ajouter 4 μL du réactif de transfection (Table des matériaux), vortex pendant 10 s et faire tourner brièvement avant utilisation. Incuber pendant 15 min à température ambiante.
- Déposer lentement 200 μL de mélange de transfection dans chaque puits (contenant des cellules CHO-K1) pour répartir uniformément. Secouez doucement la plaque à six puits pour assurer un mélange complet.
- Remplacez le milieu de transfection après 4-6 h par le milieu de croissance cellulaire constitué de milieu F12 + 10% FBS, et remettez la plaque à six puits dans l’incubateur.
- Récolter les cellules 24-48 h après la transfection.
- Digérer les cellules CHO-K1 sur le puits avec 500 μL de trypsine-EDTA à 0,05% (préchauffé à 37 °C) pendant 15 s et ajouter 1 mL de milieu de croissance composé de milieu F12 + 10% FBS. Délogez les cellules de la surface du puits en les balançant et en tapotant doucement le côté du puits.
- Transférer la suspension cellulaire dans un tube conique et centrifuger à 250 x g pendant 5 min à température ambiante. Versez le surnageant et récoltez les cellules transfectées.
REMARQUE: Avant la détermination du signal de fluorescence, vérifier les niveaux d’expression de surface cellulaire des récepteurs par ELISA comme décrit précédemment26.
- Diluer 2 μg d’ADN (étape 3.2.3) dans 200 μL de tampon de réactif de transfection (Tableau des matériaux). Mélanger en tourbillonnant pendant 10 s et en tournant brièvement avant utilisation.
- Equilibration avec le sel de D-Luciférine-potassium (Tableau des matériaux)
- Suspendre immédiatement les cellules récoltées (24 à 48 h après la transfection) avec 3 mL de tampon de dosage (c.-à-d. Solution saline équilibrée de Hank (HBSS) contenant 10 mM HEPES pH 7,4), avec une dilution supplémentaire de 3 % v/v du sel de D-Luciférine-potassium.
- Ajouter 90 μL de suspension cellulaire par puits d’une plaque de 96 puits à l’aide d’une pipette multicanal et mélanger délicatement.
- Incuber pendant 40 min à température ambiante.
- Détermination du signal de fluorescence
- Préparer à l’avance des solutions mères de 10 mM de Siponimod dissous dans du DMSO et effectuer une dilution en série à l’aide d’un tampon HBSS contenant 25 μM de forskoline avant la stimulation du ligand.
REMARQUE : À l’exception du groupe témoin sans ligand, les autres ont un gradient de concentration de 10-11-10-5 mol/L. - Stimuler avec 10 μL (par puits) de solution agoniste à différentes concentrations pendant 30 min.
- Comptez les signaux de luminescence sur un lecteur de microplaques à l’aide des paramètres logiciels associés (Table des matériaux) comme suit. Sélectionnez Luminescence pour Méthode de détection, Point de terminaison pour Type de lecture et Fibre de luminescence pour Type d’optique. Réglez le gain optique sur 255.
NOTE: Chaque mesure a été répétée dans au moins trois expériences indépendantes, chacune en trois exemplaires. - Obtenez les valeurs du signal de fluorescence, importez les données dans un tableur et traitez les données à l’aide de la fonction dose-réponse de régression non linéaire (ajustement de la courbe).
- Préparer à l’avance des solutions mères de 10 mM de Siponimod dissous dans du DMSO et effectuer une dilution en série à l’aide d’un tampon HBSS contenant 25 μM de forskoline avant la stimulation du ligand.
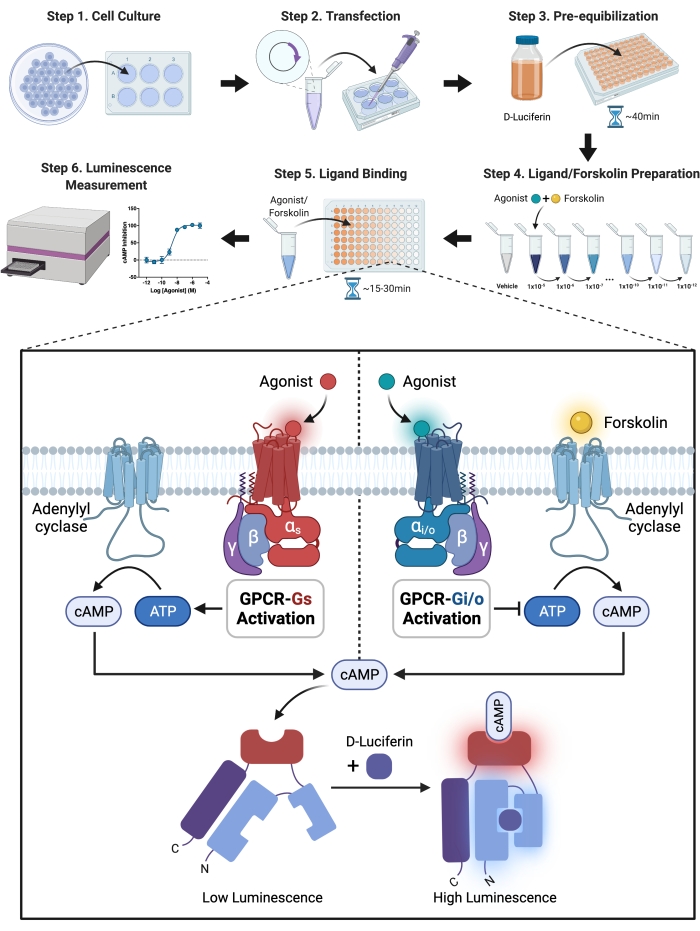
Figure 1 : Illustration schématique de l’expérience. Un guide détaillé par étapes pour la configuration et l’exécution expérimentales. En bref, le récepteur et la luciférase modifiée ont été co-exprimés transitoirement dans les cellules CHO-K1 en transfectant le récepteur et le plasmide capteur dans les cellules avec un réactif de transfection. Les cellules ont été suspendues dans une solution HBSS avec du sel de D-Luciferin-potassium, le substrat de la luciférase, et ensemencées dans une plaque de 96 puits après 24 heures. Pour permettre la perméation dans les cellules, la D-luciférine doit être pré-équilibrée avec les cellules. L’enzyme oxydative luciférase transforme la luciférine en oxyluciférine et émet de la lumière. La luciférase modifiée, d’autre part, génère de la lumière via une réaction chimique uniquement lorsqu’elle est liée à l’AMPc, et l’intensité de la lumière a une association positive avec les niveaux d’AMPc dans les cellules. Les niveaux d’AMPc ont été régulés avec des RCPG activés par agoniste. Les récepteurs couplés aux Gi ont réduit les niveaux d’AMPc, nécessitant l’ajout de forskoline pour activer l’adénylyl cyclase dans l’expérience d’inhibition de l’AMPc de Gi. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Avant de congeler l’échantillon du complexe S1PRs-Gi, l’échantillon purifié doit être séparé par chromatographie d’exclusion de taille (SEC) et analysé par chromatographie par filtration sur gel. La figure 2 montre le complexe S1PR3-Gi à titre d’exemple. La fraction maximale du complexe homogène de protéines GPCR-G était habituellement située à ~10,5 mL de la chromatographie d’exclusion de taille (Figure 2A). L’analyse SDS du complexe S1...
Discussion
Ce protocole décrit un pipeline primaire pour déterminer les structures des S1PR par cryo-EM et mesurer le pouvoir d’activation des S1PR par test d’inhibition de l’AMPc médié par Gi. Certaines étapes sont cruciales pour le succès de l’expérience.
Pour purifier le complexe S1PRs-Gi, il convient d’accorder plus d’attention à la qualité du virus et à la santé des cellules sf9 . L’expression du récepteur est considérablement réduite dans les cellules sf9
Déclarations de divulgation
Les auteurs n’ont pas de conflits d’intérêts.
Remerciements
Les données du complexe S1PRs-Gi ont été récoltées au West China Cryo-EM Center de l’Université du Sichuan et au Cryo-EM Center de la Southern University of Science and Technology (SUSTech) et traitées au Duyu High-Performance Computing Center de l’Université du Sichuan. Ce travail a été soutenu par la Natural Science Foundation of China (32100965 à L.C., 32100988 à W.Y., 31972916 à Z.S.) et le Fonds de recherche postdoctorale à temps plein de l’Université du Sichuan (2021SCU12003 à L.C.)
matériels
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
Références
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon