Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Transformation induite par un médiateur de phospholipides dans des cultures tridimensionnelles
Dans cet article
Résumé
Le présent protocole décrit la mise en place de cultures 3D « on top » d’une lignée cellulaire épithéliale mammaire non transformée, MCF10A, qui a été modifiée pour étudier la transformation induite par le facteur d’activation plaquettaire (PAF). L’immunofluorescence a été utilisée pour évaluer la transformation et est discutée en détail.
Résumé
Plusieurs modèles ont été développés pour étudier le cancer, tels que des modèles de rongeurs et des lignées cellulaires établies. Des études utilisant ces modèles ont fourni des informations précieuses sur la cancérogenèse. Les lignées cellulaires ont permis de comprendre la dérégulation de la signalisation moléculaire associée à la tumorigenèse du sein, tandis que les modèles de rongeurs sont largement utilisés pour étudier les caractéristiques cellulaires et moléculaires du cancer du sein in vivo. L’établissement de cultures 3D de cellules épithéliales et cancéreuses du sein aide à combler le fossé entre les modèles in vivo et in vitro en imitant les conditions in vivo in vitro. Ce modèle peut être utilisé pour comprendre la dérégulation d’événements complexes de signalisation moléculaire et les caractéristiques cellulaires au cours de la cancérogenèse mammaire. Ici, un système de culture 3D est modifié pour étudier une transformation induite par un médiateur phospholipide (facteur d’activation plaquettaire, PAF). Les immunomodulateurs et autres molécules sécrétées jouent un rôle majeur dans l’initiation et la progression de la tumeur dans le sein. Dans la présente étude, des cultures acineuses 3D de cellules épithéliales mammaires exposées au PAF présentaient des caractéristiques de transformation telles que la perte de polarité et des caractéristiques cellulaires altérées. Ce système de culture 3D aidera à faire la lumière sur les perturbations génétiques et / ou épigénétiques induites par diverses petites entités moléculaires dans le microenvironnement tumoral. En outre, ce système fournira également une plate-forme pour l’identification de gènes nouveaux et connus qui peuvent être impliqués dans le processus de transformation.
Introduction
Une myriade de modèles sont disponibles pour étudier la progression du cancer, chacun d’eux étant unique et représentant un sous-type de cette maladie complexe. Chaque modèle fournit des informations uniques et précieuses sur la biologie du cancer et a amélioré les moyens d’imiter l’état réel de la maladie. Les lignées cellulaires établies cultivées en monocouche ont fourni des informations précieuses sur les processus vitaux in vitro, tels que la prolifération, l’invasivité, la migration et l’apoptose1. Bien que la culture cellulaire bidimensionnelle (2D) ait été l’outil traditionnel pour étudier la réponse des cellules de mammifères à plusieurs perturbations environnementales, l’extrapolation de ces résultats pour prédire les réponses au niveau tissulaire ne semble pas suffisamment convaincante. La principale limitation des cultures 2D est que le microenvironnement créé diffère largement de celui du tissu mammaire lui-même2. La culture 2D manque de l’interaction des cellules avec la matrice extracellulaire, ce qui est vital pour la croissance de tout tissu. De plus, les forces de traction subies par la cellule dans les cultures monocouches entravent la polarité de ces cellules, modifiant ainsi la signalisation et le comportementdes cellules 3,4,5. Les systèmes de culture tridimensionnels (3D) ont ouvert une nouvelle voie dans le domaine de la recherche sur le cancer avec leur capacité à imiter les conditions in vivo in vitro. De nombreux indices microenvironnementaux cruciaux qui sont perdus dans la culture cellulaire 2D pourraient être rétablis en utilisant des cultures 3D de matrice extracellulaire riche en laminine (lrECM)6.
Diverses études ont identifié l’importance du microenvironnement tumoral dans la cancérogenèse 7,8. Les facteurs associés à l’inflammation constituent une partie importante du microenvironnement. Le facteur d’activation plaquettaire (PAF) est un médiateur phospholipide sécrété par diverses cellules immunitaires qui médie les réponses immunitairesmultiples 9,10. Des niveaux élevés de FAP sont sécrétés par différentes lignées cellulaires de cancer du sein et sont associés à une prolifération accrue11. Des études de notre laboratoire ont montré que la présence prolongée de PAF dans les cultures acineuses entraîne la transformation des cellules épithéliales du sein12. PAF active le récepteur PAF (PAFR), activant l’axe de signalisation PI3K/Akt13. Le DPAP est également associé à l’EMT, à l’invasion et aux métastases14.
Le présent protocole démontre un système modèle pour étudier la transformation induite par le PAF, en utilisant des cultures 3D de cellules épithéliales du sein, comme cela a été décrit précédemment par Chakravarty et al.12. Les cellules épithéliales mammaires cultivées sur la matrice extracellulaire (cultures 3D) ont tendance à former des sphéroïdes polarisés arrêtés par croissance. Ceux-ci sont appelés acini et ressemblent étroitement aux acini du tissu mammaire, la plus petite unité fonctionnelle de la glande mammaire, in vivo15. Ces sphéroïdes (Figure 1A,B) sont constitués d’une monocouche de cellules épithéliales polarisées étroitement tassées entourant une lumière creuse et fixées à la membrane basale (Figure 1C). Ce processus de morphogenèse a été bien décrit dans la littérature16. Lorsqu’elles sont ensemencées sur lrECM, les cellules subissent une division et une différenciation pour former un groupe de cellules, qui se polarisent ensuite à partir du jour 4. Au jour 8, les acini sont constitués d’un groupe de cellules polarisées qui sont en contact direct avec la matrice extracellulaire et d’un groupe de cellules non polarisées enfermées dans les cellules polarisées externes, sans contact avec la matrice. Ces cellules non polarisées sont connues pour subir l’apoptose au jour 12 de la culture, formant une lumière creuse. Au jour 16, les structures arrêtées par la croissance sont formées16.
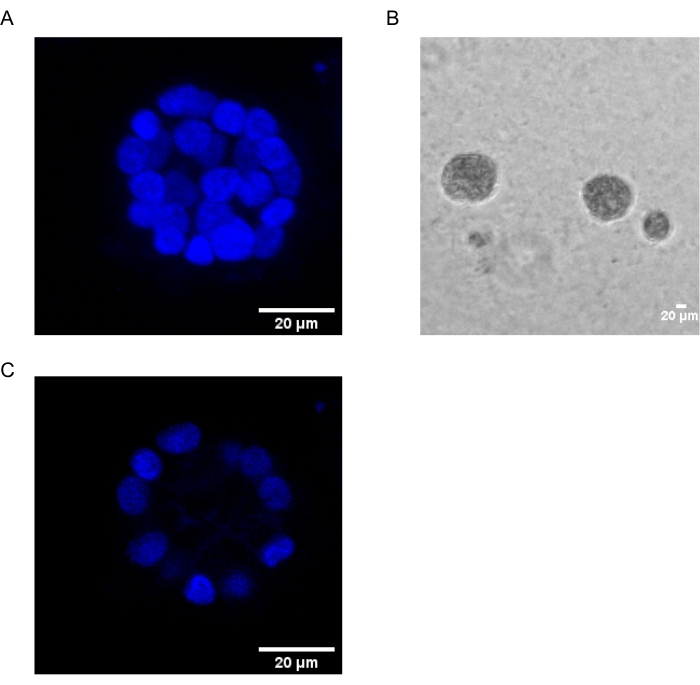
Figure 1 : Noyaux de cellules dans les acini colorés avec une coloration nucléaire. (A) Construction 3D des acini. (B) Image à contraste de phase de MCF10A acini cultivée sur Matrigel pendant 20 jours. (C) La section la plus centrale montre la présence d’une lumière creuse. Barre d’échelle = 20 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Contrairement aux cultures 2D, les cultures acineuses aident à distinguer les cellules normales et transformées par des changements morphologiques apparents. Les cellules épithéliales mammaires non transformées forment des acini avec une lumière creuse, imitant les acini mammaires humains normaux. Ces sphéroïdes, lors de la transformation, présentent une morphologie perturbée caractérisée par une perte importante de polarité (une des caractéristiques du cancer), l’absence de lumière ou une perturbation de la lumière creuse (due à l’évasion de l’apoptose) qui peut être induite en raison de la dérégulation de divers gènes17,18,19,20 . Ces transformations peuvent être étudiées à l’aide de techniques couramment utilisées telles que l’immunofluorescence. Ainsi, le modèle de culture cellulaire 3D peut fonctionner comme une méthode simple pour étudier le processus de morphogenèse acineuse du sein et de cancérogenèse mammaire. L’établissement d’un système de culture 3D pour comprendre l’effet d’un médiateur phospholipidique, PAF, aidera au criblage préclinique de médicaments à haut débit.
Ce travail a adapté le protocole de culture 3D 'on top' 16,21 pour étudier la transformation induite par PAF 22. Les changements phénotypiques induits par l’exposition des acini au médiateur phospholipide ont été étudiés par immunofluorescence. Divers marqueurs de polarité et de transition épithéliale à mésenchymateuse (EMT)12,16 ont été utilisés dans l’étude. Le tableau 1 mentionne leur localisation normale et leur phénotype attendu lors de la transformation.
| Anticorps | Marques | Localisation normale | Phénotype transformé |
| α6-Intégrine | Basolatéral | Basal avec faible coloration latérale | Forte tache latérale / apicale |
| β-Caténine | Jonction cellule-cellule | Basolatéral | Localisation anormale / nucléaire ou cytoplasmique |
| Vimentin | Emt | Présence absente / faible | Réglementation à la hausse |
Tableau 1 : Marqueurs utilisés dans l’étude. Différents marqueurs utilisés avec leur localisation en présence et en l’absence de traitement PAF.
Cette méthode peut être mieux utilisée pour étudier / cribler des médicaments plausibles et des gènes cibles pour divers sous-types de cancer du sein. Cela peut fournir des données de réponse aux médicaments plus proches du scénario in vivo , ce qui contribue à un développement de médicaments plus rapide et plus fiable. En outre, ce système peut être utilisé pour étudier la signalisation moléculaire associée à la réponse aux médicaments et à la résistance aux médicaments.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Ensemencement des cellules MCF10A dans lrECM
- Maintenir les cellules MCF10A (cellules épithéliales mammaires adhérentes) dans le milieu de croissance. Passez les cellules tous les 4 jours.
NOTE: Composition du milieu de croissance: DMEM à haute teneur en glucose sans pyruvate de sodium contenant du sérum de cheval (5%), insuline (10 μg/mL), hydrocortisone (0,5 μg/mL), facteur de croissance épidermique, EGF (20 ng/mL), de toxine cholérique (100 ng/mL) et pénicilline-streptomycine (100 unités/mL) (voir le tableau des matières). - Décongeler lrECM (voir Tableau des matériaux) sur de la glace 20 min avant le début de l’expérience. Le jour de l’ensemencement des cellules est généralement considéré comme le jour 0.
- Pour trypsiniser les cellules, aspirer le milieu et laver avec 2 ml de PBS. Ajouter 900 μL de trypsine-EDTA à 0,05% et incuber à 37 °C pendant ~15 min ou jusqu’à ce que les cellules soient complètement trypsinisées.
- Préparer un lit de lrECM dans huit puits d’une lame de verre à couvercle chambré (voir le tableau des matériaux).
- Lorsque les cellules trypsinisantes, enduisez chaque puits de 60 μL de lrECM et placez-le dans un incubateur à 37 °C CO2 pendant un maximum de 15 minutes.
- Préparez la suspension cellulaire en suivant les étapes ci-dessous.
- Après le délogement complet des cellules, ajouter 5 mL de milieu de remise en suspension pour éteindre l’activité de la trypsine et faire tourner les cellules à 112 x g pendant 10 minutes à 25 °C.
NOTE: Composition du milieu de remise en suspension: DMEM à haute teneur en glucose sans pyruvate de sodium, complété par du sérum de cheval (20%) et de la pénicilline-streptomycine (100 unités / mL). - Aspirer le milieu usé et remettre les cellules en suspension dans 2 mL de milieu de dosage. Bien mélanger la suspension pour assurer la formation d’une suspension unicellulaire.
NOTE: Composition du milieu de dosage: DMEM glycémique élevé sans pyruvate de sodium, complété par du sérum de cheval (2%), de l’hydrocortisone (0,5 μg / mL), de la toxine cholérique (100 ng / mL), de l’insuline (10 μg / mL) et de la pénicilline-streptomycine (100 unités / mL). - Compter les cellules à l’aide d’un hémocytomètre et calculer le volume de suspension cellulaire nécessaire pour ensemencer 6 x 103 cellules dans chaque puits.
REMARQUE : Il est généralement préférable d’inclure un puits supplémentaire dans le calcul pour tenir compte de toute erreur de pipetage. - Selon le nombre de puits requis, diluer la suspension cellulaire dans un milieu de recouvrement.
REMARQUE : La composition du milieu superposé pour un seul puits est la suivante : 400 μL de milieu d’essai, 8 μL de lrECM (final à 2 %) et 0,02 μL de 100 μg/mL d’EGF (5 ng/mL final).
- Après le délogement complet des cellules, ajouter 5 mL de milieu de remise en suspension pour éteindre l’activité de la trypsine et faire tourner les cellules à 112 x g pendant 10 minutes à 25 °C.
- Effectuer l’ensemencement des cellules.
- Ajouter 400 μL de la suspension cellulaire diluée aux lits d’EClr préparés (étape 1.4), en veillant soigneusement à ne pas perturber le lit de lrECM. Incuber à 37 °C dans un incubateur humidifié à 5% de CO2 .
2. Traitement PAF
- Ajouter PAF 3 h après l’ensemencement des cellules. Préparer un stock de 100 μM de PAF dans le PBS et ajouter le volume requis de 0,2 μL dans chaque puits (ce qui correspond à 200 nM).
- Ajoutez la même concentration de PAF lors de chaque changement de média.
3. Réalimentation avec des milieux frais
- Reconstituez les cellules avec du milieu frais tous les 4 jours (c.-à-d. Jour 4, Jour 8, Jour 12 et Jour 16).
4. Étude d’immunofluorescence pour détecter les changements phénotypiques induits par une exposition prolongée au FPA
- Après 20 jours de culture, pipeter soigneusement le milieu de chaque puits et laver les puits avec 400 μL de PBS préchauffé.
- Fixer les structures acineuses en ajoutant 400 μL de paraformaldéhyde à 4% (fraîchement préparé en diluant 16% de paraformaldéhyde dans 1x PBS) et en incubant pendant 20 min à température ambiante.
- Rincer les puits une fois avec du PBS glacé et perméabiliser avec du PBS contenant 0,5 % de Triton X-100 pendant 10 min à 4 °C.
- Après 10 min, extraire immédiatement mais soigneusement la solution Triton-X 100 et rincer avec 400 μL de PBS-glycine (fraîchement préparée en ajoutant une pincée de glycine dans 1x PBS). Ceci est répété trois fois pendant 15 minutes chacun.
- Ajouter 400 μL de la solution de blocage primaire comprenant 10 % de sérum de chèvre (voir tableau des matières) dans un tampon d’immunofluorescence (IF) et incuber à température ambiante pendant 60 min.
NOTE: Composition du tampon IF: 0,05% d’azoture de sodium, 0,1% BSA, 0,2% Triton-X 100 et 0,05% Tween (20 dans 1 PBS). - Retirer la solution de blocage primaire, ajouter 200 μL d’anticorps bloquant secondaire à 2 % (fragment d’anticorps F(ab')2 élevé chez la chèvre contre l’antigène de souris, voir le tableau des matières) préparé dans une solution de blocage primaire, et laisser agir pendant 45 à 60 min à température ambiante.
- Préparer l’anticorps primaire (voir le tableau des matières) dans une solution d’anticorps bloquant secondaire à 2 % dans une dilution de 1:100. Après avoir retiré la solution de blocage secondaire, ajouter l’anticorps fraîchement préparé et incuber pendant une nuit à 4 °C.
- L’étape précédente peut provoquer la liquéfaction de la membrane basale. Avant de poursuivre l’expérience, attendez que la lame atteigne la température ambiante. Pipeter soigneusement la solution d’anticorps primaires et la laver trois fois avec 400 μL de tampon IF.
- Lors du dernier lavage avec un tampon IF, préparer une dilution 1:200 de l’anticorps secondaire conjugué au fluorophore (voir le tableau des matériaux) dans la solution de blocage primaire. Incuber les lames dans la solution d’anticorps secondaire pendant 40-60 min à température ambiante.
- Rincer les lames avec 400 μL de tampon IF pendant 20 min, suivies de deux lavages avec du PBS pendant 10 min chacun.
- Contre-colorer les noyaux avec du PBS contenant 0,5 ng/mL de coloration nucléaire (voir le tableau des matières) pendant 5-6 minutes à température ambiante. Lavez les lames trois fois avec 400 μL de PBS pour enlever l’excès de tache.
- Extraire soigneusement le PBS entier et assurer l’élimination de la solution résiduelle. Ajoutez une goutte de réactif de montage (voir le tableau des matériaux) dans chaque puits et laissez-le reposer à température ambiante pendant la nuit.
- Conservez les lames à température ambiante et imagez-les dès que possible.
- Image sous grossissement 40x ou 63x sur un microscope confocal (voir Tableau des matériaux), en prenant des coupes Z optiques de 0,6 mm (NA = 1,4).
- Ouvrez les images acquises avec un logiciel de traitement d’image (voir Tableau des matériaux). Démontrer les différences morphologiques à l’aide de projections 3D.
REMARQUE: La section Z optique la plus centrale peut être mieux utilisée pour montrer les différences dans la localisation des marqueurs de polarité. Ceux-ci peuvent être quantifiés manuellement pour représenter le pourcentage de sphéroïdes montrant ce modèle de coloration spécifique. - Pour illustrer les différences dans l’expression des protéines, effectuer une analyse semi-quantitative mesurant la valeur grise moyenne ou calculant la fluorescence cellulaire totale corrigée (CTCF)23. Représenter les données sous forme de tracés de violon ou de points.
Access restricted. Please log in or start a trial to view this content.
Résultats
Les cellules MCF10A, lorsqu’elles sont exposées au traitement PAF, forment des structures acineuses avec des phénotypes très distincts. L’α6-intégrine s’est avérée mal localisée avec plus de coloration apicale. Quelques acini ont également montré une coloration discontinue (Figure 2A). Ces deux phénotypes indiquent la perte de polarité basale, comme en témoigne la littérature24,25. Des rapports antérieurs indique...
Access restricted. Please log in or start a trial to view this content.
Discussion
Les modèles établis basés sur des lignées cellulaires sont largement utilisés pour étudier le processus de cancérogenèse. Les cultures monocouches de cellules continuent de fournir des informations sur les diverses voies de signalisation moléculaire qui interviennent dans les changements caractéristiques des cellules cancéreuses32. Des études sur le rôle d’oncogènes bien connus tels que Ras, Myc et p53 muté ont d’abord été rapportées en utilisant des cultures monocouches comm...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts potentiel.
Remerciements
Nous remercions l’installation de microscopie IISER Pune pour l’accès à l’équipement et à l’infrastructure et pour le soutien aux expériences. Cette étude a été financée par une subvention du Département de biotechnologie (DBT), du gouvernement de l’Inde (BT / PR8699 / MED / 30/1018/2013), du Science and Engineering Research Board (SERB), du gouvernement de l’Inde (EMR / 2016 / 001974) et en partie par IISER, Pune Core funding. A. K. a été financé par la bourse CSIR-SRF, L.A. a été financé par la bourse DST-INSPIRE, V.C a été financé par DBT (BT / PR8699 / MED / 30/1018/2013).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin EDTA | Invitrogen | 25300062 | |
| 16% paraformaldehyde | Alfa Aesar | AA433689M | |
| Anti Mouse Alexa Flour 488 | Invitrogen | A11029 | |
| Anti Rabbit Alexa Flour 488 | Invitrogen | A-11008 | |
| BSA | Sigma | A7030 | |
| Chamber Coverglass | Nunc | 155409 | |
| Cholera Toxin | Sigma | C8052-1MG | 1 mg/mL in dH2O |
| Confocal Microscope | Leica | Leica SP8 | |
| DMEM | Gibco | 11965126 | |
| EDTA | Sigma | E6758 | |
| EGF | Sigma | E9644-0.2MG | 100 mg/mL in dH2O |
| F(ab’)2 fragment of antibody raised in goat against mouse antigen | Jackson Immunoresearch | 115-006-006 | |
| GM130 antibody | Abcam | ab52649 | |
| Goat Serum | Abcam | ab7481 | |
| Hoechst | Invitrogen | 33258 | |
| Horse Serum | Gibco | 16050122 | |
| Hydrocortisone | Sigma | H0888 | 1 mg/mL in ethanol |
| Image Processing Software | ImageJ | ||
| Insulin | Sigma | I1882 | 10 mg/mL stock dH2O |
| lrECM (Matrigel) | Corning | 356231 | |
| Mounting reagent (Slow fade Gold Anti-fade) | Invitrogen | S36937 | |
| Nuclear Stain (Hoechst) | Invitrogen | 33258 | |
| PAF | Cayman Chemicals | 91575-58-5 | Methylcarbamyl PAF C-16, procured as a 10 mg/mL in ethanol |
| Penicillin-Streptomycin | Lonza | 17-602E | |
| Sodium Azide | Sigma | S2002 | |
| Tris Base | Sigma | B9754 | |
| Triton X-100 | Sigma | T8787 | |
| Tween 20 | Sigma | P9416 | |
| Vimentin antibody | Abcam | ab92547 | |
| α6-integrin antibody | Millipore | MAB1378 |
Références
- Lacroix, M., Leclercq, G. Relevance of breast cancer cell lines as models for breast tumours: an update. Breast Cancer Research and Treatment. 83 (3), 249-289 (2004).
- Vargo-Gogola, T., Rosen, J. M. Modelling breast cancer: one size does not fit all. Nature Reviews Cancer. 7 (9), 659-672 (2007).
- Runswick, S. K., O'Hare, M. J., Jones, L., Streuli, C. H., Garrod, D. R. Desmosomal adhesion regulates epithelial morphogenesis and cell positioning. Nature Cell Biology. 3 (9), 823-830 (2001).
- Streuli, C. H., Bailey, N., Bissell, M. J. Control of mammary epithelial differentiation: basement membrane induces tissue-specific gene expression in the absence of cell-cell interaction and morphological polarity. Journal of Cell Biology. 115 (5), 1383-1395 (1991).
- Streuli, C. H., et al. Laminin mediates tissue-specific gene expression in mammary epithelia. Journal of Cell Biology. 129 (3), 591-603 (1995).
- Bissell, M. J., Kenny, P. A., Radisky, D. C. Microenvironmental regulators of tissue structure and function also regulate tumor induction and progression: the role of extracellular matrix and its degrading enzymes. Cold Spring Harbor Symposia on Quantitative Biology. 70, 343-356 (2005).
- Heinrich, E. L., et al. The inflammatory tumor microenvironment, epithelial mesenchymal transition and lung carcinogenesis. Cancer Microenvironment. 5 (1), 5-18 (2012).
- Gonda, T. A., Tu, S., Wang, T. C. Chronic inflammation, the tumor microenvironment and carcinogenesis. Cell Cycle. 8 (13), 2005-2013 (2009).
- Berdyshev, E. V., Schmid, P. C., Krebsbach, R. J., Schmid, H. H. Activation of PAF receptors results in enhanced synthesis of 2-arachidonoylglycerol (2-AG) in immune cells. The FASEB Journal. 15 (12), 2171-2178 (2001).
- Rola-Pleszczynski, M., Stankova, J. Cytokine gene regulation by PGE(2), LTB(4) and PAF. Mediators of Inflammation. 1 (2), 5-8 (1992).
- Bussolati, B., et al. PAF produced by human breast cancer cells promotes migration and proliferation of tumor cells and neo-angiogenesis. The American Journal of Pathology. 157 (5), 1713-1725 (2000).
- Chakravarty, V., et al. Prolonged exposure to platelet activating factor transforms breast epithelial cells. Frontiers in Genetics. 12, 634938(2021).
- Chen, J., et al. Platelet-activating factor receptor-mediated PI3K/AKT activation contributes to the malignant development of esophageal squamous cell carcinoma. Oncogene. 34 (40), 5114-5127 (2015).
- Chen, J., et al. Feed-forward reciprocal activation of PAFR and STAT3 regulates epithelial-mesenchymal transition in non-small cell lung cancer. Cancer Research. 75 (19), 4198-4210 (2015).
- Vidi, P. A., Bissell, M. J., Lelievre, S. A. Three-dimensional culture of human breast epithelial cells: the how and the why. Methods in Molecular Biology. 945, 193-219 (2013).
- Debnath, J., Muthuswamy, S. K., Brugge, J. S. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Barcellos-Hoff, M. H., Aggeler, J., Ram, T. G., Bissell, M. J. Functional differentiation and alveolar morphogenesis of primary mammary cultures on reconstituted basement membrane. Development. 105 (2), 223-235 (1989).
- Petersen, O. W., Ronnov-Jessen, L., Howlett, A. R., Bissell, M. J. Interaction with basement membrane serves to rapidly distinguish growth and differentiation pattern of normal and malignant human breast epithelial cells. Proceedings of the National Academy of Sciences. 89 (19), 9064-9068 (1992).
- Shaw, K. R., Wrobel, C. N., Brugge, J. S. Use of three-dimensional basement membrane cultures to model oncogene-induced changes in mammary epithelial morphogenesis. Journal of Mammary Gland Biology and Neoplasia. 9 (4), 297-310 (2004).
- Weaver, V. M., Fischer, A. H., Peterson, O. W., Bissell, M. J. The importance of the microenvironment in breast cancer progression: recapitulation of mammary tumorigenesis using a unique human mammary epithelial cell model and a three-dimensional culture assay. Biochemistry and Cell Biology. 74 (6), 833-851 (1996).
- Lee, G. Y., Kenny, P. A., Lee, E. H., Bissell, M. J. Three-dimensional culture models of normal and malignant breast epithelial cells. Nature Methods. 4 (4), 359-365 (2007).
- Bodakuntla, S., Libi, A. V., Sural, S., Trivedi, P., Lahiri, M. N-nitroso-N-ethylurea activates DNA damage surveillance pathways and induces transformation in mammalian cells. BMC Cancer. 14, 287(2014).
- Banerjee, A., et al. A rhodamine derivative as a "lock" and SCN− as a "key": visible light excitable SCN− sensing in living cells. Chemical Communications. 49 (25), 2527-2529 (2013).
- Ren, G., et al. Reduced basal nitric oxide production induces precancerous mammary lesions via ERBB2 and TGFbeta. Scientific Reports. 9 (1), 6688(2019).
- Anandi, L., Chakravarty, V., Ashiq, K. A., Bodakuntla, S., Lahiri, M. DNA-dependent protein kinase plays a central role in transformation of breast epithelial cells following alkylation damage. Journal of Cell Science. 130 (21), 3749-3763 (2017).
- Sonnenberg, A., et al. Integrin alpha 6/beta 4 complex is located in hemidesmosomes, suggesting a major role in epidermal cell-basement membrane adhesion. Journal of Cell Biology. 113 (4), 907-917 (1991).
- Pignatelli, M., Cardillo, M. R., Hanby, A., Stamp, G. W. Integrins and their accessory adhesion molecules in mammary carcinomas: loss of polarization in poorly differentiated tumors. Human Pathology. 23 (10), 1159-1166 (1992).
- Natali, P. G., et al. Changes in expression of alpha 6/beta 4 integrin heterodimer in primary and metastatic breast cancer. British Journal of Cancer. 66 (2), 318-322 (1992).
- Davis, T. L., Cress, A. E., Dalkin, B. L., Nagle, R. B. Unique expression pattern of the alpha6beta4 integrin and laminin-5 in human prostate carcinoma. Prostate. 46 (3), 240-248 (2001).
- Liu, J. S., Farlow, J. T., Paulson, A. K., Labarge, M. A., Gartner, Z. J. Programmed cell-to-cell variability in Ras activity triggers emergent behaviors during mammary epithelial morphogenesis. Cell Reports. 2 (5), 1461-1470 (2012).
- Liu, C. Y., Lin, H. H., Tang, M. J., Wang, Y. K. Vimentin contributes to epithelial-mesenchymal transition cancer cell mechanics by mediating cytoskeletal organization and focal adhesion maturation. Oncotarget. 6 (18), 15966-15983 (2015).
- Kapalczynska, M., et al. 2D and 3D cell cultures-a comparison of different types of cancer cell cultures. Archives of Medical Science. 14 (4), 910-919 (2018).
- Schulze, A., Lehmann, K., Jefferies, H. B., McMahon, M., Downward, J. Analysis of the transcriptional program induced by Raf in epithelial cells. Genes & Development. 15 (8), 981-994 (2001).
- McCarthy, S. A., Samuels, M. L., Pritchard, C. A., Abraham, J. A., McMahon, M. Rapid induction of heparin-binding epidermal growth factor/diphtheria toxin receptor expression by Raf and Ras oncogenes. Genes & Development. 9 (16), 1953-1964 (1995).
- Willis, A., Jung, E. J., Wakefield, T., Chen, X. Mutant p53 exerts a dominant negative effect by preventing wild-type p53 from binding to the promoter of its target genes. Oncogene. 23 (13), 2330-2338 (2004).
- Haupt, S., Raghu, D., Haupt, Y. Mutant p53 drives cancer by subverting multiple tumor suppression pathways. Frontiers in Oncology. 6, 12(2016).
- Langhans, S. A. Three-dimensional in vitro cell culture models in drug discovery and drug repositioning. Frontiers in Pharmacology. 9, 6(2018).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. ASSAY and Drug Development Technologies. 12 (4), 207-218 (2014).
- Lv, D., Hu, Z., Lu, L., Lu, H., Xu, X. Three-dimensional cell culture: A powerful tool in tumor research and drug discovery. Oncology Letters. 14 (6), 6999-7010 (2017).
- Jensen, C., Teng, Y. Is it time to start transitioning from 2D to 3D cell culture. Frontiers in Molecular Biosciences. 7, 33(2020).
- Saraiva, D. P., Matias, A. T., Braga, S., Jacinto, A., Cabral, M. G. Establishment of a 3D co-culture with MDA-MB-231 breast cancer cell line and patient-derived immune cells for application in the development of immunotherapies. Frontiers in Oncology. 10, 1543(2020).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon