Method Article
Génération, criblage à haut débit et biobanque de sphéroïdes cardiaques dérivés de cellules souches pluripotentes d’origine humaine
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un ensemble de protocoles pour la génération et la cryoconservation de sphéroïdes cardiaques (CS) à partir de cardiomyocytes dérivés de cellules souches pluripotentes induites par l’homme cultivés dans un format multidimensionnel à haut débit est présenté. Ce modèle tridimensionnel fonctionne comme une plate-forme robuste pour la modélisation des maladies, les criblages à haut débit et conserve sa fonctionnalité après cryoconservation.
Résumé
Les cardiomyocytes dérivés de cellules souches pluripotentes induites par l’homme (hiPSC-CM) sont d’une importance primordiale pour la modélisation et la thérapeutique des maladies cardiaques humaines. Nous avons récemment publié une stratégie rentable pour l’expansion massive des CM-HISP en deux dimensions (2D). Deux limitations majeures sont l’immaturité cellulaire et le manque d’arrangement tridimensionnel (3D) et d’évolutivité dans les plates-formes de criblage à haut débit (HTS). Pour surmonter ces limitations, les cardiomyocytes expansés forment une source cellulaire idéale pour la génération de cultures cellulaires cardiaques 3D et de techniques d’ingénierie tissulaire. Ce dernier présente un grand potentiel dans le domaine cardiovasculaire, fournissant un HTS plus avancé et physiologiquement pertinent. Ici, nous décrivons un flux de travail compatible HTS avec une évolutivité facile pour la génération, la maintenance et l’analyse optique des sphéroïdes cardiaques (CS) dans un format de 96 puits. Ces petits CS sont essentiels pour combler les lacunes présentes dans les modèles actuels de maladies in vitro et/ou la génération de plateformes d’ingénierie tissulaire 3D. Les CS présentent une morphologie, une taille et une composition cellulaire très structurées. De plus, les hiPSC-CM cultivés en tant que CS présentent une maturation accrue et plusieurs caractéristiques fonctionnelles du cœur humain, telles que la manipulation spontanée du calcium et l’activité contractile. En automatisant l’ensemble du flux de travail, de la génération de CS à l’analyse fonctionnelle, nous augmentons la reproductibilité intra et inter-lots, comme le démontrent l’imagerie à haut débit (HT) et l’analyse de la manipulation du calcium. Le protocole décrit permet de modéliser les maladies cardiaques et d’évaluer les effets médicamenteux / thérapeutiques au niveau de la cellule unique dans un environnement cellulaire 3D complexe dans un flux de travail HTS entièrement automatisé. En outre, l’étude décrit une procédure simple pour la préservation à long terme et la biobanque de sphéroïdes entiers, offrant ainsi aux chercheurs la possibilité de créer un stockage de tissus fonctionnels de nouvelle génération. Le HTS, combiné au stockage à long terme, contribuera de manière substantielle à la recherche translationnelle dans un large éventail de domaines, notamment la découverte et les essais de médicaments, la médecine régénérative et le développement de thérapies personnalisées.
Introduction
La découverte de cellules souches pluripotentes induites par l’homme (CSPhi) a offert des possibilités sans précédent d’étudier le développement humain et la maladie au niveau cellulaire. Au cours de la dernière décennie, à l’aide de leçons de développement, divers protocoles ont été établis pour assurer la différenciation efficace des CSPhi en cardiomyocytes (CM)1,2,3,4. Les cardiomyocytes dérivés des hiPSC (hiPSC-CM) peuvent servir de ressource pour modéliser les maladies cardiovasculaires génétiquement héréditaires (MCV), tester la sécurité cardiaque pour de nouveaux médicaments et les stratégies de régénération cardiaque 5,6,7,8. Malgré la différenciation cardiaque dirigée des CSPhi, le nombre indéfini de CM reste un défi dans le domaine cardiaque, car les HIPSC-CM matures ne sont généralement pas prolifératifs et les cellules humaines primaires ne sont pas disponibles en grande quantité.
Récemment, nous avons décrit que l’activation concomitante de la signalisation Wnt avec une culture à faible densité cellulaire entraînait une réponse proliférative massive (jusqu’à 250 fois) des hiPSC-CMs 9,10. Cette stratégie rentable pour l’expansion massive des CM-CSPh via le passage en série dans le format flacon de culture facilite la normalisation et le contrôle de la qualité d’un grand nombre de CM-CSP fonctionnels. De plus, pour répondre à la demande de grands lots de CM-CSPhi de divers donateurs, la biobanque de CM-HIPSC a été décrite10. Cependant, les monocouches de cardiomyocytes ensemencées dans ces boîtes de culture standard ne sont pas représentatives de la structure 3D complexe présente dans le cœur. De plus, l’immaturité des hiPSC-CM est restée un obstacle, ne permettant donc pas d’imiter le phénotype biologique et physiologique de l’environnement cardiovasculaire in vivo.
De nouveaux modèles in vitro 3D ont été développés où les hiPSC-CM montrent un comportement physiologique plus étroit tel que l’auto-organisation 11,12, le remodelage de la matrice extracellulaire (ECM) 13, la maturation améliorée 14,15,16 et la contraction synchronisée17,18,19 . Les modèles 3D ont été utilisés pour la découverte de médicaments, les tests de cardiotoxicité des médicaments, la modélisation de maladies, les thérapies régénératives et même les premiers essais cliniques 20,21,22,23,24. L’un des modèles les plus utilisés est le tissu cardiaque modifié à base de fibrine (EHT), qui présente un arrangement tissulaire et une contractilité cardiaque13,17,25. Auparavant, nous avons montré que les EHT générés à partir de hiPSC-CM étendus présentaient une contractilité comparable à celles des hiPSC-CM non développés, démontrant une fonctionnalité cellulaire sans compromis après l’expansion9. Néanmoins, même si la génération d’EHT à partir de CM-CSP a été bien établie, d’autres développements sont attendus concernant la mise en place d’une plate-forme d’évaluation de l’HT. Ici, la génération rapide d’un grand nombre de sphéroïdes cardiaques (CS) auto-agrégés au format 96 puits permet une amélioration des conditions 3D à des fins de criblage à haut débit (HTS).
Dans l’ensemble, l’avantage des CS en tant que culture cellulaire 3D est leur reproductibilité et leur évolutivité élevées. En particulier, les CS combinés à la manipulation robotisée des échantillons peuvent normaliser et automatiser la culture CS, le traitement de la toxicomanie et l’analyse à haut contenu20. Ici, nous décrivons des protocoles optimisés pour générer des CS de haute pureté et de haute qualité, qui peuvent être efficacement cryoconservés et criblés pour la fonction cardiaque en effectuant des mesures transitoires Ca2+ à l’aide d’un système optique d’acquisition et d’analyse du calcium. Ce modèle fournit un outil simple mais puissant pour effectuer des criblages à haut débit sur des centaines à des milliers de sphéroïdes17,18.
Protocole
REMARQUE : Les CM-CSPhi utilisés dans cette étude ont été générés conformément aux protocoles de culture et de différenciation des CSPh26,27 décrits précédemment. En option, les hiPSC-CM peuvent être étendus et cryoconservés comme récemment publié avant de commencer le protocole CS (section 4)10.
1. Préparation des milieux de culture cellulaire, des solutions et des aliquotes
- Préparer le milieu basal
- Équilibrer la pénicilline-streptomycine et le milieu (RPMI 1640) à température ambiante (RT) et s’assurer qu’il a complètement décongelé. Mélanger 500 mL du milieu et 5 mL de stylo/streptocoque. Conserver à 4 °C jusqu’à 8 semaines; équilibrer à 37 °C avant utilisation.
- Préparer RPMI + B27
- Équilibrez le supplément de B27 et le milieu basal à RT. Assurez-vous de décongeler complètement le supplément. Mélanger 490 mL du milieu basal et 10 mL du supplément 50x B27. Conserver à 4 °C jusqu’à 2 semaines; équilibrer à 37 °C avant utilisation.
- Préparer le support de replacage hiPSC-CM
- Ajouter un inhibiteur de protéine kinase (ROCK) associé à Rho et une bobine enroulée contenant un inhibiteur de protéine kinase (ROCK) (concentration finale de 2 μM) et un remplacement sérique knockout (KSR) à 10% au milieu RPMI + B27. Ajouter l’inhibiteur de ROCK directement au milieu RM au besoin. Ne pas stocker les milieux de culture une fois supplémentés.
- Préparer le milieu de décongélation CM
- Ajouter une concentration de 1:100 de supplément de survie cellulaire (par exemple, Revitacell) et 20% de KSR dans le milieu RPMI + B27 et équilibrer à 37 °C avant utilisation.
- Préparer le supplément de maturation
- La formule28 du milieu de maturation décrite précédemment se compose de: 3 mM de glucose, 10 mM de L-lactate, 5 mg / mL de vitamine B12, 0,82 mM de biotine, 5 mM de monohydrate de créatine, 2 mM de taurine, 2 mM de L-carnitine, 0,5 mM d’acide ascorbique, 1x NEAA, 0,5% (p / v) d’albumax, 1 x B27 et 1% de KOSR. Pour préparer un flacon plein (500 mL) de supplément de maturation, retirer 65 mL d’un flacon de DMEM sans glucose et compléter avec 2,7 g de glucose, 5,6 g de L-lactate, 0,025 mg de vitamine B12, 1 mg de biotine, 3,73 g de monohydrate de créatine, 1,25 g de taurine, 1,975 g de L-carnitine, 0,7125 g d’acide ascorbique, 50 mL de NEAA, 12,5 g d’albumax et 5 mL de pénicilline-streptomycine.
- Filtrer à travers une unité filtrante jetable stérile avec une membrane de polyéthersulfone (PES) à pores de 0,22 μm.
- Aliquote en 45 mL (pour préparer 500 mL de milieu de maturation) ou 4,5 mL (pour préparer 50 mL de milieu de maturation). Conserver à 20 °C jusqu’à 6 mois.
- Préparer les milieux de maturation
- Équilibrez le supplément de B27, le SR knockout, la pénicilline-streptomycine, le supplément de maturation28 et le milieu DMEM sans glucose à TA. Assurez-vous de décongeler complètement le supplément. Mélanger 435 mL du milieu DMEM sans glucose avec 10 mL du supplément 50x B27, 5 mL de pénicilline-streptomycine, 5 mL de SR knockout et 45 mL de supplément de maturation. Conserver à 4 °C jusqu’à 2 semaines; équilibrer à 37 °C avant utilisation.
- Préparer le milieu brillant fluor
- Équilibrer pénicilline-streptomycine et milieu DMEM fluorobrite à TA. Assurez-vous que le supplément est complètement décongelé. Mélanger 500 mL du milieu fluorbrite DMEM avec 5 mL de pénicilline-streptomycine. Conserver à 4 °C jusqu’à 1 mois; équilibrer à 37 °C avant utilisation.
- Préparer la solution de détergent non ionique
- Mélanger 20 % p/v de poudre de détergent non ionique (p. ex., F-127) avec du PBS. Filtrer à l’aide d’un filtre de 0,22 μm et conserver à 4 °C jusqu’à 6 mois; équilibrer à TA avant utilisation.
- Préparer un milieu de colorant calcique
- Mélanger la solution de détergent non ionique (concentration finale de 0,04 % v/v) et 0,1x du colorant calcique (p. ex. Cal520 AM) dans un milieu fluoré brillant. Dans un tube conique de 50 mL, ajouter 10 μL de Cal520 et 20 μL de la solution détergente non ionique. Mélanger jusqu’à dissolution complète. Gardez la solution dans l’obscurité avant de l’ajouter aux cellules.
2. Préparation des tampons
- Préparer le tampon de perméabilisation et de blocage : Ce tampon contient 10 ml de PBS, 5 % en poids/v de BSA et 0,3 % v/v de Triton-X-100.
- Préparez le tampon de cytométrie en flux : Ce tampon contient 50 ml de PBS, 1 % en poids/v de BSA et 0,3 % v/v de Triton-X-100.
- Tampon de lavage par cytométrie en flux : Ce tampon contient 50 mL de PBS et 1 % en poids/v de BSA.
- Tampon de lavage sphéroïde (SWB): Ce tampon contient 1 ml de Triton-X-100, 2 ml de FDS à 10% (p / v dans DPBS) et 2 g de BSA dans 1 L de PBS.
REMARQUE: SWB peut être conservé à 4 ° C jusqu’à 2 semaines. - Préparer la solution d’enrobage (SE) : Pour préparer 100 mL de la solution d’enrobage, mélanger 50 mL de glycérol avec 9,09 mL dedH2O, 1 mL de tampon Tris (1 M, pH 8,0) et 200 μL d’EDTA (0,5 M). Ajouter 22,7 g de fructose et mélanger à TA à l’obscurité jusqu’à dissolution. Lorsque l’on est clair, ajouter 22,2 g de fructose et mélanger jusqu’à dissolution. Ajouter ensuite 15 g d’urée et mélanger jusqu’à dissolution (conserver à 4 °C dans l’obscurité).
- Préparez le tampon PBT (PBS avec Tween-20). Ce tampon contient du PBS/Tween-20 (0,1 % v/v). Pour 1 L de PBS, ajouter 1 mL de Tween-20.
3. Préparation de petites molécules
- Reconstituer la poudre de thiazovine (inhibiteur de ROCK) dans des aliquotes de 10 mM de 50 μL dans du DMSO et conserver à -20 °C jusqu’à 6 mois. Protéger de la lumière.
- Préparer 2,5 mM d’aliquotes de 10 μL chacune de Cal-520 AM dans du DMSO et les conserver à -20 °C jusqu’à 6 mois. Protéger de la lumière.
4. Génération de sphéroïdes cardiaques
REMARQUE : Pour de plus grandes quantités de CS, ensemencez jusqu’à 1 million de CM dans une plaque de fixation ultra-basse à 6 puits avec 2 mL de média de replacage hiPSC-CM. Cette étude a utilisé un minimum de 2 500 (2,5k CS) jusqu’à 20 000 (20k CS) hiPSC-CMs par puits d’une plaque de 96 puits.
- Pour une plaque de 96 puits, préparer une culture cellulaire contenant au moins 2 x 106 cardiomyocytes dérivés de cellules souches pluripotentes induites par l’homme (hiPSC-CM)10.
- Lorsque les CM-HIPSC cultivés atteignent la confluence, ajouter 0,1 mL/cm2 de solution de décollement cardiaque stérile (p. ex. tryple) à chaque puits. Incuber la plaque à 37 °C pendant 15 min.
- À l’aide d’une pipette de 5 mL, dissocier mécaniquement les cellules en rinçant avec 2 mL de milieu basal chaud pour obtenir une suspension unicellulaire. Confirmer le détachement avec un microscope à champ clair (grossissement 4x); Les cellules auront un aspect blanc et auront une forme ronde.
- Transférer la suspension cellulaire dans un tube conique de 15 ml et centrifuger pendant 3 min à 300 x g.
- Aspirer le surnageant et remettre les cellules en suspension dans 1 mL de média de replacage hiPSC-CM.
- À l’aide d’un embout de pipette de 1 000 μL, dissocier mécaniquement la pastille de cellule. La solution semble homogène après trois ou quatre mélanges. Comptez les cellules. Transférer la quantité appropriée de cellules dans 100 μL du milieu de replacage à chaque puits à fond rond à fond rond à très faible attachement.
- Placer la plaque de CS sur un agitateur orbital à 70 tr/min dans l’incubateur pendant 24 h. Réglez les conditions de l’incubateur à 37 °C, 5 % de CO 2, 21 % O2 et 90 % d’humidité.
- Aspirer 50 μL de milieu de chaque puits et ajouter 100 μL de milieu RPMI + B27 par puits pendant les 48 premières h.
REMARQUE : Conservez toujours 50 μL du milieu dans la plaque de 96 puits pour éviter l’aspiration accidentelle et la rupture du sphéroïde. - Aspirer 100 μL du milieu de chaque puits et ajouter 100 μL du milieu de maturation par puits. Maintenir les cellules dans le milieu de maturation et rafraîchir le milieu tous les 2-3 jours.
5. Cryoconservation des CS
REMARQUE: Les CS peuvent être cryoconservés pour un stockage à long terme. La cryoconservation peut être effectuée à partir du jour 3 après la génération des CS. Les CS peuvent être cryoconservés directement dans les puits d’une plaque de 96 puits ou sous forme de suspension de CS dans des cryovials.
- Pré-refroidir la plaque en la plaçant sur de la glace pendant 10 min.
- Centrifuger la plaque sphéroïde pendant 3 min à 70 x g.
- Retirer le surnageant jusqu’à ce qu’il reste 50 μL et ajouter 200 μL de milieu de congélation hiPSC glacé par puits.
REMARQUE : Gardez la suspension CS sur la glace pendant toute la durée de la procédure. Dans le cas d’une plaque à 6 puits avec sphéroïdes, congeler un puits dans un milieu de congélation cryovial de 500 μL. - Scellez la plaque avec un film d’étanchéité de plaque.
REMARQUE: La plaque de 96 puits doit être stockée dans une boîte en polystyrène ou, lorsqu’elle n’est pas disponible, un moule en silicone peut être fabriqué comme décrit à l’étape 5.5.1. - Pour assurer un échange de chaleur uniforme entre la plaque de puits et le congélateur, placez soigneusement la plaque dans une boîte en polystyrène ou dans un moule en silicone.
- Pour préparer le moule en silicone: Mélanger vigoureusement deux composants du kit en élastomère de silicium dans un rapport de 10: 1. Dé-bulle la solution à l’aide d’une pompe à vide pendant 15-20 min. Ensuite, couler la solution à l’intérieur de la partie inférieure de la plaque de puits et débuller à l’aide d’une pompe à vide pendant 10 min. Placer le moule dans une étuve et durcir à 60 °C pendant 8 h pour obtenir un élastomère semi-flexible qui est décollé de la plaque.
- Congeler la plaque à -80 °C pendant au moins 4 h dans la boîte en polystyrène ou dans le moule en silicone préparé.
- Transférer la plaque dans un réservoir d’azote liquide ou un congélateur à -150 °C pour un stockage à long terme.
6. Décongélation des sphéroïdes cardiaques
REMARQUE : Ne décongelez pas plus d’une plaque à la fois pour assurer un processus de décongélation rapide.
- Préparer 20 mL de milieu basal préchauffé à 37 °C dans un tube conique de 50 mL.
- Recueillir la plaque cellulaire avec les CS de l’azote liquide et la placer dans l’incubateur pendant 15 min. Réglez les conditions de l’incubateur à 37 °C, 5 % de CO 2, 21 % O2 et 90 % d’humidité.
- Retirer le surnageant et les restes de granulés cellulaires, et remettre en suspension chaque puits dans un milieu basal chaud. Utilisez 200 μL de milieu par puits.
- Centrifuger pendant 3 min à 70 x g.
- Répétez les étapes 6.3 et 6.4.
- Retirer le surnageant jusqu’à ce qu’il reste la pastille de la cellule et ajouter 200 μL de milieu de décongélation CM dans chaque puits.
- Placer la plaque de CS sur un agitateur orbital à 70 tr/min dans un incubateur pendant 24 h. Réglez les conditions de l’incubateur à 37 °C, 5 % de CO 2, 21 % O2 et 90 % d’humidité.
- Aspirer 50 μL du milieu de chaque puits et ajouter 100 μL de milieu RPMI + B27 par puits pendant les 48 premières h.
- Aspirer 100 μL du milieu de chaque puits et ajouter 100 μL de milieu de maturation par puits. Maintenir les cellules dans le milieu de maturation et rafraîchir le milieu tous les 2-3 jours.
7. Évaluation des transitoires Ca2+ intracellulaires
NOTE: Les CS sont en culture pour un total de 3 semaines; 2 semaines avant la congélation et 1 semaine après la décongélation. Les contrôles « frais » sont appariés selon l’âge.
- Après 1 semaine de culture, les CS décongelés sont optimaux pour l’imagerie optique de manipulation du calcium. Utilisez un colorant calcique (par exemple, Cal520AM) pour évaluer l’absorption et la libération de Ca2+ par les cellules.
- Traitez-les avec 100 μL de milieu colorant calcique par puits et incuber pendant 60 min dans l’incubateur. Réglez les conditions de l’incubateur à 37 °C, 5 % de CO 2, 21 % O2 et 90 % d’humidité.
REMARQUE: Cal520AM est sensible à la lumière. Effectuez toutes les procédures de chargement et les expériences dans l’obscurité. - Préparer le système d’acquisition et d’analyse du calcium.
- Alimentez le microscope, en vous assurant que l’option de contrôle environnemental est activée.
- Ajustez les dimensions de l’ouverture de la caméra et du cadrage pour réduire la zone d’arrière-plan.
REMARQUE: Ici, le microscope Leica Thunder DMi8 a été utilisé; D’autres systèmes de microscope sont également applicables jusqu’à ce qu’ils permettent un taux d’échantillonnage supérieur à 30 images / seconde (fps).
- Enregistrez une vidéo avec un flux constant de 2 à 10 pics en 10 s et balayez la plaque de 96 puits, en vous déplaçant d’abord vers la gauche, puis vers le bas en zigzag pour couvrir toute la plaque. Mesurer le signal de calcium à l’aide d’un laser de 488 nm; Réglez le contraste sur un fond noir avec un signal vert vif pendant la libération de calcium.
- Après avoir acquis les transitoires Ca2+ , analysez les données avec le logiciel d’analyse des traces de fluorescence (par exemple, CyteSeer, Vala Sciences) selon les instructions du fabricant.
8. Analyses de cytométrie en flux de sphéroïdes cardiaques dissociés
REMARQUE : Dans cette étude, la cytométrie en flux a été utilisée pour déterminer la viabilité des CS avant et après le processus de décongélation.
- Recueillir les CS dans un tube conique de 15 mL à l’aide d’une pipette de 5 mL pour éviter les dommages aux sphéroïdes et centrifuger pendant 3 min à 70 x g. Aspirer le surnageant et ajouter 1 mL de PBS.
- Centrifuger pendant 3 min à 200 x g. Aspirer le surnageant et dissocier les CS en ajoutant 1 mL de solution de décollement cardiaque (p. ex. tryple). Incuber le tube à 37 °C pendant 15 min.
- À l’aide d’une pipette de 5 mL, dissocier mécaniquement les cellules en rinçant avec 2 mL de milieu basal jusqu’à ce que des cellules individuelles puissent être vues lorsqu’elles sont observées au microscope.
- Centrifuger pendant 3 min à 200 x g.
- Aspirer le surnageant et fixer les CM avec 200 μL de solution de paraformaldéhyde (PFA) à 4% dans 1x PBS. Incuber pendant 10 min à TA.
- Centrifuger pendant 3 min à 200 x g. Aspirer le surnageant et ajouter 1 mL de PBS.
REMARQUE: Point de pause: Les hiPSC-CM fixes peuvent être conservés à 4 ° C jusqu’à 4 semaines. - Transférer la suspension cellulaire dans un tube FACS et centrifuger pendant 3 min à 200 x g. Aspirer le surnageant et remettre en suspension 1 x 105 cellules dans 50 μL du tampon de perméabilisation.
- Incuber les cellules pendant 30 min à 4 °C.
- Pour l’analyse par cytométrie en flux par immunofluorescence, effectuez les étapes 8.9.1 à 8.9.4.
- Resuspendre les cellules dans le tampon de cytométrie en flux (50 μL) contenant l’anticorps α-actinine à une dilution de 1:300. Dans un autre tube FACS, remettre en suspension 1 x 105 cellules dans le tampon de cytométrie en flux (50 μL) avec le contrôle d’isotype respectif (p. ex., souris FITC IgM, isotype κ [dilution 1:200]). De même, remettre en suspension 1 x 105 cellules dans 50 μL de tampon de cytométrie de flux pour un contrôle négatif.
- Incuber les cellules pendant 30 min à 4 °C.
- Ajouter 2,5 mL de tampon de cytométrie en flux et centrifuger les cellules à 200 x g pendant 3 min à 4 °C; Jetez le surnageant et répétez cette étape de lavage deux fois.
- Resuspendre les cellules dans 50 μL de tampon de cytométrie de flux avec l’anticorps secondaire chèvre-anti-souris (dilution 1:300).
REMARQUE: Placez le tube dans l’obscurité car la solution d’anticorps secondaires est sensible à la lumière.
- Pour vérifier la viabilité avec de l’iodure de propidium (IP), ajouter 150 μL d’IP par échantillon (1:1 000) et incuber pendant 15 min.
REMARQUE: Placez le tube dans l’obscurité car la solution PI est sensible à la lumière. - Ajustez les portes selon la stratégie de contrôle standard illustrée à la figure supplémentaire 1 et analysez les cellules à l’aide d’un cytomètre en flux.
9. Coloration par immunofluorescence de sphéroïdes 3D entiers
REMARQUE: Ce protocole est basé sur le protocole d’imagerie 3D haute résolution d’organoïdes entiers lors du marquage immunofluorescent, qui a déjà été publié29 et ajusté pour les sphéroïdes cardiaques. Pendant la procédure, tous les embouts de pipettes et les tubes coniques peuvent être recouverts de 1% en poids / v BSA-PBS afin d’empêcher les sphéroïdes de coller aux plastiques. Pour enduire les matériaux, tremper dans le 1% BSA-PBS. Veillez à ne pas endommager les sphéroïdes en utilisant la pipette de 5 mL, en évitant les perturbations mécaniques.
- Recueillir les CS dans un tube revêtu de 15 mL avec une pipette de 5 mL. Les sphéroïdes sont visibles à l’œil. Recueillir ~20-50 sphéroïdes par combinaison d’anticorps. Centrifuger pendant 3 min à 70 x g et aspirer le surnageant.
- Remettez soigneusement les sphéroïdes en suspension dans 1 mL de solution glacée de paraformaldéhyde (PFA) à 4 % dans 1x PBS à l’aide d’un embout enduit de 1 mL.
- Fixer à 4 °C pendant 45 min. Après 20 min, remettre doucement les sphéroïdes en suspension à l’aide d’un embout enduit de 1 mL. Cela uniformise la fixation parmi tous les sphéroïdes.
- Ajouter 10 ml de PBS glacé dans le tube et mélanger doucement en retournant le tube. Incuber pendant 10 min à 4 °C et faire tourner vers le bas à 70 x g pendant 3 min.
REMARQUE: À partir de cette étape, le revêtement des pointes et des tubes coniques n’est généralement pas nécessaire car les CS ne collent pas à la pointe après la fixation. - Bloquer les CS en remettant en suspension la pastille dans un SWB glacé (200 μL de SWB par puits) et transférer les sphéroïdes dans une plaque de culture en suspension de 24 puits.
REMARQUE: Les CS d’un gros granulé peuvent être divisés sur plusieurs puits pour effectuer différentes colorations. Utilisez ~20-50 CS par combinaison d’anticorps. - Incuber à 4 °C pendant au moins 15 min.
- Ajouter 200 μL de SWB dans un puits vide pour servir de puits de référence.
REMARQUE: Pour la coloration par immunofluorescence, des plaques à 48 ou 96 puits peuvent également être utilisées pour réduire l’utilisation d’anticorps. Cependant, les résultats de coloration et de lavage peuvent être réduits en raison du plus petit volume par puits. - Laisser les sphéroïdes se déposer au fond de la plaque, en laissant la plaque inclinée à un angle de 45° pendant 5 min.
- Retirer le SWB en laissant les CS dans 200 μL du SWB (utiliser le puits de référence pour estimer le volume minimal de 200 μL).
- Ajouter 200 μL du SWB avec les anticorps primaires 2x concentrés (p. ex. ɑ-actinine [1:200] et troponine T [1:200]) et incuber pendant la nuit à 4 °C tout en berçant/secouant (40 rpm sur un agitateur horizontal).
- Le lendemain, ajouter 1 mL de SWB à chaque puits.
- Laisser les sphéroïdes se déposer au fond de la plaque en laissant la plaque à un angle de 45° pendant 5 min.
- Retirez le SWB en laissant 200 μL dans la plaque. Ajouter 1 mL de SWB et laver pendant 2 h en berçant/secouant lentement.
- Répétez les étapes 9.12 et 9.13 deux fois de plus.
- Laisser les CS se déposer au fond de la plaque en laissant la plaque inclinée à 45° pendant 5 min. Retirez l’ESB, laissant 200 μL dans chaque puits
- Ajouter 200 μL du SWB avec des anticorps secondaires, des anticorps conjugués et des colorants 2x concentrés (p. ex. DAPI [1 μg/mL], souris-AF488 [1:500], lapin-AF568 [1:500]), et incuber toute la nuit à 4 °C dans l’obscurité, tout en berçant ou en secouant lentement.
- Le lendemain, répétez les étapes 9.12 et 9.13 deux fois de plus.
- Transférer délicatement les CS dans un tube de 1,5 mL et faire tourner vers le bas à 70 x g pendant 3 min.
- Enlevez la plus grande partie possible de l’ESS en le pipetant sans perturber les CS.
- Ajouter la solution d’enrobage (ES; au moins 50 μL, à TA) à l’aide d’une pointe de 200 μL avec l’extrémité coupée et remettre en suspension doucement pour éviter la formation de bulles et incuber à TA pendant 20 min.
- En attendant, créez un récipient carré sur une lame de verre avec du vernis à ongles ou du mastic silicone.
- Coupez l’extrémité d’une pointe de 200 μL et transférez les CS dans ES au milieu du récipient carré.
- Placez un couvercle carré sur le dessus. Pour réduire les bulles d’air, placez d’abord un côté de la lamelle de couverture, puis abaissez lentement la lamelle de couverture d’un côté à l’autre jusqu’à ce qu’il n’y ait plus d’air emprisonné sous la surface, puis relâchez la lamelle de couverture.
- Poussez doucement sur tous les bords du couvercle pour le sceller sur le vernis à ongles ou le mastic silicone.
- Laissez la diapositive pendant la nuit à RT. Le lendemain, la diapositive est prête pour l’imagerie.
REMARQUE: L’effacement optique par le SE peut provoquer un rétrécissement mineur des tissus. Cependant, cela ne peut pas affecter la morphologie globale des CS. La procédure de coloration peut être interrompue ici en stockant les lames à 4 °C (pendant au moins 1 semaine) ou à -20 °C (pendant au moins 6 mois).
Résultats
Le protocole illustré à la figure 1A décrit la génération de CS à partir de hiPSC-CM précédemment développés. Les CS acquièrent une structure 3D au jour 1 après l’ensemencement dans des plaques à fond rond à très faible fixation et peuvent être cultivés jusqu’à 6 semaines (Figure 1B). Comme évalué par coloration par immunofluorescence, la majorité des cellules des CS âgés de 3 semaines exprimaient des protéines sarcomériques telles que la α-actinine et la troponine T et présentaient une organisation sarcomère régulière (Figure 1C). Pour la quantification des cellules positives à la α-actinine, une analyse par cytométrie de flux a été effectuée. Conformément aux résultats de l’immunofluorescence, les données de cytométrie en flux ont démontré des niveaux élevés comparables de α-actinine dans les CS du jour 0 (76,9% ± 16,6%) et des CS âgés de 3 semaines (71,1% ± 22,7%) (Figure 1D), indiquant une composition cellulaire constante et très pure pendant la culture. Il y avait une expression accrue des gènes cardiaques pour les jonctions (GJA1, JPH2 et PKP2), les desmosomes (DES) et les mitochondries (ATP5A) chez les sphéroïdes dérivés de hiPSC-CM (jour 42) par rapport aux hiPSC-CM cultivés en 2D pendant 90 jours (Figure 1E). L’expression de ces gènes est une caractéristique de l’interaction cellule-cellule et de la maturation30.
Par la suite, les propriétés fonctionnelles des CS, à savoir le taux de battement et la manipulation du Ca2+, ont été évaluées à différents moments (figure 2). Les paramètres transitoires du calcium, comme le temps de montée, le temps de pointe, le temps de désintégration et la durée transitoire du calcium (CTD90), ont été évalués comme l’indiquent les figures 2A, B. Le pourcentage de CS battus est similaire au cours des 3 premières semaines post-génération, mais a considérablement diminué au cours de la semaine 6 (Wk6) CS (Figure 2C). Le taux de battement a été significativement réduit à Wk3 par rapport à Wk1 et, comme le pourcentage de CS battus, a chuté de façon spectaculaire à Wk6 (Figure 2D). Au Wk6, une détérioration du CS a été observée, ce qui peut expliquer la baisse à la fois du taux de battement et du nombre de CS battants. La mesure des paramètres transitoires calciques a indiqué une valeur de pointe significativement plus élevée à Wk2 (Figure 2E), tandis que le temps de montée, le temps de désintégration et CTD90 ont été significativement augmentés à Wk3 par rapport à Wk1 (Figure 2F-H ). Pris ensemble, ces résultats montrent que les sphéroïdes dérivés de hiPSC-CM sont fonctionnellement optimaux vers les semaines 2 et 3 post-génération.
La figure 3 montre l’effet de la taille des sphéroïdes sur la vitesse de battement et la manipulation du calcium. Les CS ont été générés par l’ensemencement de 2,5 x 10 4, 5 x 10 4, 10 x 10 4 et 20 x 10 4 hiPSC-CM dans un puits d’une plaque de 96 puits, pour un total de 24 CS/puits par condition (figure 3A). Comme prévu, la taille des sphéroïdes a augmenté à mesure que le nombre de cellules utilisées augmentait, allant de 178 ± 36 μm à 351 ± 65 μm (figure 3A, panneau de droite). Les transitoires de Ca2+ ont été mesurés dans des CS âgés de 3 semaines aux quatre densités de semis différentes (figure 3B). Les mesures des CS battants ont indiqué que seulement environ 50 % des CS de plus petite taille (2,5K et 5K-CS) battaient, tandis que le pourcentage de CS de plus grande taille battant (10K et 20K-CS) était significativement plus élevé (environ 85 %) (figure 3C). Un taux de battement similaire (environ 28 bpm) a été montré par les 5K-, 10K-et 20K-CS, ce qui était significativement plus élevé que les 2,5K-CS (Figure 3D). Les valeurs maximales des images de calcium étaient similaires dans toutes les conditions testées (figure 3E), mais le temps de montée (figure 3F), le temps de désintégration (figure 3G) et CTD90 (figure 3H) ont été significativement augmentés dans les CS de plus grande taille (10K et 20K-CS) par rapport aux plus petits (2,5K et 5K-CS). Pris ensemble, ces résultats montrent que les sphéroïdes dérivés de hiPSC-CM sont optimaux pour le criblage de la manipulation du calcium lorsqu’une densité de semis comprise entre 10K et 20K hiPSC-CM/puits est utilisée.
Ensuite, nous avons évalué l’impact de la cryoconservation sur la viabilité et la fonction du CS. Avant l’analyse, les CS décongelés ont été maintenus en culture pendant 1 semaine (figure 4A). Comme l’ont montré les tests de viabilité cellulaire de cytométrie en flux (figure 4B) et de calcéine-AM (figure 4C), la cryoconservation n’a pas affecté la viabilité cellulaire dans les CS. De plus, les CS décongelés ont montré des niveaux d’expression similaires des protéines sarcomériques par rapport aux CS appariés par l’âge frais (Figure 4D). Ces données indiquent que les CS peuvent être cryoconservés efficacement pour une analyse ultérieure de la fonction cardiaque et un dépistage à haut débit.
Enfin, l’activité de battement et la manipulation du Ca2+ ont été mesurées dans des CS frais et cryoconservés (Figure 5). Le pourcentage de CS battus a été mesuré à différents moments après la décongélation, respectivement, à 2, 5 et 7 jours. Alors que la plupart des CS frais ont montré une activité de battement au fil du temps, il est clair que les CS cryoconservés ont eu besoin de jusqu’à 1 semaine de culture pour récupérer leur activité de battement (Figure 5B). Il n’y avait pas de changement significatif dans le taux de battement des CS décongelés par rapport aux frais; toutefois, aucune activité spontanée de battement n’a été observée chez certains CS congelés (figure 5C). Bien que les valeurs de pointe aient été significativement réduites dans les CS congelés/décongelés par rapport aux CS frais (figure 5D), aucun changement significatif n’a été observé dans le temps de montée, le temps de désintégration et le CTD90 des CS congelés/décongelés par rapport aux CS frais (figure 5E-G). Ces données indiquent qu’après la décongélation, il est important de laisser les CS récupérer dans l’incubateur pendant au moins 1 semaine avant de mesurer l’activité de battement et Ca2+ transitoire.
Pris ensemble, ces résultats montrent que la cryoconservation des sphéroïdes dérivés de hiPSC-CM préserve la viabilité des cardiomyocytes, la structure sarcomérique et leurs caractéristiques fonctionnelles telles que l’activité de battement spontané et la manipulation du calcium. Ainsi, les sphéroïdes dérivés de hiPSC-CM représentent un modèle approprié pour récapituler avec précision l’électrophysiologie cardiaque in vitro.
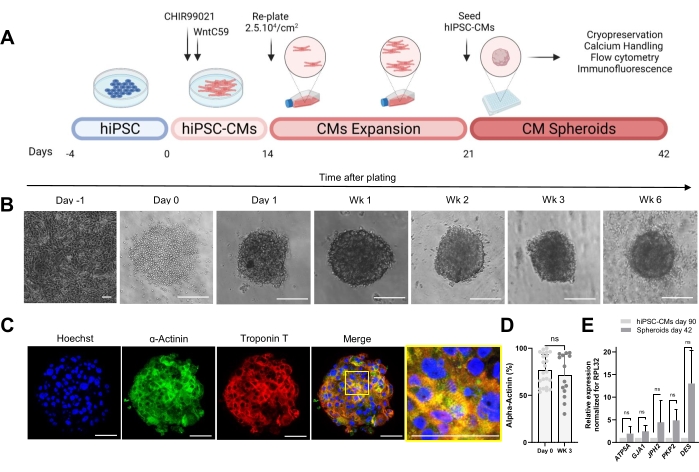
Figure 1 : Génération de sphéroïdes cardiaques. (A) Représentation schématique de la différenciation cardiaque dirigée basée sur Wnt, de l’expansion ultérieure des hiPSC-CM et de la génération de CS. Créé avec biorender.com. (B) Images en fond clair à différents points temporels de la culture CS. Barre d’échelle, 200 μm. Wk représente la semaine. (C) Images représentatives de l’immunofluorescence pour les protéines sarcomériques cardiaques α-actinine et troponine T dans les CS âgés de 3 semaines. Immunofluorescence: Hoechst (bleu), α-actinine (vert) et troponine T (rouge). Barre d’échelle, 200 μm. L’image fusionnée agrandie à droite montre l’organisation du sarcomère. Barre d’échelle, 50 μm. (D) Quantification par cytométrie en flux des cellules positives à la α-actinine avant (jour 0) et 3 semaines après la formation des CS. (n = 14-23 par condition. (E) RT-qPCR réalisée sur des hiPSC-CM cultivés pendant 90 jours (2D) et des échantillons de sphéroïdes cultivés pendant 42 jours pour établir les niveaux d’expression de différents gènes cardiaques liés aux jonctions cellulaires, aux filaments intermédiaires et aux mitochondries. (n = 1-3 lots). Les données sont représentées sous forme de moyenne ± écart-type. NS (non significatif) calculée par un test t non apparié. Veuillez cliquer ici pour voir une version agrandie de cette figure.
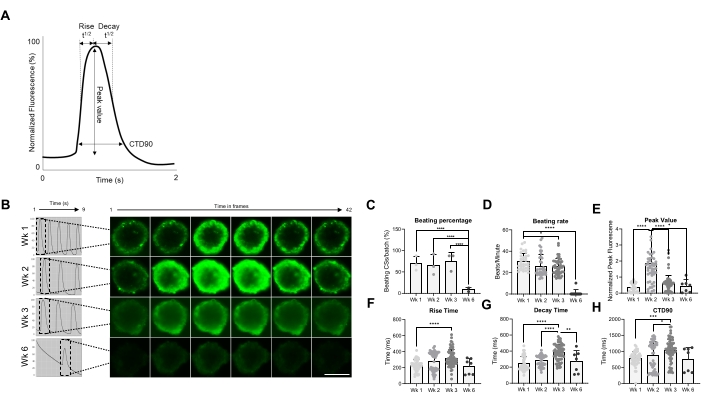
Figure 2 : Taux de battement et manipulation du calcium dans les CS à différentes semaines après la génération. (A) Exemples de paramètres transitoires calciques calculés par l’algorithme d’analyse Vala sciences de Cyteseer Software. (B) Des traces transitoires de calcium représentatives et des images accélérées des CS à différents moments (semaines) post-génération. Barre d’échelle, 200 μm. (C) La quantification de l’activité de battement spontané est exprimée en pourcentage de CS battus. (D) Taux de battement des CS pendant la culture. (E-H) Quantification des transitoires calciques montrant la valeur maximale, le temps de montée, le temps de désintégration et CTD90. Les données présentées sont des réplications moyennes ± écart-type. Réplications biologiques = trois, répétitions techniques = 38, 50, 66 et 7, respectivement. *p < 0,05, ****p < 0,001; ANOVA unidirectionnelle suivie du test de comparaisons multiples post hoc de Tukey. Abréviations; CTD = durée transitoire du calcium, semaine = semaine, CS = sphéroïdes cardiaques humains. Veuillez cliquer ici pour voir une version agrandie de cette figure.
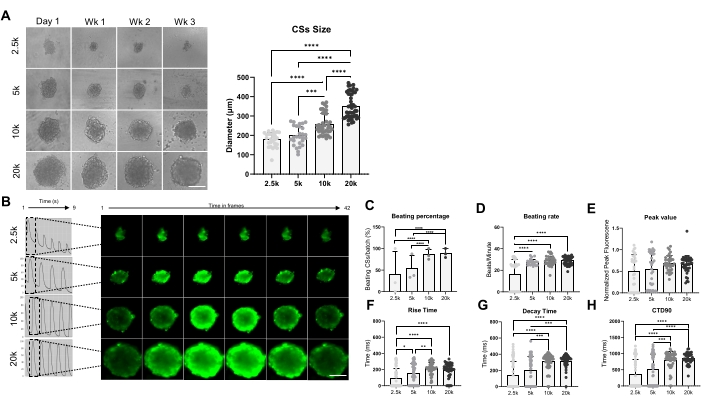
Figure 3 : Taux de battement et manipulation du calcium dans les CS générés à l’aide de différentes densités d’ensemencement cellulaire. (A) Imagerie en fond clair (à gauche) et mesures de taille (à droite) des CS générées à l’aide de différents nombres de CM-HiPSC. Barre d’échelle, 200 μm. (B) Traces transitoires de calcium représentatives et images accélérées des 2,5K-20K-CS. (C, D) Pourcentage de battement et taux de battement de 2,5K-20K-CS. (E-H) Valeur maximale, temps de montée, temps de désintégration et CTD90 dans les 2.5K-20K-CS. Les données sont moyennes ± écart-type. Réplications biologiques = trois, réplications techniques = 28-39. *p < 0,05, ****p < 0,001; ANOVA unidirectionnelle suivie du test de comparaisons multiples post hoc de Tukey. Abréviations : CTD = durée transitoire calcique, semaine = semaine, k = x 1 000 cellules, CS = sphéroïdes cardiaques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
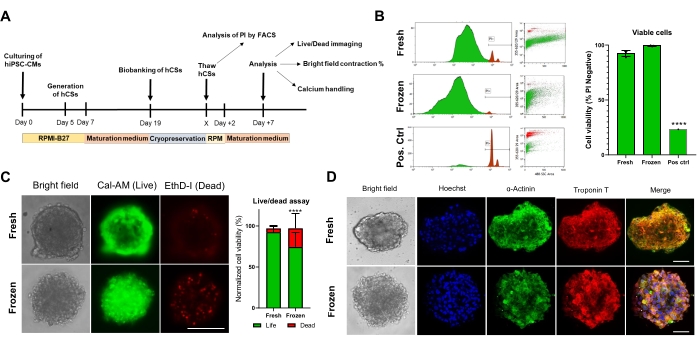
Graphique 4. Effet de la cryoconservation sur la viabilité et la structure des sphéroïdes cardiaques. (A) Représentation schématique de la génération de CS, des biobanques subséquentes et de la décongélation. (B) Test de viabilité cellulaire par cytométrie en flux dans les CS frais et cryoconservés. Comme témoin positif, un traitement avec une solution de Triton-X à 10% pendant 5 minutes a été utilisé. (n = 4 par affection). Les données sont représentées sous forme de moyenne ± écart-type. ****p < 0,001; ANOVA unidirectionnelle suivie du test de comparaisons multiples post hoc de Tukey. (C) Test de viabilité des cellules calcéine-AM dans les CS frais versus décongelés après 7 jours de culture (n = 15-17 par condition, ****p < 0,001, par test t apparié; barre d’échelle, 200 μm). (D) Coloration représentative en fond clair (à gauche) et immunofluorescence pour l’expression de la α-actinine et de la troponine T dans les CS frais et décongelés. Immunofluorescence: Hoechst (bleu), α-actinine (vert) et troponine T (rouge). Les images fusionnées à droite montrent les stries sarcomères dans les CS. Barre d’échelle, 50 μm. Abréviations : X = jour de décongélation au choix, PI = iodure de propidium, Cal-AM = calcéine-AM, EthD-I = Homodimère d’éthidium I. Veuillez cliquer ici pour voir une version agrandie de cette figure.
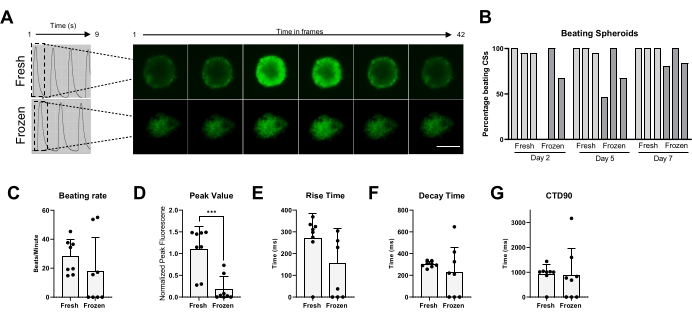
Figure 5 : Transitoires calciques dans les CS frais par rapport aux CS décongelés. (A) Traces transitoires de calcium représentatives et images accélérées des CS avant cryoconservation et 1 semaine après la décongélation. (B) Pourcentage de battement de sphéroïdes cardiaques frais et congelés/décongelés. Les barres représentent des expériences individuelles. (C) Taux de battement des sphéroïdes cardiaques frais et congelés/décongelés. (D-G) Quantification des paramètres transitoires du calcium : valeur de pointe, temps de montée, temps de désintégration et CTD90. Les données sont moyennes ± écart-type. *p < 0,05, ****p < 0,001; ANOVA unidirectionnelle suivie du test de comparaisons multiples post hoc de Tukey. Abréviations; CTD = durée transitoire calcique, CS = sphéroïdes cardiaques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Stratégies de points de contrôle représentatifs pour l’analyse de cytométrie en flux. (A) Stratégie de contrôle représentative pour les α-actinines hiPSC-CM positives dans une population pure par rapport au contrôle négatif et au contrôle isotype. Le nombre de cellules analysées positives à la α-actinine est de 25 x 105. Abréviations; SSC = diffusion latérale, PI+ = iodure de propidium positif. (B) Stratégie représentative de points de contrôle pour l’analyse de viabilité à la fois pour le témoin frais, le témoin décongelé, le témoin positif (Triton-X) et le témoin négatif (non coloré). Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La découverte de médicaments cardiaques est entravée par le recours à des modèles animaux et cellulaires non humains avec un débit et une fidélité physiologique insuffisants pour effectuer des lectures avec précision. La biologie hiPSC-CM couplée à l’instrumentation HT et aux sondes physiologiques a le potentiel de réintroduire des modèles humains dans les premières étapes de la modélisation des maladies cardiaques et de la découverte de médicaments. Nous avons développé une méthode de génération de tissu cardiaque 3D qui produit des CS fonctionnels et de haute qualité pour une plateforme optimale de modélisation des maladies cardiaques et de dépistage de médicaments. De plus, la combinaison de la technologie sphéroïde dans des systèmes de bioréacteurs 3D pour la production industrielle de véhicules électriques permet une étape nécessaire vers la traduction clinique de la thérapie à base de véhicules électriques. La méthode décrite ici repose sur plusieurs facteurs cruciaux et est une variante des protocoles existants 9,10,28,29. Ces méthodes comprennent: 1) la génération de constructions tissulaires 3D, 2) le nombre optimal de cellules et le moment avant le criblage, 3) l’amélioration de la sensibilité et de la capacité à haut débit des instruments, et 4) la possibilité de congeler les sphéroïdes avant toute analyse fonctionnelle. Contrairement aux protocoles décrits précédemment, le protocole proposé décrit la génération de jusqu’à 1 500 sphéroïdes par jour et la pertinence du HTS. L’analyse conventionnelle d’une centaine de composés sur 6 x 0,5 log doses pour 10 répétitions à l’aide de systèmes d’imagerie calcique existants à 96 puits ou de tissus cardiaques modifiés multiplexés à 24 puits nécessite environ 500 millions à 3 milliards de hiPSC-CM31,32. L’application proposée rend les dépistages cardiaques moins coûteux et moins rapides que les systèmes conventionnels puisque les plaques à 96 puits ne nécessitaient que 10 % de la densité d’ensemencement par rapport à la méthode décrite. De plus, par rapport aux protocoles précédents, tels que la méthode de la goutte suspendue, la génération de sphéroïdes par auto-agrégation dans des plaques de fixation ultra-basses permet une imagerie automatisée de haute qualité de microtissus uniques33.
Ce petit modèle 3D imite le phénotype biologique et physiologique de l’environnement cardiovasculaire in vivo . Comme démontré précédemment, les transitoires de calcium augmentent considérablement dans les constructions de tissus cardiaques 3D par rapport aux cultures cellulaires monocouches2D 34.
Ensuite, nous avons constaté que la densité de semis et le temps de culture approprié sont également des facteurs critiques pour un dépistage CS réussi. Les densités de 10K-20K hiPSC-CM par sphéroïde et le dépistage entre les semaines 2 et 3 après la génération étaient optimales, tandis que les sphéroïdes trop petits ou trop âgés présentent une manipulation perturbée du calcium (Figure 2 et Figure 3). Par conséquent, il est important de maintenir des densités de semis aussi cohérentes que possible, car la taille influence les paramètres fonctionnels. De plus, bien que cette méthode optique fournisse de bons résultats pour les cultures 3D vivantes en tant que tissu entier, il est difficile d’obtenir des données au sein de sphéroïdes plus grands au niveau (sous-)cellulaire sans recourir à des méthodes histologiques fastidieuses. Récemment, plusieurs approches ont été publiées qui utilisaient le « nettoyage optique », qui permet l’acquisition de sphéroïdes 3D entiers avec la possibilité de quantifier des marqueurs à cellule unique. Ici, nous avons adapté un protocole de 3 jours allant de la récolte CS à l’analyse d’images, optimisée pour l’imagerie 3D par microscopie confocale29 (Figure 1C et Figure 4D).
Enfin, avec l’augmentation des applications de tissus cardiaques 3D et des applications commerciales, la demande de stockage à long terme et de biobanque spécifique au patient de la part de divers donneurs augmente. La cryoconservation est une stratégie efficace pour générer des plaques HTS à partir de plusieurs lots au fil du temps. La congélation des hiPSC-CM a été décrite précédemment et n’est pas différente de celle des autres types de cellules cultivées 10,35,36. Récemment, des approches pour congeler des plaques avec des cellules 2D ont été décrites37. Ici, nous avons constaté que le kit de cryoconservation PSC est la condition la plus optimale par rapport à trois autres (données non présentées) et utilisé ce milieu pour la congélation efficace des sphéroïdes. Après cryoconservation, la viabilité reste élevée (Figure 4B,C), mais les propriétés électrophysiologiques des CS sont affectées et une période d’incubation après décongélation est nécessaire. En effet, 1 semaine après la décongélation, les CS ont montré une activité de battement spontanée et une manipulation du calcium. Cependant, il a été décrit que les hiPSC-CM fraîches et récupérées ne présentent pas toujours des propriétés moléculaires et physiologiques identiques38. Cette limitation doit être prise en compte lorsque des CM-HiPSC cryoconservés sont utilisés pour évaluer les lectures cardiaques induites par le médicament. De plus, bien que nous modulions efficacement le nombre de cellules par sphéroïde et le moment optimal de l’imagerie transitoire du calcium, les sphéroïdes cardiaques pourraient être améliorés en mélangeant des cellules cardiomyocytaires dérivées de l’hiPSC avec des cellules endothéliales, des fibroblastes, des jonctions cellule-cellule et des matrices extracellulaires, telles que le chitosane, le collagène IV, la fibronectine, la matrigel ou la laminine, imitant l’environnement cardiaque in vivo 39, 40. Dans l’ensemble, nous proposons un protocole étape par étape pour générer efficacement des CS adaptés aux applications en aval telles que la modélisation des maladies et le dépistage de médicaments HT.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous tenons à remercier VALA sciences pour le progiciel Cyteseer et l’optimisation de l’analyse 3D automatisée du calcium. Nous tenons à remercier la fondation PLN (RM) pour le soutien de la subvention. P.A.D. et F.S. sont soutenus par CUREPLaN Leducq. J.P.G.S. est soutenu par H2020-EVICARE (#725229) du Conseil Européen de la Recherche (ERC). J.W.B. est soutenu par la bourse clinique UMC Utrecht, la bourse de l’Institut de cardiologie des Pays-Bas et la bourse de jeunes talents CVON-Dosis; Fondation néerlandaise du cœur (CVON-Dosis 2014-40). N.C. est soutenu par le programme de gravitation « Materials Driven Regeneration » de l’Organisation néerlandaise pour la recherche scientifique (RegmedXB #024.003.013), et les actions Marie Skłodowska-Curie (accord de subvention RESCUE #801540). V.S.-P. est soutenu par le Fonds Alliance (UMCU, UU, TU/e). A.v.M. est soutenu par le projet BRAVE (H2020, ID:874827) financé par l’UE
matériels
| Name | Company | Catalog Number | Comments |
| 24 wells suspenion plate | Corning | 3738 | |
| 96 wells Ultra-Low Attachment Multiple Well Plate | Corning | CLS3474-24EA | |
| Albumax | Thermo Fisher Scientific | 11020021 | |
| Anti-α-Actinin (Sarcomeric) antibody | Sigma-Aldrich | A7811 | Dilution: 1:200 |
| Anti-Cardiac Troponin T antibody (ab45932) | Abcam | ab45932 | Dilution: 1:200 |
| Ascorbic acid | Sigma-Aldrich | A8960 | |
| B-27 supplement | Thermo Fisher Scientific | 17504-044 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Bovine serum albumin fraction V (BSA) | Roche | 10735086001 | |
| Cal-520, AM | Abcam | ab171868 | |
| Confocal microscope | Leica | DMi8 | |
| Confocal microscope software | Leica | Las X | |
| Conical tubes 15 mL | Greiner Bio-One | 5618-8271 | |
| Creatine monohydrate | Sigma-Aldrich | C3630 | |
| DAPI | Thermo Fisher Scientific | D3571 | Concentration: 1 µg/mL |
| DMEM no glucose | Thermo Fisher Scientific | 11966025 | |
| EDTA | Thermo Fisher Scientific | 15575020 | |
| Fructose | Sigma-Aldrich | 76050771.05 | |
| Glucose | Sigma-Aldrich | G7021 | |
| Glycerol | Boom | 76050771.05 | |
| Goat anti-mouse Alexa Fluor 488 | Invitrogen | A11029 | Dilution: 1:500 |
| Goat anti-rabbit Alexa Fluor 568 | Invitrogen | A11011 | Dilution: 1:500 |
| Horizontal shaker | IKA | 4003000 | |
| Human induced pluripotent stem cell lines | (Stanford Cardiovascular Institute (S-CVI) Biobank) | CVI-273 (control 1) | |
| Human induced pluripotent stem cell lines | Germany | 141 (control 2) 144 (control 3) | |
| Hydrochloric acid (HCl) | Ajax Firechem | 265.2.5L-PL | 10 M stock solution, corrosive |
| Isotype control, FITC mouse IgM κ isotype | BD | 556652 | |
| KnockOut Serum Replacement | Thermo Fisher Scientific | 10828 | Protect from light |
| L-carnitine | Sigma-Aldrich | C0283 | |
| Myocyte calcium and contractility system | Leica | Thunder, DMi8 | |
| Non essential amino acids (NEAA) | Thermo Fisher Scientific | 11140 | |
| Paraformaldehyde solution 4% in 1x PBS, pH 7.0–7.6 | Santa Cruz | SC281692 | Hazardous |
| PBS, pH 7.4 | Thermo Fisher Scientific | 10010023 | |
| Penicillin/streptomycin | Thermo Fisher Scientific | 15140 | |
| PES Membrane Vacuum Filter system | Corning | 431097 | |
| PI/RNase Staining Solution | Invitrogen | F10797 | Dilution: 1:1000 |
| Pluronic F-127 | Sigma-Aldrich | P2443 | |
| PSC Cryopreservation Kit | Thermo Fisher Scientific | A2644601 | |
| RevitaCell | Thermo Fisher Scientific | A2644501 | |
| RPMI 1640 medium | Thermo Fisher Scientific | 11875 | |
| Silicone Elastomer Kit | SYLGARD | 184 | |
| Sodium dodecyl sulfate solution (10%) | Sigma-Aldrich | 71736 | |
| Sodium L-Lactate | Sigma-Aldrich | 71718 | |
| Taurine | Sigma-Aldrich | T0625 | |
| Tris Fisher | Scientific | 11486631 | |
| Triton X-100 | Merck | X100-1L | Hazardous |
| Trypan blue solution, 0.4% | Thermo Fisher Scientific | 15250061 | |
| TrypLE Select Enzyme (10x) | Thermo Fisher Scientific | A1217701 | |
| Tween-20 | Sigma-Aldrich | P1379 | |
| Urea | Sigma-Aldrich | 51456 | |
| Vitamin B12 | Sigma-Aldrich | V6629 | |
| Y-27632 dihydrochloride (Rho-kinase inhibitor) | Tocris | 1254 | Protect from light |
Références
- Burridge, P. W., et al. Chemically defined and small molecule-based generation of human cardiomyocytes. Nature Methods. 11 (8), 855-860 (2014).
- Kattman, S. J., et al. Stage-specific optimization of activin/nodal and BMP signaling promotes cardiac differentiation of mouse and human pluripotent stem cell lines. Cell Stem Cell. 8 (2), 228-240 (2011).
- Lian, X., et al. Robust cardiomyocyte differentiation from human pluripotent stem cells via temporal modulation of canonical Wnt signaling. Proceedings of the National Academy of Sciences. 109 (27), 1848-1857 (2012).
- Paige, S. L., et al. Endogenous Wnt/beta-catenin signaling is required for cardiac differentiation in human embryonic stem cells. PLoS One. 5 (6), 11134 (2010).
- Gintant, G., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes in preclinical cancer drug cardiotoxicity testing: A scientific statement from the American Heart Association. Circulation Research. 125 (10), 75-92 (2019).
- Ahmed, R. E., et al. A brief review of current maturation methods for human induced pluripotent stem cells-derived cardiomyocytes. Frontiers in Cell and Developmental Biology. 19 (8), 178 (2020).
- Liu, C., et al. Generating 3D human cardiac constructs from pluripotent stem cells. EBioMedicine. 76, 103813 (2022).
- Musunuru, K., et al. Induced pluripotent stem cells for cardiovascular disease modeling and precision medicine: A scientific statement from the American Heart Association. Circulation: Genomic and Precision Medicine. 11 (1), 000043 (2018).
- Buikema, J. W., et al. Wnt activation and reduced cell-cell contact synergistically induce massive expansion of functional human iPSC-derived cardiomyocytes. Cell Stem Cell. 27 (1), 50-63 (2020).
- Maas, R. G. C., et al. Massive expansion and cryopreservation of functional human induced pluripotent stem cell-derived cardiomyocytes. Cell STAR Protocols. 2 (1), 100334 (2021).
- Tremblay, C., et al. A new construction technique for tissue-engineered heart valves using the self-assembly method. Tissue Engineering Part C: Methods. 20 (11), 905-915 (2014).
- Lewis-Israeli, Y. R., et al. Self-assembling human heart organoids for the modeling of cardiac development and congenital heart disease. Nature Communications. 12 (1), 5142 (2021).
- Goldfracht, I., et al. Engineered heart tissue models from hiPSC-derived cardiomyocytes and cardiac ECM for disease modeling and drug testing applications. Acta Biomaterialia. 1 (92), 145-159 (2019).
- Fleischer, S., et al. Comprehensive human stem cell differentiation in a 2D and 3D mode to cardiomyocytes for long-term cultivation and multiparametric monitoring on a multimodal microelectrode array setup. Biosensors and Bioelectronics. 126, 624-631 (2019).
- Branco, M. A., et al. Transcriptomic analysis of 3D cardiac differentiation of human induced pluripotent stem cells reveals faster cardiomyocyte maturation compared to 2D culture. Science Reports. 9 (1), 9229 (2019).
- Ergir, E., et al. Generation and maturation of human iPSC-derived cardiac organoids in long term culture. bioRxiv. , (2022).
- Lemoine, M. D., et al. Human iPSC-derived cardiomyocytes cultured in 3D engineered heart tissue show physiological upstroke velocity and sodium current density. Scienctific Reports. 7 (1), 5464 (2017).
- Kofron, C. M., et al. A predictive in vitro risk assessment platform for pro-arrhythmic toxicity using human 3D cardiac microtissues. Science Reports. 11 (1), 10228 (2021).
- Giacomelli, E., et al. Human-iPSC-derived cardiac stromal cells enhance maturation in 3D cardiac microtissues and reveal non-cardiomyocyte contributions to heart disease. Cell Stem Cell. 26 (6), 862-879 (2020).
- Richards, D. J., et al. Human cardiac organoids for the modelling of myocardial infarction and drug cardiotoxicity. Nature Biomedical Engineering. 4 (4), 446-462 (2020).
- Tenreiro, M. F., et al. Next generation of heart regenerative therapies: progress and promise of cardiac tissue engineering. npj Regenerative Medicine. 6 (1), 30 (2021).
- Hansen, A., et al. Development of a drug screening platform based on engineered heart tissue. Circulation Research. 107 (1), 35-44 (2010).
- McDermott-Roe, C., et al. Investigation of a dilated cardiomyopathy-associated variant in BAG3 using genome-edited iPSC-derived cardiomyocytes. Journal of Clinical Investigation Insight. 4 (22), 128799 (2019).
- National Library of Medicine (U.S.). Safety and efficacy of induced pluripotent stem cell-derived engineered human myocardium as biological ventricular assist tissue in terminal heart failure. National Library of Medicine. , (2020).
- Ronaldson-Bouchard, K., et al. Advanced maturation of human cardiac tissue grown from pluripotent stem cells. Nature. 556 (7700), 239-243 (2018).
- Oh, J. G., et al. Generation of ventricular-like HiPSC-derived cardiomyocytes and high-quality cell preparations for calcium handling characterization. Journal of Visualized Experiments. 155, 60135 (2020).
- Lian, X., et al. Directed cardiomyocyte differentiation from human pluripotent stem cells by modulating Wnt/β-catenin signaling under fully defined conditions. Nature Protocols. 8 (1), 162-175 (2013).
- Feyen, D. A. M., et al. Metabolic maturation media improve physiological function of human iPSC-derived cardiomyocytes. Cell Reports. 32 (3), 107925 (2020).
- van Ineveld, R. L., et al. Single-cell resolution three-dimensional imaging of intact organoids. Journal of Visualized Experiments. (160), e60709 (2020).
- Guo, Y., Pu, W. T. Cardiomyocyte maturation: New phase in development. Circulation Research. 126 (8), 1086-1106 (2020).
- Ding, B., et al. Three-dimensional renal organoids from whole kidney cells: Generation, optimization, and potential application in nephrotoxicology in vitro. Cell Transplantation. 29, 963689719897066 (2020).
- Denning, C., et al. Cardiomyocytes from human pluripotent stem cells: From laboratory curiosity to industrial biomedical platform. Biochimica Biophysica Acta. 1863, 1728-1748 (2016).
- Amaral, R. L. F., et al. Comparative analysis of 3D bladder tumor spheroids obtained by forced floating and hanging drop methods for drug screening. Frontiers in Physiology. 8, 605 (2017).
- Daily, N. J., et al. Improving cardiac action potential measurements: 2D and 3D cell culture. Journal of Bioengineering and Biomedical Science. 5 (3), 168 (2015).
- Preininger, M. K., et al. Cryopreservation of human pluripotent stem cell-derived cardiomyocytes: Strategies, challenges, and future directions. Advances in Experimental Medicine and Biology. 951, 123-135 (2016).
- Kim, Y. Y., et al. Cryopreservation of human embryonic stem cells derived-cardiomyocytes induced by BMP2 in serum-free condition. Reproductive Science. 18 (3), 252-360 (2011).
- Daily, M. I., et al. Cryopreservation of primary cultures of mammalian somatic cells in 96-well plates benefits from control of ice nucleation. Cryobiology. 93, 62-69 (2020).
- Zhang, J. Z., et al. Effects of cryopreservation on human induced pluripotent stem cell-derived cardiomyocytes for assessing drug safety response profiles. Stem Cell Reports. 16 (1), 168-181 (2021).
- Yeh, H. -. Y., et al. The calcium-dependent regulation of spheroid formation and cardiomyogenic differentiation for MSCs on chitosan membranes. Biomaterials. 33 (35), 8943-8954 (2012).
- Scalise, M., et al. From spheroids to organoids: The next generation of model systems of human cardiac regeneration in a dish. International Journal of Molecular Sciences. 22 (24), 13180 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon