Method Article
Segmentation basée sur l’apprentissage profond des tomogrammes cryoélectroniques
Dans cet article
Résumé
Il s’agit d’une méthode d’entraînement d’un U-Net multi-coupes pour la segmentation multi-classe de tomogrammes cryo-électroniques en utilisant une partie d’un tomogramme comme entrée d’apprentissage. Nous décrivons comment déduire ce réseau à d’autres tomogrammes et comment extraire des segmentations pour des analyses plus approfondies, telles que la moyenne des sous-tomogrammes et le traçage filamentaire.
Résumé
La cryotomographie électronique (cryo-ET) permet aux chercheurs d’imager les cellules dans leur état natif et hydraté à la plus haute résolution actuellement possible. La technique présente toutefois plusieurs limites qui rendent l’analyse des données qu’elle génère longue et difficile. La segmentation manuelle d’un seul tomogramme peut prendre de quelques heures à plusieurs jours, mais un microscope peut facilement générer 50 tomogrammes ou plus par jour. Les programmes actuels de segmentation de l’apprentissage profond pour les cryo-ET existent, mais se limitent à segmenter une structure à la fois. Ici, les réseaux neuronaux convolutifs U-Net multi-coupes sont entraînés et appliqués pour segmenter automatiquement plusieurs structures simultanément dans des cryo-tomogrammes. Avec un prétraitement approprié, ces réseaux peuvent être déduits de manière robuste à de nombreux tomogrammes sans avoir besoin de former des réseaux individuels pour chaque tomogramme. Ce flux de travail améliore considérablement la vitesse à laquelle les cryo-tomogrammes électroniques peuvent être analysés en réduisant le temps de segmentation à moins de 30 minutes dans la plupart des cas. En outre, les segmentations peuvent être utilisées pour améliorer la précision du traçage filamentaire dans un contexte cellulaire et pour extraire rapidement les coordonnées pour la moyenne des sous-tomogrammes.
Introduction
Les développements matériels et logiciels de la dernière décennie ont entraîné une « révolution de résolution » pour la cryo-microscopie électronique (cryo-EM)1,2. Avec des détecteurs 3 meilleurs et plus rapides, des logiciels pour automatiser la collecte de données4,5 et des avancées en matière d’amplification de signal telles que les plaques de phase6, la collecte de grandes quantités de données cryo-EM à haute résolution est relativement simple.
Cryo-ET offre un aperçu sans précédent de l’ultrastructure cellulaire dans un état natif et hydraté 7,8,9,10. La principale limitation est l’épaisseur de l’échantillon, mais avec l’adoption de méthodes telles que le broyage par faisceau d’ions focalisés (FIB), où des échantillons cellulaires et tissulaires épais sont amincis pour la tomographie11, l’horizon de ce qui peut être imagé avec cryo-ET est en constante expansion. Les microscopes les plus récents sont capables de produire bien plus de 50 tomogrammes par jour, et ce taux ne devrait augmenter qu’en raison du développement de systèmes de collecte rapide de données12,13. L’analyse des grandes quantités de données produites par cryo-ET reste un goulot d’étranglement pour cette modalité d’imagerie.
L’analyse quantitative de l’information tomographique nécessite qu’elle soit d’abord annotée. Traditionnellement, cela nécessite une segmentation manuelle par un expert, ce qui prend du temps; Selon la complexité moléculaire contenue dans le cryo-tomogramme, cela peut prendre des heures ou des jours d’attention particulière. Les réseaux de neurones artificiels sont une solution attrayante à ce problème car ils peuvent être formés pour effectuer la majeure partie du travail de segmentation en une fraction du temps. Les réseaux de neurones convolutifs (CNN) sont particulièrement adaptés aux tâches de vision par ordinateur14 et ont récemment été adaptés pour l’analyse des cryotomogrammesélectroniques 15,16,17.
Les CNN traditionnels nécessitent plusieurs milliers d’échantillons d’entraînement annotés, ce qui n’est pas souvent possible pour les tâches d’analyse d’images biologiques. Par conséquent, l’architecture U-Net a excellé dans cet espace18 parce qu’elle s’appuie sur l’augmentation des données pour former avec succès le réseau, minimisant ainsi la dépendance à l’égard de grands ensembles d’entraînement. Par exemple, une architecture U-Net peut être entraînée avec seulement quelques tranches d’un seul tomogramme (quatre ou cinq tranches) et déduite de manière robuste à d’autres tomogrammes sans rééducation. Ce protocole fournit un guide étape par étape pour la formation des architectures de réseaux neuronaux U-Net à segmenter les cryotomographies électroniques dans Dragonfly 2022.119.
Dragonfly est un logiciel développé commercialement utilisé pour la segmentation et l’analyse d’images 3D par des modèles d’apprentissage profond, et il est disponible gratuitement pour un usage académique (certaines restrictions géographiques s’appliquent). Il dispose d’une interface graphique avancée qui permet à un non-expert de tirer pleinement parti des pouvoirs de l’apprentissage profond pour la segmentation sémantique et le débruitage d’images. Ce protocole montre comment prétraiter et annoter des tomogrammes cryo-électroniques dans Dragonfly pour former des réseaux de neurones artificiels, qui peuvent ensuite être déduits pour segmenter rapidement de grands ensembles de données. Il traite et montre brièvement comment utiliser des données segmentées pour une analyse plus approfondie telle que le traçage filamentaire et l’extraction de coordonnées pour la moyenne des sous-tomogrammes.
Protocole
REMARQUE : Dragonfly 2022.1 nécessite une station de travail hautes performances. Les recommandations système sont incluses dans le tableau des matériaux avec le matériel du poste de travail utilisé pour ce protocole. Tous les tomogrammes utilisés dans ce protocole sont classés 4x d’une taille de pixel de 3,3 à 13,2 ang/pix. Les échantillons utilisés dans les résultats représentatifs ont été obtenus d’une entreprise (voir le tableau des matériaux) qui suit les lignes directrices sur les soins aux animaux qui s’harmonisent avec les normes éthiques de cette institution. Le tomogramme utilisé dans ce protocole et le multi-retour sur investissement généré en tant qu’entrée d’apprentissage ont été inclus en tant qu’ensemble de données groupées dans le fichier supplémentaire 1 (qui peut être trouvé à https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct) afin que l’utilisateur puisse suivre les mêmes données s’il le souhaite. Dragonfly héberge également une base de données en libre accès appelée Infinite Toolbox où les utilisateurs peuvent partager des réseaux entraînés.
1. Configuration
- Modification de l’espace de travail par défaut :
- Pour modifier l’espace de travail afin de refléter celui utilisé dans ce protocole, sur le côté gauche du panneau principal, faites défiler jusqu’à la section Propriétés de la vue de la scène et désélectionnez Afficher les légendes. Faites défiler jusqu’à la section Mise en page et sélectionnez la vue Scène unique et Quatre vues égales .
- Pour mettre à jour l’unité par défaut, accédez à Fichier | préférences. Dans la fenêtre qui s’ouvre, modifiez l’unité Par défaut de millimètres en nanomètres.
- Keybinds par défaut utiles :
- Appuyez sur Echap pour afficher le réticule dans les vues 2D et autoriser la rotation du volume 3D dans la vue 3D. Appuyez sur X pour masquer le réticule dans les vues 2D et autoriser la traduction 2D et la translation de volume 3D en vue 3D.
- Passez la souris sur le réticule pour voir les petites flèches qui peuvent être cliquées et glissées pour modifier l’angle du plan de visualisation dans les autres vues 2D.
- Appuyez sur Z pour entrer l’état de zoom dans les deux vues, ce qui permet aux utilisateurs de cliquer et de faire glisser n’importe où pour effectuer un zoom avant et arrière.
- Double-cliquez sur une vue dans la scène Quatre vues pour faire la mise au point uniquement de cette vue ; Double-cliquez à nouveau pour revenir aux quatre vues.
- Enregistrez régulièrement la progression en exportant tout ce qui se trouve dans l’onglet Propriétés en tant qu’objet ORS pour faciliter l’importation. Sélectionnez tous les objets de la liste et cliquez avec le bouton droit de la souris sur Exporter | En tant qu’objet ORS. Nommez le fichier et enregistrez. Vous pouvez également accéder à Fichier | Enregistrer la session. Pour utiliser la fonction d’enregistrement automatique du logiciel, activez-la via Fichier | Préférences | Enregistrement automatique.
2. Importation d’images
- Pour l’importation d’images, accédez à Fichier | Importer des fichiers image. Cliquez sur Ajouter, accédez au fichier image, puis cliquez sur Ouvrir | Suivant | Terminer.
REMARQUE: Le logiciel ne reconnaît pas les fichiers .rec. Tous les tomogrammes doivent avoir le suffixe .mrc. Si vous utilisez les données fournies, accédez plutôt à Fichier | Importer des objets. Accédez au fichier Training.ORSObject et cliquez sur Ouvrir, puis sur OK.
3. Prétraitement (figure 1.1)
- Créez une échelle d’intensité personnalisée (utilisée pour calibrer les intensités d’image entre les jeux de données). Aller à Utilitaires | Responsable d’unité Dimension. En bas à gauche, cliquez sur + pour créer une nouvelle unité dimensionnelle.
- Choisissez une caractéristique de haute intensité (clair) et de faible intensité (foncé) qui figure dans tous les tomogrammes d’intérêt. Donnez un nom et une abréviation à l’unité (par exemple, pour cette échelle, définissez les perles fiduciaires sur 0,0 Intensité standard et l’arrière-plan sur 100,0). Enregistrez l’unité de dimension personnalisée.
REMARQUE : Une échelle d’intensité personnalisée est une échelle arbitraire qui est créée et appliquée aux données pour s’assurer que toutes les données sont sur la même échelle d’intensité bien qu’elles soient collectées à des moments différents ou sur un équipement différent. Choisissez des caractéristiques claires et sombres qui représentent le mieux la plage dans laquelle se situe le signal. S’il n’y a pas de fiduciaires dans les données, choisissez simplement la caractéristique la plus sombre qui va être segmentée (la région la plus sombre de la protéine, par exemple). - Pour calibrer les images sur l’échelle d’intensité personnalisée, cliquez avec le bouton droit sur le jeu de données dans la colonne Propriétés sur le côté droit de l’écran et sélectionnez Calibrer l’échelle d’intensité. Dans l’onglet Principal sur le côté gauche de l’écran, faites défiler jusqu’à la section Sonde . À l’aide de l’outil de sonde circulaire avec un diamètre approprié, cliquez à quelques endroits dans la région d’arrière-plan du tomogramme et notez le nombre moyen dans la colonne Intensité brute ; Répétez l’opération pour les marqueurs fiduciaires, puis cliquez sur Calibrer. Si nécessaire, réglez le contraste pour rendre les structures à nouveau visibles à l’aide de l’outil Zone de la section Nivellement de la fenêtre de l’onglet Principal.
- Filtrage d’image :
REMARQUE: Le filtrage d’image peut réduire le bruit et augmenter le signal. Ce protocole utilise trois filtres intégrés au logiciel car ils fonctionnent le mieux pour ces données, mais de nombreux filtres sont disponibles. Une fois défini sur un protocole de filtrage d’image pour les données d’intérêt, il sera nécessaire d’appliquer exactement le même protocole à tous les tomogrammes avant la segmentation.- Dans l’onglet principal sur le côté gauche, faites défiler jusqu’au panneau Traitement d’image. Cliquez sur Avancé et attendez qu’une nouvelle fenêtre s’ouvre. Dans le panneau Propriétés , sélectionnez le jeu de données à filtrer et rendez-le visible en cliquant sur l’icône en forme d’œil à gauche du jeu de données.
- Dans le panneau Opérations , utilisez le menu déroulant pour sélectionner Égalisation de l’histogramme (sous la section Contraste ) pour la première opération. Sélectionnez Ajouter une opération | Gaussiens (sous la section Lissage ). Définissez la dimension du noyau sur 3D.
- Ajouter une troisième opération; puis sélectionnez Unsharp (sous la section Accentuation ). Laissez la sortie pour celui-ci. Appliquez à toutes les tranches et laissez le filtrage s’exécuter, puis fermez la fenêtre Traitement d’image pour revenir à l’interface principale.
4. Créer des données d’entraînement (Figure 1.2)
- Identifiez la zone d’entraînement en masquant d’abord le jeu de données non filtré en cliquant sur l’icône en forme d’œil à gauche de celui-ci dans le panneau Propriétés des données . Ensuite, affichez le jeu de données nouvellement filtré (qui sera automatiquement nommé DataSet-HistEq-Gauss-Unsharp). À l’aide du jeu de données filtré, identifiez une sous-région du tomogramme qui contient toutes les caractéristiques d’intérêt.
- Pour créer une boîte autour de la région d’intérêt, sur le côté gauche, dans l’onglet principal , faites défiler jusqu’à la catégorie Formes et sélectionnez Créer une boîte. Dans le panneau Quatre vues , utilisez les différents plans 2D pour guider/faire glisser les bords de la boîte afin de ne placer que la région d’intérêt dans toutes les dimensions. Dans la liste des données, sélectionnez la zone Zone et modifiez la couleur de la bordure pour faciliter l’affichage en cliquant sur le carré gris à côté du symbole de l’œil.
REMARQUE: La plus petite taille de patch pour un U-Net 2D est de 32 x 32 pixels; 400 x 400 x 50 pixels est une taille de boîte raisonnable pour commencer. - Pour créer un multi-ROI, sélectionnez l’onglet Segmentation | Nouveau et cochez Créer en tant que Multi-ROI. Assurez-vous que le nombre de classes correspond au nombre de fonctionnalités d’intérêt + une classe d’arrière-plan. Nommez les données d’entraînement multi-ROI et assurez-vous que la géométrie correspond au jeu de données avant de cliquer sur OK.
- Segmentation des données d’entraînement
- Faites défiler les données jusqu’à ce que les limites de la zone soient dans la zone. Sélectionnez le Multi-ROI dans le menu des propriétés à droite. Double-cliquez sur le premier nom de classe vide dans le multi-ROI pour le nommer.
- Peignez avec le pinceau 2D. Dans l’onglet de segmentation sur la gauche, faites défiler jusqu’à Outils 2D et sélectionnez un pinceau circulaire. Ensuite, sélectionnez OTSU adaptatif gaussien ou local dans le menu déroulant. Pour peindre, maintenez la touche Ctrl gauche enfoncée et cliquez. Pour effacer, maintenez la touche Maj gauche enfoncée et cliquez.
REMARQUE: Le pinceau reflétera la couleur de la classe actuellement sélectionnée. - Répétez l’étape précédente pour chaque classe d’objets dans le multi-ROI. Assurez-vous que toutes les structures de la région encadrée sont entièrement segmentées, sinon elles seront considérées comme arrière-plan par le réseau.
- Lorsque toutes les structures ont été étiquetées, cliquez avec le bouton droit sur la classe Background dans le Multi-ROI et sélectionnez Ajouter tous les voxels non étiquetés à la classe.
- Créez un nouveau retour sur investissement à classe unique nommé Mask. Assurez-vous que la géométrie est définie sur le jeu de données filtré, puis cliquez sur Appliquer. Dans l’onglet des propriétés à droite, cliquez avec le bouton droit sur la case et sélectionnez Ajouter au retour sur investissement. Ajoutez-le au retour sur investissement du masque.
- Pour couper les données d’entraînement à l’aide du masque, dans l’onglet Propriétés, sélectionnez à la fois le multi-retour sur investissement des données d’entraînement et le retour sur investissement du masque en maintenant la touche Ctrl enfoncée et en cliquant sur chacune. Ensuite, cliquez sur Intersection sous la liste des propriétés de données dans la section intitulée Opérations booléennes. Nommez le nouveau jeu de données Entrée d’apprentissage ajustée et assurez-vous que la géométrie correspond au jeu de données filtré avant de cliquer sur OK.
5. Utilisation de l’assistant de segmentation pour l’apprentissage itératif (Figure 1.3)
- Importez les données d’apprentissage dans l’assistant de segmentation en cliquant d’abord avec le bouton droit sur le jeu de données filtré dans l’onglet Propriétés , puis en sélectionnant l’option Assistant de segmentation . Lorsqu’une nouvelle fenêtre s’ouvre, recherchez l’onglet de saisie sur le côté droit. Cliquez sur Importer des images à partir d’un multi-retour sur investissement et sélectionnez l’entrée d’apprentissage rognée.
- (Facultatif) Créez un cadre de rétroaction visuelle pour suivre les progrès de la formation en temps réel.
- Sélectionnez un cadre parmi les données qui n’est pas segmenté et cliquez sur + pour l’ajouter en tant que nouveau cadre. Double-cliquez sur l’étiquette mixte à droite du cadre et remplacez-la par Surveillance.
- Pour générer un nouveau modèle de réseau neuronal, sur le côté droit de l’onglet Modèles , cliquez sur le bouton + pour générer un nouveau modèle. Sélectionnez U-Net dans la liste, puis pour Dimension d’entrée, sélectionnez 2,5D et 5 tranches, puis cliquez sur Générer.
- Pour entraîner le réseau, cliquez sur Train en bas à droite de la fenêtre SegWiz .
REMARQUE: La formation peut être arrêtée tôt sans perdre de progression. - Pour utiliser le réseau entraîné pour segmenter de nouvelles trames, lorsque la formation U-Net est terminée, créez une nouvelle trame et cliquez sur Prédire (en bas à droite). Cliquez ensuite sur la flèche vers le haut en haut à droite de l’image prévue pour transférer la segmentation à l’image réelle.
- Pour corriger la prédiction, cliquez sur deux classes en maintenant la touche Ctrl enfoncée pour modifier les pixels segmentés de l’une à l’autre. Sélectionnez les deux classes et peignez avec le pinceau pour peindre uniquement les pixels appartenant à l’une ou l’autre classe. Corrigez la segmentation dans au moins cinq nouvelles images.
REMARQUE : Peindre avec l’outil pinceau alors que les deux classes sont sélectionnées signifie qu’au lieu d’effacer Maj-clic, comme c’est normalement le cas, il convertira les pixels de la première classe en seconde. Ctrl-clic accomplira l’inverse. - Pour l’entraînement itératif, cliquez à nouveau sur le bouton Train et autorisez le réseau à s’entraîner davantage pendant 30 à 40 autres époques, après quoi arrêtez la formation et répétez les étapes 4.5 et 4.6 pour un autre cycle d’entraînement.
REMARQUE : De cette façon, un modèle peut être entraîné et amélioré de manière itérative à l’aide d’un seul jeu de données. - Pour publier le réseau, lorsque vous êtes satisfait de ses performances, quittez l’Assistant Segmentation. Dans la boîte de dialogue qui apparaît automatiquement et vous demande quels modèles publier (enregistrer), sélectionnez le réseau réussi, nommez-le, puis publiez-le pour rendre le réseau disponible pour une utilisation en dehors de l’assistant de segmentation.
6. Appliquer le réseau (Figure 1.4)
- Pour appliquer d’abord au tomogramme d’entraînement, sélectionnez le jeu de données filtré dans le panneau Propriétés . Dans le panneau Segmentation sur la gauche, faites défiler jusqu’à la section Segment avec IA . Assurez-vous que le jeu de données approprié est sélectionné, choisissez le modèle qui vient d’être publié dans le menu déroulant, puis cliquez sur Segment | Toutes les tranches. Vous pouvez également sélectionner Aperçu pour afficher un aperçu à une tranche de la segmentation.
- Pour appliquer à un jeu de données d’inférence, importez le nouveau tomogramme. Prétraiter selon l’étape 3 (Figure 1.1). Dans le panneau Segmentation, accédez à la section Segment avec IA. Assurez-vous que le tomogramme nouvellement filtré est le jeu de données sélectionné, choisissez le modèle précédemment formé, puis cliquez sur Segment | Toutes les tranches.
7. Manipulation et nettoyage de la segmentation
- Nettoyez rapidement le bruit en choisissant d’abord l’une des classes qui a segmenté le bruit et la caractéristique d’intérêt. Cliquez avec le bouton droit de la souris | Îlots de processus | Supprimer par Voxel Count | Sélectionnez une taille de voxel. Commencez petit (~200) et augmentez progressivement le nombre pour éliminer la majeure partie du bruit.
- Pour la correction de segmentation, cliquez sur deux classes tout en maintenant la touche Ctrl enfoncée pour afficher uniquement les pixels appartenant à ces classes. Ctrl-clic + glisser avec les outils de segmentation pour changer les pixels de la deuxième classe à la première et Maj-clic + glisser pour accomplir le contraire. Continuez ainsi pour corriger rapidement les pixels mal étiquetés.
- Séparer les composants connectés.
- Choisissez une classe. Cliquez avec le bouton droit sur une classe dans Multi-ROI | Séparez les composants connectés pour créer une nouvelle classe pour chaque composant qui n’est pas connecté à un autre composant de la même classe. Utilisez les boutons sous le Multi-ROI pour fusionner facilement les classes.
- Exportez le retour sur investissement au format binaire/TIFF.
- Choisissez une classe dans le Multi-ROI, puis cliquez avec le bouton droit de la souris et extrayez la classe en tant que ROI. Dans le panneau des propriétés ci-dessus, sélectionnez le nouveau retour sur investissement, cliquez avec le bouton droit de la souris | Exportation | ROI en tant que binaire (assurez-vous que l’option permettant d’exporter toutes les images dans un seul fichier est sélectionnée).
REMARQUE: Les utilisateurs peuvent facilement convertir du format tiff au format mrc à l’aide du programme IMOD tif2mrc20. Ceci est utile pour le traçage filamentaire.
- Choisissez une classe dans le Multi-ROI, puis cliquez avec le bouton droit de la souris et extrayez la classe en tant que ROI. Dans le panneau des propriétés ci-dessus, sélectionnez le nouveau retour sur investissement, cliquez avec le bouton droit de la souris | Exportation | ROI en tant que binaire (assurez-vous que l’option permettant d’exporter toutes les images dans un seul fichier est sélectionnée).
8. Génération de coordonnées pour la moyenne des sous-tomogrammes à partir du retour sur investissement
- Extraire une classe.
- Cliquez avec le bouton droit sur Classe à utiliser pour la calcul de la moyenne | Classe d’extraction en tant que ROI. Cliquez avec le bouton droit sur le retour sur investissement de la classe | Composants connectés | Nouveau Multi-ROI (26 connectés).
- Générer des coordonnées.
- Cliquez avec le bouton droit sur le nouveau Multi-ROI | Générateur scalaire. Développez Mesures de base avec jeu de données | cochez Centre de masse pondéré X, Y et Z. Sélectionnez le jeu de données et calculez. Cliquez avec le bouton droit sur Multi-ROI | Exporter les valeurs scalaires. Cochez Sélectionner tous les emplacements scalaires, puis OK pour générer les coordonnées du monde centroïde pour chaque classe du multi-ROI en tant que fichier CSV.
REMARQUE: Si les particules sont proches les unes des autres et que les segmentations se touchent, il peut être nécessaire d’effectuer une transformation de bassin versant pour séparer les composants en un multi-ROI.
- Cliquez avec le bouton droit sur le nouveau Multi-ROI | Générateur scalaire. Développez Mesures de base avec jeu de données | cochez Centre de masse pondéré X, Y et Z. Sélectionnez le jeu de données et calculez. Cliquez avec le bouton droit sur Multi-ROI | Exporter les valeurs scalaires. Cochez Sélectionner tous les emplacements scalaires, puis OK pour générer les coordonnées du monde centroïde pour chaque classe du multi-ROI en tant que fichier CSV.
9. Transformation des bassins versants
- Extraire la classe en cliquant avec le bouton droit sur la classe dans Multi-ROI à utiliser pour la moyenne | Classe d’extraction en tant que ROI. Nommez ce ROI Watershed Mask.
- (Facultatif) Fermez les trous.
- Si les particules segmentées ont des trous ou des ouvertures, fermez-les pour le bassin versant. Cliquez sur le retour sur investissement dans Propriétés des données. Dans l’onglet Segmentation (à gauche), accédez à Opérations morphologiques et utilisez la combinaison de dilate, d’érosion et de fermeture nécessaire pour obtenir des segmentations solides sans trous.
- Inverser le retour sur investissement en cliquant sur le retour sur investissement | Copier l’objet sélectionné (sous Propriétés des données). Sélectionnez le retour sur investissement copié et, sur le côté gauche, dans l’onglet Segmentation , cliquez sur Inverser.
- Créer une carte de distance en cliquant avec le bouton droit sur le retour sur investissement inversé | Créer un mappage de | Carte de distance. Pour une utilisation ultérieure, faites une copie de la carte de distance et inversez-la (clic droit | Modifier et transformer | Inverser les valeurs | Postuler). Nommez cette carte inversée Paysage.
- Créez des points de départ.
- Cachez le retour sur investissement et affichez la carte des distances. Dans l’onglet Segmentation , cliquez sur Définir la plage et diminuez la plage jusqu’à ce que seuls quelques pixels au centre de chaque point soient mis en surbrillance et qu’aucun ne soit connecté à un autre point. Au bas de la section Plage , cliquez sur Ajouter à nouveau. Nommez ce nouveau ROI Seedpoints.
- Effectuer la transformation du bassin versant.
- Cliquez avec le bouton droit sur Seedpoints ROI | Composants connectés | Nouveau Multi-ROI (26 connectés). Cliquez avec le bouton droit sur le Multi-ROI nouvellement généré | Transformation du bassin versant. Sélectionnez la carte de distance nommée Paysage et cliquez sur OK ; sélectionnez le ROI nommé Watershed Mask et cliquez sur OK pour calculer une transformation de bassin versant à partir de chaque point de départ et séparer les particules individuelles en classes distinctes dans le multi-ROI. Générez des coordonnées comme à l’étape 8.2.
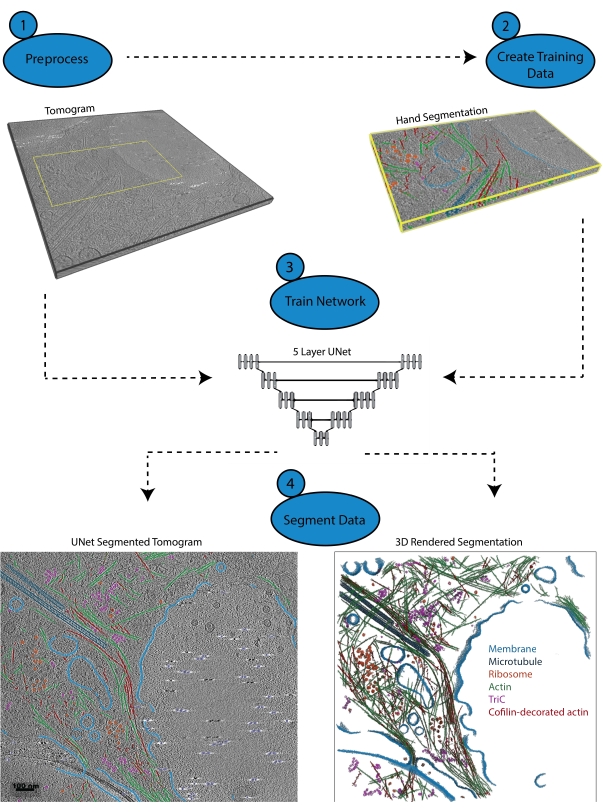
Figure 1 : Flux de travail. 1) Prétraiter le tomogramme d’entraînement en calibrant l’échelle d’intensité et en filtrant l’ensemble de données. 2) Créez les données d’entraînement en segmentant à la main une petite partie d’un tomogramme avec toutes les étiquettes appropriées que l’utilisateur souhaite identifier. 3) En utilisant le tomogramme filtré comme entrée et la segmentation de la main comme sortie d’apprentissage, un U-Net multi-couches à cinq couches est formé dans l’assistant de segmentation. 4) Le réseau entraîné peut être appliqué au tomogramme complet pour l’annoter et un rendu 3D peut être généré à partir de chaque classe segmentée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Conformément au protocole, un U-Net à cinq tranches a été formé sur un seul tomogramme (figure 2A) pour identifier cinq classes : membrane, microtubules, actine, marqueurs fiduciaires et arrière-plan. Le réseau a été entraîné de façon itérative trois fois au total, puis appliqué au tomogramme pour le segmenter et l’annoter complètement (Figure 2B,C). Un nettoyage minimal a été effectué à l’aide des étapes 7.1 et 7.2. Les trois tomogrammes suivants d’intérêt (Figure 2D,G,J) ont été chargés dans le logiciel pour le prétraitement. Avant l’importation de l’image, l’un des tomogrammes (Figure 2J) nécessitait un ajustement de la taille des pixels de 17,22 Å/px à 13,3 Å/px car il était collecté sur un microscope différent à un grossissement légèrement différent. Le programme IMOD squeezevol a été utilisé pour le redimensionnement avec la commande suivante :
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
Dans cette commande, -f fait référence au facteur par lequel modifier la taille des pixels (dans ce cas : 13,3/17,22). Après l’importation, les trois cibles d’inférence ont été prétraitées conformément aux étapes 3.2 et 3.3, puis le U-Net à cinq tranches a été appliqué. Un nettoyage minimal a de nouveau été effectué. Les segmentations finales sont présentées à la figure 2.
Les segmentations de microtubules de chaque tomogramme ont été exportées sous forme de fichiers TIF binaires (étape 7.4), converties en MRC (programme IMOD tif2mrc ), puis utilisées pour la corrélation des cylindres et le traçage des filaments. Les segmentations binaires des filaments permettent un traçage de filaments beaucoup plus robuste que le traçage sur tomogrammes. Les cartes de coordonnées du traçage des filaments (figure 3) seront utilisées pour une analyse plus approfondie, comme les mesures du plus proche voisin (garnissement des filaments) et la moyenne hélicoïdale des sous-tomogrammes le long des filaments simples pour déterminer l’orientation des microtubules.
Les réseaux infructueux ou insuffisamment formés sont faciles à déterminer. Un réseau défaillant sera incapable de segmenter des structures, alors qu’un réseau mal entraîné segmentera généralement correctement certaines structures et aura un nombre important de faux positifs et de faux négatifs. Ces réseaux peuvent être corrigés et formés de manière itérative pour améliorer leurs performances. L’assistant de segmentation calcule automatiquement le coefficient de similarité Dice d’un modèle (appelé score dans le SegWiz) après son entraînement. Cette statistique donne une estimation de la similitude entre les données d’entraînement et la segmentation U-Net. Dragonfly 2022.1 dispose également d’un outil intégré pour évaluer les performances d’un modèle accessible dans l’onglet Intelligence artificielle en haut de l’interface (voir la documentation d’utilisation).
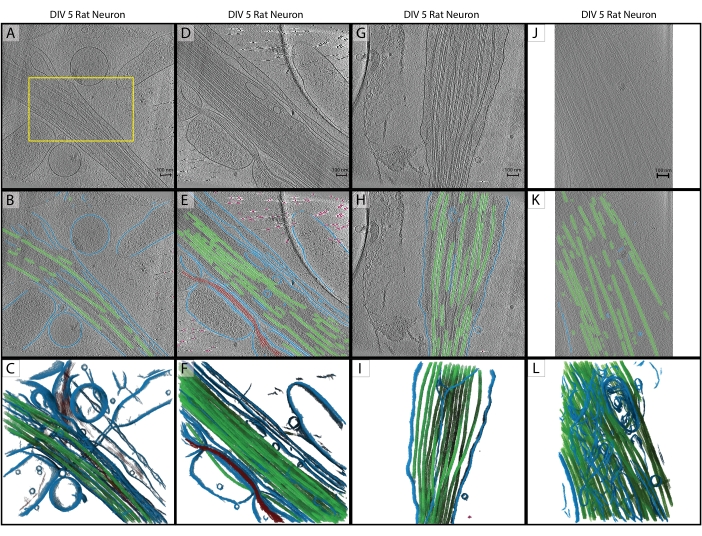
Figure 2 : Inférence. (A-C) Tomogramme d’entraînement original d’un neurone de rat hippocampique DIV 5, collecté en 2019 sur un Titan Krios. Il s’agit d’une reconstruction rétroprojetée avec correction CTF dans IMOD. (A) La case jaune représente la région où la segmentation des mains a été effectuée pour l’entrée de formation. (B) Segmentation 2D à partir du U-Net une fois la formation terminée. (C) Rendu 3D des régions segmentées montrant la membrane (bleu), les microtubules (vert) et l’actine (rouge). (D-F) Neurone de rat hippocampique DIV 5 de la même séance que le tomogramme d’entraînement. (E) Segmentation 2D à partir du U-Net sans formation supplémentaire et nettoyage rapide. Membrane (bleu), microtubules (vert), actine (rouge), fiduciaire (rose). (F) Rendu 3D des régions segmentées. (G-I) Neurone hippocampique de rat DIV 5 de la session 2019. (H) segmentation 2D à partir du U-Net avec nettoyage rapide et (I) rendu 3D. (J-L) Neurone de rat hippocampique DIV 5, collecté en 2021 sur un Titan Krios différent à un grossissement différent. La taille des pixels a été modifiée avec le programme IMOD squeezevol pour correspondre au tomogramme d’entraînement. (K) segmentation 2D à partir du U-Net avec nettoyage rapide, démontrant une inférence robuste à travers les ensembles de données avec un prétraitement approprié et (L) un rendu 3D de la segmentation. Barres d’échelle = 100 nm. Abréviations : DIV = jours in vitro; CTF = fonction de transfert de contraste. Veuillez cliquer ici pour voir une version agrandie de cette figure.
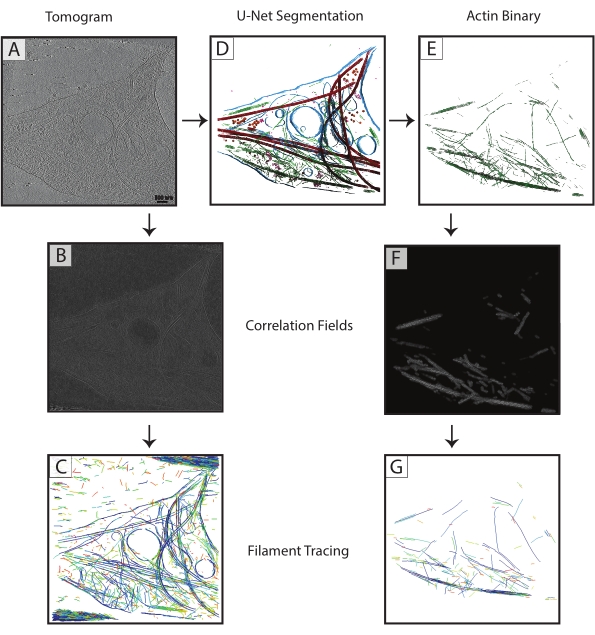
Figure 3 : Amélioration du traçage filamentaire. (A) Tomogramme d’un neurone hippocampique de rat DIV 4, recueilli sur un Titan Krios. (B) Carte de corrélation générée à partir de la corrélation du cylindre sur les filaments d’actine. (C) Traçage filamentaire de l’actine en utilisant les intensités des filaments d’actine dans la carte de corrélation pour définir les paramètres. Le traçage capture la membrane et les microtubules, ainsi que le bruit, tout en essayant de tracer uniquement l’actine. (D) Segmentation U-Net du tomogramme. Membrane surlignée en bleu, microtubules en rouge, ribosomes en orange, triC en violet et actine en vert. (E) Segmentation de l’actine extraite comme masque binaire pour le traçage filamentaire. (F) Carte de corrélation générée à partir de la corrélation cylindrique avec les mêmes paramètres de (B). (G) Amélioration significative du traçage filamentaire des filaments d’actine à partir du tomogramme. Abréviation : DIV = jours in vitro. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Fichier supplémentaire 1 : Le tomogramme utilisé dans ce protocole et le multi-ROI généré en tant qu’entrée d’apprentissage sont inclus dans un ensemble de données groupé (Training.ORSObject). Voir https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Discussion
Ce protocole définit une procédure d’utilisation du logiciel Dragonfly 2022.1 pour former un U-Net multiclasse à partir d’un seul tomogramme, et comment déduire ce réseau à d’autres tomogrammes qui n’ont pas besoin d’être du même ensemble de données. La formation est relativement rapide (peut être aussi rapide que 3-5 minutes par époque ou aussi lente que quelques heures, selon le réseau qui est formé et le matériel utilisé), et le recyclage d’un réseau pour améliorer son apprentissage est intuitif. Tant que les étapes de prétraitement sont effectuées pour chaque tomogramme, l’inférence est généralement robuste.
Un prétraitement cohérent est l’étape la plus critique pour l’inférence d’apprentissage profond. Il existe de nombreux filtres d’imagerie dans le logiciel et l’utilisateur peut expérimenter pour déterminer quels filtres fonctionnent le mieux pour des ensembles de données particuliers; Notez que quel que soit le filtrage utilisé sur le tomogramme d’entraînement, il doit être appliqué de la même manière aux tomogrammes d’inférence. Il faut également veiller à fournir au réseau des informations de formation exactes et suffisantes. Il est essentiel que toutes les fonctionnalités segmentées dans les tranches d’entraînement soient segmentées aussi soigneusement et précisément que possible.
La segmentation des images est facilitée par une interface utilisateur sophistiquée de qualité commerciale. Il fournit tous les outils nécessaires à la segmentation des mains et permet la réaffectation simple des voxels d’une classe à une autre avant la formation et le recyclage. L’utilisateur est autorisé à segmenter manuellement les voxels dans tout le contexte du tomogramme, et ils ont plusieurs vues et la possibilité de faire pivoter le volume librement. De plus, le logiciel offre la possibilité d’utiliser des réseaux multiclasses, qui ont tendance à mieux fonctionner16 et sont plus rapides que la segmentation avec plusieurs réseaux à classe unique.
Il y a, bien sûr, des limites aux capacités d’un réseau neuronal. Les données cryo-ET sont, par nature, très bruyantes et limitées dans l’échantillonnage angulaire, ce qui conduit à des distorsions spécifiques à l’orientation dans des objets identiques21. La formation repose sur un expert pour segmenter les structures avec précision, et un réseau performant est aussi bon (ou aussi mauvais) que les données de formation qui lui sont données. Le filtrage d’image pour amplifier le signal est utile pour le formateur, mais il existe encore de nombreux cas où il est difficile d’identifier avec précision tous les pixels d’une structure donnée. Il est donc important de faire très attention lors de la création de la segmentation de la formation afin que le réseau dispose des meilleures informations possibles pour apprendre pendant la formation.
Ce flux de travail peut être facilement modifié selon les préférences de chaque utilisateur. Bien qu’il soit essentiel que tous les tomogrammes soient prétraités exactement de la même manière, il n’est pas nécessaire d’utiliser les filtres exacts utilisés dans le protocole. Le logiciel dispose de nombreuses options de filtrage d’images, et il est recommandé de les optimiser pour les données particulières de l’utilisateur avant de se lancer dans un grand projet de segmentation couvrant de nombreux tomogrammes. Il existe également un certain nombre d’architectures réseau disponibles à utiliser: un U-Net multi-tranches s’est avéré fonctionner le mieux pour les données de ce laboratoire, mais un autre utilisateur pourrait trouver qu’une autre architecture (telle qu’un U-Net 3D ou un capteur 3D) fonctionne mieux. L’assistant de segmentation fournit une interface pratique pour comparer les performances de plusieurs réseaux à l’aide des mêmes données d’apprentissage.
Des outils comme ceux présentés ici feront de la segmentation manuelle des tomogrammes complets une tâche du passé. Avec des réseaux neuronaux bien entraînés et robustement inférables, il est tout à fait possible de créer un flux de travail où les données tomographiques sont reconstruites, traitées et entièrement segmentées aussi rapidement que le microscope peut les collecter.
Déclarations de divulgation
La licence en libre accès pour ce protocole a été payée par Object Research Systems.
Remerciements
Cette étude a été financée par le Penn State College of Medicine et le Département de biochimie et de biologie moléculaire, ainsi que par la subvention 4100079742-EXT du Tobacco Settlement Fund (TSF). Les services et instruments CryoEM et CryoET Core (RRID:SCR_021178) utilisés dans ce projet ont été financés, en partie, par le Pennsylvania State University College of Medicine par l’intermédiaire du Bureau du vice-doyen à la recherche et des étudiants diplômés et du ministère de la Santé de Pennsylvanie à l’aide de Tobacco Settlement Funds (CURE). Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les opinions officielles de l’Université ou du Collège de médecine. Le ministère de la Santé de Pennsylvanie décline spécifiquement toute responsabilité pour toute analyse, interprétation ou conclusion.
matériels
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Références
- Bai, X. -C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet. , Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022).
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Dragonfly 2021.3 (Computer Software). , Available from: http://www.theobjects.com/dragonfly (2021).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon